Доставка, конструирование и оптимизация систем, способов и композиций для манипуляции с последовательностями и применения в терапии - RU2721275C2
Код документа: RU2721275C2
Чертежи






















































































Описание
Родственные заявки и включение при помощи ссылки
Настоящая заявка заявляет преимущество и приоритет предварительных заявок на патенты США №№61/736527, поданной 12 декабря 2012 г.; 61/748427, поданной 2 января 2013 г.; 61/758468, поданной 30 января 2013 г., 61/769046, поданной 25 февраля 2013 г.; 61/791409 и 61/802174, поданных 15 марта 2013 г., 61/806375, поданной 28 марта 2013 г.; 61/814263, поданной 20 апреля 2013 г.; 61/819803, поданной 6 мая 2013 г.; 61/828130, поданной 28 мая 2013 г.; 61/835931 и 61/836123, поданной 17 июня 2013 г., а также 61/847537, поданной 17 июля 2013 г.
Также ссылаются на предварительные заявки на патенты США №№61/799800, поданную 15 марта 2013 г.; 61/835931, 61/835936, 61/836127, 61/836101, 61/836080 и 61/835973, поданные 17 июня 2013 г.; 61/862468 и 61/862355, поданные 5 августа 2013 г.; 61/871301, поданную 28 августа 2013 г.; 61/960777, поданную 25 сентября 2013 г., и 61/961980, поданную 28 октября 2013 г.
Вышеупомянутые заявки и все документы, упомянутые в них или во время делопроизводства по ним ("упомянутые в заявке документы"), и все документы, упомянутые или на которые ссылаются в упомянутых в заявке документах, и все документы, упомянутые или на которые ссылаются в данном документе ("упомянутые в данном документе документы"), и все документы, упомянутые или на которые ссылаются в упомянутых в данном документе документах, наравне с любыми инструкциями изготовителя, описаниями, характеристиками продуктов и описаниями продуктов для любых продуктов, упомянутых в данном документе или в любом документе, включенном при помощи ссылки в данный документ, таким образом, включены в данный документ при помощи ссылки и могут быть использованы при осуществлении на практике настоящего изобретения. Более конкретно, все документы, на которые ссылаются, включены при помощи ссылки в такой же мере, как если бы конкретно и отдельно было указано, что каждый отдельный документ включен при помощи ссылки.
Область техники
Настоящее изобретение в целом относится к доставке, конструированию, оптимизации и применениям в терапии систем, способов и композиций, используемых для контроля экспрессии генов, включающего целенаправленное воздействие на последовательность, такое как внесение изменений в геном или редактирование гена, связанное с короткими палиндромными повторами, регулярно расположенными группами (CRISPR), и их компонентами.
УТВЕРЖДЕНИЕ КАСАТЕЛЬНО ФИНАНСИРУЕМОГО ИЗ ФЕДЕРАЛЬНОГО БЮДЖЕТА ИССЛЕДОВАНИЯ
Настоящее изобретение было разработано при правительственной поддержке в рамках NIH Pioneer Award (1DP1MH100706), выданного Национальными институтами здравоохранения. Правительство обладает определенными правами на настоящее изобретение.
Предпосылки изобретения
Недавние достижения в технологиях секвенирования генома и способах анализа значительно ускорили возможность каталогизации и картирования генетических факторов, ассоциированных с широким разнообразием биологических функций и заболеваний. Технологии точного целенаправленного воздействия на геном необходимы для обеспечения системного обратного конструирования каузальных генетических изменений путем обеспечения возможности селективного внесения изменений в отдельные генетические элементы, а также для продвижения в области синтетической биологии и в области применений в биотехнологии и медицины. Несмотря на то, что технологии редактирования генома, такие как оригинальные ферменты с "цинковыми пальцами", подобные транскрипционным активаторам эффекторы (TALE) или хоминг-мегануклеазы, доступны для осуществления целенаправленного внесения изменений в геном, все еще существует необходимость в новых технологиях конструирования генома, которые являются доступными, простыми в осуществлении, масштабируемыми и характеризуются возможностью целенаправленного воздействия на несколько положений в эукариотическом геноме.
Краткое описание изобретения
Система CRISPR-Cas не требует создания индивидуализированных белков для целенаправленного воздействия на конкретные последовательности, а скорее один фермент Cas может быть запрограммирован короткой молекулой РНК для узнавания специфичной ДНК-мишени. Добавление системы CRISPR-Cas к спектру технологий секвенирования генома и способов анализа может значительно упростить методику и ускорить возможность каталогизации и картирования генетических факторов, ассоциированных с широким разнообразием биологических функций и заболеваний. Для того, чтобы использовать систему CRISPR-Cas эффективно для редактирования генома без вредных воздействий, важно понимать аспекты конструирования, оптимизации и специфичной относительно типа клетки/ткани/органа доставки этих средств для конструирования генома, которые являются аспектами заявленного изобретения.
Существует актуальная необходимость в альтернативных и функциональных системах и технологиях для целенаправленного воздействия на последовательности нуклеиновых кислот в широком спектре применений. Аспекты настоящего изобретения удовлетворяют эту необходимость и предусматривают связанные с этим преимущества. Иллюстративный комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, которая гибридизуется с целевой последовательностью в целевом полинуклеотиде. Направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизуется с tracr-последовательностью.
В одном аспекте настоящее изобретение предусматривает способы применения одного или нескольких элементов системы CRISPR-Cas. Комплекс CRISPR по настоящему изобретению обеспечивает эффективное средство модификации целевого полинуклеотида. Комплекс CRISPR по настоящему изобретению характеризуется большим разнообразием полезных свойств, включая модификацию (например, делению, вставку, транслокацию, инактивацию, активацию) целевого полинуклеотида во множестве типов клеток в различных тканях и органах. Комплекс CRISPR по настоящему изобретению как таковой имеет широкий спектр применений в, например, редактировании генов или генома, генной терапии, изыскании новых лекарственных средств, скрининге лекарственных средств, диагностике и прогнозировании заболеваний.
Аспекты настоящего изобретения относятся к ферментам Cas9, обладающим улучшенной специфичностью целенаправленного воздействия в системе CRISPR-Cas9, имеющей направляющие РНК, характеризующиеся оптимальной активностью, имеющим меньшую длину, чем ферменты Cas9 дикого типа, и к кодирующим их молекулам нуклеиновых кислот, и к химерным ферментам Cas9, а также к способам улучшения специфичности целенаправленного воздействия фермента Cas9, или разработки системы CRISPR-Cas9, включающим разработку или получение направляющих РНК, характеризующихся оптимальной активностью, и/или выбора или получения фермента Cas9, имеющего меньшие размер или длину, чем Cas9 дикого типа, при этом упаковка кодирующей его нуклеиновой кислоты в вектор доставки является более совершенной, поскольку в векторе доставки кодирующая его часть является меньшей, чем в случае Cas9 дикого типа, и/или, создания химерных ферментов Cas9.
Также предусматриваются применения последовательностей, векторов, ферментов или систем по настоящему изобретению в медицине. Также предусматриваются их применения в редактировании генов или генома.
В дополнительном аспекте настоящего изобретения фермент Cas9 может содержать одну или несколько мутаций и может применяться в качестве стандартного ДНК-связывающего белка, слитого или не слитого с функциональным доменом. Мутации могут быть мутациями, введенными искусственным образом, или мутациями приобретения или потери функции. Мутации могут включать без ограничения мутации в одном из каталитических доменов (D10 и Н840) среди каталитических доменов RuvC и HNH, соответственно. Были охарактеризованы дополнительные мутации. В одном аспекте настоящего изобретения домен активации транскрипции может представлять собой VP64. В других аспектах настоящего изобретения домен репрессии транскрипции может представлять собой KRAB или SID4X. Другие аспекты настоящего изобретения относятся к мутантному ферменту Cas9, слитому с доменами, которые включают без ограничения активатор транскрипции, репрессор транскрипции, рекомбиназу, транспозазу, фактор ремоделирования гистонов, деметилазу, ДНК-метилтрансферазу, криптохром, домен, индуцируемый/регулируемый светом, или домен, индуцируемый/регулируемый химическими веществами.
В дополнительном варианте осуществления настоящее изобретение предусматривает способы создания мутантной tracrRNA и последовательностей прямых повторов или мутантных химерных направляющих последовательностей, обеспечивающих повышение производительности этих РНК в клетках. Аспекты настоящего изобретения также предусматривают отбор указанных последовательностей.
Аспекты настоящего изобретения также предусматривают способы упрощения клонирования и доставки компонентов комплекса CRISPR. В предпочтительном варианте осуществления настоящего изобретения подходящий промотор, такой как промотор U6, амплифицируют с ДНК-олигонуклеотидом и добавляют к направляющей РНК. Полученным в результате продуктом ПЦР можно затем трансфицировать клетки для управления экспрессией направляющей РНК. Аспекты настоящего изобретения также относятся к направляющей РНК, транскрибированной in vitro или полученной от компании, проводящей синтез, и трансфицируемой напрямую.
В одном аспекте настоящее изобретение предусматривает способы улучшения активности путем применения более активной полимеразы. В предпочтительном варианте осуществления экспрессия направляющих РНК под контролем промотора Т7 управляется экспрессией полимеразы Т7 в клетке. В преимущественном варианте осуществления клетка является эукариотической клеткой. В преимущественном варианте осуществления эукариотическая клетка является клеткой человека. В более предпочтительном варианте осуществления клетка человека является индивидуальной клеткой.
В одном аспекте настоящее изобретение предусматривает способы снижения токсичности ферментов Cas. В определенных аспектах фермент Cas представляет собой любой Cas9, описанный в данном документе, например, любой встречающийся в природе бактериальный Cas9, а также любые химерные формы, мутантные формы, гомологи или ортологи. В предпочтительном варианте осуществления Cas9 доставляют в клетку в форме мРНК. Это обеспечивает транзиентную экспрессию фермента со снижением, таким образом, токсичности. В другом предпочтительном варианте осуществления настоящее изобретение также предусматривает способы экспрессии Cas9 под контролем индуцируемого промотора и конструкции, применяемые в них.
В другом аспекте настоящее изобретение предусматривает способы улучшения in vivo применений системы CRISPR-Cas. В предпочтительном варианте осуществления фермент Cas представляет собой Cas9 дикого типа или любой из модифицированных вариантов, описанных в данном документе, в том числе любой встречающийся в природе бактериальный Cas9, а также любые химерные формы, мутантные формы, гомологи или ортологи. Преимущественный аспект настоящего изобретения предусматривает отбор гомологов Cas9, которые легко упаковываются в вирусные векторы для доставки. Ортологи Cas9, как правило, имеют общую структуру, включающую 3-4 домена RuvC и домен HNH. Наиболее близкий к 5'-концу домен RuvC расщепляет некомплементарную нить, а домен HNH расщепляет комплементарную нить. Все обозначения приведены в отношении направляющей последовательности.
Каталитический остаток в 5'-домене RuvC идентифицируют посредством сравнения с целью поиска гомологии представляющего интерес Cas9 и других ортологов Cas9 (из локуса CRISPR типа II S. pyogenes, локуса 1 CRISPR S. thermophilus, локуса 3 CRISPR S. thermophilus и локуса CRISPR типа II Franciscilla novicida), и консервативный остаток Asp (D10) подвергают мутации по типу замены на аланин с превращением Cas9 в фермент, вносящий однонитевой разрыв в комплементарную нить. Аналогично, консервативные остатки His и Asn в доменах HNH подвергают мутации по типу замены на аланин с превращением Cas9 в фермент, вносящий однонитевой разрыв в некомплементарную нить. В некоторых вариантах осуществления можно осуществлять мутации из обеих групп для превращения Cas9 в неразрезающий фермент.
В некоторых вариантах осуществления фермент CRISPR представляет собой фермент CRISPR типа I или III, предпочтительно фермент CRISPR типа II. Этот фермент CRISPR типа II может быть любым ферментом Cas. Предпочтительный фермент Cas может быть идентифицирован как Cas9, поскольку он может относиться к общему классу ферментов, обладающих гомологией с самой большой нуклеазой с несколькими доменами нуклеаз системы CRISPR типа II. В наиболее предпочтительном случае фермент Cas9 получен или происходит из spCas9 или saCas9. Под происходящим заявители подразумевают, что в основе происходящего фермента главным образом лежит фермент дикого типа в том смысле, что он характеризуется высокой степенью гомологии последовательностей с этим ферментом, но был подвергнут мутации (модифицирован) некоторым образом, как описано в данном документе.
Следует иметь в виду, что выражения Cas и фермент CRISPR обычно применяются в данном документе взаимозаменяемо, если не очевидно иное. Как упоминается выше, многие из порядков нумерации остатков, применяемых в данном документе, относятся к ферменту Cas9 из локуса CRISPR типа II Streptococcus pyogenes. Однако следует иметь в виду, что настоящее изобретение включает многие другие Cas9 из других видов микроорганизмов, такие как SpCas9, SaCas9, St1Cas9 и т.д. Дополнительные примеры представлены в данном документе. Специалист в данной области будет способен определить надлежащие соответствующие остатки в ферментах Cas9, отличных от SpCas9, путем сравнения необходимых аминокислотных последовательностей. Таким образом, если конкретное аминокислотное замещение обозначается с помощью нумерации SpCas9, то, если из контекста не очевидно, что это не предназначено для применения в отношении других ферментов Cas9, подразумевается, что настоящее раскрытие охватывает соответствующие модификации в других ферментах Cas9.
Пример кодон-оптимизированной последовательности, в данном случае оптимизированной для человека (т.е. оптимизированной для экспрессии у человека), представлен в данном документе, см. кодон-оптимизированную последовательность SaCas9 для человека. Хотя это является предпочтительным, следует иметь в виду, что возможны другие примеры и что для вида-хозяина известна оптимизация кодонов.
В дополнительных вариантах осуществления настоящее изобретение предусматривает способы улучшения функционирования Cas9 посредством образования химерных белков Cas9. Химерные белки Cas9 - химерные Cas9 - могут быть новыми Cas9, содержащими фрагменты из более чем одного встречающегося в природе Cas9. Эти способы могут включать слияние N-концевых фрагментов одного гомолога Cas9 с С-концевыми фрагментами другого гомолога Cas9. Эти способы также обеспечивают отбор новых свойств, проявляемых химерными белками Cas9.
Следует иметь в виду, что в способах по настоящему изобретению, где организм представляет собой животное или растение, модификация может иметь место ex vivo или in vitro, например, в клеточной культуре, и в ряде случаев не in vivo. В других вариантах осуществления она может иметь место in vivo.
В одном аспекте настоящее изобретение предусматривает способ модификации организма или отличного от человека организма путем манипуляции с целевой последовательностью в представляющем интерес локусе генома, включающий доставку не встречающейся в природе или сконструированной композиции, содержащей:
А) - I. полинуклеотидную последовательность химерной РНК (chiRNA) системы CRISPR-Cas, где полинуклеотидная последовательность содержит:
(a) направляющую последовательность, способную гибридизоваться с целевой последовательностью в эукариотической клетке,
(b) парную tracr-последовательность и
(c) tracr-последовательность, и
П. полинуклеотидную последовательность, кодирующую фермент CRISPR, содержащий по меньшей мере одну или несколько последовательностей ядерной локализации,
где (а), (b) и (с) расположены в 5'-3' ориентации,
где при транскрипции парная tracr-последовательность гибридизуется с tracr-последовательностью, а направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью, и
где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизуется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизуется с tracr-последовательностью, а полинуклеотидная последовательность, кодирующая фермент CRISPR, представляет собой ДНК или РНК,
или
(В) I. полинуклеотиды, содержащие:
(a) направляющую последовательность, способную гибридизоваться с целевой последовательностью в эукариотической клетке, и
(b) по меньшей мере одну или несколько парных tracr-последовательностей,
II. полинуклеотидную последовательность, кодирующую фермент CRISPR, и
III. полинуклеотидную последовательность, содержащую tracr-последовательность,
где при транскрипции парная tracr-последовательность гибридизуется с tracr-последовательностью, а направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью, и
где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизуется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизуется с tracr-последовательностью, а полинуклеотидная последовательность, кодирующая фермент CRISPR, представляет собой ДНК или РНК.
Любая или все из полинуклеотидной последовательности, кодирующей фермент CRISPR, направляющей последовательности, парной tracr-последовательности или tracr-последовательности могут представлять собой РНК. Полинуклеотиды, кодирующие последовательность, кодирующую фермент CRISPR, направляющую последовательность, парную tracr-последовательность или tracr-последовательность, могут представлять собой РНК, и их могут доставлять посредством липосом, наночастиц, экзосом, микропузырьков или генной пушки.
Следует иметь в виду, что если ссылаются на полинуклеотид, который представляет собой РНК и, как говорят, 'содержит' признак, такой как парная tracr-последовательность, то последовательность РНК содержит данный признак. Если полинуклеотид представляет собой ДНК и, как говорят, содержит признак, такой как парная tracr-последовательность, то последовательность ДНК транскрибируется или может быть транскрибирована в РНК, содержащую признак, о котором идет речь. Если признак представляет собой белок, такой как фермент CRISPR, то упоминаемая последовательность ДНК или РНК транслируется или может быть транслирована (а в случае ДНК сначала транскрибируется).
Соответственно, в определенных вариантах осуществления настоящее изобретение предусматривает способ модификации организма, например, млекопитающего, в том числе человека, или отличного от человека млекопитающего или организма путем манипуляции с целевой последовательностью в представляющем интерес локусе генома, включающий доставку не встречающейся в природе или сконструированной композиции, содержащей вирусную или плазмидную векторную систему, содержащую один или несколько вирусных или плазмидных векторов, функционально кодирующих композицию для ее экспрессии, где композиция содержит: (А) не встречающуюся в природе или сконструированную композицию, содержащую векторную систему, содержащую один или несколько векторов, содержащих I. первый регуляторный элемент, функционально связанный с полинуклеотидной последовательностью химерной РНК (chiRNA) системы CRISPR-Cas, где полинуклеотидная последовательность содержит (а) направляющую последовательность, способную гибридизоваться с целевой последовательностью в эукариотической клетке, (b) парную tracr-последовательность и (с) tracr-последовательность, и II. второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей фермент CRISPR, содержащий по меньшей мере одну или несколько последовательностей ядерной локализации (или необязательно по меньшей мере одну или несколько последовательностей ядерной локализации, поскольку некоторые варианты осуществления могут не включать NLS), где (а), (b) и (с) расположены в 5'-3' ориентации, где компоненты I и II находятся в одном и том же или разных векторах системы, где при транскрипции парная tracr-последовательность гибридизуется с tracr-последовательностью, а направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью, и где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизуется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизуется с tracr-последовательностью, или (В) не встречающуюся в природе или сконструированную композицию, содержащую векторную систему, содержащую один или несколько векторов, содержащих I. первый регуляторный элемент, функционально связанный с (а) направляющей последовательностью, способной гибридизоваться с целевой последовательностью в эукариотической клетке, и (b) по меньшей мере одной или несколькими парными tracr-последовательностями, II. второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей фермент CRISPR, и III. третий регуляторный элемент, функционально связанный с tracr-последовательностью, где компоненты I, II и III находятся в одном и том же или разных векторах системы, где при транскрипции парная tracr-последовательность гибридизуется с tracr-последовательностью, а направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью, и где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизуется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизуется с tracr-последовательностью. В некоторых вариантах осуществления компоненты I, II и III находятся в одном и том же векторе. В других вариантах осуществления компоненты I и II находятся в одном и том же векторе, тогда как компонент III находится в другом векторе. В других вариантах осуществления компоненты I и III находятся в одном и том же векторе, тогда как компонент II находится в другом векторе. В других вариантах осуществления компоненты II и III находятся в одном и том же векторе, тогда как компонент I находится в другом векторе. В других вариантах осуществления каждый из компонентов I, II и III находится в отдельных векторах. Настоящее изобретение также предусматривает вирусную или плазмидную векторную систему, описанную в данном документе.
Вектор предпочтительно представляет собой вирусный вектор, как, например, векторы на основе лентивируса, или бакуловируса, или, предпочтительно, аденовируса/аденоассоциированного вируса, но известны и предусмотрены другие средства доставки (такие как дрожжевые системы, микропузырьки, генные пушки/средства прикрепления векторов к наночастицам золота). В некоторых вариантах осуществления один или несколько вирусных или плазмидных векторов можно доставлять посредством липосом, наночастиц, экзосом, микропузырьков или генной пушки.
Под манипуляцией с целевой последовательностью заявители также подразумевают эпигенетическую манипуляцию с целевой последовательностью. Она может осуществляться в отношении состояния хроматина целевой последовательности, как, например, путем модификации состояния метилирования целевой последовательности (т.е. добавление или устранение метилирования, или паттернов метилирования, или CpG-островков), модификации гистонов, повышения или снижения доступности целевой последовательности, или путем активации укладки в 3D-структуру.
Следует иметь в виду, что если ссылаются на способ модификации организма или млекопитающего, в том числе человека, или отличного от человека млекопитающего или организма путем манипуляции с целевой последовательностью в представляющем интерес локусе генома, тогда его можно использовать в отношении организма (или млекопитающего) в целом или всего лишь одной клетки или популяции клеток из этого организма (если организм является многоклеточным). В случае человека, например, заявители предусматривают, inter alia, одну клетку или популяцию клеток, и их можно предпочтительно модифицировать ex vivo и затем вводить обратно. В этом случае может быть необходим биоптат или другой образец ткани или биологической жидкости, Стволовые клетки также являются особенно предпочтительными в этом отношении. Но, разумеется, также предусматриваются варианты осуществления in vivo.
В определенных вариантах осуществления настоящее изобретение предусматривает способ лечения или подавления состояния, вызванного дефектом в целевой последовательности в представляющем интерес локусе генома у субъекта (например, млекопитающего или человека) или отличного от человека субъекта (например, млекопитающего), нуждающегося в этом, включающий модификацию субъекта или отличного от человека субъекта путем манипуляции с целевой последовательностью, и где состояние является чувствительным к лечению или подавлению путем манипуляции с целевой последовательностью, включающий обеспечение лечения, предусматривающего: доставку не встречающейся в природе или сконструированной композиции, содержащей векторную систему на основе AAV или лентивируса, содержащую один или несколько векторов на основе AAV или лентивируса, функционально кодирующих композицию для их экспрессии, где манипуляцию с целевой последовательностью осуществляют с помощью композиции при ее экспрессии, где композиция содержит: (А) не встречающуюся в природе или сконструированную композицию, содержащую векторную систему, содержащую один или несколько векторов, содержащих I. первый регуляторный элемент, функционально связанный с полинуклеотидной последовательностью химерной РНК (chiRNA) системы CRISPR-Cas, где полинуклеотидная последовательность содержит (а) направляющую последовательность, способную гибридизоваться с целевой последовательностью в эукариотической клетке, (b) парную tracr-последовательность и (с) tracr-последовательность, и II. второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей фермент CRISPR, содержащий по меньшей мере одну или несколько последовательностей ядерной локализации (или необязательно по меньшей мере одну или несколько последовательностей ядерной локализации, поскольку некоторые варианты осуществления могут не включать NLS), где (а), (b) и (с) расположены в 5'-3' ориентации, где компоненты I и II находятся в одном и том же или разных векторах системы, где при транскрипции парная tracr-последовательность гибридизуется с tracr-последовательностью, а направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью, и где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизуется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизуется с tracr-последовательностью, или (В) не встречающуюся в природе или сконструированную композицию, содержащую векторную систему, содержащую один или несколько векторов, содержащих I. первый регуляторный элемент, функционально связанный с (а) направляющей последовательностью, способной гибридизоваться с целевой последовательностью в эукариотической клетке, и (b) по меньшей мере одной или несколькими парными tracr-последовательностями, II. второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей фермент CRISPR, и III. третий регуляторный элемент, функционально связанный с tracr-последовательностью, где компоненты I, II и III находятся в одном и том же или разных векторах системы, где при транскрипции парная tracr-последовательность гибридизуется с tracr-последовательностью, а направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью, и где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизуется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизуется с tracr-последовательностью. В некоторых вариантах осуществления компоненты I, II и III находятся в одном и том же векторе. В других вариантах осуществления компоненты I и II находятся в одном и том же векторе, тогда как компонент III находится в другом векторе. В других вариантах осуществления компоненты I и III находятся в одном и том же векторе, тогда как компонент II находится в другом векторе. В других вариантах осуществления компоненты II и III находятся в одном и том же векторе, тогда как компонент I находится в другом векторе. В других вариантах осуществления каждый из компонентов I, II и III находится в отдельных векторах. Настоящее изобретение также предусматривает вирусную (например, на основе AAV или лентивируса) векторную систему, описанную в данном документе, и она может быть частью векторной системы, описанной в данном документе.
Некоторые способы по настоящему изобретению могут включать индукцию экспрессии. В некоторых способах по настоящему изобретению организм или субъект является эукариотом (в том числе млекопитающим, в том числе человеком), или отличным от человека эукариотом, или отличным от человека животным, или отличным от человека млекопитающим. В некоторых вариантах осуществления организм или субъект является отличным от человека животным и может быть членистоногим, например, насекомым, или может быть нематодой. В некоторых способах по настоящему изобретению организм или субъект является растением. В некоторых способах по настоящему изобретению организм или субъект является млекопитающим или отличным от человека млекопитающим. Отличное от человека млекопитающее может быть, например, грызуном (предпочтительно мышью или крысой), копытным или приматом. В некоторых способах по настоящему изобретению организм или субъект является водорослью, в том числе микроводорослью, или является грибом. В некоторых способах по настоящему изобретению вирусный вектор представляет собой AAV или лентивирус и может быть частью векторной системы, описанной в данном документе. В некоторых способах по настоящему изобретению фермент CRISPR представляет собой Cas9. В некоторых способах по настоящему изобретению экспрессия направляющей последовательности находится под контролем промотора Т7 и управляется экспрессией полимеразы Т7.
Настоящее изобретение в некоторых вариантах осуществления охватывает способ доставки фермента CRISPR, включающий доставку в клетку мРНК, кодирующей фермент CRISPR. В некоторых из данных способов фермент CRISPR представляет собой Cas9.
Настоящее изобретение также предусматривает способы получения векторных систем по настоящему изобретению, в частности, вирусных векторных систем, описанных в данном документе. Настоящее изобретение в некоторых вариантах осуществления охватывает способ получения AAV по настоящему изобретению, включающий трансфекцию плазмиды(плазмид), содержащих молекулу(молекулы) нуклеиновой кислоты, кодирующую AAV, или состоящих, по сути, из них, в клетки, инфицированные AAV, и обеспечение rep и/или cap AAV, обязательных для репликации и упаковки AAV. В некоторых вариантах осуществления rep и/или cap AAV, обязательные для репликации и упаковки AAV, обеспечивают путем трансфекции клеток плазмидой-помощником(плазмидами-помощниками) или вирусом-помощником(вирусами-помощниками). В некоторых вариантах осуществления вирусом-помощником является поксвирус, аденовирус, герпесвирус или бакуловирус. В некоторых вариантах осуществления поксвирус представляет собой вирус коровьей оспы. В некоторых вариантах осуществления клетки являются клетками млекопитающих. А в некоторых вариантах осуществления клетки являются клетками насекомых, а вирус-помощник представляет собой бакуловирус. В других вариантах осуществления вирус представляет собой лентивирус.
У растений патогены часто являются специфичными по отношению к хозяину. Например, Fusarium oxysporum t. sp. lycopersici вызывает фузариозный вилт томата, но поражает только томат, a F. oxysporum f. dianthii и Puccinia graminis f. sp. tritici поражают только пшеницу. Растения обладают присущими и индуцированными защитными реакциями, обеспечивающие устойчивость к большинству патогенов. Мутации и события рекомбинации в поколениях растений приводят к генетической изменчивости, которая обуславливает восприимчивость, тем более, что патогены размножаются с большей частотой, чем растения. У растений может наблюдаться устойчивость вида растения, не являющегося хозяином, например, хозяин и патоген являются несовместимыми. Также может наблюдаться горизонтальная устойчивость, например, частичная устойчивость ко всем расам патогена, обычно контролируемая многими генами, и вертикальная устойчивость, например, полная устойчивость к некоторым расам патогена, но не к другим расам, обычно контролируемая несколькими генами. На уровне взаимодействия генов растения и патогены эволюционируют совместно, а генетические изменения одного уравновешивают изменения другого. Соответственно, используя естественную изменчивость, селекционеры комбинируют наиболее применимые гены для урожайности, качества, однородности, выносливости, устойчивости. Источники генов устойчивости включают нативные или чужеродные сорта, старинные сорта, родственные дикорастущие растения и индуцированные мутации, например, при обработке растительного материала мутагенными средствами. Применяя настоящее изобретение, селекционеры растений получают новый инструмент для индукции мутаций. Соответственно, специалист в данной области может проанализировать геном источников генов устойчивости, а в отношении сортов, имеющих желаемые характеристики или признаки, использовать настоящее изобретение для индукции появления генов устойчивости с большей точностью, чем в случае применявшихся ранее мутагенных средств, и, следовательно, ускорять и улучшать программы селекции растений.
Настоящее изобретение дополнительно охватывает композицию по настоящему изобретению или ее фермент CRISPR (в том числе, или альтернативно, мРНК, кодирующую фермент CRISPR) для применения в медицине или в терапии. В некоторых вариантах осуществления настоящее изобретение охватывает композицию согласно настоящему изобретению или ее фермент CRISPR (в том числе, или альтернативно, мРНК, кодирующую фермент CRISPR) для применения в способе согласно настоящему изобретению. В некоторых вариантах осуществления настоящее изобретение предусматривает применение композиции по настоящему изобретению или ее фермента CRISPR (в том числе, или альтернативно, мРНК, кодирующей фермент CRISPR) в редактировании генов или генома ex vivo. В определенных вариантах осуществления настоящее изобретение охватывает применение композиции по настоящему изобретению или ее фермента CRISPR (в том числе, или альтернативно, мРНК, кодирующей фермент CRISPR) в производстве лекарственного препарата для редактирования генов или генома ех vivo или для применения в способе согласно настоящему изобретению. Настоящее изобретение в некоторых вариантах осуществления охватывает композицию по настоящему изобретению или ее фермент CRISPR (в том числе, или альтернативно, мРНК, кодирующую фермент CRISPR), где целевая последовательность фланкируется на своем 3'-конце РАМ-последовательностью (мотивом, прилегающим к протоспейсеру), содержащей 5'-мотив, особенно если Cas9 получен из (или происходит из) Cas9 S. pyogenes или S. aureus. Например, подходящий РАМ представляет собой 5'-NRG или 5'-NNGRR (где N представляет собой любой нуклеотид) для ферментов SpCas9 или SaCas9 (или происходящих из них ферментов), соответственно, как отмечено ниже.
Следует иметь в виду, что SpCas9 или SaCas9 получены или происходят из Cas9 S. pyogenes или S. aureus.
Аспекты настоящего изобретения охватывают улучшение специфичности фермента CRISPR, например, Cas9, опосредующей целенаправленное воздействие на гены, и снижение вероятности нецелевой модификации ферментом CRISPR, например, Cas9. Настоящее изобретение в некоторых вариантах осуществления охватывает способ модификации организма или отличного от человека организма посредством сведения к минимуму нецелевых модификаций путем манипуляции с первой и второй целевыми последовательностями на противоположных нитях ДНК-дуплекса в представляющем интерес локусе генома в клетке, включающий доставку не встречающейся в природе или сконструированной композиции, содержащей:
I. первую полинуклеотидную последовательность химерной РНК (chiRNA) - системы CRISPR-Cas, где первая полинуклеотидная последовательность содержит:
(a) первую направляющую последовательность, способную гибридизоваться с первой целевой последовательностью,
(b) первую парную tracr-последовательность и
(c) первую tracr-последовательность,
II. вторую полинуклеотидную последовательность chiRNA системы CRISPR-Cas, где вторая полинуклеотидная последовательность содержит:
(a) вторую направляющую последовательность, способную гибридизоваться со второй целевой последовательностью,
(b) вторую парную tracr-последовательность и
(c) вторую tracr-последовательность, и
III. полинуклеотидную последовательность, кодирующую фермент CRISPR, содержащий по меньшей мере одну или несколько последовательностей ядерной локализации и содержащий одну или несколько мутаций, где (а), (b) и (с) расположены в 5'-3' ориентации, где при транскрипции первая и вторая парные tracr-последовательности гибридизуются с первой и второй tracr-последовательностями, соответственно, а первая и вторая направляющие последовательности управляют специфичным к последовательности связыванием первого и второго комплексов CRISPR с первой и второй целевыми последовательностями, соответственно, где первый комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) первой направляющей последовательностью, которая гибридизуется с первой целевой последовательностью, и (2) первой парной tracr-последовательностью, которая гибридизуется с первой tracr-последовательностью, где второй комплекс CRISPR содержит фермент CRISPR, образующий комплекс со (1) второй направляющей последовательностью, которая гибридизуется со второй целевой последовательностью, и (2) второй парной tracr-последовательностью, которая гибридизуется со второй tracr-последовательностью, где полинуклеотидная последовательность, кодирующая фермент CRISPR, представляет собой ДНК или РНК, и где первая направляющая последовательность управляет расщеплением одной нити ДНК-дуплекса возле первой целевой последовательности, а вторая направляющая последовательность управляет расщеплением другой нити возле второй целевой последовательности, индуцируя двухнитевой разрыв, с модификацией, таким образом, организма или отличного от человека организма посредством сведения к минимуму нецелевых модификаций.
В некоторых способах по настоящему изобретению любая или все из полинуклеотидной последовательности, кодирующей фермент CRISPR, первой и второй направляющих последовательностей, первой и второй парных tracr-последовательностей или первой и второй tracr-последовательностей представляет собой/представляют собой РНК. В дополнительных вариантах осуществления настоящего изобретения полинуклеотиды, кодирующие последовательность, кодирующую фермент CRISPR, первую и вторую направляющие последовательности, первую и вторую парные tracr-последовательности или первую и вторую tracr-последовательности, представляют собой/представляют собой РНК, и их доставляют посредством липосом, наночастиц, экзосом, микропузырьков или генной пушки. В определенных вариантах осуществления настоящего изобретения первая и вторая парные tracr-последовательности обладают 100% идентичностью, и/или первая и вторая tracr-последовательности обладают 100% идентичностью. В некоторых вариантах осуществления полинуклеотиды могут содержаться в векторной системе, содержащей один или несколько векторов. В предпочтительных вариантах осуществления настоящего изобретения фермент CRISPR представляет собой фермент Cas9, например, SpCas9. В аспекте настоящего изобретения фермент CRISPR содержит одну или несколько мутаций в каталитическом домене, где одна или несколько мутаций выбраны из группы, состоящей из D10A, Е762А, Н840А, N854A, N863A и D986A. В особенно предпочтительном варианте осуществления фермент CRISPR имеет мутацию D10A. В предпочтительных вариантах осуществления первый фермент CRISPR имеет одну или несколько мутаций, вследствие которых фермент является ферментом, вносящим однонитевой разрыв в комплементарную нить, а второй фермент CRISPR имеет одну или несколько мутаций, вследствие которых фермент является ферментом, вносящим однонитевой разрыв в некомплементарную нить. Альтернативно, первый фермент может являться ферментом, вносящим однонитевой разрыв в некомплементарную нить, а второй фермент может являться ферментом, вносящим однонитевой разрыв в комплементарную нить.
В предпочтительных способах по настоящему изобретению первая направляющая последовательность, управляющая расщеплением одной нити ДНК-дуплекса возле первой целевой последовательности, и вторая направляющая последовательность, управляющая расщеплением другой нити возле второй целевой последовательности, обуславливают возникновение "липкого" 5'-конца. В вариантах осуществления настоящего изобретения "липкий" 5'-конец содержит не более 200 пар оснований, предпочтительно не более 100 пар оснований или более предпочтительно не более 50 пар оснований. В вариантах осуществления настоящего изобретения "липкий" 5'-конец содержит по меньшей мере 26 пар оснований, предпочтительно по меньшей мере 30 пар оснований или более предпочтительно 34-50 пар оснований.
Настоящее изобретение в некоторых вариантах осуществления охватывает способ модификации организма или отличного от человека организма посредством сведения к минимуму нецелевых модификаций путем манипуляции с первой и второй целевыми последовательностями на противоположных нитях ДНК-дуплекса в представляющем интерес локусе генома в клетке, включающий доставку не встречающейся в природе или сконструированной композиции, содержащей векторную систему, содержащую один или несколько векторов, содержащих:
I. первый регуляторный элемент, функционально связанный с
(a) первой направляющей последовательностью, способной гибридизоваться с первой целевой последовательностью, и
(b) по меньшей мере одной или несколькими парными tracr-последовательностями,
II. второй регуляторный элемент, функционально связанный со
(a) второй направляющей последовательностью, способной гибридизоваться со второй целевой последовательностью, и
(b) по меньшей мере одной или несколькими парными tracr-последовательностями,
III. третий регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей фермент CRISPR, и
IV. четвертый регуляторный элемент, функционально связанный с tracr-последовательностью,
где компоненты I, II, III и IV находятся в одном и том же или разных векторах системы, где при транскрипции парная tracr-последовательность гибридизуется с tracr-последовательностью, а первая и вторая направляющие последовательности управляют специфичным к последовательности связыванием первого и второго комплексов CRISPR с первой и второй целевыми последовательностями, соответственно, где первый комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) первой направляющей последовательностью, которая гибридизуется с первой целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизуется с tracr-последовательностью, где второй комплекс CRISPR содержит фермент CRISPR, образующий комплекс со (1) второй направляющей последовательностью, которая гибридизуется со второй целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизуется с tracr-последовательностью, где полинуклеотидная последовательность, кодирующая фермент CRISPR, представляет собой ДНК или РНК, и где первая направляющая последовательность управляет расщеплением одной нити ДНК-дуплекса возле первой целевой последовательности, а вторая направляющая последовательность управляет расщеплением другой нити возле второй целевой последовательности, индуцируя двухнитевой разрыв, с модификацией, таким образом, организма или отличного от человека организма посредством сведения к минимуму нецелевых модификаций.
Настоящее изобретение также предусматривает векторную систему, описанную в данном документе. Система может содержать один, два, три или четыре различных вектора. Компоненты I, II, III и IV могут, таким образом, находиться в одном, двух, трех или четырех различных векторах, и в данном документе предусмотрены все комбинации возможных местоположений компонентов, например: компоненты I, II, III и IV могут находиться в одном и том же векторе; каждый из компонентов I, II, III и IV может находиться в отдельном векторе; компоненты I, II, III и IV могут находиться в общей сложности в двух или трех различных векторах, при этом предусмотрены все комбинации местоположений, и т.п.
В некоторых способах по настоящему изобретению любая или все из полинуклеотидной последовательности, кодирующей фермент CRISPR, первой и второй направляющих последовательностей, первой и второй парных tracr-последовательностей или первой и второй tracr-последовательностей представляет собой/представляют собой РНК. В дополнительных вариантах осуществления настоящего изобретения первая и вторая парные tracr-последовательности обладают 100% идентичностью, и/или первая и вторая tracr-последовательности обладают 100% идентичностью. В предпочтительных вариантах осуществления настоящего изобретения фермент CRISPR представляет собой фермент Cas9, например, SpCas9. В аспекте настоящего изобретения фермент CRISPR содержит одну или несколько мутаций в каталитическом домене, где одна или несколько мутаций выбраны из группы, состоящей из D10A, Е762А, Н840А, N854A, N863A и D986A. В особенно предпочтительном варианте осуществления фермент CRISPR имеет мутацию D10A. В предпочтительных вариантах осуществления первый фермент CRISPR имеет одну или несколько мутаций, вследствие которых фермент является ферментом, вносящим однонитевой разрыв в комплементарную нить, а второй фермент CRISPR имеет одну или несколько мутаций, вследствие которых фермент является ферментом, вносящим однонитевой разрыв в некомплементарную нить. Альтернативно, первый фермент может являться ферментом, вносящим однонитевой разрыв в некомплементарную нить, а второй фермент может являться ферментом, вносящим однонитевой разрыв в комплементарную нить. В дополнительном варианте осуществления настоящего изобретения один или несколько вирусных векторов доставляют посредством липосом, наночастиц, экзосом, микропузырьков или генной пушки.
В предпочтительных способах по настоящему изобретению первая направляющая последовательность, управляющая расщеплением одной нити ДНК-дуплекса возле первой целевой последовательности, и вторая направляющая последовательность, управляющая расщеплением другой нити возле второй целевой последовательности, обуславливают возникновение "липкого" 5'-конца. В вариантах осуществления настоящего изобретения "липкий" 5'-конец содержит не более 200 пар оснований, предпочтительно не более 100 пар оснований или более предпочтительно не более 50 пар оснований. В вариантах осуществления настоящего изобретения "липкий" 5'-конец содержит по меньшей мере 26 пар оснований, предпочтительно по меньшей мере 30 пар оснований или более предпочтительно 34-50 пар оснований.
Настоящее изобретение в некоторых вариантах осуществления охватывает способ модификации представляющего интерес локуса генома посредством минимизации нецелевых модификаций путем введения в клетку, содержащую и экспрессирующую двухнитевую молекулу ДНК, кодирующую представляющий интерес продукт гена, сконструированной, не встречающейся в природе системы CRISPR-Cas, содержащей белок Cas, имеющий одну или несколько мутаций, и две направляющие РНК, которые нацеливают на первую нить и вторую нить молекулы ДНК, соответственно, при этом направляющие РНК целенаправленно воздействуют на молекулу ДНК, кодирующую продукт гена, а белок Cas вносит однонитевой разрыв в каждую из первой нити и второй нити молекулы ДНК, кодирующей продукт гена, в результате чего экспрессия продукта гена изменяется; и где белок Cas и две направляющие РНК не встречаются вместе в естественных условиях.
В предпочтительных способах по настоящему изобретению белок Cas вносит однонитевой разрыв в каждую из первой нити и второй нити молекулы ДНК, кодирующей продукт гена, что обуславливает возникновение "липкого" 5'-конца. В вариантах осуществления настоящего изобретения "липкий" 5'-конец содержит не более 200 пар оснований, предпочтительно не более 100 пар оснований или более предпочтительно не более 50 пар оснований. В вариантах осуществления настоящего изобретения "липкий" 5'-конец содержит по меньшей мере 26 пар оснований, предпочтительно по меньшей мере 30 пар оснований или более предпочтительно 34-50 пар оснований.
Варианты осуществления настоящего изобретения также охватывают направляющие РНК, содержащие направляющую последовательность, слитую с парной tracr-последовательностью и tracr-последовательностью. В аспекте настоящего изобретения белок Cas является кодон-оптимизированным для экспрессии в эукариотической клетке, предпочтительно в клетке млекопитающего или клетке человека. В дополнительных вариантах осуществления настоящего изобретения белок Cas представляет собой белок системы CRISPR-Cas типа II, например, белок Cas9. В особенно предпочтительном варианте осуществления белок Cas представляет собой белок Cas9, например, SpCas9. В аспектах настоящего изобретения белок Cas имеет одну или несколько мутаций, выбранных из группы, состоящей из D10A, Е762А, Н840А, N854A, N863A и D986A. В особенно предпочтительном варианте осуществления белок Cas имеет мутацию D10A.
Аспекты настоящего изобретения относятся к снижению экспрессии продукта гена, или к дополнительному введению матричного полинуклеотида в молекулу ДНК, кодирующую продукт гена, или к точному вырезанию вставочной последовательности путем обеспечения повторного отжига и лигирования двух "липких" 5'-концов, или к изменению активности или функции продукта гена, или к повышению экспрессии продукта гена. В варианте осуществления настоящего изобретения продукт гена представляет собой белок.
Настоящее изобретение также охватывает сконструированную, не встречающуюся в природе систему CRISPR-Cas, содержащую белок Cas с одной или несколькими мутациями, и две направляющие РНК, которые осуществляют нацеливание на первую нить и вторую нить, соответственно, двухнитевой молекулы ДНК, кодирующей продукт гена в клетке, при этом направляющие РНК осуществляют нацеливание на молекулу ДНК, кодирующую продукт гена, а белок Cas вносит однонитевой разрыв в каждую из первой нити и второй нити молекулы ДНК, кодирующей продукт гена, в результате чего экспрессия продукта гена изменяется; и где белок Cas и две направляющие РНК не встречаются вместе в естественных условиях.
В аспектах настоящего изобретения направляющие РНК могут содержать направляющую последовательность, слитую с парной tracr-последовательностью и tracr-последовательностью. В варианте осуществления настоящего изобретения белок Cas представляет собой белок системы CRISPR-Cas типа II. В аспекте настоящего изобретения белок Cas является кодон-оптимизированным для экспрессии в эукариотической клетке, предпочтительно в клетке млекопитающего или клетке человека. В дополнительных вариантах осуществления настоящего изобретения белок Cas представляет собой белок системы CRISPR-Cas типа II, например, белок Cas9. В особенно предпочтительном варианте осуществления белок Cas представляет собой белок Cas9, например, SpCas9. В аспектах настоящего изобретения белок Cas имеет одну или несколько мутаций, выбранных из группы, состоящей из D10A, Е762А, Н840А, N854A, N863A и D986A. В особенно предпочтительном варианте осуществления белок Cas имеет мутацию D10A.
Аспекты настоящего изобретения относятся к снижению экспрессии продукта гена, или к дополнительному введению матричного полинуклеотида в молекулу ДНК, кодирующую продукт гена, или к точному вырезанию вставочной последовательности путем обеспечения повторного отжига и лигирования двух "липких" 5'-концов, или к изменению активности или функции продукта гена, или к повышению экспрессии продукта гена. В варианте осуществления настоящего изобретения продукт гена представляет собой белок.
Настоящее изобретение также охватывает сконструированную, не встречающуюся в природе векторную систему, содержащую один или несколько векторов, содержащих:
a) первый регуляторный элемент, функционально связанный с каждой из двух направляющих РНК системы CRISPR-Cas, которые осуществляют нацеливание на первую нить и вторую нить, соответственно, двухнитевой молекулы ДНК, кодирующей продукт гена,
b) второй регуляторный элемент, функционально связанный с белком Cas,
где компоненты (а) и (b) находятся в одном и том же или разных векторах системы, при этом направляющие РНК осуществляют нацеливание на молекулу ДНК, кодирующую продукт гена, а белок Cas вносит однонитевой разрыв в каждую из первой нити и второй нити молекулы ДНК, кодирующей продукт гена, в результате чего экспрессия продукта гена изменяется; и где белок Cas и две направляющие РНК не встречаются вместе в естественных условиях.
В аспектах настоящего изобретения направляющие РНК могут содержать направляющую последовательность, слитую с парной tracr-последовательностью и tracr-последовательностью. В варианте осуществления настоящего изобретения белок Cas представляет собой белок системы CRISPR-Cas типа II. В аспекте настоящего изобретения белок Cas является кодон-оптимизированным для экспрессии в эукариотической клетке, предпочтительно в клетке млекопитающего или клетке человека. В дополнительных вариантах осуществления настоящего изобретения белок Cas представляет собой белок системы CRISPR-Cas типа II, например, белок Cas9. В особенно предпочтительном варианте осуществления белок Cas представляет собой белок Cas9, например, SpCas9. В аспектах настоящего изобретения белок Cas имеет одну или несколько мутаций, выбранных из группы, состоящей из D10A, Е762А, Н840А, N854A, N863A и D986A. В особенно предпочтительном варианте осуществления белок Cas имеет мутацию D10A.
Аспекты настоящего изобретения относятся к снижению экспрессии продукта гена, или к дополнительному введению матричного полинуклеотида в молекулу ДНК, кодирующую продукт гена, или к точному вырезанию вставочной последовательности путем обеспечения повторного отжига и легирования двух "липких" 5'-концов, или к изменению активности или функции продукта гена, или к повышению экспрессии продукта гена. В варианте осуществления настоящего изобретения продукт гена представляет собой белок. В предпочтительных вариантах осуществления настоящего изобретения векторы системы являются вирусными векторами. В дополнительном варианте осуществления векторы системы доставляют посредством липосом, наночастиц, экзосом, микропузырьков или генной пушки.
В одном аспекте настоящее изобретение предусматривает способ модификации целевого полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления способ включает предоставление комплексу CRISPR возможности связываться с целевым полинуклеотидом для осуществления расщепления указанного целевого полинуклеотида с модификацией, таким образом, целевого полинуклеотида, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизующейся с целевой последовательностью в указанном целевом полинуклеотиде, где указанная направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизуется с tracr-последовательностью. В некоторых вариантах осуществления указанное расщепление включает расщепление одной или двух нитей в определенной точке целевой последовательности указанным ферментом CRISPR. В некоторых вариантах осуществления указанное расщепление приводит к сниженной транскрипции целевого гена. В некоторых вариантах осуществления способ дополнительно включает репарацию указанного расщепленного целевого полинуклеотида при помощи гомологичной рекомбинации с экзогенным матричным полинуклеотидом, где указанная репарация приводит к мутации, включающей вставку, делению или замену одного или нескольких нуклеотидов указанного целевого полинуклеотида. В некоторых вариантах осуществления указанная мутация приводит к одной или нескольким аминокислотным заменам в белке, экспрессируемом с гена, содержащего целевую последовательность. В некоторых вариантах осуществления способ дополнительно включает доставку одного или нескольких векторов в указанную эукариотическую клетку, где один или несколько векторов управляют экспрессией одного или нескольких из следующих: фермента CRISPR, направляющей последовательности, связанной с парной tracr-последовательностью, и tracr-последовательности. В некоторых вариантах осуществления указанные векторы доставляют в эукариотическую клетку у субъекта. В некоторых вариантах осуществления указанная модификация имеет место в указанной эукариотической клетке в клеточной культуре. В некоторых вариантах осуществления способ дополнительно включает выделение указанной эукариотической клетки из субъекта перед указанной модификацией. В некоторых вариантах осуществления способ дополнительно включает возвращение указанной эукариотической клетки и/или клеток, происходящих из нее, указанному субъекту.
В одном аспекте настоящее изобретение предусматривает способ модификации экспрессии полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления способ включает предоставление комплексу CRISPR возможности связываться с полинуклеотидом так, что указанное связывание приводит к повышенной или пониженной экспрессии указанного полинуклеотида; где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизующейся с целевой последовательностью в указанном целевом полинуклеотиде, где указанная направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизуется с tracr-последовательностью. В некоторых вариантах осуществления способ дополнительно включает доставку одного или нескольких векторов в указанные эукариотические клетки, где один или несколько векторов управляют экспрессией одного или нескольких из следующих: фермента CRISPR, направляющей последовательности, связанной с парной tracr-последовательностью, и tracr-последовательности.
В одном аспекте настоящее изобретение предусматривает способ получения модельной эукариотической клетки, содержащей мутированный ген, ответственный за развитие заболевания. В некоторых вариантах осуществления ген, ответственный за развитие заболевания, является любым геном, ассоциированным с повышением риска наличия или развития заболевания. В некоторых вариантах осуществления способ включает (а) введение одного или нескольких векторов в эукариотическую клетку, где один или несколько векторов управляют экспрессией одного или нескольких из следующих: фермента CRISPR, направляющей последовательности, связанной с парной tracr-последовательностью, и tracr-последовательности; и (b) предоставление комплексу CRISPR возможности связываться с целевым полинуклеотидом для осуществления расщепления целевого полинуклеотида в указанном гене, ответственном за развитие заболевания, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизуется с целевой последовательностью в целевом полинуклеотиде, и (2) парной tracr-последовательностью, которая гибридизуется с tracr-последовательностью, с получением, таким образом, модельной эукариотической клетки, содержащей мутированный ген, ответственный за развитие заболевания. В некоторых вариантах осуществления указанное расщепление включает расщепление одной или двух нитей в определенной точке целевой последовательности указанным ферментом CRISPR. В некоторых вариантах осуществления указанное расщепление приводит к сниженной транскрипции целевого гена. В некоторых вариантах осуществления способ дополнительно включает репарацию указанного расщепленного целевого полинуклеотида при помощи гомологичной рекомбинации с экзогенным матричным полинуклеотидом, где указанная репарация приводит к мутации, включающей вставку, делению или замену одного или нескольких нуклеотидов указанного целевого полинуклеотида. В некоторых вариантах осуществления указанная мутация приводит к одной или нескольким аминокислотным заменам при экспрессии белка из гена, содержащего целевую последовательность.
В одном аспекте настоящее изобретение предусматривает способ отбора одной или нескольких прокариотической(прокариотических) клетки(клеток) путем введения одной или нескольких мутаций в ген одной или нескольких прокариотической(прокариотических) клетки(клеток), при этом способ включает: введение одного или нескольких векторов в прокариотическую(прокариотические) клетку(клетки), где один или несколько векторов управляют экспрессией одного или нескольких из следующих: фермента CRISPR, направляющей последовательности, связанной с парной tracr-последовательностью, tracr-последовательности и матрицы редактирования; где редактирующая матрица содержит одну или несколько мутаций, которые прекращают расщепление фермента CRISPR; обеспечение возможности гомологичной рекомбинации редактирующей матрицы с целевым полинуклеотидом в отбираемой(отбираемых) клетке(клетках); предоставление комплексу CRISPR возможности связываться с целевым полинуклеотидом для осуществления расщепления целевого полинуклеотида в указанном гене, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизуется с целевой последовательностью в целевом полинуклеотиде, и (2) парной tracr-последовательностью, которая гибридизуется с tracr-последовательностью, где связывание комплекса CRISPR с целевым полинуклеотидом индуцирует гибель клеток, с обеспечением, таким образом, возможности отбора одной или нескольких прокариотических клеток, в которые были введены одна или несколько мутаций. В предпочтительном варианте осуществления фермент CRISPR представляет собой Cas9. В другом аспекте настоящего изобретения отбираемая клетка может быть эукариотической клеткой. Аспекты настоящего изобретения предусматривают отбор конкретных клеток без необходимости наличия маркера отбора или двухстадийного способа, который может включать систему негативного отбора.
В одном аспекте настоящее изобретение предусматривает способы модификации целевого полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления способ включает предоставление комплексу CRISPR возможности связываться с целевым полинуклеотидом для осуществления расщепления указанного целевого полинуклеотида с модификацией, таким образом, целевого полинуклеотида, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизующейся с целевой последовательностью в указанном целевом полинуклеотиде, где указанная направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизуется с tracr-последовательностью.
В других вариантах осуществления настоящее изобретение предусматривает способ модификации экспрессии полинуклеотида в эукариотической клетке. Способ включает повышение или снижение экспрессии целевого полинуклеотида с помощью комплекса CRISPR, который связывается с полинуклеотидом.
При желании для осуществления модификации экспрессии в клетке один или несколько векторов, содержащих tracr-последовательность, направляющую последовательность, связанную с парной tracr-последовательностью, последовательность, кодирующую фермент CRISPR, доставляют в клетку. В некоторых способах один или несколько векторов содержат регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей указанный фермент CRISPR, содержащий последовательность ядерной локализации; и регуляторный элемент, функционально связанный с парной tracr-последовательностью и одним или несколькими сайтами встраивания для встраивания направляющей последовательности выше парной tracr-последовательности. При экспрессии направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью в клетке. Как правило, комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизуется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизуется с tracr-последовательностью.
В некоторых способах целевой полинуклеотид можно инактивировать для осуществления модификации экспрессии в клетке. Например, при связывании комплекса CRISPR с целевой последовательностью в клетке целевой полинуклеотид инактивируют таким образом, что последовательность не транскрибируется, кодируемый белок не вырабатывается или последовательность не функционирует так, как последовательность дикого типа. Например, белок или кодирующую последовательность микроРНК можно инактивировать таким образом, что белок не будет продуцироваться.
В определенных вариантах осуществления фермент CRISPR содержит одну или несколько мутаций, выбранных из группы, состоящей из D10A, Е762А, Н840А, N854A, N863A или D986A, и/или одна или несколько мутаций находятся в домене RuvC1 или HNH фермента CRISPR или представляют собой другую мутацию, обсуждаемую в данном документе. В некоторых вариантах осуществления фермент CRISPR имеет одну или несколько мутаций в каталитическом домене, где при транскрипции парная tracr-последовательность гибридизуется с tracr-последовательностью, а направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью, и где фермент дополнительно содержит функциональный домен. В некоторых вариантах осуществления функциональный домен представляет собой домен активации транскрипции, предпочтительно VP64. В некоторых вариантах осуществления функциональный домен представляет собой домен репрессии транскрипции, предпочтительно KRAB. В некоторых вариантах осуществления домен репрессии транскрипции представляет собой SID или конкатемеры SID (например, SID4X). В некоторых вариантах осуществления функциональный домен представляет собой домен, участвующий в эпигенетической модификации, так что обеспечивается фермент, участвующий в эпигенетической модификации. В некоторых вариантах осуществления функциональный домен представляет собой активаторный домен, который может представлять собой активаторный домен Р65.
В некоторых вариантах осуществления фермент CRISPR представляет собой фермент CRISPR типа I или III, но предпочтительно представляет собой фермент CRISPR типа II. Этот фермент CRISPR типа II может быть любым ферментом Cas. Фермент Cas может быть идентифицирован как Cas9, поскольку он может относиться к общему классу ферментов, обладающих гомологией с самой большой нуклеазой с несколькими доменами нуклеаз системы CRISPR типа II. В наиболее предпочтительном случае фермент Cas9 получен или происходит из spCas9 или saCas9. Под происходящим заявители подразумевают, что основой для происходящего фермента главным образом является фермент дикого типа в том смысле, что он характеризуется высокой степенью гомологии последовательностей с этим ферментом, но был подвергнут мутации (модифицирован) некоторым образом, как описано в данном документе.
Следует иметь в виду, что выражения Cas и фермент CRISPR обычно используются в данном документе взаимозаменяемо, если иное не является очевидным. Как упоминается выше, многие из порядков нумерации остатков, используемых в данном документе, относятся к ферменту Cas9 из локуса CRISPR типа II Streptococcus pyogenes. Однако, следует иметь в виду, что настоящее изобретение включает многие другие Cas9 из других видов микроорганизмов, такие как SpCas9, SaCa9, St1Cas9 и т.д.
Пример кодон-оптимизированной последовательности, в данном случае оптимизированной для человека (т.е. оптимизированной для экспрессии у человека), представлен в данном документе, см. кодон-оптимизированную последовательность SaCas9 для человека. Хотя это является предпочтительным, следует иметь в виду, что возможны другие примеры и что для вида-хозяина известна оптимизация кодонов.
Доставку предпочтительно осуществляют в форме вектора, который может представлять собой вирусный вектор, как, например, векторы на основе лентивируса, пли бакуловируса, или, предпочтительно, аденовируса/аденоассоциированного вируса, но известны и предусмотрены другие средства доставки (такие как дрожжевые системы, микропузырьки, генные пушки/средства прикрепления векторов к наночастицам золота). Вектор может означать не только представляющие интерес вирусную или дрожжевую систему (например, где нуклеиновые кислоты могут быть функционально связанными с промотором и находиться под его контролем (как, например, для получения в конечном счете процессированной РНК, если говорить об экспрессии)), но также и прямую доставку нуклеиновых кислот в клетку-хозяина. Хотя в способах в данном документе вектор может быть вирусным вектором, и он преимущественно представляет собой AAV, можно использовать другие вирусные векторы, обсуждаемые в данном документе, такие как лентивирусы. Например, бакуловирусы можно использовать для экспрессии в клетках насекомых. Эти клетки насекомых могут, в свою очередь, быть применимыми для получения больших количеств дополнительных векторов, таких как векторы на основе AAV или лентивируса, приспособленных для доставки по настоящему изобретению. Также предусматривается способ доставки фермента CRISPR по настоящему изобретению, включающий доставку в клетку мРНК, кодирующей фермент CRISPR. Следует иметь в виду, что в определенных вариантах осуществления фермент CRISPR является усеченным, и/или содержит менее одной тысячи аминокислот или менее четырех тысяч аминокислот, и/или представляет собой нуклеазу или никазу, и/или является кодон-оптимизированным, и/или содержит одну или несколько мутаций, и/или включает химерный фермент CRISPR, и/или предусматривает другие варианты, обсуждаемые в данном документе. Предпочтительными являются векторы на основе AAV и лентивируса.
В определенных вариантах осуществления целевая последовательность на своем 3'-конце фланкирована или расположена перед РАМ, подходящим для фермента CRISPR, в основном Cas и, в частности, Cas9.
Например, подходящий РАМ представляет собой 5'-NRG или 5'-NNGRR для ферментов SpCas9 или SaCas9 (или происходящих из них ферментов), соответственно.
Следует иметь в виду, что SpCas9 или SaCas9 получены или происходят из Cas9 S. pyogenes или S. aureus.
Соответственно, целью настоящего изобретения не является охват в пределах настоящего изобретения любого ранее известного продукта, способа получения продукта или способа применения продукта, так что заявители оставляют за собой право и настоящим раскрывают отказ от прав на любой ранее известный продукт, процесс или способ. Следует дополнительно отметить, что настоящее изобретение не предназначено охватывать в пределах объема настоящего изобретения любой продукт, способ получения продукта или способ применения продукта, который не соответствует письменному описанию и требованиям достаточного раскрытия сути изобретения USPTO (первый пункт §112 статьи 35 USC) или ЕРО (статья 83 ЕРС), так что заявители оставляют за собой право и настоящим раскрывают отказ от прав на любой ранее описанный продукт, способ получения продукта или способ применения продукта.
Следует отметить, что в данном раскрытии и особенно в формуле изобретения и/или абзацах выражения, такие как "содержит", "содержащийся", "содержащий" и т.п., могут иметь значение, приписываемое им в патентном законодательстве США, например, они могут означать "включает", "включенный", "включающий" и т.п., и что такие выражения, как "состоящий по сути из" и "состоит по сути из", имеют значение, приписываемое им в патентном законодательстве США, например, они допускают не указанные прямо элементы, но исключают элементы, которые обнаружены в известном уровне техники или которые влияют на основные или новые характеристики настоящего изобретения.
Эти и другие варианты осуществления раскрыты или являются очевидными из следующего подробного описания и охватываются им.
Краткое описание графических материалов
Новые признаки настоящего изобретения изложены с характерными особенностями в прилагаемой формуле изобретения. Лучшее понимание признаков и преимуществ настоящего изобретения будет доступно благодаря ссылке на следующее подробное описание, в котором изложены показательные варианты осуществления, в которых используют принципы настоящего изобретения, и на сопутствующие графические материалы.
На фигуре 1 показана схематическая модель системы CRISPR. Нуклеаза Cas9 из Streptococcus pyogenes (желтый) нацелена на геномную ДНК при помощи синтетической направляющей РНК (sgRNA), состоящей из 20-нуклеотидной направляющей последовательности (голубой) и каркаса (красный). Направляющая последовательность образует пары оснований с ДНК-мишенью (голубой) непосредственно выше соответствующего мотива, прилегающего к протоспейсеру, 5'-NGG (РАМ; пурпурный), и Cas9 опосредует двухцепочечный разрыв (DSB) на ~3 п.о. выше РАМ (красный треугольник).
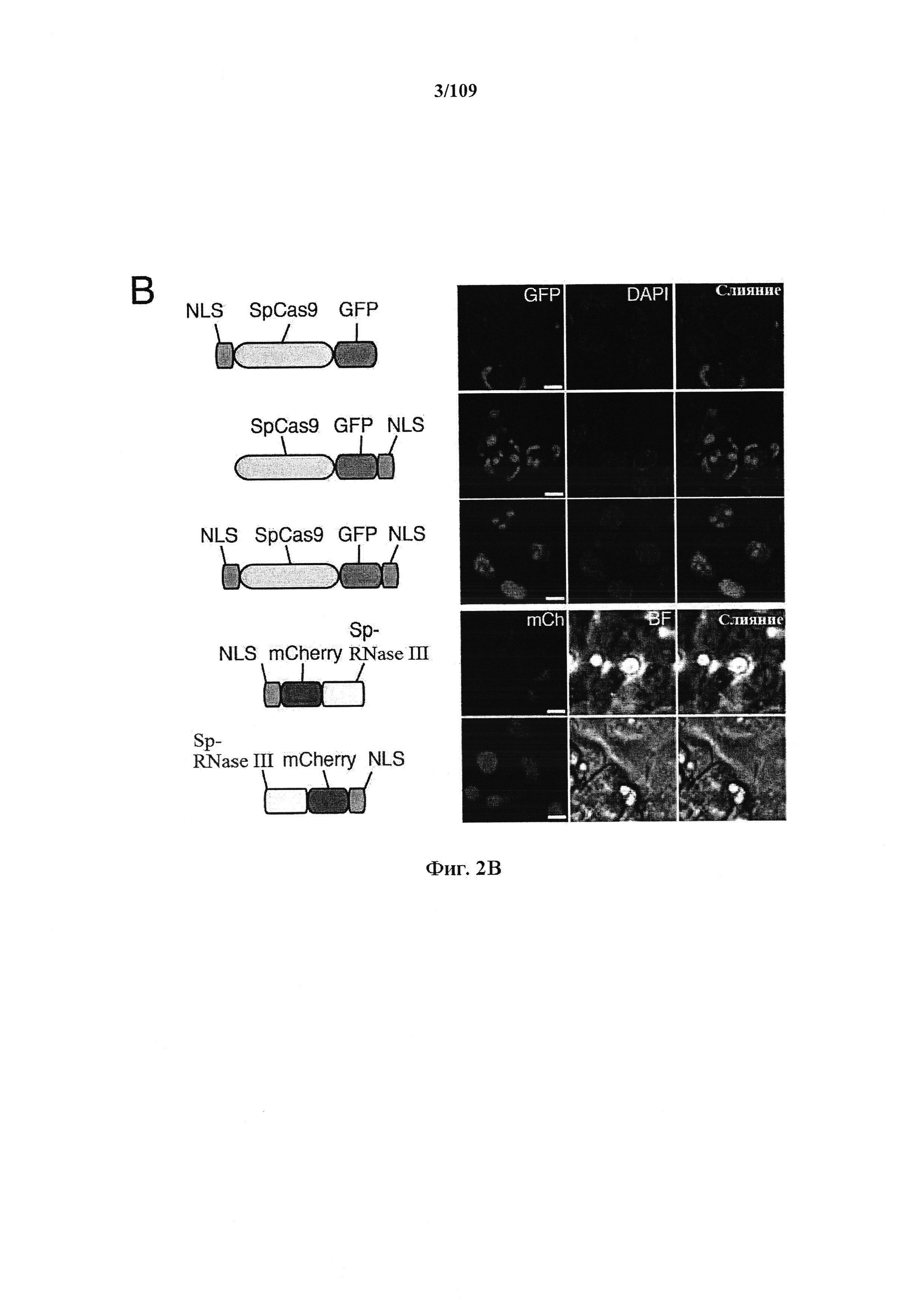
На фигурах 2A-F показана иллюстративная система CRISPR, возможный механизм действия, иллюстративная адаптация для экспрессии в эукариотических клетках и результаты тестов, оценивающих ядерную локализацию и активность CRISPR.
На фигурах 3A-D показаны результаты оценивания специфичности SpCas9 в отношении иллюстративной мишени.
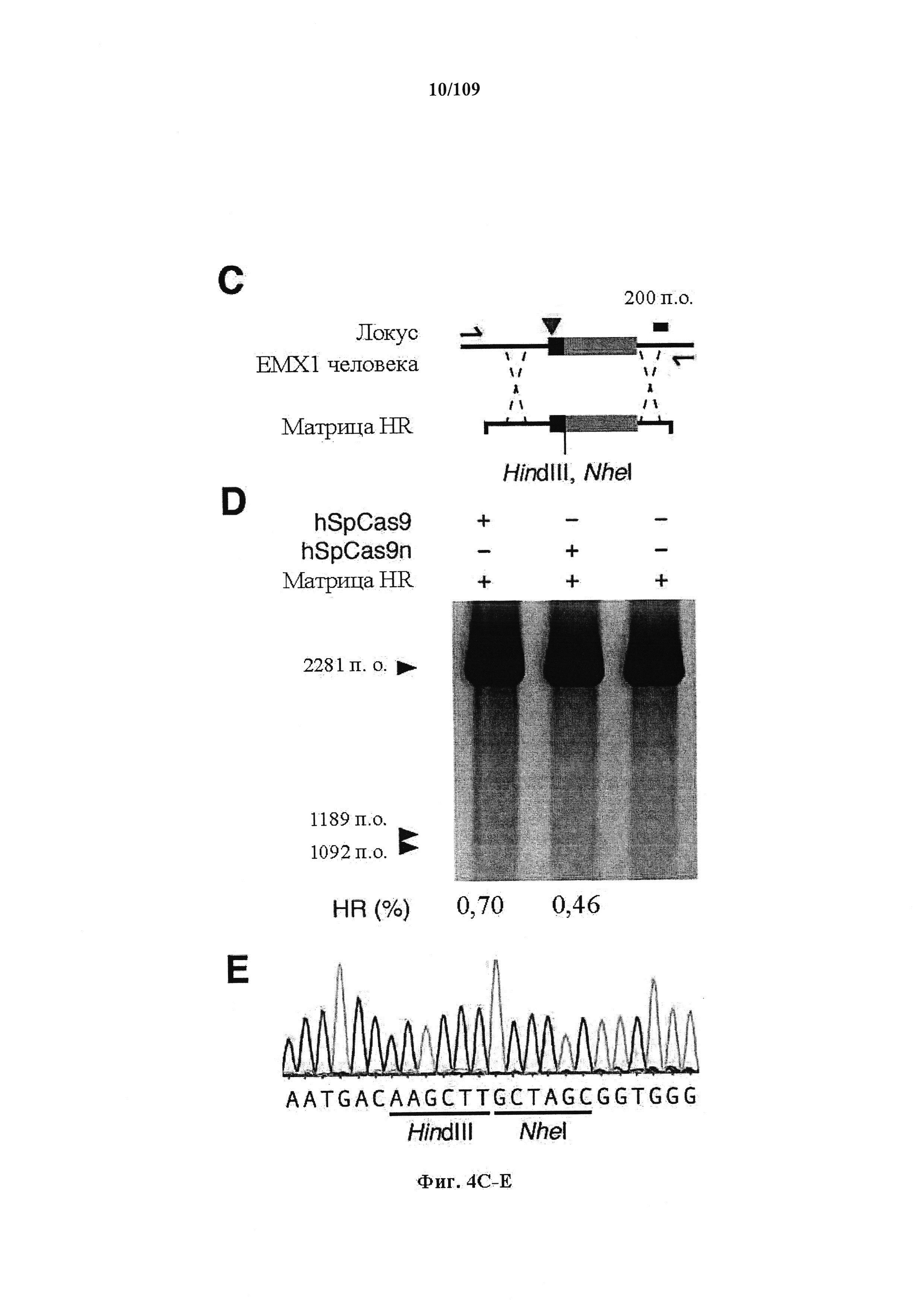
На фигурах 4A-G показана иллюстративная векторная система и результаты ее использования при управлении гомологичной рекомбинацией в эукариотических клетках.
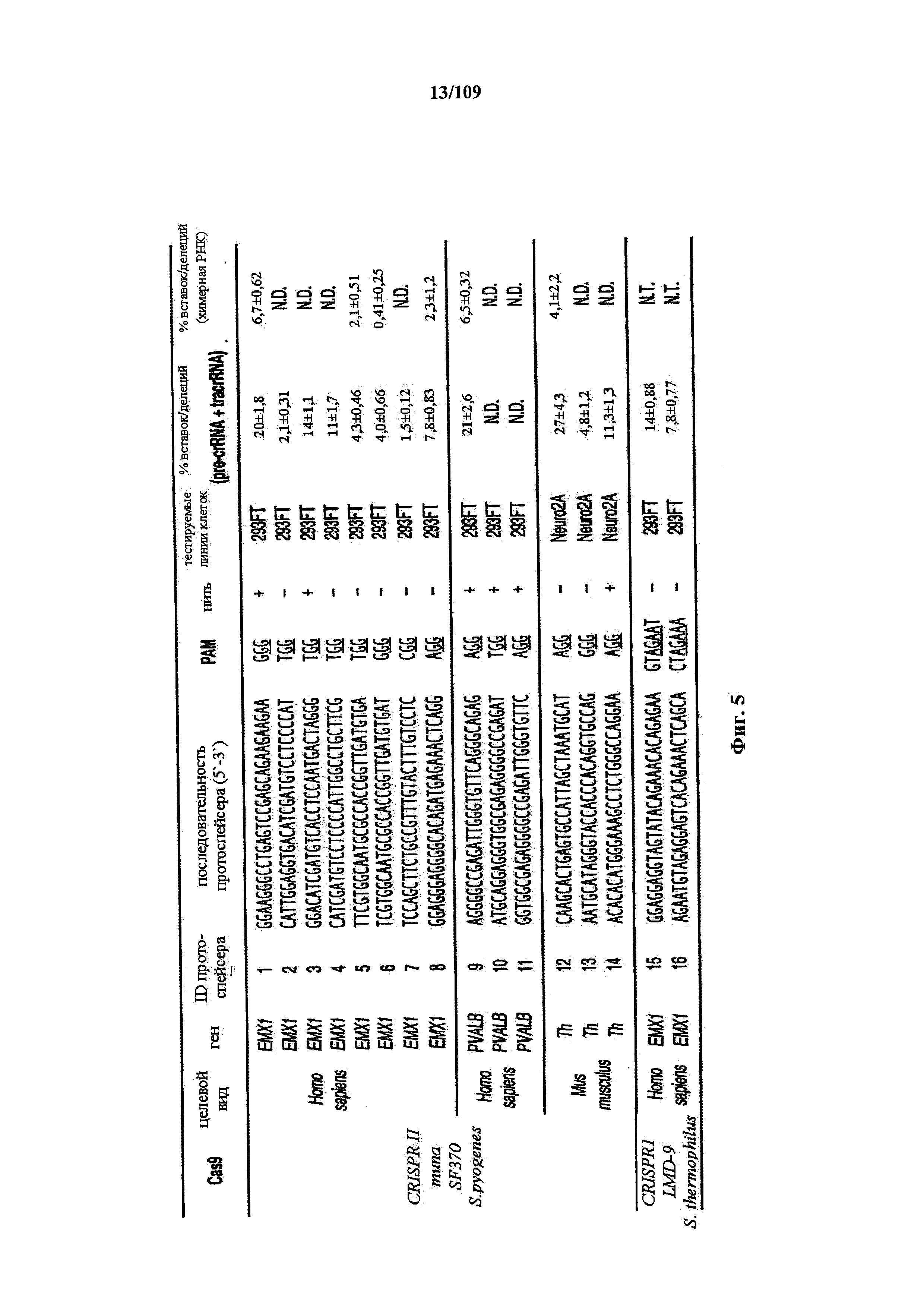
На фигуре 5 представлена таблица протоспейсерных последовательностей и обобщены результаты определения эффективности модификаций для протоспейсерных мишеней, сконструированных на основе иллюстративных систем CRISPR S. pyogenes и S. thermophilus с соответствующими РАМ, воздействующих на локусы в геномах человека и мыши. Клетки трансфицировали Cas9 и пре-crRNA/tracrRNA либо химерной РНК и анализировали через 72 часа после трансфекции. Процент вставок/делений рассчитывали на основе результатов анализа с помощью Surveyor с указанными линиями клеток (N=3 для всех протоспейсерных мишеней, ошибки представляют собой S.E.M., N.D. означает невозможно обнаружить посредством анализа с помощью Surveyor, и N.T. означает не тестировали в данном исследовании).
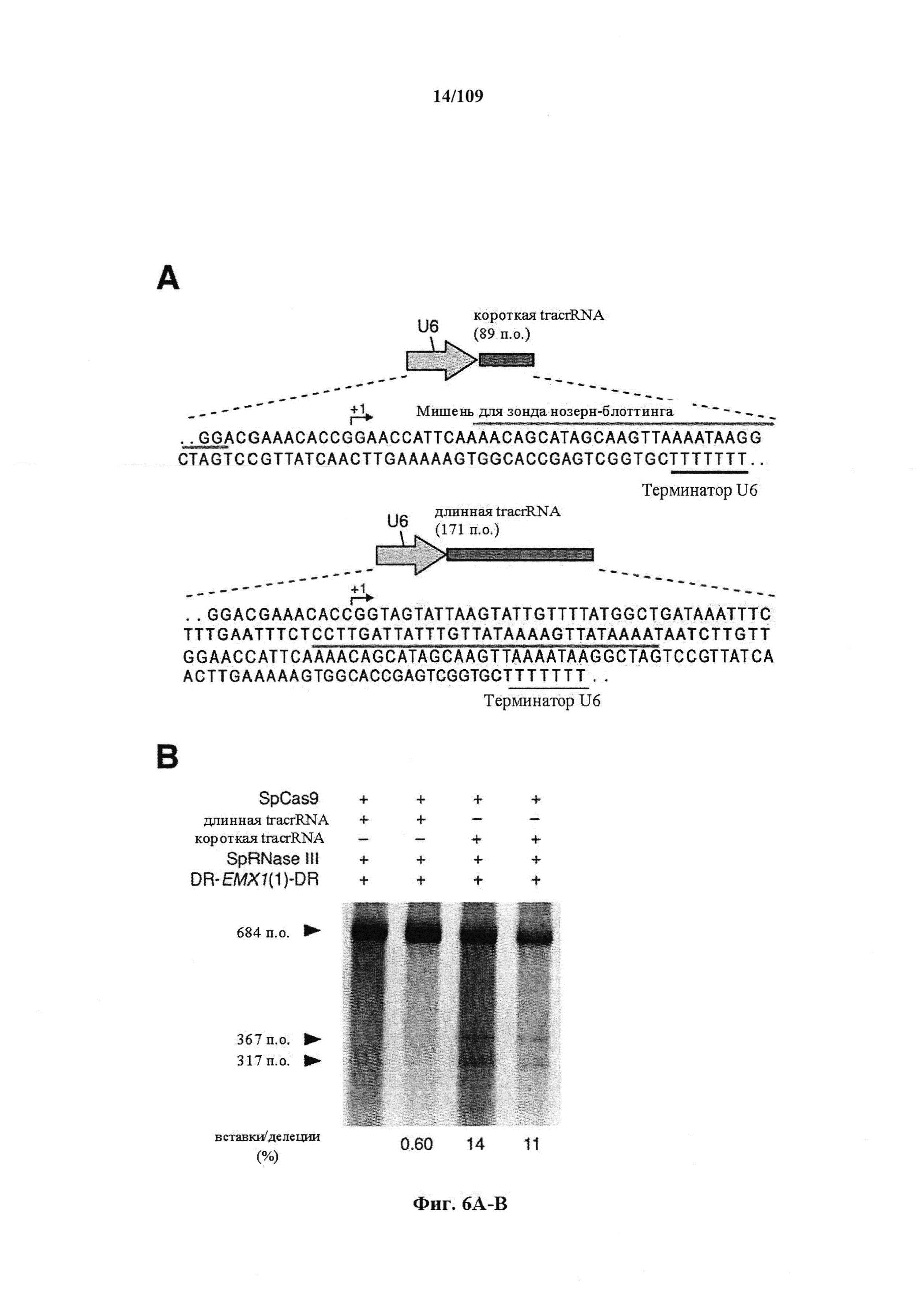
На фигурах 6А-С показано сравнение различных tracrRNA-транскриптов для Cas9-опосредованного целенаправленного воздействия на ген.
На фигуре 7 показано схематическое изображение анализа с использованием нуклеазы Surveyor для обнаружения индуцированных двухнитевым разрывом микровставок и микроделеций.
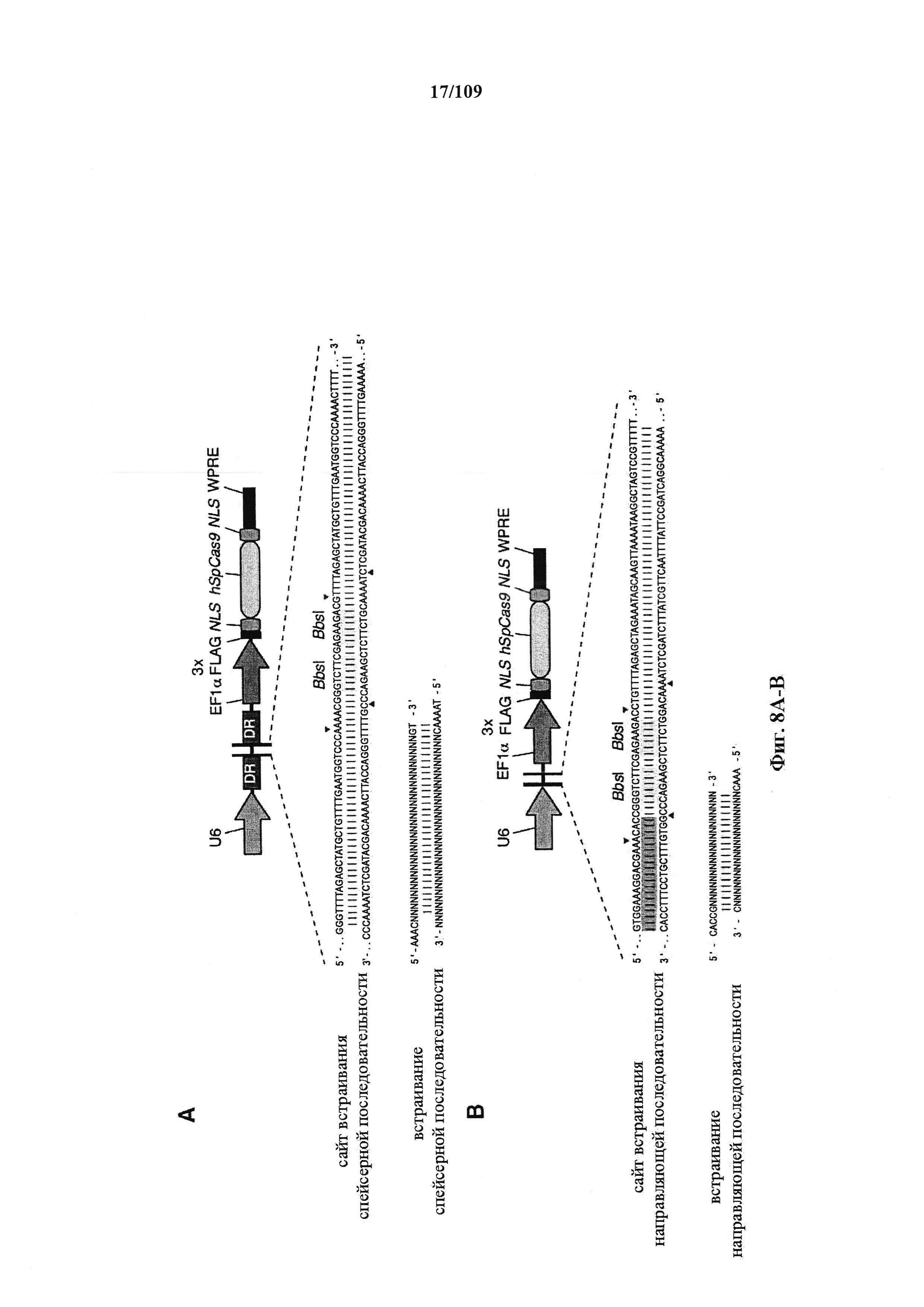
На фигурах 8А-В показаны иллюстративные бицистронные векторы экспрессии для экспрессии элементов системы CRISPR в эукариотических клетках.
На фигурах 9А-С показаны гистограммы расстояний между смежными РАМ (NGG) локуса 1 SF370 S. pyogenes (фигура 9А) и РАМ (NNAGAAW) локуса 2 LMD9 S. thermophilus (фигура 9В) в геноме человека; и расстояние для каждого РАМ в хромосомах (Chr) (фигура 9С).
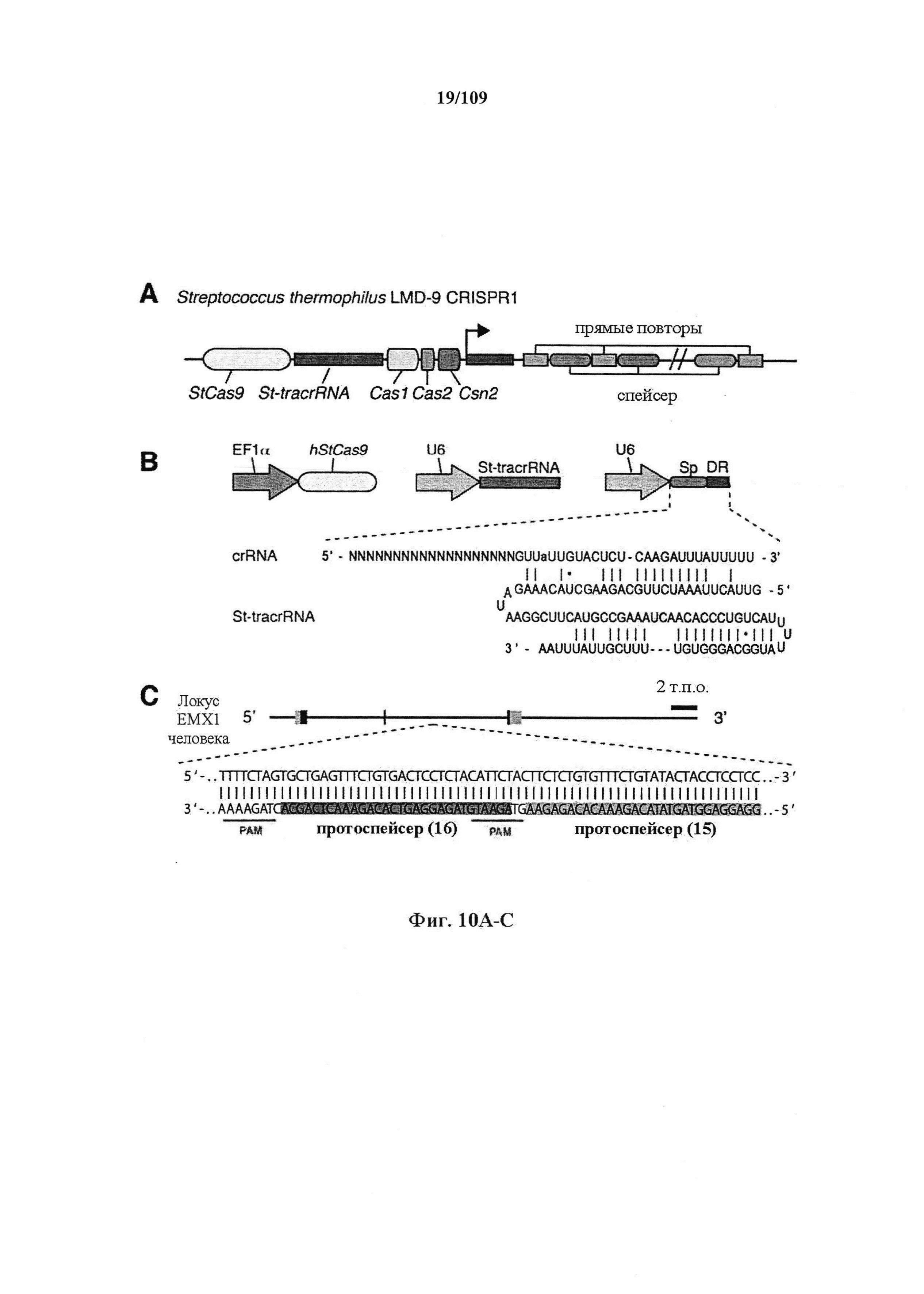
На фигурах 10A-D показана иллюстративная система CRISPR, иллюстративная адаптация для экспрессии в эукариотических клетках и результаты тестов, оценивающих активность CRISPR.
На фигурах 11А-С показаны иллюстративные манипуляции с системой CRISPR для целенаправленного воздействия на локусы генома в клетках млекопитающего.
На фигурах 12А-В показаны результаты нозерн-блот анализа процессинга crRNA в клетках млекопитающего.
На фигурах 13А-В показан иллюстративный отбор протоспейсеров в локусах PVALB человека и Th мыши.
На фигуре 14 показан иллюстративный протоспейсер и соответствующие целевые РАМ-последовательности системы CRISPR S. thermophilus в локусе ЕМХ1 человека.
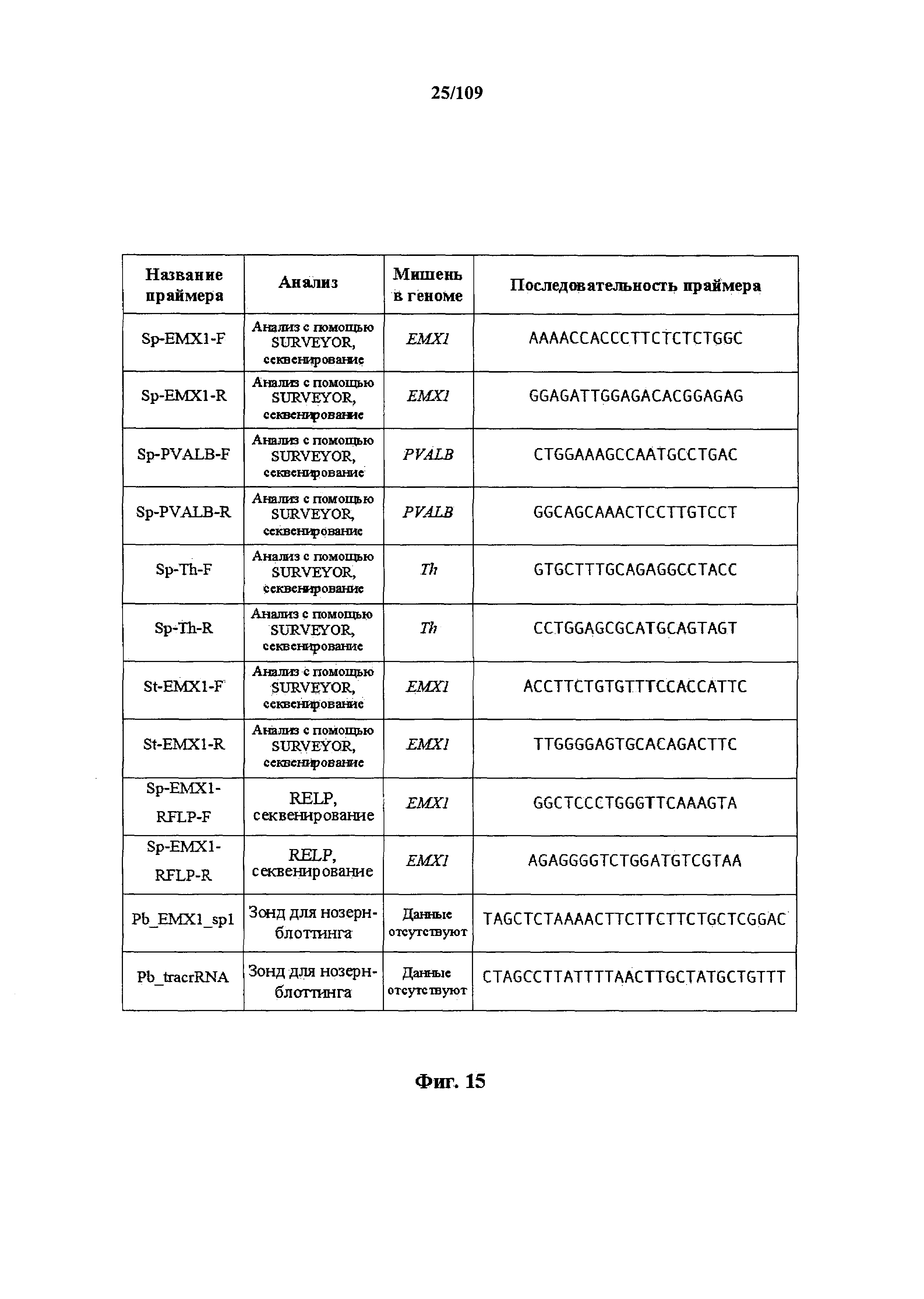
На фигуре 15 представлена таблица последовательностей для праймеров и зондов, используемых для анализа с помощью Surveyor, RFLP, геномного секвенирования и нозерн-блот анализов.
На фигурах 16А-С показана иллюстративная манипуляция с системой CRISPR с химерными РНК и результаты анализов с помощью SURVEYOR в отношении активности системы в эукариотических клетках.
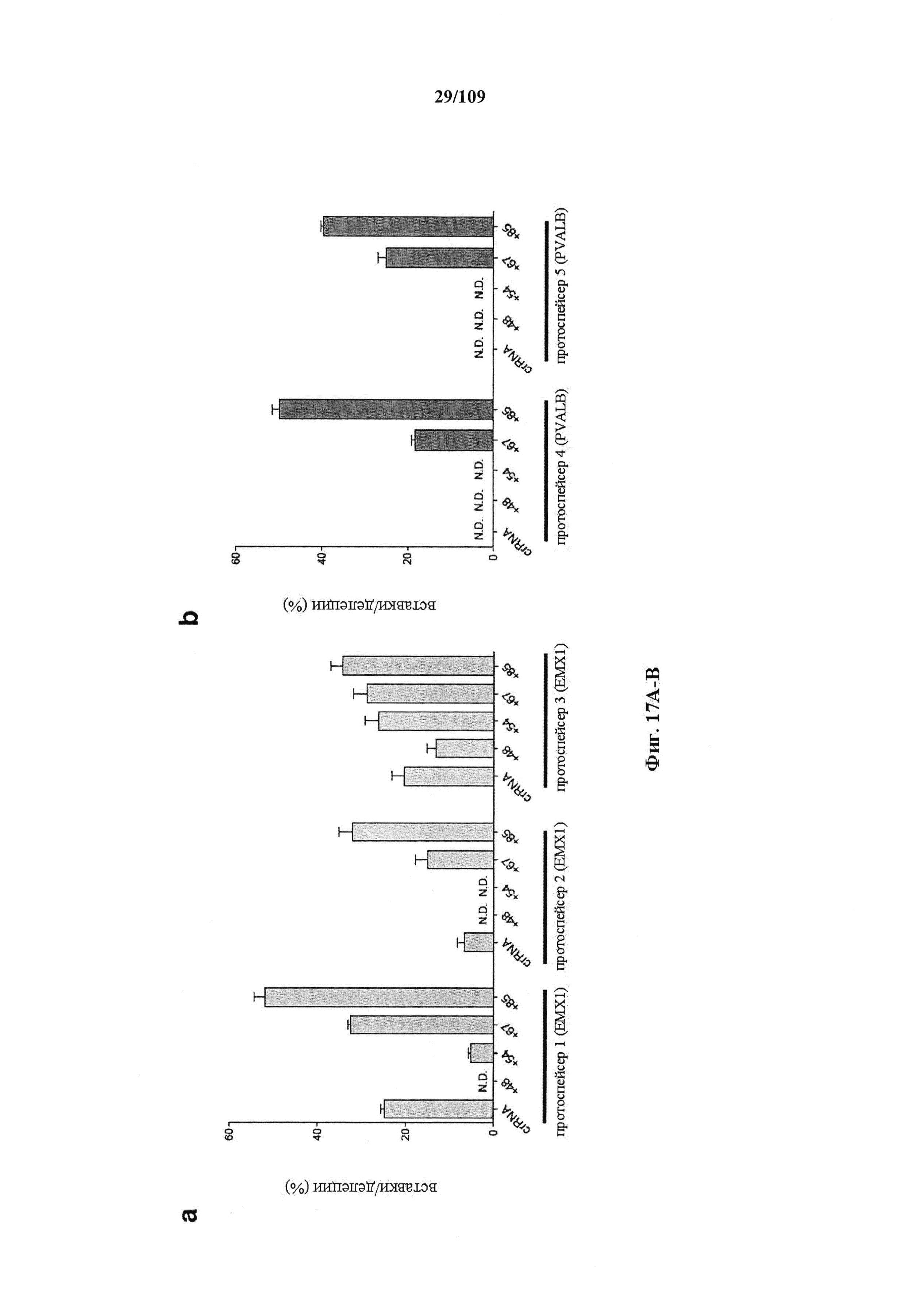
На фигурах 17А-В показано графическое изображение результатов анализов с помощью SURVEYOR в отношении активности системы CRISPR в эукариотических клетках.
На фигуре 18 показано иллюстративное отображение некоторых целевых сайтов для Cas9 S. pyogenes в геноме человека с использованием геномного браузера UCSC.
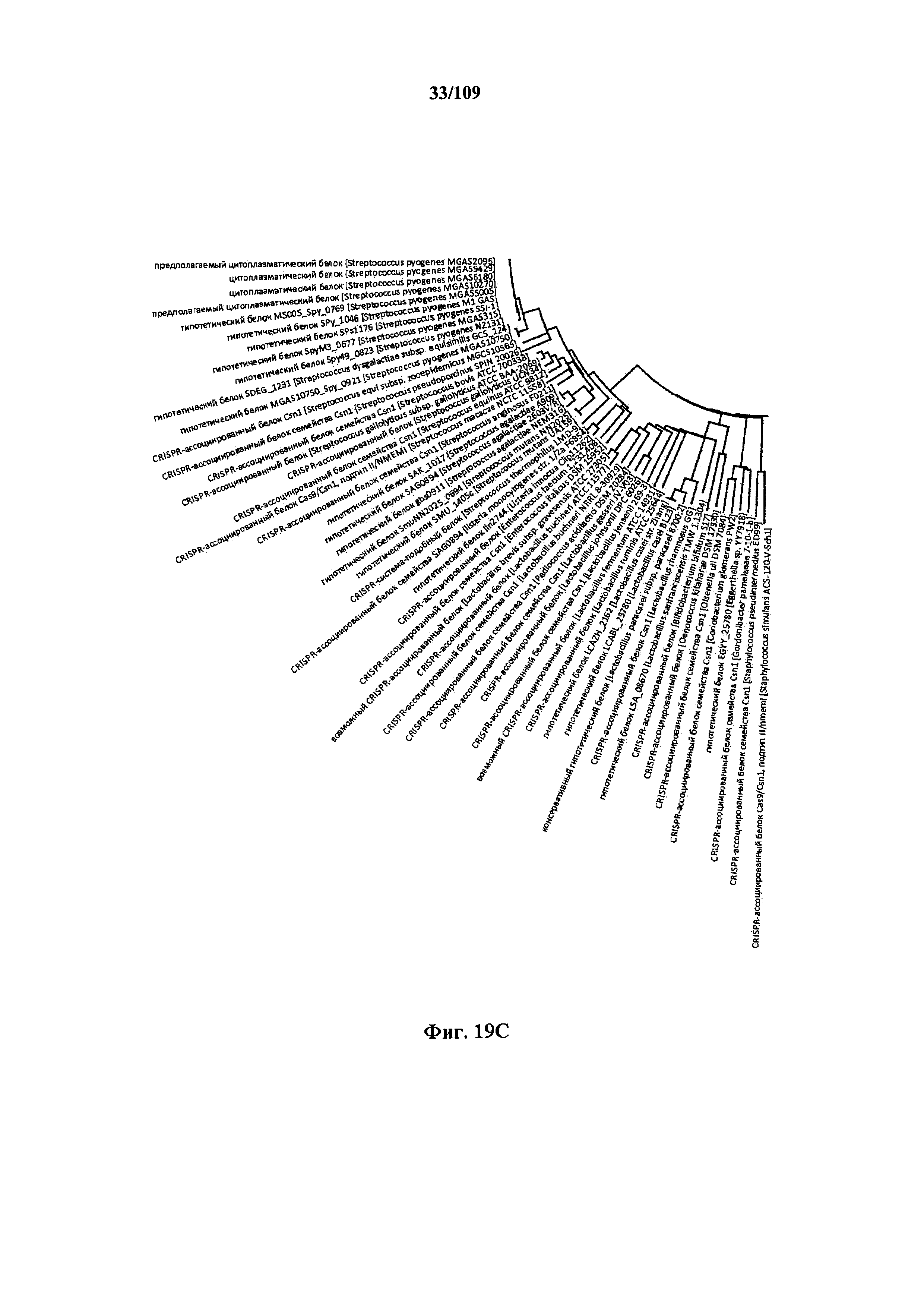
На фигурах 19A-D показано круговое представление данных филогенетического анализа, выявившего пять семейств Cas9, в том числе три группы больших Cas9 (~1400 аминокислот) и две группы малых Cas9 (~1100 аминокислот).
На фигурах 20A-F показано линейное представление данных филогенетического анализа, выявившего пять семейств Cas9, в том числе три группы большой Cas9 (~1400 аминокислот) и две группы малых Cas9 (~1100 аминокислот).
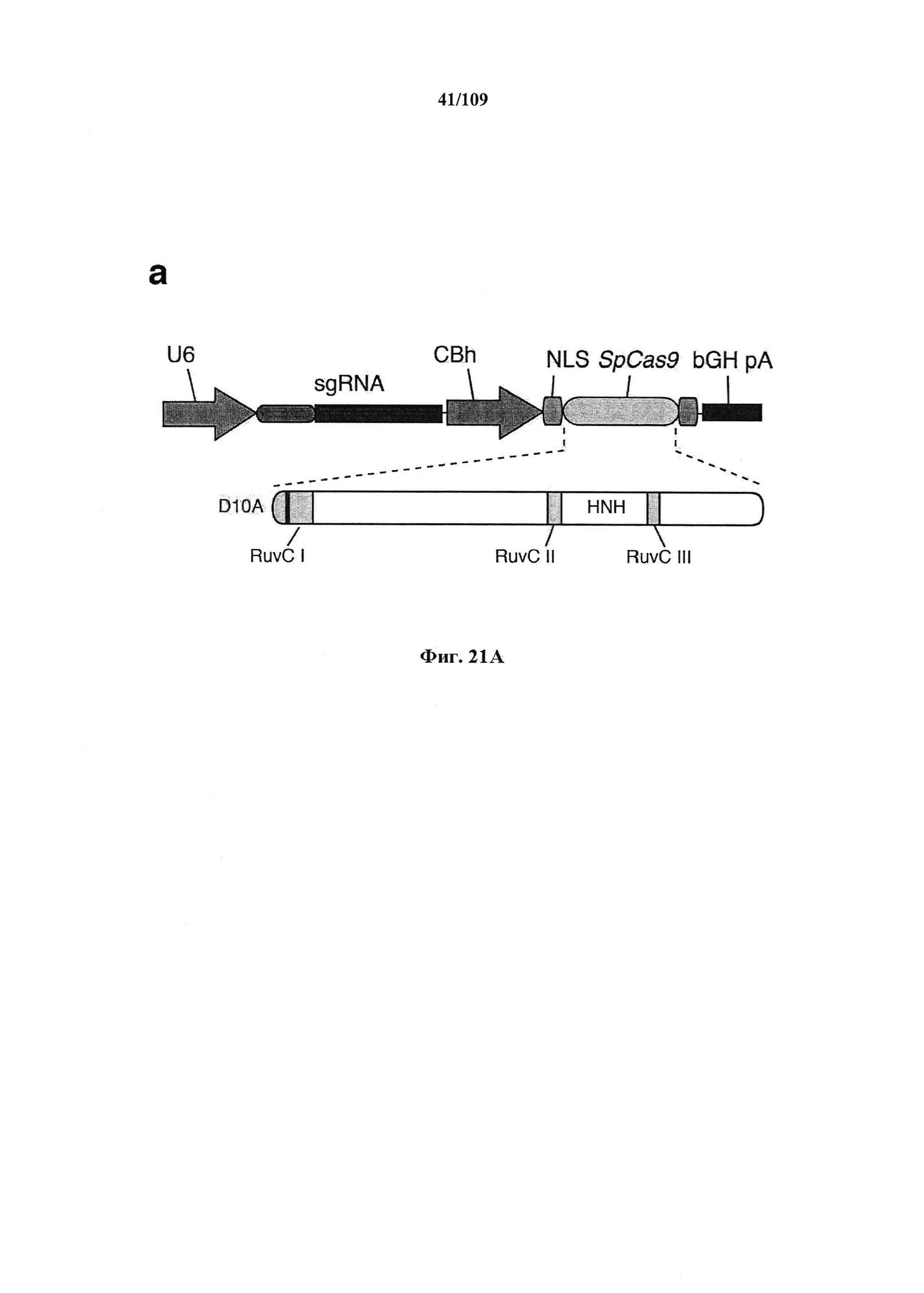
На фигурах 21A-D показано редактирование генома посредством гомологичной рекомбинации, (а) Схематическое изображение никазы SpCas9 с мутацией D10A в каталитическом домене RuvC I. (b) Схематическое представление гомологичной рекомбинации (HR) в локусе ЕМХ1 человека при использовании смысловых или антисмысловых однонитевых олигонуклеотидов в качестве матриц для репарации. Красная стрелка вверху указывает на сайт расщепления для sgRNA; праймеры для ПЦР для генотипирования (таблицы J и K) обозначены стрелками в правой панели, (с) Последовательность участка, модифицированного с помощью HR. (d) Анализ вставок/делений в целевом локусе 1 ЕМХ1 (n=3), опосредованных SpCas9 дикого типа (wt) и никазой SpCas9 (D10A), с помощью SURVEYOR. Стрелки указывают положения фрагментов ожидаемого размера.
На фигурах 22А-В показаны одновекторные структуры для SpCas9.
На фигуре 23 показана диаграмма, представляющая распределение ортологов Cas9 подлине.
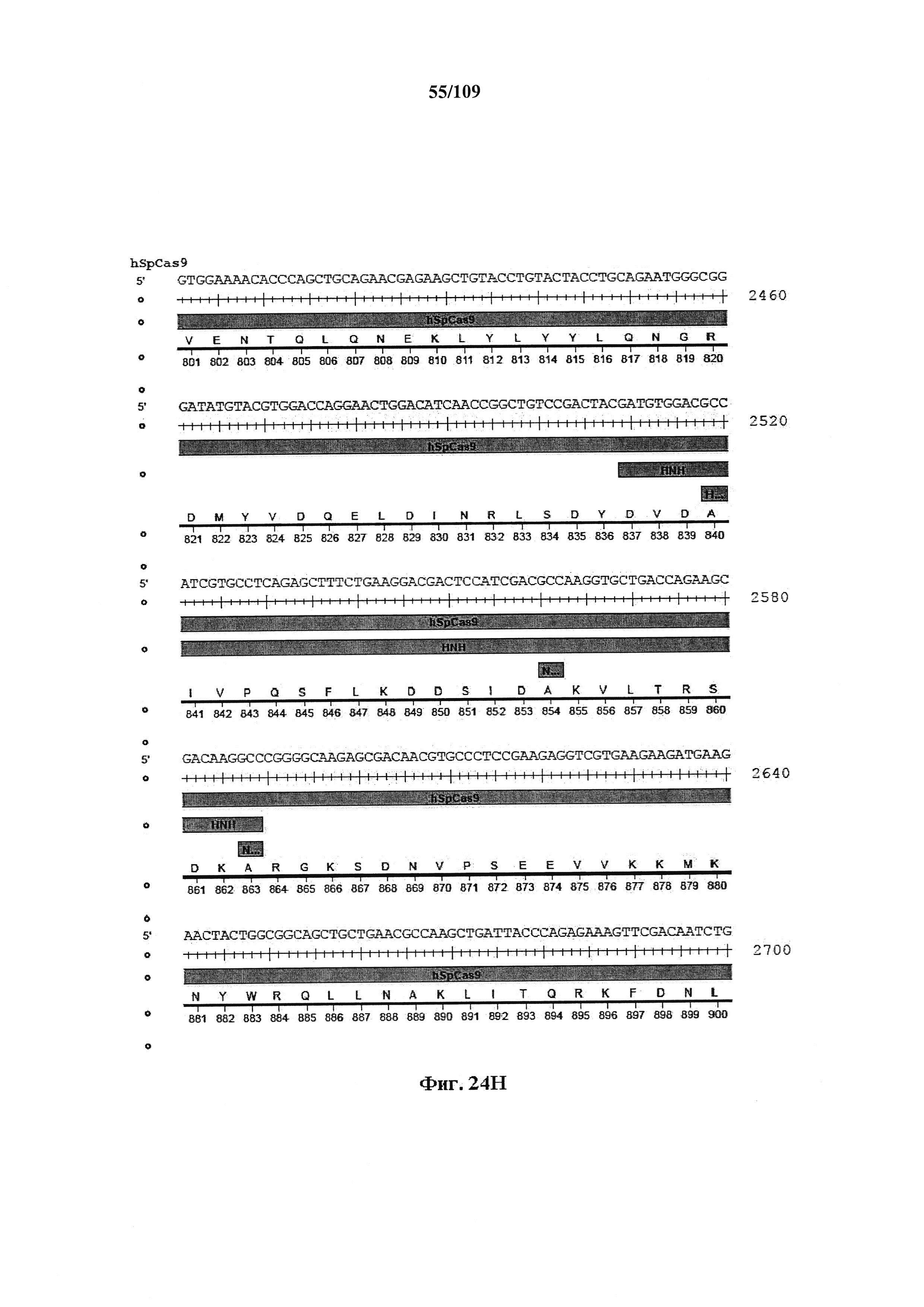
На фигурах 24А-М показаны последовательности, где положения мутаций находятся в гене SpCas9.
На фигуре 25А показана карта вектора на основе Cas9 для условного целенаправленного воздействия на Rosa26.
На фигуре 25В показана карта вектора на основе Cas9 для конститутивного целенаправленного воздействия на Rosa26.
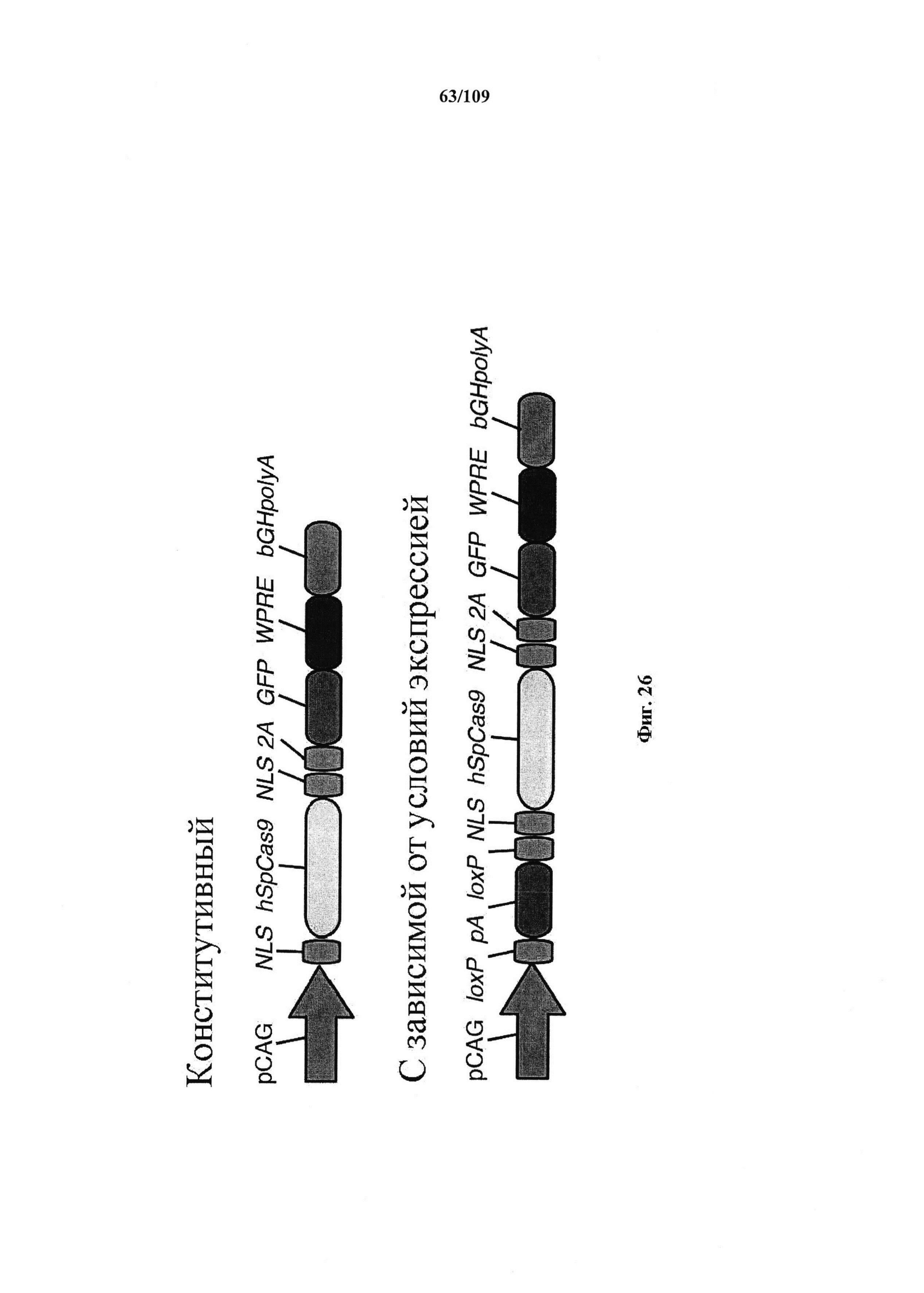
На фигуре 26 показано схематическое изображение важных элементов конструкций на основе Cas9 для конститутивного и условного целенаправленного воздействия.
На фигуре 27 показаны данные о доставке и in vivo экспрессии Cas9 в головном мозге мышей.
На фигуре 28 показана доставка Cas9 и химерной РНК в виде РНК в клетки. (А) Доставка репортера GFP в виде ДНК или мРНК в клетки Neuro-2A. (В) Доставка Cas9 и химерной РНК, воздействующих на ген Icam2, в виде РНК приводит к разрезанию одного из двух тестируемых спейсеров. (С) Доставка Cas9 и химерной РНК, воздействующих на ген F7, в виде РНК приводит к разрезанию одного из двух тестируемых спейсеров.
На фигуре 29 показано, как репарация двухнитевого разрыва ДНК (DSB) способствует редактированию генов. В пути склонного к ошибкам негомологичного соединения концов (NHEJ) концы DSB подвергаются обработке посредством эндогенных механизмов репарации ДНК и соединяются вместе, что может приводить к случайным мутациям по типу вставки или делении (вставки/делении) в месте соединения. Мутации по типу вставки/делении, имеющие место в кодирующем участке гена, могут обуславливать сдвиг рамки считывания и появление преждевременного стоп-кодона, что приводит к нокауту гена. Альтернативно, матрицу для репарации в виде плазмиды или однонитевых олигодезоксинуклеотидов (ssODN) можно предоставлять для эффективного использования пути репарации, направляемой гомологией (HDR), что обеспечивает высокое качество и точное редактирование.
На фигурах 30A-С показаны предполагаемые результаты HDR в клетках HEK и HUES9. (а) Целенаправленно воздействующую плазмиду или ssODN (смысловой или антисмысловой) с гомологичными плечами можно использовать для редактирования последовательности в целевом локусе генома, расщепляемом Cas9 (красный треугольник). Для анализа эффективности HDR вводили сайт для HindIII (красный прямоугольник) в целевой локус, который подвергали ПЦР-амплификации с праймерами, отжигаемыми за пределами участка гомологии. При расщеплении продукта ПЦР с помощью HindIII выявляют число случаев обнаружения событий HDR. (b) ssODN, ориентированные в смысловом или антисмысловом (s или а) направлении относительно представляющего интерес локуса, можно использовать в сочетании с Cas9 для достижения эффективного опосредованного HDR редактирования в целевом локусе. С каждой стороны от места модификации (красный прямоугольник) рекомендуется наличие минимального участка гомологии размером 40 п.о. и предпочтительно 90 п.о. (с) Пример эффекта ssODN в отношении HDR в локусе ЕМХ1 показан с использованием как Cas9 дикого типа, так и никазы Cas9 (D10A). Каждый ssODN содержит гомологичные плечи размером 90 п.о., фланкирующие вставку двух сайтов рестрикции размером 12 п.о.
На фигурах 31А-С показана стратегия репарации для мутации дельта-F508 гена регулятора трансмембранной проводимости при муковисцидозе.
На фигурах 32А-В (а) показано схематическое изображение экспансии GAA-повтора в интроне 1 FXN и (b) показано схематическое изображение стратегии, принятой для вырезания участка экспансии GAA с помощью системы CRISPR/Cas.
На фигуре 33 показан скрининг в отношении эффективного опосредованного SpCas9 целенаправленного воздействия на локусы генов Tet1-3 и Dnmt1, 3а и 3b. Анализ с помощью Surveyor в отношении ДНК из трансфицированных клеток N2A демонстрирует эффективное расщепление ДНК путем использования различных gRNA.
На фигуре 34 показана стратегия мультиплексного целенаправленного воздействия на геном с применением 2-векторной системы в системе доставки на основе AAV1/2. gRNA, воздействующая на Tet1-3 и Dnmt1, 3а и 3b, находится под контролем промотора U6. GFP-KASH находится под контролем промотора гена синапсина человека. Сайты рестрикции демонстрируют простую стратегию замены gRNA путем субклонирования. Показан SpCas9, меченный НА, фланкированный двумя сигналами ядерной локализации (NLS). Оба вектора доставляют в головной мозг с помощью вируса AAV1/2 в соотношении 1:1.
На фигуре 35 показано подтверждение функциональных свойств мультиплексного вектора #1 для целенаправленного воздействия на DNMT посредством анализа с помощью Surveyor. Клетки N2A котрансфипировали вектором #1 для целенаправленного воздействия на DNMT (+) и вектором, кодирующим SpCas9, для тестирования опосредованного SpCas9 расщепления локусов генов семейства DNMT. gRNA в отдельности (-) представляет собой отрицательный контроль. Клетки собирали для очистки и последующей обработки ДНК через 48 ч. после трансфекции.
На фигуре 36 показано подтверждение функциональных свойств мультиплексного вектора #2 для целенаправленного воздействия на DNMT посредством анализа с помощью Surveyor. Клетки N2A котрансфипировали вектором #1 для целенаправленного воздействия на DNMT (+) и вектором, кодирующим SpCas9, для тестирования опосредованного SpCas9 расщепления локусов генов семейства DNMT. gRNA в отдельности (-) представляет собой отрицательный контроль. Клетки собирали для очистки и последующей обработки ДНК через 48 ч. после трансфекции.
На фигуре 37 показано схематическое представление коротких промоторов и коротких вариантов поли(А), используемых для экспрессии HA-SpCas9 in vivo. Размеры кодирующих участков от L-ITR до R-ITR показаны справа.
На фигуре 38 показано схематическое представление коротких промоторов и коротких вариантов поли(А), используемых для экспрессии HA-SaCas9 in vivo. Размеры кодирующих участков от L-ITR до R-ITR показаны справа.
На фигуре 39 показана экспрессия SpCas9 и SaCas9 в клетках N2A. Иллюстративный вестерн-блоттинг меченных НА вариантов SpCas9 и SaCas9 под контролем различных коротких промоторов и с короткими поли(А)-последовательностями (spA). Тубулин представляет собой контроль загрузки. mCherry (mCh) представляет собой контроль трансфекции. Через 48 ч. после трансфекции клетки собирали и далее подвергали вестерн-блоттингу.
На фигуре 40 показан скрининг в отношении эффективного опосредованного SaCas9 целенаправленного воздействия на локус гена Tet3. Анализ с помощью Surveyor в отношении ДНК из трансфицированных клеток N2A демонстрирует эффективное расщепление ДНК путем применения различных gRNA с последовательностью PUM NNGGGT. Клетки, трансфипированные GFP, и клетки, экспрессирующие только SaCas9, являются контрольными.
На фигуре 41 показана экспрессия HA-SaCas9 в головном мозге мышей. Животным инъецировали в зубчатые извилины вирус, управляющий экспрессией HA-SaCas9 под контролем промотора гена синапсина человека. Животных умерщвляли через 2 недели после хирургической операции. НА-метку выявляли с помощью моноклонального антитела кролика C29F4 (Cell Signaling). Клеточные ядра окрашивали синим с помощью красителя DAPI.
На фигуре 42 показана экспрессия SpCas9 и SaCas9 в первичных кортикальных нейронах в культуре через 7 дней после трансдукции. Иллюстративный вестерн-блоттинг меченных НА вариантов SpCas9 и SaCas9 под контролем различных промоторов и с последовательностями bgh или короткими поли(А) (spA). Тубулин представляет собой контроль загрузки.
На фигуре 43 показано окрашивание с выявлением живых/мертвых первичных кортикальных нейронов через 7 дней после трансдукции частицами AAV1, несущими SpCas9 с различными промоторами и мультиплексные конструкции gRNA (пример показан на последней панели для DNMT). Нейроны после трансдукции с помощью AAV сравнивали с контрольными нетрансдуцированными нейронами. Красные ядра указывают на мертвые клетки с нарушенной проницаемостью мембраны (второй ряд панелей). Живые клетки отмечены зеленым цветом (третий ряд панелей).
На фигуре 44 показано окрашивание с выявлением живых/мертвых первичных кортикальных нейронов через 7 дней после трансдукции частицами AAV1, несущими SaCas9 с различными промоторами. Красные ядра указывают на мертвые клетки с нарушенной проницаемостью мембраны (второй ряд панелей). Живые клетки отмечены зеленым цветом (третий ряд панелей).
На фигуре 45 показано сравнение морфологических характеристик нейронов после трансдукции вирусом AAV1, несущим SpCas9 и мультиплексы gRNA для локусов генов ТЕТ и DNMT. Нейроны без трансдукции показаны в качестве контроля.
На фигуре 46 показано подтверждение функциональных свойств мультиплексного вектора #1 для целенаправленного воздействия на DNMT посредством анализа с помощью Surveyor в первичных кортикальных нейронах. Клетки котрансдуцировали вектором #1 для целенаправленного воздействия на DNMT и вирусами с SpCas9 с различными промоторами для тестирования опосредованного SpCas9 расщепления локусов генов семейства DNMT.
На фигуре 47 показана in vivo эффективность расщепления SpCas9 в головном мозге. Мышам инъецировали вирус AAV1/2, несущий мультиплекс gRNA, целенаправленно воздействующий на локусы генов семейства DNMT, вместе с вирусами с SpCas9 под контролем 2 различных промоторов: Меср2 мыши и Map1b крысы. Через две недели после инъекции ткань головного мозга извлекали, и ядра получали и сортировали с помощью FACS на основании экспрессии GFP под управлением промотора гена синапсина из мультиплексной конструкции gRNA. После экстракции gDNA выполняли анализ с помощью Surveyor. + означает GFP-положительные ядра, а - означает контрольные, GFP-отрицательные ядра от того же животного. Числа применительно к гелю означают оцененную эффективность SpCas9.
На фигуре 48 показана очистка меченных GFP-KASH клеточных ядер из нейронов гиппокампа. Внешняя ядерная мембрана (ONM) ядерной оболочки клетки мечена продуктом слияния GFP и белкового трансмембранного домена KASH. Наблюдается интенсивная экспрессия GFP в головном мозге через неделю после стереотаксического хирургического вмешательства и инъекции AAV1/2. Осуществляли стадию центрифугирования в градиенте плотности для очистки клеточных ядер от интактного головного мозга. Показаны очищенные ядра. Хроматин, окрашенный рубиновым красителем Vybrant® DyeCycle™, показан красным, меченные GFP ядра показаны зеленым. Иллюстративный профиль FACS GFP+ и GFP- клеточных ядер (пурпурный цвет: рубиновый краситель Vybrant® DyeCycle™, зеленый цвет: GFP).
На фигуре 49 показана эффективность расщепления SpCas9 в головном мозге мышей. Мышам инъецировали вирус AAV1/2, несущий мультиплекс gRNA, целенаправленно воздействующий на локусы генов семейства ТЕТ, вместе с вирусами с SpCas9 под контролем 2 различных промоторов: Меср2 мыши и Map1b крысы. Через три недели после инъекции ткань головного мозга извлекали, ядра получали и сортировали с помощью FACS на основании экспрессии GFP под управлением промотора гена синапсина из мультиплексной конструкции gRNA. После экстракции gDNA выполняли анализ с помощью Surveyor. + означает GFP-положительные ядра, а - означает контрольные, GFP-отрицательные ядра от того же животного. Числа применительно к гелю означают оцененную эффективность SpCas9.
На фигуре 50 показана экспрессия GFP-KASH в кортикальных нейронах в культуре. Нейроны трансдуцировали вирусом AAV1, несущим мультиплексные конструкции gRNA, целенаправленно воздействующие на локусы генов ТЕТ. Локализация самого сильного сигнала возле клеточных ядер обусловлена локализацией домена KASH.
На фигуре 51 показан (в верхней части) перечень расстояний (на которые указывает схема расположения для двух РАМ-последовательностей) между парами направляющих РНК. Только для пар направляющих РНК, удовлетворяющих паттернам 1, 2, 3, 4, проявляются вставки/делении в случае применения с никазой SpCas9(D10A). (Нижняя часть) Изображения гелей, демонстрирующие, что комбинация SpCas9(D10A) с парами направляющих РНК, удовлетворяющих паттернам 1, 2, 3, 4, приводит к образованию вставок/делений в целевом сайте.
На фигуре 52 показан перечень последовательностей обратных праймеров для U6, используемых для создания кассет экспрессии U6-направляющая РНК. Каждый праймер необходимо применять в паре с прямым праймером для U6 "gcactgagggcctatttcccatgattc" для образования ампликонов, содержащих U6 и требуемую направляющую РНК.
На фигуре 53 показана карта секвенирования генома в локусе Emx1 человека, демонстрирующая местоположения 24 паттернов, перечисленных на фигуре 33.
На фигуре 54 показано (справа) изображение геля, указывающее на образование вставок/делений в целевом сайте при наличии различных "липких" 5'-концов после расщепления никазой Cas9, нацеливаемой различными парами направляющих РНК. (Слева) Таблица, в которой указаны номера дорожек для геля справа и различные параметры, в том числе идентификация применяемых пар направляющих РНК и длина "липкого" 5'-конца, имеющегося в наличии после расщепления никазой Cas9.
На фигуре 55 показана карта секвенирования генома в локусе Emx1 человека, демонстрирующая местоположения различных пар направляющих РНК, которые обуславливают картины разделения в геле, показанные на фиг. 54 (справа), и которые дополнительно описаны в примере 35.
На фигуре 56 показано окрашивание HA-SpCas9 в дорсальной и вентральной части гиппокампа через 8 недель после инъекции вирусов, кодирующих Mecp2-HA-SpCas9 и 3xgRNA-TETS с Syn-KASH-GFP.
На фигуре 57 показано, что экспрессия Syn_GFP-KASH через 8 недель после инъекции вируса с 3xgRNA является специфичной для нейронов (NeuN-положительные клетки), но не для глиальных клеток (GFAP-положительные).
На фигуре 58 показаны поведенческие тесты, проведенные через 5 недель после опосредованного CRISPR KD ТЕТ и DNMT в зубчатой извилине (вентральная и дорсальная части), демонстрирующие повышенный уровень тревожности и нарушения способности к обучению. А) Время, проведенное в открытом рукаве во время теста в приподнятом крестообразном лабиринте. В) Тест "открытое поле", измеряли время, проведенное в центре арены, по сравнению со временем, проведенным в углах. С) Тест на распознавание нового объекта, результаты определяли через 3 ч. после окончания фазы ознакомления. D) Лабиринт Барнса; эффективность нахождения выхода в течение 3 дней тренировки. Е) Результаты теста в лабиринте Барнса. F) Замирание во время выработки обстановочного условного рефлекса в связи с переживанием чувства страха. G) Время ожидания первого эпизода замирания во время выработки обстановочного условного рефлекса в связи с переживанием чувства страха. Н) Результаты выработки следового условного рефлекса в связи с переживанием чувства страха для KD ТЕТ и KD DNMT (I). Контроль - животные, которым инъецировали вирус с SpCas9 и конструкцию GFP-KASH без gRNA. ТЕТ - животные, которым инъецировали как SpCas9, так и конструкцию, кодирующую gRNA, воздействующие на Tet1, Tet2 и Tet3. DNMT - животные, которым инъецировали как SpCas9, так и конструкцию, кодирующую gRNA, воздействующие на Dnmt1, Dnmt3a и Dnmt3b.
На фигуре 59 показана эффективность разрезания в локусах Tet в головном мозге через 8 недель после инъекции вируса с Mecp_SpCas9 по сравнению с контрольными животными, которым инъецировали только вирус с Mecp2_SpCas9.
На фигуре 60 показана эффективность разрезания в локусах Dnmt в головном мозге через 8 недель после инъекции вируса с Mecp_SpCas9 по сравнению с контрольными животными, которым инъецировали только вирус с Mecp2_SpCas9.
На фигуре 61 показано окрашивание Dnmt3a в головном мозге через 8 недель после стереотаксической инъекции вируса, кодирующего Mecp2_SpCas9 и gRNA, целенаправленно воздействующие на локусы Dnmt. На нижней панели показан увеличенный ROI, отмеченный на верхней панели.
На фигуре 62 показано окрашивание Syn_HA-SaCas9 в дорсальной части гиппокампа через 4 недели после инъекции вируса. В первом столбце показана картина для животных, которым инъецировали только Sa-Cas9, в среднем столбце - для животных, которым инъецировали как SaCas9, так и gRNA, воздействующие на локусы ТЕТ, а в правом столбце представлена картина для животных, которым инъецировали только вирус, кодирующий gRNA. Ядерная локализация SaCas9 зависит от наличия gRNA.
На фигуре 63 показан SpCas9 в клетках N2a. А) Вектор для целенаправленного воздействия и экспрессии SpCas9. В) Вестерн-блот анализ клеток N2a, экспрессирующих меченный НА SpCas9 под контролем различных промоторов. С) Эффективность разрезания в локусах Dnmt. D) Вестерн-блот анализ, демонстрирующий эффективный нокдаун Dnmt3a. е) Эффективность разрезания в локусах Tet.
На фигуре 64 показан SpCas9 в первичных нейронах. А) Схематическое представление стратегий клонирования SpCas9, применяемых в данном исследовании. Показаны короткие промоторы и короткие поли(А) для эффективной упаковки в систему доставки на основе AAV. В) Схематическое представление комбинированной стратегии мультиплексного целенаправленного воздействия и мечения ядерной оболочки. С) Вестерн-блот анализ, демонстрирующий экспрессию меченного НА SpCas9 под контролем промоторов rMap1b и mMecp2 и сигнальных последовательностей bGH и spA. D) Иммуноцитохимический анализ, демонстрирующий совместную экспрессию SpCas9 и GFP-KASH в первичных нейронах. SpCas9 под контролем промотора mMecp2 экспрессируется в нейронах (Map1b, NeuN), но не в клетках астроглии (GFAP).
На фигуре 65 показан нокдаун Dnmt3a в первичных нейронах. А) Иммуноцитохимический анализ, демонстрирующий эффективный нокдаун Dnmt3a после целенаправленного воздействия мультиплексного вектора для целенаправленного воздействия и mMecp2-SpCas9. В) Количественное представление окрашивания Dnmt3a антителами в контрольных и подвергнутых целенаправленному воздействию нейронах. С) Вестерн-блот анализ, демонстрирующий пониженный уровень белка Dnmt3a. D) Количественное представление вестерн-блот анализа, демонстрирующее полный нокдаун уровня белка Dnmt3a в смешанной культуре первичных нейронов (нейроны и астроглия), составляющий приблизительно 75%.
На фигуре 66 показан нокдаун Dnmt3a in vivo. А) Эффективность разрезания в локусах Dnmt в головном мозге через 8 недель после инъекции вируса с Mecp_SpCas9 по сравнению с контрольными животными, которым инъецировали только вирус с Mecp2_SpCas9. В) Вестерн-блот анализ, демонстрирующий пониженный уровень белка Dnmt3a в подвергнутых целенаправленному воздействию ядрах нейронов (KASH-GFP-положительных) по сравнению с контрольными ядрами (положительно окрашиваемых рубиновым красителем) после сортировки клеточных ядер с помощью FACS.
На фигуре 67 показана экспрессия SaCas9 в первичных нейронах. А) Размер вектора экспрессии SaCas9 при использовании промотора гена синапсина человека и сигнальной последовательности bGH. В) Экспрессия SaCas9 в первичных нейронах (NeuN), но не в астроглии (GFAP). С) Внеядерная локализация SaCas9 в отсутствие gRNA. С') SaCas9-положительные нейроны, показанные на С), при большем увеличении. D) Ядерная локализация SaCas9 в присутствии gRNA. D') SaCas9-положительные нейроны, показанные на D), при большем увеличении. Е) Вестерн-блот анализ, демонстрирующий экспрессию меченного НА SaCas9 и GFP-KASH. F) Эффективность разрезания в локусах Dnmt через 1 неделю после инфицирования AAV.
На фигуре 68 показана зависимая от gRNA ядерная локализация SaCas9. А) Анализ конфокальных изображений, демонстрирующий внеядерную локализацию SaCas9 в отсутствие gRNA в первичных нейронах. В) Ядерная локализация SaCas9 в присутствии gRNA. С) Анализ по методу линейного сканирования конфокального изображения А), показывающий внеядерную локализацию SaCas9 в отсутствие gRNA (красный - сигнал от SaCas9; синий - сигнал от DAPI; зеленый - сигнал от GFP-KASH). D) Анализ по методу линейного сканирования конфокального изображения В), показывающий ядерную локализацию SaCas9 в присутствии gRNA (красный - сигнал от SaCas9; синий - сигнал от DAPI; зеленый - сигнал от GFP-KASH). E) Субклеточная локализация SaCas9 и SpCas9 в условиях без (-) и с (+) gRNA в клетках N2a. Сигнал от SaCas9 при 250 кДа во фракции цитоплазмы (тубулин-положительной) указывает на димеризацию SaCas9 в цитоплазме. В присутствии gRNA визуально заметно перемещение белка SaCas9 в ядерную фракцию (Sun2-положительную). Сигнал от SaCas9 при 100 кДа указывает на зависимые от gRNA образование гомомеров SaCas9 и транспорт в клеточное ядро. В отличие от этого, SpCas9 присутствует главным образом в качестве гомомера, и его ядерная локализация не зависит от gRNA.
На фигуре 69 показан вектор AAV-Sa-Cas9, печеночноспецифический вектор AAV-Sa-Cas9 и альтернативный вектор AAV-Sa-Cas9.
На фигуре 70 показаны данные относительно оптимизированного вектора CMV-SaCas9-NLS-U6-sgRNA (последней представленной векторной структуры); по новым данным сравнивают SaCas9, меченные на N'-конце и C'-конце, и показывают повышение эффективности расщепления при применении C'-концевого мечения с помощью NLS.
На фигуре 71 показано изображение результатов анализа с помощью SURVEYOR, показывающее вставки/делении, образованные в новых мишенях Pcsk9.
На фигуре 72 показана специфичность SaCas9: нецелевые сайты во всем геноме (GWOT) предсказывают на основании 2 критериев: они содержат 4 или менее несовпадающих оснований по сравнению с предполагаемой мишенью для SaCas9 и имеют наименее ограничительный РАМ для SaCas9, NNGRR. Клетки HEK 293FT трансфицируют с помощью SpCas9 или SaCas9 с их соответствующими sgRNA, воздействующих на целевой сайт (ЕМХ1: TAGGGTTAGGGGCCCCAGGC), имеющий CGGGGT в качестве РАМ, так что он может разрезаться с помощью SpCas9 (CGG) или SaCas9 (CGGGGT). ДНК из клеток собирают и анализируют с помощью технологии секвенирования Illumina в отношении вставка/деления в целевом и 41 предсказанном нецелевом локусе (согласно протоколам от Hsu et al. Nature Biotech 2013 и системе анализа данных, разработанной David Scott и Josh Weinstein).
На фигуре 73 показано, что SaCas9 может иметь более высокий уровень нецелевой активности, чем SpCas9, в определенных локусах.
Фигуры приведены в данном документе только в целях иллюстрации, и они не обязательно должны быть вычерчены в масштабе.
Подробное описание изобретения
Настоящее изобретение относится к конструированию и оптимизации систем, способов и композиций, применяемых для контроля экспрессии генов, включающего целенаправленное воздействие на последовательность, такое как внесение изменений в геном или редактирование генов, связанное с системой CRISPR-Cas и ее компонентами. В преимущественных вариантах осуществления фермент Cas представляет собой Cas9.
Преимущество способов по настоящему изобретению заключается в том, что система CRISPR избегает нецелевого связывания и возникающих в результате этого побочных эффектов. Это достигается при применении систем, предусматривающих наличие высокой степени специфичности к последовательности в отношении целевой ДНК.
Cas9
Оптимизацию Cas9 можно применять для улучшения функционирования или для формирования новых функций, можно создавать химерные белки Cas9. Созданные заявителями образцы представлены в примере 6. Химерные белки Cas9 можно получать путем объединения фрагментов от различных гомологов Cas9. Например, в данном документе описаны два иллюстративных химерных белка Cas9 из Cas9. Например, заявители слили N-конец St1Cas9 (фрагмент из этого белка выделен жирным шрифтом) с С-концом SpCas9. Выгода от создания химерных Cas9 включает любое или все из следующего: пониженная токсичность; улучшенная экспрессия в эукариотических клетках; повышенная специфичность; сниженный молекулярный вес белка, например, при получении меньшего белка за счет объединения наименьших доменов от различных гомологов Cas9; и/или изменение требований к последовательности РАМ.
Cas9 можно использовать в качестве стандартного ДНК-связывающего белка. Например, как показано в примере 7, заявители использовали Cas9 в качестве стандартного ДНК-связывающего белка путем внесения мутации в два каталитических домена (D10 и Н840), ответственных за расщепление обеих нитей ДНК-мишени. С целью повышающей регуляции транскрипции гена в целевом локусе заявители слили домен активации транскрипции (VP64) с Cas9. Известны и другие домены активации транскрипции. Как показано в примере 17, активация транскрипции является возможной. Как также показано в примере 17, репрессия генов (в этом случае гена бета-катенина) возможна при использовании репрессора Cas9 (ДНК-связывающего домена), который связывается с последовательностью целевого гена, тем самым подавляя ее активность.
Cas9 и одну или несколько направляющих РНК можно доставлять при помощи аденоассоциированного вируса (AAV), лентивируса, аденовируса или других типов плазмидных или вирусных векторов, в частности, с применением составов и доз из, например, патентов США №№8454972 (составы, дозы для аденовируса), 8404658 (составы, дозы для AAV) и 5846946 (составы, дозы для плазмидных ДНК) и из клинических испытаний и публикаций относительно клинических испытаний с участием лентивируса, AAV и аденовируса. Например, для AAV путь введения, состав и доза могут быть определены в патенте США №8454972 и в клинических испытаниях с участием AAV. Для аденовируса путь введения, состав и доза могут быть определены в патенте США №8404658 и в клинических испытаниях с участием аденовируса. Для доставки с помощью плазмид путь введения, состав и доза могут быть такими, как определено в патенте США №5846946 и в клинических испытаниях с участием плазмид. Дозы могут быть определены в расчете на или экстраполированы на среднего индивидуума массой 70 кг и могут быть скорректированы для пациентов, субъектов, млекопитающих с другой массой и из другого вида. Частота введения входит в пределы компетенции практикующего врача или ветеринара (например, доктора, ветеринарного врача) и зависит от обычных факторов, в том числе от возраста, пола, общего состояния здоровья, других состояний пациента или субъекта и конкретных рассматриваемых состояния или симптомов.
Вирусные векторы можно инъецировать в представляющую интерес ткань. В случае модификации генома, специфической относительно типа клетки, экспрессия Cas9 может управляться промотором, специфическим относительно типа клетки. Например, при печеночноспецифической экспрессии может использоваться промотор гена альбумина, а при нейрон-специфической экспрессии может использоваться промотор гена синапсина I.
Трансгенные животные и растения
Также представлены трансгенные животные. Предпочтительные примеры включают животных, содержащих Cas9, имея при этом в виду полинуклеотиды, кодирующие Cas9, или белок сам по себе. Предпочтительными являются мыши, крысы и кролики. Для получения трансгенных мышей с конструкциями в соответствии с примерами в данном документе можно инъецировать чистую линейную ДНК в пронуклеус зиготы от псевдобеременной самки, например, самки СВ56. Особей-основателей можно затем идентифицировать, генотипировать и подвергать обратному скрещиванию с мышами СВ57. Конструкции можно затем клонировать и необязательно проверять, например, посредством секвенирования по Сэнгеру. Предусматриваются нокауты, где, например, один или несколько генов в модели подвергают нокауту. Однако, также предусматриваются нокины (в отдельности или в комбинации). Были получены иллюстративные мыши, нокинные по Cas9, и это представлено на примере, однако, нокины Cas9 являются предпочтительными. Для создания нокина Cas9 можно целенаправленно воздействовать теми же конструкциями для конститутивной и условной экспрессии на локус Rosa26, что описаны в данном документе (фиг. 25А-В и 26). Способы из публикаций заявок на патенты США №№20120017290 и 20110265198, закрепленных за Sangamo BioSciences, Inc., относящиеся к целенаправленному воздействию на локус Rosa, можно модифицировать для использования системы CRISPR-Cas по настоящему изобретению. В другом варианте осуществления способы из публикации заявки на патент США №20130236946, закрепленной за Cellectis, относящиеся к целенаправленному воздействию на локус Rosa, можно также модифицировать для использования системы CRISPR-Cas по настоящему изобретению.
Пригодность мышей с условной экспрессией Cas9: Заявители показали на клетках 293, что конструкция для условной экспрессии Cas9 может активироваться при совместной экспрессии с Cre. Заявители также показали, что подвергшиеся корректному целенаправленному воздействию mESC R1 могут иметь активный Cas9, когда Cre экспрессируется. Поскольку за Cas9 следует последовательность расщепления пептида Р2А, а затем EGFP, заявители идентифицировали успешную экспрессию путем наблюдения EGFP. Заявители показали активацию Cas9 в mESC. Эта же идея делает использование мышей с условной экспрессией Cas9 столь полезным. Заявители могут скрещивать свою мышь с условной экспрессией Cas9 с мышью, у которой повсеместно экспрессируется Cre (линия АСТВ-Cre), и могут получать мышь, которая экспрессирует Cas9 в каждой клетке. Для этого будет требоваться только доставка химерной РНК для индукции редактирования генома у мышиных эмбрионов или взрослых мышей. Примечательно то, что если мышь с условной экспрессией Cas9 скрестить с мышью, экспрессирующей Cre под контролем тканеспецифического промотора, Cas9 будет лишь в тканях, в которых также экспрессируется Cre. Этот подход можно применять для редактирования генома только в определенных тканях путем доставки химерной РНК в ту же ткань.
Как указано выше, также представлены трансгенные животные, как и трансгенные растения, в частности, сельскохозяйственные культуры и водоросли. Трансгенные растения могут быть полезными в других путях применения помимо обеспечения модели заболевания. Они могут включать производство пищи или кормопроизводство посредством биосинтеза, например, белков, углеводов, питательных веществ или витаминов на более высоких уровнях, чем будет наблюдаться в обычных условиях у дикого типа. В этом отношении предпочтительными являются трансгенные растения, в особенности зернобобовые и клубнеплоды, и животные, в особенности млекопитающие, такие как крупный рогатый скот (коровы, овцы, козы и свиньи), но также домашняя птица и съедобные насекомые.
Трансгенные водоросли или другие растения, такие как рапс, могут быть особенно применимыми в производстве растительных масел или таких видов биотоплива, как, например, спирты (особенно метанол и этанол). Они могут быть сконструированы для синтеза или сверхсинтеза масла или спиртов на высоких уровнях для применения в масложировой или биотопливной промышленности.
Аденоассоциированный вирус (AAV)
Что касается доставки in vivo, то AAV является преимущественным по сравнению с другими вирусными векторами по двум причинам:
Низкая токсичность (она может быть обусловлена способом очистки, не требующим ультрацентрифугирования клеточных частиц, которые могут активировать иммунный ответ),
Низкая вероятность вызывания инсерционного мутагенеза, поскольку он не интегрируется в геном хозяина.
AAV имеет предел упаковки, составляющий 4,5 или 4,75 т.п.о. Это означает, что все из Cas9, а также промотора и терминатора транскрипции должны вмещаться в один вирусный вектор. Конструкции, размер которых превышает 4,5 или 4,75 т.п.о., будут обуславливать значительное снижение продуцирования вируса. SpCas9 является довольно большим, размер гена самого по себе превышает 4,1 т.п.о., что осложняет его упаковку в AAV. Следовательно, варианты осуществления настоящего изобретения включают использование более коротких гомологов Cas9. Например:


Эти виды, таким образом, в целом являются предпочтительными видами для получения Cas9. Заявители показали данные о доставке и in vivo экспрессии Cas9 в головном мозге мышей.
Предпочтительными являются два способа упаковки молекул нуклеиновых кислот, кодирующих Cas9, например, ДНК, в вирусные векторы для опосредования модификации генома in vivo:
Для обеспечения опосредованного NHEJ нокаута гена:
Один вирусный вектор:
вектор, содержащий две или более кассеты экспрессии:
промотор-молекула нуклеиновой кислоты, кодирующая Cas9-терминатор;
промотор-gRNA1-терминатор;
промотор-gRNA2-терминатор;
промотор-gRNA(N)-терминатор (до предельного размера вектора).
Два вирусных вектора:
вектор 1, содержащий одну кассету экспрессии для управления экспрессией Cas9;
промотор-молекула нуклеиновой кислоты, кодирующая Cas9-терминатор;
вектор 2, содержащий одну или несколько кассет экспрессии для управления экспрессией одной или нескольких направляющих РНК;
промотор-gRNA1-терминатор;
промотор-gRNA(N)-терминатор (до предельного размера вектора).
Для опосредования репарации, направляемой гомологичной рекомбинацией. В дополнение к подходам с одним и двумя вирусными векторами, описанными выше, используют дополнительный вектор для доставки матрицы для репарации, направляемой гомологичной рекомбинацией.
Промотор, используемый для управления экспрессией молекулой нуклеиновой кислоты, кодирующей Cas9, может включать следующее:
ITR AAV может служить в качестве промотора: это является преимущественным для устранения необходимости в дополнительном промоторном элементе (который может занимать пространство в векторе). Освободившееся дополнительное пространство можно задействовать для управления экспрессией дополнительных элементов (gRNA и т.д.). Также активность ITR является относительно более слабой, поэтому ее можно использовать для снижения токсичности, обусловленной сверхэкспрессией Cas9.
Для повсеместной экспрессии можно использовать следующие промоторы: CMV, CAG, CBh, PGK, SV40, генов тяжелой или легкой цепей ферритина и т.д.
Для экспрессии в головном мозге использовать применять следующие промоторы: гена синапсина I для всех нейронов, гена CaMKII-альфа для возбуждающих нейронов, GAD67, или GAD65, или VGAT для GABA-эргических нейронов и т.д.
Для экспрессии в печени можно использовать промотор гена альбумина.
Для экспрессии в легких можно использовать SP-B.
Для эндотелиальных клеток можно использовать ICAM.
Для гемопоэтических клеток можно использовать промотор гена IFN-бета или CD45.
Для остеобластов можно использовать OG-2.
Промотор, используемый для управления направляющей РНК, может включать следующее:
промоторы для Pol III, такие как U6 или H1.
Использование промотора для Pol II и интронных кассет для экспрессии gRNA.
Что касается AAV, AAV может представлять собой AAV1, AAV2, AAV5 или любую их комбинацию. Можно выбрать AAV из AAV с учетом клеток, подлежащих целенаправленному воздействию, например, можно выбрать AAV серотипов 1, 2, 5, или гибрид или капсид AAV1, AAV2, AAV5, или любую их комбинацию для целенаправленного воздействия на головной мозг или нейроны; и можно выбрать AAV4 для целенаправленного воздействия на сердечную ткань. AAV8 применим для доставки в печень. Вышеуказанные промоторы и векторы являются предпочтительными по отдельности.
Доставка РНК также является применимым способом доставки in vivo. На фигуре 27 показаны данные о доставке и in vivo экспрессии Cas9 в мозгу мышей. Возможно доставлять Cas9 и gRNA (и, например, матрицу для HR-репарации) в клетки посредством липосом или наночастиц. Таким образом, доставка фермента CRISPR, такого как Cas9, и/или доставка РНК по настоящему изобретению может осуществляться в форме РНК и посредством микропузырьков, липосом или наночастиц. Например, мРНК Cas9 и gRNA могут быть упакованы в липосомные частицы для доставки in vivo. Реагенты для липосомной трансфекции, такие как Lipofectamine от Life Technologies, и другие реагенты, имеющиеся в продаже, могут эффективно доставлять молекулы РНК в печень.
Повышение эффективности NHEJ или HR также способствует доставке. Предпочтительно, чтобы эффективность NHEJ повышали посредством совместной экспрессии ферментов для обработки концов, таких как Trex2 (Dumitrache et al. Genetics. 2011 August; 188(4): 787-797). Предпочтительно, чтобы эффективность HR повышалась путем транзиентного ингибирования компонентов механизма NHEJ, таких как Ku70 и Ku86. Эффективность HR также можно повысить путем совместной экспрессии прокариотических или эукариотических ферментов гомологичной рекомбинации, таких как RecBCD, RecA.
Различные средства доставки описаны в данном документе и дополнительно обсуждаются в данном разделе.
Доставка с помощью вируса: фермент CRISPR, например, Cas9, и/или любую из РНК по настоящему изобретению, например, направляющую РНК, можно доставлять с помощью аденоассоциированного вируса (AAV), лентивируса, аденовируса или других типов вирусных векторов или их комбинаций. Cas9 и одну или несколько направляющих РНК можно упаковать в один или несколько вирусных векторов. В некоторых вариантах осуществления вирусный вектор доставляют в представляющую интерес ткань посредством, например, внутримышечной инъекции, тогда как в других случаях доставка с помощью вируса осуществляется посредством внутривенного, трансдермального, интраназального, перорального, трансмукозального или других способов доставки. Такая доставка может осуществляться в виде однократной дозы или многократных доз. Специалист в данной области понимает, что фактическая доза, подлежащая доставке в данном документе, может в значительной степени варьировать в зависимости от ряда факторов, таких как выбранный вектор, целевые клетка, организм или ткань, общее состояние субъекта, подлежащего лечению, степень желаемой трансформации/модификации, путь введения, способ введения, тип желаемой трансформации/модификации и т.п.
Такая доза может дополнительно содержать, например, носитель (воду, солевой раствор, этанол, глицерин, лактозу, сахарозу, фосфат кальция, желатин, декстран, агар, пектин, арахисовое масло, кунжутное масло и т.д.), разбавитель, фармацевтически приемлемый носитель (например, фосфатно-солевой буфер), фармацевтически приемлемый наполнитель, адъювант для усиления антигенности, иммуностимулирующие соединение или молекулу и/или другие соединения, известные из уровня техники. Адъювант в данном документе может включать суспензию минеральных веществ (квасцы, гидроксид алюминия, фосфат алюминия), на которых адсорбирован антиген; или эмульсию типа "вода в масле", в которой раствор антигена эмульгирован в масле (MF-59, неполный адъювант Фрейнда), иногда содержит убитые микобактерии (полный адъювант Фрейнда) для дополнительного усиления антигенности (ингибирует распад антигена и/или вызывает миграцию макрофагов). Адъюванты также включают иммуностимулирующие молекулы, такие как цитокины, костимулирующие молекулы и, например, иммуностимулирующие молекулы ДНК или РНК, такие как CpG-олигонуклеотиды. Такой дозированный состав может без труда определить специалист в данной области. Доза может дополнительно содержать одну или несколько фармацевтически приемлемых солей, таких как, например, соль неорганической кислоты, такая как гидрохлорид, гидробромид, фосфат, сульфат и т.д.; и соли органических кислот, такие как ацетаты, пропионаты, малонаты, бензоаты и т.д. Дополнительно, в данном документе также могут присутствовать вспомогательные вещества, такие как смачиватели или эмульгаторы, буферные вещества, поддерживающие рН, гели или гелеобразующие материалы, ароматизаторы, красители, микросферы, полимеры, суспендирующие средства и т.д. В дополнение, также могут присутствовать один или несколько других традиционных фармацевтических ингредиентов, таких как консерванты, увлажнители, суспендирующие средства, поверхностно-активные вещества, антиоксиданты, средства против слеживания, заполнители, хелатообразователи, покрывающие средства, химические стабилизаторы и т.д., особенно если лекарственная форма представляет собой растворимую форму. Пригодные иллюстративные ингредиенты включают микрокристаллическую целлюлозу, натрий-карбоксиметилцеллюлозу, полисорбат 80, фенилэтиловый спирт, хлорбутанол, сорбат калия, сорбиновую кислоту, диоксид серы, пропилгаллат, парабены, этилванилин, глицерин, фенол, парахлорфенол, желатин, альбумин и их комбинацию. Подробное обсуждение фармацевтически приемлемых наполнителей доступно в REMINGTON'S PHARMACEUTICAL SCIENCES (Mack Pub. Co., N.J. 1991), включенном в данный документ посредством ссылки.
В варианте осуществления в данном документе доставку осуществляют посредством аденовируса, который может находиться в однократной бустерной дозе, содержащей по меньшей мере 1×105 частиц (также называемых единичными частицами, pu) аденовирусного вектора. В варианте осуществления в данном документе доза предпочтительно составляет по меньшей мере приблизительно 1×106 частиц (например, приблизительно 1×106-1×1012 частиц), более предпочтительно по меньшей мере приблизительно 1×107 частиц, более предпочтительно по меньшей мере приблизительно 1×108 частиц (например, приблизительно 1×108-1×1011 частиц или приблизительно 1×108-1×1012 частиц), и наиболее предпочтительно по меньшей мере приблизительно 1×100 частиц (например, приблизительно 1×109-1×1010 частиц или приблизительно 1×109-1×1012 частиц) или даже по меньшей мере приблизительно 1×1010 частиц (например, приблизительно 1×1010-1×1012 частиц) аденовирусного вектора. Альтернативно, доза содержит не более приблизительно 1×1014 частиц, предпочтительно не более приблизительно 1×1013 частиц, еще более предпочтительно не более приблизительно 1×1012 частиц, еще более предпочтительно не более приблизительно 1×1011 частиц и наиболее предпочтительно не более приблизительно 1×1010 частиц (например, не более приблизительно 1×109 частиц). Таким образом, доза может включать в себя однократную дозу аденовирусного вектора с, например, приблизительно 1×106 единичных частиц (pu), приблизительно 2×106 pu, приблизительно 4×106 pu, приблизительно 1×107 pu, приблизительно 2×107 pu, приблизительно 4×107 pu, приблизительно 1×108 pu, приблизительно 2×108 pu, приблизительно 4×108 pu, приблизительно 1×109 pu, приблизительно 2×109 pu, приблизительно 4×109 pu, приблизительно 1×1010 pu, приблизительно 2×1010 pu, приблизительно 4×1010 pu, приблизительно 1×1011 pu, приблизительно 2×1011 pu, приблизительно 4×1011 pu, приблизительно 1×1012 pu, приблизительно 2×1012 pu или приблизительно 4×1012 pu аденовирусного вектора. См., например, аденовирусные векторы в патенте США №8454972 В2 Nabel, et. al., выданном 4 июня 2013 г.; включенном в данный документ посредством ссылки, и дозы в столбце 29, строках 36-58 данного патента. В варианте осуществления в данном документе аденовирус доставляют посредством многократных доз.
В варианте осуществления в данном документе доставку осуществляют посредством AAV. Полагают, что терапевтически эффективная доза для in vivo доставки AAV человеку находится в диапазоне от приблизительно 20 до приблизительно 50 мл физиологического раствора, содержащего от приблизительно 1×1010 до приблизительно 1×1010 функциональных частиц AAV/мл раствора. Дозу можно скорректировать для уравновешивания терапевтической пользы и любых побочных эффектов. В варианте осуществления в данном документе доза AAV, как правило, находится в диапазоне концентраций от приблизительно 1×105 до 1×1050 геномов AAV, от приблизительно 1×108 до 1×1020 геномов AAV, от приблизительно 1×1010 до приблизительно 1×1016 геномов или от приблизительно 1×1011 до приблизительно 1×1016 геномов AAV. Доза для человека может составлять приблизительно 1×1013 геномов AAV. Такие концентрации можно доставлять в от приблизительно 0,001 мл до приблизительно 100 мл, от приблизительно 0,05 до приблизительно 50 мл или от приблизительно 10 до приблизительно 25 мл раствора носителя. Другие эффективные дозы может без труда установить один из средних специалистов в данной области посредством стандартных испытаний с построением кривых зависимости "доза-эффект". См., например, патент США №8404658 В2 Hajjar, et al., выданный 26 марта 2013 г., в столбце 27, строках 45-60.
В варианте осуществления в данном документе доставку осуществляют посредством плазмиды. В таких плазмидных композициях доза должна быть количеством плазмид, достаточным для того, чтобы вызвать эффект. Например, подходящее количество плазмидной ДНК в плазмидных композициях может составлять от приблизительно 0,1 до приблизительно 2 мг или от приблизительно 1 мкг до приблизительно 10 мкг.
Дозы в данном документе определяются в расчете на среднего индивидуума массой 70 кг. Частота введения находится в пределах компетенции практикующего врача или ветеринара (например, доктора, ветеринарного врача) или ученого, являющегося специалистом в данной области.
Лентивирус
Лентивирусы являются сложными ретровирусами, которые обладают способностью инфицировать как митотические, так и постмитотические клетки и экспрессировать в них свои гены. Наиболее широко известным лентивирусом является вирус иммунодефицита человека (HIV), который использует гликопротеины оболочки других вирусов для целенаправленного воздействия на широкий спектр типов клеток.
Лентивирусы можно получить следующим образом. После клонирования pCasES10 (которая содержит остов лентивирусной плазмиды-переносчика), HEK293FT прошедшие малое количество пассажей (р=5), высевали в колбу Т-75 до 50% конфлюентности за день до трансфекции в DMEM с 10% фетальной бычьей сывороткой и без антибиотиков. Через 20 часов среду заменяли на среду OptiMEM (бессывороточную), и 4 часа спустя проводили трансфекцию. Клетки трансфицировали с помощью 10 мкг лентивирусной плазмиды-переносчика (pCasES10) и следующих пакующих плазмид: 5 мкг pMD2.G (псевдотип VSV-g) и 7,5 мкг psPAX2 (gag/pol/rev/tat). Трансфекцию проводили в 4 мл OptiMEM со средством доставки на основе катионного липида (50 мкл Lipofectamine 2000 и 100 мкл реагента Plus). Через 6 часов среду заменяли на DMEM, не содержащую антибиотиков, с 10% фетальной бычьей сывороткой.
Лентивирус можно очистить следующим образом. Вируссодержащие надосадочные жидкости отбирали через 48 часов. Надосадочные жидкости сперва очищали от дебриса и фильтровали через 0,45 мкм фильтр с низкой степенью связывания белка (PVDF). Затем их центрифугировали на ультрацентрифуге в течение 2 часов при 24000 об./мин. Вируссодержащие осадки ресуспендировали в 50 мкл DMEM в течение ночи при 4°С. Затем их разделяли на аликвоты и сразу же замораживали при -80°С.
В другом варианте осуществления также предусмотрены минимальные лентивирусные векторы для отличных от приматов организмов на основе вируса инфекционной анемии лошадей (EIAV), особенно для генной терапии глаз (см., например, Balagaan, J Gene Med 2006; 8: 275-285, опубликовано в Интернете 21 ноября 2005 г. в Wiley InterScience (www.interscience.wiley.com). DOI: 10.1002/jgm.845). В другом варианте осуществления также предусмотрен RetinoStat®, лентивирусный вектор на основе вируса инфекционной анемии лошадей для генной терапии, экспрессирующий ангиостатические белки эндостатин и ангиостатин, который доставляют посредством субретинальной инъекции для лечения влажной формы возрастной макулодистрофии (см., например, Binley et al., HUMAN GENE THERAPY 23: 980-991 (September 2012)), который может быть модифицирован для системы CRISPR-Cas по настоящему изобретению.
В другом варианте осуществления самоинактивирующиеся лентивирусные векторы с siRNA, целенаправленно воздействующими на общий экзон, который имеют tat/rev HTV, сигналом ядрышковой локализации TAR-ловушкой и специфичным к CCR5 рибозимом в виде головки молотка (см., например, DiGiusto et al. (2010) Sci Transl Med 2: 36ra43), можно использовать и/или приспосабливать к системам CRISPR-Cas по настоящему изобретению. Не менее 2,5×106 CD34+ клеток на килограмм массы пациента можно собирать и предварительно стимулировать в течение 16-20 часов в среде X-VIVO 15 (Lonza), содержащей 2 мМ L-глутамин, фактор стволовых клеток (100 нг/мл), лиганд Flt-3 (Flt-3L) (100 нг/мл) и тромбопоэтин (10 нг/мл) (CellGenix) при плотности 2×106 клеток/мл. Предварительно стимулированные клетки можно трансдуцировать лентивирусом при множественности заражения 5 в течение 16-24 часов во флаконах с культурой тканей на 75 см2, покрытых фибронектином (25 мг/см2) (RetroNectin, Takara Bio Inc.).
Лентивирусные векторы были раскрыты в отношении лечения болезни Паркинсона, см., например, публикацию заявки на патент США №20120295960 и патенты США №№7303910 и 7351585. Лентивирусные векторы также были раскрыты в отношении лечения заболеваний глаз, см., например, публикации заявок на патенты США №№20060281180, 20090007284, US 20110117189; US 20090017543; US 20070054961, US 20100317109. Лентивирусные векторы также были раскрыты в отношении доставки в головной мозг, см., например, публикации заявок на патенты США №№ US 20110293571; US 20110293571, US 20040013648, US 20070025970, US 20090111106 и патент США № US 7259015.
Доставка РНК
Доставка РНК: фермент CRISPR, например, Cas9, и/или любую из РНК по настоящему изобретению, например, направляющую РНК, также можно доставлять в форме РНК. мРНК Cas9 можно получить с помощью in vitro транскрипции. Например, мРНК Cas9 можно синтезировать с помощью кассеты для ПЦР, содержащей следующие элементы: промотор Т7_последовательность Козак (GCCACC)-Cas9-3'-UTR гена бета-глобина-поли(А)-хвост (цепь из 120 или более адениновых остатков). Кассету можно использовать для транскрипции под действием полимеразы Т7. Направляющие РНК также можно транскрибировать с помощью in vitro транскрипции с кассеты, содержащей промотор Т7_GG-последовательность направляющей РНК.
Для повышения экспрессии и снижения токсичности фермент CRISPR и/или направляющую РНК можно модифицировать с помощью псевдо-U или 5-метил-С.
Способы доставки мРНК в настоящее время являются особенно перспективными для доставки в печень. В частности, для AAV8 особенно предпочтительной является доставка в печень.
Наночастицы
mРНК фермента CRISPR и направляющую РНК можно доставлять одновременно с помощью наночастиц или липидных оболочек.
Например, у Su X, Fricke J, Kavanagh DG, Irvine DJ ("In vitro and in vivo mRNA delivery using lipid-enveloped pH-responsive polymer nanopardcles" Mol Pharm. 2011 Jun 6; 8(3): 774-87. doi: 10.1021/mp100390w. Epub 2011 Apr 1) раскрываются биоразлагаемые наночастицы со структурой ядро/оболочка с ядром из сложного поли(β-аминоэфира) (РВАЕ), окруженным фосфолипидной двуслойной оболочкой. Они были разработаны для in vivo доставки мРНК. Чувствительный к рН компонент РВАЕ был выбран для содействия разрушению эндосом, тогда как поверхностный липидный слой был выбран для сведения к минимуму токсичности поликатионного ядра. Они, таким образом, являются предпочтительными для доставки РНК по настоящему изобретению.
В одном варианте осуществления предусмотрены наночастицы на основе самособирающихся биоадгезивных полимеров со свойством самосборки, которые можно использовать для пероральной доставки пептидов, внутривенной доставки пептидов и интраназальной доставки пептидов, во всех случаях в головной мозг. Также предусмотрены другие варианты осуществления, такие как абсорбция при пероральном применении и внутриглазная доставка гидрофобных лекарственных средств. Технология молекулярных оболочек предусматривает сконструированную полимерную оболочку, защищающую и доставляющую в очаг заболевания (см., например, Mazza, M. et al. ACSNano, 2013. 7(2): 1016-1026; Siew, A., et al. Mol Pharm, 2012. 9(1): 14-28; Lalatsa, A., et al. J Contr Rel, 2012. 161(2): 523-36; Lalatsa, A., et al., Mol Pharm, 2012. 9(6): 1665-80; Lalatsa, A., et al. Mol Pharm, 2012. 9(6): 1764-74; Garrett, N.L., et al. J Biophotonics, 2012. 5(5-6): 458-68; Garrett, N.L., et al. J Raman Spect, 2012. 43(5): 681-688; Ahmad, S., et al. J Royal Soc Interface 2010. 7: S423-33; Uchegbu, I.F. Expert Opin Drug Deliv, 2006. 3(5): 629-40; Qu, X., et al. Biomacromolecules, 2006. 7(12): 3452-9 и Uchegbu, I.F., et al. Int J Pharm, 2001. 224: 185-199). Предусмотрены дозы, составляющие приблизительно 5 мг/кг, которые в зависимости от целевой ткани будут однократными или многократными дозами.
В одном варианте осуществления наночастицы, которые могут доставлять РНК в раковые клетки для прекращения роста опухолей, разработанные в лаборатории Дэна Андерсона в MIT, можно использовать для систем CRISPR-Cas по настоящему изобретению и/или приспосабливать к ним. В частности, в лаборатории Андерсона были разработаны полностью автоматизированные, комбинаторные системы для синтеза, очистки, исследования и составления новых биоматериалов и наносоставов. См., например, Alabi et al., Proc Natl Acad Sci USA. 2013 Aug 6; 110(32): 12881-6; Zhang et al., Adv Mater. 2013 Sep 6; 25(33): 4641-5; Jiang et al., Nano Lett. 2013 Mar 13; 13(3): 1059-64; Karagiannis et al., ACS Nano. 2012 Oct 23; 6(10): 8484-7; Whitehead et al., ACS Nano. 2012 Aug 28; 6(8): 6922-9 и Lee et al., Nat Nanotechnol. 2012 Jun 3; 7(6): 389-93.
Заявка на патент США 20110293703 относится к липидоподобным соединениям, также являющимся особенно применимыми при введении полинуклеотидов, которые можно использовать для доставки системы CRISPR-Cas по настоящему изобретению. В одном аспекте аминоспиртовые липидоподобные соединения объединяют со средством, подлежащим введению в клетку или субъекту, с образованием микрочастиц, наночастиц, липосом или мицелл. Средство, подлежащее доставке с помощью частиц, липосом или мицелл, может быть в форме газа, жидкости или твердого вещества, и средство может представлять собой полинуклеотид, белок, пептид или малую молекулу. Аминоспиртовые липидоподобные соединения можно объединять с другими аминоспиртовыми липидоподобными соединениями, полимерами (синтетическими или природными), поверхностно-активными веществами, холестерином, углеводами, белками, липидами и т.д. с образованием частиц. Эти частицы можно затем необязательно объединять с фармацевтическим наполнителем с образованием фармацевтической композиции.
В публикации заявки на патент США №0110293703 также представлены способы получения аминоспиртовых липидоподобных соединений. Одному или нескольким эквивалентам амина позволяют вступать в реакцию с одним или несколькими эквивалентами соединения с концевыми эпоксидными группами в подходящих условиях с образованием аминоспиртового липидоподобного соединения по настоящему изобретению. В определенных вариантах осуществления все аминогруппы амина полностью реагируют с соединением с концевыми эпоксидными группами с образованием третичных аминов. В других вариантах осуществления не все аминогруппы амина полностью вступают в реакцию с соединением с концевыми эпоксидными группами с образованием третичных аминов, в результате чего, таким образом, образуются первичные или вторичные аминогруппы аминоспиртового липидоподобного соединения. Эти первичные или вторичные аминогруппы оставляют в существующем состоянии или могут вводить в реакцию с другим электрофилом, таким как другое соединение с концевыми эпоксидными группами. Специалисту в данной области следует принять во внимание, что введение амина в реакцию с меньшим, чем избыточное, количеством соединения с концевыми эпоксидными группами приведет к получению множества различных аминоспиртовых липидоподобных соединений с различным количеством "хвостов". Определенные амины могут быть полностью функционализированными с помощью двух "хвостов" соединений, полученных из эпоксидов, тогда как другие молекулы могут быть не полностью функционализированными с помощью "хвостов" соединений, полученных из эпоксидов. Например, диамин или полиамин может содержать один, два, три или четыре "хвоста" соединений, полученных из эпоксидов, у различных аминофрагментов молекулы, в результате чего образуются первичные, вторичные и третичные аминогруппы. В определенных вариантах осуществления все аминогруппы являются не полностью функционализированными. В определенных вариантах осуществления используют два соединения с концевыми эпоксидными группами одного типа. В других вариантах осуществления используют два или более различных соединения с концевыми эпоксидными группами. Синтез аминоспиртовых липидоподобных соединений осуществляют с помощью или без растворителя, и синтез можно осуществлять при более высоких температурах, варьирующих в диапазоне от 30 до 100°С, предпочтительно при примерно 50-90°С. Получаемые аминоспиртовые липидоподобные соединения можно необязательно очищать. Например, смесь аминоспиртовых липидоподобных соединений можно очищать с получением аминоспиртового липидоподобного соединения с определенным количеством "хвостов" соединений, полученных из эпоксидов. Или же смесь можно очищать с получением определенного стерео- или региоизомера. Аминоспиртовые липидоподобные соединения можно также алкилировать с помощью алкилгалогенида (например, йодистого метила) или другого алкилирующего средства, и/или их можно ацилировать.
В публикации заявки на патент США №0110293703 также представлены библиотеки аминоспиртовых липидоподобных соединений, полученных согласно способам по настоящему изобретению. Эти аминоспиртовые липидоподобные соединения можно получать и/или подвергать скринингу с применением высокопроизводительных методик, предусматривающих использование дозаторов жидкостей, роботов, планшетов для микротитрования, компьютеров и т.д. В определенных вариантах осуществления аминоспиртовые липидоподобные соединения подвергают скринингу в отношении их способности к трансфекции полинуклеотидов или других средств (например, белков, пептидов, малых молекул) в клетку.
Публикация заявки на патент США №20130302401 относится к классу поли(бета-аминоспиртов) (РВАА), получаемых при помощи комбинаторных методик полимеризации. РВАА по настоящему изобретению можно применять в биотехнологии и медико-биологических путях применения в качестве покрытий (таких как пленочные покрытия или многослойные пленки для медицинских инструментов или имплантатов), добавок, материалов, наполнителей, средств против биологического обрастания, средств для формирования микроструктуры и средств для инкапсулирования клеток. В случае применения в качестве поверхностных покрытий эти РВАА вызывают различные уровни воспаления как in vitro, так и in vivo в зависимости от их химических структур. Большое химическое разнообразие этого класса материалов позволяет идентифицировать полимерные покрытия, ингибирующие активацию макрофагов in vitro. Кроме того, эти покрытия уменьшают мобилизацию воспалительных клеток и уменьшают выраженность фиброза после подкожной имплантации микрочастиц карбоксилированного полистирола. Эти полимеры можно использовать для образования капсул на основе полиэлектролитных комплексов для инкапсулирования клеток. Настоящее изобретение также может иметь много других путей применения в биологии, таких как получение противомикробных покрытий, доставка ДНК или siRNA и тканевая инженерия с применением стволовых клеток. Идеи, изложенные в публикации заявки на патент США №20130302401, можно применять к системе CRISPR-Cas по настоящему изобретению.
В другом варианте осуществления предусмотрены липидные наночастицы (LNP). В частности, малые интерферирующие РНК, воздействующие на транстиретин, инкапсулированные в липидных наночастицах (см., например, Coelho et al., N Engl J Med 2013; 369: 819-29), можно применять в отношении системы CRISPR-Cas по настоящему изобретению. Предусмотрены дозы, составляющие от приблизительно 0,01 до приблизительно 1 мг на кг массы тела, вводимые внутривенно. Предусмотрены лекарственные препараты для снижения риска возникновения инфузионных реакций, такие как дексаметазон, ацетаминофен, дифенгидрамин или цетиризин и ранитидин. Также предусмотрены многократные дозы, состоящие из пяти доз по приблизительно 0,3 мг на килограмм, принимаемых каждые 4 недели.
Было показано, что LNP являются высокоэффективными в доставке siRNA в печень (см., например, Tabernero et al., Cancer Discovery, April 2013, Vol. 3, No. 4, pages 363-470) и, таким образом, предусмотрены для доставки CRISPR-Cas в печень. Может быть предусмотрен режим дозирования с приемом приблизительно четырех доз по 6 мг/кг LNP каждые две недели. Tabernero и соавт. продемонстрировали, что после первых 2 циклов дозирования LNP при 0,7 мг/кг наблюдалась регрессия опухоли, а к концу 6 циклов у пациента достигался частичный ответ с полной регрессией метастазов в лимфатических узлах и значительным уменьшением размеров опухолей в печени. У данного пациента, у которого сохранялась ремиссия и который завершил лечение после получения доз в течение 26 месяцев, полный ответ достигался после приема 40 доз. У двух пациентов с RCC и внепеченочными очагами заболевания, включающими почку, легкое и лимфатические узлы, в которых наблюдалось прогрессирование после предшествующей терапии ингибиторами сигнального пути VEGF, наблюдалось стабильное заболевание во всех очагах в течение примерно 8-12 месяцев, а пациент с PNET и метастазами в печени продолжал участие в расширенном исследовании в течение 18 месяцев (36 доз) при стабильном заболевании.
Однако, следует принимать во внимание заряд LNP. Так, объединение катионных липидов с отрицательно заряженными липидами индуцирует образование структур, не являющихся двуслойными, которые облегчают внутриклеточную доставку. Поскольку заряженные LNP быстро выводятся из кровотока после внутривенной инъекции, были разработаны ионизируемые катионные липиды со значениями pKa ниже 7 (см., например, Rosin et al. Molecular Therapy, vol. 19, no. 12, pages 1286-2200, Dec. 2011). Отрицательно заряженные полимеры, такие как siRNA-олигонуклеотиды, можно загружать в LNP при низких значениях рН (например, рН 4), где ионизируемые липиды проявляют положительный заряд. Однако, при физиологических значениях рН LNP проявляют низкий поверхностный заряд, совместимый с большими значениями времени пребывания в кровотоке. Основное внимание было сосредоточено на четырех молекулах ионизируемых катионных липидов, а именно 1,2-дилинолеоил-3-диметиламмонийпропан (DLinDAP), 1,2-дилинолеилокси-3-N,N-диметиламинопропан (DLinDMA), 1,2-дилинолеилоксикето-N,N-диметил-3-аминопропан (DLinKDMA) и 1,2-дилинолеил-4-(2-диметиламиноэтил)-[1,3]-диоксолан (DLinKC2-DMA). Было показано, что системы LNP с siRNA, содержащие эти липиды, проявляют существенно отличающиеся свойства сайленсинга генов в гепатоцитах in vivo, при этом их активность изменяется в ряду DLinKC2-DMA>DLinKDMA>DLinDMA>>DLinDAP, при использовании модели сайленсинга гена фактора VII (см., например. Rosin et al. Molecular Therapy, vol. 19, no. 12, pages 1286-2200, Dec. 2011). Могут быть предусмотрены уровни дозы 1 мкг/мл, особенно для состава, содержащего DLinKC2-DMA.
Получение LNP и инкапсулирование CRISPR-Cas можно применять и/или приспосабливать согласно Rosin et al. Molecular Therapy, vol. 19, no. 12, pages 1286-2200, Dec. 2011. Катионные липиды 1,2-дилинолеоил-3-диметиламмонийпропан (DLinDAP), 1,2-дилинолеилокси-3-N,N-диметиламинопропан (DLinDMA), 1,2-дилинолеилоксикето-N,N-диметил-3-аминопропан (DLinK-DMA), 1,2-дилинолеил-4-(2-диметиламиноэтил)-[1,3]-диоксолан (DLinKC2-DMA), (3-о-[2''-(метоксиполиэтиленгликоль 2000)-сукциноил]-1,2-димиристоил-sn-гликоль (PEG-S-DMG) и R-3-[((ω-метоксиполи(этиленгликоль)2000)-карбамоил]-1,2-димиристилоксипропил-3-амин (PEG-C-DOMG) могут быть предоставлены Tekmira Pharmaceuticals (Ванкувер, Канада) или синтезированы. Холестерин можно приобрести у Sigma (Сент-Луис, Миссури). Конкретную РНК CRISPR-Cas можно инкапсулировать в LNP, содержащую DLinDAP, DLinDMA, DLinK-DMA и DLinKC2-DMA (катионный липид : DSPC : холестерин : PEG-S-DMG или PEG-C-DOMG в молярном соотношении 40:10:40:10). При необходимости можно включать в состав 0,2% SP-DiOC18 (Invitrogen, Берлингтон, Канада) для определения клеточного поглощения, внутриклеточной доставки и биораспределения. Инкапсулирование можно осуществлять путем растворения липидных смесей, содержащих катионный липид : DSPC : холестерин : PEG-C-DOMG (молярное соотношение 40:10:40:10) в этаноле до конечной концентрации липидов 10 ммоль/л. Этот раствор липидов в этаноле можно добавлять по каплям к 50 ммоль/л цитрата, рН 4,0, с образованием многослойных пузырьков до получения конечной концентрации этанола 30% об./об. Крупные однослойные пузырьки можно формировать после экструзии многослойных пузырьков через два установленных один над другим поликарбонатных фильтра Nuclepore на 80 нм при помощи экструдера (Northern Lipids, Ванкувер, Канада). Инкапсулирование можно осуществлять путем добавления РНК, растворенной при 2 мг/мл в 50 ммоль/л цитрата, рН 4,0, содержащего 30% этанол об./об., по каплям к экструдированным предварительно сформированным крупным однослойным пузырькам и инкубирования при 31°С в течение 30 минут при постоянном перемешивании до конечного весового соотношения РНК/липид 0,06/1 вес./вес. Удаление этанола и нейтрализацию буфера для получения состава проводили путем диализа против фосфатно-солевого буфера (PBS), рН 7,4, в течение 16 часов при помощи диализных мембран Spectra/Por 2 из регенерированной целлюлозы. Распределение наночастиц по размеру можно определить посредством динамического рассеяния света с использованием измерителя размера частиц NICOMP 370, режимов объема пузырьков/интенсивности рассеянного света и аппроксимации функцией Гаусса (Nicomp Particle Sizing, Санта-Барбара, Калифорния). Размер частиц для всех трех систем LNP может составлять ~70 нм в диаметре. Эффективность инкапсулирования siRNA можно определить путем удаления свободной siRNA из образцов, отобранных до или после диализа, с помощью колонок VivaPureD MiniH (Sartorius Stedim Biotech). Инкапсулированную РНК можно экстрагировать из элюированных наночастиц и подвергать количественной оценке при 260 нм. Соотношение siRNA и липидов определяли путем измерения содержания холестерина в пузырьках с помощью ферментативного анализа Cholesterol E от Wako Chemicals USA (Ричмонд, Виргиния).
Получение крупных LNP можно применять и/или адаптировать согласно Rosin et al. Molecular Therapy, vol. 19, no. 12, pages 1286-2200, Dec. 2011. Раствор предварительно приготовленной смеси липидов (общая концентрация липидов 20,4 мг/мл) можно получать в этаноле, содержащем DLinKC2-DMA, DSPC и холестерин в молярном соотношении 50:10:38,5. К предварительно приготовленной смеси липидов можно добавлять ацетат натрия в молярном соотношении 0,75:1 (ацетат натрия : DLinKC2-DMA). Липиды затем можно гидрировать путем объединения смеси с 1,85 объема нитратного буфера (10 ммоль/л, рН 3,0) при энергичном перемешивании, вызывая самопроизвольное образование липосом в водном буфере, содержащем 35% этанол. Раствор липосом можно инкубировать при 37°С для обеспечения зависимого от времени увеличения размера частиц. Можно отбирать аликвоты в различные моменты времени в ходе инкубирования для изучения изменений размера липосом посредством динамического рассеяния света (Zetasizer Nano ZS, Malvern Instruments, Вустершир, Великобритания). По достижении желаемого размера частиц к смеси липосом можно добавлять водный раствор конъюгатов PEG-липид (исходный раствор = 10 мг/мл PEG-DMG в 35% (об./об.) этаноле) с получением конечной молярной концентрации PEG 3,5% от общего количества липидов. После добавления конъюгатов PEG-липид липосомы должны свой размер с эффективным подавлением дальнейшего роста. К "пустым" липосомам можно затем добавить РНК при соотношении siRNA и общих липидов, составляющем примерно 1:10 (вес : вес), с последующим инкубированием в течение 30 минут при 37°C с образованием нагруженных LNP. Смесь можно затем подвергнуть диализу в течение ночи в PBS и отфильтровать через шприцевой фильтр с диаметром пор 0,45 мкм.
Конструкции сферических нуклеиновых кислот (SNA™) и другие наночастицы (в частности, наночастицы золота) также предусмотрены в качестве средств доставки системы CRISPR/Cas к предполагаемым мишеням. Существенные данные показывают, что конструкции сферических нуклеиновых кислот (SNA™) AuraSense Therapeutics на основе наночастиц золота, функционализированных нуклеиновыми кислотами, превосходят альтернативные платформы руководствуясь несколькими следующими ключевыми факторами успеха:
Высокая стабильность in vivo. По причине их плотной загрузки большинство молекул-карго (ДНК или siRNA) остаются связанными с конструкциями внутри клеток, что придает нуклеиновым кислотам стабильность и устойчивость к ферментативному расщеплению.
Возможность доставки. Для всех изучаемых типов клеток (например, нейронов, линий опухолевых клеток и т.д.) конструкции демонстрируют 99% эффективность трансфекции без необходимости в носителях или средствах для трансфекции.
Терапевтическое целенаправленное воздействие. Уникальная аффинность связывания с мишенью и специфичность конструкций обеспечивают превосходную специфичность в отношении совпадающих целевых последовательностей (т.е. с ограниченными нецелевыми эффектами).
Превосходящая эффективность. Конструкции значительно превосходят ведущие традиционные реагенты для трансфекции (Lipofectamine 2000 и Cytofectin).
Низкая токсичность. Конструкции могут поступать в ряд культивируемых клеток, первичных клеток и тканей без видимой токсичности.
Отсутствие значительного иммунного ответа. Конструкции вызывают минимальные изменения глобальной экспрессии генов согласно измерениям в полногеномных микроматричных исследованиях и специфичных в отношении цитокинов анализах белков.
Способность получения заданных химических свойств. Для получения заданных свойств поверхности конструкций можно использовать любое количество отдельных или комбинированных средств (например, белков, пептидов, малых молекул).
Данная платформа для терапевтических средств на основе нуклеиновых кислот может быть применима к многочисленным болезненным состояниям, включающим воспаление и инфекционное заболевание, рак, кожные нарушения и сердечно-сосудистое заболевание.
Литературные источники, на которые можно ссылаться, включают: Cutler et al., J. Am. Chem. Soc. 2011 133: 9254-9257, Hao et al., Small. 2011 7: 3158-3162, Zhang et al., ACS Nano. 2011 5: 6962-6970, Cutler et al., J. Am. Chem. Soc. 2012 134: 1376-1391, Young et al., Nano Lett. 2012 12: 3867-71, Zheng et al., Proc. Natl. Acad. Sci. USA. 2012 109: 11975-80, Mirkin, Nanomedicine 2012 7: 635-638, Zhang et al., J. Am. Chem. Soc. 2012 134: 16488-1691, Weintraub, Nature 2013 495: S14-S16, Choi et al., Proc. Natl. Acad. Sci. USA. 2013 110(19): 7625-7630, Jensen et al., Sci. Transl. Med. 5, 209ra152 (2013) и Mirkin, et al., Small, doi.org/10.1002/smll.201302143.
Самособирающиеся наночастицы с siRNA могут быть сконструированы с помощью полиэтиленимина (PEI), пегилированного пептидным лигандом Arg-Gly-Asp (RGD), присоединенным к дистальному концу полиэтиленгликоля (PEG), например, в качестве средства для нацеливания на новообразованные сосуды опухоли, в которых экспрессируются интегрины, и использоваться для доставки siRNA, ингибирующих экспрессию рецептора фактора роста эндотелия сосудов-2 (VEGF R2) и, таким образом, ангиогенеза опухоли (см., например, Schiffelers et al., Nucleic Acids Research, 2004, Vol. 32, No. 19). Наноплексы можно получать путем смешивания равных объемов водных растворов катионного полимера и нуклеиновой кислоты с получением чистого молярного избытка ионизируемого азота (полимера) относительно фосфата (нуклеиновой кислоты) в диапазоне от 2 до 6. Электростатические взаимодействия между катионными полимерами и нуклеиновой кислотой приводили к образованию подиндексов, характеризующихся распределением частиц по размеру со средним размером, составляющим приблизительно 100 нм, называемых здесь, таким образом, наноплексами. Для доставки в самособирающихся наночастицах согласно Schiffelers и соавт. предполагается доза, составляющая приблизительно от 100 до 200 мг CRISPR-Cas.
Наноплексы по Bardett et al. (PNAS, September 25, 2007, vol. 104, no. 39) также можно применять в настоящем изобретении. Наноплексы по Bartlett и соавт. получают путем смешивания равных объемов водных растворов катионного полимера и нуклеиновой кислоты с получением чистого молярного избытка ионизируемого азота (полимера) относительно фосфата (нуклеиновой кислоты) в диапазоне от 2 до 6. Электростатические взаимодействия между катионными полимерами и нуклеиновой кислотой приводили к образованию полиплексов, характеризующихся распределением частиц по размеру со средним размером, составляющим приблизительно 100 нм, называемых здесь, таким образом, наноплексами. Конъюгаты DOTA-siRNA no Bartlett et al. синтезировали следующим образом: сложный моно(N-гидроксисукцинимидный эфир) 1,4,7,10-тетраазациклододекан-1,4,7,10-тетрауксусной кислоты (сложный эфир DOTA-NHS) заказывали у Macrocyclics (Даллас, Техас). В микроцентрифужную пробирку добавляли аминомодифицированную смысловую нить РНК со 100-кратным молярным избытком сложного эфира DOTA-NHS в карбонатном буфере (рН 9). Содержимое вводили в реакцию путем перемешивания в течение 4 ч. при комнатной температуре. Конъюгат DOTA-смысловая нить РНК осаждали этанолом, ресуспендировали в воде и отжигали с немодифицированной антисмысловой нитью с получением конъюгата DOTA-siRNA. Все жидкости предварительно обрабатывали с помощью Chelex-100 (Bio-Rad, Геркулес, Калифорния) для удаления следовых количеств металлических примесей. Нацеленные на Tf или ненацеленные наночастицы с siRNA можно получать с помощью поликатионов, содержащих циклодекстрин. Как правило, наночастицы получают в воде при соотношении зарядов 3 (+/-) и концентрации siRNA 0,5 г/литр. Один процент молекул конъюгатов адамантан-PEG на поверхности нацеленных наночастиц модифицировали с помощью Tf (адамантан-PEG-Tf). Наночастицы суспендировали в 5% (вес./об.) глюкозе в качестве раствора-носителя для инъекции.
Davis et al. (Nature, Vol 464, 15 April 2010) проводят клиническое испытание с siRNA, в котором используют систему доставки на основе нацеленных наночастиц (регистрационный номер клинического испытания NCT00689065). Пациентам с солидными формами рака, трудно поддающимися стандартным методикам лечения, вводили дозы нацеленных наночастиц в дни 1, 3, 8 и 10 21-дневного цикла посредством 30-минутной внутривенной инфузии. Наночастицы состоят из синтетической системы доставки, содержащей: (1) линейный полимер на основе циклодекстрина (CDP), (2) лиганд, нацеливающийся на белок трансферрин человека (TF), представленный на внешней поверхности наночастиц, который входит в контакт с рецепторами TF (TFR) на поверхности раковых клеток, (3) гидрофильный полимер (полиэтиленгликоль (PEG), используемый для обеспечения стабильности наночастиц в биологических жидкостях), и (4) siRNA, предназначенную для снижения экспрессии RRM2 (последовательность, применяемая в клинической практике, ранее была обозначена как siR2B+5). Давно известно, что в злокачественных клетках повышена экспрессия TFR, a RRM2 является общепризнанной мишенью для противораковой терапии. Было показано, что эти наночастицы (клинический вариант обозначен как CALAA-01) хорошо переносятся в исследованиях с использованием многократных доз у отличных от человека приматов. Даже при том, что отдельным пациентам с хроническим миелоидным лейкозом вводили siRNA посредством доставки с помощью липосом, клиническое испытание по Davis и соавт. является первым испытанием с участием человека, в котором проводят системную доставку siRNA с помощью системы нацеленной доставки и лечат пациентов с солидным раком. Для того, чтобы выяснить, может ли система нацеленной доставки обеспечивать эффективную доставку функциональных siRNA в опухоли человека, Davis и соавт. исследовали биоптаты от трех пациентов из трех различных групп дозирования; пациентов А, В и С, все из которых имели метастазирующую меланому и получали дозы CALAA-01 с 18, 24 и 30 мгм-2 siRNA, соответственно. Аналогичные дозы также могут быть предусмотрены для системы CRISPR-Cas по настоящему изобретению. Доставку по настоящему изобретению можно осуществлять с помощью наночастиц, содержащих линейный полимер на основе циклодекстрина (CDP), лиганд, нацеливающийся на белок трансферрин человека (TF), представленный на внешней поверхности наночастиц, который входит в контакт с рецепторами TF (TFR) на поверхности раковых клеток, и/или гидрофильный полимер (например, полиэтиленгликоль (PEG), применяемый для обеспечения стабильности наночастиц в биологических жидкостях).
Экзосомы
Экзосомы являются эндогенными нанопузырьками, переносящими РНК и белки, которые могут доставлять короткие интерферирующие РНК (siRNA) в головной мозг мышей. Для снижения иммуногенности Alvarez-Erviti et al. (2011, Nat Biotechnol 29: 341) использовали аутогенные дендритные клетки для получения экзосом. Нацеливания достигали путем конструирования дендритных клеток, экспрессирующих Lamp2b, мембранный белок экзосом, слитый с нейрон-специфическим пептидом RVG3. Очищенные экзосомы нагружали экзогенной siRNA путем электропорации. Меченные RVG нацеленные экзосомы, инъецируемые внутривенно, осуществляли специфическую доставку siRNA для GAPDH в нейроны, микроглию, олигодендроциты в головном мозге, обуславливая нокдаун конкретного гена. Предварительное воздействие меченных RVG экзосом не ослабляло выраженность нокдауна, и неспецифическое поглощение в других тканях не наблюдалось. Терапевтические возможности опосредованной экзосомами доставки siRNA были продемонстрированы сильно выраженным нокдауном мРНК (60%) и белка (62%) ВАСЕ1, терапевтической мишени при болезни Альцгеймера.
Для получения пула иммунологически инертных экзосом Alvarez-Erviti и соавт. отбирали костный мозг у инбредных мышей C57BL/6 с гомогенным гаплотипом главного комплекса гистосовместимости (МНС). Поскольку незрелые дендритные клетки вырабатывают большие количества экзосом, лишенных активаторов Т-клеток, таких как MHC-II и CD86, Alvarez-Erviti и соавт. производили отбор дендритных клеток с гранулоцитарно-макрофагальным колониестимулирующим фактором (GM-CSF) в течение 7 дней. Экзосомы очищали от культуральной надосадочной жидкости на следующий день с применением общепринятых протоколов ультрацентрифугирования. Вырабатываемые экзосомы были физически однородными и характеризовались распределением по размеру с пиком при 80 нм в диаметре, как определяли с помощью анализа отслеживания наночастиц (NTA) и электронной микроскопии. Alvarez-Erviti и соавт. получали 6-12 мкг экзосом (измерено по концентрации белка) на 106 клеток.
Затем Alvarez-Erviti и соавт. исследовали возможность загрузки модифицированных экзосом экзогенными молекулами-карго с применением протоколов электропорации, приспособленных для путей применения на наномасштабном уровне. Поскольку электропорация для мембранных частиц в нанометрическом масштабе изучена недостаточно хорошо, для эмпирической оптимизация протокола электропорации использовали неспецифичную меченную Су5 siRNA. Количество инкапсулированной siRNA анализировали после ультрацентрифугирования и лизиса экзосом. Электропорация при 400 В и 125 мкФ приводила к наибольшему удержанию siRNA и применялась для всех последующих экспериментов.
Alvarez-Erviti и соавт. вводили по 150 мкг каждой siRNA для ВАСЕ1, инкапсулированной в 150 мкг меченных RVG экзосом, нормальным мышам C57BL/6 и сравнивали эффективность нокдауна с таковой в четырех контрольных группах: необработанные мыши, мыши, которым инъецировали только меченные RVG экзосомы, мыши, которым инъецировали siRNA для ВАСЕ1, образующие комплекс с реагентом на основе катионных липосом для in vivo доставки, и мыши, которым инъецировали siRNA для ВАСЕ1, образующие комплекс с RVG-9R, пептидом RVG, конъюгированным с 9 остатками D-аргинина, который электростатически связывается с siRNA. Образцы кортикальной ткани анализировали через 3 дня после введения, и как у обработанных siRNA-RVG-9R, так и у обработанных меченными RVG экзосомами с siRNA мышей наблюдали значительный нокдаун белка (45%, Р<0,05 и 62%, Р<0,01), обусловленный значительным снижением уровней мРНК ВАСЕ1 (66% [+ или -] 15%, Р<0,001 и 61% [+ или -] 13%, Р<0,01, соответственно). Кроме того, заявители продемонстрировали значительное снижение (55%, Р<0,05) общих уровней [бета]-амилоидного пептида 1-42, основного компонента амилоидных бляшек в патологическом процессе при болезни Альцгеймера у животных, обработанных меченными RVG экзосомами. Наблюдавшееся снижение было большим, чем снижение уровней β-амилоидного пептида 1-40, демонстрируемое у нормальных мышей после внутрижелудочковой инъекции ингибиторов ВАСЕ1. Alvarez-Erviti и соавт. проводили быструю амплификацию 5'-концов кДНК (RACE) в отношении продукта расщепления ВАСЕ1, что свидетельствовало об опосредованном РНКи нокдауне с помощью siRNA.
Наконец, Alvarez-Erviti и соавт. исследовали, индуцируют ли меченные RVG экзосомы с siRNA иммунные ответы in vivo, путем определения концентраций IL-6, IP-10, TNFα и IFN-α в сыворотке. После обработки с помощью меченных RVG экзосом с siRNA для всех цитокинов регистрировали незначительные изменения подобно обработке реагентом для трансфекции с siRNA и в отличие от siRNA-RVG-9R, который активно стимулировал секрецию IL-6, что подтверждало иммунологическую инертность как особенность обработки экзосомами. С учетом того, что экзосомы инкапсулируют только 20% siRNA, доставка с помощью меченных RVG экзосом, по-видимому, является более эффективной, чем доставка с помощью RVG-9R, поскольку с использованием в пять раз меньшего количества siRNA без соответствующего уровня стимуляции иммунного ответа достигали сопоставимого нокдауна мРНК и большего нокдауна белка. Данный эксперимент продемонстрировал терапевтические возможности технологии меченных RVG экзосом, которая потенциально подходит для долговременного сайленсинга генов, связанных с нейродегенеративными заболеваниями. Систему доставки на основе экзосом по Alvarez-Erviti и соавт. можно использовать для доставки системы CRISPR-Cas по настоящему изобретению к терапевтическим мишеням, особенно при нейродегенеративных заболеваниях. В настоящем изобретении может быть предусмотрена доза, составляющая приблизительно 100-1000 мг CRISPR-Cas, инкапсулированных в приблизительно 100-1000 мг меченных RVG экзосом.
El-Andaloussi et al. (Nature Protocols 7, 2112-2126(2012)) раскрывают, как экзосомы, полученные из культивируемых клеток, можно приспособить для доставки siRNA in vitro и in vivo. В данном протоколе впервые описано создание нацеленных экзосом посредством трансфекции вектором экспрессии, содержащим экзосомный белок, слитый с пептидным лигандом. Затем El-Andaloussi и соавт. объясняют, как очищать и характеризовать экзосомы из надосадочной жидкости с трансфицированными клетками. Затем El-Andaloussi и соавт. подробно описывают важнейшие стадии загрузки siRNA в экзосомы. Наконец, El-Andaloussi и соавт. излагают в общих чертах, как использовать экзосомы для эффективной доставки siRNA in vitro и in vivo в головной мозг мышей. Также приведены примеры предполагаемых результатов, в которых опосредованная экзосомами доставка siRNA оценивается посредством функциональных анализов и визуализации. Выполнение полного протокола занимает ~3 недели. Доставку или введение согласно настоящему изобретению можно осуществлять с помощью экзосом, полученных из аутогенных дендритных клеток.
В другом варианте осуществления предусмотрены плазменные экзосомы по Wahlgren et al. (Nucleic Acids Research, 2012, Vol. 40, No. 17 e130). Экзосомы представляют собой наноразмерные пузырьки (размером 30-90 нм), вырабатываемые многими типами клеток, в том числе дендритными клетками (DC), В-клетками, Т-клетками, тучными клетками, эпителиальными клетками и опухолевыми клетками. Данные пузырьки образуются путем внутреннего почкования поздних эндосом, а затем высвобождаются во внеклеточную среду при слиянии с плазматической мембраной. Поскольку экзосомы в естественных условиях переносят РНК между клетками, это свойство может быть полезным в генной терапии.
Экзосомы из плазмы получают путем центрифугирования лейкоцитарной пленки при 900 g в течение 20 мин. для выделения плазмы с последующим сбором надосадочной жидкости с клетками, центрифугированием при 300 g в течение 10 мин. для удаления клеток и при 16500 g в течение 30 мин. с последующей фильтрацией через фильтр с диаметром пор 0,22 мм. Экзосомы осаждают путем ультрацентрифугирования при 120000 g в течение 70 мин. Введение siRNA в экзосомы посредством химической трансфекции проводят согласно инструкциям производителя в наборе РНКи Human/Mouse Starter Kit (Quiagen, Хильден, Германия). SiRNA добавляют к 100 мл PBS в конечной концентрации 2 ммоль/мл. После добавления реагента для трансфекции HiPerFect смесь инкубируют в течение 10 мин. при RT. С целью удаления избытка мицелл экзосомы повторно выделяют при помощи латексных частиц с альдегидными/сульфатными группами. Введение CRISPR-Cas в экзосомы посредством химической трансфекции можно проводить аналогично введению siRNA. Экзосомы можно совместно культивировать с моноцитами и лимфоцитами, выделенными из периферической крови здоровых доноров. Таким образом, может быть предусмотрено, чтобы экзосомы, содержащие CRISPR-Cas, можно было вводить в моноциты и лимфоциты и подвергать аутологическому обратному введению в организм человека. Соответственно, доставку или введение согласно настоящему изобретению можно осуществлять с помощью плазменных экзосом.
Липосомы
Доставку или введение согласно настоящему изобретению можно осуществлять с помощью липосом. Липосомы являются сферическими везикулярными структурами, содержащими одно- или многослойный липидный бислой, окружающий внутренние водные компартменты, и относительно непроницаемый внешний липофильный фосфолипидный бислой. Липосомы получили значительное внимание в качестве носителей для доставки лекарственных средств, поскольку они являются биологически совместимыми, нетоксичными, могут доставлять как гидрофильные, так и липофильные молекулы лекарственных средств, защищают свою молекулу-карго от расщепления плазматическими ферментами и переносят свой "груз" через биологические мембраны и гематоэнпефалический барьер (ВВВ) (для обзора см., например, Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi:10.1155/2011/469679).
Липосомы можно получать из нескольких различных типов липидов; однако, для создания липосом в качестве носителей лекарственных средств чаще всего применяют фосфолипиды. Хотя образование липосом является самопроизвольным при смешивании липидной пленки с водным раствором, его также можно ускорить путем приложения силы в виде встряхивания посредством применения гомогенизатора, ультразвукового диспергатора или экструзионного аппарата (для обзора см., например, Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi:10.1155/2011/469679).
К липосомам можно добавлять некоторые другие добавки с целью модификации их структуры и свойств. Например, холестерин либо сфингомиелин можно добавлять к смеси липосом в целях содействия стабилизации структуры липосом и предотвращения утечки внутренних молекул-карго липосом. Дополнительно, липосомы получают из гидрогенизированного яичного фосфатидилхолина или яичного фосфатидилхолина, холестерина и диацетилфосфата, и их средние размеры пузырьков доводили до приблизительно 50 и 100 нм (для обзора см., например, Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi:10.1155/2011/469679).
Традиционный липосомный состав содержит главным образом природные фосфолипиды и липиды, такие как 1,2-дистеароил-sn-глицеро-3-фосфатидилхолин (DSPC), сфингомиелин, формы яичного фосфатидилхолина и моносиалоганглиозид. Поскольку данный состав состоит только из фосфолипидов, липосомные составы сталкиваются со многими проблемами, одной из которых является нестабильность в плазме. Было предпринято несколько попыток преодоления данных проблем, в частности, посредством манипуляции с липидной мембраной. Одна из этих попыток направлена на манипуляцию с холестерином. Добавление холестерина к традиционным составам уменьшает быстрое высвобождение инкапсулированного биологически активного соединения в плазму, а 1,2-диолеоил-sn-глицеро-3-фосфоэтаноламин (DOPE) повышает стабильность (для обзора см., например, Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi:10.1155/2011/469679).
В особенно преимущественном варианте осуществления желательными являются липосомы "троянские кони" (также известные как "молекулярные троянские кони"), и протоколы можно найти на http://cshprotocols.cshlp.org/content/2010/4/pdb.prot5407.long. Эти частицы обеспечивают доставку трансгена в головной мозг в целом после внутрисосудистой инъекции. Без ограничений полагают, что нейтральные липидные частицы со специфичными антителами, конъюгированными с поверхностью, обеспечивают проникновение через гематоэнцефалический барьер посредством эндоцитоза. Заявитель теоретически допускает использование липосом "троянских коней" для доставки нуклеаз семейства CRISPR в головной мозг посредством внутрисосудистой инъекции, что будет обеспечивать получение животных с трансгенами во всем головном мозге без необходимости в манипуляции с эмбрионами. Может быть предусмотрено приблизительно 1-5 г ДНК для in vivo введения в липосомы.
В другом варианте осуществления систему CRISPR-Cas можно вводить в липосомы, такие как стабильная частица из нуклеиновой кислоты и липидов (SNALP) (см., например, Morrissey et al., Nature Biotechnology, Vol. 23, No. 8, August 2005). Предусматриваются ежедневные внутривенные инъекции приблизительно 1, 3 или 5 мг/кг/день специфичной целенаправленно воздействующей CRISPR-Cas в SNALP. Обработку можно осуществлять ежедневно в течение приблизительно трех дней и затем еженедельно в течение приблизительно пяти недель. В другом варианте осуществления также предусмотрена специфичная CRISPR-Cas, инкапсулированная в SNALP, вводимая посредством внутривенной инъекции в дозах, составляющих приблизительно 1 или 2,5 мг/кг (см., например, Zimmerman et al., Nature Letters, Vol. 441, 4 May 2006). Состав на основе SNALP может содержать липиды 3-N-[(ω-метоксиполи(этиленгликоль)2000)карбамоил]-1,2-димиристилоксипропиламин (PEG-C-DMA), 1,2-дилинолеилокси-N,N-диметил-3-аминопропан (DLinDMA), 1,2-дистеароил-sn-глицеро-3-фосфохолин (DSPC) и холестерин в молярном процентном соотношении 2:40:10:48 (см., например, Zimmerman et al., Nature Letters, Vol. 441, 4 May 2006).
В другом варианте осуществления было подтверждено, что стабильные частицы из нуклеиновой кислоты и липидов (SNALP) являются эффективными молекулами для доставки в высоковаскуляризированные опухоли печени, происходящие из HepG2, но не в слабо васкуляризированные опухоли печени, происходящие из НСТ-116 (см., например, Li, Gene Therapy (2012) 19, 775-780). SNALP-липосомы можно получать путем составления D-Lin-DMA и PEG-C-DMA с дистеароилфосфатидилхолином (DSPC), холестерином и siRNA с использованием соотношения липид/siRNA 25:1 и молярного соотношения холестерин/D-Lin-DMA/DSPC/PEG-C-DMA 48/40/10/2. Полученные в результате SNALP-липосомы имеют размер приблизительно 80-100 нм.
В еще одном варианте осуществления SNALP может содержать синтетический холестерин (Sigma-Aldrich, Сент-Луис, Миссури, США), дипальмитоилфосфатидилхолин (Avanti Polar Lipids, Алабастер, Алабама, США), 3-N-[(ω-метоксиполи(этиленгликоль)2000)карбамоил]-1,2-димиристилоксипропиламин и катионный 1,2-дилинолеилокси-3-N,N-диметиламинопропан (см., например, Geisbert et al., Lancet 2010; 375: 1896-905). Может предусматриваться режим дозирования с приемом приблизительно 2 мг/кг общего количества CRISPR-Cas на дозу, вводимую, например, в виде болюсной внутривенной инфузии.
В еще одном варианте осуществления SNALP может содержать синтетический холестерин (Sigma-Aldrich), 1,2-дистеароил-sn-глицеро-3-фосфохолин (DSPC; Avanti Polar Lipids Inc.), PEG-C-DMA и 1,2-дилинолеилокси-3-(N,N-диметил)аминопропан (DLinDMA) (см., например. Judge, J. Clin. Invest. 119: 661-673 (2009)). Составы, используемые для in vivo исследований, могут содержать липиды и РНК в конечном массовом соотношении, составляющем приблизительно 9:1.
Профиль безопасности нанопрепаратов, работающих на основе РНК-интерференции, был рассмотрен Barros and Gollob из Ainylam Pharmaceuticals (см., например, Advanced Drug Delivery Reviews 64 (2012) 1730-1737). Стабильная частица из нуклеиновой кислоты и липидов (SNALP) содержит четыре различных липида - ионизируемый липид (DLinDMA), который является катионным при низком рН, нейтральный липид-помощник, холестерин и диффундирующий конъюгат полиэтиленгликоль (PEG)-липид. Частица имеет диаметр примерно 80 нм и является электронейтральной при физиологическом значении рН. Во время составления ионизируемый липид служит для конденсации липида с анионной siRNA в ходе образования частиц. Будучи положительно заряженным в условиях возрастающей кислотности в эндосомах, ионизируемый липид также опосредует слияние SNALP с мембраной эндосомы, обеспечивая высвобождение siRNA в цитоплазму. Конъюгат PEG-липид стабилизирует частицу и уменьшает агрегацию во время составления, а также впоследствии обеспечивает нейтральную гидрофильную наружную поверхность, улучшающую фармакокинетические свойства.
К настоящему времени была начата реализация двух программ клинических исследований с применением составов на основе SNALP с siRNA. В Tekmira Pharmaceuticals недавно завершили фазу I однодозового исследования SNALP-ApoB с участием взрослых добровольцев с повышенным уровнем холестерина LDL. АроВ преимущественно экспрессируется в печени и тонкой кишке и является ключевым для сборки и секреции VLDL и LDL. Семнадцать субъектов получали однократную дозу SNALP-ApoB (повышение дозы, охватывающее 7 уровней дозирования). Не наблюдалось свидетельств гепатотоксичности (предполагаемой в качестве возможной дозолимитирующей токсичности на основании доклинических исследований). Один (или два) субъекта при наиболее высокой дозе испытывали симптомы гриппоподобных заболеваний, указывающие на стимуляцию иммунной системы, и было принято решение завершить испытание.
В Alnylam Pharmaceuticals аналогичным образом успешно провели исследование ALN-TTR01, который использует технологию SNALP, описанную выше, и целенаправленно воздействует на выработку гепатоцитами как мутантного TTR, так и TTR дикого типа, для лечения опосредованного TTR амилоидоза (ATTR). Были описаны три синдрома при ATTR: семейная амилоидическая полинейропатия (FAP) и семейная амилоидическая кардиомиопатия (FAC) - оба из которых обусловлены аутосомно-доминантными мутациями в TTR; и старческий системный амилоидоз (SSA), обусловленный отложением TTR дикого типа. Недавно завершили I фазу плацебо-контролируемого испытания с повышением однократной дозы ALN-TTR01 с участием пациентов с ATTR. Введение ALN-TTR01 осуществляли в виде 15-минутной IV инфузии 31 пациенту (исследуемое лекарственное средство для 23 и плацебо для 8) в диапазоне доз 0,01-1,0 мг/кг (из расчета siRNA). Лечение хорошо переносилось без значительного повышения показателей печеночных проб. Инфузионные реакции отмечались у 3 из 23 пациентов при ≥0,4 мг/кг; все они реагировали на замедление скорости инфузии и все они продолжали исследование. Минимальные и временные повышения уровней цитокинов IL-6, IP-10 и IL-1ra в сыворотке отмечались у двух пациентов при наиболее высокой дозе 1 мг/кг (как предполагалось на основании доклинических исследований и исследований с участием NHP). Снижение уровня TTR в сыворотке, ожидаемый фармакодинамический эффект ALN-TTR01, наблюдалось при 1 мг/кг.
В еще одном варианте осуществления SNALP можно получить путем солюбилизации катионного липида, DSPC, холестерина и конъюгата PEG-липид, которые солюбилизируют в этаноле в молярном соотношении 40:10:40:10, соответственно (см. Semple et al., Nature Biotechnology, Volume 28 Number 2 February 2010, pp. 172-177). Смесь липидов добавляли к водному буферу (50 мМ цитрат, рН 4) с перемешиванием до конечной концентрации этанола и липидов 30% (об./об.) и 6,1 мг/мл, соответственно, и ей позволяли уравновешиваться при 22°С в течение 2 мин. перед экструзией. Гидрированные липиды экструдировали через два установленных один над другим фильтра с размером пор 80 нм (Nuclepore) при 22°С при помощи экструдера Lipex (Northern Lipids) до достижения диаметра пузырьков 70-90 нм, определяемого посредством анализа по методу динамического рассеяния света. Для этого обычно требовалось 1-3 прохождения. SiRNA (солюбилизированную в водном растворе, содержащем 30% этанол, с 50 мМ цитратом, рН 4) добавляли к предварительно уравновешенным (35°С) пузырькам со скоростью ~5 мл/мин, при перемешивании. После достижения конечного целевого соотношения siRNA/липиды 0,06 (вес/вес) смесь инкубировали в течение дополнительных 30 мин. при 35°С для обеспечения реорганизации пузырьков и инкапсулирования siRNA. Этанол затем удаляли, а внешний буфер заменяли на PBS (155 мМ NaCl, 3 мМ Na2HPO4, 1 мМ KH2PO4, рН 7,5) путем диализа либо тангенциальной поточной диафильтрации. SiRNA инкапсулировали в SNALP посредством регулируемого способа по методу ступенчатого разбавления. Липидные составляющие KC2-SNALP представляли собой DLin-KC2-DMA (катионный липид), дипальмитоилфосфатидилхолин (DPPC; Avanti Polar Lipids), синтетический холестерин (Sigma) и PEG-C-DMA, используемые в молярном соотношении 57,1:7,1:34,3:1,4. После образования нагруженных частиц SNALP подвергали диализу против PBS и стерилизации путем фильтрации через фильтр с диаметром пор 0,2 мкм перед применением. Средние значения размера частиц составляли 75-85 нм, и 90-95% siRNA были инкапсулированы в липидных частицах. Конечное соотношение siRNA/липиды в составах, используемых для in vivo тестирования, составляло ~0,15 (вес/вес). Системы LNP-siRNA, содержащие siRNA для фактора VII, разбавляли до соответствующих концентраций в стерильном PBS непосредственно перед применением, и составы вводили внутривенно через латеральную хвостовую вену в общем объеме 10 мл/кг. Данный способ можно экстраполировать на систему CRISPR-Cas по настоящему изобретению.
Другие липиды
Другие катионные липиды, такие как аминолипид 2,2-дилинолеил-4-диметиламиноэтил-[1,3]-диоксолан (DLin-KC2-DMA), можно использовать для инкапсулирования CRISPR-Cas аналогично siRNA (см., например, Jayaraman, Angew. Chem. Int. Ed. 2012, 51, 8529-8533). Может быть предусмотрен предварительно сформированный пузырек со следующим составом липидов: аминолипид, дистеароилфосфатидилхолин (DSPC), холестерин и (R)-2,3-бис(октадецилокси)пропил-1-(метоксиполи(этиленгликоль)2000)пропилкарбамат (конъюгат PEG-липид) в молярном соотношении 40/10/40/10, соответственно, и с соотношением siRNA для FVII/общие липиды, составляющим примерно 0,05 (вес/вес). Для обеспечения узкого распределения частиц по размеру в диапазоне 70-90 нм и низкого коэффициента полидисперсности 0,11±0,04 (n=56) частицы можно экструдировать до трех раз через мембраны с диаметром пор 80 нм перед добавлением РНК CRISPR-Cas. Можно использовать частицы, содержащие высокоактивный аминолипид 16, в которых молярное соотношение четырех липидных компонентов 16, DSPC, холестерина и конъюгата PEG-липид (50/10/38,5/1,5) можно дополнительно оптимизировать для повышения in vivo активности.
Michael S D Kormann et al. ("Expression of therapeutic proteins after delivery of chemically modified mRNA in mice: Nature Biotechnology, Volume: 29, Pages: 154-157 (2011), Published online 09 January 2011) описывает применение липидных оболочек для доставки РНК. Применение липидных оболочек также является предпочтительным в настоящем изобретении.
В другом варианте осуществления липиды можно составлять с системой CRISPR-Cas по настоящему изобретению с образованием липидных наночастиц (LNP). Липиды включают без ограничения DLin-KC2-DMA4, С 12-200 и совместно действующие липиды дистеароилфосфатидилхолин, холестерин и PEG-DMG, которые можно составлять с CRISPR-Cas вместо siRNA (см., например, Novobrantseva, Molecular Therapy-Nucleic Acids (2012) 1, e4; doi:10.1038/mtna.2011.3) с помощью процедуры самопроизвольного образования пузырьков. Молярное соотношение компонентов может составлять приблизительно 50/10/38,5/1,5 (DLin-KC2-DMA или С12-200/дистеароилфосфатидилхолин/холестерин/PEG-DMG). Конечное весовое соотношение липиды : siRNA может составлять ~12:1 и 9:1 в случае липидных наночастиц (LNP) на основе DLin-KC2-DMA и С12-200, соответственно. Составы могут характеризоваться средними диаметрами частиц ~80 нм при >90% эффективности включения. Может быть предусмотрена доза 3 мг/кг.
Tekmira имеет портфель из примерно 95 семейств патентов-аналогов, выданных в США и за границей, которые направлены на различные аспекты LNP и составы на основе LNP (см., например, патенты США №№7982027; 7799565; 8058069; 8283333; 7901708; 7745651; 7803397; 8101741; 8188263; 7915399; 8236943 и 7838658 и европейские патента №№1766035; 1519714; 1781593 и 1664316), все из которых можно применять в настоящем изобретении и/или приспосабливать к нему.
Систему CRISPR-Cas можно доставлять инкапсулированной в микросферах на основе PLGA, таких как дополнительно описанные в опубликованных заявках на патенты США 20130252281, и 20130245107, и 20130244279 (закрепленных за Moderna Therapeutics), которые относятся к аспектам составления композиций, содержащих модифицированные молекулы нуклеиновых кислот, которые могут кодировать белок, предшественник белка или частично или полностью процессированную форму белка или предшественника белка. Состав может характеризоваться молярным соотношением 50:10:38,5:1,5-3,0 (катионный липид: фузогенный липид: холестерин: конъюгат PEG-липид). Конъюгат PEG-липид может быть выбран без ограничения из PEG-C-DOMG, PEG-DMG. Фузогенный липид может представлять собой DSPC. См. также Schrum et al., Delivery and Formulation of Engineered Nucleic Acids, опубликованная заявка на патент США 20120251618.
Технология Nanomerics преодолевает проблемы, связанные с биологической доступностью, для широкого спектра терапевтических средств, в том числе низкомолекулярных гидрофобных лекарственных средств, пептидов и терапевтических средств на основе нуклеиновых кислот (плазмид, siRNA, микроРНК). Конкретные пути введения, для которых технология продемонстрировала очевидные преимущества, включают пероральный путь, перенос через гематоэнцефалический барьер, доставка в солидные опухоли, а также в глаз. См., например, Mazza et al., 2013, ACS Nano. 2013 Feb 26; 7(2): 1016-26; Uchegbu and Siew, 2013, J Pharm Sci. 102(2): 305-10 и Lalatsa et al., 2012, J Control Release. 2012 Jul 20; 161(2): 523-36.
В публикации заявки на патент США №20050019923 описаны катионные дендримеры для доставки биологически активных молекул, таких как молекулы полинуклеотидов, пептиды и полипептиды и/или фармацевтические средства, в организм млекопитающего. Дендримеры подходят для обеспечения нацеленной доставки биологически активных молекул в, например, печень, селезенку, легкое, почку или сердце. Дендримеры являются синтетическими 3-мерными макромолекулами, получаемыми ступенчатым способом из простых разветвленных мономерных звеньев, природу и количество функциональных групп которых можно легко регулировать и изменять. Дендримеры синтезируют путем повторяющегося присоединения "строительных блоков" в направлении от сердцевины с несколькими функциональными группами (дивергентный подход к синтезу) или к сердцевине с несколькими функциональными группами (конвергентный подход к синтезу), и каждое присоединение 3-мерной оболочки из "строительных блоков" приводит к образованию дендримеров более высокой генерации. Синтез полипропилениминовых дендримеров начинается с диаминобутановой сердцевины, к которой присоединяют удвоенное количество аминогрупп посредством двойного присоединения по Михаэлю ацетонитрила к первичным аминогруппам с последующим гидрированием нитрильных групп. Это обуславливает удвоение количества аминогрупп. Полилропилениминовые дендримеры содержат 100% протонируемых атомов азота и до 64 концевых аминогрупп (генерация 5, DAB 64). Протонируемые группы обычно представляют собой аминогруппы, способные принимать протоны при нейтральном рН. Применение дендримеров в качестве средств для доставки геном в основном ориентировано на использование полиамидоамина и фосфорсодержащих соединений со смесью из амина/амида или N-P(O2)S в качестве конъюгирующих единиц, соответственно. При этом в работах не сообщалось о применении полипропилениминовых дендримеров низкой генерации для доставки генов. Полипропилениминовые дендримеры также изучали в качестве рН-чувствительных систем с контролируемым высвобождением для доставки лекарственных средств и для инкапсулирования в них гостевых молекул в случае химической модификации периферических аминокислотных групп. Также изучали цитотоксичность и взаимодействие полипропилениминовых дендримеров с ДНК, а также эффективность трансфекции с помощью DAB 64.
Публикация заявки на патент США №20050019923 основана на наблюдении того, что в противоположность более ранним сообщениям катионные дендримеры, такие как Полипропилениминовые дендримеры, проявляют надлежащие свойства, такие как специфичное нацеливание и низкая токсичность, для применения в нацеленной доставке биологически активных молекул, таких как генетический материал. В дополнение, производные катионного дендримера также проявляют подходящие свойства для нацеленной доставки биологически активных молекул. См. также "Биологически активные полимеры", публикация заявки на патент США 20080267903, в которой раскрыто следующее: "Показано, что различные полимеры, в том числе катионные полиаминные полимеры и дендримерные полимеры, обладают антипролиферативной активностью и могут, таким образом, быть применимыми для лечения нарушений, характеризующихся нежелательной пролиферацией клеток, таких как неоплазии и опухоли, воспалительные нарушения (в том числе аутоиммунные нарушения), псориаз и атеросклероз. Полимеры можно применять в отдельности в качестве активных средств или в качестве средств доставки других терапевтических средств, таких как молекулы лекарственных средств или нуклеиновые кислоты, для генной терапии. В таких случаях присущая полимерам собственная противоопухолевая активность может дополнять активность средства, подлежащего доставке".
Белки с избыточным зарядом
Белки с избыточным зарядом представляют собой класс сконструированных или встречающихся в природе белков с необычно высоким положительным или отрицательным теоретическим суммарным зарядом. Белки как с избыточным отрицательным, так и с избыточным положительным зарядом проявляют особое свойство устойчивости к термически или химически индуцированной агрегации. Белки с избыточным положительным зарядом также способны проникать в клетки млекопитающих. Ассоциация молекул-карго, таких как плазмидная ДНК, siRNA, с этими белками или другими белками может обеспечивать функциональную доставку данных макромолекул в клетки млекопитающих как in vitro, так и in vivo. В лаборатории Дэвида Лю сообщили о создании и определении характеристик белков с избыточным зарядом в 2007 г. (Lawrence et al., 2007, Journal of the American Chemical Society 129, 10110-10112).
Невирусная доставка siRNA и плазмидной ДНК в клетки млекопитающих является значимой как в исследованиях, так и в терапевтических применениях (Akinc et al., 2010, Nat. Biotech. 26, 561-569). Очищенный белок GFP с зарядом +36 (или другой белок с избыточным положительным зарядом) смешивают с siRNA в подходящей бессывороточной среде, и обеспечивают возможность образования ими комплекса перед добавлением к клеткам. Включение сыворотки на этой стадии ингибирует образование комплексов белок с избыточным зарядом-siRNA и снижает эффективность обработки. Было обнаружено, что следующий протокол является эффективным для ряда линий клеток (McNaughton et al., 2009, Proc. Natl. Acad. Sci. USA 106, 6111-6116). Однако, в целях оптимизации процедуры для конкретных линий клеток следует проводить предварительные эксперименты с различными дозами белка и siRNA.
(1) За один день до обработки высеять 1×105 клеток на лунку в 48-луночный планшет.
(2) В день обработки разбавить очищенный белок GFP с зарядом +36 в бессывороточной среде до конечной концентрации 200 нМ. Добавить siRNA до конечной концентрации 50 нМ. Перемешать в вихревой мешалке и инкубировать при комнатной температуре в течение 10 мин.
(3) Во время инкубирования аспирировать среду от клеток и промыть один раз с помощью PBS.
(4) После инкубирования GFP с зарядом +36 и siRNA добавить к клеткам комплексы белок-siRNA.
(5) Инкубировать клетки с комплексами при 37°С в течение 4 ч.
(6) После инкубирования аспирировать среду и промыть три раза с помощью 20 ЕД/мл гепарина в PBS. Инкубировать клетки в сывороточной среде в течение дополнительных 48 ч. или дольше в зависимости от анализа нокдауна.
(7) Анализировать клетки с помощью иммуноблоттинга, количественной ПЦР, фенотипического анализа или другого соответствующего способа.
В лаборатории Дэвида Лю дополнительно обнаружили, что GFP с зарядом +36 является эффективным реагентом для доставки плазмид в ряд клеток. Поскольку плазмидная ДНК является более крупной молекулой-карго, чем siRNA, то для образования эффективного комплекса с плазмидами требуется пропорционально больше белка GFP с зарядом +36. Для эффективной доставки плазмид заявители разработали вариант GFP с зарядом +36, несущий С-концевую пептидную метку НА2, известный пептид, разрушающий эндосомы, происходящий из белка гемагглютинина вируса гриппа. Следующий протокол был эффективным для многих клеток, но, как указано выше, рекомендуется, чтобы дозы плазмидной ДНК и белка с избыточным зарядом были оптимизированы для конкретных линий клеток и путей применения в доставке.
(1) За один день до обработки высеять 1×105 на лунку в 48-луночный планшет.
(2) В день обработки разбавить очищенный белок GFP с зарядом

(3) Во время инкубирования аспирировать среду от клеток и промыть один раз с помощью PBS.
(4) После инкубирования GFP с зарядом

(5) Инкубировать клетки с комплексами при 37°С в течение 4 ч.
(6) После инкубирования аспирировать среду и промыть с помощью PBS. Инкубировать клетки в сывороточной среде и инкубировать в течение дополнительных 24-48 ч.
(7) При необходимости проанализировать доставку плазмид (например, посредством экспрессии генов, обусловленной плазмидами).
См. также, например, McNaughton et al., Proc. Natl. Acad. Sci. USA 106, 6111-6116 (2009); Cronican et al., ACS Chemical Biology 5, 747-752 (2010); Cronican et al., Chemistry & Biology 18, 833-838 (2011); Thompson et al., Methods in Enzymology 503, 293-319 (2012); Thompson, D.B., et al., Chemistry & Biology 19 (7), 831-843 (2012). Способы использования белков с избыточным зарядом можно применять для доставки системы CRISPR-Cas по настоящему изобретению и/или приспосабливать к ней.
Имплантируемые устройства
В другом варианте осуществления также предусмотрены имплантируемые устройства для доставки системы CRISPR-Cas. Например, в публикации заявки на патент США 20110195123 раскрыто представленное имплантируемое медицинское устройство, высвобождающее лекарственное средство локально и в течение длительного периода, в том числе несколько типов такого устройства, реализуемые методы лечения и способы имплантации. Устройство содержит полимерный субстрат, такой как матрица, например, применяемый в качестве корпуса устройства, и лекарственные средства, и в некоторых случаях дополнительные трехмерные подложки-носители, такие как металлы или дополнительные полимеры, и материалы для улучшения видимости и визуализации. В основе выбора лекарственного средства лежит преимущество высвобождения лекарственного средства, происходящего локально и в течение длительного периода, где лекарственное средство высвобождается непосредственно во внеклеточный матрикс (ЕСМ) пораженной заболеванием области, как, например, в случае опухоли, воспаления, дегенерации, или в целях симптоматической терапии, или в пораженные гладкомышечные клетки, или для предупреждения. Один вид лекарственных средств представляет собой лекарственные средства для сайленсинга генов на основе РНК-интерференции (РНКи), включающие без ограничения siRNA, shRNA или антисмысловые РНК/ДНК, рибозимы и нуклеозидные аналоги. Таким образом, данную систему можно применять для системы CRISPR-Cas по настоящему изобретению и/или адаптировать к ней. Методы имплантации в некоторых вариантах осуществления представляют собой существующие процедуры имплантации, разработанные и применяемые в настоящее время для других видов лечения, в том числе для брахитерапии и пункционной биопсии. В таких случаях размеры нового имплантата, описанного в настоящем изобретении, аналогичны размерам первоначального имплантата. Как правило, в ходе одной процедуры лечения имплантируют несколько устройств.
Как описано в публикации заявки на патент США 20110195123, представлена имплантируемая или вставная система для доставки лекарственных средств, в том числе системы, применимые для введения в полость, такую как брюшная полость, и/или для любого другого типа введения, в которой система для доставки лекарственных средств не закреплена и не присоединена, содержащая биоустойчивый, и/или разлагаемый, и/или биопоглощаемый полимерный субстрат, который может, например, необязательно представлять собой матрицу. Следует отметить, что выражение "вставка" также включает имплантацию. Система для доставки лекарственных средств преимущественно реализуется как "Loder", описанный в публикации заявки на патент США 20110195123.
Полимер или множество полимеров, содержащие средство и/или множество средств, являются биосовместимыми, обеспечивая высвобождение средства с контролируемой скоростью, где общий объем полимерного субстрата, такого как матрица, например, в некоторых вариантах осуществления необязательно и предпочтительно не превосходит максимальный объем, позволяющий достигнуть терапевтического уровня средства. В качестве неограничивающего примера, такой объем предпочтительно находится в диапазоне от 0,1 м3 до 1000 мм3, как того требует объем загруженного средства. Loder необязательно может иметь больший размер, например, будучи включенным в состав устройства, размер которого определяется функциональным назначением, например, без ограничения коленного сустава, внутриматочного или шеечного кольца и т.п.
Система для доставки лекарственных средств (для доставки композиции) в некоторых вариантах осуществления предназначена для предпочтительного использования разлагаемых полимеров, где основным механизмом высвобождения является объемная эрозия; или же в некоторых вариантах осуществления применяются неразлагаемые или медленно разлагаемые полимеры, где основным механизмом высвобождения является диффузия, а не объемная эрозия, так что их наружная часть функционирует в качестве мембраны, а их внутренняя часть функционирует в качестве депо лекарственного средства, которое практически не подвергается воздействию окружения в течение продолжительного периода (например, от приблизительно недели до приблизительно нескольких месяцев). Также можно необязательно применять комбинации различных полимеров с различными механизмами высвобождения. Градиент концентраций на поверхности предпочтительно эффективно поддерживается постоянным в течение значительного периода в ходе общего периода высвобождения лекарственного средства, и, таким образом, скорость диффузии (называемой "диффузией нулевого порядка") является эффективно постоянной. Под выражением "постоянный" подразумевают скорость диффузии, которая предпочтительно поддерживается выше нижнего порога терапевтической эффективности, но которая, тем не менее, может необязательно характеризоваться начальным всплеском и/или колебаться, например, повышаясь и понижаясь в некоторой степени. Скорость диффузии предпочтительно поддерживается таким образом в течение длительного периода, и до определенного уровня она может считаться постоянной для оптимизации терапевтически эффективного периода, например, эффективного периода сайленсинга.
Система для доставки лекарственных средств необязательно и предпочтительно предназначена для защиты нуклеотидного терапевтического средства от деградации, химической по своей природе или обусловленной воздействием ферментов и других факторов в организме субъекта.
Система для доставки лекарственных средств, описанная в публикации заявки на патент США 20110195123, необязательно связана с чувствительными и/или активирующими приборами, функционирующими во время и/или после имплантации устройства посредством неинвазивных и/или минимально инвазивных способов активации и/или ускорения/замедления, например, необязательно включающих без ограничения способы или устройства с применением термического нагревания и охлаждения, лазерных пучков и ультразвука, в том числе фокусированного ультразвука, и/или RF (радиочастот).
Согласно некоторым вариантам осуществления публикации заявки на патент США 20110195123 участок для локальной доставки может необязательно включать целевые участки, характеризующиеся высокой аномальной пролиферацией клеток и подавлением апоптоза, в том числе опухоли, очаги активного и/или хронического воспаления и инфекции, включающих аутоиммунные болезненные состояния, ткань с дегенеративными изменениями, включающую мышечную и нервную ткань, очаги хронической боли, участки с дегенеративными изменениями, и местоположения переломов костей, и другие местоположения ран, для усиления регенерации ткани, а также поврежденные сердечные, гладкие и поперечно-полосатые мышцы. Участок для локальной доставки также может необязательно включать участки, позволяющие осуществлять профилактические мероприятия, в том числе при беременности, путем предупреждения инфекции и для пожилых людей.
Участок для имплантации композиции, или целевой участок, предпочтительно характеризуется радиусом, площадью и/или объемом, достаточно малыми для нацеленной локальной доставки. Например, целевой участок необязательно имеет диаметр в диапазоне от приблизительно 0,1 мм до приблизительно 5 см.
Местоположение целевого участка предпочтительно выбирают для достижения максимальной терапевтической эффективности. Например, композицию системы для доставки лекарственных средств (необязательно вместе с устройством для имплантации, описанным выше) необязательно и предпочтительно имплантируют в опухолевое окружение, или рядом с ним, или в кровеносную сеть, связанную с ним.
Например, композицию (необязательно вместе с устройством) необязательно имплантируют в поджелудочную железу, предстательную железу, молочную железу, печень или рядом с ними, через сосок, в сосудистую систему и т.д.
Целевое местоположение необязательно выбирают из группы, состоящей из (только в качестве неограничивающих примеров, поскольку любой участок в организме необязательно может подходить для имплантации Loder): 1. участков головного мозга с дегенеративными изменениями, таких как базальные ганглии, белое и серое вещество, при болезни Паркинсона или Альцгеймера; 2. спинного мозга, как в случае бокового амиотрофического склероза (ALS); 3. шейки матки для предупреждения HPV-инфекции; 4. суставов с активным или хроническим воспалением; 5. дермы, как в случае псориаза; 6. участков симпатических и чувствительных нервов для обезболивающего эффекта; 7. участков внутрикостной имплантации; 8. очагов острой и хронической инфекции; 9. интравагинальных участков; 10. внутреннего уха слуховой системы, лабиринта внутреннего уха, вестибулярной системы; 11. внутритрахеальных участков; 12. внутрисердечных участков; участков коронарных сосудов, эпикардиальных участков; 13. мочевого пузыря; 14. желчевыделительной системы; 15. паренхимной ткани, включающей, без ограничения, почку, печень, селезенку; 16. лимфатических узлов; 17. слюнных желез; 18. десен вокруг зубов; 19. внутрисуставных участков (имплантация в суставы); 20. внутриглазных участков; 21. ткани головного мозга; 22. желудочков головного мозга; 23. полостей, в том числе брюшной полости (например, без ограничения, для лечения рака яичника); 24. внутрипищеводных участков и 25. внутрипрямокишечных участков.
Вставка системы (например, устройства, содержащего композицию) необязательно связана с инъекцией материала в ЕСМ целевого участка и окружение этого участка для воздействия на локальные рН, и/или температуру, и/или другие биологические факторы, влияющие на диффузию лекарственного средства и/или кинетику лекарственного средства в ЕСМ целевого участка и окружении такого участка.
Согласно некоторым вариантам осуществления высвобождение указанного средства необязательно может быть связано с чувствительными и/или активирующими приборами, функционирующими до, и/или во время, и/или после вставки посредством неинвазивных, и/или минимально инвазивных, и/или других способов активации и/или ускорения/замедления, включающих способы или устройства с применением лазерных пучков, излучения, термического нагревания и охлаждения, и ультразвука, в том числе фокусированного ультразвука, и/или RF (радиочастот), а также химических активаторов.
Согласно другим вариантам осуществления в публикации заявки на патент США 20110195123 лекарственное средство предпочтительно включает биологическое лекарственное средство для сайленсинга генов на основе РНКи, например, для лечения случаев локализованного рака молочной железы, поджелудочной железы, головного мозга, почки, мочевого пузыря, легкого и предстательной железы, описанных ниже. Кроме того, многие лекарственные средства, отличные от siRNA, применимы для инкапсулирования в Loder и могут применяться в контексте настоящего изобретения, поскольку такие лекарственные средства могут быть инкапсулированы в субстрат Loder, такой как, например, матрица. Такие лекарственные средства включают одобренные лекарственные средства, доставляемые в настоящее время с помощью способов, отличных от способов по настоящему изобретению, включающие амфотерицин В для лечения грибковой инфекции; антибиотики, как, например, при остеомиелите; обезболивающие средства, такие как наркотические средства; антидегенеративные средства, как, например, при болезни Альцгеймера или Паркинсона, в Loder, имплантируемом вблизи спинного мозга в случае боли в спине. Такую систему можно применять для доставки системы CRISPR-Cas по настоящему изобретению и/или приспосабливать к ней.
Например, для конкретных путей применения, таких как предупреждение роста или возобновления роста гладкомышечных клеток (поврежденных в ходе процедуры стентирования и вследствие этого склонных к пролиферации), лекарственное средство может необязательно представлять собой siRNA, осуществляющую сайленсинг в гладкомышечных клетках, в том числе сайленсинг H19, или лекарственное средство, выбранное из группы, состоящей из таксола, рапамицина и аналогов рапамицина. В таких случаях Loder предпочтительно представляет собой стент, выделяющий лекарственное средство (DES), с пролонгированным высвобождением при постоянной скорости либо выделенное устройство, которое имплантируют отдельно, совместно со стентом. Все из этого можно применять для системы CRISPR-Cas по настоящему изобретению и/или приспосабливать к ней.
В качестве другого примера конкретного применения, нейро- и миодегенеративные заболевания развиваются в связи с аномальной экспрессией генов. Локальная доставка РНК для сайленсинга может иметь терапевтические свойства, препятствующие такой аномальной экспрессии генов. Локальная доставка антиапоптотических, противовоспалительных и антидегенеративных лекарственных средств, в том числе низкомолекулярных лекарственных средств и макромолекул, может также необязательно быть терапевтической. В таких случаях Loder применяют для пролонгированного высвобождения при постоянной скорости и/или посредством выделенного устройства, которое имплантируют отдельно. Все из этого можно применять для системы CRISPR-Cas по настоящему изобретению и/или приспосабливать к ней.
В качестве еще одного примера конкретного применения, психические и когнитивные расстройства лечат с помощью модификаторов генов. Нокдаун гена с помощью РНК для сайленсинга является возможным методом лечения. Применение Loder для локальной доставки нуклеотидных средств в участки центральной нервной системы является возможным методом терапии психических и когнитивных расстройств, в том числе без ограничения психоза, биполярных расстройств, невротических расстройств и расстройств поведения. Loder могут также обеспечивать локальную доставку лекарственных средств, в том числе низкомолекулярных лекарственных средств и макромолекул, при имплантации в конкретные участки головного мозга. Все из этого можно применять для системы CRISPR-Cas по настоящему изобретению и/или приспосабливать к ней.
В качестве другого примера конкретного применения, сайленсинг генов медиаторов врожденного и/или приобретенного иммунного ответа в локальных участках обеспечивает предупреждение отторжения трансплантированного органа. Локальная доставка РНК для сайленсинга и иммуномодулирующих реагентов с помощью Loder, имплантированного в трансплантированный орган и/или участок имплантации, активирует подавление местного иммунитета в отношении трансплантированного органа путем отвлечения иммунных клеток, таких как CD8+. Все из этого можно применять для системы CRISPR-Cas по настоящему изобретению и/или приспосабливать к ней.
В качестве другого примера конкретного применения, факторы роста сосудов, в том числе VEGF, и ангиогенин, и другие, являются существенно важными для неоваскуляризации. Локальная доставка факторов, пептидов, пептидомиметиков или подавление их репрессоров является важным терапевтическим воздействием; сайленсинг репрессоров и локальная доставка факторов, пептидов, макромолекул и низкомолекулярных лекарственных средств, стимулирующих ангиогенез, с помощью Loder являются терапевтическими мерами в отношении заболевания периферических сосудов, системного заболевания сосудов и заболевания сосудов сердца.
Способ вставки, такой как имплантация, необязательно можно еще применять для других типов имплантации в ткань, и/или для вставок, и/или для отбора образцов тканей, необязательно без модификаций или, альтернативно, необязательно лишь с незначительными модификациями таких способов. Такие способы необязательно включают без ограничения способы брахитерапии, биопсию, эндоскопию с применением ультразвуковых технологий и/или без них, такую как ERCP, стереотаксические способы в отношении ткани головного мозга, лапароскопию, в том числе имплантацию с помощью лапароскопа в суставы, органы брюшной полости, стенку мочевого пузыря и полости тела.
МРНК Фермента CRISPR и направляющая РНК
МРНК фермента CRISPR и направляющую РНК можно также доставлять по отдельности. МРНК фермента CRISPR можно доставлять перед направляющей РНК, чтобы предоставить время для экспрессии фермента CRISPR. МРНК фермента CRISPR можно вводить за 1-12 часов (предпочтительно примерно за 2-6 часов) до введения направляющей РНК.
Альтернативно, мРНК фермента CRISPR и направляющую РНК можно вводить совместно. Вторую бустерную дозу направляющей РНК можно преимущественно вводить через 1-12 часов (предпочтительно примерно через 2-6 часов) после первого введения мРНК фермента CRISPR + направляющей РНК.
Введение дополнительных доз мРНК фермента CRISPR и/или направляющей РНК может быть применимым для достижения наиболее эффективных уровней модификации генома.
Для сведения к минимуму токсичности и нецелевого эффекта будет важной регуляция концентрации доставляемых мРНК фермента CRISPR и направляющей РНК. Оптимальные концентрации мРНК фермента CRISPR и направляющей РНК можно определить путем тестирования различных концентраций в клеточной или животной модели и применения глубокого секвенирования для анализа степени модификации в возможных нецелевых локусах генома. Например, для направляющей последовательности, целенаправленно воздействующей на 5'-GAGTCCGAGCAGAAGAAGAA-3' в гене ЕМХ1 генома человека, можно применять глубокое секвенирование для определения уровня модификации в следующих двух нецелевых локусах - 1: 5'-GAGTCCTAGCAGGAGAAGAA-3' и 2: 5'-GAGTCTAAGCAGAAGAAGAA-3'. Для in vivo доставки следует выбрать концентрацию, дающую наиболее высокий уровень целевой модификации при минимизации уровня нецелевой модификации.
Альтернативно, для минимизации уровня токсичности и нецелевого эффекта можно доставлять мРНК фермента CRISPR никазы (например, Cas9 S. pyogenes с мутацией D10A) вместе с парой направляющих РНК, которые осуществляют нацеливание на сайт, представляющий интерес. Две направляющие РНК необходимо расположить следующим образом. Направляющие последовательности показаны красным (одинарное подчеркивание) и синим (двойное подчеркивание), соответственно (эти примеры основаны на требованиях к РАМ для Cas9 Streptococcus pyogenes).





Дополнительное детальное исследование системы предоставило заявителям свидетельство наличия "липкого" 5'-конца (см., например. Ran et al., Cell. 2013 Sep 12; 154(6): 13 80-9 и предварительную заявку на патент США с регистрационным номером 61/871301, поданную 28 августа 2013 г.). Заявители дополнительно идентифицировали параметры, связанные с эффективным расщеплением мутантной никазой Cas9 в сочетании с двумя направляющими РНК, и эти параметры включают, без ограничения, длину "липкого" 5'-конца. В вариантах осуществления настоящего изобретения "липкий" 5'-конец содержит не более 200 пар оснований, предпочтительно не более 100 пар оснований или более предпочтительно не более 50 пар оснований. В вариантах осуществления настоящего изобретения "липкий" 5'-конец содержит по меньшей мере 26 пар оснований, предпочтительно по меньшей мере 30 пар оснований или более предпочтительно 34-50 пар оснований или 1-34 пары оснований. В других предпочтительных способах по настоящему изобретению первая направляющая последовательность, управляющая расщеплением одной нити ДНК-дуплекса возле первой целевой последовательности, и вторая направляющая последовательность, управляющая расщеплением другой нити возле второй целевой последовательности, обуславливают возникновение "тупого" конца или "липкого" 3'-конца. В вариантах осуществления настоящего изобретения "липкий" 3'-конец содержит не более 150, 100 или 25 пар оснований или не менее 15, 10 или 1 пары оснований. В предпочтительных вариантах осуществления "липкий" 3'-конец содержит 1-100 пар оснований.
Аспекты настоящего изобретения относятся к снижению экспрессии продукта гена, или к дополнительному введению матричного полинуклеотида в молекулу ДНК, кодирующую продукт гена, или к точному вырезанию вставочной последовательности путем обеспечения повторного отжига и дотирования двух "липких" 5'-концов, или к изменению активности или функции продукта гена, или к повышению экспрессии продукта гена. В варианте осуществления настоящего изобретения продукт гена представляет собой белок.
Только пары sgRNA, создающие "липкие" 5'-концы, с перекрыванием между направляющими последовательностями, составляющим менее 8 п.о. (смещение превышает -8 п.о.), были способны опосредовать выявляемое образование вставок/делений. Важно, что каждая направляющая последовательность, применяемая в данных анализах, способна эффективно индуцировать образование вставок/делений при спаривании с Cas9 дикого типа, что указывает на то, что взаимное расположение пар направляющих последовательностей является наиболее важным параметром для предсказания активности внесения двойных однонитевых разрывов.
Поскольку Cas9n и Cas9H840A вносят однонитевые разрывы в противоположные нити ДНК, замена Cas9n и Cas9H840A указанной парой sgRNA должна обуславливать инверсию типа "липкого" конца. Например, пара sgRNA, которая с Cas9n будет образовывать "липкий" 5'-конец, теоретически будет образовывать вместо этого соответствующий "липкий" 3'-конец. Таким образом, пары sgRNA, обуславливающие с Cas9n образование "липкого" 3'-конца, можно применять с Cas9H840A для образования "липкого" 5'-конца. Заявители тестировали Cas9H840A с набором пар sgRNA, предназначенных для образования "липких" как 5'-, так и 3'-концов (диапазон смещений от -278 до +58 п.о.), но, вопреки ожиданиям, не могли наблюдать образование вставок/делений. Может потребоваться дополнительная работа, для определения необходимых правил конструирования пар sgRNA для обеспечения внесения двойных однонитевых разрывов с помощью Cas9H840A.
Печень, пропротеинконвертаза субтилизин/кексин 9 (PCSK9)
Пропротеинконвертаза субтилизин/кексин 9 (PCSK9) является представителем семейства сериновых протеаз субтилизинового типа. PCSK9 экспрессируется главным образом в печени и является критически важным для снижения экспрессии рецепторов LDL в гепатоцитах. Уровни LDL-C в плазме значительно повышены у людей с мутациями приобретения функции в PCSK9, вследствие чего их считают имеющими тяжелую гиперхолестеринемию. Таким образом, PCSK9 является привлекательной мишенью для CRISPR. Целенаправленно воздействующие на PCS9K CRISPR можно составлять в липидных частицах и вводить, например, при приблизительно 15, 45, 90, 150, 250 и 400 мкг/кг внутривенно (см., например, http://www.alnylam.com/capella/wp-content/uploads/2013/08/ALN-PCS02-001-Protocol-Lancet.pdf).
Bailey et al. (J Mol Med (Berl). 1999 Jan; 77(1): 244-9) раскрывает доставку инсулина посредством генной терапии соматических клеток ex vivo, включающей выделение отличных от В-клеток соматических клеток (например, фибробластов) у пациента с диабетом и генетическое изменение их in vitro для выработки и секреции инсулина. Клетки можно выращивать в культуре и возвращать донору в качестве источника заменителя инсулина. Клетки, модифицированные таким образом, можно оценивать перед имплантацией, а резервные запасы можно подвергать криоконсервапии. В случае применения собственных клеток пациента процедура будет избавлена от необходимости в подавлении иммунитета и преодолеет проблему снабжения тканей, избегая при этом повторного разрушения клеток. Для генной терапии соматических клеток ex vivo требуется доступный и устойчивый тип клеток, поддающийся нескольким трансфекциям и подвергаемый регулируемой пролиферации. Особые проблемы, связанные с применением отличных от В-клеток соматических клеток, включают процессинг проинсулина в инсулин и придание чувствительности стимулируемому глюкозой биосинтезу проинсулина и регулируемому высвобождению инсулина. Предварительные исследования с применением фибробластов, гипофизарных клеток, клеток почек (COS) и клеток яичников (СНО) позволяют предположить, что с этими проблемами можно столкнуться и что генная терапия соматических клеток ех vivo предлагает возможный подход к заместительной терапии инсулином. Систему по Bailey и соавт. можно применять для доставки системы CRISPR-Cas по настоящему изобретению в печень и/или адаптировать к ней.
Способы по Sato и соавт.(Nature Biotechnology Volume 26 Number 4 April 2008, Pages 431-442) можно применять для доставки системы CRISPR-Cas по настоящему изобретению в печень. Sato и соавт. обнаружили, что виды лечения с помощью связанных с витамином А липосом, содержащих siRNA, практически полностью устраняли фиброз печени и продлевали период выживаемости у крыс со смертельным в иных случаях циррозом печени, индуцированным диметилнитрозамином, в зависимости от дозы и продолжительности. Катионные липосомы (Lipotrust), содержащие O,O'-дитетрадеканоил-N-(а-триметиламмонийацетил)диэтаноламинохлорид (DC-6-14) в качестве катионного липида, холестерин и диолеоилфосфатидилэтаноламин в молярном соотношении 4:3:3 (демонстрировавшие высокую эффективность трансфекции в условиях сывороточной среды для доставки генов in vitro и in vivo), приобретали у Hokkaido System Science. Липосомы изготовляли посредством способа с применением подвергнутых сублимационной сушке "пустых" липосом и получали в концентрации 1 мМ (DC-16-4) путем добавления бидистиллированной воды (DDW) к лиофилизированной смеси липидов при перемешивании вихревым способом перед применением. Для получения связанных с VA липосом 200 нмоль витамина А (ретинол, Sigma), растворенного в DMSO, смешивали с суспензиями липосом (в виде 100 нмоль DC-16-4) путем перемешивания вихревым способом в пробирке на 1,5 мл при 25°С. Для получения связанных с VA липосом, несущих siRNAgp46 (VA-липосома-siRNAgp46), раствор siRNAgp46 (580 пмоль/мл в DDW) добавляли к раствору связанных с ретинолом липосом при перемешивании при 25°С. Соотношение siRNA и DC-16-4 составляло 1:11,5 (моль/моль), а соотношение siRNA и липосом (вес/вес) составляло 1:1. Любой свободный витамин А или siRNA, не поглощенную липосомами, отделяли от липосомальных препаратов при помощи системы микроразделения (концентратор VTVASPIN 2, MWCO 30000, PES, VIVASCIENCE). Суспензию липосом добавляли на фильтры и центрифугировали при 1500 g в течение 5 мин. 3 раза при 25°С. Фракции собирали, и материал, захваченный фильтром, разводили в PBS с получением желаемой дозы для применения in vitro или in vivo. Крысам проводили три инъекции по 0,75 мг/кг siRNA через день. Систему по Sato и соавт. можно применять для доставки системы CRISPR-Cas по настоящему изобретению в печень посредством доставки приблизительно 0,5-1 мг/кг РНК CRISPR-Cas в липосомах, как описано Sato и соавт. в отношении людей, и/или адптировать к ней.
Способы по Rozema et al. (PNAS, August 7, 2007, vol. 104, no. 32) в отношении средств доставки siRNA в гепатоциты как in vitro, так и in vivo, которые Rozema и соавт. назвали динамичными поликонъюгатами (Dynamic PolyConjugates) siRNA, можно также применять в настоящем изобретении. Ключевые особенности технологии динамичных поликонъюгатов включают применение мембраноактивного полимера, способность к обратимой маскировке активности данного полимера до достижения им кислой среды эндосом и способность к специфичному целенаправленному воздействию этого модифицированного полимера и его молекулы-карго siRNA на гепатоциты in vivo после простой i.v. инъекции под низким давлением. SATA-модифицированные siRNA синтезируют с помощью реакции 5'-аминомодифицированной siRNA с 1 весовым эквивалентом (вес. экв.) реагента N-сукцинимидил-S-ацетилтиоацетата (SATA) (Pierce) и 0,36 вес. экв. NaHCO3 в воде при 4°С в течение 16 ч. Модифицированные siRNA затем осаждают путем добавления 9 об. этанола и инкубирования при 80°С в течение 2 ч. Осадок ресуспендируют в 1X буфере для siRNA (Dharmacon) и оценивают количественно путем измерения поглощения при длине волны 260 нм. PBAVE (30 мг/мл в 5 мМ TAPS, рН 9) модифицируют путем добавления 1,5 вес. % SMPT (Pierce). После инкубирования в течение 1 ч. 0,8 мг SMPT-PBAVE добавляли к 400 мкл изотонического раствора глюкозы, содержащего 5 мМ TAPS (рН 9). К этому раствору добавляли 50 мкг SATA-модифицированной siRNA. Для экспериментов по изучению зависимости "доза-эффект", где [PBAVE] была постоянной, добавляют различные количества siRNA. Смесь затем инкубируют в течение 16 ч. К раствору затем добавляют 5,6 мг свободного основания HEPES, а после этого смесь 3,7 мг CDM-NAG и 1,9 мг CDM-PEG. Раствор затем инкубируют в течение по меньшей мере 1 ч. при комнатной температуре перед инъекцией. CDM-PEG и CDM-NAG синтезируют из хлорангидрида, образующегося посредством применения оксалилхлорида. К хлорангидриду добавляют 1,1 молярного эквивалента монометилового эфира полиэтиленгликоля (средняя молекулярная масса 450) с образованием CDM-PEG или (аминоэтокси)этокси-2-(ацетиламино)-2-дезокси-β-D-глюкопиранозида с образованием CDM-NAG. Конечный продукт очищают с помощью обращенно-фазовой HPLC с 0,1% TFA в градиенте вода/ацетонитрил. Мышам доставляли приблизительно 25-50 мкг siRNA. Систему по Rozema и соавт. можно применять для доставки системы CRISPR-Cas по настоящему изобретению в печень, например, предусматривая дозу от приблизительно 50 до приблизительно 200 мг CRISPR-Cas для доставки человеку.
Кость
Oakes and Lieberman (Clin Orthop Relat Res. 2000 Oct; (379 Suppl): S101-12) обсуждают доставку генов в кость. Путем переноса генов в клетки в конкретном анатомическом участке можно использовать остеоиндуктивные свойства факторов роста в физиологических дозах в течение длительного периода для содействия более значительной реакции заживления. Конкретный анатомический участок, качество кости и мягкотканный футляр влияют на выбор целевых клеток для регионарной генной терапии. Векторы для генной терапии, доставляемые в обрабатываемый участок в остеоиндуктивных носителях, давали многообещающие результаты. Некоторые исследователи показали впечатляющие результаты, применяя регионарную генную терапию в животных моделях ex vivo и in vivo. Такую систему можно применять для доставки системы CRISPR-Cas в кость и/или адаптировать к ней.
Головной мозг
Варианты доставки в головной мозг включают инкапсулирование фермента CRISPR и направляющей РНК в форме ДНК или РНК в липосомы и конъюгацию с "молекулярными троянскими конями" для доставки через гематоэнцефалический барьер (ВВВ). Было показано, что "молекулярные троянские кони" являются эффективными для доставки векторов экспрессии B-gal в головной мозг отличных от человека приматов. Этот же подход можно применять для доставки векторов, содержащих фермент CRISPR и направляющую РНК. Например, Xia CF and Boado RJ, Pardridge WM ("Antibody-mediated targeting of siRNA via the human insulin receptor using avidin-biotin technology." Mol Pharm. 2009 May-Jun; 6(3): 747-51. dot: 10.1021/mp800194) описывают возможность доставки коротких интерферирующих РНК (siRNA) в клетки в культуре и in vivo в случае комбинированного применения моноклонального антитела (mAb), специфичного к рецептору, и авидин-биотиновой технологии. Авторы также сообщают, что, поскольку в случае применения авидин-биотиновой технологии связь между нацеливающим mAb и siRNA является устойчивой, после внутривенного введения целенаправленно воздействующей siRNA наблюдаются эффекты РНКи in vivo в отдаленных участках, таких как головной мозг.
Zhang et al. (Mol Ther. 2003 Jan; 7(1): 11-8.)) описывают, как экспрессионные плазмиды, кодирующие репортеры, такие как люцифераза, инкапсулировали во внутреннее пространство "искусственного вируса", включающего пегилированную иммунолипосому размером 85 нм, нацеливаемую на головной мозг макака-резуса in vivo с помощью моноклонального антитела (MAb) к рецептору инсулина человека (HIR). MAb к HIR. позволяет липосоме, несущей экзогенный ген, подвергаться трансцитозу через гематоэнцефалический барьер и эндоцитозу через плазматическую мембрану нейронов после внутривенной инъекции. Уровень экспрессии гена люциферазы в головном мозге у макака-резуса был в 50 раз выше по сравнению с крысой. Широко распространенная экспрессия гена бета-галактозидазы в нейронах головного мозга приматов была продемонстрирована с помощью как гистохимического анализа, так и конфокальной микроскопии. Авторы указывают, что данный подход позволяет достичь обратимой экспрессии трансгена у взрослых животных в течение 24 часов. Соответственно, применение иммунолипосом является предпочтительным. Их можно использовать в сочетании с антителами для нацеливания на конкретные ткани или белки клеточной поверхности.
Также являются предпочтительными другие средства доставки РНК, как, например, посредством наночастиц (Cho, S., Goldberg, M., Son, S., Xu, Q., Yang, F., Mei, Y., Bogatyrev, S., Langer, R. and Anderson, D., Lipid-like nanoparticles for small interfering RNA delivery to endothelial cells. Advanced Functional Materials, 19: 3112-3118, 2010) или экзосом (Schroeder, A., Levins, С., Cortez, С., Langer, R., and Anderson, D., Lipid-based nanotherapeutics for siRNA delivery. Journal of Internal Medicine, 267: 9-21, 2010, PMID: 20059641).
Действительно, было показано, что экзосомы являются особенно применимыми в доставке siRNA, системы, в некоторой степени сходной с системой CRISPR. Например, El-Andaloussi S, et al. ("Exosome-mediated доставка of siRNA in vitro and in vivo." Nat Protoc. 2012 Dec; 7(12): 2112-26. dot: 10.1038/nprot.2012.131. Epub 2012Nov 15.) описывают, как экзосомы, являющиеся перспективными инструментами доставки лекарственных средств через различные биологические барьеры, можно приспособить для доставки siRNA in vitro и in vivo. Данный подход заключается в создании нацеленных экзосом посредством трансфекции вектором экспрессии, содержащим экзосомный белок, слитый с пептидным лигандом. Экзосомы затем очищают от надосадочной жидкости с трансфицированными клетками и характеризуют, а затем в экзосомы загружают siRNA. Доставку или введение согласно настоящему изобретению можно осуществлять с помощью экзосом, в частности, без ограничения, в головной мозг.
Витамин Е (α-токоферол) можно конъюгировать с CRISPR-Cas и доставлять в головной мозг вместе с липопротеином высокой плотности (HDL), например, аналогично тому, как это было выполнено Uno et al. (HUMAN GENE THERAPY 22:711-719 (June 2011)) для доставки коротких интерферирующих РНК (siRNA) в головной мозг. Мышам проводили инфузию с помощью осмотических мининасосов (модель 1007D; Alzet, Купертино, Калифорния), наполненных фосфатно-солевым буфером (PBS) или свободной Toc-siBACE или Toc-siBACE/HDL, и соединенных с набором 3 для инфузий в головной мозг (Alzet). Канюлю для инфузий в головной мозг размещали приблизительно на 0,5 мм кзади от брегмы на средней линии для инфузий в дорсальную часть третьего желудочка. Uno и соавт. обнаружили, что всего 3 нмоля Toc-siRNA с HDL в том же способе ICV инфузий могут индуцировать сравнимую степень целевого снижения. Аналогичная доза CRISPR-Cas, конъюгированной с α-токоферолом и вводимой совместно с HDL, нацеливающейся на головной мозг, может предусматриваться в настоящем изобретении для людей, например, может предусматриваться от приблизительно 3 нмоль до приблизительно 3 мкмоль CRISPR-Cas, нацеливающейся на головной мозг.
Zou et al. ((HUMAN GENE THERAPY 22: 465-475 (April 2011)) описывают способ опосредованной лентивирусами доставки коротких шпилечных РНК, целенаправленно воздействующих на PKCγ, для in vivo сайленсинга генов в спинном мозге крыс. Zou и соавт. вводили приблизительно 10 мкл рекомбинантного лентивируса с титром 1×109 трансдуцирующих единиц (ТЕ)/мл с помощью интратекального катетера. Аналогичная доза экспрессируемой CRISPR-Cas в лентивирусном векторе, нацеливающемся на головной мозг, может предусматриваться в настоящем изобретении для людей, например, может предусматриваться приблизительно 10-50 мл CRISPR-Cas, нацеливающейся на головной мозг, в лентивирусе с титром 1×109 трансдуцирующих единиц (ТЕ)/мл.
Направленная деления, терапевтические применения
Направленная деления генов является предпочтительной. Примеры проиллюстрированы в примере 18. Таким образом, предпочтительными являются гены, вовлеченные в биосинтез холестерина, биосинтез жирных кислот и другие метаболические нарушения, гены, кодирующие неправильно свернутые белки, вовлеченные в амилоидоз и другие заболевания, онкогены, приводящие к трансформации клеток, гены латентных вирусов и гены, приводящие к доминантно-негативным нарушениям среди прочих нарушений. Как проиллюстрировано здесь, заявители отдают предпочтение доставке гена системы CRISPR-Cas в ткань печени, головного мозга, глазную, эпителиальную, кроветворную или иную ткань субъекта или пациента, нуждающегося в этом, страдающего от метаболических нарушений, амилоидоза и заболеваний, связанных с агрегацией белков, трансформации клеток, возникающей в результате генетических мутаций и транслокаций, доминантно-негативных эффектов генных мутаций, латентных вирусных инфекций и других связанных симптомов, с использованием либо вирусных систем доставки, либо систем доставки на основе наночастиц.
Пути применения системы CRISPR-Cas в терапии, в том числе при глаукоме, амилоидозе и болезни Хантингтона. Они проиллюстрированы в примере 20, и особенности, описанные там, являются предпочтительными по отдельности или в комбинации.
Другой пример заболевания, характеризующегося экспансией полиглутаминовых повторов, которое можно лечить с помощью настоящего изобретения, включает спинально-церебеллярную атаксию 1 типа (SCA1). При внутримозжечковой инъекции векторы на основе рекомбинантного аденоассоциированного вируса (AAV), экспрессирующие короткие шпилечные РНК, существенно улучшают координацию движений, восстанавливают морфологические характеристики мозжечка и устраняют характерные включения атаксина 1 в клетках Пуркинье мышей с SCA1 (см., например, Xia et al., Nature Medicine, Vol. 10, No. 8, Aug. 2004). В частности, предпочтительными являются векторы AAV1 и AAV5, и желаемыми являются титры AAV, составляющие приблизительно 1×1012 геномов векторов/мл.
В качестве примера, можно лечить или предупреждать хроническую инфекцию, вызываемую HIV-1. Для выполнения этой задачи можно создать направляющие РНК для CRISPR-Cas, которые осуществляют нацеливание на подавляющее большинство геномов HIV-1 с учетом вариантов штаммов HIV-1 для максимального охвата и эффективности. Можно осуществлять доставку системы CRISPR-Cas с помощью общепринятой, опосредованной аденовирусом или лентивирусом инфекции иммунной системы хозяина. В зависимости от подхода иммунные клетки хозяина могут быть а) выделены, трансдуцированы CRISPR-Cas, подвергнуты отбору и повторно введены хозяину; или b) трансдуцированы in vivo путем системной доставки системы CRISPR-Cas. Первый подход обеспечивает возможность получения популяции устойчивых иммунных клеток, тогда как второй, по всей вероятности, целенаправленно воздействует на резервуары латентного вируса в хозяине. Более подробно это обсуждается в разделе "Примеры".
В другом примере публикация заявки на патент США №20130171732, закрепленная за Sangamo BioSciences, Inc., относится к вставке в геном трансгена, действующего против HTV, способы которой можно применять к системе CRISPR-Cas по настоящему изобретению. В другом варианте осуществления можно целенаправленно воздействовать на ген CXCR4, и систему TALE согласно публикации заявки на патент США №20100291048, закрепленной за Sangamo BioSciences, Inc., можно модифицировать с получением системы CRISPR-Cas по настоящему изобретению. Способ согласно публикациям заявок на патенты США №№20130137104 и 20130122591, закрепленным за Sangamo BioSciences, me., и публикации заявки на патент США №20100146651, закрепленной за Cellectis, может быть в более общем смысле применимым к экспрессии трансгена, поскольку он включает модификацию локуса гена гипоксантингуанинфосфорибозилтрансферазы (HPRT) для повышения частоты модификации гена.
В настоящем изобретении также предусматривается создание клеточной библиотеки нокаутных генов. Каждая клетка может иметь один нокаутный ген. Это проиллюстрировано в примере 23.
Можно получить библиотеку клеток ES, в которой каждая клетка имеет один нокаутный ген, и в полной библиотеке клеток ES все без исключения гены будут нокаутными. Эта библиотека применима для скрининга функции гена в клеточных процессах, а также при заболеваниях. Для получения этой клеточной библиотеки заявители могут интегрировать Cas9, управляемый индуцируемым промотором (например, индуцируемым доксициклином промотором), в клетку ES. Кроме того, можно интегрировать одну направляющую РНК, осуществляющую нацеливание на конкретный ген в клетке ES. Для создания библиотеки клеток ES можно просто смешать клетки ES с библиотекой генов, кодирующих направляющие РНК, которые осуществляют нацеливание на каждый ген в геноме человека. Сначала можно ввести один сайт attB для ВхВ1 в локус AAVS1 в человеческих клетках ES. Затем можно использовать интегразу ВхВ1 для облегчения интеграции отдельных генов направляющих РНК в сайт attB для ВхВ1 в локусе AAVS1. Для облегчения интеграции каждый ген направляющей РНК можно размещать на плазмиде, которая несет один сайт attP. Таким образом, ВхВ1 будет рекомбинировать сайт attB в геноме с сайтом attP на плазмиде, содержащей направляющую РНК. Для создания клеточной библиотеки можно взять библиотеку клеток, имеющих отдельные интегрированные направляющие РНК, и индуцировать экспрессию Cas9. После индукции Cas9 опосредует двухнитевой разрыв в сайтах, определенных направляющей РНК.
Длительное введение белковых терапевтических средств может вызвать нежелательные иммунные ответы на данный белок. Иммуногенность белковых лекарственных средств может объясняться наличием нескольких иммунодоминантных эпитопов для Т-лимфоцитов-хелперов (HTL). Путем снижения аффинности связывания этих эпитопов для HTL, содержащихся в данных белках, с МНС можно создавать лекарственные средства с более низкой иммуногенностью (Tangri S, et al. ("Rationally engineered therapeutic proteins with reduced immunogenicity" J Immunol. 2005 Mar 15; 174(6): 3187-96.) В настоящем изобретении иммуногенность фермента CRISPR, в частности, можно снизить, следуя подходу, впервые изложенному Tangri и соавт. в отношении эритропоэтина и впоследствии получившему развитие. Соответственно, для снижения иммуногенности фермента CRISPR (например, Cas9) у вида-хозяина (человека или другого вида) можно применять направленную эволюцию или рациональное проектирование.
В примере 28 заявители применяют 3 направляющие РНК, представляющие интерес, и могут визуализировать эффективное расщепление ДНК in vivo, имеющее место лишь в небольшой субпопуляции клеток. В сущности, здесь заявители показали направленное расщепление in vivo. В частности, это предоставляет подтверждение концепции, заключающейся в том, что у высших организмов, таких как млекопитающие, также можно достичь специфичного целенаправленного воздействия. Это также подчеркивает множественность аспекта в том отношении, что несколько направляющих последовательностей (т.е. воздействующих на отдельные мишени) можно использовать одновременно (в смысле совместной доставки). Другими словами, заявители применяли многосторонний подход с несколькими различными последовательностями, целенаправленно воздействующими в одно и то же время, но независимо.
Подходящий пример протокола получения AAV, предпочтительного вектора согласно настоящему изобретению, приведен в примере 34.
Нарушения, связанные с экспансией тринуклеотидных повторов, являются предпочтительными состояниями для лечения. Они также проиллюстрированы в данном документе.
Например, в публикации заявки на патент США №20110016540 описывается применение нуклеаз с "цинковыми пальцами" для генетической модификации клеток, животных и белков, ассоциированных с нарушениями, связанными с экспансией тринуклеотидных повторов. Нарушения, связанные с экспансией тринуклеотидных повторов, являются комплексными прогрессирующими нарушениями, затрагивающими биологию развития нервной системы и часто нарушающими когнитивные функции, а также сенсомоторные функции.
Белки, связанные с экспансией тринуклеотидных повторов, представляют собой разнородную группу белков, ассоциированных с восприимчивостью к развитию нарушения, связанного с экспансией тринуклеотидных повторов, наличием нарушения, связанного с экспансией тринуклеотидных повторов, тяжестью нарушения, связанного с экспансией тринуклеотидных повторов, или любой их комбинацией. Нарушения, связанные с экспансией тринуклеотидных повторов, подразделяют на две категории, определяемые типом повтора. Наиболее распространенным повтором является триплет CAG, который в случае наличия в кодирующем участке гена кодирует аминокислоту глутамин (Q). Таким образом, эти нарушения называются нарушениями, связанными с экспансией полиглутаминовых повторов (поли-Q), и включают следующие заболевания: болезнь Хантингтона (HD); спинобульбарную мышечную атрофию (SBMA); формы спинально-церебеллярной атаксии (SCA типов 1, 2, 3, 6, 7 и 17) и дентато-рубро-паллидо-льюисову атрофию (DRPLA). Остальные нарушения, связанные с экспансией тринуклеотидных повторов, при которых триплет CAG не вовлечен либо триплет CAG находится не в кодирующем участке гена, называются, таким образом, нарушениями, не связанными с экспансией полиглутаминовых повторов. Нарушения, не связанные с экспансией полиглутаминовых повторов, включают синдром ломкой Х-хромосомы (FRAXA); синдром умственной отсталости, сцепленный с ломкой Х-хромосомой (FRAXE); атаксию Фридрейха (FRDA); миотоническую дистрофию (DM) и формы спинально-церебеллярной атаксии (SCA типов 8 и 12).
Белки, ассоциированные с нарушениями, связанными с экспансией тринуклеотидных повторов, обычно выбирают на основании экспериментального изучения взаимосвязи белка, ассоциированного с нарушением, связанным с экспансией тринуклеотидных повторов, и нарушения, связанного с экспансией тринуклеотидных повторов. Например, скорость образования или концентрация в кровотоке белка, ассоциированного с нарушением, связанным с экспансией тринуклеотидных повторов, может быть повышенной или пониженной в популяции, имеющей нарушение, связанное с экспансией тринуклеотидных повторов, по сравнению с популяцией, не имеющей нарушения, связанного с экспансией тринуклеотидных повторов. Различия по уровням белка можно оценить при помощи протеомных методик, в том числе без ограничения вестерн-блоттинга, иммуногистохимического окрашивания, твердофазного иммуноферментного анализа (ELISA) и масс-спектрометрии. В альтернативном случае, белки, связанные с сердечно-сосудистым заболеванием, можно идентифицировать путем получения профилей генной экспрессии для генов, кодирующих белки, при помощи методик геномного анализа, в том числе без ограничения микроматричного анализа ДНК, последовательного анализа экспрессии генов (SAGE) и количественной полимеразной цепной реакции в режиме реального времени (Q-PCR).
Неограничивающие примеры белков, ассоциированных с нарушениями, связанными с экспансией тринуклеотидных повторов, включают AR (андрогенный рецептор), FMR1 (белок 1, ассоциированный с умственной отсталостью, сцепленной с ломкой Х-хромосомой), НТТ (хантингтин), DMPK (протеинкиназу, ассоциированную с миотонической дистрофией), FXN (фратаксин), ATXN2 (атаксин 2), ATN1 (атрофин 1), FEN1 (структуроспецифичную флэп-эндонуклеазу 1), TNRC6A (белок, кодируемый геном 6А, содержащим тринуклеотидные повторы), PABPN1 (ядерный поли(А)-связывающий белок 1), JPH3 (юнктофилин 3), MED15 (субъединицу 15 медиаторного комплекса), ATXN1 (атаксин 1), ATXN3 (атаксин 3), ТВР (ТАТА-бокс-связывающий белок), CACNA1A (альфа-1A-субъединицу потенциал-зависимого кальциевого канала P/Q-типа), ATXN80S (белок, синтезируемый с противоположной нити ATXN8 (не кодирующей белок)), PPP2R2B (бета-изоформу регуляторной субъединицы В протеинфосфатазы 2), ATXN7 (атаксин 7), TNRC6B (белок, кодируемый геном 6 В, содержащим тринуклеотидные повторы), TNRC6C (белок, кодируемый геном 6С, содержащим тринуклеотидные повторы), CELF3 (CUGBP, член 3 семейства Elav-подобных белков), MAB21L1 (mab-21-подобный белок 1 (С. elegans)), MSH2 (гомолог 2 mutS, ассоциированный с неполипозным колоректальным раком типа 1 (Е. coli)), TMEM185A (трансмембранный белок 185А), SIX5 (белок, кодируемый гомеобоксом 5 SEX), CNPY3 (гомолог Canopy 3 (данио-рерио)), FRAXE (белок, ассоциированный с "редким" ломким сайтом, проявляющимся при недостатке фолиевой кислоты, fra(X)(q28) Е), GNB2 (бета-полипептид 2 белка, связывающего гуаниновые нуклеотиды (G-белка)), RPL14 (рибосомный белок L14), ATXN8 (атаксин 8), INSR (инсулиновый рецептор), TTR (транстиретин), ЕР400 (E1A-связывающий белок р400), GIGYF2 (белок GYF 2, взаимодействующий с GRB10), OGG1 (8-оксогуанин-ДНК-гликозилазу), STC1 (станниокальцин 1), CNDP1 (карнозиндипептидазу 1 (металлопептидазу семейства М20)), C10orf2 (белок, кодируемый открытой рамкой считывания 2 хромосомы 10), MAML3 (mastermind-подобный белок 3 (Drosophila)), DKC1 (белок 1, ассоциированный с врожденным дискератозом, дискерин), PAXIP1 (белок 1, взаимодействующий с PAX (с доменом активации транскрипции)), CASK (кальпий/кальмодулин-зависимую сериновую протеинкиназу (семейства MAGUK)), МАРТ (белок tau, ассоциированный с микротрубочками), SP1 (фактор транскрипции Sp1), POLG (полимеразу гамма (ДНК-направленную)), AFF2 (член 2 семейства AF4/FMR2), THBS1 (тромбоспондин 1), ТР53 (опухолевый белок р53), ESR1 (эстрогеновый рецептор 1), CGGBP1 (белок 1, связывающий триплетный повтор CGG), ABT1 (активатор 1 базальной транскрипции), KLK3 (родственную калликреину пептидазу 3), PRNP (белок приона), JUN (онкоген jun), KCNN3 (кальций-активируемый калиевый канал средней/малой проводимости, представитель 3 подсемейства N), ВАХ (BCL2-ассоциированный белок X), FRAXA (белок, ассоциированный с "редким" ломким сайтом, проявляющимся при недостатке фолиевой кислоты, fra(X)(q27.3) А (макроорхидизм, умственная отсталость)), KBTBD10 (белок 10, содержащий повтор Kelch и домен ВТВ (POZ)), MBNL1 (muscleblind-подобный белок (Drosophila)), RAD51 (гомолог RAD51 (гомолог RecA, E.coli) (S. cerevisiae)), NCOA3 (коактиватор 3 ядерных рецепторов), ERDA1 (белок с экспансией повторяющихся доменов, CAG/CTG 1), TSC1 (белок 1, ассоциированный с туберозным склерозом), СОМР (олигомерный матриксный белок хряща), GCLC (каталитическую субъединицу глутаматпистеинлигазы), RRAD (Ras-родственный белок, ассоциированный с сахарным диабетом), MSH3 (гомолог 3 mutS (E. coli)), DRD2 (дофаминовый рецептор D2), CD44 (молекулу CD44 (система групп крови Indian)), CTCF (СССТС-связывающий фактор (белок с "цинковыми пальцами")), CCND1 (циклин D1), CLSPN (гомолог класпина (Xenopus laevis)), MEF2A (энхансерный фактор 2А миоцитов), PTPRU (протеинтирозинфосфатазу рецепторного типа U), GAPDH (глицеральдегид-3-фосфатдегидрогеназу), TRIM22 (белок 22, содержащий тройной мотив), WT1 (белок 1 опухоли Вильмса), AHR (арил-углеводородный рецептор), GPX1 (глутатионпероксидазу 1), ТРМТ (тиопурин-S-метилтрансферазу), NDP (белок, ассоциированный с болезнью Норри (псевдоглиомой)), ARX (белок, кодируемый гомеобоксом гена, родственного aristaless), MUS81 (гомолог эндонуклеазы MUS81 (S. cerevisiae)), TYR (тирозиназу (глазокожный альбинизм IA)), EGR1 (белок 1 раннего ростового ответа), UNG (урацил-ДНК-гликозилазу), NUMBL (белок, подобный гомологу numb (Drosophila)), FABP2 (белок 2, связывающий жирные кислоты в кишечнике), EN2 (белок, кодируемый гомеобоксом engrailed 2), CRYGC (гамма-С-кристаллин), SRP14 (гомологичный РНК-связывающий белок Alu (размером 14 кДа из частицы узнавания сигнала)), CRYGB (гамма-В-кристаллин), PDCD1 (белок 1 запрограммированной гибели клеток), НОХА1 (белок, кодируемый гомеобоксом A1), ATXN2L (атаксин-2-подобный белок), PMS2 (PMS2, белок 2, противодействующий повышению уровня постмейотической сегрегации (S. cerevisiae)), GLA (альфа-галактозидазу), CBL (белок, кодируемый последовательностью, трансформирующей с экотропным ретровирусом Cas-Br-М (мышей)), FTH1 (полипептид 1 тяжелой субъединицы ферритина), IL12RB2 (бета-2-субъединицу рецептора интерлейкина 12), ОТХ2 (белок, кодируемый гомеобоксом orthodenticle 2), НОХА5 (белок, кодируемый гомеобоксом А5), POLG2 (вспомогательную гамма-2-субъединицу полимеразы (ДНК-направленной)), DLX2 (белок, кодируемый гомеобоксом distal-less 2), SIRPA (сигнально-регуляторный белок альфа), ОТХ1 (белок, кодируемый гомеобоксом orthodenticle 1), AHRR (репрессор арил-углеводородного рецептора), MANF (мезэнцефальный нейротрофический фактор, происходящий из астроцитов), ТМЕМ158 (трансмембранный белок 158 (ген/псевдоген)) и ENSG00000078687.
Предпочтительные белки, ассоциирующиеся с нарушениями, связанными с экспансиями тринуклеотидных повторов, включают НТТ (хантингтин), AR (андрогенный рецептор), FXN (фратаксин), Atxn3 (атаксин), Atxn1 (атаксин), Atxn2 (атаксин), Atxn7 (атаксин), Atxn10 (атаксин), DMPK (миотонин-протеинкиназу), Atn1 (атрофин 1), СВР (creb-связывающий белок), VLDLR (рецептор липопротеинов очень низкой плотности) и их любую комбинацию.
В соответствии с другим аспектом предусматривается способ генной терапии для лечения субъекта, имеющего мутацию в гене CFTR, который включает введение терапевтически эффективного количества частиц с CRISPR-Cas для генной терапии, необязательно посредством биологически совместимого фармацевтического носителя, в клетки субъекта. Предпочтительно целевая ДНК содержит мутацию deltaF508. Обычно предпочтительно, чтобы у мутантов восстанавливался дикий тип. В этом случае мутация представляет собой делецию трех нуклеотидов, включающих в себя кодон фенилаланина (F), в положении 508. Соответственно, репарация в данном случае требует повторного введения мутанту недостающего кодона.
Для реализации этой стратегии репарации генов предпочтительно, чтобы векторную систему на основе аденовируса/AAV вводили в клетку-хозяина, в клетки или пациенту. Система предпочтительно содержит Cas9 (или никазу Cas9) и направляющую РНК вместе с векторной системой на основе аденовируса/AAV, содержащей матрицу для репарации путем гомологичной рекомбинации, содержащую остаток F508. Ее можно вводить субъекту посредством одного из обсуждаемых ранее способов доставки. Система CRISPR-Cas может направляться химерной направляющей РНК для дельта-508 CFTR. Она целенаправленно воздействует на конкретный сайт локуса генома CFTR, подлежащий внесению одноцепочечного разрыва или расщеплению. После расщепления матрицу для репарации вставляют в сайт расщепления посредством гомологичной рекомбинации, исправляющей делению, которая приводит к муковисцидозу или вызывает связанные с муковисцидозом симптомы. Данную стратегию для управления доставкой и обеспечения системного встраивания систем CRISPR с помощью соответствующих направляющих РНК можно использовать для целенаправленного воздействия на генные мутации, чтобы редактировать или проводить иного рода манипуляции с генами, которые вызывают метаболические заболевания и нарушения, заболевания и нарушения печени, почек и заболевания и нарушения, связанные с белками, такие как приведенные в таблице В.
Редактирование генома
Системы CRISPR/Cas9 по настоящему изобретению можно применять для коррекции генетических мутаций, попытки которой с ограниченным успехом ранее предпринимались с применением TALEN и ZFN. Например, в опубликованной заявке Duke University WO 2013163628 A2 "Генетическая коррекция мутантных генов" описаны попытки коррекции, например, мутации по типу сдвига рамки считывания, которая вызывает появление преждевременного стоп-кодона и усечение продукта гена, которую можно откорректировать посредством опосредованного нуклеазами негомологичного соединения концов, как, например, обуславливающей мышечную дистрофию Дюшенна ("DMD"), рецессивное смертельное сцепленное с Х-хромосомой нарушение, приводящее к мышечной дегенерации в связи с мутациями гена дистрофина. Большинство мутаций гена дистрофина, вызывающих DMD, представляют собой делеции экзонов, нарушающие рамку считывания и вызывающие преждевременную терминацию трансляции гена дистрофина. Дистрофии представляет собой цитоплазматический белок, обеспечивающий стабильность структуры дистрогликанового комплекса клеточной мембраны, отвечающего за регуляцию целостности и функционирования мышечных клеток. Ген дистрофина или "ген DMD", как взаимозаменяемо используется в данном документе, образован 2,2 миллиона пар оснований в локусе Хр21. Размер первичного транскрипта составляет приблизительно 2400 т.п.о., при этом размер зрелой мРНК составляет приблизительно 14 п.о. 79 экзонов кодируют белок, образованный более 3500 аминокислотами. Экзон 51 часто является смежным с положениями делеции, нарушающих рамку считывания, у пациентов с DMD, и в клинических испытаниях на него был направлен пропуск экзона, основанный на применении олигонуклеотидов. Недавно в клиническом испытании с пропуском экзона 51 с помощью соединения этерлипсена сообщали о значительном положительном функциональном эффекте в течение 48 недель со средним количеством дистрофин-положительных волокон 47% по сравнению с исходным уровнем. Мутации в экзоне 51 идеально подходят для устойчивой коррекции посредством редактирования генома на основе NHEJ.
Способы согласно публикации заявки на патент США №20130145487, закрепленной за Cellectis, которые относятся к вариантам мегануклеаз для расщепления целевой последовательности гена дистрофина человека (DMD), также можно модифицировать для системы CRISPR-Cas по настоящему изобретению.
Кровь
Настоящее изобретение также предусматривает доставку системы CRISPR-Cas в кровь.
Плазменные экзосомы по Wahlgren et al. (Nucleic Acids Research, 2012, Vol. 40, No. 17 e130) были описаны ранее и могут быть использованы для доставки системы CRISPR-Cas в кровь.
Система CRISPR-Cas по настоящему изобретению также предусматривается для лечения гемоглобинопатий, таких как формы талассемии и серповидно-клеточная анемия. См., например, международную публикацию заявки на патент WO 2013/126794 в отношении потенциальных мишеней, на которые может целенаправленно воздействовать система CRISPR-Cas по настоящему изобретению.
Публикации заявок на патенты США №№20110225664, 20110091441, 20100229252, 20090271881 и 20090222937, закрепленные за Cellectis, относятся к вариантам CREI, где по меньшей мере один из двух мономеров I-CreI имеет по меньшей мере две замены, по одной в каждом из двух функциональных субдоменов сердцевинного домена LAGLIDADG, расположенных, соответственно, в положениях 26-40 и 44-77 I-CreI, при этом указанный вариант способен расщеплять целевую последовательность ДНК гена гамма-цепи рецептора интерлейкина-2 человека (IL2RG), также называемого геном общей гамма-цепи рецепторов цитокинов или геном гамма-С. Целевые последовательности, указанные в публикациях заявок на патенты США №№20110225664, 20110091441, 20100229252, 20090271881 и 20090222937, можно использовать для системы CRISPR-Cas по настоящему изобретению.
Тяжелый комбинированный иммунодефицит (SCID) возникает в результате нарушения созревания Т-лимфоцитов, во всех случаях ассоциированного с нарушением функционирования В-лимфоцитов (Cavazzana-Calvo et al., Annu. Rev. Med., 2005, 56, 585-602; Fischer et al., Immunol. Rev., 2005, 203, 98-109). Общая заболеваемость по оценкам составляет 1 на 75000 родившихся. Пациенты с нелеченым SCID подвержены множественным инфекциям, вызываемым условно-патогенными микроорганизмами, и живут, как правило, не более одного года. SCID можно лечить путем аллогенного переноса гемопоэтических стволовых клеток от донора-родственника. Степень гистосовместимости с донором может варьировать в широких пределах. В случае аденозиндезаминазной (ADA) недостаточности, одной из форм SCID, пациентов можно лечить с помощью инъекции рекомбинантного фермента аденозиндезаминазы.
Поскольку было показано, что ген ADA у пациентов с SCID является мутантным (Giblett et al., Lancet, 1972, 2, 1067-1069), были идентифицированы некоторые другие гены, вовлеченные в SCID (Cavazzana-Calvo et al., Annu. Rev. Med., 2005, 56, 585-602; Fischer et al., Immunol. Rev., 2005, 203, 98-109). Существуют четыре основные причины SCID: (i) Наиболее часто встречающуюся форму SCID, SCID-X1 (SCID, сцепленный с X-хромосомой, или X-SCID), вызывает мутация в гене IL2RG, которая приводит к отсутствию зрелых Т-лимфопитов и NK-клеток. IL2RG кодирует белок гамма-С (Noguchi, et al., Cell, 1993, 73, 147-157), общий компонент по меньшей мере пяти рецепторных комплексов интерлейкинов. Данные рецепторы активируют несколько мишеней с помощью киназы JAK3 (Macchi et al., Nature, 1995, 377, 65-68), инактивация которой приводит к возникновению того же синдрома, что и инактивация гамма-С. (ii) Мутация в гене ADA приводит к нарушению метаболизма пуринов, вызывающему гибель предшественников лимфоцитов, что, в свою очередь, приводит к кажущемуся отсутствию В-, Т- и NK-клеток. (iii) V(D)J-рекомбинация является существенным этапом созревания иммуноглобулинов и рецепторов Т-лимфопитов (TCR). Мутации в генах, активирующих рекомбинацию, 1 и 2 (RAG1 и RAG2) и Artemis, трех генах, участвующих в этом процессе, приводят к отсутствию зрелых Т- и В-лимфоцитов. (iv) Также сообщали о мутациях в других генах, таких как CD45, участвующих в специфичной передаче сигналов в Т-клетках, хотя они представляют меньшинство случаев (Cavazzana-Calvo et al., Annu. Rev. Med., 2005, 56, 585-602; Fischer et al., Immunol. Rev., 2005, 203, 98-109).
С тех пор, как были выявлены их генетические основы, различные формы SCID стали модельными для подходов к генной терапии (Fischer et al., Immunol. Rev., 2005, 203, 98-109) по двум основным причинам. Во-первых, как и при всех заболеваниях крови, может быть предусмотрено лечение ex vivo. Можно выделить гемопоэтические стволовые клетки (HSC) из костного мозга и сохранять их свойства плюрипотентности в течение нескольких клеточных делений. Таким образом, их можно обрабатывать in vitro, а затем повторно инъецировать пациенту, где они повторно заселяют костный мозг. Во-вторых, поскольку созревание лимфоцитов у пациентов с SCID ухудшено, скорректированные клетки имеют селективное преимущество. Таким образом, небольшое количество скорректированных клеток может восстановить функционирование иммунной системы. Эта гипотеза была неоднократно подтверждена (i) частичным восстановлением иммунных функций, связанным с реверсией мутаций у пациентов с SCID (Hirschhorn et al., Nat. Genet, 1996, 13, 290-295; Stephan et al., N. Engl. J. Med., 1996, 335, 1563-1567; Bousso et al., Proc. Natl., Acad. Sci. USA, 2000, 97, 274-278; Wada et al., Proc. Natl. Acad. Sci. USA, 2001, 98, 8697-8702; Nishikomori et al., Blood, 2004, 103, 4565-4572), (ii) коррекцией форм недостаточности SCID-X1 in vitro в гемопоэтических клетках (Candotti et al., Blood, 1996, 87, 3097-3102; Cavazzana-Calvo et al., Blood, 1996, Blood, 88, 3901-3909; Taylor et al., Blood, 1996, 87, 3103-3107; Hacein-Bey et al., Blood, 1998, 92, 4090-4097), (iii) коррекцией форм недостаточности SCID-X1 (Soudais et al., Blood, 2000, 95, 3071-3077; Tsai et al., Blood, 2002, 100, 72-79), JAK-3 (Bunting et al., Nat. Med., 1998, 4, 58-64; Bunting et al., Hum. Gene Ther., 2000, 11, 2353-2364) и RAG2 (Yates et al., Blood, 2002, 100, 3942-3949) in vivo в животных моделях и (iv) результатом клинических испытаний генной терапии (Cavazzana-Calvo et al., Science, 2000, 288, 669-672; Aiuti et al., Nat. Med., 2002; 8, 423-425; Caspar et al., Lancet, 2004, 364, 2181-2187).
Публикация заявки на патент США №20110182867, закрепленная за Children's Medical Center Corporation и президентом и членами управляющего совета Гарвардского университета, относится к способам модулирования экспрессии фетального гемоглобина (HbF) и путям ее применения в гемопоэтических клетках-предшественниках с помощью ингибиторов экспрессии или активности BCL11A, таких как интерферирующие РНК (РНКи) и антитела. На мишени, раскрытые в публикации заявки на патент США №20110182867, такие как BCL11A, можно целенаправленно воздействовать с помощью системы CRISPR-Cas по настоящему изобретению для модулирования экспрессии фетального гемоглобина. См. также Bauer et al. (Science 11 October 2013: Vol. 342 no. 6155 pp. 253-257) и Xu et al. (Science 18 November 2011: Vol. 334 no. 6058 pp. 993-996) в отношении дополнительных мишеней BCL11A.
Уши
Настоящее изобретение также предусматривает доставку системы CRISPR-Cas в одно ухо или оба уха.
Исследователи рассматривают вопрос о том, можно ли применять генную терапию для содействия существующим способам лечения глухоты - а именно, применению кохлеарных имплантатов. Глухоту часто вызывают утрата или повреждение волосковых клеток, которые не могут передавать сигналы слуховым нейронам. В таких случаях можно применять кохлеарные имплантаты для обеспечения реакции на звук и передачи электрических сигналов нервным клеткам. Однако эти нейроны часто дегенерируют и подвергаются ретракции отростков в улитке, поскольку пораженные волосковые клетки высвобождают меньше факторов роста.
В заявке на патент США 20120328580 описана инъекция фармацевтической композиции в ухо (например, путем ушного введения), как, например, в просветы улитки (например, в проток улитки, лестницу преддверия и барабанную лестницу улитки), например, с помощью шприца, например, шприца с однократной дозой. Например, одно или несколько соединений, описанных в данном документе, можно вводить путем интратимпанальной инъекции (например, в среднее ухо) и/или инъекций в наружное, среднее и/или внутреннее ухо. Такие способы регулярно применяются в данной области, например, для введения стероидов и антибиотиков в уши людей. Инъекция может осуществлять, например, через круглое окно уха или через капсулу улитки. В данной области известны и другие способы введения во внутреннее ухо (см., например, Salt and Plontke, Drug Discovery Today, 10: 1299-1306, 2005).
В другом способе введения фармацевтическую композицию можно вводить in situ с помощью катетера или насоса. Катетер или насос могут, например, направлять фармацевтическую композицию в просветы улитки, или круглое окно уха, и/или просвет толстой кишки. Иллюстративный аппарат для доставки лекарственных средств и способы, подходящие для введения одного или нескольких соединений, описанных в данном документе, в ухо, например, в ухо человека, описаны McKenna и соавт. (публикация заявки на патент США №2006/0030837) и Jacobsen и соавт. (патент США №7206639). В некоторых вариантах осуществления катетер или насос могут быть расположены, например, в ухе (например, в наружном, среднем и/или внутреннем ухе) пациента во время хирургического вмешательства. В некоторых вариантах осуществления катетер или насос могут быть расположены, например, в ухе (например, в наружном, среднем и/или внутреннем ухе) пациента без необходимости в хирургическом вмешательстве.
Альтернативно или в дополнение, одно или несколько соединений, описанных в данном документе, можно вводить в сочетании с механическим устройством, таким как кохлеарный имплантат или слуховой аппарат, которое носят в наружном ухе. Иллюстративный кохлеарный имплантат, подходящий для применения в настоящем изобретении, описан Edge и соавт. (публикация заявки на патент США №2007/0093878).
В некоторых вариантах осуществления способы введения, описанные выше, можно комбинировать в любом порядке и можно применять одновременно или попеременно.
Альтернативно или в дополнение, настоящее изобретение можно применять согласно любому из способов, одобренных Управлением по контролю качества пищевых продуктов и лекарственных средств, например, описанных в справочнике стандартов CDER, версия номер 004 (доступном по адресу fda.give/cder/dsm/DRG/drg00301.htm).
В целом, способы клеточной терапии, описанные в заявке на патент США 20120328580, можно применять для стимуляции полной или частичной дифференцировки клеток в определенный тип зрелых клеток внутреннего уха (например, в волосковые клетки) или в его направлении in vitro. Клетки, полученные в результате осуществления таких способов, можно затем трансплантировать или имплантировать пациенту, нуждающемуся в таком лечении. Способы культивирования клеток, необходимые для осуществления на практике этих способов, включающие способы идентификации и отбора подходящих типов клеток, способы стимуляции полной или частичной дифференцировки выбранных клеток, способы идентификации полностью или частично дифференцированных типов клеток и способы имплантации полностью или частично дифференцированных клеток, описаны ниже.
Клетки, подходящие для применения в настоящем изобретении, включают без ограничения клетки, способные к полной или частичной дифференцировке в зрелые клетки внутреннего уха, например, в волосковые клетки (например, внутренние и/или наружные волосковые клетки), при контакте, например, in vitro, с одним или несколькими соединениями, описанными в данном документе. Иллюстративные клетки, способные к дифференцировке в волосковые клетки, включают без ограничения стволовые клетки (например, стволовые клетки внутреннего уха, взрослые стволовые клетки, стволовые клетки костномозгового происхождения, эмбриональные стволовые клетки, мезенхимальные стволовые клетки, стволовые клетки кожи, iPS-клетки и стволовые клетки жировой ткани), клетки-предшественники (например, клетки-предшественники внутреннего уха), поддерживающие клетки (например, клетки Дейтерса, столбовые клетки, внутренние фаланговые клетки, тектальные клетки и клетки Гензена) и/или зародышевые клетки. Применение стволовых клеток для замещения чувствительных клеток внутреннего уха описано Li и соавт.(публикация заявки на патент США №2005/0287127) и Li и соавт. (патент США с регистрационным №11/953797). Применение стволовых клеток костномозгового происхождения для замещения чувствительных клеток внутреннего уха описано Edge и соавт. в PCT/US2007/084654. iPS-клетки описаны, например, в Takahashi et al., Cell, Volume 131, Issue 5, Pages 861-872 (2007); Takahashi and Yamanaka, Cell 126, 663-76 (2006); Okita et al., Nature 448, 260-262 (2007); Yu, J. et al., Science 318(5858): 1917-1920 (2007); Nakagawa et al., Nat. Biotechnol. 26: 101-106 (2008) и Zaehres and Scholer, Cell 131(5): 834-835 (2007).
Такие подходящие клетки можно идентифицировать путем анализа (например, качественного или количественного) наличия одного или нескольких тканеспецифичных генов. Например, экспрессию гена можно выявить путем выявления белкового продукта одного или нескольких тканеспецифичных генов. Методики выявления белков включают окрашивание белков (например, с использованием клеточных экстрактов или цельных клеток) с помощью антител к соответствующему антигену. В данном случае соответствующий антиген является белковым продуктом экспрессии тканеспецифичного гена. Хотя, в принципе, меченым может быть первое антитело (т.е. антитело, связывающее антиген), более распространенным (и улучшающим визуализацию) является применение второго антитела, направленного против первого (например, антитела к IgG). Данное второе антитело конъюгируют с флуорохромами, или соответствующими ферментами для колориметрических реакций, или гранулами золота (для электронной микроскопии), или с системой биотин-авидин, так что можно определить местоположение первичного антитела и, следовательно, антигена.
Молекулы CRISPR-Cas по настоящему изобретению можно доставлять в ухо путем непосредственного внесения фармацевтической композиции в наружное ухо с применением модифицированных композиций из опубликованной заявки на патент США 20110142917. В некоторых вариантах осуществления фармацевтическую композицию вносят в наружный слуховой проход. Доставка в ухо может также называться внутриушной или ушной доставкой.
В некоторых вариантах осуществления молекулы РНК по настоящему изобретению доставляют в липосомных составах или составах на основе Lipofectin и т.п., и их можно получить с помощью способов, хорошо известных специалистам в данной области. Такие способы описаны, например, в патентах США №№5593972, 5589466 и 5580859, включенных в данный документ посредством ссылки.
Были разработаны системы доставки, специально предназначенные для повышения эффективности и улучшения доставки siRNA в клетки млекопитающих (см., например, Shen et al FEBS Let. 2003, 539: 111-114; Xia et al., Nat. Biotech. 2002, 20: 1006-1010; Reich et al., Mol. Vision. 2003, 9: 210-216; Sorensen et al., J. Mol. Biol. 2003, 327: 761-766; Lewis et al., Nat. Gen. 2002, 32: 107-108 и Simeoni et al., NAR 2003, 31, 11: 2717-2724), и их можно применять в настоящем изобретении. siRNA недавно успешно применяли для ингибирования экспрессии генов у приматов (см., например, Tolentino et al., Retina 24(4): 660), и их также можно применять в настоящем изобретении.
Qi и соавт. раскрывают способы эффективного введения siRNA во внутреннее ухо через неповрежденное круглое окно путем трансфекции с помощью новой технологии доставки протеидов, которую можно применять в отношении к системе CRISPR-Cas по настоящему изобретению (см., например, Qi et al., Gene Therapy (2013), 1-9). В частности, успешным было применение доменов ТАТ, связывающих двухнитевую РНК (ТАТ-DRBD), с помощью которых можно трансфицировать меченную Су3 siRNA в клетки внутреннего уха, в том числе внутренние и наружные волосковые клетки, ампулярный гребешок, пятно эллиптического мешочка и пятно сферического мешочка, посредством проникновения через неповрежденное круглое окно, для доставки двухнитевых siRNA in vivo для лечения различных болезней внутреннего уха и сохранения слуховой функции. Приблизительно 40 мкл 10 мМ РНК может быть предусмотрено в качестве дозы для введения в ухо.
Согласно Rejali et al. (Hear Res. 2007 Jun; 228(1-2): 180-7), функционирование кохлеарных имплантатов можно улучшить путем надлежащего сохранения нейронов спирального ганглия, которые являются мишенью для электростимуляции имплантатом, и ранее было показано, что нейротрофический фактор головного мозга (BDNF) повышает выживаемость спирального ганглия в ушах с экспериментально индуцированной глухотой. Rejali и соавт. тестировали модифицированную конструкцию электрода кохлеарного имплантата, имеющего покрытие из клеток-фибробластов, трансдуцированных вирусным вектором со вставкой гена BDNF. Для осуществления данного типа переноса генов ex vivo Rejali и соавт. трансдуцировали фибробласты морской свинки аденовирусом со вставкой кассеты с геном BDNF, и определили, что эти клетки секретируют BDNF, а затем прикрепили клетки, секретирующие BDNF, к электроду кохлеарного имплантата с помощью агарозного геля и имплантировали электрод в барабанную лестницу улитки. Rejali и соавт. определили, что электроды с экспрессией BDNF были способны обеспечивать сохранение значительно большего количества нейронов спирального ганглия в базальных витках улитки через 48 дней после имплантации по сравнению с контрольными электродами и демонстрировали возможность осуществления терапии с применением кохлеарных имплантатов в комбинации с переносом генов ех vivo для повышения выживаемости нейронов спирального ганглия. Такую систему можно применять для доставки системы CRISPR-Cas по настоящему изобретению в ухо.
Mukherjea и соавт. (Antioxidants & Redox Signaling, Volume 13, Number 5, 2010) документально подтверждают, что нокдаун NOX3 с помощью короткой интерферирующей (si) РНК нейтрализовал ототоксичность цисплатина, о чем свидетельствует защита ОНС от повреждения и снижение величин сдвига порогов стволомозговых слуховых вызванных потенциалов (ABR). Крысам вводили различные дозы siNOX3 (0,3, 0,6 и 0,9 мкг), и экспрессию NOX3 оценивали с помощью ОТ-ПЦР в режиме реального времени. Наименьшая применяемая доза siRNA для NOX3 (0,3 мкг) не демонстрировала какого-либо ингибирования мРНК NOX3 по сравнению с транстимпанальным введением скремблированной siRNA или отсутствием обработки улиток. Введение более высоких доз siRNA для NOX3 (0,6 и 0,9 мкг), однако, снижало экспрессию NOX3 по сравнению с контрольной скремблированной siRNA. Такую систему можно применять для транстимпанального введения системы CRISPR-Cas по настоящему изобретению в дозе от приблизительно 2 мг до приблизительно 4 мг CRISPR-Cas для введения человеку.
Jung et al. (Molecular Therapy, vol. 21 no. 4, 834-841 apr. 2013) демонстрируют, что уровни Hes5 в эллиптическом мешочке снижались после внесения siRNA и что количество волосковых клеток в этих эллиптических мешочках было значительно большим, чем после контрольной обработки. Данные позволяют предположить, что технология siRNA может быть применимой для индукции восстановления и регенерации во внутреннем ухе и что сигнальный путь Notch является потенциально применимой мишенью для ингибирования экспрессии конкретного гена. Jung и соавт. инъецировали 8 мкг siRNA для Hes5 в объеме 2 мкл, полученном путем добавления стерильного нормального физиологического раствора к лиофилизированной siRNA, в вестибулярный эпителий уха. Такую систему можно применять для введения системы CRISPR-Cas по настоящему изобретению в вестибулярный эпителий уха в дозе от приблизительно 1 до приблизительно 30 мг CRISPR-Cas для введения человеку.
Глаза
Настоящее изобретение также предусматривает доставку системы CRISPR-Cas в один глаз или оба глаза.
В еще одном аспекте настоящего изобретения систему CRISPR-Cas можно использовать для коррекции офтальмологических дефектов, которые являются результатом нескольких генетических мутаций, дополнительно описанных в Genetic Diseases of the Eye, Second Edition, под редакцией Elias I. Trabouisi, Oxford University Press, 2012.
Для введения в глаз особенно предпочтительными являются лептивирусные векторы, в частности, вирусы инфекционной анемии лошадей (EIAV).
В другом варианте осуществления также предусмотрены минимальные лентивирусные векторы для отличных от приматов организмов на основе вируса инфекционной анемии лошадей (EIAV), особенно для генной терапии глаз (см., например, Balagaan, J Gene Med 2006; 8: 275-285, опубликовано в Интернете 21 ноября 2005 г. в Wiley InterScience (www.interscience.wiley.com). DOI: 10.1002/jgm.845). Предусмотрено, что векторы имеют промотор цитомегаловируса (CMV), управляющий экспрессией целевого гена. Также предусмотрена любая из внутрикамерной, субретинальной, внутриглазной и интравитреальной инъекций (см., например, Balagaan, J Gene Med 2006; 8: 275-285, опубликовано в Интернете 21 ноября 2005 г. в Wiley InterScience (www.interscience.wiley.com). DOI: 10.1002/jgm.845). Внутриглазные инъекции можно осуществлять с помощью операционного микроскопа. В случае субретинальной и интравитреальной инъекций можно выпятить глаза путем осторожного надавливания пальцами и визуализировать глазное дно с помощью системы контактных линз, состоящей из капли раствора контактной среды на роговице, накрытой покровным стеклом для микропрепаратов. При субретинальных инъекциях наконечник иглы 34 калибра на 10 мм, закрепленной на 5-мкл шприце Hamilton, можно при непосредственной визуализации продвигать через экваториальную область верхней части склеры тангенциально к заднему полюсу глазного яблока, пока в субретинальном пространстве не будет видна апертура иглы. Затем можно инъецировать 2 мкл суспензии вектора, вызывая буллезное верхнее отслоение сетчатки, что, таким образом, подтверждает субретинальное введение вектора. В данном подходе производят самогерметизирующийся разрез склеры, позволяющий суспензии вектора удерживаться в субретинальном пространстве до поглощения ее RPE, обычно в течение 48 ч. после процедуры. Эту процедуру можно повторить в нижнем полушарии, вызывая нижнее отслоение сетчатки. Данная методика обуславливает воздействие суспензии вектора на приблизительно 70% нейросенсорной части сетчатки и RPE. В случае интравитреальных инъекций можно продвигать наконечник иглы через склеру на 1 мм кзади от корнеосклерального лимба и инъецировать 2 мкл суспензии вектора в полость стекловидного тела. В случае внутрикамерных инъекций можно продвигать наконечник иглы через парацентез корнеосклерального лимба в направлении центральной части роговицы и можно инъецировать 2 мкл суспензии вектора. В случае внутрикамерных инъекций можно продвигать наконечник иглы через парацентез корнеосклерального лимба в направлении центральной части роговицы и можно инъецировать 2 мкл суспензии вектора. Эти векторы можно инъецировать в титрах 1,0-1,4×1010 или 1,0-1,4×109 трансдуцирующих единиц (ТЕ)/мл.
В другом варианте осуществления также предусмотрен RetinoStat®, лентивирусный вектор на основе вируса инфекционной анемии лошадей для генной терапии, экспрессирующий ангиостатические белки эндостатин и ангиостатин, который доставляют посредством субретинальной инъекции для лечения влажной формы возрастной макулодистрофии (см., например, Binley et al., HUMAN GENE THERAPY 23: 980-991 (September 2012)). Такой вектор может быть модифицирован для системы CRISPR-Cas по настоящему изобретению. Каждый глаз можно обрабатывать любым RetinoStat® в дозе 1,1×105 трансдуцирующих единиц на глаз (ТЕ/глаз) в общем объеме 100 мкл.
В другом варианте осуществления может быть предусмотрен аденовирусный вектор с делецией Е1 и Е4 и частичной делецией Е3 для доставки в глаз. Двадцать восемь пациентов с неоваскулярной возрастной макулодистрофией на поздней стадии (AMD) получали однократную интравитреальную инъекцию аденовирусного вектора с делецией Е1 и Е4 и частичной делецией Е3, экспрессирующего фактор пигментного эпителия человека (AdPEDF.ll) (см., например, Campochiaro et al., Human Gene Therapy 17: 167-176 (February 2006)). Исследовали дозы, варьирующие в диапазоне от 106 до 109,5 единичных частиц (PU), и не наблюдали серьезных нежелательных явлений, связанных с AdPEDF.ll, и дозолимитирующей токсичности (см., например, Campochiaro et al., Human Gene Therapy 17: 167-176 (February 2006)). Опосредованный аденовирусными векторами перенос генов в глаза, по-видимому, является действенным подходом для лечения нарушений со стороны органов зрения и может применяться для системы CRISPR-Cas.
В другом варианте осуществления систему sd-rxRNA® от RXi Pharmaceuticals можно применять для доставки CRISPR-Cas в глаз и/или приспосабливать к ней. В этой системе однократное интравитреальное введение 3 мкг sd-rxRNA приводит к специфичному относительно последовательности снижению уровней мРНК PPIB в течение 14 дней. Систему sd-rxRNA® можно применять для системы CRISPR-Cas по настоящему изобретению, предусматривая введение человеку дозы CRISPR, составляющей приблизительно 3-20 мг.
Millington-Ward et al. (Molecular Therapy, vol. 19 no. 4, 642-649 apr. 2011) описывают векторы на основе аденоассоциированного вируса (AAV) для доставки супрессора родопсина, функционирующего на основе РНК-интерференции (РНКи), и замещающего гена родопсина с модифицированными кодонами, устойчивого к супрессии в связи с нуклеотидными изменениями в вырожденных положениях в целевом сайте для РНКи. Осуществляли субретинальную инъекцию 6,0×108 vp или 1,8×1010 vp AAV в глаза согласно Millington-Ward и соавт. Векторы на основе AAV согласно Millington-Ward и соавт. можно применять для системы CRISPR-Cas по настоящему изобретению, предусматривая введение человеку дозы, составляющей от приблизительно 2×1011 до приблизительно 6×1013 vp.
Dalkara et al. (Sci Transi Med 5, 189ra76 (2013)) также обращаются к направленной эволюции in vivo для конструирования вектора на основе AAV, доставляющего варианты дефектных генов дикого типа по всей сетчатке после безвредной инъекции в жидкую часть стекловидного тела глаза. Dalkara описывает дисплейную библиотеку 7-мерных пептидов и библиотеку AAV, сконструированную посредством ДНК-шаффлинга генов cap AAV1, 2, 4, 5, 6, 8 и 9. Упаковывали библиотеки rcAAV и векторы на основе rAAV, экспрессирующие GFP под контролем промотора CAG или Rho, и с помощью количественной ПЦР получали титры геномов, устойчивых к действию дезоксирибонуклеаз. Библиотеки объединяли, и проводили два цикла эволюции, каждый из которых состоял из диверсификации исходной библиотеки с последующими тремя этапами отбора in vivo. На каждом таком этапе мышам Р30, экспрессирующим rho-GFP, интравитреально инъецировали 2 мл очищенной йодиксанолом и подвергнутой диализу против фосфатно-солевого буфера (PBS) библиотеки с титром геномов приблизительно 1×1012 vg/мл. Векторы на основе AAV согласно Dalkara и соавт. можно применять для системы CRISPR-Cas по настоящему изобретению, предусматривая введение человеку дозы, составляющей от приблизительно 1×1015 до приблизительно 1×1016 vg/мл.
В другом варианте осуществления можно целенаправленно воздействовать на ген родопсина для лечения пигментного ретинита (RP), где систему согласно публикации заявки на патент США №20120204282, закрепленной за Sangamo BioSciences, Inc., можно модифицировать по образу системы CRISPR-Cas по настоящему изобретению.
В другом варианте осуществления способы согласно публикации заявки на патент США №20130183282, закрепленной за Cellectis, направленной на способы расщепления целевой последовательности гена родопсина человека, можно также модифицировать для системы CRISPR-Cas по настоящему изобретению.
Публикация заявки на патент США №20130202678, закрепленная за Academia Sinica, относится к способам лечения форм ретинопатии и офтальмологических нарушений с угрозой потери зрения, относящимся к доставке гена Puf-A (экспрессируемого в ганглиозных и пигментных клетках сетчатки в тканях глаза и проявляющего уникальную антиапоптотическую активность) в субретинальное или интравитреальное пространство глаза. В частности, желаемые мишени представляют собой zgc: 193933, prdm1a, spata2, tex10, rbb4, ddx3, zp2.2, Blimp-1 и HtrA2, на все из которых можно целенаправленно воздействовать с помощью системы CRISPR-Cas по настоящему изобретению.
Wu (Cell Stem Cell, 13: 659-62, 2013) разработал направляющую РНК, которая нацеливает Cas9 на местоположение мутации в одной паре оснований, вызывающей формы катаракты у мышей, где он индуцирует расщепление ДНК. Затем с помощью другого аллеля дикого типа или олигонуклеотидов, вводимых в зиготы, механизмы репарации корригируют последовательность поврежденного аллеля и корригируют генетический дефект, вызывающий катаракту, у мутантной мыши.
В публикации заявки на патент США №20120159653 описывается применение нуклеаз с "цинковыми пальцами" для генетической модификации клеток, животных и белков, ассоциированных с макулодистрофией (MD). Макулодистрофия (MD) является основной причиной ухудшения зрения у лиц пожилого возраста, однако также является характерным симптомом детских заболеваний, таких как болезнь Штаргардта, дистрофия глазного дна Сорсби и смертельные детские нейродегенеративные заболевания, при этом начало заболеваний проявляется уже в младенческом возрасте. Макулодистрофия приводит к потере зрения в центре поля зрения (желтом пятне) по причине поражения сетчатки. Существующие в настоящее время животные модели не воспроизводят основные отличительные признаки заболевания, как это наблюдается у людей. В доступных животных моделях, предусматривающих мутантные гены, кодирующие белки, ассоциированные с MD, также получают крайне изменчивые фенотипы, переходя к проблематике заболевания человека и разработки способов терапии.
Один аспект публикации заявки на патент США №20120159653 относится к редактированию любых хромосомных последовательностей, которые кодируют белки, ассоциированные с MD, что можно применять по отношению к системе CRISPR-Cas по настоящему изобретению. Белки, ассоциированные с MD, как правило, выбирают, исходя из экспериментально установленной взаимосвязи белка, ассоциированного с MD, с нарушением по типу MD. Например, скорость образования или циркулирующая концентрация белка, связанного с MD, может быть повышенной или пониженной в популяции с нарушением по типу MD по сравнению с популяцией без нарушения по типу MD. Различия по уровням белка можно оценить при помощи протеомных методик, в том числе без ограничения вестерн-блоттинга, иммуногистохимического окрашивания, твердофазного иммуноферментного анализа (ELISA) и масс-спектрометрии. Альтернативно, белки, ассоциированные с MD, можно идентифицировать путем получения профилей экспрессии генов для генов, кодирующих белки, при помощи методик геномного анализа, в том числе без ограничения микроматричного анализа ДНК, последовательного анализа экспрессии генов (SAGE) и количественной полимеразной цепной реакции в режиме реального времени (Q-PCR).
В качестве неограничивающего примера, белки, ассоциированные с MD, включают, без ограничения, следующие белки: представитель 4 подсемейства А (АВС1) АТФ-связывающей кассеты (АВСА4), АСНМ1 - белок 1, ассоциированный с ахроматопсией (палочковым монохроматизмом), АроЕ - аполипопротеин Е (АроЕ), C1QTNF5 (CTRP5) - C1q/белок 5, родственный фактору некроза опухолей (C1QTNF5), С2 - компонент 2 системы комплемента (С2), компонент С3 системы комплемента (С3), CCL2 - хемокиновый лиганд 2 (с мотивом С-С) (CCL2), CCR2 - рецептор хемокина 2 (с мотивом С-С) (CCR2), CD36 - кластер дифференцировки 36, CFB - фактор В системы комплемента, CFH - фактор Н системы комплемента (CFH), CFHR1 - белок 1, родственный фактору Н системы комплемента, CFHR3 - белок 3, родственный фактору Н системы комплемента, CNGB3 - бета-3-субъединица ионного канала, регулируемого циклическими нуклеотидами, СР - церулоплазмин (СР), CRP - С-реактивный белок (CRP) CST3 - цистатин С или цистатин 3 (CST3), CTSD - катепсин D (CTSD), CX3CR1 - рецептор хемокина 1 (с мотивом С-Х3-С), ELOVL4 - белок 4, отвечающий за удлинение жирных кислот с очень длинной цепью, ERCC6 - белок эксцизионной репарации, вступающий в перекрестную комплементацию, корректирующий дефицит репарации у грызунов, комплементационная группа 6, FBLN5 - фибулин-5, FBLN5 - фибулин 5, FBLN6 - фибулин 6, FSCN2 - фасции (FSCN2), HMCN1 - гемицентрин 1, HMCN1 - гемицентрин 1, HTRA1 - сериновая пептидаза HtrA 1 (HTRA1), HTRA1 - сериновая пептидаза HtrA 1, IL-6 - интерлейкин 6, IL-8 - интерлейкин 8, LOC387715 - гипотетический белок, PLEKHA1 - белок, содержащий плекстрин-гомологичный домен, представитель 1 семейства А (PLEKHA1), PROM1 - проминин 1 (PROM1 или CD133), PRPH2 - периферин-2, RPGR - регулятор ГТФазы, ассоциированный с пигментным ретинитом, SERPING1 - ингибитор сериновой пептидазы, представитель 1 клады G (С1-ингибитор), TCOF1 - Treacle, TIMP3 - ингибитор 3 металлопротеиназ (TIMP3), TLR3 - Toll-подобный рецептор 3.
Идентичность белка, ассоциированного с MD, редактирование хромосомной последовательности которого осуществляют, может и будет варьировать. В предпочтительном варианте осуществления белки, ассоциированные с MD, редактирование хромосомных последовательностей которых осуществляют, могут представлять собой белок представитель 4 (АВСА4) подсемейства А (АВС1) АТФ-связывающей кассеты, кодируемый геном ABCR, белок аполипопротеин Е (АРОЕ), кодируемый геном АРОЕ, белок хемокиновый лиганд 2 (с мотивом С-С) (CCL2), кодируемый геном CCL2, белок рецептор хемокина 2 (с мотивом С-С) (CCR2), кодируемый геном CCR2, белок церулоплазмин (СР), кодируемый геном СР, белок катепсин D (CTSD), кодируемый геном CTSD, или белок ингибитор 3 металлопротеиназ (TIMP3), кодируемый геном TIMP3. В иллюстративном варианте осуществления генетически модифицированное животное представляет собой крысу, и редактируемые хромосомные последовательности, кодирующие белок, ассоциированный с MD, могут быть следующими: NM_000350 (АВСА4) для представителя 4 подсемейства А (АВС1) АТФ-связывающей кассеты, NM_138828 (АРОЕ) для аполипопротеина Е АРОЕ, NM_031530 (CCL2) для хемокинового лиганда 2 (с мотивом С-С) CCL2, NM_021866 (CCR2) для рецептора хемокина 2 (с мотивом С-С) CCR2, NM_012532 (СР) для церулоплазмина СР, NM_34334 (CTSD) для катепсина D CTSD, NM_012886 (TIMP3) для ингибитора 3 металлопротеиназ TIMP3. Животное или клетка могут содержать 1, 2, 3, 4, 5, 6, 7 или более хромосомных последовательностей с нарушенной структурой, кодирующих белок, ассоциированный с MD, и ноль, 1, 2, 3, 4, 5, 6, 7 или более интегрированных в хромосомы последовательностей, кодирующих белок с нарушенной структурой, ассоциированный с MD.
Отредактированную или интегрированную хромосомную последовательность можно модифицировать так, чтобы она кодировала измененный белок, ассоциированный с MD. Некоторые мутации в хромосомных последовательностях, связанных с MD, были ассоциированы с MD. Неограничивающие примеры мутаций в хромосомных последовательностях, ассоциированные с MD, включают те, которые могут вызывать MD, в том числе в белке ABCR - E471K (т.е. глутамат в положении 471 заменен на лизин), R1129L (т.е. аргинин в положении 1129 заменен на лейцин), Т1428М (т.е. треонин в положении 1428 заменен на метионин), R1517S (т.е. аргинин в положении 1517 заменен на серин), I1562T (т.е. изолейцин в положении 1562 заменен на треонин) и G1578R (т.е. глицин в положении 1578 заменен на аргинин); в белке CCR2 - V64I (т.е. валин в положении 192 заменен на изолейцин); в белке СР - G969B (т.е. глицин в положении 969 заменен на аспарагин или аспартат); в белке TIMP3 - S156C (т.е. серин в положении 156 заменен на цистеин), G166C (т.е. глицин в положении 166 заменен на цистеин), G167C (т.е. глицин в положении 167 заменен на цистеин), Y168C (т.е. тирозин в положении 168 заменен на цистеин), S170C (т.е. серин в положении 170 заменен на цистеин), Y172C (т.е. тирозин в положении 172 заменен на цистеин) и S181C (т.е. серин в положении 181 заменен на цистеин). В данной области известны и другие взаимосвязи генных вариантов генов, ассоциированных с MD, и заболевания.
Сердце
Настоящее изобретение также предусматривает доставку системы CRISPR-Cas в сердце. Для сердца предпочтительным является тропный к миокарду аденоассоциированный вирус (AAVM), в частности AAVM41, при использовании которого продемонстрирован преимущественный перенос генов в сердце (см., например, Lin-Yanga et al., PNAS, March 10, 2009, vol. 106, no. 10). Введение может быть системным или местным. Доза приблизительно 1-10×1014 векторных геномов предусматривается для системного введения. См. также, например, Eulalio et al. (2012) Nature 492: 376 и Somasuntharam et al. (2013) Biomaterials 34: 7790.
Например, в публикации патентного документа США №20110023139 описывается применение нуклеаз с "цинковыми пальцами" для генетической модификации клеток, животных и белков, ассоциированных с сердечно-сосудистым заболеванием. Сердечнососудистые заболевания, как правило, включают высокое кровяное давление, сердечные приступы, сердечную недостаточность и инсульт, а также TIA. Любую хромосомную последовательность, связанную с сердечно-сосудистым заболеванием, или белок, кодируемый любой хромосомной последовательностью, связанной с сердечнососудистым заболеванием, можно использовать в способах, описанных в настоящем раскрытии. Белки, связанные с сердечно-сосудистым заболеванием, обычно выбирают исходя из экспериментально подтвержденной ассоциации белка, связанного с сердечнососудистым заболеванием, с развитием сердечно-сосудистого заболевания. Например, скорость образования или концентрация в кровотоке белка, связанного с сердечнососудистым заболеванием, может быть повышенной или пониженной в популяции с сердечно-сосудистым заболеванием по сравнению с популяцией без сердечно-сосудистого заболевания. Различия по уровням белка можно оценить при помощи протеомных методик, в том числе без ограничения вестерн-блоттинга, иммуногистохимического окрашивания, твердофазного иммуноферментного анализа (ELISA) и масс-спектрометрии. В альтернативном случае, белки, связанные с сердечно-сосудистым заболеванием, можно идентифицировать путем получения профилей генной экспрессии для генов, кодирующих белки, при помощи методик геномного анализа, в том числе без ограничения микроматричного анализа ДНК, последовательного анализа экспрессии генов (SAGE) и количественной полимеразной цепной реакции в режиме реального времени (Q-PCR).
В качестве примера, хромосомная последовательность может содержать без ограничения следующие: IL1B (интерлейкин 1, бета), XDH (ксантиндегидрогеназа), ТР53 (опухолевый белок р53), PTGIS (простагландин 12 (простациклин) синтаза), MB (миоглобин), IL4 (интерлейкин 4), ANGPT1 (ангиопоэтин 1), ABCG8 (АТФ-связывающая кассета, подсемейство G (WHITE), представитель 8), CTSK (катепсин K), PTGIR (рецептор простагландина 12 (простациклина) (IP)), KCNJ11 (входящий калиевый канал, подсемейство J, представитель 11), INS (инсулин), CRP (С-реактивный белок, родственный пентраксину), PDGFRB (полипептид рецептора тромбоцитарного фактора роста бета), CCNA2 (циклин А2), PDGFB (полипептид тромбоцитарного фактора роста бета (гомолог онкогена вируса саркомы обезьян (v-sis))), KCNJ5 (входящий калиевый канал, подсемейство J, представитель 5), KCNN3 (кальций-активируемый калиевый канал средней/малой проводимости, подсемейство N, представитель 3), CAPN10 (кальпаин 10), PTGES (простагландин Е синтаза), ADRA2B (адренергический, альфа-2 В-, рецептор), ABCG5 (АТФ-связывающая кассета, подсемейство G (WHITE), представитель 5), PRDX2 (пероксиредоксин 2), CAPN5 (кальпаин 5), PARP14 (семейство полимераз поли(АДФ-рибозы), представитель 14), МЕХ3С (гомолог С mex-3 (С. elegans)), АСЕ, ангиотензинпревращающего фермента I (пептидилдипептидаза А) 1), TNF (фактор некроза опухоли (суперсемейство TNF, представитель 2)), IL6 (интерлейкин 6 (интерферон, бета 2)), STN (статин), SERPINE1 (ингибитор серпиновой пептидазы, клада Е (нексин, ингибитор активатора плазминогена типа 1), представитель 1), ALB (альбумин), ADIPOQ (адипонектин, содержащий C1Q и коллагеновый домен), АРОВ (аполипопротеин В (в том числе антиген Ag(x))), АРОЕ (аполипопротеин Е), LEP (лептин), MTHFR (5,10-метилентетрагидрофолатредуктаза (NADPH)), APOA1 (аполипопротеин A-I), EDN1 (эндотелии 1), NPPB (предшественник В натрийуретического пептида), NOS3 (синтаза 3 оксида азота (эндотелиальная клетка)), PPARG (рецептор гамма, активируемый пролифератором пероксисом), PLAT (активатор плазминогена, ткань), PTGS2 (простагландин-эндопероксидсинтаза 2 (простагландин G/H синтаза и циклооксигеназа)), СЕТР (транспортный белок сложного эфира холестерина, плазма), AGTR1 (рецептор ангиотензина II, тип 1), HMGCR (редуктаза 3-гидрокси-3-метилглутарилкофермента A), IGF1 (инсулиноподобный фактор роста 1 (соматомедин С)), SELE (селектин Е), REN (ренин), PPARA (альфа-рецептор, активируемый пролифератором пероксисом), PON1 (параоксоназа 1), KNG1 (кининоген 1), CCL2 (лиганд 2 хемокина (мотив С-С)), LPL (липопротеинлипаза), VWF (фактор фон Виллебранда), F2 (фактор коагуляции II (тромбин)), ICAM1 (внутриклеточная молекула адгезии 1), TGFB1 (трансформирующий фактор роста, бета 1), NPPA (предшественник А натрийуретического пептида), IL10 (интерлейкин 10), ЕРО (эритропоэтин), SOD1 (супероксиддисмутаза 1, растворимая), VCAM1 (молекула адгезии сосудистого эндотелия типа 1), IFNG (интерферон, гамма), LPA (липопротеин, Lp(a)), MPO (миелопероксидаза), ESR1 (эстрогеновый рецептор 1), MAPK1 (митоген-активируемая протеинкиназа 1), HP (гаптоглобин), F3 (фактор коагуляции III (тромбопластин, тканевой фактор)), CST3 (цистатин С), COG2 (компонент олигомерного комплекса Гольджи 2), ММР9 (матриксная металлопептидаза 9 (желатиназа В, 92 кДа желатиназа, 92 кДа коллагеназа типа IV)), SERPINC1 (ингибитор серпиновой пептидазы, клада С (антитромбин), представитель 1), F8 (фактор коагуляции VIII, прокоагулянтный компонент), НМОХ1 (гемоксигеназа (дециклизирующая) 1), АРОС3 (аполипопротеин С-III), IL8 (интерлейкин 8), PROK1 (прокинетицин 1), CBS (цистатион-бета-синтаза), NOS2 (синтаза 2 оксида азота, индуцируемая), TLR4 (толл-подобный рецептор 4), SELP (селектин Р (гранулярный мембранный белок 140 кДа, антиген CD62)), АВСА1 (АТФ-связывающая кассета, подсемейство А (АВС1), представитель 1), AGT (ангиотензиноген (ингибитор серпиновой пептидазы, клада А, представительн 8)), LDLR (рецептор липопротеинов низкой плотности), GPT (глутамат-пируватная трансаминаза (аланинаминотрансфераза)), VEGFA (фактор роста эндотелия сосудов A), NR3C2 (ядерный рецептор, подсемейство 3, группа С, представитель 2), IL18 (интерлейкин 18 (интерферон-гамма-индуцирующий фактор)), NOS1 (синтаза 1 оксида азота (нейрональная)), NR3C1 (ядерный рецептор, подсемейство 3, группа С, представитель 1 (глюкокортикоидный рецептор)), FGB (бета-цепь фибриногена), HGF (фактор роста гепатоцитов (гепапоэтин А; рассеивающий фактор)), ILIA (интерлейкин 1, альфа), RETN (резистин), AKT1 (гомолог 1 онкогена v-akt вируса тимомы мышей), LIPC (липаза, печеночная), HSPD1 (60 кДа белок теплового шока 1 (шаперонин)), MAPK14 (митоген-активируемая протеинкиназа 14), SPP1 (секретируемый фосфопротеин 1), ITGB3 (интегрин, бета 3 (гликопротеин 111a тромбоцитов, антиген CD61)), CAT (каталаза), UTS2 (уротензин 2), THBD (тромбомодулин), F10 (фактор коагуляции X), СР (церулоплазмин (ферроксидаза)), TNFRSF11B (суперсемейство рецепторов фактора некроза опухоли, представитель 11b), EDNRA (рецептор эндотелина типа A), EGFR (рецептор эпидермального фактора роста (гомолог онкогена вируса эритробластического лейкоза (v-erb-b), птичий)), ММР2 (матриксная металлопептидаза 2 (желатиназа А, 72 кДа желатиназа, 72 кДа коллагеназа типа IV)), PLG (плазминоген), NPY (нейропептид Y), RHOD (гомолог белка семейства генов ras, представитель D), MAPK8 (митоген-активируемая протеинкиназа 8), MYC (гомолог онкогена v-myc вируса миелоцитоматоза (птичий)), FN1 (фибронектин 1), СМА1 (химаза 1, тучная клетка), PLAU (активатор плазминогена, урокиназа), GNB3 (белок, связывающий гуаниновый нуклеотид (G-белок), бета-полипептид 3), ADRB2 (адренергический, бета-2-, рецептор, поверхностный), АРОА5 (аполипопротеин A-V), SOD2 (супероксиддисмутаза 2, митохондриальная), F5 (фактор коагуляции V (проакцелерин, лабильный фактор)), VDR (рецептор витамина D (1,25-дигидроксивитамин D3)), ALOX5 (арахидонат-5-липоксигеназа), HLA-DRB1 (главный комплекс гистосовместимости, класс II, DR-бета 1), PARP1 (полимераза 1 поли(ADP-рибозы)), CD40LG (лиганд CD40), PON2 (параоксоназа 2), AGER (рецептор, специфичный к конечным продуктам усиленного гликозилирования), IRS1 (субстрат 1 инсулинового рецептора), PTGS1 (простагландин-эндопероксидсинтаза 1 (простагландин G/H синтаза и циклооксигеназа)), ЕСЕ1 (эндотелин-превращающий фермент 1), F7 (фактор коагуляции VII (сывороточный ускоритель превращения протромбина)), URN (антагонист рецептора интерлейкина 1), ЕРНХ2 (эпоксидгидролаза 2, цитоплазматическая), IGFBP1 (белок 1, связывающий инсулиноподобный фактор роста), MAPK10 (митоген-активируемая протеинкиназа 10), FAS (Fas (суперсемейство рецепторов TNF, представитель 6)), АВСВ1 (АТФ-связывающая кассета, подсемейство В (MDR/TAP), представитель 1), JUN (онкоген jun), IGFBP3 (белок 3, связывающий инсулиноподобный фактор роста), CD14 (молекула CD14), PDE5A (фосфодиэстераза 5А, cGMP-специфичная), AGTR2 (рецептор ангиотензина II, тип 2), CD40 (молекула CD40, член 5 суперсемейства рецепторов TNF), LCAT (лецитинхолестеринацилтрансфераза), CCR5 (рецептор 5 хемокина (мотив С-С)), ММР1 (матриксная металлопептидаза 1 (интерстициальная коллагеназа)), TIMP1 (ингибитор 1 металлопептидазы TIMP), ADM (адреномедуллин), DYT10 (белок 10, связанный с дистонией), STAT3 (переносчик сигнала и активатор транскрипции 3 (фактор ответа острой фазы)), ММР3 (матриксная металлопептидаза 3 (стромелизин 1, прожелатиназа)), ELN (эластин), USF1 (транскришщонный фактор 1, расположенный выше промотора), CFH (фактор комплемента Н), HSPA4 (70 кДа белок теплового шока 4), ММР12 (матриксная металлопептидаза 12 (эластаза макрофагов)), ММЕ (мембранная металлоэндопептидаза), F2R (рецептор фактора коагуляции II (тромбина)), SELL (селектин L), CTSB (катепсин В), ANXA5 (аннексии А5), ADRB1 (адренергический, бета-1-, рецептор), CYBA (цитохром b-245, альфа-полипетид), FGA (альфа-цепь фибриногена), GGT1 (гамма-глутамилтрансфераза 1), LIPG (липаза, эндотелиальная), HIF1A (фактор 1, индуцируемый гипоксией, альфа-субъединица (транскрипционный фактор с основным доменом типа спираль-петля-спираль)), CXCR4 (рецептор 4 хемокина (мотив С-Х-С)), PROC (белок С (инактиватор факторов коагуляции Va и VIIIa)), SCARB1 (класс В фагоцитарных рецепторов, член 1), CD79A (молекула CD79a, иммуноглобулин-ассоциированная альфа), PLTP (белок, переносящий фосфолипиды), ADD1 (аддупин 1 (альфа)), FGG (гамма-цепь фибриногена), SAA1 (сывороточный амилоид A1), KCNH2 (калиевый потенциалзависимый канал, подсемейство Н (eag-родственный), представитель 2), DPP4 (дипептидилпептидаза 4), G6PD (глюкозо-6-фосфатдегидрогеназа), NPR1 (рецептор А натрийуретического пептида/гуанилатциклаза А (рецептор А атрионатрийуретического пептида)), VTN (витронектин), KIAA0101 (KIAA0101), FOS (гомолог онкогена вируса остеосаркомы мышей FBJ), TLR2 (толл-подобный рецептор 2), PPIG (пептидилпролилизомераза G (циклофилин G)), IL1R1 (рецептор интерлейкина 1, тип I), AR (андрогеновый рецептор), CYP1A1 (цитохром Р450, семейство 1, подсемейство А, полипептид 1), SERPINA1 (ингибитор серпиновой пептидазы, клада А (альфа-1 антипротеиназа, антитрипсин), член 1), MTR (5-метилтетрагидрофолатгомоцистеинметилтрансфераза), RBP4 (белок 4, связывающий ретинол, плазма), АРОА4 (аполипопротеин A-IV), CDKN2A (ингибитор 2А циклин-зависимой киназы (меланома, р16, ингибирует CDK4)), FGF2 (фактор 2 роста фибробластов (основной)), EDNRB (рецептор эндотелина типа В), ITGA2 (интегрин, альфа 2 (CD49B, субъединица альфа 2 рецептора VLA-2)), CABIN1 (белок 1, связывающий кальциневрин), SHBG (глобулин, связывающий половые гормоны), HMGB1 (группа белков с высокой подвижностью 1), HSP90B2P (90 кДа белок теплового шока бета (Grp94), представитель 2 (псевдоген)), CYP3A4 (цитохром Р450, семейство 3, подсемейство А, полипептид 4), GJA1 (белок межклеточных щелевых контактов, альфа 1, 43 кДа), CAV1 (кавеолин 1, кавеолярный белок, 22 кДа), ESR2 (эстрогеновый рецептор 2 (ER-бета)), LTA (лимфотоксин-альфа (суперсемейство TNF, представитель 1)), GDF15 (фактор 15 роста и дифференцировки), BDNF (нейротрофический фактор головного мозга), CYP2D6 (цитохром Р450, семейство 2, подсемейство D, полипептид 6), NGF (фактор роста нервов (бета-полипептид)), SP1 (транскрипционный фактор Sp1), TGIF1 (гомеобокс 1 TGFB-индуцируемого фактора), SRC (гомолог онкогена v-src вируса саркомы (Schmidt-Ruppin А-2) (птичий)), EGF (эпидермальный фактор роста (бета-урогастрон)), PIK3CG (фосфоинозитид-3-киназа, каталитическая, гамма-полипептид), HLA-A (главный комплекс гистосовместимости, класс I, A), KCNQ1 (калиевый потенциалзависимый канал, подсемейство KQT-подобных белков, представитель 1), CNR1 (каннабиноидный рецептор 1 (головной мозг)), FBN1 (фибриллин 1), CHKA (холинкиназа альфа), BEST1 (бестрофин 1), АРР (белок-предшестенник бета-амилоида (А4)), CTNNB1 (катенин (кадгерин-ассоциированный белок), бета 1, 88 кДа), IL2 (интерлейкин 2), GD36 (молекула CD36 (рецептор тромбоспондина)), PRKAB1 (протеинкиназа, АМР-активируемая, бета 1, некаталитическая субъединица), ТРО (тиреоидная пероксидаза), ALDH7A1 (семейство альдегиддегидрогеназы 7, представитель A1) CX3CR1 (рецептор 1 хемокина (мотив С-Х3-С)), ТН (тирозингидроксилаза), F9 (фактор коагуляции DC), GH1 (гормон роста 1), TF (трансферрин), HFE (гемохроматоз), IL17A (интерлейкин 17А), PTEN (гомолог фосфатазы и тензина), GSTM1 (глутатион-S-трансфераза mu I), DMD (дистрофин), GATA4 (GATA-связывающий белок 4), F13A1 (фактор коагуляции XIII, полипептид A1), TTR (транстиретин), FABP4 (белок 4, связывающий жирные ксилоты, адипоцитарный), PON3 (параоксоназа 3), АРОС1 (аполипопротеин C-I), INSR (инсулиновый рецептор), TNFRSF1B (суперсемейство рецепторов фактора некроза опухоли, представитель 1В), HTR2A (5-гидрокситриптаминовый (серотониновый) рецептор 2А), CSF3 (колониестимулирующий фактор 3 (гранулоцитарный)), CYP2C9 (цитохром Р450, семейство 2, подсемейство С, полипептид 9), TXN (тиоредоксин), CYP11B2 (цитохром Р450, семейство 11, подсемейство В, полипептид 2), РТН (паратиреоидный гормон), CSF2 (колониестимулирующий фактор 2 (гранулоцитарно-макрофагальный)), KDR (рецептор вставочного домена киназы (рецептор тирозинкиназы III типа)), PLA2G2A (фосфолипаза А2, группа IIA (тромбоциты, синовиальная жидкость)), В2М (бета-2-микроглобулин), THBS1 (тромбоспондин 1), GCG (глюкагон), RHOA (гомолог белка семейства генов ras, член A), ALDH2 (семейство альдегиддегидрогеназы 2 (митохондриальная)), TCF7L2 (белок 2, подобный транскрипционному фактору 7 (специфический по отношению к Т-клеткам, HMG-бокс)), BDKRB2 (брадикининовый рецептор В2), NFE2L2 (белок 2, подобный ядерному фактору (эритроидному 2)), NOTCH 1 (гомолог Notch 1, ассоциированный с транслокацией (Drosophila)), UGT1A1 (семейство UDP-глюкуронозилтрансферазы 1, полипептид A1), IFNA1 (интерферон, альфа 1), PPARD (рецептор дельта, активируемый пролифератором пероксисом), SIRT1 (сиртуин (гомолог регулятора 2 экспрессии, обеспечивающего сайленсинг) 1 (S. cerevisiae)), GNRH1 (гонадотропин-рилизинг гормон 1 (лютеинизирующий рилизинг-гормон)), РАРРА (ассоциированный с беременностью белок плазмы А, паппализин 1), ARR3 (аррестин 3, ретинальный (Х-аррестин)), NPPC (предшественник С натрийуретического пептида), AHSP (белок, стабилизирующий альфа-гемоглобин), PTK2 (PTK2 тирозиновая протеинкиназа 2), IL13 (интерлейкин 13), MTOR (механистическая мишень рапамицина (серин/треонинкиназа)), ITGB2 (интегрин, бета 2 (субъединица рецептора 3 и 4 компонента комплемента 3)), GSTT1 (глутатион-S-трансфераза тета 1), IL6ST (переносчик сигнала интерлейкина 6 (gp130, рецептор онкостатина М)), СРВ2 (карбоксипептидаза В2 (плазма)), CYP1A2 (цитохром Р450, семейство 1, подсемейство А, полипептид 2), HNF4A (гепатопитарный ядерный фактор 4, альфа), SLC6A4 (семейство переносчиков растворенных веществ 6 (транспортер нейротрансмиттеров, серотониновый), представитель 4), PLA2G6 (фосфолипаза А2, группа VI (цитозольная, кальций-независимая)), TNFSF11 (суперсемейство фактора некроза опухоли (лиганд), член 11), SLC8A1 (семейство переносчиков растворенных веществ 8 (натрий/кальциевый обменник), представитель 1), F2RL1 (белок 1, подобный рецептору фактора коагуляции II (тромбина)), AKR1A1 (семейство 1 альдокеторедуктаз, представитель A1 (альдегидредуктаза)), ALDH9A1 (семейство альдегиддегидрогеназы 9, член Al), BGLAP (гамма-карбоксиглутаматный (gla) белок кости), МТТР (микросомальный белок-переносчик триглицеридов), MTRR (редуктаза 5-метилтетрагидрофолат-гомоцистеинметилтрансферазы), SULT1A3 (семейство сульфотрансфераз, цитозольная, 1А, преимущественно воздействующих на фенол, представитель 3), RAGE (антиген опухоли почек), С4В (компонент комплемента 4В (группа крови Chido), P2RY12 (пуринергический рецептор P2Y, связанный с G-белком, 12), RNLS (реналаза, FAD-зависимая аминоксидаза), CREB1 (белок 1, связывающий сАМР-чувствительный элемент), РОМС (проопиомеланокортин), RAC1 (родственный ras субстрат 1 ботулотоксина С3 (семейство rho, малый GTP-связывающий белок Rac1)), LMNA (ламин NC), CD59 (молекула CD59, регуляторный белок комплемента), SCN5A (натриевый канал, потенциалзависимый, тип V, альфа-субъединица), CYP1B1 (цитохром Р450, семейство 1, подсемейство В, полипептид 1), MIF (фактор ингибирования миграции макрофагов (фактор, ингибирующий гликозилирование)), ММР13 (матриксная металлопептидаза 13 (коллагеназа 3)), TIMP2 (ингибитор 2 металлопептидазы TIMP), CYP19A1 (цитохром Р450, семейство 19, подсемейство А, полипептид 1), CYP21A2 (цитохром Р450, семейство 21, подсемейство А, полипептид 2), PTPN22 (тирозиновая протеинфосфатаза, нерецепторного типа 22 (лимфоидная)), MYH14 (миозин, тяжелая цепь 14, отличный от мышечного), MBL2 (лектин, связывающий маннозу (белок С) 2, растворимый (опсонический дефект)), SELPLG (лиганд селектина Р), АОС3 (аминоксидаза, белок 3, содержащий медь (васкулярный адгезивный белок 1)), CTSL1 (катепсин L1), PCNA (ядерный антиген пролиферирующих клеток), IGF2 (инсулиноподобный фактор роста 2 (соматомедин A)), ITGB1 (интегрин, бета 1 (рецептор фибронектина, бета-полипептид, антиген CD29, включающий MDF2, MSK12)), CAST (кальпастатин), CXCL12 (лиганд 12 хемокина (мотив С-Х-С) (стромальный клеточный фактор 1)), IGHE (константный участок тяжелой эпсилон-цепи иммуноглобулина), KCNE1 (калиевый потенциалзависимый канал, Isk-родственное семейство, представитель 1), TFRC (рецептор трансферрина (р90, CD71)), COL1A1 (коллаген, тип I, альфа 1), COL1A2 (коллаген, тип I, альфа 2), IL2RB (рецептор интерлейкина 2, бета), PLA2G10 (фосфолипаза А2, группа X), ANGPT2 (ангиопоэтин 2), PROCR (рецептор белка С, эндотелиальный (EPCR)), NOX4 (NADPH-оксидаза 4), HAMP (противомикробный пептид гепсидин), PTPN11 (тирозиновая протеинфосфатаза, нерецепторного типа 11), SLC2A1 (семейство 2 переносчиков растворенных веществ (транспортер, обеспечивающий облегченный перенос глюкозы), предствитель 1), IL2RA (рецептор интерлейкина 2, альфа), CCL5 (лиганд 5 хемокина (мотив С-С)), IRF1 (регуляторный фактор 1 интерферона), CFLAR (CASP8 и FADD-подобный регулятор апоптоза), CALCA (полипептид альфа, родственный кальцитонину), EIF4E (эукариотический фактор инициации трансляции 4Е), GSTP1 (глутатион-S-трансфераза пи 1), JAK2 (Janus киназа 2), CYP3A5 (цитохром Р450, семейство 3, подсемейство А, полипептид 5), HSPG2 (гепарансульфат-протеогликан 2), CCL3 (лиганд 3 хемокина (мотив С-С)), MYD88 (белок гена первичного ответа, связанного с дифференцировкой миелоидных клеток (88)), VIP (вазоактивный интестинальный пептид), SOAT1 (стерол-O-ацилтрансфераза 1), ADRBK1 (адренергическая, бета, рецепторная киназа 1), NR4A2 (ядерный рецептор, подсемейство 4, группа А, представитель 2), ММР8 (матриксная металлопептидаза 8 (коллагеназа нейтрофилов)), NPR2 (рецептор В натрийуретического пептида/гуанилатпиклаза В (рецептор В атрионатрийуретического пептида)), GCH1 (GTP-циклогидролаза 1), EPRS (глутамилпролил-тРНК-синтетаза), PPARGC1A (гамма-рецептор, активируемый пролифератором пероксисом, коактиватор 1 альфа), F12 (фактор коагуляции XII (фактор Хагемана)), РЕСАМ1 (молекула клеточной адгезии эндотелиальных клеток/тромбоцитов), CCL4 (лиганд 4 хемокина (мотив С-С)), SERPINA3 (ингибитор серпиновой пептидазы, клада А (альфа-1 антипротеиназа, антитрипсин), предтавитель 3), CASR (кальций-чувствительный рецептор), GJA5 (белок межклеточных щелевых контактов, альфа 5, 40 кДа), FABP2 (белок 2, связывающий жирные кислоты, кишечный), TTF2 (фактор терминации транскрипции, РНК-полимераза II), PROS1 (белок S (альфа)), CTF1 (кардиотрофин 1), SGCB (саркогликан, бета (43 кДа дистрофин-ассоциированный гликопротеин)), YME1L1 (YME1-подобный белок 1 (S. cerevisiae)), CAMP (противомикробный пептид кателицидин), ZC3H12A (белок 12А СССН-типа, содержащий "цинковые пальцы"), AKR1B1 (семейство 1 альдокеторедуктаз, представитель В1 (альдозоредуктаза)), DES (десмин), ММР7 (матриксная металлопептидаза 7 (матрилизин, маточный)), AHR (арилуглеводородный рецептор), CSF1 (колониестимулирующий фактор 1 (макрофагальный)), HDAC9 (деацетилаза гистонов 9), CTGF (фактор роста соединительной ткани), KCNMA1 (кальций-активируемый калиевый канал большой проводимости, подсемейство М, альфа, член 1), UGT1A (семейство UDP-глюкуронозилтрансферазы 1, полипептид А сложного локуса), PRKCA (протеинкиназа С, альфа), СОМТ (катехол-бета-метилтрансфераза), S100B (S100 кальций-связывающий белок В), EGR1 (белок, связанный с ранними стадиями роста 1), PRL (пролактин), IL15 (интерлейкин 15), DRD4 (допаминовый рецептор D4), CAMK2G (кальций/кальмодулин-зависимая протеинкиназа II гамма), SLC22A2 (семейство 22 переносчиков растворенных веществ (транспортер органических катионов), представитель 2), CCL11 (лиганд 11 хемокина (мотив С-С)), PGF (B321 плацентарный фактор роста), ТНРО (тромбопоэтин), GP6 (гликопротеин VI (тромбоцитарный)), TACR1 (тахикининовый рецептор 1), NTS (нейротензин), HNF1A (HNF1 гомеобокс A), SST (соматостатин), KCND1 (калиевый потенциалзависимый канал, Shal-родственное подсемейство, представитель 1), LOC646627 (ингибитор фосфолипазы), TBXAS1 (тромбоксан А синтаза 1 (тромбоцитарная)), CYP2J2 (цитохром Р450, семейство 2, подсемейство J, полипептид 2), TBXA2R (рецептор тромбоксана А2), ADH1C (алкогольдегидрогеназа 1C (класс I), гамма-полипептид), ALOX12 (арахидонат 12-липоксигеназа), AHSG (альфа-2-HS-гликопротеин), ВНМТ (бетаин-гомоцистеинметилтрансфераза), GJA4 (белок межклеточных щелевых контактов, альфа 4, 37 кДа), SLC25A4 (семейство 25 переносчиков растворенных веществ (митохондриальный переносчик; переносчик адениновых нуклеотидов), член 4), ACLY (АТР-цитратлиаза), ALOX5AP (арахидонат-5-липоксигеназа-активирующий белок), NUMA1 (белок 1 ядерного митотического аппарата), CYP27B1 (цитохром Р450, семейство 27, подсемейство В, полипептид 1), CYSLTR2 (цистеиниллейкотриеновый рецептор 2), SOD3 (супероксиддисмутаза 3, внеклеточная), LTC4S (лейкотриен-С4-синтаза), UCN (урокортин), GHRL (препропептид грелин/обестатин), АРОС2 (аполипопротеин C-II), CLEC4A (семейство 4 лектиновых доменов С-типа, представитель A), KBTBD10 (белок 10, содержащий повтор kelch и домен ВТВ (POZ)), TNC (тенасцин С), TYMS (тимидилатсинтетаза), SHC1 (SHC (гомолог 2, содержащий домен Src) трансформирующий белок 1), LRP1 (белок 1, родственный рецептору липопротеинов низкой плотности), SOCS3 (супрессор цитокинового сигнала 3), ADH1B (алкогольдегидрогеназа 1В (класс I), бета-полипептид), KLK3 (калликреин-родственная пептидаза 3), HSD11B1 (гидроксистероидная (11-бета) дегидрогеназа 1), VKORC1 (комплекс эпоксидредуктазы и витамина K, субъединица 1), SERPINB2 (ингибитор серпиновой пептидазы, клада В (овальбумин), представитель 2), TNS1 (тензин 1), RNF19A (белок 19А с доменом ring), EPOR (рецептор эритропоэтина), ITGAM (интегрин, альфа М (субъединица рецептора 3 компонента комплемента 3)), PITX2 (гомеодомен 2, подобный парному), MAPK7 (митоген-активируемая протеинкиназа 7), FCGR3A (Fc-фрагмент IgG, белок 111a с низким сродством, рецептор (CD16a)), LEPR (лептиновый рецептор), ENG (эндоглин), GPXL (глутатионпероксидаза 1), GOT2 (глутамат-оксалоацетат-трансаминаза 2, митохондриальная (аспартатаминотрансфераза 2)), HRH1 (гистаминовый рецептор H1), NR112 (ядерный рецептор, подсемейство 1, группа I, представитель 2), CRH (кортикотропин-рилизинг-гормон), HTR1A (5-гидрокситриптаминовый (серотониновый) рецептор 1А), VDAC1 (потенциалзависимый анионный канал 1), HPSE (гепараназа), SFTPD (сурфактантный белок D), TAP2 (транспортер 2, АТР-связывающая кассета, подсемейство В (MDR/TAP)), RNF123 (белок 123 с доменом ring), PTK2B (PTK2B тирозиновая протеинкиназа 2 бета), NTRK2 (нейротрофическая тирозинкиназа, рецептор, тип 2), IL6R (рецептор интерлейкина 6), ACHE (ацетилхолинэстераза (группа крови Yt)), GLP1R (рецептор глюкагон-подобного пептида 1), GHR (рецептор гормона роста), GSR (глутатионредуктаза), NQO1 (NAD(Р)Н-дегидрогеназа, хинон 1), NR5A1 (ядерный рецептор, подсемейство 5, группа А, представитель 1), GJB2 (белок межклеточных щелевых контактов, бета 2, 26 кДа), SLC9A1 (семейство 9 переносчиков растворенных веществ (натрий/водородный обменник), представитель 1), МАОА (моноаминоксидаза А), PCSK9 (пробелок-конвертаза субтилизин/кексин типа 9), FCGR2A (Fc-фрагмент IgG, белок IIa с низким сродством, рецептор (CD32)), SERPINF1 (ингибитор серпиновой пептидазы, клада F (альфа-2 антиплазмин, фактор из пигментного эпителия), представитель 1), EDN3 (эндотелин 3), DHFR (дигидрофолатредуктаза), GAS6 (белок 6, специфический по отношению к остановке роста), SMPD1 (сфингомиелинфосфодиэстераза 1, кислая лизосомная), UCP2 (разобщающий белок 2 (митохондриальный, переносчик протонов)), TFAP2A (транскрипционный фактор АР-2 альфа (активирующий энхансер-связывающий белок 2 альфа)), С4ВРА (компонент комплемента 4 связывающий белок, альфа), SERPINF2 (ингибитор серпиновой пептидазы, клада F (альфа-2 антиплазмин, фактор из пигментного эпителия), представитель 2), TYMP (тимидинфосфорилаза), ALPP (щелочная фосфатаза, плацентарная (изофермент Regan)), CXCR2 (рецептор 2 хемокина (мотив С-Х-С)), SLC39A3 (семейство 39 переносчиков растворенных веществ (цинковый транспортер), представитель 3), ABCG2 (АТР-связывающая кассета, подсемейство G (WHITE), представитель 2), ADA (аденозиндезаминаза), JAK3 (Janus киназа 3), HSPA1A (70 кДа белок теплового шока 1А), FASN (синтаза жирных кислот), FGF1 (фактор 1 роста фибробластов (кислый)), F11 (фактор коагуляции XI), АТР7А (АТФаза, Cu++-транспортирующая, альфа-полипетид), CR1 (рецептор 1 компонента комплемента (3b/4b) (группа крови Knops)), GFAP (глиальный фибриллярный кислый белок), ROCK1 (Rho-ассоциированная, протеинкиназа 1, содержащая суперспираль), МЕСР2 (метил-CpG-связывающий белок 2 (синдром Ретта)), MYLK (киназа легкой цепи миозина), ВСНЕ (бутирилхолинэстераза), LIPE (липаза, гормон-чувствительная), PRDX5 (пероксиредоксин 5), ADORA1 (рецептор аденозина A1), WRN (синдром Вернера, белок, подобный хеликазе RecQ), CXCR3 (рецептор 3 хемокина (мотив С-Х-С)), CD81 (молекула CD81), SMAD7 (представитель 7 семейства SMAD), LAMC2 (ламинин, гамма 2), MAP3K5 (митоген-активируемая протеинкиназа 5), CHGA (хромогранин А (паратиреоидный секреторный белок 1)), LAPP (островковый амилоидный полипептид), RHO (родопсин), ENPP1 (эктонуклеотидпирофосфатаза/фосфодиэстераза 1), PTHLH (гормон, подобный паратиреоидному гормону), NRG1 (нейрегулин 1), VEGFC (фактор роста эндотелия сосудов С), ENPEP (глутамиламинопептидаза (аминопептидаза А)), СЕВРВ (ССААТ/энхансер-связывающий белок (С/ЕВР), бета), NAGLU (N-ацетилглюкозаминидаза, альфа-), F2RL3 (белок 3, подобный рецептору фактора коагуляции II (тромбина)), CX3CL1 (лиганд 1 хемокина (мотив С-Х3-С)), BDKRB1 (брадикининовый рецептор B1), ADAMTS13 (ADAM металлопептидаза с мотивом тромбоспондина типа 1, 13), ELANE (эластаза, экспрессируемая нейтрофилами), ENPP2 (эктонуклеотидпирофосфатаза/фосфодиэстераза 2), CISH (индуцируемый цитокинами белок, содержащий SH2), GAST (гастрин), MYOC (миоциллин, обеспечивающий индуцируемую чувствительность трабекулярной сети к глюкокортикоидам), АТР1А2 (АТФаза, На+/K+-транспортирующая, альфа 2 полипептид), NF1 (нейрофибромин 1), GJB1 (белок межклеточных щелевых контактов, бета 1, 32 кДа), MEF2A (энхансерный фактор миоцитов 2А), VCL (винкулин), BMPR2 (рецептор костного морфогенетического белка, тип II (серин/треонинкиназа)), TUBB (тубулин, бета), CDC42 (белок 42 цикла клеточного деления (GTP-связывающий белок, 25 кДа)), KRT18 (кератин 18), HSF1 (транскрипционный фактор 1 теплового шока), MYB (гомолог онкогена v-myb вируса миелобластоза (птичий)), PRKAA2 (протеинкиназа, АМР-активируемая, альфа 2 каталитическая субъединица), ROCK2 (Rho-ассоциированная, протеинкиназа 2, содержащая суперспираль), TFPI (ингибитор пути тканевого фактора (липопротеин-ассоциированный ингибитор коагуляции)), PRKG1 (протеинкиназа, cGMP-зависимая, тип I), BMP2 (костный морфогенетический белок 2), CTNND1 (катенин (кадгерин-ассоциированный белок), дельта 1), СТН (цистатионаза (цистатион гамма-лиаза)), CTSS (катепсин S), VAV2 (фактор обмена гуаниновых нуклеотидов vav 2), NPY2R (рецептор Y2 нейропептида Y), IGFBP2 (белок 2, связывающий инсулиноподобный фактор роста, 36 кДа), CD28 (молекула CD28), GSTA1 (глутатион-S-трансфераза альфа 1), PPIA (пептидилпролилизомераза А (циклофилин А)), АРОН (аполипопротеин Н (бета-2-гликопротеин I)), S100A8 (S100 кальций-связывающий белок А8), IL11 (интерлейкин 11), ALOX15 (арахидонат-15-липоксигеназа), FBLN1 (фибулин 1), NR1H3 (ядерный рецептор, подсемейство 1, группа Н, представитель 3), SCD (стеароил-СоА-десатураза (дельта-9-десатураза)), GIP (желудочный ингибиторный полипептид), CHGB (хромогранин В (секретогранин 1)), PRKCB (протеинкиназа С, бета), SRD5A1 (стероид-5-альфа-редуктаза, альфа-полипетид 1 (3-оксо-5-альфа-стероид-дельта-4-дегидрогеназа альфа 1)), HSD11B2 (гидроксистероид (11-бета) дегидрогеназа 2), CALCRL (белок, подобный кальцитониновому рецептору), GALNT2 (UDP-N-ацетил-альфа-D-галактозамин: полипептид N-ацетилгалактозаминилтрансфераза 2 (GalNAc-T2)), ANGPTL4 (белок 4, подобный ангиопоэтину), KCNN4 (кальций-активируемый калиевый канал средней/малой проводимости, подсемейство N, представитель 4), PIK3C2A (фосфоинозитид-3-киназа, класс 2, альфа-полипетид), HBEGF (гепарин-связывающий EGF-подобный фактор роста), CYP7A1 (цитохром Р450, семейство 7, подсемейство А, полипептид 1), HLA-DRB5 (главный комплекс гистосовместимости, класс II, DR бета 5), BNIP3 (19 кДа белок 3 аденовируса Е1В, взаимодействующий с BCL2), GCKR (регулятор глюкокиназы (гексокиназы 4)), S100A12 (S100 кальций-связывающий белок А12), PAD14 (пептидиларгининдезиминаза, тип IV), HSPA14 (70 кДа белок 14 теплового шока), CXCR1 (рецептор 1 хемокина (мотив С-Х-С)), H19 (H19, подвергающийся импринтингу, экспрессируемый с материнской хромосомы транскрипт (не кодирующий белок)), KRTAP19-3 (кератин-ассоциированный белок 19-3), IDDM2 (инсулин-зависимый белок 2, связанный с сахарным диабетом), RAC2 (родственный ras субстрат 2 ботулотоксина С3 (семейство rho, малый GTP-связывающий белок Rac2)), RYR1 (рианодиновый рецептор 1 (скелетный)), CLOCK (гомолог clock (мышь)), NGFR (суперсемейство рецепторов фактора роста нервов (TNFR, представитель 16)), DBH (допамин-бета-гидроксилаза (допамин-бета-монооксигеназа)), CHRNA4 (холинергический рецептор, никотиновый, альфа 4), CACNA1C (кальциевый канал, потенциалзависимый, L-тип, субъединица альфа 1C), PRKAG2 (протеинкиназа, АМР-активируемая, некаталитическая субъединица гамма 2), CHAT (холинацетилтрансфераза), PTGDS (21 кДа простагландин-D2-синтаза (головной мозг)), NR1H2 (ядерный рецептор, подсемейство 1, группа Н, представитель 2), TEK (тирозинкиназа TEK, эндотелиальная), VEGFB (фактор роста эндотелия сосудов В), MEF2C (энхансерный фактор миопитов 2С), MAPKAPK2 (митоген-активируемая протеинкиназа-активируемая протеинкиназа 2), TNFRSF11A (суперсемейство рецепторов фактора некроза опухоли, представитель 11а, активатор NFKB), HSPA9 (70 кДа белок 9 теплового шока (морталин)), CYSLTR1 (цистеиниллейкотриеновый рецептор 1), МАТ1А (метионинаденозилтрансфераза I, альфа), OPRL1 (белок 1, подобный опиатному рецептору), IMPA1 (инозитол(мио)-1(или 4)-монофосфатаза 1), CLCN2 (хлорный канал 2), DLD (дигидролипоамиддегидрогеназа), PSMA6 (субъединица протеасомы (просома, макропаин), тип альфа, 6), PSMB8 (субъединица протеасомы (просома, макропаин), тип бета, 8 (большая мультифункциональная пептидаза 7)), CHI3L1 (белок 1, подобный хитиназе 3 (хрящевой гликопротеин-39)), ALDH1B1 (семейство альдегиддегидрогеназы 1, представитель B1), PARP2 (полимераза 2 поли(ADP-рибозы)), STAR (стероидогенный острый регуляторный белок), LBP (липополисахарид-связывающий белок), АВСС6 (АТФ-связывающая кассета, подсемейство C(CFTR/MRP), представитель 6), RGS2 (регулятор 2 передачи сигнала G-белком, 24 кДа), EFNB2 (эфрин-В2), GJB6 (белок межклеточных щелевых контактов, бета 6, 30 кДа), АРОА2 (аполипопротеин A-II), AMPD1 (аденозинмонофосфатдезаминаза 1), DYSF (дисферлин, белок 2В, связанный с тазово-плечевой мышечной дистрофией (аутосомно-рецессивное наследование)), FDFT1 (фарнезилдифосфатфарнезилтрансфераза 1), EDN2 (эндотелин 2), CCR6 (рецептор 6 хемокина (мотив С-С)), GJB3 (белок межклеточных щелевых контактов, бета 3, 31 кДа), IL1RL1 (белок 1, подобный рецептору интерлейкина 1), ENTPD1 (эктонуклеозидтрифосфатдифосфогидролаза 1), BBS4 (белок 4, связанный с синдромом Барде-Бидля), CELSR2 (кадгерин, рецептор 2 G-типа с доменами EGF и LAG (гомолог flamingo, Drosophila)), F11R (рецептор F11), RAPGEF3 (фактор обмена гуаниновых нуклеотидов Rap (GEF) 3), HYAL1 (гиалуроноглюкозаминидаза 1), ZNF259 (белок 259, содержащий "цинковые пальцы"), АТОХ1 (гомолог антиоксидантного белка 1 АТХ1 (дрожжи)), ATF6 (активирующий транскрипционный фактор 6), KHK (кетогексокиназа (фруктокиназа)), SAT1 (спермидин/спермин-N1-ацилтрансфераза 1), GGH (гамма-глутамилгидролаза (конъюгаза, фолилполигаммаглутамилгидролаза)), TIMP4 (ингибитор 4 металлопептидазы TIMP), SLC4A4 (семейство 4 переносчиков растворенных веществ, котранспортер бикарбоната натрия, представитель 4), PDE2A (фосфодиэстераза 2А, cGMP-стимулируемая), PDE3B (фосфодиэстераза 3В, cGMP-ингибируемая), FADS1 (десатураза 1 жирных кислот), FADS2 (десатураза 2 жирных кислот), TMSB4X (тимозин бета 4, Х-сцепленный), TXNIP (белок, взаимодействующий с тиоредоксином), LIMS1 (домены 1, подобные антигенам LIM и сенесцентных клеток), RHOB (гомолог белка семейства генов ras, представитель В), LY96 (лимфоцитарный антиген 96), FOXO1 (белок forkhead box O1), PNPLA2 (белок 2, содержащий домен фосфолипазы, подобной пататину), TRH (тиреотропин-рилизинг гормон), GJC1 (белок межклеточных щелевых контактов, гамма 1, 45 кДа), SLC17A5 (семейство 17 переносчиков растворенных веществ (анионный/сахарный транспортер), представитель 5), FTO (белок, ассоциированный с жировой массой и ожирением), GJD2 (белок межклеточных щелевых контактов, дельта 2, 36 кДа), PSRC1 (белок 1 с суперспиралью, богатой пролином/серином), CASP12 (каспаза 12 (ген/псевдоген)), GPBAR1 (G белок-связанный рецептор 1 желчной кислоты), PXK (серин/треонинкиназа, содержащая домен РХ), IL33 (интерлейкин 33), TRIB1 (гомолог 1 белка tribbles (Drosophila)), PBX4 (гомеобокс 4, связанный с лейкозом с вовлечением В-клеток-предшественников), NUPR1 (ядерный белок, регулятор транскрипции, 1), 15-Sep (15 кДа селенопротеин), CILP2 (хрящевой белок 2 промежуточного слоя), TERC (РНК-компонент теломеразы), GGT2 (гамма-глутамилтрансфераза 2), МТ-CO1 (кодируемая митохондриальным геномом цитохром с-оксидаза I) и UOX (уратоксидаза, псевдоген).
В дополнительном варианте осуществления хромосомная последовательность также может быть выбрана из следующих: Pon1 (параоксоназа 1), LDLR (рецептор LDL), АроЕ (аполипопротеин Е), Аро В-100 (аполипопротеин В-100), АроА (аполипопротеин(а)), АроА1 (аполипопротеин A1), CBS (цистатион-В-синтаза), гликопротеин IIb/IIb, MTHRF (5,10-метилентетрагидрофолатредуктаза (NADPH) и их комбинаций. В одном случае хромосомные последовательности и белки, кодируемые хромосомными последовательностями, связанные с сердечно-сосудистым заболеванием, могут быть выбраны из Cacna1C, Sod1, Pten, Ppar(альфа), Аро Е, лептина и их комбинаций.
Почки
Настоящее изобретение также предусматривает доставку системы CRISPR-Cas в почку. Стратегии доставки для индукции поглощения клетками терапевитической нуклеиновой кислоты предусматривают использование физических сил или векторных систем, например, доставка с использованием вирусов, липидов, или комплексов, или наноносителей. Исходя из первоначальных применений, имеющих незначительную возможную клиническую значимость, в случае доставки нуклеиновых кислот в клетки почки при помощи гидродинамической системной инъекции с созданием высокого давления, широкий диапазон вирусных носителей и носителей, отличных от вирусных, уже применяется для целенаправленного воздействия на посттранскрипционные события в различных животных моделях заболевания почек in vivo (Csaba






Yuan et al. (Am J Physiol Renal Physiol 295: F605-F617, 2008) исследовали, может ли in vivo доставка малых интерферирующих РНК (siRNA), целенаправленно воздействующих на 12/15-липоксигеназный (12/15-LO) путь метаболизма арахидоновой кислоты, приводить к уменьшению повреждения почек и диабетической нефропатии (DN) в модели диабета 1 типа на мышах, которым вводили стрептозотопин путем инъекции. Для достижения большей in vivo доступности и экспрессии siRNA в почке. Yuan et al. использовал двухцепочечные олигонуклеотиды siRNA к 12/15-LO, конъюгированные с холестерином. Приблизительно 400 мкг siRNA вводили мышам путем подкожной инъекции. Способ согласно Yuang и соавт. можно применять по отношению к системе CRISPR Cas согласно настоящему изобретению, что предусматривает подкожную инъекцию человеку 1-2 г CRISPR Cas, конъюгированной с холестерином, для доставки в почки.
Molitoris et al. (J Am Soc Nephrol 20: 1754-1764, 2009) использовал клетки проксимальных канальцев (РТС) в качестве участка реабсорбпии олигопуклеотидов в почке для исследования эффективности siRNA, целенаправленно воздействующей на р53, ключевой белок в апоптическом пути, для предупреждения повреждения почки. "Оголенная" синтетическая siRNA к р53, которую вводили путем внутривенной инъекции через 4 ч после ишемического повреждения, обеспечивала максимальную защиту как РТС, так и функции почки. Данные Molitoris и соавт. указывают на быструю доставку siRNA в клетки проксимальных канальцев после внутривенного введения. Для анализа зависимости эффекта от дозы крысам вводили путем инъекции дозы siP53 0,33; 1, 3 или 5 мг/кг, которые предоставляли в одни и те же четыре момента времени, что давало в результате суммарные дозы 1,32; 4, 12 и 20 мг/кг, соответственно. Все исследованные дозы siRNA приводили к эффекту, связанному с уменьшением SCr, в день один, причем более высокие дозы являлись эффективными в течение приблизительно пяти дней по сравнению с обработанными PBS контрольными крысами с ишемией. Суммарные дозы 12 и 20 мг/кг обеспечивали наилучший защитный эффект. Способ согласно Molitoris и соавт. можно применять по отношению к системе CRISPR Cas согласно настоящему изобретению, что предусматривает введение человеку суммарных доз 12 и 20 мг/кг для доставки в почки.
Thompson et al. (Nucleic Acid Therapeutics, Volume 22, Number 4, 2012) сообщил токсикологические и фармакокинетические свойства синтетических малых интерферирующих РНК I5NP после внутривенного введения грызунам и приматам, отличным от человека. I5NP разработан так, чтобы действовать посредством пути РНК-интерференции (РНКи) для временного ингибирования экспрессии проапоптического белка р53 и создан для защиты клеток от повреждений, связанных с острой ишемией/реперфузией, например, острое повреждение почки, что может возникать при обширной операции на сердце, и отсроченная функция трансплантата, что может возникать после пересадки почки. Дозы 800 мг/кг I5NP для грызунов и 1000 мг/кг I5NP для приматов, отличных от человека, требовались для того, чтобы вызвать нежелательные эффекты, которые у обезьян сводились к непосредственному воздействию на кровь, которое включало бессимптомную активацию комплемента и несколько увеличенное время свертывания крови. У крыс не наблюдали дополнительных нежелательных эффектов при использовании аналога I5NP, предназначенного для крыс, что указывало на то, что эти эффекты, вероятно, представляют собой эффекты, связанные с классом синтетических РНК-дуплексов, а не с токсичностью, обусловленной целевой фармакологической активностью I5NP. Взятые вместе, эти данные согласуются с клиническим исследованием с внутривенным введением I5NP для сохранения функции почек после повреждения, связанного с острой ишемией/реперфузией. Уровень, при котором не наблюдали нежелательных эффектов (NOAEL) у обезьян, составлял 500 мг/кг. Не наблюдали эффектов в отношении параметров сердечно-сосудистой, дыхательной и нервной системы у обезьян после внутривенного введения при уровнях дозы до 25 мг/кг. Следовательно, аналогичная доза может предусматриваться для внутривенного введения CRISPR Cas в почки человека.
Shimizu et al. (J Am Soc Nephrol 21: 622-633, 2010) разработали систему для целенаправленной доставки siRNA в клубочки при помощи средств на основе поли(этиленгликоля) и поли(L-лизина). Диаметр комплекса siRNA/наноноситель составлял от приблизительно 10 до 20 нм, причем данный размер будет позволять ему проходить через окончатый эндотелий для того, чтобы попасть в мезангий. После интраперитонеальной инъекции флуоресцентно меченых комплексов siRNA/наноноситель, Shimizu и соавт. выявляли siRNA в кровотоке в течение длительного времени. Повторное интраперитонеальное введение комплекса siRNA к митоген-активируемой протеинкиназе 1 (MAPK1)/наноноситель подавляло экспрессию мРНК и белка MAPK1 в клубочках в мышиной модели гломерулонефрита. Для исследования накопления siRNA Cy5-меченые siRNA в комплексе с PIC наноносителями (0,5 мл, содержание siRNA 5 нмоль), "оголенные" Су5-меченые siRNA (0,5 мл, 5 нмоль) или Cy5-меченые siRNA, инкапсулированные в HVJ-E (0,5 мл, содержание siRNA 5 нмоль), вводили мышам BALB-c. Способ согласно Shimizu и соавт. можно применять по отношению к системе CRISPR Cas согласно настоящему изобретению, что предусматривает дозу приблизительно 10-20 мкмоль CRISPR Cas в комплексе с наноносителями на приблизительно 1-2 литра для интраперитонеального введения человеку и доставки в почки.
Легкие
Настоящее изобретение также предусматривает доставку системы CRISPR-Cas в одно легкое или оба легких.
Несмотря на то, что векторы на основе AAV-2 были изначально предложены для доставки CFTR в дыхательные пути при CF, другие серотипы, например, AAV-1, AAV-5, AAV-6 и AAV-9, демонстрировали улучшенную эффективность переноса генов в ряде моделей эпителия легких (см., например, Li et al., Molecular Therapy, vol. 17 no. 12, 2067-2077 Dec 2009). Как было продемонстрировано, AAV-1 являлся в -100 раз более эффективным, чем AAV-2 и AAV-5 при in vitro трансдукции эпителиальных клеток дыхательных путей человека, хотя эффективность трансдукции эпителия трахеи мышей in vivo при помощи AAV-1 была равной таковой для AAV-5. Другие исследования продемонстрировали, что AAV-5 являлся в 50 раз более эффективным, чем AAV-2, при доставке генов в эпителий дыхательных путей человека (НАЕ) in vitro и значительно более эффективным в эпителии воздухоносных путей легких мышей in vivo. Также было продемонстрировано, что AAV-6 являлся более эффективным, чем AAV-2, в эпителиальных клетках дыхательных путей человека in vitro и дыхательных путях мышей in vivo. Как было показано, изолят, обнаруженный позже, AAV-9, продемонстрировал большую эффективность переноса генов, чем AAV-5, в назальном и альвеолярном эпителии мышей in vivo, причем экспрессию гена выявляли в течение более 9 месяцев, что позволяет предположить, что AAV может обеспечивать длительную экспрессию генов in vivo, являющуюся необходимым свойством для вектора для доставки гена CFTR. Более того, было продемонстрировано, что AAV-9 можно повторно вводить в легкие мышей без потери экспрессии CFTR и с минимальными последствиями, связанными с иммунной системой. Культуры НАЕ с CF и без CF можно инокулировать на апикальной поверхности 100 мкл векторов AAV в течение нескольких часов (см., например, Li et al., Molecular Therapy, vol. 17 no. 12, 2067-2077 Dec 2009). MOI может варьировать от 1×103 до 4×105 векторных геномов/клетка, в зависимости от концентрации вируса и целей экспериментов. Упомянутые выше векторы предусматриваются для доставки и/или введения согласно настоящему изобретению.
Zamora et al. (Am J Respir Crit Care Med Vol 183. pp 531-538, 2011) представили пример применения терапевтического средства на основе РНК-интерференции для лечения инфекционных заболеваний человека, а также рандомизированного исследования противовирусного лекарственного средства у реципиентов трансплантата легкого, инфицированного респираторным синцитиальным вирусом (RSV). Zamora и соавт. провели рандомизированное, двойное слепое, плацебо-контролируемое исследование у реципиентов LTX с инфекцией дыхательных путей RSV. Пациентам давали возможность получать стандартное лечение RSV. ALN-RSV01 в форме аэрозоля (0,6 мг/кг) или плацебо вводили ежедневно в течение 3 дней. Это исследование продемонстрировало, что терапевтическое средство на основе РНКи, целенаправленно воздействующее на RSV, можно вводить без риска реципиентам LTX с инфекцией RSV. Три ежедневные дозы ALN-RSV01 не приводили в результате к какому-либо обострению симптомов в дыхательных путях или нарушению функции легких и не проявляли каких-либо системных провоспалительных эффектов, таких как индукция цитокинов или CRP. Фармакокинетические исследования продемонстрировали только низкий уровень временного системного воздействия после ингаляции, что согласуется с данными доклинических исследований на животных, демонстрирующих, что ALN-RSV01, вводимый внутривенно или путем ингаляции, подвергается быстрому клиренсу из кровотока при помощи опосредованного экзонуклеазами расщепления и почечной экскреции. Способ согласно Zamora и соавт. можно применять по отношению к системе CRISPR Cas согласно настоящему изобретению и при этом CRISPR Cas в форме аэрозоля, например, при дозе 0,6 мг/кг, может предусматриваться в соответствии с настоящим изобретением.
Что касается примера химерной направляющей РНК к CFTR с мутацией дельта508, см. пример 22, в котором демонстрируется перенос генов или доставка генов системы CRISPR-Cas в дыхательные пути нуждающегося в этом субъекта или пациента, страдающего от муковисцидоза или от связанных с муковисцидозом (CF) симптомов, с использованием частиц аденоассоциированного вируса (AAV). В частности, на примере представлена стратегия репарации для мутации дельта-F508 при муковисцидозе. Этот тип стратегии может применяться у всех организмов. В особенности, что касается CF, подходящие пациенты могут включать следующих: человек, не относящийся к человеку примат, собака, кошка, корова, лошадь и другие домашние животные. В этом случае, авторы настоящего изобретения использовали систему CRISPR-Cas, содержащую фермент Cas9, для целенаправленного воздействия на дельтаF508 или другие мутации, индуцирующие CFTR.
Подвергающиеся лечению субъекты в данном случае получали фармацевтически эффективное количество векторной системы на основе AAV в форме аэрозоля на легкое, доставляемое эндобронхиально при непринужденном дыхании. Таким образом, в общем, доставка в форме аэрозоля является предпочтительной для доставки AAV. Аденовирус или частицу AAV можно использовать для доставки. Подходящие конструкции с генами, каждый из которых функционально связан с одной или несколькими регуляторными последовательностями, можно клонировать в вектор доставки. В этом случае, следующие конструкции представлены в качестве примеров: промотор Cbh или EF1a для Cas9, промотор U6 или HI для химерной направляющей РНК). Предпочтительной схемой является применение химерной направляющей, целенаправленно воздействующей на CFTR с мутацией дельта508, матрицы для репарации мутации дельтаF508 и кодон-оптимизированного фермента Cas9 (предпочтительными Cas9 являются ферменты с нуклеазной или никазной активностью) необязательно с одним или несколькими сигналами или последовательностями ядерной локализации (NLS), например, с двумя (2) NLS. Также предусматриваются конструкции без NLS.
Для идентификации целевого сайта Cas9 авторы настоящего изобретения анализировали геномный локус CFTR человека и идентифицировали целевой сайт Cas9. Предпочтительно, как правило и в случае CF, РАМ может содержать мотив NGG или NNAGAAW.
Соответственно, в случае CF, способ по настоящему изобретению предусматривает манипуляцию с целевой последовательностью в представляющем интерес геномном локусе, включающую доставку не встречающейся в природе или сконструированной композиции, содержащей вирусную векторную систему, содержащую один или несколько вирусных векторов, функционально кодирующих композицию для их экспрессии, где композиция содержит:
не встречающуюся в природе или сконструированную композицию, содержащую векторную систему, содержащую один или несколько векторов, содержащих
I. первый регуляторный элемент, функционально связанный с полинуклеотидной последовательностью химерной РНК (chiRNA) системы CRISPR-CAS, где полинуклеотидная последовательность содержит
(a) направляющую последовательность, способную гибридизироваться с целевой последовательностью, связанной с CF, в подходящей клетке млекопитающего,
(b) парную tracr-последовательность, и
(c) tracr-последовательность, и
II. второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей фермент CRISPR, содержащей по меньшей мере одну или несколько последовательностей ядерной локализации,
где (а), (b) и (с) расположены в ориентации 5'-3',
где компоненты I и II находятся в одном и том же или разных векторах системы,
где при транскрипции парная tracr-последовательность гибридизуется с tracr-последовательностью, а направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью, и
где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизуется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизуется с tracr-последовательностью. Что касается CF, предпочтительные целевые последовательности ДНК содержат CFTR с мутацией дельта508. Предпочтительный РАМ описан выше. Предпочтительным ферментом CRISPR является любой Cas (описанный в данном документе, но, в особенности, описанный в примере 22).
Альтернативы CF включают любое наследственное заболевание и их примеры хорошо известны. Другим предпочтительным способом или применением согласно настоящему изобретению является исправление дефектов в генах ЕМР2А и ЕМР2В, которые, как было идентифицировано, ассоциированы с болезнью Лафора.
В некоторых вариантах осуществления "направляющая последовательность" может отличаться от "направляющей РНК". Направляющая последовательность может относиться к последовательности, составляющей приблизительно 20 п.о., в пределах направляющей РНК, которая определяет целевой сайт.
В некоторых вариантах осуществления Cas9 представляет собой (или происходит из) SpCas9. В этих вариантах осуществления предпочтительными мутациями являются мутации по любому или всем из положений 10, 762, 840, 854, 863 и/или 986 в SpCas9 или соответствующим положениям в других Cas9 (которые могут быть определены, например, при помощи стандартных инструментов сравнения последовательностей. В частности, любые или все из следующих мутаций являются предпочтительными для SpCas9: D10A, Е762А, Н840А, N854A, N863A и/или D986A; а также предусматривается консервативная замена для любого аминокислотного замещения. То же самое (или консервативные замены по этим мутациям) также является предпочтительным в соответствующих положениях других Cas9. Особенно предпочтительными являются D10 и Н840 в SpCas9. Однако, в других Cas9 остатки, соответствующие SpCas9 D10 и Н840, также являются предпочтительными. Преимущественными являются те, которые обеспечивают никазную активность. Эти мутации можно применять согласно всем аспектам настоящего изобретения, не только для лечения CF.
Schwank et al. (Cell Stem Cell, 13: 653-58, 2013) использовали CRISPR/Cas9 для исправления дефекта, ассоциированного с муковисцидозом, в стволовых клетках человека. Целью исследователей являлся ген ионного канала, рецептора трансмембранной проводимости, связанного с муковисцидозом (CFTR). Делеция в CFTR приводит к неправильной укладке белка у пациентов с муковисцидозом. С использованием культивируемых стволовых клеток кишечника, полученных из образцов клеток от двух детей с муковисцидозом, Schwank и соавт. могли исправить дефект с использованием CRISPR вместе с донорной плазмидой, содержащей репаративную последовательность, подлежащую вставке. Исследователи затем вырастили клетки до "органоидов" кишечника или кишок небольшого размера и продемонстрировали, что они нормально функционировали. В этом случае, приблизительно половина клональных органоидов подвергалась надлежащей коррекции наследственного материала.
Мышцы
Настоящее изобретение также предусматривает доставку системы CRISPR-Cas в мышцу (мышцы).
Bortolanza et al. (Molecular Therapy vol. 19 no. 11, 2055-2064 Nov. 2011) продемонстрировали, что системная доставка кассет экспрессии для РНК-интерференции у мышей FRG1 после начала проявления плече-лопаточно-лицевой мышечной дистрофии (FSHD) приводила к дозозависимому длительному нокдауну FRG1 без симптомов токсичности. Bortolanza и соавт. обнаружили, что однократная внутривенная инъекция 5×1012 vg (векторных геномов) rAAV6-sh1FRG1 восстанавливает гистопатологические характеристики мышц и функцию мышц у мышей FRG1. Более подробно, 200 мкл, содержащие 2×1012 или 5×1012 vg вектора в физиологическом растворе, вводили путем инъекции в хвостовую вену с использованием шприца Terumo с иглой 25-ого калибра. Способ согласно Bortolanza и соавт. можно применять в отношении AAV, экспрессирующему CRISPR Cas, и вводить его человеку путем инъекции в дозе приблизительно 2×1015 или 2×1016 vg вектора.
Dumonceaux et al. (Molecular Therapy vol. 18 no. 5, 881-887 May 2010) осуществлял ингибирование пути миостатина с применением методики РНК-интерференции, направленной против мРНК рецептора миостатина AcvRIIb (sh-AcvRIIb). Восстановление квази-дистрофина было опосредовано методикой направленного U7 пропуска экзона (U7-DYS). Векторы на основе аденоассоциированных вирусов, несущих либо только конструкцию sh-AcvrIIb, либо только конструкцию U7-DYS, или комбинацию обоих конструкций вводили путем инъекции в переднюю большеберцовую (ТА) мышцу мышей mdx с дистрофией. Инъекции осуществляли с использованием 1011 геномов вируса AAV. Способ согласно Dumonceaux и соавт. можно применять в отношении AAV, экспрессирующему CRISPR Cas, и вводить его человеку путем инъекции, например, при дозе от приблизительно 1014 до приблизительно 1015 vg вектора.
Kinouchi et al. (Gene Therapy (2008) 15, 1126-1130) сообщили об эффективности in vivo доставки siRNA в скелетные мышцы нормальных или больных мышей посредством образования наночастиц не модифицированной химически siRNA с ателоколлагеном (ATCOL). ATCOL-опосредованное местное применение siRNA, целенаправленно воздействующей на миостатин, отрицательный регулятор роста скелетных мышц, при введении в скелетные мышцы мышей или внутривенно приводило к существенному увеличению мышечной массы в течение нескольких недель после применения. Эти результаты указывают на то, что ATCOL-опосредованное применение siRNA является мощным инструментом для дальнейшего терапевтического применения для лечения заболеваний, в том числе мышечной атрофии. Mst-siRNA (конечная концентрация, 10 мМ) смешивали с ATCOL (конечная концентрация для местного введения, 0,5%) (AteloGene, Kohken, Токио, Япония) в соответствии с инструкциями производителя. После анестезии мышей (самцы C57BL/6 в возрасте 20 недель) при помощи нембутала (25 мг/кг, интраперитонеально) комплекс Mst-siRNA/ATCOL complex вводили путем инъекции в жевательные мышцы и двуглавую мышцу бедра. Способ согласно Kinouchi и соавт. можно применять в отношении CRISPR Cas и вводить ее человеку путем инъекции, например, в дозе от приблизительно 500 до 1000 мл 40 мкМ раствора в мышцу.
Hagstrom et al. (Molecular Therapy Vol. 10, No. 2, August 2004) описывали интраваскулярную методику без использования вируса, которая обеспечивает эффективную и воспроизводимую доставку нуклеиновых кислот в мышечные клетки (мышечные волокна) мышц конечностей млекопитающих. Методика включает инъекцию "оголенной" плазмидной ДНК или siRNA в вену дистальной части конечности, временно изолированную при помощи жгута или пневматической манжеты. Доставка нуклеиновой кислоты в мышечные волокна обеспечивается посредством ее быстрого введения путем инъекции при объеме, достаточном для обеспечения просачивания раствора нуклеиновой кислоты в мышечную ткань. Высокие уровни экспрессии трансгена в скелетной мышце достигались как у мелких, так и у крупных животных при минимальной токсичности. Также были получены доказательства доставки siRNA в мышцу конечности. Для внутривенной инъекции плазмидной ДНК макаку-резусу трехходовый кран присоединяли к двум шприцевым насосам (Model PHD 2000; Harvard Instruments), в каждый из которых помещали один шприц. Через пять минут после инъекции папаверина вводили путем инъекции pDNA (15,5-25,7 мг в 40-100 мл физиологического раствора) при скорости 1,7 или 2,0 мл/с. Это можно воспроизводить в увеличенном масштабе для плазмидной ДНК, экспрессирующей CRISPR Cas согласно настоящему изобретению, причем с инъекцией человеку от приблизительно 300 до 500 мг в 800-2000 мл физиологического раствора. Для инъекции аденовирусного вектора крысе 2×109 инфекционных частиц в 3 мл физиологического солевого раствора (NSS) вводили путем инъекции. Это можно воспроизводить в увеличенном масштабе для аденовирусного вектора, экспрессирующего CRISPR Cas согласно настоящему изобретению, причем путем инъекции человеку приблизительно 1×1013 инфекционных частиц в 10 литрах NSS. Что касается siRNA, крысе вводили путем инъекции в большую подкожную вену 12,5 мкг siRNA, а примату вводили путем инъекции в большую подкожную вену 750 мкг siRNA. Это можно воспроизводить в увеличенном масштабе для CRISPR Cas согласно настоящему изобретению, например, с инъекцией от приблизительно 15 до приблизительно 50 мг в большую подкожную вену человека.
Кожа
Настоящее изобретение также предусматривает доставку системы CRISPR-Cas в кожу.
Hickerson et al. (Molecular Therapy-Nucleic Acids (2013) 2, e129) имеет отношение к снабженному приводом устройству с матрицей микроигл для доставки в кожу, предназначенному для самостоятельной (sd) доставки siRNA в кожу человека и мыши. Основной проблемой, связанной с переносом терапевтических средств на основе siRNA для кожи, в клиническую практику, является разработка эффективных систем доставки. Значительные усилия были приложены к созданию ряда методик доставки в кожу с ограниченным успехом. В клиническом исследовании, в котором кожу обрабатывали siRNA, острая боль, связанная с инъекцией при помощи иглы для подкожных инъекций, препятствовала включению дополнительных пациентов в исследование, что придает большое значение потребности в улучшенных, более "удобных для пациента" (т.е. слабая боль или без боли) средствах доставки. Микроиглы представляют эффективный способ доставки крупных заряженных молекул-карго, включающих siRNA, через первичный барьер, роговой слой, и, как правило, считаются причиняющими меньшую боль, чем обычные иглы для подкожных инъекций. Снабженные приводом устройства "штамповочного типа" с микроиглами, в том числе снабженное приводом устройство с матрицей микроигл (MMNA), используемое Hickerson и соавт., как было продемонстрировано, были безопасными в исследованиях на бесшерстных мышах и причиняли слабую боль или не причиняли боли, о чем свидетельствует (i) широкое применение в косметологии и (ii) ограниченное тестирование, в котором практически все добровольцы считали применение устройства причиняющим намного меньшую боль, чем при вакцинации против гриппа, что позволяет предположить, что результатом доставки siRNA с применением этого устройства будет намного меньшая болезненность, чем испытываемая в предшествующих клинических исследованиях с применением игл для подкожных инъекций. Устройство MMNA (имеющееся в продаже как Triple-M или Tri-M от Bomtech Electronic Co, Сеул, Южная Корея) приспособили для доставки siRNA в кожу мыши и человека. Раствор sd-siRNA (до 300 мкл 0,1 мг/мл РНК) вводили в камеру одноразового инъекционного картриджа с иглами Tri-M (Bomtech), которые устанавливали на глубину 0,1 мм. Для обработки кожи человека деидентифицированную кожу (полученную непосредственно после хирургических процедур) растягивали вручную и прикалывали к пробковому столу перед обработкой. Все интрадермальные инъекции осуществляли при помощи инсулинового шприца с 0,5-дюймовой иглой 28 калибра. Устройство MMNA и способ согласно Hickerson и соавт. можно применять и/или адаптировать для доставки CRISPR Cas согласно настоящему изобретению, например, в дозе до 300 мкл 0,1 мг/мл CRISPR Cas, в кожу.
Leachman et al. (Molecular Therapy, vol. 18 no. 2, 442-446 Feb. 2010) имеет отношение к клиническому исследованию фазы Ib применительно к лечению редкого кожного заболевания врожденной пахионихии (PC), аутосомно-доминантного синдрома, который включает повреждение подошвенной кератодермы, с использованием первого терапевтического средства на основе короткой интерферирующей РНК (siRNA) для кожи. Эта siRNA, под названием TD101, специфично и эффективно целенаправленно воздействует на мРНК мутантного кератина 6а (K6a) N171K, не оказывая влияния на мРНК K6a дикого типа. Схема с увеличением дозы представлена ниже:

Вначале, 0,1 мл 1,0 мг/мл раствора TD101 или только среды (фосфатно-солевого буферного раствора Dulbecco без кальция или магния) вводили в симметрично расположенные мозоли. Определяли шесть возрастающих объемов дозы без нежелательной реакции на увеличения: 0,1, 0,25, 0,5, 1,0, 1,5 и 2,0 мл 1,0 мг/мл раствора TD101 на инъекцию. Так как наиболее высокий запланированный объем (2,0 мл) был хорошо переносимым, концентрацию TD101 затем увеличивали каждую неделю от 1 мг/мл до конечной концентрации 8,5 мг/мл. Аналогичные дозы предусматриваются для введения CRISPR Cas, которая специфично и эффективно целенаправленно воздействует на мРНК мутантного кератина 6а (K6a) N171K.
Zheng et al. (PNAS, July 24, 2012, vol. 109, no. 30, 11975-11980) продемонстрировал, что конъюгаты сферических наночастиц с нуклеиновой кислотой (SNA-NC), причем ядра из золота окружены плотной оболочкой из строго ориентированных, ковалентно иммобилизованных siRNA, свободно проникают практически в 100% кератиноцитов in vitro, в коже мыши и в эпидермисе человека в течение нескольких часов после применения. Zheng et al. продемонстрировал, что однократное применение 25 нМ SNA-NC к рецептору эпидермального фактора роста (EGFR) в течение 60 ч продемонстрировало эффективный нокдаун гена в коже человека. Аналогичная доза может предусматриваться для CRISPR Cas, иммобилизованной в SNA-NC для введения в кожу.
Вирусы гепатита
Настоящее изобретение также можно применять для лечения вируса гепатита В (HBV). Однако, система CRISPR Cas должна быть приспособлена для того, чтобы избежать недостатков РНКи, таких как риск перенасыщения эндогенных путей малых РНК, с помощью, например, оптимизации дозы и последовательности (см., например, Grimm et al., Nature vol. 441, 26 May 2006). Например, предусматриваются низкие дозы, например, приблизительно 1-10×1014 частиц на человека.
В другом варианте осуществления систему CRISPR Cas, направленную против HBV, можно вводить в липосомы, например, стабильная частица из нуклеиновой кислоты и липидов (SNALP) (см., например, Morrissey et al., Nature Biotechnology, Vol. 23, No. 8, August 2005). Предусматриваются ежедневные внутривенные инъекции приблизительно 1, 3 или 5 мг/кг/день CRISPR Cas, целенаправленно воздействующей на РНК HBV, в SNALP. Ежедневное лечение можно осуществлять в течение приблизительно трех дней и затем еженедельно в течение приблизительно пяти недель.
В другом варианте осуществления систему согласно Chen et al. (Gene Therapy (2007) 14, 11-19) можно применять и/или приспособить к системе CRISPR Cas согласно настоящему изобретению. Chen et al. использовали двухцепочечный псевдотипированный вектор на основе аденоассоциированного вируса 8 (dsAAV2/8) для доставки короткой шпилечной (shRNA). Однократное введение вектора dsAAV2/8 (1×1012 векторных геномов на мышь), несущего специфичную к HBV shRNA, эффективно подавляло стабильный уровень белка, мРНК и репликативной ДНК HBV в печени трансгенных мышей с HBV, что приводило к снижению нагрузки HBV в кровотоке на вплоть до 2-3 log10. Значительное подавление HBV продолжалось в течение по меньшей мере 120 дней после введения вектора. Терапевтический эффект shRNA зависел от целевой последовательности и не включал активацию интерферона. В соответствии с настоящим изобретением систему CRISPR Cas, направленную в отношении HBV, можно клонировать в вектор AAV, например, вектор dsAAV2/8, и вводить человеку, например, в дозе от приблизительно 1×1015 векторных геномов до приблизительно 1×1016 векторных геномов на человека.
В другом варианте осуществления способ согласно Wooddell et al. (Molecular Therapy vol. 21 no. 5, 973-985 May 2013) можно использовать и/или адаптировать к системе CRISPR Cas согласно настоящему изобретению. Woodell et al. продемонстрировал, что простая совместная инъекция целенаправленно воздействующего на гепатопиты, конъюгированного с N-ацетилгалактозамином мелиттин-подобного пептида (NAG-MLP) с тропной к печени конъюгированной с холестерином siRNA (chol-siRNA), целенаправленно воздействующей на фактор коагуляции VII (F7), приводит в результате к эффективному нокдауну F7 у мышей и приматов, отличных от человека, без изменений клинических химических показателей или индукции цитокинов. Используя временные и трансгенные мышиные модели инфекции HBV, Wooddell et al. продемонстрировали, что однократная совместная инъекция NAG-MLP с активной chol-siRNA, целенаправленно воздействующей на консервативные последовательности HBV, приводила в результате к многократной репрессии вирусной РНК, белков и вирусной ДНК с большой продолжительностью эффекта. Внутривенные совместные инъекции, например, приблизительно 6 мг/кг NAG-MLP и 6 мг/кг специфичной к HBV CRISPR Cas, могут предусматриваться в соответствии с настоящим изобретением. В альтернативном случае, приблизительно 3 мг/кг NAG-MLP и 3 мг/кг специфичной к HBV CRISPR Cas могут доставляться в день один с последующим введением приблизительно 2-3 мг/кг NAG-MLP и 2-3 мг/кг специфичной к HBV CRISPR Cas две недели спустя.
Настоящее изобретение также можно применять для лечения вируса гепатита С (HCV). Способы согласно Roelvinki et al. (Molecular Therapy vol. 20 no. 9, 1737-1749 Sep 2012) можно применять по отношению к системе CRISPR Cas. Например, вектор AAV, такой как AAV8, может быть предполагаемым вектором и может предусматриваться, например, доза от приблизительно 1,25×1011 до 1,25×1013 векторных геномов на килограмм массы тела (vg/кг).
Будет очевидно, что организм-хозяин с другими заболеваниями можно лечить подобным образом. Некоторые примеры наследственных заболеваний, вызванных мутациями, приведены в данном документе, но известно их намного больше. Изложенную выше стратегию можно применять для лечения этих заболеваний.
Болезнь Хантингтона (HD)
РНК-интерференция (РНКи) предоставляет терапевтические возможности для лечения этого нарушения посредством уменьшения экспрессии НТТ, гена, приводящего к развитию болезни Хантингтона (см., например, McBride et al., Molecular Therapy vol. 19 no. 12 Dec. 2011, pp. 2152-2162), следовательно, заявитель настоящего изобретения предполагает, что ее можно использовать и/или приспособить к системе CRISPR-Cas. Систему CRISPR-Cas можно получить с использованием алгоритма для уменьшения возможности нецелевого воздействия антисмысловых последовательностей. Последовательности CRISPR-Cas могут целенаправленно воздействовать либо на последовательность в экзоне 52 хантингтина мыши, макака-резуса или человека и экспрессироваться вирусным вектором, например, AAV. Животным, в том числе человеку, можно вводить путем приблизительно трех микроинъекций на полушарие (всего шесть инъекций): первая на 1 мм ростральнее передней спайки (12 мкл) и две оставшиеся инъекции (12 мкл и 10 мкл, соответственно) на расстоянии 3 и 6 мм каудальнее по отношению к первой инъекции, причем с 1е12 vg/мл AAV при скорости приблизительно 1 мкл/минута, при этом иглу оставляли на месте в течение дополнительных 5 минут для обеспечения диффузии вводимого вещества с наконечника иглы.
DiFiglia et al. (PNAS, October 23, 2007, vol. 104, no. 43, 17204-17209) наблюдал, что однократное введение в полосатое тело взрослого организма siRNA, целенаправленно воздействующей на Htt, может привести к сайленсингу Htt, ослаблению нейрональной патологии и задержке развития аномального поведенческого фенотипа, наблюдаемого в модели HD на трансгенных мышах, полученной с использованием вируса, с быстрым началом проявления. DiFiglia инъецировал мышам в полосатое тело 2 мкл Су3-меченых cc-siRNA-Htt или неконъюгированных siRNA-Htt при 10 мкМ. Аналогичная доза CRISPR Cas, целенаправленно воздействующей на Htt, может предусматриваться в настоящем изобретении для человека, например, от приблизительно 5 до 10 мл 10 мкМ CRISPR Cas, целенаправленно воздействующей на Htt, можно инъецировать в полосатое тело.
В другом примере Boudreau et al. (Molecular Therapy vol. 17 no. 6 June 2009) инъецировал 5 мкл рекомбинантных векторов на основе вируса AAV серотипа 2/1, экспрессирующих htt-специфичное средство для РНКи (при 4×1012 вирусных геномов/мл) в полосатое тело. Аналогичная доза CRISPR Cas, целенаправленно воздействующей на Htt, может предусматриваться в настоящем изобретении для человека, например, от приблизительно 10 до 20 мл 4×1012 вирусных геномов/мл), причем CRISPR Cas, целенаправленно воздействующую на Htt, можно инъецировать в полосатое тело.
В другом примере CRISPR Cas, целенаправленно воздействующую на HTT, можно вводить непрерывно (см., например, Yu et al., Cell 150, 895-908, August 31, 2012). Yu et al. использовал доставку при помощи осмотических насосов, обеспечивающих 0,25 мл/ч (Model 2004), для доставки 300 мг/день ss-siRNA или фосфатно-солевого буферного раствора (PBS) (Sigma Aldrich) в течение 28 дней, и насосы, выполненные с возможностью доставки 0,5 мкл/ч (Model 2002), испольозовали для доставки 75 мг/день МОЕ ASO положительного контроля в течение 14 дней. Насосы (Durect Corporation) заполняли ss-siRNA или МОЕ, разведенным стерильным PBS, а затем инкубировали при 37°С в течение 24 или 48 (Model 2004) часов перед имплантацией. Мышей анестезировали 2,5% изофлураном и делали срединный разрез у основания черепа. Используя стереотаксические зонды имплантировали канюлю в боковой правый желудочек и закрепляли при помощи клея Loctite. Катетер, прикрепленный к осмотическому мининасосу Alzet, прикрепляли к канюле, и насос размещали подкожно между лопатками. Разрез закрывали швами, используя нейлон 5,0. Аналогичная доза CRISPR Cas, целенаправленно воздействующей на Htt, может предусматриваться в настоящем изобретении для человека, например, можно вводить от приблизительно 500 до 1000 г/день CRISPR Cas, целенаправленно воздействующей на Htt.
В другом примере непрерывной инфузии Stiles et al. (Experimental Neurology 233 (2012) 463-471) имплантировал интрапаренхиматозный катетер с титановым наконечником иглы в правую скорлупу. Катетер присоединяли к насосу SynchroMed® II (Medtronic Neurological, Миннеаполис, Миннесота), подкожно имплантированному в области живота. После 7 дней инфузии фосфатно-солевого буферного раствора при 6 мкл/день насосы повторно заполняли исследуемым препаратом и программировали на непрерывную доставку в течение 7 дней. От приблизительно 2,3 до 11,52 мг/день siRNA вводили путем инфузии при различных значениях скорости инфузии от приблизительно 0,1 до 0,5 мкл/мин. Аналогичная доза CRISPR Cas, целенаправленно воздействующей на Htt, может предусматриваться в настоящем изобретении для человека, например, можно вводить от приблизительно 20 до 200 мг/день CRISPR Cas, целенаправленно воздействующей на Htt.
В другом примере способы согласно публикации патентного документа США №20130253040, закрепленного за Sangamo, также можно адаптировать исходя из TALES к системе CRISPR Cas согласно настоящему изобретению для лечения болезни Хантингтона.
Нуклеиновые кислоты, аминокислоты и белки
В настоящем изобретении нуклеиновые кислоты используются для связывания целевых последовательностей ДНК. Это является преимущественным, поскольку получать нуклеиновые кислоты намного легче и дешевле, чем белки, и специфичность может варьировать в зависимости от длины фрагмента, если необходима гомология. Например, не требуется сложное 3-D определение положений многочисленных доменов.
Выражения "полинуклеотид", "нуклеотид", "нуклеотидная последовательность", "нуклеиновая кислота" и "олигонуклеотид" используют взаимозаменяемо. Они обозначают полимерную форму нуклеотидов любой длины, как дезоксирибонуклеотидов, так и рибонуклеотидов или их аналогов. Полинуклеотиды могут обладать любой пространственной структурой и могут выполнять любую функцию, известную или неизвестную. Неограничивающими примерами полинуклеотидов являются следующие:
кодирующие или некодирующе участки гена или фрагмента гена, локусы (локус), определенные в результате анализа сцепления, экзоны, интроны, информационная РНК (иРНК), транспортная РНК, рибосомная РНК, короткая интерферирующая РНК (siRNA), короткая шпилечная РНК (shRNA), микроРНК (miRNA), рибозимы, кДНК, рекомбинантные полинуклеотиды, разветвленные полинуклеотиды, плазмиды, векторы, выделенная ДНК любой последовательности, выделенная РНК любой последовательности, зонды для нуклеиновых кислот и праймеры. Выражение также охватывает структуры, подобные нуклеиновым кислотам с синтетическими остовами, см., например, Eckstein, 1991; Baserga et al., 1992; Milligan, 1993; WO 97/03211; WO 96/39154; Mata, 1997; Strauss-Soukup, 1997 и Samstag, 1996. Полинуклеотид может содержать один или несколько модифицированных нуклеотидов, как, например, метилированные нуклеотиды и аналоги нуклеотидов. При наличии, модификации в нуклеотидную структуру могут быть внесены до или после сборки полимера. Последовательность нуклеотидов может прерываться отличными от нуклеотидов компонентами. Полинуклеотид можно дополнительно модифицировать после полимеризации, как, например, путем соединения с компонентом для мечения.
Используемое в данном документе выражение "дикий тип" является выражением из данной области техники, понятным специалисту в данной области, и означает типичную форму организма, штамма, гена или характеристики, которые встречаются в природе, в отличие от мутантных или вариантных форм.
Используемое в данном документе выражение "вариант" следует понимать как означающее проявление качеств, которые характеризуются паттерном, отличающимся от такового, который встречается в природе.
Выражения "не встречающийся в природе" или "сконструированный" используют взаимозаменяемо, и оно указывает на вмешательство человека. Выражения, в тех случаях, когда они касаются молекул нуклеиновых кислот или полипептидов, означают, что молекула нуклеиновой кислоты или полипептид по меньшей мере практически не содержат по меньшей мере один отличный компонент, с которым они естественным образом связаны в природе и встречаются в природе.
"Комплементарность" означает способность нуклеиновой кислоты образовывать водородную(ые) связь(и) с другой последовательностью нуклеиновой кислоты при помощи либо традиционного спаривания оснований по Уотсону-Крику, либо других нетрадиционных типов. Процент комплементарности показывает процентную долю остатков в молекуле нуклеиновой кислоты, которые могут образовывать водородные связи (к примеру, образование пар по Уотсону-Крику) со второй последовательностью нуклеиновой кислоты (к примеру, при этом 5, 6, 7, 8, 9, 10 из 10 будут на 50%, 60%, 70%, 80%, 90% и 100% комплементарны). "Точная комплементарность" означает, что все смежные остатки последовательности нуклеиновой кислоты будут связаны водородными связями с тем же количеством смежных остатков во второй последовательности нуклеиновой кислоты. Выражение "практически комплементарный", используемое в данном документе, означает степень комплементарности, которая составляет по меньшей мере 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 97%, 98%, 99% или 100% в пределах участка из 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 30, 35, 40, 45, 50 или более нуклеотидов, или относится к двум нуклеиновым кислотам, которые гибридизуются при жестких условиях.
Используемые в данном документе "жесткие условия" в отношении гибридизации означают условия, при которых нуклеиновая кислота с комплементарностью к целевой последовательности преимущественно гибридизуется с целевой последовательностью и практически не гибридизуется с нецелевыми последовательностями. Жесткие условия, как правило, являются зависимыми от последовательности и изменяются в зависимости от ряда факторов. Как правило, чем длиннее последовательность, тем выше температура, при которой последовательность специфично гибридизуется с целевой последовательностью. Неограничивающие примеры жестких условий описаны подробно в Tijssen (1993), Laboratory Techniques In Biochemistry And Molecular Biology-Hybridization With Nucleic Acid Probes Part I, Second Chapter "Overview of principles of hybridization and the strategy of nucleic acid probe assay", Elsevier, N.Y. Если предполагается полинуклеотидная последовательность, то также предусматриваются комплементарные или частично комплементарные последовательности. Они предпочтительно способны гибридизоваться с эталонной последовательностью при очень жестких условиях. Как правило, для доведения до максимума скорости гибридизации, выбирают условия гибридизации относительно низкой жесткости: температура на от приблизительно 20 до 25°С ниже температуры точки плавления (Tm). Tm представляет собой температуру, при которой 50% специфичной целевой последовательности гибридизуется с точно комплементарным зондом в растворе при определенной ионной силе и рН. Как правило, если требуется по меньшей мере приблизительно 85% нуклеотидная комплементарность гибридизованных последовательностей, выбирают очень жесткие условия отмывки с температурой на от приблизительно 5 до 15°С ниже, чем Tm. Если требуется по меньшей мере приблизительно 70% нуклеотидная комплементарность гибридизованных последовательностей, выбирают умеренно жесткие условия отмывки с температурой на от приблизительно 15 до 30°С ниже, чем Tm. Высоко пермиссивные (очень низкой жесткости) условия отмывки могут характеризоваться наименьшей температурой на 50°С ниже Tm, что допускает высокий уровень ошибочных совпадений между гибридизованными последовательностями. Специалисты в данной области поймут, что другие физические и химические параметры на стадиях гибридизации и отмывки также можно изменять для того, чтобы повлиять на получаемый в результате выявляемый сигнал гибридизации исходя из конкретного уровня гомологии между целевой последовательностью и последовательностью зонда. Предпочтительные условия высокой жесткости предусматривают инкубирование в 50% формамиде, 5×SSC и 1% SDS при 42°С или инкубирование в 5×SSC и 1% SDS при 65°C с отмывкой в 0,2×SSC и 0,1% SDS при 65°С.
"Гибридизация" означает реакцию, при которой один или несколько полинуклеотидов реагируют с образованием комплекса, который стабилизируется посредством образования водородных связей между основаниями нуклеотидных остатков. Образование водородных связей может происходить по принципу спаривания оснований по Уотсону-Крику, Хугстиновского связывания или любым другим специфичным к последовательности образом. Комплекс может содержать две нити, образующие дуплексную структуру, три или более нитей, образующих многонитевой комплекс, одиночную самогибридизующуюся нить или любую их комбинацию. Реакция гибридизации может представлять собой стадию в более обширном способе, такую как начальная стадия ПЦР или расщепление полинуклеотида при помощи фермента. Последовательность, способную гибридизоваться с данной последовательностью, называют "комплементарной последовательностью" данной последовательности.
Используемое в данном документе выражение "геномный локус" или "локус" (форма множественного числа локусы) представляет собой специфическое положение гена или последовательности ДНК на хромосоме. "Ген" относится к фрагментам ДНК или РНК, которые кодируют цепь полипептида или РНК, которые играют функциональную роль в организме, и, следовательно, представляют собой молекулярную единицу наследственности в живых организмах. Для цели настоящего изобретения может считаться, что гены содержат участки, которые регулируют образование продукта гена, независимо от того являются ли регуляторные последовательности смежными с кодирующими и/или транскрибируемыми последовательностями или нет. Соответственно, ген содержит, но без обязательного ограничения, промоторные последовательности, терминаторы, регуляторные последовательности трансляции, например, участки связывания рибосомы и участки внутренней посадки рибосомы, энхансеры, сайленсеры, инсуляторы, граничные элементы, точки начала репликации, участки прикрепления к матриксу и регуляторные участки локуса.
Используемое в данном документе выражение "экспрессия геномного локуса" или "экспрессия гена" относится к процессу, в ходе которого информация гена используется в синтезе функционального продукта гена. Продукты экспрессии генов часто представляют собой белки, но у генов, не кодирующих белки, например, гены рРНК или гены тРНК, продукт представляет собой функциональную РНК. Процесс экспрессии генов используется всеми известными живыми организмами - эукариотами (в том числе многоклеточными организмами), прокариотами (бактериями и археями) и вирусами для образования функциональных продуктов, необходимых для выживания. Используемое в данном документе выражение "экспрессия" гена или нуклеиновой кислоты охватывает не только экспрессию генов в клетках, но также транскрипцию и трансляцию нуклеиновой кислоты(нуклеиновых кислот) в системах клонирования и в любом другом контексте. Используемое в данном документе выражение "экспрессия" означает процесс, при котором полинуклеотид транскрибируется с ДНК-матрицы (как, например, с образованием мРНК или другого РНК-транскрипта), и/или процесс, при помощи которого транскрибированная мРНК далее транслируется с образованием пептидов, полипептидов или белков. Транскрипты и закодированные полипептиды можно в совокупности называть "продуктом гена". Если полинуклеотид получен из геномной ДНК, то экспрессия может включать сплайсинг мРНК в эукариотической клетке.
Выражения "полипептид", "пептид" и "белок" используют взаимозаменяемо в данном документе для обозначения полимеров из аминокислот любой длины. Полимер может быть линейным или разветвленным, он может содержать модифицированные аминокислоты, и его структура может прерываться отличными от аминокислот компонентами. Выражения также охватывают полимер из аминокислот, который был модифицирован; например, образованием дисульфидных связей, гликозилированием, липидизацией, ацетилированием, фосфорилированием или любой другой манипуляцией, такой как соединение с компонентом для мечения. Используемое в данном документе выражение "аминокислота" включает природные, и/или отличные от природных, или синтетические аминокислоты, в том числе глицин и как D-, так и L-оптические изомеры, и аналоги аминокислот, и пептидомиметики.
Используемое в данном документе выражение "домен" или "домен белка" относится к части последовательности белка, которая может существовать и функционировать независимо от остальной части белковой цепи.
Как описано в аспектах согласно настоящему изобретению, идентичность последовательности относится к гомологии последовательности. Сравнения гомологии можно проводить на глаз или, что делается чаще, с помощью легко доступных программ для сравнения последовательностей. При помощи этих коммерчески доступных компьютерных программ можно рассчитывать процент (%) гомологии между двумя или более последовательностями и также можно рассчитывать идентичность последовательности между двумя или более аминокислотными последовательностями или последовательностями нуклеиновых кислот. В некоторых предпочтительных вариантах осуществления участок кэпирования dTALE, описанный в данном документе, имеет последовательности по меньшей мере на 95% идентичные или обладающие идентичностью с участком кэпирования аминокислотных последовательностей, представленных в данном документе.
Значения гомологии последовательности можно получить при помощи любой из ряда компьютерных программ, известных из уровня техники, например, BLAST или FASTA и т.д. Подходящей компьютерной программой для осуществления такого выравнивания является пакет программ GCG Wisconsin Bestfit (Университет Висконсина, США; Devereux et al., 1984, Nucleic Acids Research 12: 387). Примеры другого программного обеспечения, при помощи которого можно осуществлять сравнения последовательностей, включают без ограничения пакет программ BLAST (см. Ausubel et al., 1999 ibid - Chapter 18), FASTA (Atschul et al., 1990, J. Mol. Biol., 403-410) и набор инструментов для сравнения GENEWORKS. Как в BLAST, так и в FASTA доступны оффлайн- и онлайн-поиск (см. Ausubel et al., 1999 ibid, pages 7-58 to 7-60). Однако, предпочтительным является использование программы GCG Bestfit.
Процент (%) гомологии последовательности можно рассчитывать для непрерывных последовательностей, т.е. одну последовательность выравнивают с другой последовательностью и каждую аминокислоту или нуклеотид в одной последовательности непосредственно сравнивают с соответствующей аминокислотой или нуклеотидом в другой последовательности, один остаток за один раз. Это называется выравниванием "без гэпов". Как правило, такие выравнивания без гэпов осуществляют только для относительно малого числа остатков.
Несмотря на то, что этот способ является очень простым и соответствующим, при его применении не учитывается то, что, например, в паре последовательностей, которые в остальном являются идентичными, одна вставка или делеция может привести к тому, что следующие за ней аминокислотные остатки не будут учитываться при выравнивании, что, таким образом, потенциально приводит в результате к значительному уменьшению % гомологии при осуществлении глобального выравнивания. Следовательно, большинство способов сравнения последовательностей разработаны для получения оптимальных выравниваний, в которых учитываются возможные вставки и делении без наложения чрезмерного штрафа на общую гомологию или балл идентичности. Это достигается путем вставки "гэпов" в выравнивание последовательностей в попытке доведения до максимума локальной гомологии или идентичности.
Однако, в этих более сложных способах назначаются "штрафы за внесения гэпа" для каждого гэпа, который встречается при выравнивании, таким образом, для одинакового количества идентичных аминокислот выравнивание последовательностей с наименьшим количеством гэпов, что отражает высокую степень родства между двумя сравниваемыми последовательностями, может привести в результате к более высокому баллу, чем выравнивание с большим количеством гэпов. Как правило, используют "значения аффинного штрафа за внесение гэпа" для родственных последовательностей", с использованием которых отчисляют относительно высокое значение за существование гэпа и меньший штраф за каждый последующий остаток в гэпе. Это наиболее часто используемая система оценки гэпов. Высокие штрафы за внесение гэпа, конечно, могут привести к оптимизированным выравниваниям с меньшим количеством гэпов. В большинстве программ выравнивания допускается изменение штрафов за внесение гэпа. Однако, предпочтительно использовать значения по умолчанию при использовании такого программного обеспечения для сравнений последовательностей. Например, при использовании пакета программ GCG Wisconsin Bestfit штраф за внесение гэпа по умолчанию для аминокислотной последовательности составляет -12 для гэпа и -4 за каждый остаток его продолжения.
Для расчета максимального % гомологии, следовательно, изначально требуется получение оптимального выравнивания с учетом штрафов за внесение гэпа. Подходящая компьютерная программа для осуществления такого выравнивания представляет собой пакета программ GCG Wisconsin Bestfit (Devereux et al., 1984 Nuc. Acids Research 12 p387). Примеры другого программного обеспечения, при помощи которого можно осуществлять сравнения последовательностей, включают без ограничения пакет программ BLAST (см. Ausubel et al., 1999 Short Protocols in Molecular Biology, 4й Ed. - Chapter 18), FASTA (Altschul et al., 1990 J. Mol. Biol. 403-410) и набор инструментов для сравнения GENEWORKS. Как в BLAST, так и в FASTA доступны оффлайн- и онлайн-поиск (см. Ausubel et al., 1999, Short Protocols in Molecular Biology, pages 7-58 to 7-60). Однако, для некоторых задач предпочтительно использовать программу GCG Bestfit. Новый инструмент под названием BLAST 2 Sequences также доступен для сравнения белковых и нуклеотидных последовательностей (см. FEMS Microbiol Lett. 1999 174(2): 247-50; FEMS Microbiol Lett. 1999 177(1): 187-8 и веб-сайт Национального центра биотехнологической информации на веб-сайте Национального института здоровья).
Несмотря на то, что конечный % гомологии можно измерять в единицах идентичности, способ выравнивания сам по себе, как правило, не основывается на сравнении пар по типу "все или ничего". Вместо этого, как правило, используется матрица замен со шкалой сходства, с использованием которой назначаются баллы для каждого попарного сравнения исходя из химического сходства или эволюционного расстояния. Примером этой матрицы, используемой чаще всего, является матрица BLOSUM62 - матрица по умолчанию для набора программ BLAST. В программах GCG Wisconsin, как правило, используются либо общедоступные значения по умолчанию или специальные таблицы сравнения символов, если предоставляются (дополнительные подробности см. в руководстве пользователя). Для некоторых задач, предпочтительным является применение общедоступных значений по умолчанию для пакета программ GCG пли, в случае другого программного обеспечения, матрицы по умолчанию, например, BLOSUM62.
В альтернативном случае, процентные значения гомологии можно рассчитывать с использованием функции множественного выравнивания в DNASIS™ (Hitachi Software) с применением алгоритма, аналогичного CLUSTAL (Higgins DG & Sharp PM (1988), Gene 73(1), 237-244). После того, как программное обеспечение предоставит оптимальное выравнивание, возможно рассчитать % гомологии, предпочтительно % идентичности последовательности. Программное обеспечение, как правило, осуществляет это в ходе сравнения последовательностей и выдает численный результат.
Последовательности также могут иметь делении, вставки или замены аминокислотных остатков, которые приводят к молчащему изменению и приводят в результате к функционально эквивалентному веществу. Преднамеренные аминокислотные замены могут быть сделаны исходя из сходства свойств аминокислот (например, полярность, заряд, растворимость, гидрофобность, гидрофильность и/или амфинатическая природа остатков) и, следовательно, они являются применимыми для того, чтобы сгруппировать аминокислоты в функциональные группы. Аминокислоты можно сгруппировать исходя из свойств только их боковых цепей. Однако, также более полезно включить данные о мутациях. Группы аминокислот, полученные таким образом, вероятно, будут консервативными по структурным причинам. Эти группы могут быть описаны в форме диаграммы Венна (Livingstone C.D. and Barton G.J. (1993) "Protein sequence alignments: a strategy for the hierarchical analysis of residue conservation" Comput. Appl. Biosci. 9: 745-756) (Taylor W.R. (1986) "The classification of amino acid conservation" J. Theor. Biol. 119; 205-218). Консервативные замены могут быть сделаны, например, в соответствии с таблицей, представленной ниже, в которой описывается общепринятая группировка аминокислот в форме диаграммы Венна.

Варианты осуществления согласно настоящему изобретению охватывают последовательности (как полинуклеотидные, так и полипептидные), которые могут содержать гомологичную замену (используемые в данном документе, как замена, так и замещение означают обмен существующего аминокислотного остатка или нуклеотида на альтернативный остаток или нуклеотид), которая может происходить, т.е., в случае аминокислот, замена на аналогичную, например, основная на основную, кислая на кислую, полярная на полярную и т.д. Также может происходить негомологичная замена, т.е. остатка из одного класса на остаток из другого или, в альтернативном случае, связанная с включением аминокислот, отличных от природных, например, орнитина (далее в данном документе называемого Z), орнитиндиаминомасляной кислоты (далее в данном документе называемой В), норлейпинорнитина (далее в данном документе называемого О), пиридилаланина, тиенилаланина, нафтилаланина и фенилглицина.
Вариантные аминокислотные последовательности могут содержать подходящие спейсерные группы, которые могут быть вставлены между двумя аминокислотными остатками последовательности, в том числе алкильные группы, например, метальная, этильная или пропильная группы, в дополнение к аминокислотным спейсерам, таким как глициновые или β-аланиновые остатки. Другая форма вариации, которая включает присутствие одного или нескольких аминокислотных остатков в пептоидной форме, может быть хорошо понятна специалистам в данной области. Для того, чтобы избежать сомнений, "пептоидная форма" используется для обозначения вариантных аминокислотных остатков, где замещающая группа α-углерода расположена на атоме азота остатка, а не на α-углероде. Способы получения пептидов в пептоидной форме известны из уровня техники, например, Simon RJ et al., PNAS (1992) 89(20), 9367-9371 и Horwell DC, Trends Biotechnol. (1995) 13(4), 132-134.
Практическое применение настоящего изобретения предусматривает, если не указано иное, традиционные методики иммунологии, биохимии, химии, молекулярной биологии, микробиологии, клеточной биологии, геномики и технологию рекомбинантной ДНК, которые находятся в пределах квалификации специалиста в данной области. См. Sambrook, Fritsch and Maniatis, MOLECULAR CLONING: A LABORATORY MANUAL, 2nd edition (1989); CURRENT PROTOCOLS IN MOLECULAR BIOLOGY (F. M. Ausubel, et al. eds., (1987)); серия METHODS IN ENZYMOLOGY (Academic Press, Inc.): PCR 2: A PRACTICAL APPROACH (M.J. MacPherson, B.D. Hames and G.R. Taylor eds. (1995)), Harlow and Lane, eds. (1988) ANTIBODIES, A LABORATORY MANUAL, и ANIMAL CELL CULTURE (R.I. Freshney, ed. (1987)).
Векторы
В одном аспекте настоящее изобретение предусматривает векторы, которые применяются при конструировании и оптимизации систем CRISPR-Cas.
Как используется в данном документе, "вектор" представляет собой инструмент, который дает возможность или обеспечивает перенос объекта из одной среды в другую. В репликон, такой как плазмида, фаг или космида, может быть встроен другой сегмент ДНК для осуществления, таким образом, репликации встроенного сегмента. Как правило, вектор способен к репликации, если ассоциирован с соответствующими регуляторными элементами. В целом, выражение "вектор" относится к молекуле нуклеиновой кислоты, способной переносить другую нуклеиновую кислоту, с которой он был связан. Векторы включают без ограничения молекулы нуклеиновых кислот, которые являются одноцепочечными, двухцепочечными или частично двухцепочечными; молекулы нуклеиновых кислот, которые содержат один или несколько свободных концов, не содержат свободных концов (к примеру, кольцевые); молекулы нуклеиновых кислот, которые содержат ДНК, РНК или и ту, и другую; и другие разновидности полинуклеотидов, известных из уровня техники. Одним типом вектора является "плазмида", которая означает кольцевую петлю двухцепочечной ДНК, в которую можно встраивать дополнительные сегменты ДНК, как, например, при помощи стандартных технологий молекулярного клонирования. Другим типом вектора является вирусный вектор, где полученные из вируса последовательности ДНК или РНК присутствуют в векторе для упаковки в вирус (к примеру, ретровирусы, ретровирусы с дефектной системой репликации, аденовирусы, аденовирусы с дефектной системой репликации и аденоассоциированные вирусы (AAV)). Вирусные векторы также включают полинуклеотиды, переносимые вирусами для трансфекции клетки-хозяина. Определенные векторы способны к саморегулируемой репликации в клетке-хозяине, в которую они введены (к примеру, бактериальные векторы с бактериальной точкой начала репликации и эписомные векторы для млекопитающих). Другие векторы (к примеру, векторы для млекопитающих, отличные от эписомных) интегрируются в геном клетки-хозяина после введения в клетку-хозяина и, таким образом, реплицируются наряду с геномом хозяина. Более того, определенные векторы способны управлять экспрессией генов, с которыми они функционально связаны. Такие векторы в данном документе называют "векторами экспрессии". Общепринятые пригодные для технологий рекомбинантной ДНК векторы экспрессии часто находятся в форме плазмид.
Рекомбинантные векторы экспрессии могут содержать нуклеиновую кислоту согласно настоящему изобретению в форме, подходящей для экспрессии нуклеиновой кислоты в клетке-хозяине, что означает, что рекомбинантные векторы экспрессии включают один или несколько регуляторных элементов, которые могут быть выбраны с учетом клеток-хозяев, которые предполагается использовать для экспрессии, которые функционально связаны с последовательностью нуклеиновой кислоты, подлежащей экспрессии. В контексте рекомбинантного вектора экспрессии выражение "функционально связанный" предназначено для обозначения того, что представляющая интерес нуклеотидная последовательность связана с регуляторным(и) элементом(ами) таким образом, при котором обеспечивается экспрессия нуклеотидной последовательности (к примеру, в in vitro системе транскрипции/трансляции или в клетке-хозяине, когда вектор вводят в клетку-хозяина). Что касается способов рекомбинации и клонирования, следует упомянуть заявку на патент США №10/815730, опубликованную 2 сентября 2004 г. как US 2004-0171156 А1, содержание которой включено в данный документ посредством ссылки во всей полноте.
Аспекты настоящего изобретения относятся к бипистронным векторам для химерной РНК и Cas9. Бицистронные векторы экспрессии для химерной RNA и Cas9 являются предпочтительными. В общем и конкретно в данном варианте осуществления Cas9 предпочтительно управляется промотором CBh. Химерная РНК предпочтительно может управляться промотором U6. Оптимальным является их сочетание. Химерная направляющая РНК, как правило, состоит из направляющей последовательности (Ns) из 20 п.о., которая может быть соединена с tracr-последовательностью (проходящей от первого "U" в нижней нити к концу транскрипта). В разных указанных положениях tracr-последовательность может быть усечена. Направляющие и tracr-последовательности разделены парной tracr-последовательностью, которая может представлять собой GUUUUAGAGCUA. За ней может следовать показанная последовательность петли GAAA. Обе из них являются предпочтительными примерами. Авторы настоящего изобретения продемонстрировали Са89-опосредованные вставки/делении в локусах ЕМХ1 и PVALB человека при помощи анализов с использованием SURVEYOR. ChiRNA показаны путем обозначения их "+n", a crRNA относится к гибридной РНК, в которой направляющие и tracr-последовательности экспрессируются в виде раздельных транскриптов. По всей данной заявке химерная РНК также может называться одиночной направляющей или синтетической направляющей РНК (sgRNA). Петля предпочтительно представляет собой GAAA, но не ограничивается этой последовательностью или действительно ее длина составляет только 4 п.о. Действительно, предпочтительные петлеобразующие последовательности для использования в "шпилечных" структурах имеют длину четыре нуклеотида и наиболее предпочтительно имеют последовательность GAAA. Однако, можно использовать более короткие или длинные последовательности петли, а также альтернативные последовательности. Последовательности предпочтительно включают нуклеотидный триплет (например, ААА) и дополнительный нуклеотид (например, С или G). Примеры петлеобразующих последовательностей включают СААА и AAAG.
Выражение "регуляторный элемент" предназначено включать промоторы, энхансеры, участки внутренней посадки рибосомы (IRES) и другие контролирующие экспрессию элементы (к примеру, сигналы терминации транскрипции, такие как сигналы полиаденилирования и поли-U-последовательности). Такие регуляторные элементы описаны, например, в Goeddel, GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY 185, Academic Press, San Diego, Calif. (1990). Регуляторные элементы включают такие, которые управляют конститутивной экспрессией нуклеотидной последовательности во многих типах клеток-хозяев, и такие, которые управляют экспрессией нуклеотидной последовательности только в определенных клетках-хозяевах (к примеру, тканеспецифичные регуляторные последовательности). Тканеспецифичный промотор может управлять экспрессией преимущественно в представляющей интерес целевой ткани, такой как мышца, нейрон, кость, кожа, кровь, конкретных органах (к примеру, печени, поджелудочной железе) или определенных типах клеток (к примеру, лимфоцитах). Регуляторные элементы также могут управлять экспрессией зависимым от времени образом, как, например, зависимым от клеточного цикла или зависимым от стадии развития образом, который может быть или может не быть также тканеспецифичным или специфичным к типу клеток. В некоторых вариантах осуществления вектор содержит один или несколько промоторов pol III (к примеру, 1, 2, 3, 4, 5 или более промоторов pol III), один или несколько промоторов pol II (к примеру, 1, 2, 3, 4, 5 или более промоторов pol II), один или несколько промоторов pol I (к примеру, 1, 2, 3, 4, 5 или более промоторов pol I) или их комбинации. Примеры промоторов pol III включают без ограничения промоторы U6 и H1. Примеры промоторов pol II включают без ограничения ретровирусный промотор LTR вируса саркомы Рауса (RSV) (необязательно с энхансером RSV), промотор цитомегаловируса (CMV) (необязательно с энхансером CMV) [см., например, Boshart et al. Cell, 41: 521-530 (1985)], промотор SV40, промотор дигидрофолатредуктазы, промотор β-актина, промотор глицерофосфаткиназы (PGK) и промотор EF1α. Также выражением "регуляторный элемент" охватываются энхансерные элементы, такие как WPRE; энхансеры CMV; сегмент R-U5' в LTR HTLV-I (Mol. Cell. Biol., Vol. 8(1), p. 466-472, 1988); энхансер SV40; и интронная последовательность между экзонами 2 и 3 β-глобина кролика (Proc. Natl. Acad. Sci. USA., Vol. 78(3), p. 1527-31, 1981). Специалистам в данной области техники будет понятно, что структура вектора экспрессии может зависеть от таких факторов, как выбор клетки-хозяина, подлежащей трансформации, желательный уровень экспрессии и т.п. Вектор можно вводить в клетки-хозяева с получением, таким образом, транскриптов, белков или пептидов, в том числе слитых белков или пептидов, кодируемых нуклеиновыми кислотами, которые описаны в данном документе (к примеру, транскриптов коротких палиндромных повторов, регулярно расположенных группами (CRISPR), белков, ферментов, их мутантных форм, их слитых белков и т.п.). Что касается регуляторных последовательностей, следует упомянуть заявку на патент США №10/491026, содержание которой включено в данный документ посредством ссылки во всей ее полноте. Что касается промоторов, следует упомянуть РСТ публикацию WO 2011/028929 и заявку на патент США №12/511940, содержание которых включено в данный документ посредством ссылки во их полноте.
Векторы можно разработать для экспрессии транскриптов CRISPR (к примеру, транскриптов нуклеиновых кислот, белков или ферментов) в прокариотических или эукариотических клетках. Например, транскрипты CRISPR могут экспрессироваться в бактериальных клетках, например, Escherichia coli, клетках насекомых (с использованием бакуловирусных векторов экспрессии), клетках дрожжей или клетках млекопитающих. Подходящие клетки-хозяева дополнительно рассматриваются в Goeddel, GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY 185, Academic Press, San Diego, Calif. (1990). В качестве альтернативы, рекомбинантный вектор экспрессии может транскрибироваться и транслироваться in vitro, например, при помощи регуляторных последовательностей промотора Т7 и полимеразы Т7.
Векторы можно вводить и размножать в прокариоте или прокариотической клетке. В некоторых вариантах осуществления прокариота используют для амплификации копий вектора, который предполагается вводить в эукариотическую клетку, или в качестве промежуточного вектора при получении вектора, который предполагается вводить в эукариотическую клетку (к примеру, путем амплификации плазмиды как части системы упаковки вирусного вектора). В некоторых вариантах осуществления прокариота используют для амплификации копий вектора и экспрессии одной или нескольких нуклеиновых кислот, как, например, для обеспечения источника одного или нескольких белков для доставки в клетку-хозяина или организм-хозяин. Экспрессию белков в прокариотах наиболее часто осуществляют в Escherichia coli векторами, содержащими конститутивные или индуцибельные промоторы, управляющие экспрессией либо слитых белков, либо белков, отличных от слитых. В слитых векторах добавляют некоторое количество аминокислот к белку, закодированному в них, как, например, к амино-концу рекомбинантного белка. Такие слитые векторы могут служить для одной или нескольких целей, как например: (i) для повышения экспрессии рекомбинантного белка; (ii) для повышения растворимости рекомбинантного белка и (iii) для содействия очистке рекомбинантного белка посредством функционирования в качестве лиганда при аффинной очистке. Часто в слитые векторы экспрессии вводят сайт протеолитического расщепления в месте соединения фрагмента слияния и рекомбинантного белка для облегчения отделения рекомбинантного белка от фрагмента слияния после очистки слитого белка. Такие ферменты и их когнатные распознающие последовательности включают фактор Ха, тромбин и энтерокиназу. Иллюстративные слитые векторы экспрессии включают pGEX (Pharmacia Biotech Inc; Smith and Johnson, 1988. Gene 67: 31-40), pMAL (New England Biolabs, Беверли, Массачусетс) и pRIT5 (Pharmacia, Пискатауэй, Нью-Джерси), в которых глутатион-S-трансфераза (GST), мальтоза-связывающий белок Е или белок А, соответственно, слиты с целевым рекомбинантным белком.
Примеры подходящих индуцибельных, не являющихся слитыми векторов экспрессии для Е. coli включают pTrc (Amrann et al., (1988) Gene 69: 301-315) и рЕТ lid (Studier et al., GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY 185, Academic Press, San Diego, Calif. (1990) 60-89).
В некоторых вариантах осуществления вектор является вектором экспрессии для дрожжей. Примеры векторов для экспрессии в дрожжах Saccharomyces cerivisae включают pYepSec1 (Baldari, et al., 1987. EMBO J. 6: 229-234), pMFa (Kuijan and Herskowitz, 1982. Cell 30: 933-943), pJRY88 (Schultz et al., 1987. Gene 54: 113-123), pYES2 (Invitrogen Corporation, Сан-Диего, Калифорния) и picZ (InVitrogen Corp, Сан-Диего, Калифорния).
В некоторых вариантах осуществления вектор управляет экспрессией белка в клетках насекомых с применением бакуловирусных векторов экспрессии. Бакуловирусные векторы, доступные для экспрессии белков в культивируемых клетках насекомых (к примеру, клетках SF9), включают группу рАс (Smith, et al., 1983. Mol. Cell. Biol. 3: 2156-2165) и группу pVL (Lucklow and Summers, 1989. Virology 170: 31-39).
В некоторых вариантах осуществления вектор способен управлять экспрессией одной или нескольких последовательностей в клетках млекопитающих при помощи вектора экспрессии для млекопитающих. Примеры векторов экспрессии для млекопитающих включают pCDM8 (Seed, 1987. Nature 329: 840) и pMT2PC (Kaufman, et al., 1987. EMBO J. 6: 187-195). При использовании клеток млекопитающих функции контроля вектора экспрессии, как правило, обеспечиваются одним или несколькими регуляторными элементами. Например, широко используемые промоторы получают из вируса полиомы, аденовируса 2, цитомегаловируса, вакуолизирующего обезьяньего вируса-40 и других, раскрытых в данном документе и известных из уровня техники. Что касается других подходящих систем экспрессии как для прокариотических, так и для эукариотических клеток, см., к примеру, главы 16 и 17 в Sambrook, et al., MOLECULAR CLONING: A LABORATORY MANUAL. 2nd ed., Cold Spring Harbor Laboratory, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y., 1989.
В некоторых вариантах осуществления рекомбинантные векторы экспрессии для млекопитающих способны управлять экспрессией нуклеиновой кислоты преимущественно в определенном типе клеток (к примеру, тканеспецифичные регуляторные элементы используют для экспрессии нуклеиновый кислот). Тканеспецифичные регуляторные элементы известны из уровня техники. Неограничивающие примеры подходящих тканеспецифичных промоторов включают промотор гена альбумина (печень-специфичный; Pinkert, et al., 1987. Genes Dev. 1: 268-277), специфичные к лимфоидной ткани промоторы (Calame and Eaton, 1988. Adv. Immunol. 43: 235-275), в частности, промоторы рецепторов Т-клеток (Winoto and Baltimore, 1989. EMBO J. 8: 729-733) и иммуноглобулины (Baneiji, et al., 1983. Cell 33: 729-740; Queen and Baltimore, 1983. Cell 33: 741-748), нейрон-специфичные промоторы (к примеру, промотор гена нейрофиламента; Byrne and Ruddle, 1989. Proc. Natl. Acad. Sci. USA 86: 5473-5477), специфичные к клеткам поджелудочной железы промоторы (Edlund, et al., 1985. Science 230: 912-916) и специфичные к клеткам молочной железы промоторы (к примеру, промотор молочной сыворотки; патент США №4873316 и публикация европейской заявки №264166). Регулируемые стадией развития промоторы также охвачены, к примеру, промоторы генов box мыши (Kessel and Gruss, 1990. Science 249: 374-379) и промотор гена α-фетопротеина (Campes and Tilghman, 1989. Genes Dev. 3: 537-546). Что касается этих прокариотических и эукариотических векторов, следует упомянуть патент США №6750059, содержание которого включено в данный документ посредством ссылки во всей его полноте. Другие варианты осуществления согласно настоящему изобретению могут относиться к вирусным векторам, которые упоминаются в заявке на патент США №13/092085, содержание которой включено в данный документ посредством ссылки во всей ее полноте. Тканеспецифичные регуляторные элементы известны из уровня техники и, в связи с этим, следует упомянуть патент США №7776321, содержание которого включено в данный документ посредством ссылки во всей его полноте.
Регуляторные элементы
В некоторых вариантах осуществления регуляторный элемент является функционально связанным с одним или несколькими элементами системы CRISPR с тем, чтобы управлять экспрессией одного или нескольких элементов системы CRISPR. В целом, CRISPR (короткие палиндромные повторы, регулярно расположенные группами), также известные как SPIDR (прерываемые спейсерами прямые повторы), составляют семейство локусов ДНК, которые, как правило, специфичны для определенного вида бактерий. Локус CRISPR включает определенный класс чередующихся коротких повторов последовательностей (SSR), которые были обнаружены у Е. coli (Ishino et al., J. Bacteriol., 169: 5429-5433 [1987]; и Nakata et al., J. Bacteriol., 171: 3553-3556 [1989]), и ассоциированные гены. Подобные чередующиеся SSR были идентифицированы у Haloferax mediterranei. Streptococcus pyogenes, Anabaena и Mycobacterium tuberculosis (см. Groenen et al., Mol. Microbiol., 10: 1057-1065 [1993]; Hoe et al., Emerg. Infect. Dis., 5: 254-263 [1999]; Masepohl et al., Biochim. Biophys. Acta 1307: 26-30 [1996]; и Mojica et al., Mol. Microbiol., 17: 85-93 [1995]). Локусы CRISPR, как правило, отличаются от других SSR по структуре повторов, которые были названы короткими повторами с регулярными интервалами (SRSR) (Janssen et al., OMICS J. Integ. Biol., 6: 23-33 [2002]; и Mojica et al., Mol. Microbiol., 36: 244-246 [2000]). В целом, повторы являются короткими элементами, которые встречаются группами, которые регулярно разделены уникальными вставочными последовательностями с практически постоянной длинной (Mojica et al., [2000], выше). Несмотря на то, что последовательности повторов высоко консервативны между штаммами, некоторое количество чередующихся повторов и последовательностей спейсерных участков, как правило, отличаются от штамма к штамму (van Embden et al., J. Bacteriol., 182: 2393-2401 [2000]). Локусы CRISPR были идентифицированы у более чем 40 видов прокариот (см., к примеру, Jansen et al., Mol. Microbiol., 43: 1565-1575 [2002]; и Mojica et al., [2005]), в том числе без ограничения у Aeropymm, Pyrobaculum, Sulfolobus, Archaeoglobus, Halocarcula, Methanobacterium, Methanococcus, Methanosarcina, Methanopyrus, Pyrococcus, Picrophilus, Thermoplasma, Corynebacterium, Mycobacterium, Streptomyces, Aquifex, Porphyromonas, Chlorobium, Thermus, Bacillus, Listeria, Staphylococcus, Clostridium, Thermoanaerobacter, Mycoplasma, Fusobacterium, Azarcus, Chromobacterium, Neisseria, Nitrosomonas, Desulfovibrio, Geobacter, Myxococcus, Campylobacter, Wolinella, Acinetobacter, Erwinia, Escherichia, Legionella, Methylococcus, Pasteurella, Photobacterium, Salmonella, Xanthomonas, Yersinia, Treponema и Thennotoga.
В целом, "система CRISPR" означает в совокупности транскрипты и другие элементы, участвующие в экспрессии CRISPR-ассоциированных ("Cas") генов или управлении их активностью, в том числе последовательности, кодирующие ген Cas, tracr (транс-активируемую CRISPR) последовательность (к примеру, tracrRNA или активную частичную tracrRNA), парную tracr-последовательность (охватывающую "прямой повтор" и tracrRNA-процессированный неполный прямой повтор в контексте эндогенной системы CRISPR), направляющую последовательность (также называемую "спейсером" в контексте эндогенной системы CRISPR) или другие последовательности и транскрипты с локуса CRISPR. В вариантах осуществления согласно настоящему изобретению выражения направляющая последовательность и направляющая РНК используются взаимозаменяемо. В некоторых вариантах осуществления один или несколько элементов системы CRISPR получены из системы CRISPR типа I, типа II или типа III. В некоторых вариантах осуществления один или несколько элементов системы CRISPR получены из определенного организма, содержащего эндогенную систему CRISPR, как, например, Streptococcus pyogenes. В целом, система CRISPR характеризуется элементами, которые способствуют образованию комплекса CRISPR в сайте целевой последовательности (также называемой протоспейсером в контексте эндогенной системы CRISPR). В контексте образования комплекса CRISPR "целевая последовательность" означает последовательность, по отношению к которой направляющая последовательность разработана так, чтобы обладать комплементарностью, где гибридизация между целевой последовательностью и направляющей последовательностью способствует образованию комплекса CRISPR. Целевая последовательность может содержать любой полинуклеотид, как, например, ДНК- или РНК-полинуклеотиды. В некоторых вариантах осуществления целевая последовательность расположена в ядре или цитоплазме клетки.
В некоторых вариантах осуществления, прямые повторы можно идентифицировать in silico посредством поиска повторяющихся мотивов, которые соответствуют любым или всем из следующих критериев:
1. обнаруживаются в окне размером 2 т.п.о. геномной последовательности, фланкирующей CRISPR типа II;
2. имеют протяженность от 20 до 50 п.о. и
3. расположены с интервалами от 20 до 50 п.о. В некоторых вариантах осуществления можно использовать 2 из этих критериев, например, 1 и 2, 2 и 3 или 1 и 3. В некоторых вариантах осуществления можно использовать все 3 критерия.
В некоторых вариантах осуществления кандидатные tracrRNA можно впоследствии предсказать с использованием последовательностей, которые соответствуют любым или всем из следующих критериев:
1. гомология последовательности с прямыми повторами (поиск мотивов в Geneious с несовпадениями вплоть до 18 п.о.);
2. наличие предсказанного Rho-независимого терминатора транскрипции по направлению транскрипции; и
3. стабильная шпилечная вторичная структура между tracrRNA и прямым повтором.
В некоторых вариантах осуществления можно использовать 2 из этих критериев, например, 1 и 2, 2 и 3 или 1 и 3. В некоторых вариантах осуществления можно использовать все 3 критерия.
В некоторых вариантах осуществления химерные структуры синтетических направляющих РНК (sgRNA) могут включать по меньшей мере 12 п.о. дуплексной структуры между прямым повтором и tracrRNA.
В предпочтительных вариантах осуществления согласно настоящему изобретению система CRISPR представляет собой систему CRISPR типа II и фермент Cas представляет собой Cas9, который катализирует расщепление ДНК. Ферментативная активность Cas9, полученной из Streptococcus pyogenes, или любой близкородственной Cas9, приводит к образованию двухцепочечных разрывов в последовательностях целевого сайта, который гибридизуется с 20 нуклеотидами направляющей последовательности и который содержит последовательность смежного с протоспейсером мотива (РАМ) (примеры включают NGG/NRG или РАМ, который можно определить, как описано в данном документе) после 20 нуклеотидов целевой последовательности. Активность CRISPR посредством Cas9 по отношению к сайт-специфическому распознаванию и расщеплению ДНК определяется направляющей последовательностью, tracr-последовательностью, которая частично гибридизуется с направляющей последовательностью и последовательностью РАМ. Больше аспектов системы CRISPR описаны в Karginov and Hannon, The CRISPR system: small RNA-guided defence in bacteria and archaea. Mole Cell 2010, January 15; 37(1): 7.
Локус CRISPR типа II происходит из Streptococcus pyogenes SF370, который содержит кластер из 4 генов Cas9, Cas1, Cas2 и Csn1, а также 2 некодирующих элементов РНК, tracrRNA и характерный массив повторяющихся последовательностей (прямых повторов), чередующихся с короткими фрагментами неповторяющихся последовательностей (спейсерами, приблизительно 30 п.о. каждый). В данной системе целенаправленный двухнитевой разрыв (DSB) ДНК образуется в ходе четырех последовательных стадий (фигура 2А). Во-первых, две некодирующие РНК, массив pre-crRNA и tracrRNA транскрибируются с локуса CRISPR. Во-вторых, tracrRNA гибридизуется с прямыми повторами в pre-crRNA, которая затем процессируется в зрелые crRNA, содержащие индивидуальные спейсерные последовательности. В-третьих, комплекс зрелая crRNA : tracrRNA направляет Cas9 к ДНК-мишени, состоящей из протоспейсера и соответствующего РАМ, посредством образования гетеродуплекса между спейсерным участком crRNA и протоспейсерной ДНК. И наконец, Cas9 опосредует расщепление целевой ДНК выше РАМ с образованием DSB внутри протоспейсера (фигура 2А). На фигуре 2В представлена ядерная локализация кодон-оптимизированной Cas9. Для того, чтобы способствовать точной инициации транскрипции, промотор U6 на основе РНК-полимеразы III выбирали для управления экспрессией tracrRNA (фигура 2С). Подобным образом, конструкцию на основе промотора U6 разработали для экспрессии массива pre-crRNA, состоящего из одного спейсера, фланкированного двумя прямыми повторами (DR, также охватываются выражением "парные tracr-последовательности"; фигура 2С). Исходный спейсер был сконструирован для целенаправленного воздействия на целевой сайт размером 33 пары оснований (п.о.) (протоспейсер из 30 п.о., а также последовательность мотива CRISPR (РАМ) из 3 п.о., соответствующая мотиву узнавания NGG у Cas9) в локусе ЕМХ1 человека (фигура 2С), ключевом гене в развитии коры головного мозга.
Как правило, в контексте эндогенной системы CRISPR образование комплекса CRISPR (содержащего направляющую последовательность, гибридизующуюся с целевой последовательностью и образующую комплекс с одним или несколькими белками Cas) приводит к расщеплению одной или обеих нитей в или около (к примеру, в пределах 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 50 или более пар оснований от) целевой последовательности. Не вдаваясь в теорию полагают, что tracr-последовательность, которая может содержать или состоять из всей или части tracr-последовательности дикого типа (к примеру, приблизительно или более чем приблизительно 20, 26, 32, 45, 48, 54, 63, 67, 85 или более нуклеотидов tracr-последовательности дикого типа), может также образовывать часть комплекса CRISPR, как, например, путем гибридизации вдоль по меньшей мере части tracr-последовательности со всей или частью парной tracr-последовательности, которая функционально связана с направляющей последовательностью. В некоторых вариантах осуществления один или несколько векторов, управляющих экспрессией одного пли нескольких элементов системы CRISPR, вводят в клетку-хозяина, так что экспрессия элементов системы CRISPR управляет образованием комплекса CRISPR на одном или нескольких целевых сайтах. Например, каждое из фермента Cas, направляющей последовательности, связанной с парной tracr-последовательностью, и tracr-последовательности может быть функционально связано с отдельными регуляторными элементами в отдельных векторах. В качестве альтернативы, два или более элементов, экспрессируемых с одних и тех же или различных регуляторных элементов, можно объединять в одном векторе, с одним или несколькими дополнительными векторами, обеспечивающими любые компоненты системы CRISPR, не включенные в первый вектор. Элементы системы CRISPR, которые объединены в один вектор, могут быть расположены в любой удобной ориентации, как, например, один элемент, расположенный 5' ("выше") относительно или 3' ("ниже") относительно второго элемента. Кодирующая последовательность одного элемента может быть расположена па той же или противоположной нити по отношению к кодирующей последовательности второго элемента и направлена в том же или противоположном направлении. В некоторых вариантах осуществления один промотор управляет экспрессией транскрипта, кодирующего фермент CRISPR, и одной или нескольких из направляющей последовательности, парной tracr-последовательности (необязательно функционально связанной с направляющей последовательностью) и tracr-последовательности, встроенных в одну или несколько интронных последовательностей (к примеру, каждая в разном интроне, две или более по меньшей мере в одном интроне или все в одном интроне). В некоторых вариантах осуществления фермент CRISPR, направляющая последовательность, парная tracr-последовательность и tracr-последовательность функционально связаны с одним и тем же промотором и экспрессируются с него.
В некоторых вариантах осуществления вектор содержит один или несколько сайтов встраивания, как, например, последовательность узнавания рестрикционной эндонуклеазой (также называемая "сайтом клонирования"). В некоторых вариантах осуществления один или несколько сайтов встраивания (к примеру, приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более сайтов встраивания) расположены выше и/или ниже одного или нескольких элементов последовательности одного или нескольких векторов. В некоторых вариантах осуществления вектор содержит сайт встраивания выше парной tracr-последовательности и необязательно ниже регуляторного элемента, функционально связанного с парной tracr-последовательностью, так что после встраивания направляющей последовательности в сайт встраивания и при экспрессии направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью в эукариотической клетке. В некоторых вариантах осуществления вектор содержит два или более сайта встраивания, при этом каждый сайт встраивания расположен между двумя парными tracr-последовательностями с тем, чтобы обеспечить возможность встраивания направляющей последовательности в каждый сайт. При таком расположении две или более направляющих последовательностей могут содержать две или более копий одной направляющей последовательности, две или более различных направляющих последовательностей или их комбинации. В тех случаях, когда применяют несколько различных направляющих последовательностей, можно использовать одну экспрессирующую конструкцию для целенаправленного воздействия активности CRISPR на несколько различных соответствующих целевых последовательностей в клетке. Например, один вектор может содержать приблизительно или более приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20 или более направляющих последовательностей. В некоторых вариантах осуществления приблизительно или более приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более таких содержащих направляющие последовательности векторов могут быть предусмотрены и необязательно доставлены в клетку.
В некоторых вариантах осуществления вектор содержит регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей фермент CRISPR, как, например, белок Cas. Неограничивающие примеры белков Cas включают Cas1, Cas1B, Cas2, Cas3, Cas4, Cas5, Cas6, Cas7, Cas8, Cas9 (также известный как Csn1 и Csx12), Cas10, Csy1, Csy2, Csy3, Cse1, Cse2, Csc1, Csc2, Csa5, Csn2, Csm2, Csm3, Csm4, Csm5, Csm6, Cmr1, Cmr3, Cmr4, Cmr5, Cmr6, Csb1, Csb2, Csb3, Csx17, Csx14, Csx10, Csx16, CsaX, Csx3, Csx1, Csx15, Csf1, Csf2, Csf3, Csf4, их гомологи или их модифицированные варианты. В некоторых вариантах осуществления немодифицированный фермент CRISPR характеризуется активностью в отношении расщепления ДНК, как, например, Cas9. В некоторых вариантах осуществления фермент CRISPR управляет расщеплением одной или обеих нитей в определенной точке целевой последовательности, как, например, в пределах целевой последовательности и/или в пределах комплементарной последовательности целевой последовательности. В некоторых вариантах осуществления фермент CRISPR управляет расщеплением одной или обеих нитей в пределах приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 50, 100, 200, 500 или более пар оснований от первого или последнего нуклеотида целевой последовательности. В некоторых вариантах осуществления вектор кодирует фермент CRISPR, который является мутированным по отношению к соответствующему ферменту дикого типа, так что у мутированного фермента CRISPR отсутствует способность расщеплять одну или обе нити целевого полинуклеотида, содержащего целевую последовательность. Например, замена аспартата - на - аланин (D10A) в каталитическом домене RuvC I Cas9 из S. pyogenes трансформирует Cas9 из нуклеазы, которая расщепляет обе нити, в никазу (расщепляет одну нить). Другие примеры мутаций, которые превращают Cas9 в никазу, включают без ограничения Н840А, N854A и N863A. В качестве дополнительного примера два или более каталитических доменов Cas9 (RuvC I, RuvC II и RuvC III или домен HNH) можно подвергать мутациям с получением мутированного Cas9, у которого практически отсутствует вся активность для расщепления ДНК. В некоторых вариантах осуществления мутацию D10A объединяют с одной или несколькими из мутаций Н840А, N854A или N863A с получением фермента Cas9, у которого практически отсутствует вся активность для расщепления ДНК. В некоторых вариантах осуществления фермент CRISPR рассматривают как такой, у которого практически отсутствует вся активность для расщепления ДНК, в случаях, когда активность для расщепления ДНК мутированного фермента составляет менее приблизительно 25%, 10%, 5%, 1%, 0,1%, 0,01% или меньше по отношению к его форме, отличающейся от мутированной. Если фермент не является SpCas9, мутации можно осуществлять по любому или всем остаткам, соответствующим положениям 10, 762, 840, 854, 863 и/или 986 SpCas9 (которые могут быть определены, например, при помощи стандартных инструментов сравнения последовательностей). В частности, любые или все из следующих мутаций являются предпочтительными для SpCas9: D10A, Е762А, Н840А, N854A, N863A и/или D986A; а также предусматривается консервативная замена для любого аминокислотного замещения. То же самое (или консервативные замены по этим мутациям) также является предпочтительным в соответствующих положениях других Cas9. Особенно предпочтительными являются D10 и Н840 в SpCas9. Однако, в других Cas9 остатки, соответствующие SpCas9 D10 и Н840, также являются предпочтительными.
Замену аспартат-на-аланин (D10A) в каталитическом домене RuvC I в SpCas9 производили с применением методик генной инженерии для превращения нуклеазы в никазу (SpCas9n) (см., например, Sapranauskas et al., 2011, Nucleic Acids Research, 39: 9275; Gasiunas et al., 2012, Proc. Natl. Acad. Sci. USA, 109:E2579) так, чтобы надрезанная геномная ДНК подвергалась высокоточной репарации с использованием гомологичной рекомбинации (HDR). Анализ с помощью Surveyor подтвердил, что SpCas9n не создает вставок/делений в мишени-протоспейсере ЕМХ1. Коэкспрессия целенаправленно воздействующей на ЕМХ1 химерной crRNA (также содержащей компонент tracrRNA) с SpCas9 приводила к вставкам/делениям в целевом сайте, тогда как коэкспрессия с SpCas9n не приводила (n=3). Более того, секвенирование 327 ампликонов не привело к обнаружению каких-либо вставок/делеций, индуцированных SpCas9n. Для исследования опосредованной CRISPR HR при совместной трансфекции клеток HEK 293FT химерной РНК, целенаправленно воздействующей на ЕМХ1, hSpCas9 или hSpCas9n, выбирали тот же локус, также как и матрицу для HR для введения пары сайтов рестрикции (HindIII и NheI) возле протоспейсера.
Предпочтительные ортологи описаны в данном документе. Фермент Cas может быть идентифицирован как Cas9, поскольку он может относиться к общему классу ферментов, обладающих гомологией с самой большой нуклеазой с несколькими доменами нуклеаз системы CRISPR типа II. В наиболее предпочтительном случае фермент Cas9 получен или происходит из SpCas9 или saCas9. Под происходящим авторы настоящего изобретения подразумевают, что в основе происходящего фермента, главным образом, лежит фермент дикого типа в том смысле, что он характеризуется высокой степенью гомологии последовательности с этим ферментом, по он был некоторым образом подвергнут мутации (модифицирован), как описано в данном документе.
Следует иметь в виду, что выражения Cas и фермент CRISPR обычно используются в данном документе взаимозаменяемо, если не очевидно иное. Как упоминается выше, многие из порядков нумерации остатков, используемых в данном документе, относятся к ферменту Cas9 из локуса CRISPR типа II Streptococcus pyogenes. Однако, следует иметь в виду, что настоящее изобретение включает многие другие Cas9 из других видов микроорганизмов, такие как SpCas9, SaCa9, St1Cas9 и т.д.
Оптимизация кодонов
Пример кодон-оптимизированной последовательности, в данном случае оптимизированной для человека (т.е. оптимизированной для экспрессии у человека), представлен в данном документе, см. кодон-оптимизированную последовательность SaCas9 для человека. Хотя это является предпочтительным, следует иметь в виду, что возможны другие примеры и что для вида-хозяина известна оптимизация кодонов.
В некоторых вариантах осуществления кодирующая фермент последовательность, кодирующая фермент CRISPR, является кодон-оптимизированной для экспрессии в определенных клетках, как, например, эукариотических клетках. Эукариотические клетки могут быть клетками определенного организма или полученными из него, как, например, млекопитающего, в том числе, без ограничения, человека, мыши, крысы, кролика, собаки или отличного от человека млекопитающего или примата. В некоторых вариантах осуществления могут не допускаться способы модификации генетической идентичности зародышевой линии человека и/или способы модификации генетической идентичности животных, которые, вероятно, могут причинить им страдания без какой-либо значительной медицинской пользы для человека или животных, а также животные, являющиеся результатом таких способов.
В целом, оптимизация кодонов означает способ модификации последовательности нуклеиновой кислоты для повышения экспрессии в представляющих интерес клетках-хозяевах путем замещения по меньшей мере одного кодона (к примеру, приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 10, 15, 20, 25, 50 или более кодонов) нативной последовательности кодонами, которые чаще или наиболее часто используются в генах этой клетки-хозяина, в то же время с сохранением нативной аминокислотной последовательности. Разные виды проявляют определенное "предпочтение" в отношении конкретных кодонов определенной аминокислоты. "Предпочтение" кодонов (различия в частоте использования кодонов между организмами) часто соотносят с эффективностью трансляции матричной РНК (мРНК), которая, в свою очередь, как полагают, зависит, среди прочего, от свойств кодонов, которые транслируются, и доступности конкретных молекул транспортной РНК (тРНК). Преобладание выбранных тРНК в клетке, как правило, является отражением кодонов, используемых наиболее часто при синтезе пептидов. Соответственно, гены могут быть приспособлены для оптимальной экспрессии генов в данном организме с использованием оптимизации кодонов. Таблицы частоты использования кодонов общедоступны, например, в "Базе данных частот использования кодонов" ("Codon Usage Database"), доступной в Интернете по адресу www.kazusa.orjp/codon/ (просмотрена 9 июля 2002 г.), и эти таблицы можно адаптировать различными способами. См. Nakamura, Y., et al. "Codon usage tabulated from the international DNA sequence databases: status for the year 2000" Nucl. Acids Res. 28: 292 (2000). Также доступны компьютерные алгоритмы для оптимизации кодопов определенной последовательности для экспрессии в определенной клетке-хозяине, как, например, также доступный Gene Forge (Aptagen; Джакобус, Пенсильвания). В некоторых вариантах осуществления один или несколько кодонов (к примеру, 1, 2, 3, 4, 5,10, 15, 20, 25, 50 или более или все кодоны) в последовательности, кодирующей фермент CRISPR, соответствуют наиболее часто используемому кодону для определенной аминокислоты.
Последовательности ядерной локализации (NLS)
В некоторых вариантах осуществления вектор кодирует фермент CRISPR, содержащий одну или несколько последовательностей ядерной локализации (NLS), как, например, приблизительно или более приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более NLS. В некоторых вариантах осуществления фермент CRISPR содержит приблизительно или более приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более NLS на амино-конце или рядом с ним, приблизительно или более приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более NLS на карбокси-конце или рядом с ним или комбинацию этого (к примеру, одну или несколько NLS на амино-конце и одну или несколько NLS на карбокси-конце). В тех случаях, когда присутствуют несколько NLS, каждая может быть выбрана независимо от других, так что одна NLS может присутствовать в нескольких копиях и/или в комбинации с одной или несколькими другими NLS, присутствующими в одной или нескольких копиях. В предпочтительном варианте осуществления настоящего изобретения фермент CRISPR содержит не более 6 NLS. В некоторых вариантах осуществления считают, что NLS находится рядом с N- или С-концом в тех случаях, когда самая близкая аминокислота NLS находится в пределах приблизительно 1, 2, 3, 4, 5, 10, 15, 20, 25, 30, 40, 50 или более аминокислот вдоль полипетидной цепи от N- или С-конца. Неограничивающие примеры NLS включают NLS-последовательности, полученные из следующих: NLS из большого Т-антигена вируса SV40 с аминокислотной последовательностью PKKKRKV; NLS из нуклеоплазмина (к примеру, двойной NLS из нуклеоплазмина с последовательностью KRPAATKKAGQAKKKK); NLS из c-myc с аминокислотной последовательностью PAAKRVKLD или RQRRNELKRSP; NLS из hRNPA1 M9 с последовательностью NQSSNFGPMKGGNFGGRSSGPYGGGGQYFAKPRNQGGY; последовательности RMRIZFKNKGKDTAELRRRRVEVSVELRKAKKDEQILKRRNV домена IBB из импортина-альфа; последовательности VSRKRPRP и PPKKARED из Т-белка миомы; последовательность POPKKKPL из р53 человека; последовательность SALIKKKKKMAP из с-abl IV мыши; последовательности DRLRR и PKQKKRK из NS1 вируса гриппа; последовательность RKLKKKIKKL из дельта-антигена вируса гепатита; последовательность REKKKFLKRR из белка Мх1 мыши; последовательность KRKGDEVDGVDEVAKKKSKK из поли(АДФ-рибоза)-полимеразы человека и последовательность RKCLQAGMNLEARKTKK из рецепторов стероидных гормонов для глюкокортикоидов (человека).
В целом, одна или несколько NLS являются достаточно эффективными, чтобы управлять накоплением фермента CRISPR в обнаруживаемом количестве в ядре эукариотической клетки. В целом, степень проявления активности ядерной локализации может быть результатом следующего: количества NLS в ферменте CRISPR, конкретных(ой) используемых(ой) NLS или комбинации этих факторов. Обнаружение накопления в ядре можно выполнять при помощи любой подходящей методики. Например, детектируемый маркер может быть слит с ферментом CRISPR, так что расположение в клетке может быть визуализировано, как, например, в комбинации со средствами для обнаружения расположения в ядре (к примеру, красителем, специфичным к ядру, таким как DAPI). Ядра клеток также можно выделять из клеток, причем их содержимое можно затем анализировать при помощи любого подходящего способа для обнаружения белка, как, например, иммуногистохимии, вестерн-блоттинга или анализа активности фермента. Накопление в ядре также можно определить опосредованно, как, например, при помощи анализа эффекта образования комплекса CRISPR (к примеру, анализа на расщепление ДНК или мутацию в целевой последовательности или анализа активности экспрессии генов, измененной посредством образования комплекса CRISPR и/или активности фермента CRISPR) по сравнению с контролем, который не подвергали воздействию фермента или комплекса CRISPR или подвергали воздействию фермента CRISPR, у которого отсутствуют одна или несколько NLS.
Направляющая последовательность
В целом, направляющая последовательность представляет собой любую полинуклеотидную последовательность, обладающую достаточной комплементарностью с целевой полинуклеотидной последовательностью для гибридизации с целевой последовательностью и управления специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью. В некоторых вариантах осуществления степень комплементарности между направляющей последовательностью и ее соответствующей целевой последовательностью при оптимальном выравнивании с использованием подходящего алгоритма выравнивания составляет приблизительно или более приблизительно 50%, 60%, 75%, 80%, 85%, 90%, 95%, 97,5%, 99% или более. Оптимальное выравнивание можно определять при помощи любого подходящего алгоритма для выравниваемых последовательностей, неограничивающие примеры которого включают алгоритм Смита-Ватермана, алгоритм Нидлмана-Вунша, алгоритмы, основанные на преобразовании Барроуза-Уилера (к примеру. Burrows Wheeler Aligner), ClustalW, Clustal X, BLAT, Novoalign (Novocraft Technologies; доступный на www.novocraft.com), ELAND (Illumina, Сан-Диего, Калифорния), SOAP (доступный на soap.genomics.org.cn) и Maq (доступный на maq.sourceforge.net). В некоторых вариантах осуществления направляющая последовательность составляет приблизительно или более приблизительно 5, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 75 или более нуклеотидов в длину. В некоторых вариантах осуществления направляющая последовательность составляет менее приблизительно 75, 50, 45, 40, 35, 30, 25, 20, 15, 12 или менее нуклеотидов в длину. Способность направляющей последовательности управлять специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью можно оценить при помощи любого подходящего анализа. Например, компоненты системы CRISPR, достаточные для образования комплекса CRISPR, в том числе направляющая последовательность, которую необходимо исследовать, могут быть доставлены в клетку-хозяина с соответствующей целевой последовательностью, как, например, при помощи трансфекции векторами, кодирующими компоненты последовательности CRISPR, с последующей оценкой предпочтительного расщепления в пределах целевой последовательности, как, например, при помощи анализа с помощью Surveyor, который описан в данном документе. Подобным образом, расщепление целевой полинуклеотидной последовательности может быть установлено в пробирке путем обеспечения целевой последовательности, компонентов комплекса CRISPR, в том числе направляющей последовательности, которую необходимо исследовать, и контрольной направляющей последовательности, отличной от тестовой направляющей последовательности, и сравнения воздействий тестовой и контрольной направляющей последовательности на связывание или скорость расщепления целевой последовательности. Другие анализы возможны и будут очевидны специалисту в данной области.
Направляющая последовательность может быть выбрана для целенаправленного воздействия на любую целевую последовательность. В некоторых вариантах осуществления целевая последовательность является последовательностью в пределах генома клетки. Иллюстративные целевые последовательности включают те, которые являются уникальными в целевом геноме. Например, для Cas9 S. pyogenes уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 в виде MMMMMMMMNNNNNNNNNNNNXGG, где NNNNNNNNNNNNXGG (N представляет собой A, G, Т или С; и Х может быть любым) характеризуется единичным появлением в геноме. Уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 S. pyogenes в виде MMMMMMMMMNNNNNNNNNNNXGG, где NNNNNNNNNNNXGG (N представляет собой A, G, Т или С; и X может быть любым) характеризуется единичным появлением в геноме. Для Cas9 CRISPR1 S. fhermophilus уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 в виде MMMMMMMMNNNNNNNNNNNNXXAGAAW, где NNNNNNNNNNNNXXAGAAW (N представляет собой А, G, Т или С; Х может быть любым; и W представляет собой А или Т) характеризуется единичным появлением в геноме. Уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 CRISPR1 S. thermophilus в виде MMMMMMMMMNNNNNNNNNNNXXAGAAW, где NNNNNNNNNNNXXAGAAW (N представляет собой А, G, Т или С; Х может быть любым; и W представляет собой А или Т) характеризуется «единичным появлением в геноме. Для Cas9 S.pyogenes уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 в виде MMMMMMMMNNNNNNNNNNNNXGGXG, где NNNNNNNNNNNNXGGXG (N представляет собой А, G, Т или С; и Х может быть любым) характеризуется единичным появлением в геноме. Уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 S.pyogenes в виде MMMMMMMMMNNNNNNNNNNNXGGXG, где NNNNNNNNNNNXGGXG (N представляет собой А, G, Т или С; и Х может быть любым) характеризуется единичным появлением в геноме. В каждой из этих последовательностей "М" может представлять собой А, G, Т или С и не должен учитываться при идентификации последовательности как уникальной.
В некоторых вариантах осуществления направляющая последовательность выбрана для снижения доли вторичной структуры в направляющей последовательности. В некоторых вариантах осуществления приблизительно или менее приблизительно 75%, 50%, 40%, 30%, 25%, 20%, 15%, 10%, 5%, 1% или меньше нуклеотидов направляющей последовательности участвуют в самокомплементарном спаривании оснований при оптимальном сворачивании. Оптимальное сворачивание можно определить при помощи любого подходящего алгоритма сворачивания полинуклеотида. Некоторые программы основаны на вычислении минимальной свободной энергии Гиббса. Примером одного такого алгоритма является mFold, который описан Zuker and Stiegler (Nucleic Acids Res. 9 (1981), 133-148). Другим примером алгоритма сворачивания является доступный в режиме онлайн веб-сервер RNAfold, разработанный в Институте теоретической химии при Венском университете, в котором используется алгоритм прогнозирования структуры на основе центроидного метода (см., к примеру, A.R. Gruber et al., 2008, Cell 106(1): 23-24; и PA Carr and GM Church, 2009, Nature Biotechnology 27(12); 1151-62).
Парная tracr-последовательность
В общем, парная tracr-последовательность включает любую последовательность, которая характеризуется достаточной комплементарностью с tracr-последовательностью для содействия одному или нескольким из следующих: (1) вырезание направляющей последовательности, фланкированной парными tracr-последовательностями, в клетке, содержащей соответствующую tracr-последовательность; и (2) образование комплекса CRISPR на целевой последовательности, где комплекс CRISPR содержит парную tracr-последовательность, гибридизующуюся с tracr-последовательностью. В общем, степень комплементарности указана на основании оптимального выравнивания парной tracr-последовательности и tracr-последовательности по длине более короткой из двух последовательностей. Оптимальное выравнивание можно определить при помощи любого подходящего алгоритма выравнивания и можно дополнительно рассчитать для вторичных структур, как, например, самокомплементарность в пределах либо tracr-последовательности, либо парной tracr-последовательности. В некоторых вариантах осуществления степень комплементарности между tracr-последовательностью и парной tracr-последовательностью по длине более короткой из двух при оптимальном выравнивании составляет приблизительно или более приблизительно 25%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 97,5%, 99% или более. В некоторых вариантах осуществления tracr-последовательность составляет приблизительно или более приблизительно 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 40, 50 или более нуклеотидов в длину. В некоторых вариантах осуществления tracr-последовательность и парная tracr-последовательность содержатся в одном транскрипте, так что гибридизация между ними двумя дает транскрипт со вторичной структурой, такой как "шпилька". В одном варианте осуществления настоящего изобретения транскрипт или транскрибированная полинуклеотидная последовательность характеризуются по меньшей мере двумя или более "шпильками". В предпочтительных вариантах осуществления транскрипт характеризуется двумя, тремя, четырьмя или пятью "шпильками". В дополнительном варианте осуществления настоящего изобретения транскрипт характеризуется самое большее пятью "шпильками". В "шпилечной" структуре часть последовательности в 5' направлении по отношению к концевому "N" и выше петли соответствует парной tracr-последовательности, а часть последовательности в 3' направлении по отношению к петле соответствует tracr-последовательности. Дополнительными неограничивающими примерами отдельных полинуклеотидов, содержащих направляющую последовательность, парную tracr-последовательность и tracr-последовательность, являются следующие (перечисленные от 5' к 3'), где "N" представляет собой основание направляющей последовательности, первый блок букв нижнего регистра представляет собой парную tracr-последовательность, а второй блок букв нижнего регистра представляет собой tracr-последовательность, и конечная поли-Т-последовательность представляет собой терминатор транскрипции:

Матрица для рекомбинации
В некоторых вариантах осуществления также предусмотрена матрица для рекомбинации. Матрица для рекомбинации может быть компонентом другого вектора, который описан в данном документе, может содержаться в отдельном векторе или предусматриваться в виде отдельного полинуклеотида. В некоторых вариантах осуществления матрица для рекомбинации разработана так, чтобы служить в качестве матрицы при гомологичной рекомбинации, как, например, в пределах или рядом с целевой последовательностью, надрезанной или расщепленной ферментом CRISPR в качестве части комплекса CRISPR. Матричный полинуклеотид может быть любой подходящей длины, как, например, приблизительно или более приблизительно 10, 15, 20, 25, 50, 75, 100, 150, 200, 500, 1000 или более нуклеотидов в длину. В некоторых вариантах осуществления матричный полинуклеотид комплементарен части полинуклеотида, содержащего целевую последовательность. При оптимальном выравнивании матричный полинуклеотид может перекрываться с одним или несколькими нуклеотидами целевых последовательностей (к примеру, с приблизительно или более чем приблизительно 1, 5, 10, 15, 20 или более нуклеотидами). В некоторых вариантах осуществления при оптимальном выравнивании матричной последовательности и полинуклеотида, содержащего целевую последовательность, наиболее близкий нуклеотид матричного полинуклеотида находится в пределах приблизительно 1, 5, 10, 15, 20, 25, 50, 75, 100, 200, 300, 400, 500, 1000, 5000, 10000 или более нуклеотидов от целевой последовательности.
Белок слияния
В некоторых вариантах осуществления фермент CRISPR является частью белка слияния, содержащего один или несколько доменов гетерологичного белка (к примеру, приблизительно или более приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более доменов в дополнение к ферменту CRISPR). Белок слияния, содержащий фермент CRISPR, может содержать любую дополнительную последовательность белка и необязательно линкерную последовательность между любыми двумя доменами. Примеры белковых доменов, которые могут быть слиты с ферментом CRISPR, включают, без ограничения, эпитопные метки, последовательности из генов-репортеров и белковые домены с одной или несколькими из следующих активностей: метилазная активность, деметилазная активность, активность для активации транскрипции, активность для репрессии транскрипции, активность фактора освобождения при транскрипции, активность для модификации гистонов, активность для расщепления РНК и активность для связывания нуклеиновой кислоты. Неограничивающие примеры эпитопных меток включают гистидиновые (His) метки, V5-метки, FLAG-метки, метки гемагглютинина вируса гриппа (НА), Мус-метки, VSV-G-метки и тиоредоксиновые (Trx) метки. Примеры генов-репортеров включают без ограничения глутатион-S-трансферазу (GST), пероксидазу хрена (HRP), хлорамфеникол-ацетилтрансферазу (CAT), бета-галактозидазу, бета-глюкуронидазу, люциферазу, зеленый флуоресцентный белок (GFP), HcRed, DsRed, голубой флуоресцентный белок (CFP), желтый флуоресцентный белок (YFP) и автофлуоресцирующие белки, в том числе синий флуоресцентный белок (BFP). Фермент CRISPR может быть слит с последовательностью гена, кодирующей белок или фрагмент белка, которые связываются с молекулой ДНК или связываются с другими клеточными молекулами, в том числе без ограничения связывающий мальтозу белок (МВР), S-метка, продукты слияния Lex А и ДНК-связывающего домена (DBD), продукты слияния GAL4 и ДНК-связывающего домена и продукты слияния белка ВР16 вируса простого герпеса (HSV). Дополнительные домены, которые могут образовывать часть белка слияния, содержащего фермент CRISPR, описаны в US 20110059502, включенном в данный документ при помощи ссылки. В некоторых вариантах осуществления меченый фермент CRISPR используют для идентификации расположения целевой последовательности.
Индуцируемая система
В некоторых вариантах осуществления фермент CRISPR может образовывать компонент индуцируемой системы. Индуцируемая природа системы будет обеспечивать возможность пространственно-временного контроля редактирования генов или экспрессии генов с использованием определенной формы энергии. Форма энергии может включать, но без ограничения, электромагнитное излучение, звуковую энергию, химическую энергию и тепловую энергию. Примеры индуцируемой системы включают индуцируемые тетрациклином промоторы (Tet-On или Tet-Off), двухгибридные системы активации транскрипции с использованием малых молекул (FKBP, АВА и т.д.) или индуцируемые светом системы (фитохром, LOV домены или криптохром). В одном варианте осуществления фермент CRISPR может быть частью индуцируемого светом транскрипционного эффектора (LITE) для управления изменениями транскрипционной активности специфичным к последовательности образом. Компоненты светом системы могут включать фермент CRISPR, чувствительный к свету гетеродимер цитохрома (например, из Arabidopsis thaliana) и домен активации/репрессии транскрипции. Дополнительные примеры индуцируемых ДНК-связывающих белков и способы их применения представлены в US 61/736465 и US 61/721283, которые включены в данный документ посредством ссылки во всей полноте.
Доставка
В некоторых аспектах настоящее изобретение предусматривает способы, включающие доставку одного или нескольких полинуклеотидов, как, например, или одного, или нескольких векторов, которые описаны в данном документе, одного или нескольких их транскриптов и/или одного или белков, транскрибируемых с них, в клетку-хозяина. В некоторых аспектах настоящее изобретение дополнительно предусматривает клетки, полученные при помощи таких способов, и животных, содержащих такие клетки или полученных из них. В некоторых вариантах осуществления фермент CRISPR в комбинации с (и необязательно образующий комплекс с) направляющей последовательностью доставляют в клетку. Традиционные способы переноса генов с использованием вирусов и без использования вирусов можно применять для введения нуклеиновых кислот в клетки млекопитающих или целевые ткани. Такие способы можно использовать для введения нуклеиновых кислот, кодирующих компоненты системы CRISPR, в клетки в культуре и в организме-хозяине. Системы доставки на основе векторов, отличных от вирусных включают ДНК-плазмиды, РНК (к примеру, транскрипт вектора, описанного в данном документе), "оголенную" нуклеиновую кислоту и нуклеиновую кислоту, образующую комплекс со средством доставки, например, липосому. Системы доставки на основе вирусного вектора включают ДНК- и РНК-вирусы, которые имеют либо эписомальные, либо интегрированные геномы после доставки в клетку. В отношении обзора процедур генной терапии см. Anderson, Science 256: 808-813 (1992); Nabel & Felgner, TIBTECH 11: 211-217 (1993); Mitani & Caskey, TIBTECH 11: 162-166 (1993); Dillon, TIBTECH 11: 167-175 (1993); Miller, Nature 357: 455-460 (1992); Van Brunt, Biotechnology 6(10): 1149-1154 (1988); Vigne, Restorative Neurology and Neuroscience 8: 35-36 (1995); Kremer & Perricaudet, British Medical Bulletin 51(1): 31-44 (1995); Haddada et al., в Current Topics in Microbiology and Immunology, Doerfler and

Способы отличной от вирусной доставки нуклеиновых кислот включают липофекцию, микроинъекцию, баллистическую трансфекцию, виросомы, липосомы, иммунолипосомы; поликатион или конъюгаты липид : нуклеиновая кислота, "оголенную" ДНК, искусственные вирионы и повышенное при помощи средства поглощение ДНК. Липофекция описана, например, в патентах США №№5049386, 4946787 и 4897355), и реагенты для липофекции реализуются в промышленных масштабах (к примеру, Transfectam™ и Lipofectin™). Катионные и нейтральные липиды, которые подходят для эффективной липофекции полинуклеотидов с узнаванием рецептора, включают таковые из Felgner, WO 91/17424; WO 91/16024. Доставка может осуществляться в клетки (к примеру, in vitro или ex vivo введение) или целевые ткани (к примеру, in vivo введение).
Получение комплексов липид : нуклеиновая кислота, в том числе целенаправленно воздействующих липосом, как, например, иммунолипидных комплексов, хорошо известно специалистам в данной области (см., к примеру. Crystal, Science 270: 404-410 (1995); Blaese et al., Cancer Gene Ther. 2: 291-297 (1995); Behr et al., Bioconjugate Chem. 5: 382-389 (1994); Remy et al., Bioconjugate Chem. 5: 647-654 (1994); Gao et al., Gene Therapy 2: 710-722 (1995); Ahmad et al., Cancer Res. 52: 4817-4820 (1992); патенты США №№4186183, 4217344, 4235871, 4261975, 4485054, 4501728, 4774085, 4837028 и 4946787).
При применении систем на основе РНК- и ДНК-вирусов для доставки нуклеиновых кислот используют тщательно разработанные способы обеспечения целенаправленного воздействия вируса на конкретные клетки в организме и перемещения полезных последовательностей вируса в ядро. Вирусные векторы можно вводить непосредственно пациентам (in vivo) или их можно использовать для обработки клеток in vitro, и модифицированные клетки можно необязательно вводить пациентам (ех vivo). Традиционные системы на основе вирусов могут включать ретровирусные, лентивирусные, аденовирусные векторы, векторы на основе аденоассоциированного вируса и вируса простого герпеса для переноса генов. Интеграция в геном хозяина возможна с применением способов переноса генов на основе ретровируса, лентивируса и аденоассоциированного вируса, что часто приводит к длительной экспрессии встроенного трансгена. Кроме того, высокие показатели эффективности трансдукции наблюдали у многих различных типов клеток и целевых тканей.
Тропизм ретровирусов может быть изменен путем включения чужеродных белков оболочки с расширением возможной целевой популяции целевых клеток. Лентивирусные векторы являются ретровирусными векторами, которые способны трансфицировать или инфицировать неделящиеся клетки и, как правило, дают высокие вирусные титры. Выбор системы переноса генов на основе ретровирусов, таким образом, будет зависеть от целевой ткани. Ретровирусные векторы состоят из действующих в цис-положении длинных концевых повторов с упаковывающей емкостью до 6-10 п.о. чужеродной - последовательности. Минимальных действующих в цис-положении LTR достаточно для репликации и упаковки векторов, которые затем используют для интеграции терапевтического гена в целевую клетку с получением постоянной экспрессии трансгена. Широко применяемые ретровирусные векторы включают такие, которые основаны на вирусе лейкоза мышей (MuLV), вирусе лейкоза гиббонов (GaLV), вирусе иммунодефицита обезьян (SIV), вирусе иммунодефицита человека (HTV) и их комбинациях (см., к примеру, Buchscher et al., J. Virol. 66: 2731-2739 (1992); Johann et al., J. Virol. 66: 1635-1640 (1992); Sommnerfelt et al., Virol. 176: 58-59 (1990); Wilson et al., J. Virol. 63: 2374-2378 (1989); Miller et al., J. Virol. 65: 2220-2224 (1991); PCT/US94/05700).
В другом варианте осуществления предусматриваются псевдотипированные ретровирусные векторные частицы на основе оболочки везикуловируса Кокал (см., например, публикацию патентного документа США №20120164118, закрепленного за Fred Hutchinson Cancer Research Center (Онкологический исследовательский центр Фреда Хатчинсона)). Вирус Кокал относится к роду Vesiculovirus и является возбудителем везикулярного стоматита у млекопитающих. Вирус Кокал изначально был выделен из клещей в Тринидаде (Jonkers et al., Am. J. Vet. Res. 25: 236-242 (1964)), и инфекции были идентифицированы в Тринидаде, Бразилии и Аргентине у насекомых, крупного рогатого скота и лошадей. Многие везикуловирусы, которые инфицируют млекопитающих, были выделены у инфицированных в естественных условиях членистоногих, что позволяет предположить, что они являются трансмиссивными. Антитела к везикуловирусам распространены у людей, живущих в сельской местности, где вирусы являются эндемичными и лабораторными; причем инфекции у человека обычно приводят к гриппоподобным симптомам. Гликопротеин оболочки вируса Кокал обладает идентичностью 71,5% на аминокислотном уровне с VSV-G Indiana, и при этом филогенетическое сравнение генов оболочки везикуловирусов продемонстрировало, что вирус Кокал серологически отличается, но является наиболее близкородственным со штаммами VSV-G Indiana среди везикуловирусов. Jonkers et al., Am. J. Vet. Res. 25: 236-242 (1964) и Travassos da Rosa et al., Am. J. Tropical Med. & Hygiene 33: 999-1006 (1984). Псевдотипированные ретровирусные векторные частицы на основе оболочки везикуловируса Кокал могут включать, например, лентивирусные, альфаретровирусные, бетаретровирусные, гаммаретровирусные, дельтаретровирусные и эпсилонретровирусные векторные частицы, которые могут содержать ретровирусный Gag, Pol и/или один или несколько акцессорных белков и белок оболочки везикуловируса Кокал. В некоторых аспектах этих вариантов осуществления Gag, Pol и акцессорные белки являются лентивирусными и/или гаммаретровирусными.
В случаях, при которых временная экспрессия является предпочтительной, можно применять системы на основе аденовирусов. Векторы на основе аденовирусов способны проявлять очень высокую эффективность трансдукции во многих типах клеток и не требуют деления клеток. С применением таких векторов были получены высокие титры и уровни экспрессии. Такой вектор можно получать в больших количествах в относительно простой системе.
Векторы на основе аденоассоциированного вируса ("AAV") также можно использовать для трансдукции клеток целевыми нуклеиновыми кислотами, к примеру, при получении in vitro нуклеиновых кислот и пептидов, и для процедур генной терапии in vivo и ex vivo (см., к примеру. West et al., Virology 160: 38-47 (1987); патент США №4797368; WO 93/24641; Kotin, Human Gene Therapy 5: 793-801 (1994); Muzyczka, J. Clin. Invest. 94: 1351 (1994). Создание рекомбинантных AAV-векторов описано в ряде публикаций, в том числе в патенте США №5173414; Tratschin et al., Mol. Cell. Biol. 5: 3251-3260 (1985); Tratschin, et al., Mol. Cell. Biol. 4: 2072-2081 (1984); Hermonat & Muzyczka, PNAS 81: 6466-6470 (1984); и Samulski et al., J. Virol. 63: 03822-3828 (1989).
Упаковывающие клетки, как правило, используют для получения вирусных частиц, которые способны инфицировать клетку-хозяина. Такие клетки включают клетки 293, которые упаковывают аденовирус, и клетки ψ2 или клетки РА317, которые упаковывают ретровирус. Вирусные векторы, используемые в генной терапии, как правило, создают путем получения линии клеток, которые упаковывают вектор на основе нуклеиновой кислоты в вирусную частицу. Векторы обычно содержат минимальные вирусные последовательности, необходимые для упаковки и последующей интеграции в хозяина, при этом другие вирусные последовательности замещены кассетой экспрессии для экспрессии полинуклеотида(ов). Отсутствующие вирусные функции, как правило, обеспечивают в другом объекте при помощи линии упаковывающих клеток. Например, AAV-векторы, применяемые в генной терапии, как правило, имеют только ITR-последовательности из генома AAV, которые необходимы для упаковки и интеграции в геном хозяина. Вирусная ДНК упакована в линию клеток, которая содержит вспомогательную плазмиду, кодирующую другие гены AAV, а именно rep и cap, но без ITR-последовательностей. Линия клеток также может быть инфицирована аденовирусом в качестве вируса-помощника. Вирус-помощник способствует репликации AAV-вектора и экспрессии генов AAV из вспомогательной плазмиды. Вспомогательная плазмида не упакована в значительном количестве в связи с отсутствием ITR-последовательностей. Контаминация аденовирусом может быть снижена, к примеру, при помощи тепловой обработки, к которой аденовирус более чувствителен, чем AAV.
Соответственно, AAV считается оптимальным кандидатом для применения в качестве трансдуцирующего вектора. Такие трансдуцирующие векторы па основе AAV могут содержать достаточные функции, действующие в цис-положении, для репликации в присутствии вспомогательных функций аденовируса, или герпесвируса, или поксвируса (например, вируса коровьей оспы), предоставленных в другом объекте. Рекомбинантный AAV (rAAV) можно использовать для переноса экзогенных генов в клетки различных клеточных линий. В этих векторах гены cap и/или rep AAV удалены из вирусного генома и замещены выбранным сегментом ДНК. Современные векторы на основе AAV могут вмещать до 4300 оснований встроенной ДНК.
Существует ряд способов получения rAAV и настоящее изобретение предусматривает rAAV и способы получения rAAV. Например, плазмидой (плазмидами), содержащими или практически состоящими из необходимой вирусной конструкции, трансфицируют клетки, инфицированные AAV. Кроме того, эти клетки совместно трансфицируют второй или дополнительной вспомогательной плазмидой с обеспечением генов rep и/или cap AAV, которые являются обязательными для репликации и упаковки рекомбинантной вирусной конструкции. В этих условиях белки rep и/или cap AAV в другом объекте обеспечивают стимуляцию репликации и упаковки конструкции rAAV. Через два-три дня после трансфекции собирают rAAV. Традиционно, rAAV собирают из клеток вместе с аденовирусом. Контаминирующий аденовирус затем инактивируют при помощи тепловой обработки. В настоящем изобретении rAAV преимущественно собирают не из самих клеток, а из супернатанта клеток. Соответственно, в первом аспекте настоящего изобретения предусматривается получение rAAV, и в дополнение к вышеупомянутому, rAAV можно получить при помощи способа, который включает или состоит по сути из следующего: инфицирование восприимчивых клеток AAV, содержащим экзогенную ДНК, в том числе ДНК для экспрессии, и вирусом-помощником (например, аденовирусом, герпесвирусом, поксвирусом, например, вирусом коровьей оспы), причем rAAV не содержит функциональный cap и/или rep (и вирус-помощник (например, аденовирус, герпесвирус, поксвирус, например, вирус коровьей оспы) обеспечивает функцию cap и/или rep, которую не содержит rAAV); или инфицирование восприимчивых клеток rAAV, содержащим экзогенную ДНК, в том числе ДНК для экспрессии, причем рекомбинантная конструкция не содержит функциональный cap и/или rep, и трансфекция указанных клеток плазмидой, предоставляющей функцию cap и/или rep, которую не содержит rAAV; или инфицирование восприимчивых клеток rAAV, содержащим экзогенную ДНК, в том числе ДНК для экспрессии, причем рекомбинантная конструкция не содержит функциональный cap и/или rep, при этом указанные клетки предоставляют функцию cap и/или rep, которую не содержит рекомбинантная конструкция; или трансфекция восприимчивых клеток AAV, не содержащим функциональный cap и/или rep, и плазмидами для встраивания экзогенной ДНК в рекомбинантную конструкцию, так что экзогенная ДНК экспрессируется рекомбинантной конструкцией, и для предоставления функций rep и/или cap, причем трансфекция приводит в результате к rAAV, содержащему экзогенную ДНК, в том числе ДНК для экспрессии, который не содержит функциональный cap и/или rep.
RAAV можно получить исходя из AAV, как описано в данном документе, и преимущественно он может представлять собой rAAV1, rAAV2, AAV5 или rAAV, имеющий гибридную структуру или капсид, что может предусматривать AAV1, AAV2, AAV5 или любую их комбинацию. Можно выбрать AAV из rAAV с учетом клеток, подлежащих целенаправленному воздействию с применением rAAV, например, можно выбрать AAV серотипов 1, 2, 5, или гибридную структуру или капсид AAV1, AAV2, AAV5, или любую их комбинацию для целенаправленного воздействия на головной мозг или нейроны; и при этом можно выбрать AAV4 для целенаправленного воздействия на сердечную ткань.
Кроме клеток 293, при осуществлении на практике настоящего изобретения можно применять другие клетки, и при этом относительная инфицирующая способность определенных серотипов AAV in vitro по отношению к этим клеткам (см. Grimm, D. et al, J. Virol. 82: 5887-5911 (2008)) представлена ниже.
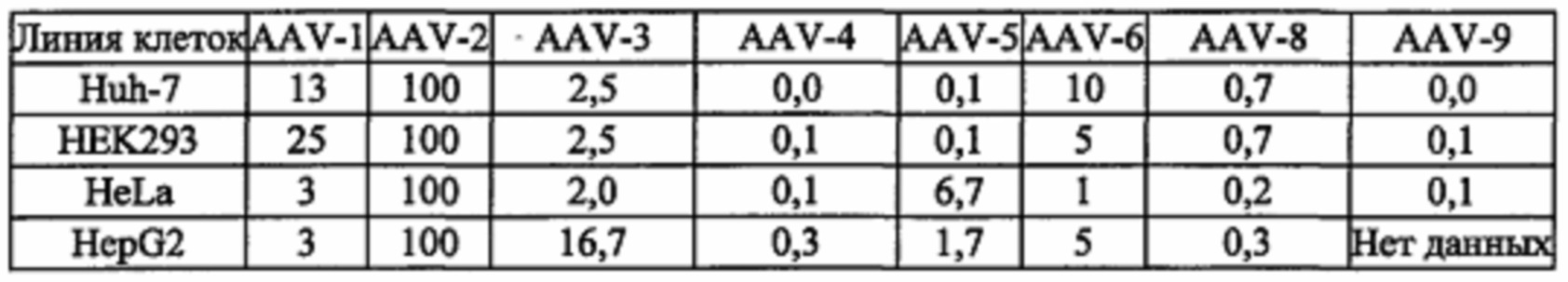

Настоящее изобретение предусматривает rAAV, который содержит или состоит по сути из экзогенной молекулы нуклеиновой кислоты, кодирующей систему CRISPR (короткие палиндромные повторы, регулярно расположенные группами), например, множество кассет, содержащих или состоящих из первой кассеты, содержащей или состоящей по сути из промотора, молекулы нуклеиновой кислоты, кодирующей CRISPR-ассоциированный (Cas) белок (предполагаемые нуклеазные или хеликазные белки), например, Cas9 и терминатор, и две или более, преимущественно до предела упаковки вектора, например, всего (включая первую кассету) пять кассет, содержащих или состоящих по сути из промотора, молекулы нуклеиновой кислоты, кодирующей направляющую РНК (gRNA) и терминатор (например, каждая кассета схематически представлена как промотор-gRNA1-терминатор, промотор-gRNA2-терминатор … Промотор-gRNA(N)-терминатор (где N относится к тому количеству, которое можно встроить, находящемуся на верхней границе предела упаковки вектора), или два или более отдельных rAAV, причем каждый содержит одну или несколько кассет системы CRISPR, например, первый rAAV, содержащий первую кассету, содержащую или состоящую по сути из промотора, молекулы нуклеиновой кислоты, кодирующей Cas, например, Cas9, и терминатора, и второй rAAV, содержащий множество, а именно четыре кассеты, содержащие или состоящие по сути из промотора, молекулы нуклеиновой кислоты, кодирующей направляющую РНК (gRNA), и терминатора (например, каждая кассета схематически представлена как промотор-gRNA1-терминатор, промотор-gRNA2-терминатор … промотор-gRNA(N)-терминатор (где N относится к количеству того, что можно встроить, находящемуся на верхней границе предела упаковки вектора). Поскольку rAAV представляет собой ДНК-содержащий вирус, молекулы нуклеиновой кислоты в изложенном в данном документе обсуждении в отношении AAV или rAAV преимущественно представляют собой ДНК. В некоторых вариантах осуществления промотор преимущественно представляет собой промотор синапсина I человека (hSyn).
Дополнительные способы доставки нуклеиновых кислот в клетки известны специалистам в данной области. См., например, US 20030087817, включенный в данный документ при помощи ссылки.
В некоторых вариантах осуществления клетка-хозяин транзиентно или нетранзиентно трасфицируется одним или несколькими векторами, описанными в данном документе. В некоторых вариантах осуществления клетка трансфицирована так, как это в естественных условиях происходит у субъекта. В некоторых вариантах осуществления клетка, которую трансфицируют, взята от субъекта. В некоторых вариантах осуществления клетка получена из клеток, взятых от субъекта, как, например, линия клеток. Широкий спектр линий клеток для культуры тканей известен из уровня техники. Примеры линий клеток включают без ограничения С8161, CCRF-CEM, MOLT, mIMCD-3, NHDF, HeLa-S3, Huh1, Huh4, Huh7, HUVEC, HASMC, HEKn, HEKa, MiaPaCell, Panc1, PC-3, TF1, CTLL-2, C1R, Rat6, CV1, RPTE, A10, T24, J82, A375, ARH-77, Calu1, SW480, SW620, SKOV3, SK-UT, CaCo2, P388D1, SEM-K2, WEHI-231, HB56, TIB55, Jurkat, J45.01, LRMB, Bc1-1, BC-3, IC21, DLD2, Raw264.7, NRK, NRK-52E, MRC5, MEF, Hep G2, HeLa B, HeLa T4, COS, COS-1, COS-6, COS-M6A, эпителиальные клетки почки обезьяны BS-C-1, эмбриональные фибробласты мыши BALB/ 3Т3, 3Т3 Swiss, 3T3-L1, фетальные фибробласты человека 132-d5; фибробласты мыши 10.1, 293-Т, 3Т3, 721, 9L, А2780, A2780ADR, A2780cis, А172, А20, А253, А431, А-549, ALC, В1б, В35, клетки ВСР-1, BEAS-2B, bEnd.3, BHK-21, BR 293, ВхРС3, С3Н-10Т1/2, С6/36, Cal-27, СНО, СНО-7, CHO-IR, CHO-K1, СНО-K2, СНО-Т, СНО Dhfr -/-, COR-L23, COR-L23/CPR, COR-L23/5010, COR-L23/R23, COS-7, COV-434, CML T1, CMT, CT26, D17, DH82, DU145, DuCaP, EL4, EM2, EM3, EMT6/AR1, EMT6/AR10.0, FM3, H1299, H69, HB54, HB55, HCA2, HEK-293, HeLa, Hepa1c1c7, HL-60, HMEC, HT-29, Jurkat, клетки JY, клетки K562, Ku812, KCL22, KG1, KYO1, LNCap, Ma-Mel 1-48, MC-38, MCF-7, MCF-10A, MDA-MB-231, MDA-MB-468, MDA-MB-435, MDCK II, MDCK II, MOR/0.2R, MONO-MAC 6, MTD-1A, MyEnd, NCI-H69/CPR, NCI-H69/LX10, NCI-H69/LX20, NCI-H69/LX4, NIH-3T3, NALM-1, NW-145, линии клеток OPCN/OPCT, Peer, PNT-1A / PNT 2, RenCa, RIN-5F, RMA/RMAS, клетки Saos-2, Sf-9, SkBr3, T2, T-47D, T84, линия клеток ТНР1, U373, U87, U937, VCaP, клетки Vero, WM39, WT-49, X63, YAC-1, YAR и их трансгенные разновидности. Линии клеток доступны из ряда источников, известных специалистам в данной области (см., к примеру. Американская коллекция типовых культур (АТСС) (Манассас, Вирджиния)). В некоторых вариантах осуществления клетку, трансфицированную одним или несколькими векторами, описанными в данном документе, используют для получения новой линии клеток, содержащей одну или несколько полученных из вектора последовательностей. В некоторых вариантах осуществления клетку, транзиентно трансфицированную компонентами системы CRISPR, которая описана в данном документе (как, например, путем транзиентной трансфекции одним или несколькими векторами или трансфекции РНК), и модифицированную при помощи активности комплекса CRISPR, используют для получения новой линии клеток, содержащей клетки, которые содержат модификацию, но у которых отсутствует любая другая экзогенная последовательность. В некоторых вариантах осуществления клетки, транзиентно или нетранзиентно трансфицированные одним или несколькими векторами, описанными в данном документе, или линии клеток, полученные из таких клеток, использовали при оценивании одного или нескольких тестовых соединений.
В некоторых вариантах осуществления один или несколько векторов, описанных в данном документе, используют для получения отличного от человека трансгенного животного или трансгенного растения. В некоторых вариантах осуществления трансгенным животным является млекопитающее, как, например, мышь, крыса или кролик. Способы получения трансгенных животных и растений известны из уровня техники и, как правило, начинаются со способа трансфекции клетки, такого как описанный в данном документе.
В другом варианте осуществления может предусматриваться устройство для доставки жидкости с матрицей игл (см., например, публикацию патентного документа США №20110230839, закрепленного за Fred Hutchinson Cancer Research Center (Онкологический исследовательский центр Фреда Хатчинсона)), для доставки CRISPR Cas в плотную ткань. Устройство согласно публикации патентного документа США №20110230839 для доставки жидкости в плотную ткань может содержать множество игл, расположенных в виде матрицы; множество емкостей, каждая из которых находится в жидкостном соединении с соответствующей одной иглой из множества игл; и множество приводов, функционально связанных с соответствующими емкостями из множества емкостей и выполненных с возможностью регулирования давления жидкости в емкости. В определенных вариантах осуществления каждый из множества приводов может содержать один из множества поршней, причем первая концевая часть каждого из множества поршней находится в соответствующей одной емкости из множества емкостей, и в определенных дополнительных вариантах осуществления поршни из множества поршней функционально связаны вместе по соответствующим вторым концевым частям с обеспечением возможности одновременного нажатия. В определенных других дополнительных вариантах осуществления может предусматриваться управляющий элемент для поршней, сконфигурированный с возможностью выборочного нажатия всех из множества поршней с различной скоростью. В других вариантах осуществления каждый из множества приводов может содержать одну из множества жидкостных поточных линий, имеющих первую и вторую концевые части, причем первая концевая часть каждой из множества жидкостных поточных линий соединена с соответствующей одной емкостью из множества емкостей. В других вариантах осуществления устройство может содержать источник давления жидкости, и при этом каждый из множества приводов предусматривает жидкостное соединение между источником давления жидкости и соответствующей одной емкостью из множества емкостей. В дополнительных вариантах осуществления источник давления жидкости может предусматривать по меньшей мере одно из следующих: компрессор, вакуумный накопитель, перистальтический насос, основной цилиндр, микрожидкостный насос и клапана. В другом варианте осуществления каждая из множества игл может содержать множество отверстий, распределенных вдоль ее длины.
Модификация мишени
В одном аспекте настоящее изобретение предусматривает способы модификации целевого полинуклеотида в эукариотической клетке, что может происходить in vivo, ex vivo или in vitro. В некоторых вариантах осуществления способ включает забор клетки или популяции клеток от человека, или отличного от человека животного, или растения и модификацию клетки или клеток. Культивирование можно осуществлять на любой стадии ех vivo. Клетку или клетки можно даже повторно вводить отличному от человека животному или в растение. Что касается повторно вводимых клеток, особенно предпочтительно, чтобы эти клетки являлись стволовыми клетками.
В некоторых вариантах осуществления способ включает обеспечение связывания комплекса CRISPR с целевым полинуклеотидом для осуществления расщепления указанного целевого полинуклеотида с модификацией, таким образом, целевого полинуклеотида, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизующейся с целевой последовательностью в указанном целевом полинуклеотиде, где указанная направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизуется с tracr-последовательностью.
В одном аспекте настоящее изобретение предусматривает способ модификации экспрессии полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления способ включает обеспечение связывания комплекса CRISPR с полинуклеотидом так, что указанное связывание приводит к повышенной или пониженной экспрессии указанного полинуклеотида; где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизующейся с целевой последовательностью в указанном полинуклеотиде, где указанная направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизуется с tracr-последовательностью. Аналогичные факторы и условия распространяются на способы модификации целевого полинуклеотида, как изложено выше. Фактически, эти варианты отбора образцов, культивирования и повторного введения охватывают аспекты настоящего изобретения.
Действительно, в любом аспекте настоящего изобретения комплекс CRISPR может содержать фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизующейся с целевой последовательностью, где указанная направляющая последовательность может быть связана с парной tracr-последовательностью, которая, в свою очередь, может гибридизоваться с tracr-последовательностью. Аналогичные факторы и условия распространяются на способы модификации целевого полинуклеотида, как изложено выше.
Наборы
В одном аспекте настоящее изобретение предусматривает наборы, содержащие любой один или несколько из элементов, раскрытых в приведенных выше способах и композициях. Элементы могут быть предоставлены отдельно или в комбинациях и могут быть предоставлены в любом подходящем контейнере, как, например, ампуле, флаконе или пробирке. В некоторых вариантах осуществления набор включает инструкции на одном или нескольких языках, например, на более чем одном языке.
В некоторых вариантах осуществления набор содержит один или несколько реагентов для применения в способе, в котором используется один или несколько элементов, описанных в данном документе. Реагенты могут быть предоставлены в любом подходящем контейнере. Например, набор может предусматривать один или несколько реакционных буферов или буферов для хранения. Реагенты могут быть предоставлены в форме, которая применима в конкретном анализе, или в форме, которая предусматривает добавление одного или нескольких других компонентов перед применением (к примеру, в форме концентрата или лиофилизированной форме). Буфер может быть любым буфером, в том числе без ограничения натрий-карбонатным буфером, натрий-бикарбонатным буфером, боратным буфером, Tris-буфером, буфером MOPS, буфером HEPES и их комбинациями. В некоторых вариантах осуществления буфер является щелочным. В некоторых вариантах осуществления буфер имеет значение рН от приблизительно 7 до приблизительно 10. В некоторых вариантах осуществления набор содержит один или несколько олигонуклеотидов, соответствующих направляющей последовательности, для встраивания в вектор с тем, чтобы имела место функциональная связь направляющей последовательности и регуляторного элемента. В некоторых вариантах осуществления набор содержит матричный полинуклеотид для гомологичной рекомбинации. В некоторых вариантах осуществления набор содержит один или несколько векторов и/или один или несколько полинуклеотидов, описанных в данном документе. Преимущественно набор может предоставлять все элементы системы по настоящему изобретению.
Комплекс CRISPR
В одном аспекте настоящее изобретение предусматривает способы применения одного или нескольких элементов системы CRISPR. Комплекс CRISPR по настоящему изобретению представляет собой эффективное средство модификации целевого полинуклеотида. Комплекс CRISPR по настоящему изобретению характеризуется большим разнообразием полезных свойств, включая модификацию (например, делецию, вставку, транслокацию, инактивацию, активацию) целевого полинуклеотида во множестве типов клеток. Комплекс CRISPR по настоящему изобретению как таковой имеет широкий спектр применений, к примеру, в генной терапии, скрининге лекарственных средств, диагностике и прогнозировании развития заболеваний. Иллюстративный комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, которая гибридизуется с целевой последовательностью в целевом полинуклеотиде. Направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизуется с tracr-последовательностью.
В одном варианте осуществления настоящее изобретение предусматривает способ расщепления целевого полинуклеотида. Способ предусматривает модификацию целевого полинуклеотида с применением комплекса CRISPR, который связывается с целевым полинуклеотидом и осуществляет расщепление указанного целевого полинуклеотида. Как правило, комплекс CRISPR согласно настоящему изобретению при введении в клетку создает разрыв (например, одноцепочечный или двухцепочечный разрыв) в геномной последовательности. Например, способ можно применять для расщепления гена, ответственного за развитие заболевания, в клетке.
Репарация разрыва, созданного комплексом CRISPR, может осуществляться посредством способа репарации, например, путем склонного к ошибкам негомологичного соединения концов (NHEJ) или высокоточной репарации с использованием гомологичной рекомбинации (HDR) (фигура 29). В ходе данного способа репарации в геномную последовательность может быть введен экзогенный матричный полинуклеотид. В некоторых способах способ HDR используют для модификации геномной последовательности. Например, в клетку вводят экзогенный матричный полинуклеотид, содержащий последовательность, подлежащую встраиванию, фланкированную последовательностью, расположенной выше, и последовательностью, расположенной ниже. Последовательности, расположенные выше и ниже, характеризуются сходством последовательности с каждой стороной сайта встраивания в хромосоме.
При необходимости донорный полинуклеотид может представлять собой ДНК, например, ДНК-плазмиду, бактериальную искусственную хромосому (ВАС), искусственную хромосому дрожжей (YAC), вирусный вектор, линейный фрагмент ДНК, ПЦР-фрагмент, "оголенную" нуклеиновую кислоту или нуклеиновую кислоту в комплексе со средством доставки, например, липосомой или полоксамером.
Экзогенный" матричный полинуклеотид содержит последовательность, подлежащую встраиванию (например, мутированный ген). Последовательность, предназначенная для встраивания, может представлять собой последовательность, эндогенную или экзогенную по отношению к клетке. Примеры последовательности, подлежащей встраиванию, включают полинуклеотиды, кодирующие белок или некодирующую РНК (например, микроРНК). Таким образом, последовательность, предназначенная для встраивания, может быть функционально связанной с соответствующей регуляторной последовательностью или последовательностями. В альтернативном случае, последовательность, подлежащая встраиванию, может обеспечивать регуляторную функцию.
Последовательности, расположенные выше и ниже, в экзогенном матричном полинуклеотиде выбраны так, чтобы способствовать рекомбинации между хромосомной последовательностью, представляющей интерес, и донорным полинуклеотидом. Последовательность, расположенная выше, представляет собой последовательность нуклеиновой кислоты, которая обладает сходством последовательности с геномной последовательностью, расположенной выше целевого сайта интеграции. Аналогично, последовательность, расположенная ниже, представляет собой последовательность нуклеиновой кислоты, которая обладает сходством последовательности с хромосомной последовательностью, расположенной ниже целевого сайта интеграции. Последовательности, расположенные выше и ниже, в экзогенном матричном полинуклеотиде могут иметь 75%, 80%, 85%, 90%, 95% или 100% идентичность последовательности с целевой геномной последовательностью. Предпочтительно последовательности, расположенные выше и ниже, в экзогенном матричном полинуклеотиде могут иметь приблизительно 95%, 96%, 97%, 98%, 99% или 100% идентичность последовательности с целевой геномной последовательностью. В некоторых способах последовательности, расположенные выше и ниже, в экзогенном матричном полинуклеотиде имеют приблизительно 99% или 100% идентичность последовательности с целевой геномной последовательностью.
Последовательность, расположенная выше или ниже, может содержать от приблизительно 20 п.о. до приблизительно 2500 п.о., например, приблизительно 50, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 1600, 1700, 1800, 1900, 2000, 2100, 2200, 2300, 2400 или 2500 п.о. В некоторых способах иллюстративная последовательность, расположенная выше или ниже, имеет от приблизительно 200 п.о. до приблизительно 2000 п.о., от приблизительно 600 п.о. до приблизительно 1000 п.о. или, в частности, от приблизительно 700 п.о. до приблизительно 1000 п.о.
В некоторых способах экзогенный матричный полинуклеотид может дополнительно содержать маркер. Такой маркер может облегчать скрининг на предмет целевых интеграции. Примеры подходящих маркеров включают сайты рестрикции, флуоресцентные белки или селектируемые маркеры. Экзогенный матричный полинуклеотид согласно настоящему изобретению можно сконструировать с применением методик рекомбинантной ДНК (см., например, Sambrook et al., 2001 и Ausubel et al., 1996).
В иллюстративном способе модификации целевого полинуклеотида посредством интеграции экзогенного матричного полинуклеотида в геномную последовательность вводят двухцепочечный разрыв при помощи комплекса CRISPR, осуществляют репарацию разрыва посредством гомологичной рекомбинации с участием экзогенного матричного полинуклеотида, так что матрица интегрируется в геном. Наличие двухцепочечного разрыва обеспечивает интеграцию матрицы.
В других вариантах осуществления настоящее изобретение предусматривает способ модификации экспрессии полинуклеотида в эукариотической клетке. Способ включает повышение или снижение экспрессии целевого полинуклеотида при помощи комплекса CRISPR, который связывается с полинуклеотидом.
В некоторых способах целевой полинуклеотид можно инактивировать для осуществления модификации экспрессии в клетке. Например, при связывании комплекса CRISPR с целевой последовательностью в клетке целевой полинуклеотид инактивируется таким образом, что последовательность не транскрибируется, кодируемый белок не продуцируется или последовательность не функционирует так, как последовательность дикого типа. Например, последовательность, кодирующую белок или микроРНК, можно инактивировать таким образом, что белок не будет продуцироваться.
В некоторых способах регуляторную последовательность можно инактивировать так что она более не функционирует в качестве регуляторной последовательности. Как используется в данном документе, "регуляторная последовательность" относится к любой последовательности нуклеиновой кислоты, которая оказывает влияние на транскрипцию, трансляцию или доступность последовательности нуклеиновой кислоты. Примеры регуляторной последовательности включают промотор, терминатор транскрипции и энхансер, которые являются регуляторными последовательностями.
Инактивированная целевая последовательность может содержать мутацию по типу делении (т.е. деления одного или нескольких нуклеотидов), мутацию по типу вставки (т.е. вставка одного или нескольких нуклеотидов) или нонсенс-мутацию (т.е. замена одного нуклеотида другим нуклеотидом, так что вводится стоп-кодон). В некоторых способах инактивация целевой последовательности приводит в результате к "нокауту" целевой последовательности.
Модели заболеваний
Способ по настоящему изобретению можно применять для создания растения, животного или клетки, которые можно использовать в качестве модели заболевания. Как используется в данном документе, "заболевание" относится к заболеванию, нарушению или симптому у субъекта. Например, способ по настоящему изобретению можно использовать для создания животного или клетки, которые содержат модификацию одной или нескольких последовательностей нуклеиновой кислоты, ассоциированных с заболеванием, или растения, животного или клетки, в которых изменена экспрессия одной или нескольких последовательностей нуклеиновой кислоты, ассоциированных с заболеванием. Такая последовательность нуклеиновой кислоты может кодировать последовательность белка, ассоциированного с заболеванием или может представлять собой регуляторную последовательность, ассоциированную с заболеванием. Соответственно, подразумевается, что в вариантах осуществления по настоящему изобретению растение, субъект, пациент, организм или клетка может относиться к субъекту, отличному от человека, пациенту, организму или клетке. Таким образом, настоящее изобретение предусматривает растение, животное или клетку, полученные при помощи способа по настоящему изобретению, или их потомство. Потомство может представлять собой клон полученного растения или животного или его можно получить при помощи полового размножения посредством скрещивания с другими индивидами того же вида для придания дополнительных желательных признаков их потомкам. Клетка может находиться in vivo или ex vivo в случае многоклеточных организмов, в частности, животных или растений. В случае, если клетка находится в культуре, можно получить линию клеток при выполнении соответствующих условий культивирования, и предпочтительно, если клетка соответствующим образом приспособлена для этой цели (например, стволовая клетка). Также предусматриваются линии бактериальных клеток, полученные согласно настоящему изобретению. Следовательно, также предусматриваются линии клеток.
В некоторых способах модель заболевания можно использовать для исследования влияния мутаций на животное или клетку и развитие и/или прогрессирование заболевания с применением показателей, обычно используемых при исследовании заболевания. В альтернативном случае, такая модель заболевания является применимой для исследования воздействия фармацевтически активного соединения на заболевание.
В некоторых способах модель заболевания можно использовать для оценки эффективности потенциальной стратегии генной терапии. Таким образом, ген или полинуклеотид, ассоциированный с заболеванием, можно модифицировать, так что развитие и/или прогрессирование заболевания замедляется или уменьшается. В частности, способ предусматривает модификацию гена или полинуклеотида, ассоциированного с заболеванием, так что продуцируется измененный белок, и в результате у животного или клетки наблюдается ответ, связанный с этим изменением. Соответственно, в некоторых способах генетически модифицированное животное можно сравнивать с животным, предрасположенным к развитию заболевания, так что можно оценить эффект осуществления генной терапии.
В другом варианте осуществления настоящее изобретение предусматривает способ получения биологически активного средства, которое модулирует процесс передачи сигнала в клетке, ассоциированный с геном, ответственным за развитие заболевания. Способ предусматривает приведение в контакт тестового соединения с клеткой, содержащей один или несколько векторов, которые управляют экспрессией одного или нескольких из фермента CRISPR, направляющей последовательности, связанной с парной tracr-последовательностью, и tracr-последовательности; и обнаружение изменения при считывании, которое свидетельствует об уменьшении или усилении процесса передачи сигнала в клетке, ассоциированного, например, с мутацией в гене, ответственном за развитие заболевания, который содержится в клетке.
Клеточную модель или животную модель можно сконструировать в сочетании со способом по настоящему изобретению для скрининга изменения клеточной функции. Такую модель можно использовать для исследования влияния геномной последовательности, модифицированной при помощи комплекса CRISPR согласно настоящему изобретению, на клеточную функцию, представляющую интерес. Например, модель клеточной функции можно использовать для исследования воздействия модифицированной геномной последовательности на внутриклеточную передачу сигнала или внеклеточную передачу сигнала. В альтернативном случае, модель клеточной функции можно использовать для исследования воздействий модифицированной геномной последовательности на сенсорную чувствительность. В некоторых таких моделях одна или несколько геномных последовательностей, ассоциированных с биохимическим путем передачи сигнала, в модели является модифицированной.
Несколько моделей заболеваний были специально исследованы. Они включают гены CHD8, KATNAL2 и SCN2A, связанные с риском развития аутизма de novo; и ген UBE3A, связанный с синдромным аутизмом (синдром Ангельмана). Эти гены и полученные в результате модели аутизма, разумеется, являются предпочтительными, но служат для - того, чтобы продемонстрировать широкую применимость настоящего изобретения по отношению к генам и соответствующим моделям.
Измененную экспрессию одной или нескольких геномных последовательностей, ассоциированных с биохимическим путем передачи сигнала, можно определять при помощи анализа различия по уровням мРНК соответствующих генов между клетками исследуемой модели и контрольными клетками, если их приводят в контакт с кандидатным средством. В альтернативном случае, различную экспрессию последовательностей, ассоциированных с биохимическим путем передачи сигнала, определяют посредством выявления различия по уровню кодируемого полипептида или продукта гена.
Для анализа индуцированного определенным средством изменения уровня мРНК-транскриптов или соответствующих полинуклеотидов, нуклеиновую кислоту, которая содержится в образце, вначале экстрагируют в соответствии со стандартными способами из уровня техники. Например, мРНК можно выделять с использованием различных литических ферментов или химических растворов, в соответствии с методиками, изложенными в Sambrook et al. (1989), или экстрагировать при помощи смол, связывающих нуклеиновые кислоты, в соответствии с прилагаемыми инструкциями, предоставленными производителями. МРНК, которая содержалась в экстрагированном образце нуклеиновой кислоты, затем выявляли при помощи методик амплификации или общепринятых гибридизационных анализов (например, анализа с помощью нозерн-блоттинга) в соответствии со способами, широко известными из уровня техники или основанными на способах, проиллюстрированных в данном документе.
Для целей настоящего изобретения амплификация означает любой способ с использованием праймера и полимеразы, способной обеспечивать репликацию целевой последовательности с достаточной точностью. Амплификацию можно осуществлять при помощи природных или рекомбинантных ДНК-полимераз, таких как TaqGold™, ДНК-полимераза Т7, фрагмент Кленова ДНК-полимеразы Е. coli и обратная транскриптаза. Предпочтительным способом амплификации является ПЦР. В частности, выделенную РНК можно подвергать анализу с обратной транскрипцией, который объединен с количественной полимеразной цепной реакцией (RT-PCR) для количественного определения уровня экспрессии последовательности, ассоциированной с биохимическим путем передачи сигнала.
Выявление уровня экспрессии генов можно осуществлять в анализе амплификации в режиме реального времени. В одном аспекте амплифицированные продукты можно непосредственно визуализировать при помощи флуоресцентных ДНК-связывающих средств, в том числе без ограничения ДНК-интеркаляторов и средств, связывающихся с бороздкой спирали ДНК. Поскольку количество интеркаляторов, включенных в двухцепочечные молекулы ДНК, как правило, является пропорциональным количеству амплифицированных ДНК-продуктов, специалист в данной области техники может беспрепятственно определить количество амплифицированных продуктов путем количественного определения флуоресценции интеркалирующего красителя с применением общепринятых оптических систем из уровня техники. ДНК-связывающий краситель, подходящий для этой задачи, охватывает SYBR green, SYBR blue, DAPI, пропидий иодид, Hoeste, SYBR gold, этидий бромид, акридины, профлавин, акридиновый оранжевый, акрифлавин, фторкумарин, эллиптицин, дауномицин, хлорохин, дистамицин D, хромомицин, хомидий, митрамицин, рутений полипиридил, антрамицин и т.п.
В другом аспекте можно использовать другие флуоресцентные метки, например, зонды, специфичные по отношению к последовательности, в реакции амплификации для обеспечения выявления и количественного определения амплифицированных продуктов. Количественная амплификация с использованием зонда основана на специфичном по отношению к последовательности выявлении требуемого амплифицированного продукта. Используются флуоресцентные зонды, специфичные по отношению к мишени (например, зонды TaqMan®), что приводит в результате к увеличению специфичности и чувствительности. Способы осуществления количественной амплификации с использованием зонда являются общепринятыми в данной области техники и описаны в патенте США №5210015.
В еще одном аспекте можно осуществлять общепринятый гибридизационный анализ с использованием гибридизационных зондов, которые характеризуются гомологией последовательности с последовательностями, ассоциированными с биохимическим путем передачи сигнала. Как правило, в реакции гибридизации зондам дают возможность образовать стабильные комплексы с последовательностями, ассоциированными с биохимическим путем передачи сигнала, которые содержатся в биологическом образце, полученном от исследуемого субъекта. Специалист в данной области поймет, что если антисмысловая нуклеиновая кислота используется в качестве зонда, то целевые полинуклеотиды, предоставленные в образце, выбирают так, чтобы они были комплементарными последовательностям антисмысловых нуклеиновых кислот. Напротив, если нуклеотидный зонд является смысловой нуклеиновой кислотой, то целевой полинуклеотид выбирают так, чтобы он был комплементарным последовательностям смысловой нуклеиновой кислоты.
Гибридизацию можно осуществлять в условиях различной жесткости. Подходящие условия гибридизации для осуществления на практике настоящего изобретения являются такими, что обеспечивающее распознавание взаимодействие зонда с последовательностями, ассоциированными с биохимическим путем передачи сигнала, является как достаточно специфичным, так и достаточно стабильным. Условия, которые приводят к увеличению жесткости реакции гибридизации, хорошо известны и опубликованы в уровне техники. См., например (Sambrook, et al., (1989); Nonradioactive In Situ Hybridization Application Manual, Boehringer Mannheim, second edition). Гибридизационный анализ можно осуществлять с использованием зондов, иммобилизованных на любой твердой подложке, в том числе без ограничения нитроцеллюлозной, стеклянной, кремниевой, и ряда ДНК-чипов. Предпочтительный гибридизационный анализ проводят на генных чипах высокой плотности, описанных в патенте США №5445934.
Для удобного выявления комплексов зонд-мишень, образованных в ходе гибридизационного анализа, осуществляют конъюгирование нуклеотидного зонда с детектируемой меткой. Детектируемые метки, подходящие для применения в настоящем изобретении, включают любую композицию, выявляемую при помощи фотохимических, биохимических, спектроскопических, иммунохимических, электрических, оптических или химических средств. Широкий спектр соответствующих детектируемых меток известен из уровня техники, причем он включает флуоресцентные или хемилюминисцентные метки, метки на основе радиоактивных изотопов, ферментные или другие лиганды. В предпочтительных вариантах осуществления, вероятно, предпочтительной будет флуоресцентная метка или ферментная метка, например, дигоксигенин, β-галактозидаза, уреаза, щелочная фосфатаза или пероксидаза, комплекс авидин/биотин.
Способы выявления, применяемые для выявления или количественного определения интенсивности гибридизации, как правило, будут зависеть от метки, выбранной выше. Например, радиоактивные метки можно выявлять с использованием фотографической пленки или фосфовизуализатора. Флуоресцентные маркеры можно выявлять и проводить количественное определение с использованием фотодетектора для выявления излучаемого света. Ферментные метки, как правило, выявляют посредством предоставления субстрата для фермента и измерения количества продукта реакции, образованного при воздействии фермента на субстрат; и при этом, в конечном итоге, колориметрические метки выявляют посредством простой визуализации цветной метки.
Индуцированное определенным средством изменение экспрессии последовательностей, ассоциированных с биохимическим путем передачи сигнала, также можно определять посредством исследования соответствующих продуктов генов. Определение уровня белка, как правило, включает а) приведение в контакт белка, содержащегося в биологическом образце, со средством, которое специфично связывается с белком, ассоциированным с биохимическим путем передачи сигнала; и (b) идентификацию любого комплекса средство: белок, образованного таким образом. В одном аспекте данного варианта осуществления средство, которое специфически связывает белок, ассоциированный с биохимическим путем передачи сигнала, представляет собой антитело, предпочтительно моноклональное антитело.
Реакцию осуществляют посредством приведения в контакт средства с образцом белков, ассоциированных с биохимическим путем передачи сигнала, полученным из исследуемых образцов при условиях, которые обеспечивают возможность образования комплекса между средством и белками, ассоциированными с биохимическим путем передачи сигнала. Образование комплекса можно выявлять непосредственно или опосредованно в соответствии со стандартными методиками из уровня техники. В способе непосредственного выявления средства предоставляют с детектируемой меткой и из комплекса можно удалять непрореагировавшие средства; причем количество оставшейся метки, таким образом, отражает количество образованного комплекса. Для такого способа предпочтительно выбирать метки, которые остаются прикрепленными к средствам даже при жестких условиях отмывки. Предпочтительно, чтобы метка не препятствовала реакции связывания. В альтернативном случае, для методики опосредованного выявления можно использовать средство, которое содержит метку, введенную либо химическим, либо ферментативным путем. Желаемая метка, как правило, не препятствует связыванию или стабильности полученного в результате комплекса средство : полипептид. Однако, метка, как правило, разработана так, чтобы она была доступной для эффективного связывания с антителом и, следовательно, выработки детектируемого сигнала.
Широкий спектр меток, подходящих для выявления уровней белка, известен из уровня техники. Неограничивающие примеры включают радиоактивные изотопы, ферменты, коллоидные металлы, флуоресцентные соединения, биолюминисцентные соединения и хемилюминисцентные соединения.
Количество комплексов средство лолипептид, образованных в ходе реакции связывания, можно количественно определять при помощи стандартных количественных анализов. Как проиллюстрировано выше, образование комплекса средство лолипептид можно измерить непосредственно по количеству метки, оставшейся в участке связывания. В альтернативном случае, белок ассоциированный с биохимическим путем передачи сигнала, исследуют в отношении его способности конкурировать с меченым аналогом за участки связывания на специфическом средстве. В этом конкурентном анализе количество захваченной метки является обратно пропорциональным количеству последовательностей белка, ассоциированного с биохимическим путем передачи сигнала, присутствующих в исследуемом образце.
Ряд методик анализа белка, основанных на общих принципах, изложенных выше, доступен из уровня техники. Они включают без ограничения радиоиммунные анализы, ELISA (твердофазные ферментные иммунорадиометрические анализы), "сэндвич"-иммуноанализы, иммунорадиометрические анализы, in situ иммуноанализы (с применением, например, коллоидного золота, фермента или радиоизотопных меток), вестерн-блот анализ, иммунопреципитационные анализы, иммунофлуоресцентные анализы и SDS-PAGE.
Антитела, которые обеспечивают специфичное распознавание или связываются с белками, ассоциированными с биохимическим путем передачи сигнала, являются предпочтительными для осуществления вышеупомянутых анализов белка. При необходимости можно использовать антитела, которые обеспечивают распознавание конкретного типа посттрансляционных модификаций (например, модификации, индуцируемые биохимическим путем передачи сигнала). Посттрансляпионные модификации включают без ограничения гликозилирование, липидизацию, ацетилирование и фосфорилирование. Эти антитела можно приобрести у коммерческих поставщиков. Например, антитела к фосфотирозину, которые обеспечивают специфичное распознавание фосфорилированных по тирозину белков, доступны от ряда поставщиков, включая Invitrogen и Perkin Elmer. Антитела к фосфотирозину являются особенно применимыми при выявлении белков, которые различным образом фосфорилируются по их тирозиновым остаткам в ответ на стресс ER (эндоплазматического ретикулума). Такие белки включают без ограничения эукариотический фактор инициации трансляции 2 альфа (eIF-2α). В альтернативном случае, эти антитела можно получить при помощи общепринятых методик поликлональных или моноклональных антител посредством иммунизации животного-хозяина или клетки, продуцирующей антитела, целевым белком, который характеризуется необходимой посттрансляционной модификацией.
При осуществлении заявленного способа на практике, может быть необходимо определить профиль экспрессии белка, ассоциированного с биохимическим путем передачи сигнала, в различных тканях организма, в различных типах клеток и/или в различных субклеточных структурах. Данные исследования можно проводить с применением тканеспецифичных, специфичных в отношении определенных клеток или специфичных в отношении определенных субклеточных структур антител, способных связываться с белковыми маркерами, которые преимущественно экспрессируются в определенных тканях, типах клеток или субклеточных структурах.
Измененную экспрессию гена, ассоциированного с биохимическим путем передачи сигнала, также можно определять при помощи исследования изменения активности продукта гена по сравнению с контрольной клеткой. Анализ индуцированного определенным средством изменения активности белка, ассоциированного с биохимическим путем передачи сигнала, будет зависеть от биологической активности и/или исследуемого пути передачи сигнала. Например, если белок представляет собой киназу, изменение его способности фосфорилировать субстрат(субстраты) на последующих стадиях можно определять посредством ряда анализов, известных из уровня техники. Типичные анализы включают без ограничения иммуноблоттинг и иммунопреципитацию с использованием антител, таких как антитела к фосфотирозину, которые обеспечивают распознавание фосфорилированных белков. Кроме того, активность киназы можно выявлять при помощи высокопроизводительных хемилюминиспентных анализов, например, AlphaScreen™ (доступный от Perkin Elmer) и анализ eTag™ (Chan-Hui, et al. (2003) Clinical Immunology 111: 162-174).
Если белок, ассоциированный с биохимическим путем передачи сигнала, является частью сигнального каскада, который приводит к колебанию внутриклеточных условий рН, молекулы, чувствительные к рН, например, флуоресцентные рН-чувствительные красители, можно использовать в качестве репортерных молекул. В другом примере, если белок, ассоциированный с биохимическим путем передачи сигнала, представляет собой ионный канал, можно отслеживать колебания мембранного потенциала и/или внутриклеточной концентрации ионов. Ряд коммерческих наборов и высокопроизводительных устройств являются особенно подходящими для быстрого и надежного скрининга модуляторов ионных каналов. Типичные инструменты включают FLIPRTM (Molecular Devices, Inc.) и VIPR (Aurora Biosciences). Эти инструменты способны обеспечивать одновременное выявление реакций в более 1000 лунках с образцом в микропланшете и обеспечивать измерение в реальном времени и функциональные данные в течение секунды или даже миллисекунды.
При осуществлении на практике любых способов, раскрытых в данном документе, подходящий вектор можно вводить в клетку или эмбрион посредством одного или нескольких способов, известных из уровня техники, в том числе без ограничения микроинъекции, электропорации, сонопорации, баллистической трансфекции, трансфекции, опосредованной фосфатом кальция, трансфекции с помощью катионных липидных частиц, липосомной трансфекции, трансфекции при помощи дендримеров, трансфекции посредством теплового шока, трансфекции посредством нуклеофекции, магнитофекции, липофекции, импалефекции, оптической трансфекции, поглощения нуклеиновых кислот, стимулируемого проприетарным средством, и доставки при помощи липосом, иммунолипосом, виросом или искусственных вирионов. В некоторых способах вектор вводят в эмбрион посредством микроинъекции. Можно осуществлять микроинъекцию вектора или векторов в ядро или цитоплазму эмбриона. В некоторых способах вектор или векторы можно вводить в клетку посредством нуклеофекции.
Целевой полинуклеотид комплекса CRISPR может быть любым полинуклеотидом, эндогенным или экзогенным по отношению к эукариотической клетке. Например, целевой полинуклеотид может быть полинуклеотидом, находящимся в ядре эукариотической клетки. Целевой полинуклеотид может быть последовательностью, кодирующей продукт гена (к примеру, белок), или некодирующей последовательностью (к примеру, регуляторным полинуклеотидом или избыточной ДНК).
Примеры целевых полинуклеотидов включают последовательность, ассоциированную с биохимическими путями передачи сигнала, к примеру, ген или полинуклеотид ассоциированный с биохимическими путями передачи сигнала. Примеры целевых полинуклеотидов включают ген или полинуклеотид, ассоциированный с заболеванием. "Ассоциированный с заболеванием" ген или полинуклеотид означает любой ген или полинуклеотид, который обеспечивает продукты транскрипции или трансляции при отклоняющемся от нормы уровне или в отклоняющейся от нормы форме в клетках, полученных из пораженных заболеванием тканей, по сравнению с тканями или клетками контроля без заболевания. Это может быть ген, который начинает экспрессироваться при аномально высоком уровне; это может быть ген, который начинает экспрессироваться при аномально низком уровне, где измененная экспрессия коррелирует с появлением и/или развитием заболевания. Ассоциированный с заболеванием ген также означает ген, несущий мутацию(и) или генетическое изменение, который непосредственно ответственен или находится в неравновесном сцеплении с геном(ами), который(е) ответственен(ответственны) за этиологию заболевания. Транскрибируемые или транслируемые продукты могут быть известными или неизвестными и могут быть на нормальном уровне или на отклоняющемся от нормального уровне.
Целевым полинуклеотидом комплекса CRISPR может быть любой полинуклеотид, эндогенный или экзогенный по отношению к эукариотической клетке. Например, целевой полинуклеотид может быть полинуклеотидом, находящимся в ядре эукариотической клетки. Целевой полинуклеотид может быть последовательностью, кодирующей продукт гена (к примеру, белок), или некодирующей последовательностью (к примеру, регуляторным полинуклеотидом или избыточной ДНК). Не вдаваясь в теорию полагают, что целевая последовательность должна быть ассоциирована с РАМ (мотивом, смежным с протоспейсером); то есть короткой последовательностью, узнаваемой комплексом CRISPR. Определенные требования в отношении последовательности и длины РАМ различаются в зависимости от используемого фермента CRISPR, но РАМ, как правило, является последовательностью в 2-5 пары оснований, прилегающей к протоспейсеру (то есть целевой последовательности). Примеры последовательностей РАМ приведены в разделе "Примеры" ниже, и специалист в данной области сможет выявить дополнительные последовательности РАМ для применения с данным ферментом CRISPR.
Целевой полинуклеотид комплекса CRISPR может включать некоторое количество ассоциированных с заболеваниями генов и полинуклеотидов, а также генов и полинуклеотидов, ассоциированных с биохимическими путями передачи сигнала, которые перечислены в предварительных заявках на патент США 61/736527 и 61/748427 с общей ссылкой BI-2011/008/WSGR, номер в реестре 44063-701.101, и BI-2011/008/WSGR, номер в реестре 44063-701.102, соответственно, обе озаглавленные "СИСТЕМЫ, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ" (SYSTEMS METHODS AND COMPOSITIONS FOR SEQUENCE MANIPULATION), поданные 12 декабря 2012 г. и 2 января 2013 г., соответственно, причем содержание всех из которых включено в данный документ при помощи ссылки в полном их объеме.
Примеры целевых полинуклеотидов включают последовательность, ассоциированную с биохимическими путями передачи сигнала, к примеру, ген или полинуклеотид ассоциированный с биохимическими путями передачи сигнала. Примеры целевых полинуклеотидов включают ген или полинуклеотид, ассоциированный с заболеванием. "Ассоциированный с заболеванием" ген или полинуклеотид означает любой ген или полинуклеотид, который обеспечивает продукты транскрипции или трансляции при отклоняющемся от нормы уровне или в отклоняющейся от нормы форме в клетках, полученных из пораженных заболеванием тканей, по сравнению с тканями или клетками контроля без заболевания. Это может быть ген, который начинает экспрессироваться при аномально высоком уровне; это может быть ген, который начинает экспрессироваться при аномально низком уровне, где измененная экспрессия коррелирует с появлением и/или развитием заболевания. Ассоциированный с заболеванием ген также означает ген, несущий мутацию(и) или генетическое изменение, который непосредственно ответственен или находится в неравновесном сцеплении с геном(генами), который(е) ответственен(ответственны) за этиологию заболевания. Транскрибируемые или транслируемые продукты могут быть известными или неизвестными и могут быть на нормальном уровне или на отклоняющемся от нормального уровне.
Примеры ассоциированных с заболеваниями генов и полинуклеотидов перечислены в таблицах А и В. Конкретная информация в отношении заболеваний доступна от Института генетической медицины Маккьюсика-Натанса (McKusick-Nathans Institute of Genetic Medicine) при Университете Джонса Хопкинса (Johns Hopkins University) (Балтимор, Мэриленд) и Национального центра биотехнологической информации (National Center for Biotechnology Information), Национальной библиотеки медицины (National Library of Medicine) (Бетесда, Мэриленд), доступных во всемирной сети Интернет. Примеры ассоциированных с биохимическими путями передачи сигнала генов и полинуклеотидов перечислены в таблице С.
Мутации в этих генах и путях могут приводить к продуцированию несоответствующих белков или белков в несоответствующих количествах, которые воздействуют на функцию. Дополнительные примеры генов, заболеваний и белков, таким образом, включены при помощи ссылки из предварительной заявки на патент США 61/736527, поданной 12 декабря 2012 г. Такие гены, белки и пути могут относиться к целевому полинуклеотиду комплекса CRISPR.
















Варианты осуществления настоящего изобретения также относятся к способам и композициям, связанным с нокаутированием генов, амплифицированием генов и репарацией конкретных мутаций, ассоциированных с нестабильностью ДНК-повторов и неврологическими нарушениями (Robert D. Wells, Tetsuo Ashizawa, Genetic Instabilities and Neurological Diseases, Second Edition, Academic Press, Oct 13, 2011 - Medical). Как было обнаружено, определенные аспекты последовательностей тандемных повторов ответственны за более чем двадцать заболеваний человека (New insights into repeat instability: role of RNA⋅DNA hybrids. Mclvor EI, Polak U, Napierala M. RNA Biol. 2010 Sep-Oct; 7(5): 551-8). Система CRISPR-Cas может быть приспособлена для корректировки таких дефектов геномной нестабильности.
Дополнительный аспект настоящего изобретения относится к использованию системы CRISPR-Cas для корригирования дефектов в генах ЕМР2А и ЕМР2В, которые, как было обнаружено, ассоциированы с болезнью Лафора. Болезнь Лафора представляет собой аутосомно-рецессивное состояние, которое характеризуется прогрессирующей миоклонус-эпилепсией, которая может начинаться с эпилептических приступов в подростковом возрасте. Некоторые случаи заболевания могут быть вызваны мутациями в генах, которые уже были выявлены. Заболевание вызывает приступы, мышечные спазмы, затрудненную ходьбу, слабоумие и, в конечном итоге, смерть. В настоящее время не существует терапии, которая показала эффективность в отношении прогрессирования заболевания. На другие генетические расстройства, ассоциированные с эпилепсией, также можно целенаправленно воздействовать при помощи системы CRISPR-Cas, и лежащие в основе генетические механизмы дополнительно описаны в Genetics of Epilepsy and Genetic Epilepsies, edited by Giuliano Avanzini, Jeffrey L. Noebels, Mariani Foundation Paediatric Neurology: 20; 2009).
Способы согласно публикации патентного документа США №20110158957, закрепленного за Sangamo BioSciences, Inc., связанные с инактивацией генов Т-клеточного рецептора (TCR), также можно модифицировать для применения с системой CRISPR Cas согласно настоящему изобретению. В другом примере способы согласно публикации патентного документа США №20100311124, закрепленного за Sangamo BioSciences, Inc. и публикации патентного документа США №20110225664, закрепленного за Cellectis, оба из которых связаны с инактивацией экспрессии генов глутаминсинтетазы, также можно модифицировать для применения с системой CRISPR Cas согласно настоящему изобретению.
Некоторые дополнительные аспекты настоящего изобретения связаны с корригированном дефектов, ассоциированных с широким спектром наследственных заболеваний, которые дополнительно описаны на веб-сайте Национальных институтов здравоохранения (National Institutes of Health) в тематическом подразделе "Наследственные заболевания" ("Genetic Disorders") (веб-сайт по адресу health.nih.gov/topic/GeneticDisorders). Наследственные заболевания головного мозга могут включать без ограничения адренолейкодистрофию, агенезию мозолистого тела, синдром Айкарди, синдром Альперса, болезнь Альцгеймера, синдром Барта, болезнь Баттена, CADASIL, мозжечковую дегенерацию, болезнь Фабри, синдром Герстмана-Штраусслера-Шейнкера, болезнь Гентингтона и другие связанные с триплетным повтором нарушения, болезнь Лея, синдром Леша-Найхана, болезнь Менкеса, типы митохондриальной миопатии и кольпоцефалию по критериям NINDS. Такие заболевания дополнительно описаны на веб-сайте Национальных институтов здравоохранения (National Institutes of Health) в тематическом подразделе "Наследственные заболевания головного мозга" ("Genetic Brain Disorders").
В некоторых вариантах осуществления состоянием может быть неоплазия. В некоторых вариантах осуществления, где состоянием является неоплазия, гены, на которые целенаправленно воздействуют, являются любыми из перечисленных в таблице А (в данном случае PTEN и так далее). В некоторых вариантах осуществления состоянием. может быть возрастная дегенерация желтого пятна. В некоторых вариантах осуществления состоянием может быть шизофреническое нарушение. В некоторых вариантах, осуществления состоянием может быть связанное с тринуклеотидным повтором нарушение. В некоторых вариантах осуществления состоянием может быть синдром ломкой Х-хромосомы. В некоторых вариантах осуществления состоянием может быть связанное с секретазой нарушение. В некоторых вариантах осуществления состоянием может быть связанное с прионами нарушение. В некоторых вариантах осуществления состоянием может быть ALS. В некоторых вариантах осуществления состоянием может быть привыкание к наркотическим средствам. В некоторых вариантах осуществления состоянием может быть аутизм. В некоторых вариантах осуществления состоянием может быть болезнь Альцгеймера. В некоторых вариантах осуществления состоянием может быть воспаление. В некоторых вариантах осуществления состоянием может быть болезнь Паркинсона.
Например, в публикации заявки на патент США №20110023145 описывается применение нуклеаз с "цинковыми пальцами" для генетической модификации клеток, животных и белков, ассоциированных с расстройствами аутистического спектра (ASD). Расстройства аутистического спектра (ASD) представляют собой группу расстройств, характеризующихся качественным нарушением социального взаимодействия и коммуникации, а также ограниченными повторяющимися и стереотипными паттернами поведения, интересов и видов деятельности. Три расстройства, аутизм, синдром Аспергера (AS) и неспецифическое первазивное расстройство развития (PDD-NOS) относятся к одному и тому же расстройству с различными степенями тяжести, ассоциированными с умственной деятельностью и медицинскими состояниями. ASD преимущественно являются расстройствами, которые предопределены наследственными факторами, с наследуемостью приблизительно 90%.
В публикации патентного документа США №20110023145 предусматривается; редактирование любых хромосомных последовательностей, которые кодируют белки, ассоциированные с ASD, что можно применять по отношению к системе CRISPR Cas согласно настоящему изобретению. Белки, ассоциированные с ASD, как правило, выбирают исходя из экспериментально установленной ассоциации белка, ассоциированного с ASD, с возникновением или симптомом ASD. Например, скорость образования или концентрация в кровотоке белка, связанного с ASD, может быть повышенной или пониженной в популяции с ASD по сравнению с популяцией без ASD. Различия по уровням белка можно оценить при помощи протеомных методик, в том числе без ограничения вестерн-блоттинга, иммуногистохимического окрашивания, твердофазного иммуноферментного анализа (ELISA) и масс-спектрометрии. Альтернативно, белки, ассоциированные с ASD, можно идентифицировать путем получения профилей экспрессии генов для генов, кодирующих белки, при помощи методик геномного анализа, в том числе без ограничения микроматричного анализа ДНК, последовательного анализа экспрессии генов (SAGE) и количественной полимеразной цепной реакции в режиме реального времени (Q-PCR).
Неограничивающие примеры болезненных состояний или расстройств, которые могут быть ассоциированы с белками, ассоциированными с ASD, включают аутизм, синдром Аспергера (AS), неспецифическое первазивное расстройство развития (PDD-NOS), синдром Ретта, туберозный склероз, фенилкетонурию, синдром Смита-Лемли-Опитца и синдром ломкой Х-хромосомы. В качестве неограничивающего примера, белки, ассоциированные с ASD, включают, но без ограничения, следующие белки: АТР10С аминофосфолипид-транспортирующая АТФаза (АТР10С) MET МЕТ-рецепторная тирозинкиназа BZRAP1 MGLUR5 (GRM5) метаботропный глутаматный рецептор 5 (MGLUR5) CDH10 кадгерин-10 MGLUR6 (GRM6) метаботропный глутаматный рецептор 6 (MGLUR6) CDH9 кадгерин-9 NLGN1 нейролигин-1 CNTN4 контактин-4 NLGN2 нейролигин-2 CNTNAP2 контактин-ассоциированный подобный белку 2 (CNTNAP2) SEMA5A нейролигип-3 DHCR7 7-дегидрохолестеринредуктаза (DHCR7) NLGN4X нейролигин-4 Х-связанный NLGN4Y нейролигин-4 Y-связанный DOC2A двойной С2-подобный домен содержащий альфа-белок DPP6 дипептидил подобный аминопептидазе белок 6 NLGN5 нейролигин-5, EN2 белок 2 кодируемый гомеобоксом (EN2) NRCAM молекула адгезии нейронов (NRCAM), MDGA2, ассоциированный с умственной отсталостью, сцепленной с ломкой Х-хромосомой (MDGA2), NRXN1 нейрексин-1 FMR2 (AFF2) представитель 2 семейства AF4/FMR2 OR4M2 4М2 рецептор обонятельных луковиц FOXP2 белок Forkhead box P2 (FOXP2) OR4N4 4N4 рецептор обонятельных луковиц FXR1 связанный с умственной отсталостью, аутосомный гомолог 1 сцепленной с ломкой Х-хромосомой (FXR1), OXTR окситоциновый рецептор (OXTR), FXR2 связанный с умственной отсталостью, сцепленной с ломкой Х-хромосомой РАН, содержащий фенилаланин, аутосомная гидроксилаза (РАН), гомолог 2 (FXR2), GABRA1 рецепторная субъединица альфа-1 гамма-аминомасляной кислоты (GABRA1) PTEN фосфатаза и гомолог тензина (PTEN) GABRA5 GABAA (гамма-аминомасляной кислоты) рецепторная субъединица альфа 5 (GABRA5) PTPRZ1 тирозин протеинфосфатаза дзета рецепторного типа (PTPRZ1), GABRB1 рецепторная субъединица бета-1 гамма-аминомасляной кислоты (GABRB1) RELN рилин GABRB3 GABAA (гамма-аминомасляной кислоты) рецепторная субъединица бета 3 (GABRB3) RPL10 60S рибосомальный белок L10 GABRG1 гамма-аминомасляной кислоты рецепторная субъединица гамма-1 (GABRG1) SEMA5A семафорин-5А (SEMA5A) HIRIP3 HIRA-взаимодействующий белок 3 SEZ6L2 гомолог 2 подобный белку 6 связанному с приступами (мышь) НОХА1 белок гомеобокса Нох-А1 (НОХА1) SHANK3 SH3 и несколько повторяющихся доменов 3 анкирина (SHANK3) IL6 интерлейкин-6 SHBZRAP1 SH3 и несколько повторяющихся доменов 3 анкирина (SHBZRAP1) LAMB1 ламинин субъединица бета-1 (LAMB1), SLC6A4 серотонпновый транспортер (SERT) MAPK3 митоген-активируемая протеинкиназа 3 TAS2R1 вкусовой рецептор тип 2 представитель 1 TAS2R1 MAZ Мус-ассоциировапный белок с "цинковыми пальцами" TSC1 белок 1 связанный с туберозным склерозом MDGA2 гликозилфосфатидилинозитол-связанный белок 2 якорная форма 2 содержащий домен МАМ (MDGA2) TSC2 белок 2 связанный с туберозным склерозом МЕСР2 метил-CpG связывающий белок 2 (МЕСР2) лигаза Е3А UBE3A убиквитинового белка 2 (UBE3A) МЕСР2 метил-CpG связывающий белок 2 (МЕСР2) WNT2 сайт интеграции MMTV тип Wingless представитель 2 семейства (WNT2).
Идентичность белка, ассоциированного с ASD, редактирование хромосомной последовательности которого осуществляют, может и будет варьировать. В предпочтительных вариантах осуществления белки, ассоциированные с ASD, редактирование хромосомной последовательности которых осуществляют, могут представлять собой белок 1, ассоциированный с периферическим бензодиазепиновым рецептором (BZRAP1), кодируемый геном BZRAP1, белок 2, представитель семейства AF4/FMR2 (AFF2), кодируемый геном AFF2 (также называемый MFR2), белок-аутосомный гомолог 1, ассоциированный с умственной отсталостью, связанной с ломкой Х-хромосомой (FXR1), кодируемый геном FXR1, или белок-аутосомный гомолог 2, ассоциированный с умственной отсталостью, связанной с ломкой Х-хромосомой (FXR2), кодируемый геном FXR2, гликозилфосфатидилинозитол-связанный белок, содержащий домен МАМ, якорная форма 2 (MDGA2), кодируемый геном MDGA2, метил-CpG связывающий белок 2 (МЕСР2), кодируемый геном МЕСР2, метаботропный глутаматный рецептор 5 (MGLUR5), кодируемый геном MGLUR5-1 (также называемый GRM5), белок нейрексин 1, кодируемый геном NRXN1, или белок семафорин-5А (SEMA5A), кодируемый геном SEMA5A. В иллюстративном варианте осуществления генетически модифицированное животное представляет собой крысу, и редактируемые хромосомные последовательности, кодирующие белок, ассоциированный с ASD, перечислены ниже: BZRAP1 - бензодиазепиновый рецептор ХМ_002727789, (периферический) ассоциированный с ХМ_213427, белок 1 (BZRAP1) ХМ_002724533, ХМ_001081125, AFF2 (FMR2), представитель 2 семейства AF4/FMR2 ХМ_219832, (AFF2) ХМ_001054673, FXR1, аутосомный гомолог 1, ассоциированный с умственной отсталостью, связанной с ломкой Х-хромосомой, NM_001012179 (FXR1), FXR2, аутосомный гомолог 2, ассоциированный с умственной отсталостью, связанной с ломкой Х-хромосомой NM_001100647 (FXR2), MDGA2 гликозилфосфатидилинозитол-связанный белок, содержащий домен МАМ, якорная форма 2, NM_199269 (MDGA2), МЕСР2 метил-CpG связывающий белок 2 NM_022673 (МЕСР2), MGLUR5 - метаботропный глутаматный рецептор 5 NM_017012 (GRM5) (MGLUR5), NRXN1 - нейрексин-1 NM_021767, SEMA5A - семафорин-5А (SEMA5A) NM_001107659.
Иллюстративные животные или клетки могут содержать одну, две, три, четыре, пять, шесть, семь, восемь, или девять, или более инактивированных хромосомных последовательностей, кодирующих белок, ассоциированный с ASD, и нуль, одну, две, три, четыре, пять, шесть, семь, восемь, или девять, или более интегрированных в хромосому последовательностей, кодирующих белки, ассоциированные с ASD. Отредактированную или интегрированную хромосомную последовательность можно модифицировать так, чтобы она кодировала измененный белок, ассоциированный с ASD. Неограничивающие примеры мутаций в белках, ассоциированных с ASD, включают мутацию L18Q в нейрексине 1, где лейцин в положении 18 замещен глутамином, мутацию R451C в нейролигине 3, где аргинин в положении 451 замещен цистеином, мутацию R87W в нейролигине 4, где аргинин в положении 87 замещен триптофаном, и мутацию I425V в серотониновом транспортере, где изолейцин в положении 425 замещен валином. Ряд других мутаций и хромосомных перестроек в связанных с ASD хромосомных последовательностях ассоциирован с ASD, и они известны из уровня техники. См., например, Freitag et al. (2010) Eur. Child. Adolesc. Psychiatry 19: 169-178, и Bucan et al. (2009) PLoS Genetics 5: e1000536, раскрытие которых включено в данный документ посредством ссылки во всей их полноте.
Примеры белков, ассоциированных с болезнью Паркинсона, включают без ограничения α-синуклеин, DJ-1, LRRK2, PINK1, паркин, UCHL1, синфилин-1 и NURR1.
Примеры связанных с привыканием белков могут включать, например, АВАТ.
Примеры связанных с воспалением белков могут включать, например, моноцитарный хемоаттрактантный белок-1 (МСР1), кодируемый геном Ccr2, C-C рецептор хемокина 5 типа (CCR5), кодируемый геном Ccr5, IgG-рецептор IIB (FCGR2b, также называемый CD32), кодируемый геном Fcgr2b, или белок Fc-эпсилон-R1g (FCER1g), кодируемый геном Fcer1g.
Примеры ассоциированных с заболеваниями сердечно-сосудистой системы белков могут включать, например, IL1B (интерлейкин 1, бета), XDH (ксантиндегндрогеназу), ТР53 (опухолевый белок р53), PTGIS (простагландин-I2(простациклин)-синтазу), MB (миоглобин), IL4 (интерлейкин 4), ANGPT1 (ангиопоэтин 1), ABCG8 (АТФ-связывающую кассету, подсемейство G (WHITE), представитель 8) или CTSK (катепсин K).
Например, в публикации патентного документа США №20110023153 описывается применение нуклеаз с "цинковыми пальцами" для генетической модификации клеток, животных и белков, ассоциированных с болезнью Альцгеймера. В случае модификации клетки и животных можно дополнительно исследовать с применением известных способов для исследования воздействия целенаправленных мутаций на развитие и/или прогрессирование AD с использованием показателей, обычно применяемых в исследовании AD - таких как без ограничения обучение и память, тревожность, депрессия, привыкание и сенсомоторные функции, а также анализов, при помощи которых измеряют поведенческие, функциональные, патологические, метаболические и биохимические характеристики.
Настоящее раскрытие предусматривает редактирование любых хромосомных последовательностей, которые кодируют белки, ассоциированные с AD. Белки, связанные с AD, обычно выбирают исходя из экспериментально подтвержденной ассоциации белка, связанного с AD, с заболеванием AD. Например, скорость образования или концентрация в кровотоке белка, связанного с AD, может быть повышенной или пониженной в популяции с AD по сравнению с популяцией без AD. Различия по уровням белка можно оценить при помощи протеомных методик, в том числе без ограничения вестерн-блоттинга, иммуногистохимического окрашивания, твердофазного иммуноферментного анализа (ELISA) и масс-спектрометрии. В альтернативном случае, белки, связанные с AD, можно идентифицировать путем получения профилей генной экспрессии для генов, кодирующих белки, при помощи методик геномного анализа, в том числе без ограничения микроматричного анализа ДНК, последовательного анализа экспрессии генов (SAGE) и количественной полимеразной цепной реакции в режиме реального времени (Q-PCR).
Примеры ассоциированных с болезнью Альцгеймера белков могут включать, например, белок, представляющий собой рецептор липопротепнов очень низкой плотности (VLDLR), кодируемый геном VLDLR, убиквитин-подобный модификатор-активирующий фермент 1 (UBA1), кодируемый геном UBA1, или белок, являющийся каталитической субъединицей NEDD8-активирующего фермента E1 (UBE1C), кодируемый геном UBA3.
В качестве неограничивающего примера, белки, ассоциированные с AD, включают без ограничения белки, перечисленные ниже: кодируемый хромосомной последовательностью белок ALAS2 дельта-аминолевулинатсинтаза 2 (ALAS2) АВСА1 АТФ-связывающая кассета транспортер (АВСА1) АСЕ ангиотензин I-превращающий фермент (АСЕ) АРОЕ предшественник аполипопротеина Е (АРОЕ) АРР белок-предшественник амилоида (АРР), AQP1 белок аквапорин 1 (AQP1) BIN1 Мус box-зависимый взаимодействующий белок 1 или адаптерный белок-интегратор 1 (BIN1) BDNF нейротрофический фактор головного мозга (BDNF) BTNL8 белок 8 подобный бутирофилину (BTNL8) C10RF49 открытая рамка считывания 49 хромосомы 1 CDH4 кадгерин-4 CHRNB2 нейрональный ацетилхолиновый рецептор субъединица бета-2 CKLFSF2 CKLF-подобный белок 2 содержащий трансмембранный домен MARVEL (CKLFSF2) CLEC4E пектиновый домен С-типа семейство 4 представитель е (CLEC4E) CLU кластериновый белок (также известный как аполипопротеин J) CR1 эритропитарный рецептор комплемента 1 (CR1, также известный как CD35, рецептор C3b/C4b и рецептор иммунной адгезии) CR1L эритропитарный рецептор комплемента 1 (CR1L) CSF3R рецептор гранулоцитарного колониестимулирующего фактора 3 (CSF3R) CST3 цистатин С или цистатин 3 CYP2C цитохром Р450 2С DAPK1 ассоциированная с клеточной гибелью протеинкиназа 1 (DAPK1) ESR1 эстрогеновый рецептор 1 FCAR Fc-фрагмент рецептора для IgA (FCAR, также известный как CD89) FCGR3B Fc-фрагмент рецептора IIIb для IgG с низким сродством (FCGR3B или CD16b) FFA2 рецептор 2 свободных жирных кислот (FFA2) FGA фибриноген (фактор I), GAB2 GRB2-ассоциированный связывающий белок 2 (GAB2) GAB2 GRB2-ассоциированный связывающий белок 2 (GAB2) GALP галанин-подобный пептид GAPDHS глицеральдегид-3-фосфатдегидрогеназа, сперматогенные клетки (GAPDHS) GMPB GMBP HP гаптоглобин (HP) HTR7 5-гидрокситриптаминовый (серотониновый) рецептор 7 (связанный с аденилатциклазой) IDE фермент, разрушающий инсулин IF127 IF127, IFI6 интерферон альфа-индуцируемый белок 6 (IFI6) IFIT2 интерферон-индуцируемый белок с тетратрикопептидными повторами 2 (IFIT2) IL1RN антагонист рецептора интерлейкина-1 (IL-1RA) IL8RA рецептор интерлейкина 8, альфа (IL8RA или CD 181), IL8RB рецептор интерлейкина 8 бета (IL8RB) JAG1 белок Jagged I (JAG1) KCNJ15 входящий калиевый канал подсемейство J представитель 15 (KCNJ15) LRP6 белок 6 родственный рецептору липопротеинов низкой плотности (LRP6) МАРТ белок tau ассоциированный с микротрубочками (МАРТ) MARK4 киназа 4 регулирующая сродство к МАР/микротрубочкам (MARK4) MPHOSPH1 фосфобелок 1 М-фазы MTHFR 5,10-метилентетрагидрофолатредуктаза МХ2 интерферон-индупируемый GTP-связывающий белок Мх2 NBN нибрин, также известный как NBN NCSTN никастрин NIACR2 рецептор 2 ниацина (NIACR2, также известный как GPR109B) NMNAT3 никотинамиднуклеотидаденилилтрансфераза 3 NTM нейротримин (или HNT) ORM1 орозомукоид 1 (ORM1) или альфа-1-кислый гликопротеин 1 P2RY13 P2Y пуринорецептор 13 (P2RY13) PBEF1 никотинамидфосфорибозилтрансфераза (NAmPRTаза или Nampt), также известная как колониестимулирующий фактор 1 пре-В-клеток (PBEF1) или висфатин PCK1 фосфоенолпируваткарбоксикиназа PICALM фосфатидилинозит-связывающий белок вовлеченный в формирование клатриновых комплексов (PICALM) PLAU активатор плазминогена урокиназного типа (PLAU) PLXNC1 плексин C1 (PLXNC1) PRNP прионный белок PSEN1 белок пресенилин 1 (PSEN1) PSEN2 белок пресенилин 2 (PSEN2) PTPRA белок рецептора тирозинфосфатазы типа A (PTPRA) RALGPS2 Ral GEF с доменом РН и SH3 связывающим мотивом 2 (RALGPS2) RGSL2 белок 2, подобный регулятору передачи сигнала при помощи G-белка (RGSL2) SELENBP1 селенсвязывающий белок 1 (SELNBP1) SLC25A37 митоферрин-1 SORL1 родственный сортилину рецептор L (класс DLR) белок, содержащий повторы А (SORL1) TF трансферрин TFAM митохондриальный транскрипционный фактор A TNF фактор некроза опухоли TNFRSF10C суперсемейство рецепторов фактора некроза опухоли представитель 10С (TNFRSF10C) TNFSF10 суперсемейство рецепторов фактора некроза опухоли (TRAIL) представитель 10а (TNFSF10) UBA1 фермент 1, активирующий убнквитин-подобный модификатор (UBA1) UBA3 белок каталитической субъединицы NEDD8-активирующего фермента E1 (UBE1C), UBB белок убиквитин В (UBB) UBQLN1 убиквилин-1 UCHL1 белок эстеразы карбокси-конца убиквитина L1 (UCHL1) UCHL3 белок, относящийся к изоферменту L3 гидролазы карбокси-конца убиквитина (UCHL3) VLDLR белок рецептора липопротеинов очень низкой плотности (VLDLR).
В иллюстративных вариантах осуществления белки, ассоциированные с AD, редактирование хромосомной последовательности которых осуществляют, могут представлять собой белок рецептора липопротеинов очень низкой плотности (VLDLR), кодируемый геном VLDLR, фермент 1, активирующий убиквитин-подобный модификатор (UBA1), кодируемый геном UBA1, белок каталитической субъединицы NEDD8-активирующего фермента E1 (UBE1C), кодируемый геном UBA3, белок аквапорин 1 (AQP1), кодируемый геном AQP1, белок эстеразы карбокси-конца убиквитина L1 (UCHL1), кодируемый геном UCHL1, белок, относящийся к изоферменту L3 гидролазы карбокси-конца убиквитина (UCHL3), кодируемый геном UCHL3, белок убиквитин В (UBB), кодируемый геном UBB, белок tau, ассоциированный с микротрубочками (МАРТ), кодируемый геном МАРТ, белок рецептора тирозинфосфатазы типа A (PTPRA), кодируемый геном PTPRA, фосфатидилинозит-связывающий белок, вовлеченный в формирование клатриновых комплексов (PICALM), кодируемый геном PICALM, кластериновый белок (также известный как аполипопротеин J), кодируемый геном CLU, белок пресенилин 1, кодируемый геном PSEN1, белок пресенилин 2, кодируемый геном PSEN2, родственный сортилину рецептор L (класс DLR, NM_053519), белок, содержащий повторы A (SORL1), кодируемый геном SORL1, белок-предшественник амилоида (АРР), кодируемый геном АРР, предшественник аполипопротеина Е (АРОЕ), кодируемый геном АРОЕ, пли нейротрофический фактор головного мозга (BDNF), кодируемый геном BDNF. В иллюстративном варианте осуществления генетически модифицированное животное представляет собой крысу, и редактируемые хромосомные последовательности, кодирующие белок, ассоциированный с AD, являются следующими: АРР белок-предшественник амилоида (АРР) NM_019288 AQP1 белок аквапорин 1 (AQP1) NM_0127 BDNF нейротрофический фактор головного мозга NM_012513 CLU кластериновый белок (также известный как аполипопротеин J NM_053021) МАРТ белок tau ассоциированный с микротрубочками NM_017212 (МАРТ) PICALM фосфатидилинозит-связывающий белок вовлеченный в формирование клатриновых комплексов NM_053554 (PICALM) PSEN1 белок пресенилин 1 (PSEN1) NM_019163 PSEN2 белок пресенилин 2 (PSEN2) NM_031087 PTPRA белок рецептора тирозинфосфатазы типа A NM_012763 (PTPRA) SORL1 родственный сортилину рецептор L (класс DLR Nm_053519), белок, содержащий повторы А ХМ_00106550б (SORL1) ХМ_217115 UBA1 фермент 1 активирующий убиквитин-подобный модификатор NM_001014080 (UBA1) UBA3 белок каталитической субъединицы NEDD8-активирующего фермента E1 NM_057205 (UBE1C) UBB белок убиквитин В (UBB) NM_138895 UCHL1 белок эстеразы карбокси-конца убиквитина L1 NM_017237 (UCHL1) UCHL3 белок относящийся к изоферменту L3 гпдролазы карбокси-конца убиквитина NM_001110165 (UCHL3) VLDLR белок рецептора липопротеинов очень низкой плотности NM_013155 (VLDLR).
Животное или клетка может содержать 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 или более хромосомных последовательностей с нарушенной структурой, кодирующих белок, ассоциированный с AD, и нуль, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 или более интегрированных в хромосомы последовательностей, кодирующих белок, ассоциированный с AD.
Отредактированную или интегрированную хромосомную последовательность можно модифицировать так, чтобы она кодировала измененный белок, ассоциированный с AD. Рад мутаций в хромосомных последовательностях, связанных с AD, были ассоциированы с AD. Например, миссенс-мутация V7171 (т.е. валин в положении 717 заменен изолейцином) в АРР приводит к семейной форме AD. Несколько мутаций в белке пресенилин-1, например, H163R (т.е. гистидин в положении 163 заменен аргинином), А246Е (т.е. аланин в положении 246 заменен глутаматом), L286V (т.е. лейцин в положении 286 заменен валином) и C410Y (т.е. цистеин в положении 410 заменен тирозином) приводят к семейной форме болезни Альцгеймера типа 3. Мутации в белке пресенилип-2, например, N141 I (т.е. аспарагин в положении 141 заменен изолейцином), M239V (т.е. метионин в положении 239 заменен валином) и D439A (т.е. аспартат в положении 439 заменен аланином) приводят к семейной форме болезни Альцгеймера типа 4. Другие ассоциации генных вариантов генов, ассоциированных с AD, и заболевания известны из уровня техники. См., например, Waring et al. (2008) Arch. Neurol. 65: 329-334, раскрытие которой включено в данный документ посредством ссылки во всей их полноте.
Примеры белков, ассоциированных с расстройствами аутистического спектра, могут включать, например, белок 1, ассоциированный с периферическим бензодиазепиновым рецептором (BZRAP1), кодируемый геном BZRAP1, белок 2, представитель семейства AF4/FMR2 (AFF2), кодируемый геном AFF2 (также называемый MFR2), белок-аутосомный гомолог 1, ассоциированный с умственной отсталостью, связанной с ломкой Х-хромосомой (FXR1), кодируемый геном FXR1, или белок-аутосомный гомолог 2, ассоциированный с умственной отсталостью, связанной с ломкой Х-хромосомой (FXR2), кодируемый геном FXR2.
Примеры белков, ассоциированных с дегенерацией желтого пятна, могут включать, например, АТФ-связывающую кассету, белок 4 (АВСА4), представителя подсемейства А (АВС1), кодируемого геном ABCR, белок аполипротеин Е (АРОЕ), кодируемый геном АРОЕ, или белок-лиганд 2 хемокина (С-С мотив) (CCL2), кодируемый геном CCL2.
Примеры белков, ассоциированных с шизофренией, могут включать NRG1, ErbB4, CPLX1, ТРН1, ТРН2, NRXN1, GSK3A, BDNF, DISC1, GSK3B и их комбинации.
Примеры белков, вовлеченных в подавление опухоли, могут включать, например, ATM (мутированный, атаксия-телеангиэктазия), ATR (атаксия-телеангиэктазия и Rad3-родственный), EGFR (рецептор эпидермального фактора роста), ERBB2 (гомолог 2 онкогена v-erb-b2 вируса эритробластического лейкоза), ERBB3 (гомолог 3 онкогена v-erb-b2 вируса эритробластического лейкоза), ERBB4 (гомолог 4 онкогена v-erb-b2 вируса эритробластического лейкоза). Notch 1, Notch2, Notch 3 или Notch 4.
Примеры белков, ассоциированных с нарушением, связанным с активностью секретазы, могут включать, например, PSENEN (гомолог усилителя пресенилина 2 (С. elegans)), CTSB (катепсин В), PSEN1 (пресенилин 1), АРР (белок-предшественник бета-амилоида (А4)), АРН1 В (гомолог В дефектного белка 1 переднего отдела глотки (С. elegans)), PSEN2 (пресенилин 2 (болезнь Альцгеймера 4)) или ВАСЕ1 (фермент 1, расщепляющий АРР по бета-сайту).
Например, в публикации патентного документа США №20110023146 описывается применение нуклеаз с "цинковыми пальцами" для генетической модификации клеток, животных и белков, ассоциированных с нарушением, связанным с активностью секретазы. Секретазы необходимы для процессинга белков-предшественников с образованием их биологически активных форм. Дефекты различных компонентов секретазных путей связаны со многими нарушениями, в особенности, с характерным амилоидогенезом или амилоидными бляшками, например, болезнь Альцгеймера (AD).
Что касается нарушения, связанного с активностью секретазы, белки, ассоциированные с этими нарушениями, представляют собой разнородную группу белков, которые оказывают влияние на восприимчивость ко многим нарушениям, наличие нарушения, тяжесть нарушения или любую их комбинацию. Настоящее раскрытие предусматривает редактирование любых хромосомных последовательностей, которые кодируют белки, ассоциированные с нарушением, связанным с активностью секретазы. Белки, ассоциированные с нарушением, связанным с активностью секретазы, как правило, выбирают исходя из экспериментально установленной ассоциации белков, родственных секретазе, с развитием нарушения, связанного с активностью секретазы. Например, скорость образования или концентрация в кровотоке белка, ассоциированного с нарушением, связанным с активностью секретазы, может быть повышенной или пониженной в популяции с нарушением, связанным с активностью секретазы, по сравнению с популяцией без нарушения, связанного с активностью секретазы. Различия по уровням белка можно оценить при помощи протеомных методик, в том числе без ограничения вестерн-блоттинга, иммуногистохимического окрашивания, твердофазного иммуноферментного анализа (ELISA) и масс-спектрометрии. Альтернативно, белок, ассоциированный с нарушением, связанным с активностью секретазы, можно идентифицировать путем получения профилей экспрессии генов для генов, кодирующих белки, при помощи методик геномного анализа, в том числе без ограничения микроматричного анализа ДНК, последовательного анализа экспрессии генов (SAGE) и количественной полимеразной цепной реакции в режиме реального времени (Q-PCR).
В качестве неограничивающего примера, белки, ассоциированные с нарушением, связанным с активностью секретазы, включают PSENEN (гомолог усилителя пресенилина 2 (С. elegans)), CTSB (катепсин В), PSEN1 (пресенилин 1), АРР (белок-предшественник бета-амилоида (А4)), АРН1 В (гомолог В дефектного белка 1 переднего отдела глотки (С. elegans)), PSEN2 (пресенилин 2 (болезнь Альцгеймера 4 типа)), ВАСЕ1 (фермент 1, расщепляющий АРР по бета-сайту), ITM2B (интегральный мембранный белок 2 В), CTSD (катепсин D), NOTCH 1 (гомолог Notch 1, ассоциированный с транслокацией (Drosophila)), TNF (фактор некроза опухоли (суперсемейство TNF, представитель 2)), INS (инсулин), DYT10 (белок 10, связанный с дистонией), ADAM17 (ADAM, металлопептидазный домен 17), АРОЕ (аполипопротеин Е), АСЕ (ангиотензин 1-превращающий фермент (пептидилдипептидаза А) 1), STN (статин), ТР53 (опухолевый белок р53), IL6 (интерлейкин 6 (интерферон, бета 2)), NGFR (рецептор фактора роста нервов (суперсемейство TNFR, представитель 16)), IL1B (интерлейкин 1, бета), ACHE (ацетилхолинэстераза (группа крови Yt)), CTNNB1 (катенин (кадгерин-ассоциированный белок), бета 1, 88 кДа), IGF1 (инсулиноподобный фактор роста 1 (соматомеднн С)), IFNG (интерферон, гамма), NRG1 (нейрегулин 1), CASP3 (каспаза 3, связанная с апоптозом цистеиновая пептидаза), MAPK1 (митоген-активируемая протеинкиназа 1), CDH1 (кадгерин 1, тип 1, Е-кадгерин (эпителиальные клетки)), АРВВ1 (белок, связывающий предшественник бета-амилоида (А4), семейство В, представитель 1 (Fe65)), HMGCR (редуктаза 3-гидрокси-3-метилглутарилкофермента A), CREB1 (белок 1, связывающий сАМР-чувствительный элемент), PTGS2 (простагландин-эндопероксидсинтаза 2 (простагландин G/H синтаза и циклооксигеназа)), HES1 (белок 1 hairy and enhancer of split (Drosophila)), CAT (каталаза), TGFB1 (трансформирующий фактор роста, бета 1), ENO2 (енолаза 2 (гамма, нейрональная)), ERBB4 (гомолог 4 онкогена v-erb-a вируса эритробластического лейкоза (птичий)), TRAPPC10 (комплекс 10 транспортного белка частиц), МАОВ (моноаминоксидаза В), NGF (фактор роста нервов (бета-полипептид)), ММР12 (матриксная металлопептидаза 12 (эластаза макрофагов)), JAG1 (белок Jagged 1 (синдром Алажиля)), CD40LG (лиганд CD40), PPARG (гамма-рецептор, активируемый пролифератором пероксисом), FGF2 (фактор 2 роста фиброблаетов (основный)), IL3 (интерлейкин 3 (колониестимулирующий фактор, множественый)), LRP1 (белок 1, родственный рецептору липопротеинов низкой плотности), NOTCH4 (гомолог Notch 4 (Drosophila)), MAPK8 (митоген-активируемая протеинкиназа 8), PREP (пролилэндопептидаза), NOTCH3 (гомолог Notch 3 (Drosophila)), PRNP (прионный белок), CTSG (катепсин G), EGF (эпидермальный фактор роста (бета-урогастрон)), REN (ренин), CD44 (молекула CD44 (система групп крови Indian)), SELP (селектин Р (гранулярный мембранный белок 140 кДа, антиген CD62)), GHR (рецептор гормона роста), ADCYAP1 (полипептид 1, активирующий аденилатциклазу (гипофиз)), INSR (инсулиновый рецептор), GFAP (глиальный фибриллярный кислый белок), ММР3 (матриксная металлопептидаза 3 (стромелизин 1, прожелатиназа)), MAPK10 (митоген-активируемая протеинкиназа 10), SP1 (транскрипционный фактор Sp1), MYC (гомолог онкогена v-myc вируса миелопитоматоза (птичий)), CTSE (катепсин Е), PPARA (альфа-рецептор, активируемый пролифератором пероксисом), JUN (онкоген jun), TIMP1 (ингибитор 1 металлопептидазы TIMP), IL5 (интерлейкин 5 (колониестимулирующий фактор, эозинофилы)), ILIA (интерлейкип 1, альфа), ММР9 (матриксная металлопептидаза 9 (желатиназа В, 92 кДа желатиназа, 92 кДа коллагеназа IV типа)), HTR4 (5-гидрокситриптаминовый (серотониновый) рецептор 4), HSPG2 (гепарансульфат-протеогликан 2), KRAS (гомолог онкогена v-Ki-ras2 вируса саркомы Кирстена крысы), CYCS (цитохром с, соматические клетки), SMG1 (гомолог SMG1, киназа, родственная фосфатидилинозитол-3-киназе (С. elegans)), IL1R1 (рецептор интерлейкина 1, I типа), PROK1 (прокинетицин 1), MAPK3 (митоген-активируемая протеинкиназа 3), NTRK1 (нейротрофическая тирозинкиназа, рецептор, тип 1), IL13 (интерлейкин 13), ММЕ (мембранная металлоэндопептидаза), TKT (транскетолаза), CXCR2 (рецептор 2 хемокина (мотив С-Х-С)), IGF1R (рецептор 1 инсулиноподобного фактора роста), RARA (рецептор ретиноевой кислоты, альфа), CREBBP (CREB-связывающий белок), PTGS1 (простагландин-эндопероксидсинтаза 1 (простагландин G/H синтаза и циклооксигеназа)), GALT (галактозо-1-фосфатуридилтрансфераза), CHRM1 (холинергический рецептор, мускариновый 1), ATXN1 (атаксин 1), PAWR (PRKC, связанный с апоптозом, WT1, регулятор), NOTCH2 (гомолог Notch 2 (Drosophila)), M6PR (рецептор маннозо-6-фосфата (катион-зависимый)), CYP46A1 (цитохром Р450, семейство 46, подсемейство А, полипептид 1), CSNK1 D (казеинкиназа 1, дельта), MAPK14 (митоген-активируемая протеинкиназа 14), PRG2 (протеогликан 2, костный мозг (активатор клеток натуральных киллеров, главный основный белок гранул эозинофилов)), PRKCA (протеинкиназа С, альфа), L1 САМ (молекула клеточной адгезии L1), CD40 (молекула CD40, представитель 5 суперсемейства рецепторов TNF), NR1I2 (ядерный рецептор, подсемейство 1, I группа, представитель 2), JAG2 (белок Jagged 2), CTNND1 (катенин (кадгерин-ассоциированный белок), дельта 1), CDH2 (кадгерин 2, тип 1, N-кадгерин (нейрональный)), СМА1 (химаза 1, тучная клетка), SORT1 (сортилин 1), DLK1 (гомолог Delta-подобного белка 1 (Drosophila)), THEM4 (представитель 4 суперсемейства тиоэстераз), JUP (плакоглобин адгезионных контактов), CD46 (молекула CD46, регуляторный белок комплемента), CCL11 (лиганд 11 хемокина (мотив С-С)), CAV3 (кавеолин 3), RNASE3 (рибонуклеаза, семейство РНКазы А, 3 (катионный белок эозинофилов)), HSPA8 (70 кДа белок 8 теплового шока), CASP9 (каспаза 9, связанная с апоптозом цистеинпептидаза), CYP3A4 (цитохром Р450, семейство 3, подсемейство А, полипептид 4), CCR3 (рецептор 3 хемокина (мотив С-С)), TFAP2A (транскрипционный фактор АР-2 альфа (активирующий энхансер-связывающий белок 2 альфа)), SCP2 (стерол-переносящий белок 2), CDK4 (циклин-зависимая киназа 4), HIF1A (фактор 1, индуцируемый гипоксией, альфа-субъединица (транскрипционный фактор с основным доменом типа спираль-петля-спираль)), TCF7L2 (белок 2, подобный транскрипционному фактору 7 (специфический по отношению к Т-клеткам, HMG-бокс)), IL1R2 (рецептор интерлейкина 1, II типа), B3GALTL (белок, подобный бета-1,3-галактозилтрансферазе), MDM2 (гомолог Mdm2 белка, связывающего р53 (мышь)), RELA (гомолог А онкогена v-rel вируса ретикулоэндотелиоза (птичий)), CASP7 (каспаза 7, связанная с апоптозом цистеинпептидаза), IDE (фермент, разрушающий инсулин), FABP4 (белок 4, связывающий жирные кислоты, адипопитарный), CASK (кальций/кальмодулин-зависимая сериновая протеинкиназа (семейство MAGUK)), ADCYAP1R1 (рецептор I типа полипептида 1, активирующего аденилатпиклазу (гипофиз)), ATF4 (активирующий транскрипционный фактор 4 (tax-чувствительный энхансерный элемент В67)), PDGFA (полипетид тромбопитарпого фактора роста альфа), С21 или f33 (открытая рамка считывания 33 хромосомы 21), SCG5 (секретогранин V (белок 7 В2)), RNF123 (белок 123 с доменом ring), NFKB1 (ядерный фактор 1 энхансера гена полипептида каппа легкой цепи иммуноглобулина в В-клетках), ERBB2 (гомолог 2 онкогена v-erb-b2 вируса эритробластического лейкоза, гомолог онкогена, полученный из нейро/глиобластомы (птичий)), CAV1 (кавеолин 1, кавеолярный белок, 22 кДа), ММР7 (матриксная металлопептидаза 7 (матрилизин, маточный)), TGFA (трансформирующий фактор роста, альфа), RXRA (рецептор ретиноида X, альфа), STX1A (синтаксин 1А (головной мозг)), PSMC4 (26S субъединица протеасомы (просома, макропаин), АТФаза, 4), P2RY2 (пуринергический рецептор P2Y, связанный с G-белком, 2), TNFRSF21 (суперсемейство рецепторов фактора некроза опухоли, представитель 21), DLG1 (discs, гомолог 1 large (Drosophila)), NUMBL (белок, подобный гомологу numb (Drosophila)), SPN (сиалофорин), PLSCR1 (фосфолипидскрамблаза 1), UBQLN2 (убиквилин 2), UBQLN1 (убиквилин 1), PCSK7 (пробелок-конвертаза субтилизин/кексин типа 7), SPON1 (спондин 1, белок внеклеточного матрикса), SILV (гомолог silver (мышь)), QPCT (глутаминилпептидциклотрансфераза), HESS (белок 5 hairy and enhancer of split (Drosophila)), GCC1 (GRIP и белок 1, содержащий домен в виде суперспирали) и любую их комбинацию.
Генетически модифицированное животное или клетка могут содержать 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более хромосомных последовательностей с нарушенной структурой, кодирующих белок, ассоциированный с нарушением, связанным с активностью секретазы, и нуль, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более интегрированных в хромосомы последовательностей, кодирующих белок с нарушенной структурой, ассоциированный с нарушением, связанным с активностью секретазы.
Примеры белков, ассоциированных с амиотрофическим латеральным склерозом, могут включать, например, SOD1 (супероксиддисмутазу 1), ALS2 (белок 2, ассоциированный с амиотрофическим латеральным склерозом), FUS (РНК-связывающий белок FUS (fused in sarcoma)), TARDBP (TAR-ДНК связывающий белок), VAGFA (фактор роста эндотелия сосудов A), VAGFB (фактор роста эндотелия сосудов В) и VAGFC (фактор роста эндотелия сосудов С) и любую их комбинацию.
Например, в публикации заявки на патент США №20110023144 описывается применение нуклеаз с "цинковыми пальцами" для генетической модификации клеток, животных и белков, ассоциированных с заболеванием, связанным с амиотрофическим латеральным склерозом (ALS). ALS характеризуется постепенной прогрессирующей дегенерацией определенных нервных клеток в коре головного мозга, стволе головного мозга и спинном мозге, связанных с произвольными движениями.
Что касается нарушения, связанного с двигательными нейронами, белки, ассоциированные с этими нарушениями, представляют собой разнородную группу белков, которые оказывают влияние на восприимчивость к развитию нарушения, связанного с двигательными нейронами, наличие нарушения, связанного с двигательными нейронами, тяжесть нарушения, связанного с двигательными нейронами, или любую их комбинацию. Настоящее раскрытие предусматривает редактирование любых хромосомных последовательностей, которые кодируют белки, ассоциированные с заболеванием, связанным с ALS, специфическим нарушением, связанным с двигательными нейронами. Белки, ассоциированные с ALS, как правило, выбирают исходя из экспериментально установленной взаимосвязи белков, связанных с ALS, с нарушением по типу ALS. Например, скорость образования или концентрация в кровотоке белка, ассоциированного с ALS, может быть повышенной или пониженной в популяции с ALS по сравнению с популяцией без ALS. Различия по уровням белка можно оценить при помощи протеомных методик, в том числе без ограничения вестерн-блоттинга, иммуногистохимического окрашивания, твердофазного иммуноферментного анализа (ELISA) и масс-спектрометрии. Альтернативно, белки, ассоциированные с ALS, можно идентифицировать путем получения профилей экспрессии генов для генов, кодирующих белки, при помощи методик геномного анализа, в том числе без ограничения микроматричного анализа ДНК, последовательного анализа экспрессии генов (SAGE) и количественной полимеразной цепной реакции в режиме реального времени (Q-PCR).
В качестве неограничивающего примера, белки, ассоциированные с ALS, включают, без ограничения, следующие белки: SOD1 супероксиддисмутаза 1, ALS3 белок 3, связанный с амиотрофическим латеральным склерозом SETX сенатаксин ALS5 белок 5, связанный с амиотрофическим латеральным склерозом FUS РНК-связывающий белок FUS (fused in sarcoma) ALS7 белок 7, связанный с амиотрофическим латеральным склерозом ALS2 белок 2, связанный с амиотрофическим латеральным склерозом DPP6 дипептидилпептидаза 6, NEFH тяжелый полипептид нейрофиламента PTGS1 простагландин-эндопероксидсинтазы 1 SLC1A2 семейство 1 переносчиков растворенных веществ (глутаматный транспортер глиальных клеток с высоким сродством), представитель 2 TNFRSF10B фактор некроза опухоли, суперсемейство рецепторов представитель 10b PRPH периферии HSP90AA1 90 кДа белок теплового шока альфа (цитозольный), класс А представитель 1, GRIA2 глутаматный рецептор, ионотропный, АМРА 2, IFNG интерферон, гамма S100B S100 кальций-связывающий белок В FGF2 фактор 2 роста фибробластов АОХ1 альдегидоксидаза 1 CS цитратсинтаза TARDBP TAR ДНК-связывающий белок, TXN тиоредоксин, RAPH1 Ras-ассоциированный белок, (RaIGDS/AF-б) и киназа 5 с доменами 1, характеризующимися гомологией с плекстрином, MAP3K5 митоген-активируемая протеинкиназа, NBEAL1 белок 1, подобный нейробичину, GPX1 глутатионпероксидаза 1, ICA1L подобный 1,69 кДа-аутоантигену островковых клеток, RAC1 ras-родственный белок, подобный субстрату 1 ботулинического С3 токсина, МАРТ белок tau, ассоциированный с микротрубочками, ITPR2 рецептор инозитол-1,4,5-трифосфата, тип 2, ALS2CR4 кандидатный участок 4 хромосомы для белка 2, связанного с амиотрофическим латеральным склерозом (ювенильным), GLS глутаминаза, ALS2CR8 кандидатный участок 8 хромосомы для белка 2, связанного с амиотрофическим латеральным склерозом (ювенильным), CNTFR рецептор для цилиарного нейротрофического фактора, ALS2CR11 кандидатный участок 11 хромосомы для белка 2, связанного с амиотрофическим латеральным склерозом (ювенильным), FOLH1 фолатгидролаза 1, FAM117B семейство белков со сходством последовательности с белком 117, представитель В, Р4НВ пролил-4-гидроксилаза, полипептид бета, CNTF цилиарный нейротрофический фактор, SQSTM1 секвестосома 1, STRADB STE20-родственная киназа, бета-адаптерная, Nин-3-монооксигAIP семейство NLR, связанный с апоптозом ингибиторный белок, YWHAQ тирозеназа/триптофан-5-монооксигеназа активирующий белок, полипептид тета, SLC33A1 семейство 33 переносчиков растворенных веществ (ацетил-СоА транспортеры), представитель 1, TRAK2 транспортный белок, кинезин-связывающий 2, FIG. 4 FIG. гомолог 4, содержащий домен фосфатазы липидов SAC1, NIF3L1 NIF3 NGG1-взаимодействующий фактор 3, подобный 1, INA интернексин, нейрональный промежуточный филаментный белок, альфа, PARD3B белок par-3 (partitioning defective 3), гомолога В, СОХ8А цитохром с оксидаза, субъединица VIIIA, CDK15 циклин-зависимая киназа, HECW1 НЕСТ белок, содержащий домен С2 и WW 15, Е3 лигаза 1 убиквитинового белка, NOS1 синтаза 1 оксида азота, MET протоонкоген met, SOD2 митохондриальная супероксиддисмутаза 2, HSPB1 27 кДа белок 1 теплового шока, NEFL легкий полипептид нейрофиламента, CTSB катепсин В, ANG ангиогенин, рибонуклеаза ANG ангиогенин, рибонуклеаза, РНКаза семейства 5, HSPA8 70 кДа белок теплового шока 8, VAPB VAMP (ассоциированный с везикулами мембранный белок)-ассоциированные белки В и С, ESR1 эстрогеновый рецептор 1, SNCA синуклеин, альфа, HGF фактор роста гепатоцитов, CAT каталаза, АСТВ актин, бета, NEFM среднего размера полипептид нейрофиламента, ТН тирозингидроксилаза BCL2 белок 2 В-клеток связанный с CLL/лимфомой FAS Fas (суперсемейство рецепторов TNF, представитель 6) CASP3 каспаза 3, связанная с апоптозом цистеинпептидаза, CLU кластерин, SMN1 белок, связанный с выживанием двигательных нейронов, G6PD глюкозо-6-фосфатдегидрогеназа 1 ВАХ BCL2-ассоциированный белок Х HSF1 транскрипционный фактор 1 белка теплового шока, RNF19A белок 19А с доменом ring, JUN онкоген jun, ALS2CR12 кандидатный участок 12 хромосомы для белка 2, связанного с амиотрофическим латеральным склерозом (ювенильным), HSPA5 70 кДа белок 5 теплового шока, MAPK14 митоген-активируемая протеинкиназа 14, IL10 интерлейкин 10, АРЕХ1 тАРЕХ-нуклеаза (мультифункциональный фермент репарации ДНК), TXNRD1 тиоредоксинредуктаза 1, NOS2 индуцируемая синтаза 2 оксида азота, TIMP1 TIMP ингибитор 1 металлопептпдазы, CASP9 каспаза 9, связанная с апоптозом цистеинпептидаза, XIAP Х-сцепленный ингибитор апоптоза, GLG1 глнкопротеин 1 комплекса Гольджи, ЕРО эритропоэтин, VEGFA фактор роста эндотелия сосудов A, ELN эластин, GDNF нейротрофический фактор, полученный из глиальных клеток, NFE2L2 белок 2 подобный ядерному фактору (эритроидному), SLC6A3 представитель 3 семейства 6 переносчиков растворенных веществ (транспортер нейротрансмиттеров, допаминовый), HSPA4 70 кДа белок 4 теплового шока, АРОЕ аполипопротеин Е, PSMB8 субъединица протеасомы (просома, макропаин), тип бета, 8, DCTN1 динактин 1, ТТМР3 TIMP ингибитор 3 металлопептидазы KIFAP3 кинезин-ассоциированный белок 3 SLC1A1 представитель 1 семейства 1 переносчиков растворенных веществ (глутаматный транспортер нейронов/эпителиальных клеток с высоким сродством, система Xag) SMN2 центромерный белок 2 выживания двигательных нейронов CCNC циклин С МРР4 пальмитоилированный мембранный белок 4 STUB1 белок 1, гомологичный STIP1 и содержащий U-box ALS2 белок-предшественник амилоида бета (А4) PRDX6 пероксиредоксин 6 SYP синаптофизин CABIN1 кальциневрин-связывающий белок 1 CASP1 каспаза 1, связанная с апоптозом цистеинпептидаза GART фосфорибозилглицинамидформилтрансфераза, фосфорибозилглицинамидсинтетаза, фосфорибозиламиноимидазолсинтетаза, CDK5 циклин-зависимая киназа 5 ATXN3 атаксии 3 RTN4 ретикулон 4 C1QB компонент комплемента 1 субкомпонент q цепь В VEGFC рецептор фактора роста нервов НТТ хантингтин PARK7 белок 7 связанный с болезнью Паркинсона XDH ксантиндегидрогеназа GFAP глиальный фибриллярный кислый белок МАР2 белок 2 ассоциированный с микротрубочками CYCS цитохром с, соматические клетки FCGR3B Fc-фрагмент рецептора IIIb для IgG с низким сродством, CCS медь-содержащий шаперон супероксиддисмутазы UBL5 белок 5 подобный убиквитину ММР9 матриксная металлопептидаза 9 SLC18A3 представитель 3 семейства 18 переносчиков растворенных веществ (везикулярный, ацетилхолиновый) TRPM7 катионный канал транзиентного рецепторного потенциала подсемейство М представитель 7 HSPB2 27 кДа белок 2 теплового шока АКТ1 гомолог 1 онкогена v-akt вируса тимомы мышей DERL1 представитель 1 семейства белков с Der1-подобным доменом CCL2 лиганд 2 хемокина (С-С мотив) NGRN неугрин, ассоциированный с ростом аксонов GSR глутатионредуктаза ТРРР3 представитель 3 семейства белков способствующих полимеризации тубулина APAF1 фактор 1 активирующий апоптическую пептидазу BTBD10 белок 10 содержащий домен ВТВ (POZ) GLUD1 глутаматдегидрогеназа 1 CXCR4 рецептор 4 хемокина (С-Х-С мотив) SLC1A3 представитель 3 семейства 1 переносчиков растворенных веществ (глутаматный транспортер глиальных клеток с высоким сродством) FLT1 тирозинкиназа 1 родственная fins PON1 параоксоназа 1 AR андрогеповый рецептор LIF ингибиторный фактор связанный с лейкозом ERBB3 гомолог 3 онкогена v-erb-b2 вируса эритробластического лейкоза, LGALS1 лектин галактозид-связывающий растворимый белок 1, CD44 молекула CD44, ТР53 опухолевый белок р53 TLR3 толл-подобный рецептор 3 GRIA1 глутаматный рецептор, ионотропный, АМРА 1, GAPDH глицеральдегид-3-фосфатдегидрогеназа, GRIK1 глутаматный рецептор, ионотропный, каинатный белок 1, DES десмин, CHAT холинацетилтрансфераза, FLT4 тирозинкиназа 4, родственная fins, CHMP2B белок 2В, модифицирующий хроматин, BAG1 BCL2-ассоциированный атаноген, МТ3 металлотионеин 3, CHRNA4 холинергический рецептор никотиновый альфа 4 GSS глутатионсинтетаза BAK1 BCL2-антагонист/киллер 1 KDR рецептор вставочного домена киназы (рецептор тирозинкиназы III типа) GSTP1 глутатион-S-трансфераза пи 1 OGG1 8-оксогуанин-ДНК-гликозилаза IL6 интерлейкин 6 (интерферон, бета 2).
Животное или клетка могут содержать 1, 2, 3, 4, 5, 6, 7, 8, 9 или более хромосомных последовательностей с нарушенной структурой, кодирующих белок, ассоциированный с ALS, и нуль, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более интегрированных в хромосомы последовательностей, кодирующих белок, ассоциированный с ALS. Предпочтительные белки, ассоциированные с ALS, включают SOD1 (супероксиддисмутазу 1), ALS2 (белок 2, ассоциированный с амиотрофическим латеральным склерозом), FUS (РНК-связывающий белок FUS), TARDBP (TAR-ДНК связывающий белок), VAGFA (фактор роста эндотелия сосудов A), VAGFB (фактор роста эндотелия сосудов В) и VAGFC (фактор роста эндотелия сосудов С) и любую их комбинацию.
Примеры белков, ассоциированных с прионными болезнями, могут включать SOD1 (супероксиддисмутазу 1), ALS2 (белок 2, ассоциированный с амиотрофическим латеральным склерозом), FUS (РНК-связывающий белок FUS), TARDBP (TAR-ДНК связывающий белок), VAGFA (фактор роста эндотелия сосудов А), VAGFB (фактор роста эндотелия сосудов В) и VAGFC (фактор роста эндотелия сосудов С) и любую их комбинацию.
Примеры белков, связанных с нейродегенеративными состояниями при прионных болезнях, могут включать, например, А2М (альфа-2-макроглобулин), AATF (фактор транскрипции, противодействующий апоптозу), АСРР (простатоспецифическую кислую фосфатазу), АСТА2 (альфа-актин 2 гладкой мускулатуры аорты), ADAM22 (ADAM, металлопептидазный домен), ADORA3 (аденозиновый рецептор типа A3) или ADRA1D (альфа-1D адренергический рецептор для альфа-1D адренорецептора).
Примеры белков, ассоциированных с иммунодефицитом, могут включать, например, А2М [альфа-2-макроглобулин]; AANAT [арилалкилампн-N-ацетилтрансферазу]; АВСА1 [АТФ-связывающую кассету, подсемейство А (АВС1), представитель 1]; АВСА2 [АТФ-связывающую кассету, подсемейство А (АВС1), представитель 2] или АВСА3 [АТФ-связывающую кассету, подсемейство А (АВС1), представитель 3].
Примеры белков, ассоциированных с нарушениями, связанными с тринуклеотидными повторами, включают, например, AR (андрогеновый рецептор), FMR1 (белок 1, ассоциированный с умственной отсталостью, связанной с ломкой X-хромосомой), НТТ (хантингтин) или DMPK (протеинкиназу, ассоциированную с мышечной дистрофией), FXN (фратаксин), ATXN2 (атаксин 2).
Примеры белков, ассоциированных с нарушениями передачи нервных импульсов, включают, например, SST (соматостатин), NOS1 (синтазу оксида азота 1 (нейрональную)), ADRA2A (альфа-2А-адренергический рецептор), ADRA2C (альфа-2С-адренергический рецептор), TACR1 (тахикининовый рецептор 1) или HTR2c (5-гидрокситриптаминовый (серотониновый) рецептор 2С).
Примеры последовательностей, ассоциированных с неврологическим развитием, включают, например, А2ВР1 [атаксии 2-связывающий белок 1], AADAT [аминоадипатаминотрансферазу], AANAT [арилалкиламин-N-ацетилтрансферазу], АВАТ [4-аминобутиратаминотрансферазу], АВСА1 [АТФ-связывающую кассету, подсемейство А (АВС1), представитель 1] или АВСА13 [АТФ-связывающую кассету, подсемейство А (АВС1), представитель 13].
Дополнительные примеры предпочтительных состояний, которые подлежат лечению с помощью данной системы, включают те, которые могут быть выбраны из синдрома Айкарди-Гутьереса; болезни Александера; синдрома Аллана-Херндона-Дадли; связанных с геном POLG нарушений; альфа-маннозидоза (II и III тип); синдрома Альстрема; синдрома Ангельмана; атаксии-телеангиэктазии; нейронного восковидного липофусциноза; бета-талассемии; двусторонней атрофии зрительного нерва и (инфантильной) атрофии зрительного нерва 1 типа; ретинобластомы (двусторонней); болезни Канавана; церебро-окуло-фацио-скелетного синдрома 1 [COFS1]; церебротендинального ксантоматоза; синдрома Корнелии де Ланге; связанных с геном МАРТ нарушений; наследственных прионных болезней; синдрома Драве; семейной болезни Альцгеймера с ранним началом; атаксии Фридрейха [FRDA]; сипдрома Фринса; фукозидоза; врожденной мышечной дистрофии Фукуямы; галактосиалидоза; болезни Гоше; органической апидемии; гемофагоцитарного лимфогистиоцитоза; синдрома прогерии Гетчинсона-Гилфорда; муколипидоза II; инфантильной болезни накопления свободной сиаловой кислоты; ассоциированной с геном PLA2G6 нейродегенерации; синдрома Джервелла-Ланге-Нильсена; узелкового врожденного буллезного эпидермолиза; болезни Гентингтона; болезни Краббе (инфантильной); ассоциированного с митохондриальной ДНК синдрома Ли и NARP; синдрома Леша-Найхана; ассоциированной с геном LIS1 лиссэнцефалии; синдрома Лоу; болезни "кленового сиропа"; синдрома дупликации МЕСР2; связанных с геном АТР7А нарушений обмена меди; связанной с геном LAMA2 мышечной дистрофии; недостаточности арилсульфатазы А; мукополисахаридоза I, II или III типов; связанных с биогенезом пероксисом нарушений, спектра заболеваний по типу синдрома Цельвегера; нарушений по типу нейродегенерации с накоплением железа в головном мозге; недостаточности кислой сфингомиелиназы; болезни Ниманна-Пика типа С; глициновой энцефалопатии; связанных с геном ARX нарушений; нарушений орнитинового цикла; связанного с геном COL1A1/2 несовершенного остеогенеза; синдромов удаления митохондриальной ДНК; связанных с геном PLP1 нарушений; синдрома Перри; синдрома Фелана-МакДермида; болезни накопления гликогена II типа (болезни Помпе) (инфантильной); связанных с геном МАРТ нарушений; связанных с геном МЕСР2 нарушений; эпифизарной точечной хондродисплазии 1 типа костей верхних конечностей или бедренной кости; синдрома Робертса; болезни Сандхоффа; болезни Шиндлера - 1 типа; аденозиндезаминазной недостаточности; синдрома Смита-Лемли-Опитца; спинальной мышечной атрофии; спинально-церебеллярной атаксии с возникновением в младенческом возрасте; недостаточности гексозаминидазы; танатофорной дисплазии 1 типа; связанных с геном коллагена VI типа нарушений; синдрома Ашера I типа; врожденной мышечной дистрофии; синдрома Вольфа-Хиршхорна; недостаточности лизосомной кислой липазы и пигментной ксеродермы.
Как будет понятно, предусматривается, что настоящую систему можно использовать для целенаправленного воздействия на любую представляющую интерес полинуклеотидную последовательность. Некоторые состояния или заболевания, которые можно эффективно лечить с использованием настоящей системы, включены в таблицы выше, и примеры известных на данный момент генов, ассоциированных с такими состояниями, также представлены в них. Тем не менее, гены, приведенные в качестве примеров, не являются исчерпывающими.
Например, "StCas9 дикого типа" относится к Cas9 дикого типа из S. thermophilus, при этом последовательность его белка представлена в базе данных SwissProt под номером доступа G3ECR1. Аналогично, S. pyogenes Cas9 включен в SwissProt под номером доступа Q99ZW2.
Возможность применения системы CRISPR-Cas для осуществления эффективного и экономичного редактирования генов и манипуляции с ними обеспечит возможность быстрого выбора и сравнения одиночных и мультиплексных генетических манипуляций для трансформирования таких геномов в отношении увеличенной продукции и улучшенных признаков. В связи с этим ссылаются на патенты США и публикации: патент США №6603061 - опосредованный агробактериями способ трансформации растений (Agrobacterium-Mediated Plant Transformation Method); патент США №7868149 -последовательности генома растений и их применение (Plant Genome Sequences and Uses Thereof) и US 2009/0100536 - трансгенные растения с улучшенными агротехническими признаками (Transgenic Plants with Enhanced Agronomic Traits), все содержание и раскрытие каждого из которых включено в данный документ при помощи ссылки в полном объеме. При осуществлении на практике настоящего изобретения содержание и раскрытие Morrell et al "Crop genomics:advances and applications" Nat Rev Genet. 2011 Dec 29; 13(2): 85-96 также включено в данный документ при помощи ссылки в полном объеме.
Примеры
Следующие примеры приведены с целью иллюстрации различных вариантов осуществления настоящего изобретения, и не предполагается, что они ограничивают настоящее изобретение каким-либо образом. Данные примеры совместно со способами, описанными в данном документе, в настоящее время отражают предпочтительные варианты осуществления, являются иллюстративными и не предназначены для ограничения объема настоящего изобретения. Изменения в данном документе и другие применения, которые охватываются сущностью настоящего изобретения, как определено объемом формулы изобретения, будут очевидны специалистам в данной области.
Пример 1. Активность комплекса CMSPR в ядре эукариотической клетки
Примером системы CRISPR типа II является локус CRISPR типа II из Streptococcus pyogenes SF370, который содержит кластер из 4 генов Cas9, Cas1, Cas2 и Csn1, а также 2 некодирующих элемента РНК, tracrRNA и характерный массив повторяющихся последовательностей (прямых повторов), чередующихся с короткими фрагментами неповторяющихся последовательностей (спейсерами, приблизительно 30 п.о. каждый). В данной системе целенаправленный двухнитевой разрыв (DSB) целевой ДНК образуется в ходе четырех последовательных стадий (фигура 2А). Во-первых, две некодирующие РНК, массив pre-crRNA и tracrRNA транскрибируются с локуса CRISPR. Во-вторых, tracrRNA гибридизуется с прямыми повторами в pre-crRNA, которая затем процессируется в зрелые crRNA, содержащие индивидуальные спейсерные последовательности. В-третьих, комплекс зрелая crRNA: tracrRNA направляет Cas9 к ДНК-мишени, состоящей из протоспейсера и соответствующей РАМ, посредством образования гетеродуплекса между спейсерным участком crRNA и протоспейсерной ДНК. И наконец, Cas9 опосредует расщепление целевой ДНК выше РАМ с образованием DSB внутри протоспейсера (фигура 2А). В данном примере описывается иллюстративный способ для приспособления этой РНК-программируемой нуклеазной системы к управлению активностью комплекса CRISPR в ядрах эукариотических клеток.
Клеточная культура и трансфекция
Клеточную линию почки человеческого эмбриона (HEK), HEK 293FT (Life Technologies), поддерживали в среде Игла, модифицированной Дульбекко (DMEM), которую дополняли 10% фетальной бычьей сыворотки (HyClone), 2 мМ GlutaMAX (Life Technologies), 100 ед./мл пенициллина и 100 мкг/мл стрептомицина, инкубируя при 37°С, 5% CO2. Клеточную линию нейробластомы мышей (N2A) (АТСС) поддерживали в DMEM, дополненной 5% фетальной бычьей сывороткой (HyClone), 2 мМ GlutaMAX (Life Technologies), 100 ед/мл пенициллина и 100 мкг/мл стрептомицина, при 37°С, 5% СО2.
Клетки HEK 293FT или N2A высевали в 24-луночные планшеты (Corning) за один день до трансфекции с плотностью 200000 клеток на лунку. Клетки трансфицировали с использованием Lipofectamine 2000 (Life Technologies), следуя рекомендованному производителем протоколу. Для каждой лунки 24-луночного планшета использовали в общей сложности 800 нг плазмид.
Анализ с помощью Surveyor и анализ с помощью секвенирования на предмет наличия модификации генома
Клетки HEK 293FT или N2A трансфицировали плазмидной ДНК, как описано выше. После трансфекции клетки инкубировали при 37°С в течение 72 часов перед экстракцией геномной ДНК. Геномную ДНК экстрагировали с помощью набора QuickExtract DNA extraction kit (Epicentre), следуя протоколу производителя. Вкратце, клетки ресуспендировали в растворе QuickExtract и инкубировали при 65°С в течение 15 минут и при 98°С в течение 10 минут. Экстрагированную геномную ДНК подвергали немедленной обработке или хранили при -20°С.
Геномный участок, окружающий целевой сайт CRISPR, для каждого гена подвергали ПЦР амплификации и продукты очищали с использованием колонки QiaQuick Spin Column (Qiagen), следуя протоколу производителя. В общей сложности 400 нг очищенных ПЦР-продуктов смешивали с 2 мкл 10Х ПЦР-буфера для Taq-полимеразы (Enzymatics) и водой сверхвысокой чистоты до конечного объема 20 мкл и подвергали процессу повторной гибридизации для обеспечения образования гетеродуплекса: 95°С в течение 10 мин, линейное снижение температуры с 95°С до 85°С со скоростью - 2°С/с, с 85°С до 25°С со скоростью 0,25°С/с и с выдерживанием при 25°С в течение 1 минуты. После повторной гибридизации продукты обрабатывали нуклеазой Surveyor и энхансером S Surveyor (Transgenomics), следуя рекомендованному производетелем протоколу, и анализировали в 4-20% полиакриламидных гелях Novex TBE (Life Technologies). Гели окрашивали ДНК-красителем SYBR Gold (Life Technologies) в течении 30 минут и визуализировали с помощью системы для визуализации гелей Gel Doc gel imaging (Bio-rad). Количественное определение основывалось на значениях относительной интенсивности окрашивания полос в качестве единицы измерения фракции расщепленной ДНК. На фигуре 7 представлена схематическая иллюстрация данного анализа с помощью Surveyor.
Анализ полиморфизма длины рестрикционных фрагментов для выявления гомологичной рекомбинации.
Клетки HEK 293FT и N2A трансфицировали плазмидной ДНК и инкубировали при 37°С в течение 72 часов перед экстракцией геномной ДНК, как описано выше. Целевой геномный участок подвергали ПЦР амплификации с использованием праймеров за пределами гомологичных плечей матрицы для гомологичной рекомбинации (HR). ПЦР-продукты разделяли в 1% агарозном геле и экстрагировали с помощью набора MinElute GelExtraction Kit (Qiagen). Очищенные продукты расщепляли с помощью HindIII (Fermentas) и анализировали в 6% полиакриламидном геле Novex TBE (Life Technologies).
Прогнозирование и анализ вторичной структуры РНК
Прогнозирование вторичной структуры РНК осуществляли с использованием доступного в режиме онлайн веб-сервера RNAfold, разработанного в Институте теоретической химии при Венском университете, с использованием алгоритма прогнозирования структуры на основе центроидного метода (см., например, A.R. Gruber et al., 2008, Cell 106(1): 23-24; и PA Carr and GM Church, 2009, Nature Biotechnology 27(12): 1151-62).
Очистка РНК
Клетки HEK 293FT поддерживали и трансфицировали, как указано выше. Клетки собирали путем трипсинизации с последующим промыванием в фосфатно-солевом буферном растворе (PBS). Общую клеточную РНК экстрагировали с помощью реагента TRI (Sigma), следуя протоколу производителя. Общую выделенную РНК количественно определяли с использованием Naonodrop (Thermo Scientific) и нормализовали по отношению к такой же концентрации.
Анализ экспрессии crRNA и tracrRNA в клетках млекопитающих с помощью нозерн-блоттинга
РНК смешивали с равными объемами 2Х загрузочного буфера (Ambion), нагревали до 95°С в течение 5 мил, охлаждали на льду в течение 1 мин, а затем загружали в 8% денатурирующие полиакриламидные гели (SequaGel, National Diagnostics) после предварительного прогона геля в течение по меньшей мере 30 минут. Образцы подвергали электрофорезу в течение 1,5 часа при предельной мощности 40 Вт. После этого РНК переносили на мембрану Hybond N+ (GE Healthcare) при силе тока 300 мА в устройстве полусухого переноса (Bio-rad) при комнатной температуре в течение 1,5 часа. РНК пришивали к мембране с использованием кнопки "автоматического сшивания" на приборе для автоматического сшивания с помощью ультрафиолета Stratagene UV Crosslinker the Stratalinker (Stratagene). Мембрану подвергали предварительной гибридизации в буфере для гибридизации ULTRAhyb-Oligo Hybridization Buffer (Ambion) в течение 30 мин с вращением при 42°С, а затем добавляли зонды и проводили гибридизацию в течение ночи. Зонды заказывали у IDT и метили [гамма-32P] АТР (Perkin Elmer) с использованием полинуклеотид-киназы Т4 (New England Biolabs). Мембрану промывали один раз предварительно подогретым (42°С) 2×SSC, 0,5% SDS в течение 1 мин с последующими двумя промываниями по 30 минут при 42°С. Мембрану экспонировали на люминесцентном экране в течение одного часа или в течение ночи при комнатной температуре, а затем сканировали с использованием устройства для формирования изображения на люминесцентном фосфорном покрытии (Typhoon).
Конструирование и оценка бактериальной системы CRISPR
Элементы локуса CRISPR, в том числе tracrRNA, Cas9 и лидерную последовательность, подвергали ПЦР амплификации из геномной ДНК Streptococcus pyogenes SF370 с фланкирующими гомологичными плечами для сборки по методу Гибсона. Два сайта BsaI IIS типа вводили между двумя прямыми повторами для облегчения простой вставки спейсеров (фигура 8). ПЦР продукты клонировали в расщепленный с помощью EcoRV pACYC184 ниже промотора tet с использованием мастер-микса Гибсону Gibson Assembly Master Mix (NEB). Другие эндогенные элементы системы CRISPR пропускали за исключением последних 50 п.о. Csn2. Олигонуклеотиды (Integrated DNA Technology), кодирующие спейсеры с комплементарными выступающими частями клонировали в расщепленный с помощью BsaI вектор pDC000 (NEB), а затем лигировали с использованием лигазы Т7 (Enzymatics) с получением плазмид pCRISPR. Контрольные плазмиды, содержащие спейсеры с экспрессией РАМ.
В клетках млекопитающих (экспрессионные конструкции, проиллюстрированные на фигуре 6А, с функциональностью, которая определена по результатам анализа с использованием Surveyor, показанным на фигуре 6В). Сайты начала транскрипции помечены как +1, а также показаны терминатор транскрипции и последовательность, являющаяся зондом для нозерн-блоттинга. Экспрессию подвергнутой процессингу tracrRNA также подтверждали с помощью нозерн-блотгинга. На фигуре 6С показаны результаты анализа с помощью нозерн-блоттинга общей РНК, экстрагированной из 293FT клеток, трансфицированных экспрессионными конструкциями U6, несущими длинную или короткую tracrRNA, а также SpCas9 и DR-EMX1(1)-DR. Левая и правая секции получены с клетками 293FT, трансфицированными без или с SpRNase III, соответственно. U6 являются показателем для контроля загрузки при блоттинге с зондом, нацеленным на малую ядерную РНК (snRNA) U6 человека. Трансфекция экспрессионной конструкции с короткой tracrRNA приводит к избыточным уровням процессированной формы tracrRNA (~75 п.о.). Очень низкие количества длинной tracrRNA выявляли при нозерн-блоттинге.
Для стимуляции точной инициации транскрипции промотор U6 на основе РНК-полимеразы III выбирали для управления экспрессией tracrRNA (фигура 2С). Подобным образом, конструкцию на основе промотора U6 разработали для экспрессии массива pre-crRNA, состоящей из одного спейсера, фланкированного двумя прямыми повторами (DR, также включены в термин "парные tracr-последовательности"; фигура 2С). Исходный спейсер был сконструирован для целенаправленного воздействия на целевой сайт размеров 33 пары оснований (п.о.) (протоспейсер из 30 п.о., а также последовательность мотива CRISPR (РАМ) из 3 п.о., соответствующую мотиву узнавания NGG у Cas9) в локусе ЕМХ1 человека (фигура 2С), ключевом гене в развитии коры головного мозга.
Клетки HEK 293FT трансфицировали комбинациями компонентов CRISPR для того, чтобы определить, возможно ли при гетерологичной экспрессии системы CRISPR (SpCas9, SpRNase III, tracrRNA и pre-crRNA) в клетках млекопитающих достичь целенаправленного расщепления хромосом млекопитающего. Поскольку DSB в ядрах млекопитающих подвергаются частичной репарации с помощью пути негомологичного соединения концов (NHEJ), который приводит к образованию вставок/делений, анализ с помощью Surveyor использовали для выявления возможной активности в отношении расщепления в целевом локусе ЕМХ1 (фигура 7) (см., например, Guschin et al., 2010, Methods Mol Biol 649: 247). Совместная трансфекция всех четырех компонентов CRISPR была способна индуцировать расщепление в протоспейсере на уровне до 5,0% (см. фигуру 2D). Совместная трансфекция всех компонентов CRISPR, за исключением SpRNase III, также индуцировала образование вставок/делений в протоспейсере на уровне до 4,7%, указывая на то, что могут существовать эндогенные РНКазы млекопитающих, которые способны помогать созреванию crRNA, такие как, например, родственные ферменты Dicer и Drosha. Удаление любого из трех остальных компонентов ликвидировало активность системы CRISPR в отношении расщепления генома (фигура 2D). Секвенирование по Сэнгеру ампликонов, содержащих целевой локус подтверждало активность в отношении расщепления: на 43 подвергшихся секвенированию клонов было обнаружено 5 аллелей с мутацией (11,6%). В подобных экспериментах с использованием ряда направляющих последовательностей процентные значения содержания вставок/делений составляют вплоть до 29% (см. фигуры 3-6, 10 и 11). Эти результаты определяют трехкомпонентную систему для эффективной опосредованной CRISPR модификации генома в клетках млекопитающих. Для оптимизации эффективности расщепления авторы настоящего изобретения также определяли, влияют ли различные изоформы tracrRNA на эффективность расщепления, и обнаружили, что в этой иллюстративной системе только короткая (89 п.о.) форма транскрипта была способна опосредовать расщепление локуса генома ЕМХ1 человека (фигура 6В).
На фигуре 12 представлен дополнительный анализ процессинга crRNA в клетках млекопитающих с помощью нозерн-блоттинга. На фигуре 12А проиллютрировано схематическое изображение вектора экспрессии для одного спейсера, фланкированного двумя прямыми повторами (DR-EMX1(1)-DR). Спейсер из 30 п.о., целенаправленно воздействующий на протоспейсер 1 локуса ЕМХ1 человека (см. фигура 6), и последовательности прямых повторов показаны в последовательности внизу фигуры 12А. Линия указывает на участок, обратно комплементарную последовательность которого использовали для создания зондов для нозерн-блоттинга для выявления crRNA ЕМХ1(1). На фигуре 12В показаны результаты анализа с помощью нозерн-блоттинга общей РНК, экстрагированной из клеток 293FT, которые трансфицировали экспрессионными конструкциями U6, несущими DR-EMX1(1)-DR. Левая и правая секции получены с клетками 293FT, трансфицированными без или с SpRNase III, соответственно. DR-EMX1(1)-DR подвергался процессингу в зрелые crRNA только в присутствии SpCas9 и короткой tracrRNA и не зависел от присутствия SpRNase III. Зрелая crRNA, выявляемая в общей РНК трансфицированных 293FT, имела длину ~33 п.о. и была короче зрелой crRNA из S. pyogenes длиной 39-42 п.о. Данные результаты демонстрируют, что систему CRISPR можно пересадить в эукариотические клетки и перепрограммировать для облегчения расщепления эндогенных целевых полинуклеотидов млекопитающих.
На фигуре 2 проиллюстрирована бактериальная система CRISPR, описанная в этом примере. На фигуре 2А проиллюстрировано схематическое изображение локуса 1 CRISPR из Streptococcus pyogenes SF370 и предполагаемый механизм опосредованного CRISPR расщепления ДНК с помощью этой системы. Зрелая crRNA, подвергшаяся процессингу из массива прямых повторов-спейсеров, направляет Cas9 к мишеням в геноме, состоящим из комплементарных протоспейсеров и протоспейсерного смежного мотива (РАМ). При спаривании оснований мишень-спейсер Cas9 опосредует двухнитевой разрыв в целевой ДНК. На фигуре 2В проиллюстрировано конструирование Cas9 (SpCas9) S. pyogenes и RNase III (SpRNase III) с сигналами ядерной локализации (NLS) для обеспечения возможности импорта в ядро млекопитающих. На фигуре 2С проиллюстрирована экспрессия SpCas9 и SpRNase III у млекопитающих, управляемая конститутивным промотором EF1a, и массива tracrRNA и, pre-crRNA (DR-спейсер-DR), управляемая промотором U6 РНК-полимеразы 3 для стимуляции точной инициации и терминации транскрипции. Протоспейсер из локуса ЕМХ1 человека с удовлетворительной последовательностью РАМ использовали в качестве спейсера в массиве pre-crRNA. На фигуре 2D проиллюстрирован анализ с помощью нуклеазы Surveyor в отношении опосредованных SpCas9 минорных вставок и делеций. SpCas9 экспрессировался с и без SpRNase III, tracrRNA и массива pre-crRNA, несущего целевой спейсер для ЕМХ1. На фигуре 2Е проиллюстрировано схематическое изображение спаривания оснований между целевым локусом и целенаправленно воздействующей на ЕМХ1 crRNA, а также иллюстративная хроматограмма, показывающая микроделецию, расположенную смежно с сайтом расщепления SpCas9. На фигуре 2F проиллюстрированы мутированные аллели, идентифицированные в результате анализа секвенирования 43 клональных ампликонов, показывающие разнообразие микровставок и микроделеций. Штрихами указаны удаленные основания, а невыровненные или несовпадающие основания указывают на вставки или мутации. Масштабная метка = 10 мкм.
Для дальнейшего упрощения трехкомпонентной системы адаптировали химерную crRNA-tracrRNA гибридную структуру, в которой зрелую crRNA (содержащую направляющую последовательность) можно слить с активной частью tracrRNA через структуру по типу стебель-петля для имитации естественного дуплекса crRNA: tracrRNA. Для повышения эффективности совместной доставки создавали бицистронный вектор экспрессии для управления коэкспрессией химерной РНК и SpCas9 в трансфицированных клетках. Параллельно, бицистронные векторы использовали для экспрессии pre-crRNA (DR-направляющая последовательность-DR) с SpCas9, чтобы индуцировать процессинг в crRNA с участием отдельно экспрессируемой tracrRNA (сравнение на фигуре 11В: верхняя часть и нижняя часть). На фигуре 8 представлены схематические иллюстрации бицистронных векторов экспрессии для массива pre-crRNA (фигура 8А) или химерных crRNA (представлена короткой линией ниже сайта встраивания направляющей последовательности и выше промотора EF1α на фигуре 8В) с hSpCas9, показывающие положение различных элементов и точки встраивания направляющей последовательности. Расширенная последовательность вокруг положения сайта встраивания направляющей последовательности на фигуре 8В также показывает часть последовательности DR (GTTTAGAGCTA) и часть последовательность tracrRNA (TAGCAAGTTAAAATAAGGCTAGTCCGTTTTT). Направляющие последовательности можно встроить между сайтами BbsI с использованием гибридизованных олигонуклеотидов. Структуры последовательностей для олигонуклеотидов показаны ниже схематических иллюстраций на фигуре 8 с указанными подходящими адаптерами лигирования. WPRE представляет собой посттранскрипционный регуляторный элемент вируса гепатита сурков. Эффективность опосредованного химерной РНК расщепления исследовали путем целенаправленного воздействия на тот же локус ЕМХ1, описанный выше. С использованием как анализа с помощью Surveyor, так и секвенирования ампликонов по Сэнгеру авторы настоящего изобретения подтвердили, что химерная струкутура РНК облегчает расщепление локуса ЕМХ1 человека со степенью модификации примерно 4,7% (фигура 3).
Генерализованность опосредованного CRISPR расщепления в эукариотических клетках исследовали путем целенаправленного воздействия на дополнительные локусы генома как в человеческих, так и мышиных клетках путем конструирования химерных РНК, целенаправленно воздействующих на множественные сайты в ЕМХ1 и PVALB человека, а также на локусы Th мыши. На фигуре 13 проиллюстрирован выбор некоторых дополнительных служащих в качестве мишени протоспейсеров в локусах PVALB человека (фигура 13А) и Th мыши (фигура 13В). Приведены схематические изображения локусов генов и положения трех протоспейсеров в последнем экзоне каждого из них. Подчеркнутые последовательности включают 30 п.о. последовательности протоспейсера и 3 п.о. на 3' конце, соответствующую последовательностям РАМ. Протоспейсеры на смысловой и антисмысловой нитях указаны выше и ниже последовательностей ДНК, соответственно. Степень модификации 6,3% и 0,75% достигали для локусов PVALB человека и Th мыши, соответственно, демонстрируя широкую применимость системы CRISPR при модификации различных локусов у нескольких организмов (фигура 5). Хотя при использовании химерных конструкций расщепление выявлялось только с одним из трех спейсеров для каждого локуса, все целевые последовательности расщеплялись с эффективностью получения вставок/делений, достигающей 27% при использовании схемы с коэкспрессируемой pre-crRNA (фигуры 6 и 13).
На фигуре 11 представлена дополнительная иллюстрация того, что SpCas9 можно перепрограммировать для целенаправленного воздействия на множественные локусы генома в клетках млекопитающих. На фигуре 11А представлено схематическое изображение локуса ЕМХ1 человека, показывающего положение пяти протоспейсеров, указанных подчеркнутыми последовательностями. На фигуре 11В представлено схематическое изображение комплекса pre-crRNA/trcrRNA, показывающего гибридизацию между участком прямого повтора в pre-crRNA и tracrRNA (вверху), и схематическое изображение химерной структуры РНК, содержащей направляющую последовательность из 20 п.о., и парную tracr-последовательность, и tracr-последовательность, состоящие из неполного прямого повтора и последовательностей tracrRNA, гибридизованных в шпильковую структуру (внизу). Результаты анализа с помощью a Surveyor со сравнением эффективности опосредованного Cas9 расщепления в пяти протоспейсерах в локусе ЕМХ1 человека проиллюстрированы на фигуре 11С. Целенаправленное воздействие на каждый протоспейсер осуществляется либо с использованием подвергнутого процессингу комплекса pre-crRNA/tracrRNA (crRNA), либо с использованием химерной РНК (chiRNA).
Поскольку вторичная структура РНК может быть критически значимой для межмолекулярных взаимодействий, алгоритм прогнозирования структуры на основе минимальной свободной энергии и ансамбля взвешенных структур по Больцману использовали для сравнения прогнозируемых вторичных структур всех направляющих последовательностей, используемых в экспериментах с целенаправленным воздействием на геном (см., например, Gruber et al., 2008, Nucleic Acids Research, 36: W70). Анализ выявил, что в большинстве случаев эффективные направляющие последовательности в контексте химерной crRNA, по сути, не содержали мотивов вторичной структуры, тогда как неэффективные направляющие последовательности с большей вероятностью образуют внутренние вторичные структуры, которые могут препятствовать спариванию оснований с ДНК целевого протоспейсера. Таким образом, возможно, что вариабельность во вторичной структуре спейсера может оказывать воздействие на эффективность опосредованной CRISPR интерференции при использовании химерной crRNA.
Структуры дополнительных векторов для SpCas9 показаны на фигуре 22, на которой проиллюстрированы отдельные векторы экспрессии, включающие промотор U6, сцепленный с сайтом встраивания для направляющего олигонуклеотида, и промотор Cbh, сцепленный с кодирующей последовательностью SpCas9. Вектор, показанный на фигуре 22b, включает кодирующую последовательность tracrRNA, сцепленную с промотором H1.
При анализе у бактерий все спейсеры облегчали эффективную интерференцию CRISPR (фигура 3С). Эти результаты указывают, на то, что могут существовать дополнительные факторы, воздействующие на эффективность активности CRISPR в клетках млекопитающих.
Для исследования специфичности опосредованного CRISPR расщепления эффект однонуклеотидных мутаций в направляющей последовательности в отношении расщепления протоспейсера в геноме млекопитающих анализировали с использованием ряда целенаправленно воздействующих на ЕМХ1 химерных crRNA с единичными точковыми мутациями (фигура 3А). На фигуре 3В проиллюстрированы результаты анализа с помощью нуклеазы Surveyor со сравнением эффективности расщепления Cas9 при спаривании с различными мутантными химерными РНК. Несовпадение одного основания в участке вплоть до 12 п.о. с 5' в РАМ, по сути, прекращало расщепление генома SpCas9, тогда как спейсеры с мутациями в положениях, расположенных в более отдаленных положениях выше относительно хода транскрипции сохраняли активность в отношении исходной мишени-протоспейсера (фигура 3В). В дополнение к РАМ, SpCas9 характеризуется специфичностью в отношении одного основания в последних 12 п.о. спейсера. Кроме того, CRISPR способен опосредовать расщепление генома столь же эффективно, как и пара нуклеаз TALE (TALEN), целенаправленно воздействующих на тот же протоспейсер ЕМХ1. На фигуре 3С представлено схематическое изображение, показывающее структуру TALEN, целенаправленно воздействующих на ЕМХ1, и на фигуре 3D показано сравнение эффективности TALEN и Cas9 (n=3) при разгонке в геле продуктов, полученных в результате анализа с помощью Surveyor.
Установив набор компонентов для достижения опосредованного CRISPR редактирования генов в клетках млекопитающих посредством подверженного ошибкам механизма NHEJ, исследовали способность CRISPR к стимуляции гомологичной рекомбинации (HR), высокоточный путь репарации генов для создания точных редакционных изменений в геноме. SpCas9 дикого типа способен опосредовать сайт-специфические DSB, которые могут репарироваться как с помощью NHEJ, так и HR. Кроме того, замену аспартат-на-аланин (D10A) в каталитическом домене RuvC I в SpCas9 конструировали для превращения нуклеазы в никазу (SpCas9n; проиллюстрировано на фигуре 4А) (см., например, Sapranausaks et al., 2011, Nucleic Acids Research, 39: 9275; Gasiunas et al., 2012, Proc. Natl. Acad. Sci. USA, 109: E2579) так, чтобы надрезанная геномная ДНК подвергалась высокоточной репарации с использованием гомологичной рекомбинации (HDR). Анализ с помощью Surveyor подтвердил, что SpCas9n не создает вставок/делений в мишени-протоспейсере ЕМХ1. Как иллюстрируется па фигуре 4В, коэкспрессия целенаправленно воздействующей на ЕМХ1 химерной crRNA с SpCas9 дает вставки/делеции в целевом сайте, тогда как коэкспрессия с SpCas9n - нет (n=3). Более того, секвенирование 327 ампликонов не выявило каких-либо вставок/делений, индуцированных SpCas9n. Для исследования опосредованной CRISPR HR при совместной трансфекции клеток HEK 293FT химерной РНК, целенаправленно воздействующей на ЕМХ1, hSpCas9 или hSpCas9n, выбирали тот же локус, также как и матрицу для HR для введения пары сайтов рестрикции (HindIII и NheI) около протоспейсера. На фигуре 4С приведена схематическая иллюстрация стратегии HR с относительными положениями точек рекомбинации и последовательностей для гибридизации праймеров (стрелки). SpCas9 и SpCas9n действительно катализируют интеграцию матрицы HR в локус ЕМХ1. ПЦР амплификация целевого участка с последующим рестрикционным расщеплением HindIII выявила продукты расщепления, соответствующие ожидаемым размерам фрагментов (стрелки на результатах анализа полиморфизма длин рестрикционных фрагментов с помощью гель-электрофореза, показанных на фигуре 4D), причем SpCas9 и SpCas9n опосредуют подобные уровни эффективности HR. Заявители дополнительно подтверждали HR с использованием секвенирования геномных ампликонов по Сэнгеру (фигура 4Е). Эти результаты демонстрировали пригодность CRISPR для облегчения целенаправленной вставки генов в геном млекопитающего. С учетом специфичности к мишени в 14 п.о. (12 п.о. от спейсера и 2 п.о. от РАМ) SpCas9 дикого типа доступность никазы может значительно снизить вероятность нецелевой модификации, поскольку одноцепочечные разрывы не являются субстратами для подверженного ошибкам пути NHEJ.
Экспрессионные конструкции, имитирующие естественную архитектуру локусов CRISPR с собранными в массив спейсерами (фигура 2А), конструировали для исследования возможности мультиплексного целенаправленного воздействия на последовательности. При использовании одного массива CRISPR, кодирующего пару спейсеров, целенаправленно воздействующих на ЕМХ1 и PVALB, выявлялось эффективное расщепление в обоих локусах (фигура 4F, на которой показаны как схематическая структура массива crRNA, так и блот, полученный после анализа расщепления Surveyor, показывающий эффективное опосредованно расщепления). Также исследовали целенаправленную делению геномных участков большего размера посредством одновременных DSB с использованием спейсеров против двух мишеней в ЕМХ1, разделенных 119 п.о., и выявили эффективность делении 1,6% (3 из 182 ампликонов; фигура 4G). Это демонстрирует, что система CRISPR может опосредовать мультиплексное редактирование в пределах одного генома.
Пример 2. Модификации и альтернативы системы CRISPR
Возможность применения РНК для программирования специфичного к последовательности расщепления ДНК определяет новый класс инструментов для конструирования генома для разнообразных исследовательских и промышленных применений. Несколько аспектов системы CRISPR можно дополнительно улучшить для повышения эффективности и универсальности целенаправленного воздействия с помощью CRISPR. Оптимальная активность Cas9 может зависеть от доступности несвязанного Mg2+ на уровнях, которые превышают имеющиеся в ядре млекопитающего (см., например, Jinek et at., 2012, Science, 337: 816), и предпочтение в отношении NGG мотива непосредственно ниже протоспейсера ограничивает способность к целенаправленному воздействию, в среднем, на каждые 12 п.о. в геноме человека (фигура 9, оценка как плюс-, так и минус-нитей в хромосомных последовательностях человека). Некоторые из этих затруднений можно преодолеть путем изучения разнообразия локусов CRISPR в микробном метагеноме (см., например, Makarova et al., 2011, Nat Rev Microbiol, 9: 467). Другие локусы CRISPR можно пересадить в микроокружение клетки млекопитающего с помощью способа, подобного описанному в примере 1. Например, на фигуре 10 иллюстрируется адаптация системы CRISPR II типа из CRISPR I Streptococcus thermophilus LMD-9 для гетерологичной экспрессии в клетках млекопитающих, чтобы достичь опосредованного CRISPR редактирования генома. На фигуре 10А приведена схематическая иллюстрация CRISPR 1 из S. thermophilus LMD-9. На фигуре 10В иллюстрируется структура системы экспрессии для системы CRISPR S. thermophilus. Кодон-оптимизированный hStCas9 человека экспрессируется с помощью конститутивного промотора EF1α. Зрелые варианты tracrRNA и crRNA экспрессируются с помощью промотора U6 для стимуляции точной инициации транскрипции. Проиллюстрированы последовательности из зрелых crRNA и tracrRNA. Одно основание, обозначенное буквой "а" в нижнем регистре в последовательности crRNA используют для удаления последовательности polyU, которая служит в качестве терминатора транскрипции РНК polIII. На фигуре 10С приведено схематическое изображение направляющих последовательностей, нацеленных на локус ЕМХ1 человека. На фигуре 10D показаны результаты опосредованного hStCas9 расщепления в целевом локусе с использованием анализа с помощью Surveyor. РНК направляющих спейсеров 1 и 2 индуцировали 14% и 6,4%, соответственно. Статистический анализ активности в отношении расщепления по биологическим копиям в этих двух протоспейсерных сайтах также приведен на фигуре 5. На фигуре 14 приведено схематическое изображение дополнительного протоспейсера и соответствующих последовательностей РАМ, являющихся мишенями для системы CRISPR S. thermophilus, в локусе ЕМХ1 человека. Выделены последовательности двух протоспейсеров и их соответствующие последовательности РАМ, удовлетворяющие мотиву NNAGAAW, обозначены путем подчеркивания в направлении 3' относительно соответствующей выделенной последовательности. Оба протоспейсера нацелены на антисмысловую нить.
Пример 3. Алгоритм выбора образцов целевой последовательности
Создали компьютерную программу для идентификации кандидатных целевых последовательностей CRISPR на обеих нитях вводимой последовательности ДНК на основе длины желаемой направляющей последовательности и последовательности мотива CRISPR (РАМ) для определенного фермента CRISPR. Например, целевые сайты для Cas9 из S. pyogenes с последовательностями РАМ NGG можно идентифицировать путем поиска в отношении 5'-Nx-NGG-3' как на вводимой последовательности, так и на последовательности, обратно-комплементарной вводимой. Подобным образом, целевые сайты для Cas9 CRISPR1 S. thermophilus с последовательностью РАМ NNAGAAW, можно идентифицировать путем поиска в отношении 5'-Nx-NNAGAAW-3' как на вводимой последовательности, так и на последовательности, обратно-комплементарной вводимой. Подобным образом, целевые сайты для Cas9 CRISPR3 S. thermophilus с последовательностью РАМ NGGNG можно идентифицировать путем поиска в отношении 5'-Nx-NGGNG-3' как на вводимой последовательности, так и на последовательности, обратно-комплементарной вводимой. Значение "х" в Nx может фиксироваться программой или может быть определено пользователем, как например, 20.
Поскольку несколько случаев появления целевого сайта ДНК в геноме могут приводить к неспецифическому редактированию генома, после идентификации всех возможных сайтов программа профильтровывает последовательности, исходя из количества раз, когда они встречаются в подходящем эталонном геноме. Для тех ферментов CRISPR, для которых специфичность последовательности определяется 'затравочной' последовательностью, такой как находящаяся в 11-12 п.о. в направлении 5' от последовательности РАМ, в том числе сама последовательность РАМ, стадия фильтрования может основываться на затравочной последовательности. Следовательно, во избежание редактирования в дополнительных локусах генома результаты фильтруют, исходя из числа случаев обнаружения последовательности затравки : РАМ в подходящем геноме. Пользователь может иметь возможность выбора длины затравочной последовательности. Пользователь также может иметь возможность определять число случаев обнаружения последовательности затравки : РАМ в геноме применительно к прохождению фильтра. По умолчанию установлен скрининг в отношении уникальных последовательностей. Уровень фильтрования изменяют путем изменения как длины затравочной последовательности, так и числа случаев обнаружения последовательности в геноме. В качестве дополнения или альтернативы, программа может обеспечивать последовательность направляющей последовательности, комплементарную сообщенной(ым) целевой(ым) последовательностям) путем обеспечения последовательности, обратно комплементарной идентифицированной(ым) целевой(ым) последовательностям). Иллюстративная визуализация некоторых целевых сайтов в геноме человека представлена на фигуре 18.
Дальнейшие детали способов и алгоритмов для оптимизации выбора последовательности можно найти в заявке на патент США с порядковым номером 61/064798 (номер дела у патентного поверенного 44790.11.2022; расширенная ссылка BI-2012/084); включенной в данный документ по ссылке.
Пример 4. Оценка гибридов нескольких химерных crRNA-tracrRNA
В данном примере описаны результаты, полученные для химерных РНК (chiRNA; содержащие направляющую последовательность, парную tracr-последовательность и tracr-последовательность в одном транскрипте), имеющих tracr-последовательности, которые включают фрагменты последовательности tracrRNA дикого типа с разной длиной. На фигуре 16а проиллюстрировано схематическое изображение бицистронного вектора экспрессии для химерной РНК и Cas9. Cas9 управляется промотором CBh, а химерная РНК управляется промотором U6. Химерная направляющая РНК состоит из направляющей последовательности (Ns) из 20 п.о., соединенной с tracr-последовательностью (проходящей от первого "U" в нижней нити к концу транскрипта), которая усечена в разных указанных положениях. Направляющие и tracr-последовательности разделены парной tracr-последовательностью GUUUUAGAGCUA, за которой следует последовательность петли GAAA. Результаты анализов с помощью SURVEYOR в отношении опосредованных Cas9 вставок/делений в локусах ЕМХ1 и PVALB человека проиллюстрированы на фигурах 16b и 16с, соответственно. Стрелки указывают на ожидаемые фрагменты, полученные в результате расщепления SURVEYOR. ChiRNA показаны путем обозначения их "+n", a crRNA относится к гибридной РНК, в которой направляющие и tracr-последовательности экспрессируются в виде раздельных транскриптов. Количественное определение этих результатов, выполненное в трех повторностях, проиллюстрировано гистограммой на фигурах 17а и 17b, соответствующих фигурам 16b и 16с, соответственно ("N.D." указывает на отсутствие выявленных вставок/делений). ID (идентификационные данные) протоспейсеров и их соответствующей геномной мишени, последовательность протоспейсера, последовательность РАМ и положение нити приведены в таблице D. Направляющие последовательности сконструированы так, чтобы они были комплементарны полной последовательности протоспейсера в случае отдельных транскриптов в гибридной системе, или только подчеркнутой части в случае химерных РНК.

Дополнительные детали для оптимизации направляющих последовательностей можно найти в заявке на патент США с порядковым номером 61/836127 (номер дела у патентного поверенного 44790.08.2022; расширенная ссылка BI-2013/004G); включенной в данный документ по ссылке.
Первоначально целенаправленному воздействию подвергали три сайта в пределах локуса ЕМХ1 у клеток HEK 293FT человека. Эффективность модификации генома каждой chiRNA оценивали с использованием анализа с помощью нуклеазы SURVEYOR, который выявляет мутации, возникающие в результате двухнитевых разрывов (DSB) ДИК и их последующей репарации с помощью пути репарации повреждения ДНК путем негомологичного соединения концов (NHEJ). В конструкциях, обозначенных chiRNA(+n), указывается, что нуклеотиды в количестве до +n нуклеотида в tracrRNA дикого типа включены в химерную РНК конструкцию, при этом для n используются значения 48, 54, 67 и 85. Химерные РНК, содержащие более длинные фрагменты tracrRNA дикого типа (chiRNA(+67) и chiRNA(+85)), опосредовали расщепление ДНК во всех трех целевых сайтах ЕМХ1, причем chiRNA(+85), в частности, демонстрировал значительно более высокие уровни расщепления ДНК, чем соответствующие гибриды crRNA/tracrRNA, у которых направляющие и tracr-последовательности экспрессируются в отдельных транскриптах (фигуры 16b и 17а). Два сайта в локусе PVALB, которые не давали выявляемого расщепления с использованием гибридной системы (направляющая последовательность и tracr-последовательность, экспрессируемые в виде отдельных транскриптов), также подвергались целенаправленному воздействию с использованием chiRNA. ChiRNA(+67) и chiRNA(+85) были способны опосредовать значительное расщепление в двух протоспейсерах в PVALB (фигуры 16с и 17b).
Для всех пяти мишеней в локусах ЕМХ1 и PVALB наблюдали соответствующее повышение эффективности модификации генома с увеличением длины tracr-последовательности. Не вдаваясь в какую-либо теорию, вторичная структура, формируемая 3' концом tracrRNA, может играть роль в увеличении скорости образования комплекса CRISPR.
Пример 5. Разнообразие Cas9
Система CRISPR-Cas является адаптивным иммунным механизмом в отношении внедряющейся экзогенной ДНК, используемым разнообразными видами из числа бактерий и архей. Система CRISPR-Cas9 типа II состоит из набора генов, кодирующих белки, ответственные за "захват" чужеродной ДНК в локус CRISPR, а также из набора генов, кодирующих "выполнение" механизма расщепления ДНК; они включают ДНК-нуклеазу (Cas9), некодирующую транс-активирующую cr-RNA (tracrRNA) и массив полученных из чужеродной ДНК спейсеров, фланкированных прямыми повторами (crRNA). При созревании под действием Cas9 дуплекс tracRNA n crRNA направляет нуклеазу Cas9 к целевой последовательности ДНК, определенной спейсерными направляющими последовательностями и опосредует двухнитевые разрывы в ДНК вблизи короткого мотива последовательности в целевой ДНК, которые требуются для расщепления и являются специфичными для каждой системы CRISPR-Cas. Системы CRISPR-Cas II типа обнаруживаются повсеместно в царстве бактерий и являются очень разнообразными по последовательности и размеру белка Cas9, последовательности прямого повтора tracrRNA и crRNA, организации этих элементов в геноме и требованиям к мотиву для целенаправленного расщепления. Один вид может иметь несколько отдельных систем CRISPR-Cas.
Заявители оценивали 207 предполагаемых Cas9 из видов бактерий, идентифицированных на основе гомологии последовательности с известными Cas9 и структурами, ортблогичными известным субдоменам, в том числе домену эндонуклеазы HNH и доменам эндонуклеазы RuvC [информация от Eugene Koonin и Kira Makarova]. Филогенетический анализ на основе консервативности белковой последовательности в этом наборе выявил пять семейств Cas9, включающие три группы больших Cas9 (~1400 аминокислот) и две группы небольших Cas9 (~1100 аминокислот) (см. фигуры 19 и 20А-F).
Дополнительные детали Cas9 и мутации в ферменте Cas9 для превращения в никазу или ДНК-связывающий белок и их применение с измененной функциональностью можно найти в заявках на патент США с порядковыми номерами 61/836101 и 61/835936 (номера дел у патентного поверенного 44790.09.2022 и 4790.07.2022 и расширенные ссылки BI-2013/004E и BI-2013/004F, соответственно), включенных в данный документ по ссылке.
Пример 6. Ортологи Cas9
Заявители анализировали ортологи Cas9 для идентификации подходящих последовательностей РАМ и соответствующей химерной направляющей РНК. Наличие расширенного набора РАМ обеспечивает более расширенное целенаправленное воздействие по геному, а также значительно повышает число уникальных целевых сайтов и обеспечивает потенциал для идентификации новых Cas9 с повышенными уровнями специфичности в геноме.
Специфичность ортологов Cas9 можно оценить с помощью исследования способности каждого Cas9 допускать несовпадения между направляющей РНК и ДНК-мишенью. Например, специфичность SpCas9 была охарактеризовала с помощью исследования эффекта мутаций в направляющей РНК в отношении эффективности расщепления. Создавали библиотеки направляющих РНК с одним или несколькими несовпадениями между направляющей последовательностью и целевой ДНК. Исходя из этих данных, целевые сайты для SpCas9 можно выбрать на основе следующих рекомендаций.
Для максимального повышения специфичности SpCas9 в отношении редактирования конкретного гена, специалисту следует выбрать целевой сайт в пределах представляющего интерес локуса таким образом, чтобы возможные 'нецелевые' геномные последовательности соблюдали следующие четыре ограничения: первое и главное, за ними не должен следовать РАМ с любой из последовательностей 5'-NGG или NAG; второе, глобальное сходство их последовательности с целевой последовательностью должно быть сведено к минимуму; третье, максимальное число несовпадений должно находиться в пределах ближайшего к РАМ участка в нецелевом сайте; наконец, максимальное число несовпадений должны быть последовательными или отделенными друг от друга менее чем четырьмя основаниями.
Подобные способы можно использовать для оценки специфичности других ортологов Cas9 и для установления критериев для выбора специфических целевых сайтов в геномах целевых видов. Как упоминалось ранее, филогенетический анализ на основе консервативности белковой последовательности в этом наборе выявил пять семейств Cas9, включающие три группы больших Cas9 (~1400 аминокислот) и две группы небольших Cas9 (~1100 аминокислот) (см. фигуры 19 и 20A-F). Дальнейшие детали в отношении ортологов Cas можно выявить заявках на патент США с порядковыми номерами 61/836101 и 61/835936 (номера дел у патентного поверенного 44790.09.2022 и 4790.07.2022 и расширенные ссылки BI-2013/004E и BI-2013/004F, соответственно), включенных в данный документ по ссылке.
Пример 7. Методические улучшения для упрощения клонирования и доставки
Вместо того чтобы закодировать промотор U6 и направляющую РНК на плазмиде, заявители амплифицировали промотор U6 с олигонуклеотидом ДНК для достройки направляющей РНК. Полученный в результате ПЦР продукт можно трансфицировать в клетки для управления экспрессией направляющей РНК.
Иллюстративная пара праймеров, которая обеспечивает возможность образования ПЦР продукта, состоящего из промотора U6: направляющей РНК, целенаправленно воздействующих на локус Emx1 человека:
прямой праймер: AAACTCTAGAgagggcctatttcccatgattc,
обратный праймер (несущий направляющую РНК, которая подчеркнута).

Пример 8. Методическое улучшение для улучшения активности
Вместо применения промоторов pol3, в частности, РНК полимеразы III (например, промоторов U6 или H1), для экспрессии направляющих РНК в эукариотических клетках, заявители обеспечивали экспрессию полимеразы Т7 в эукариотических клетках для управления экспрессией направляющих РНК с использованием промотора Т7.
Один пример этой системы может включать введение трех частей ДНК:
1. вектора экспрессии для Cas9;
2. вектора экспрессии для полимеразы Т7;
3. вектора экспрессии, содержащего направляющую РНК, слитую с промотором Т7.
Пример 9. Методическое улучшение для снижения токсичности Cas9: доставка Cas9 в форме мРНК
Доставка Cas9 в форме мРНК обеспечивает возможность временной экспрессии Cas9 в клетках для снижения токсичности. Например, гуманизированный SpCas9 можно амплифицировать с использованием следующей пары праймеров:
прямой праймер (для достройки промотора Т7 для транскрипции in vitro):

обратный праймер (для достройки полиА-хвоста):
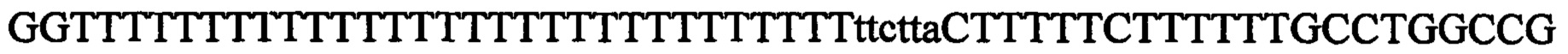
Заявители трансфицировали мРНК Cas9 в клетки направляющей РНК в форме кассет или РНК, или ДНК для управления экспрессией направляющей РНК в эукариотических клетках.
Пример 10. Методическое улучшение для снижения токсичности Cas9: применение индуцируемого промотора
Заявители временно включали экспрессию Cas9 только когда она требовалась для осуществления модификации генома. Примеры индуцируемой системы включают индуцируемые тетрациклином промоторы (Tet-On пли Tet-Off), системы для двугибридной активации транскрипции с использованием малых молекул (FKBP, АВА и т.д.) или индуцируемые светом системы (фитохром, LOV домены или криптохром).
Пример 11. Улучшение системы Cas9 для применения in vivo
Заявители проводили поиск с использованием метагеномного подхода в отношении Cas9 с малым молекулярным весом. Большинство гомологов Cas9 являются достаточно большими. Например, SpCas9 имеет длину около 1368 а.к., что слишком много для легкой упаковки в вирусные векторы для доставки. График, представляющий распределение длины гомологов Cas9, получали на основе последовательностей, депонированных в GenBank (фигура 23). Некоторые из последовательностей могли быть неверно аннотированы, и, таким образом, точная частота каждой длины не обязательно может быть достоверной. Тем не менее она дает некоторое представление о распределении белков Cas9 и указывает на то, что существуют более короткие гомологи Cas9.
С помощью анализа на основе расчетов заявители обнаружили, что у штамма бактерии Campylobacter присутствуют два белка Cas9 с менее чем 1000 аминокислот. Последовательность для одного Cas9 из Campylobacter jejuni представлена ниже. При такой длине CjCas9 может быть легко упакован в AAV, лентивирусы, аденовирусы и другие вирусные векторы для надежной доставки в первичные клетки и in vivo в животных моделях. В предпочтительном варианте осуществления настоящего изобретения применяют белок Cas9 из S. aureus.
>Cas9 Campylobacter jejuni (CjCas9)

Предполагаемый элемент tracrRNA для данного CjCas9 представляет собой:
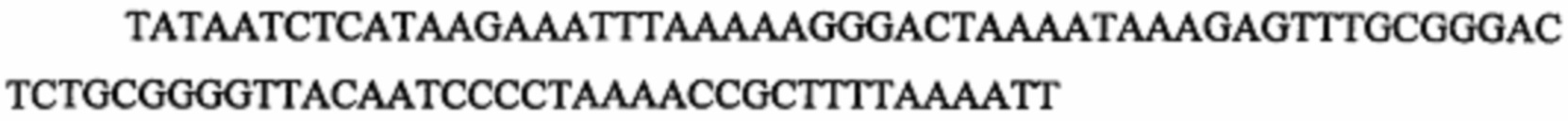
Последовательность прямого повтора представляет собой:
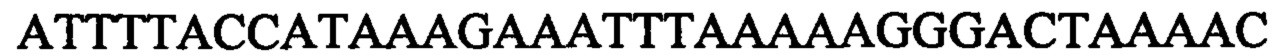
Пример химерной направляющей РНК для CjCas9 представляет собой:
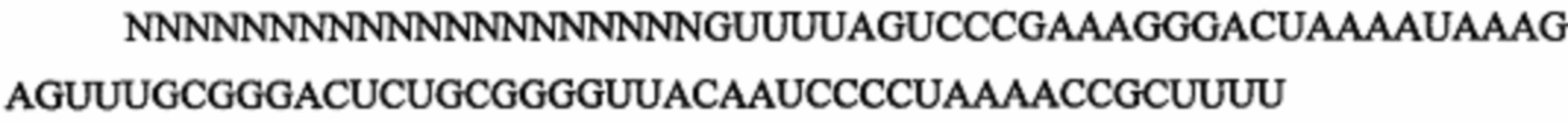
Пример 12. Оптимизация Cas9
Для усиленного функционирования или для развития новых функций заявители создали химерные белки Cas9 путем объединения фрагментов от различных гомологов Cas9. Например, два иллюстративных химерных белка Cas9:
Например, заявители слили N-конец St1Cas9 (фрагмент из этого белка выделен жирным шрифтом) с С-концом SpCas9 (фрагмент из этого белка подчеркнут).
>St1(N)Sp(C)Cas9

>Sp(N)St1(C)Cas9


Выгода от создания химерного Cas9 включает:
снижение токсичности;
улучшение экспрессии в эукариотических клетках;
повышение специфичности;
снижение молекулярного веса белка, делающее белок меньше за счет объединения наименьших доменов от различных гомологов Cas9.
Изменение требований к последовательности РАМ.
Пример 13. Использование Cas9 в качестве стандартного ДНК-связывающсго белка
Заявители использовали Cas9 в качестве стандартного ДНК-связывающего белка путем внесения мутации в два каталитических домена (D10 и Н840), ответственных за расщепление обеих нитей ДНК-мишени. С целью повышающей регуляции транскрипции гена в целевом локусе заявители слили домен активации транскрипции (VP64) с Cas9. Заявители предположили, что будет важно увидеть строгую ядерную локализацию слитого белка Cas9-VP64, поскольку эффективность активации транскрипционным фактором зависит от времени, проведенном у мишени. Таким образом, заявители клонировали набор Cas9-VP64-GFP конструкций, переносили их в клетки 293 и оценивали их локализацию под флуоресцентным микроскопом через 12 часов после трансфекции.
Те же конструкции клонировали в виде 2A-GFP, а не в виде прямого слияния, с целью функционального исследования конструкций без помех от массового присутствия GFP. Заявители выбрали для целенаправленного воздействия локус Sox2 с трансактиватором Cas9, поскольку он может быть полезен для перепрограммирования клеток, и данный локус уже был подтвержден как мишень для опосредованной TALE-TF активации транскрипции. Для локуса Sox2 заявители выбрали восемь целей рядом с сайтом инициации транскрипции (TSS). Каждая мишень имела длину 20 п.о. с расположенным рядом с мотивом, прилегающим к протоспейсеру (РАМ) NGG. Каждую конструкцию Cas9-VP64 совместно трансфицировали с каждой полученной с помощью ПЦР химерной crispr РНК (chiRNA) в клетки 293. Через 72 часов после трансфекции активацию транскрипции оценивали с помощью RT-qPCR.
Для дополнительной оптимизации активатора транскрипции, заявители подбирали соотношение chiRNA (Sox2.1 и Sox2.5) к Cas9 (NLS-VP64-NLS-hSpCas9-NLS-VP64-NLS), трансфицировали в клетки 293 и количественно определяли с помощью RT-qPCR. Эти результаты указывают на то, что Cas9 можно использовать в качестве стандартного ДНК-связывающего домена для повышающей регуляции транскрипции гена в целевом локусе.
Заявители разработали второе поколение конструкций. (таблица ниже).

Заявители применяли эти конструкции для оценки активации (слитые конструкции с VP64) и репрессии (только Cas9) транскрипции с помощью RT-qPCR. Заявители оценивали клеточную локализацию каждой конструкции с использованием антитела к His, нуклеазную активность с использованием анализа с помощью нуклеазы Surveyor и аффинность связывания с ДНК с использованием анализа задержки электрофоретического сдвига в геле. В предпочтительном варианте осуществления настоящего изобретения анализ задержки электрофоретической сдвига в геле представляет собой анализ изменения электрофоретической подвижности EMSA.
Пример 14. Мыши, трансгенные по Cas9 и с нокином по Cas9
Для получения мыши, которая экспрессирует нуклеазу Cas9, заявители рассматривали две общих стратегии, трансгенез и нокин. Эти стратегии можно применять для получения любого другого модельного организма, представляющего интерес, например, крысы. Для каждой из общих стратегий заявители получили конститутивно активный Cas9 и Cas9, который экспрессируется при определенных условиях (зависимый от рекомбиназы Cre). Конститутивно активная нуклеаза Cas9 экспрессируется в следующем контексте: pCAG-NLS-Cas9-NLS-P2A-EGFP-WPRE-bGHpolyA. pCAG представляет собой промотор, NLS представляет собой сигнал ядерной локализации, Р2А представляет собой последовательность расщепления пептида, EGFP представляет собой улучшенный зеленый флуоресцентный белок, WPRE представляет собой посттранскрипционный регуляторный элемент вируса гепатита сурков, и bGHpolyA представляет собой поли-А сигнальную последовательность бычьего гормона роста (фигуры 25А-В). Активный при определенных условиях вариант имел один дополнительный элемент в виде стоп-кассеты, loxP-SV40 polyA х3-loxP, после промотора и перед NLS-Cas9-NLS (т.е. pCAG-loxP-SV40polyAx3-loxP-NLS-Cas9-NLS-P2A-EGFP-WPRE-bGHpolyA). Важные экспрессионные элементы наглядно представлены на фигуре 26. Конститутивная конструкция должна экспрессироваться во всех типах клеток в ходе развития, тогда как экспрессируемая при определенных условиях конструкция будет обеспечивать возможность экспрессии Cas9 только когда та же клетка будет экспрессировать рекомбиназу Cre. Этот последний вариант будет обеспечивать возможность тканеспецифичной экспрессии Cas9, когда Cre находится под воздействием экспрессии тканеспецифичного промотора. Более того, экспрессию Cas9 можно индуцировать у взрослых мышей путем помещения Cre под воздействие экспрессии индуцируемого промотора, такого как система ТЕТ on или off.
Валидация конструкций Cas9: каждую плазмиду подвергают функциональной валидации тремя способами: 1) временная трансфекция в клетки 293 с последующим подтверждением экспрессии GFP; 2) временная трансфекция в клетки 293 с последующей иммунофлуоресценцией с использованием антитела, узнающего последовательность Р2А; и 3) временная трансфекция с последующим анализом с помощью нуклеазы Surveyor. Клетки 293 могут представлять собой клетки 293FT или 293 Т в зависимости от того, какие клетки представляют интерес. В предпочтительном варианте осуществления клетки представляют собой клетки 293FT. Результаты расщепления с помощью Surveyor разделяли на верхней и нижней лунках геля для конструкции, чья экспрессия активируется при определенных условиях, и для конститутивной конструкции, соответственно. Каждую из них исследовали в присутствии и в отсутствие химерной РНК, целенаправленно воздействующей на локус hEMX1 (химерная РНК hEMX1.1). Результаты указывают на то, что конструкция может успешно целенаправленно воздействовать на локус hEMX1 только в присутствии химерной РНК (и Cre в случае с экспрессией при определенных условиях). Гели использовали для количественного определения, и результаты представлены в виде средней эффективности разрезания и стандартного отклонения для трех образцов.
Трансгенные по Cas9 мыши Для получения трансгенных мышей с конструкциями заявители инъецировали чистую линейную ДНК в пронуклеус зиготы от псевдобеременной самки СВ56. Особей-основателей идентифицировали, генотипировали и подвергали обратному скрещиванию с мышами СВ57. Конструкции успешно клонировали и подтверждали путем секвенирования по Сэнгеру.
Мыши с нокином по Cas9 Для создания нокина по Cas9 у мышей заявители целенаправленно воздействовали теми же конститутивными конструкциями и конструкциями, активными при определенных условиях, на локус Rosa26. Заявители осуществили это путем клонирования каждого из них в целенаправленно воздействующий на Rosa26 вектор со следующими элементами: короткое гомологичное плечо Rosa26 - кассета для конститутивной/активирующейся при определенных условиях экспрессии Cas9 - длинное гомологичное плече pPGK-Neo-Rosa26 - pPGK-DTA. pPGK представляет собой промотор для маркера положительного отбора Neo, который придает устойчивость к неомицину, короткое плечо длиной 1 т.п.о., длинное плече длиной 4,3 т.п.о. и дифтерийный токсин (DTA), управляемый PGK, для отрицательного отбора.
Две конструкции вводили посредством электропорации в mESC R1 и обеспечивали возможность роста в течение 2 дней до применения отбора с помощью неомицина. Отдельные колонии, которые выжили к 5-7 дню, собирали и выращивали в отдельных лунках. 5-7 дней спустя колонии собирали, половину из них замораживали, а другую половину использовали для генотипирования. Генотипирование выполняли с помощью геномной ПЦР, когда один праймер отжигали в донорской плазмиде (AttpF), а второй за пределами короткого гомологичного плеча (Rosa26-R). Из 22 колоний, собранных для случаев экспрессии при определенных условиях, 7 были положительными (слева). Из 27 колоний, собранных для случаев конститутивной экспрессии, ноль были положительных (справа). Вероятно, что Cas9 приводит к некоторому уровню токсичности в mESC, и по этой причине положительные клоны не присутствовали. Для изучения этого факта заявители вводили плазмиду для экспрессии Cre в клетках с точным целенаправленным воздействием экспрессируемой при определенных условиях Cas9 и выявили очень низкую токсичность после большого количества дней в культуре. Уменьшенного числа копий Cas9 в клетках с корректным целенаправленным воздействием экспрессируемой при определенных условиях Cas9 (1-2 копии на клетку) достаточно для обеспечения возможности стабильной экспрессии и относительного отсутствия цитотоксичности. Более того, эти данные указывают на то, что число копий Cas9 определяет токсичность. После электропорации каждая клетка должна получить несколько копий Cas9, и, вероятно, вследствие этого не обнаруживалось положительных колоний в случае конститутивной конструкции Cas9. Это обеспечивает убедительное доказательство того, что использование стратегии экспрессии при определенных условиях, Cre-зависимой стратегии, должно показать пониженную токсичность. Заявители вводили клетки, подвергнутые точному целенаправленному воздействию, в бластоцисту и имплантировали в самку мыши. Химерных особей идентифицировали и подвергали обратному скрещиванию. Особей-основателей идентифицировали и генотипировали.
Использование мышей с экспрессией Cas9 при определенных условиях Заявители показали на клетках 293, что конструкция для условной экспрессии Cas9 может активироваться при коэкспрессии с Cre. Заявители также показали, что подвергшиеся точному целенаправленному воздействию mESC R1 могут иметь активный Cas9, когда Cre экспрессируется. Поскольку за Cas9 следует последовательность расщепления пептида Р2А, а затем EGFP, заявители идентифицировали успешную экспрессию путем наблюдения EGFP. Эта же идея делает использование мышей с экспрессией Cas9 при определенных условиях столь полезным. Заявители могут скрещивать свою мышь с экспрессией Cas9 при определенных условиях с мышью, у которой повсеместно экспрессируется Cre (линия АСТВ-Cre), и могут получать мышь, которая экспрессирует Cas9 в каждой клетке. Для этого потребуется только доставка химерной РНК для индукции редактирования генома у мышиных эмбрионов или взрослых мышей. Что интересно, если мышь с экспрессией Cas9 при определенных условиях скрестить с мышью, экспрессирующей Cre под контролем тканеспецифического промотора, Cas9 будет лишь в тканях, в которых также экспрессируется Cre. Этот подход можно применять для редактирования генома только в определенных тканях путем доставки химерной РНК в ту же ткань.
Пример 15. Разнообразие Cas9 и химерные РНК
Система CRISPR-Cas является адаптивным иммунным механизмом в отношении внедряющейся экзогенной ДНК, используемым разнообразными видами из числа бактерий и архей. Система CRISPR-Cas типа II состоит из набора генов, кодирующих белки, ответственные за "захват" чужеродной ДНК в локус CRISPR, а также из набора генов, кодирующих "выполнение" механизма расщепления ДНК; они включают ДНК-нуклеазу (Cas9), некодирующую транс-активирующую cr-RNA (tracrRNA) и массив полученных из чужеродной ДНК спейсеров, фланкированных прямыми повторами (crRNA). При созревании под действием Cas9 дуплекс tracrRNA и crRNA направляет нуклеазу Cas9 к целевой последовательности ДНК, определенной спейсерными направляющими последовательностями и опосредует двухнитевые разрывы в ДНК вблизи короткого мотива последовательности в целевой ДНК, которые требуются для расщепления и являются специфичными для каждой системы CRISPR-Cas. Системы CRISPR-Cas II типа обнаруживаются повсеместно в царстве бактерий и являются очень разнообразными по последовательности и размеру белка Cas9, последовательности прямого повтора tracrRNA и crRNA, организации этих элементов в геноме и требованиям к мотиву для целенаправленного расщепления. Один вид может иметь несколько отдельных систем CRISPR-Cas.
Заявители оценивали 207 предполагаемых Cas9 из видов бактерий, идентифицированных на основе гомологии последовательности с известными Cas9 и структурами, ортологичными известным субдоменам, в том числе домену эндонуклеазы HNH и доменам эндонуклеазы RuvC [информация от Eugene Koonin и Kira Makarova]. Филогенетический анализ на основе консервативности белковой последовательности в этом наборе выявил пять семейств Cas9, включающие три группы больших Cas9 (~1400 аминокислот) и две группы небольших Cas9 (~1100 аминокислот) (фигуры 19A-D и 20A-F).
Заявители также получали оптимизированную направляющую РНК для Cas9 с использованием способов in vitro.
Пример 16. Мутации Cas9
В этом примере заявители показали, что следующие мутации могут превратить SpCas9 в надрезающий фермент: D10A, Е762А, Н840А, N854A, N863A, D986A.
Заявители представили последовательности, показывающие где расположены точки мутаций в гене SpCas9 (фигура 24А-М). Заявители также показали, что никазы при этом все еще способны опосредовать гомологичную рекомбинацию. Более того, заявители показали, что SpCas9 с этими мутациями (отдельно) не индуцировал двухнитевой разрыв.
Все ортологи Cas9 имеют подобную общую организацию из 3-4 доменов RuvC и домена HNH. Наиболее близкий к 5' домен RuvC расщепляет некомплементарную нить, а домен HNH расщепляет комплементарную нить. Все обозначения приведены в отношении направляющей последовательности.
Каталитический остаток в 5'-домене RuvC идентифицировали посредством сравнения с целью поиска гомологии Cas9, представляющего интерес, и других ортологов Cas9 (из локуса CRISPR типа II S. pyogenes, локуса 1 CRISPR S. thennophilus, локуса 3 CRISPR S. thermophilus и локуса CRISPR типа II Franciscilla novicida), и консервативный остаток Asp подвергали мутации замены на аланин с превращением Cas9 в фермент, вносящий однонитевой разрыв в комплементарную нить. Аналогично, консервативные остатки His и Asn в доменах HNH подвергают мутации по типу замены на аланин с превращением Cas9 в фермент, вносящий однонитевой разрыв в некомплементарную нить.
Пример 17. Активация транскрипции Cas9 и репрессор Cas9
Активация транскрипции Cas9
Конструировали и исследовали второе поколение конструкций (таблица 1). Данные конструкции использовали для оценки активации транскрипции (слитые конструкции с VP64) и репрессии (только Cas9) с помощью RT-qPCR. Заявители оценивали клеточную локализацию каждой конструкции с использованием антитела к His, нуклеазную активность с использованием анализа с помощью нуклеазы Surveyor и аффинность связывания с ДНК с использованием анализа задержки электрофоретического сдвига в геле.
Репрессор Cas
Ранее было показано, что dCas9 можно использовать в качестве стандартного ДНК-связывающего домена для репрессии экспрессии гена. Заявители описывают улучшенную структуру dCas9, а также слияния dCas9 с репрессорными доменами KRAB и SID4x. Из плазмидной библиотеки, созданной для модуляции транскрипции с использованием Cas9 в таблице 1, с помощью qPCR были функционально охарактеризованы следующие репрессорные плазмиды: pXRP27, pXRP28, pXRP29, pXRP48, pXRP49, pXRP50, pXRP51, pXRP52, pXRP53, pXRP56, pXRP58, pXRP59, pXRP61 и pXRP62.
Каждую репрессорную плазмиду для dCas9 котрансфицировали двумя направляющими РНК, осуществляющими нацеливание на кодирующую нить гена бета-катенина. РНК выделяли через 72 часа после трансфекции и экспрессию гена количественно определяли с помощью RT-qPCR. Эндогенным контрольным геном был GAPDH. Две валидированные shRNA использовали в качестве положительных контролей. Отрицательными контролями были определенные плазмиды, трансфицированные без gRNA (направляющей РНК), они обозначены как "pXRP## контроль". Плазмиды pXRP28, pXRP29, pXRP48 и pXRP49 могут репрессировать ген бета-катенина при использовании определенной стратегии целенаправленного воздействия. Эти плазмиды соответствуют dCas9 без функционального домена (pXRP28 и pXRP28) и dCas9, слитому с SID4x (pXRP48 и pXRP49).
В дальнейших работах проводятся следующие исследования: повторение вышеуказанного эксперимента, целенаправленное воздействие на различные гены, использование других gRNA для определения оптимального положения для целенаправленного воздействия и мультиплексная репрессия.


Пример 18. Направленная делеция генов, вовлеченных в биосинтез холестерина, биосинтез жирных кислот и другие метаболические нарушения, генов, кодирующих подвергающиеся неправильному фолдингу белки, вовлеченные в амилоидные и другие заболевания, онкогенов, приводящих к клеточной трансформации, латентных вирусных генов и генов, приводящих к доминантно-негативным нарушениям среди прочих нарушений
Заявители продемонстрировали доставку гена системы CRISPR-Cas в ткань печени, головного мозга, глазную, эпителиальную, кроветворную или иную ткань субъекта или пациента, нуждающегося в этом, страдающего от метаболических нарушений, амилоидоза и заболеваний, связанных с агрегацией белков, трансформации клеток, возникающей в результате генетических мутаций и транслокаций, доминантно-негативных эффектов генных мутаций, латентных вирусных инфекций и других связанных симптомов, с использованием либо вирусных систем доставки, либо систем доставки на основе наночастиц.
План исследования Нуждающиеся в лечении субъекты или пациенты, страдающие от метаболических нарушений, амилоидоза и заболевания, связанного с агрегацией белков, включают без ограничения человека, не относящегося к человеку примата, собаку, кошку, корову, лошадь, других домашних животных и родственных млекопитающих. Система CRISPR-Cas направляется химерной направляющей РНК и оказывает целенаправленное воздействие на специфический сайт в локусах генома человека, которые нужно расщепить. После расщепления и опосредованной негомологичным соединением концов репарации, мутация сдвига рамки считывания приводит к нокауту генов.
Заявители выбирали направляющие РНК, оказывающие целенаправленное воздействие на гены, вовлеченные в вышеупомянутые нарушения, которые являются специфичными к эндогенным локусам при минимальной нецелевой активности. Две или более направляющих РНК могут быть закодированы в одном массиве CRISPR для индукции одновременных двухнитевых разрывов в ДНК, приводящих к микроделециям в подвергшихся воздействию генах или хромосомных участках.
Идентификация и конструирование генов-мишеней
Для каждого связанного с заболеванием кандидатного гена заявители выбирали представляющие интерес последовательности ДНК, включающие кодирующие белок экзоны, последовательности, включающие и фланкирующие известные сайты доминатно-негативных мутаций, последовательности, включающие и фланкирующие повторяющиеся последовательности, связанные с патологией. Для подходов, в которых используется нокаутирование генов, ранние кодирующие экзоны, наиболее близкие к старт-кодону, предлагают наилучшие варианты для достижения полного нокаута и сведения к минимуму возможности сохранения усеченными белковыми продуктами частичной функции.
Заявители анализировали представляющие интерес последовательности в отношении всех возможных поддающихся целенаправленному воздействию последовательностей из 20 п.о. непосредственно в 5' положении относительно мотива NGG (для системы SpCas9) или NNAGAAW (для системы St1Cas9). Заявители выбирали последовательности для исключительного направляемого одной РНК распознавания Cas9 в геноме для сведения к минимуму нецелевых эффектов на основе вычислительного алгоритма для определения специфичности.
Клонирование направляющих последовательностей в систему доставки
Направляющие последовательности синтезировали в воде двухнитевых олигонуклеотидов из 20-24 п.о. После обработки нуклеотидов 5'-фосфорилированием и гибридизации с образованием дуплексов, олигонуклеотиды лигируют в подходящий вектор в зависимости от способа доставки:
Способы доставки на основе вирусов
Векторы на основе AAV (PX260, 330, 334, 335) были описаны в других местах.
В векторах на основе лентивирусов используется подобная стратегия клонирования прямого дотирования направляющих последовательностей в один вектор, несущий каркас управляемой промотором U6 химерной РНК и управляемую промотором EF1a Cas9 или Cas9-никазу.
Получение вируса описано в других местах.
Способы доставки РНК на основе наночастиц
1. Направляющие последовательности синтезировали в виде дуплекса олигонуклеотидов, кодирующих промотор Т7 - направляющую последовательность - химерную РНК. Промотор Т7 добавляют в положении 5' относительно Cas9 с помощью способа ПЦР.
2. РНК управляемой Т7 Cas9 и химерные РНК направляющей последовательности транскрибировали in vitro, и мРНК Cas9 подвергали дополнительному копированию и присоединению А-хвоста с использованием коммерческих наборов. РНК-продукты очищали согласно инструкциям к наборам.
Способы гидродинамической доставки через хвостовую вену (для мыши)
Направляющие последовательности клонировали в плазмиды на основе AAV, которые описаны выше и в других местах в данной заявке.
In vitro валидация на клеточных линиях
Трансфекция
1. Трансфекция ДНК-плазмидами
Плазмиды, несущие направляющие последовательности, трансфицировали в клетки почки человеческого эмбриона (HEK293T) или человеческие эмбриональные стволовые (hES) клетки, другие подходящие типы клеток с использованием способов на основе липидов, химических средств или электропорации. Для трансфекции клеток HEK293T в 24-луночном формате (~260000 клеток) 500 нг общей ДНК трансфицировали в каждую отдельную лунку с использованием Lipofectamine 2000. Для трансфекции клеток hES в 12-луночном формате 1 мкг общей ДНК трансфицировали в отдельную лунку с использованием Fugene HD.
2. Трансфекция РНК
Описанную выше очищенную РНК использовали для трансфекции в клетки HEK293T. 1-2 мкг РНК можно трансфицировать в ~260000 с использованием Lipofectamine 2000 согласно инструкциям производителя. Доставка РНК Cas9 и химерной РНК показана на фигуре 28.
Анализ создания вставок/делеций in vitro
Клетки собирали через 72 часа после трансфекции и анализировали в отношении возникновения вставок/делеций в качестве признака двухнитевых разрывов.
Вкратце, участок генома вокруг целевой последовательности амплифицировали с помощью ПЦР (размер ампликона ~400-600 п.о.) с использованием высокоточной полимеразы. Продукты очищали, приводили к равной концентрации и медленно гибридизовали при температуре от 95°С до 4°С для обеспечения возможности образования гетеродуплексов ДНК. После отжига фермент Cel-I использовали для расщепления гетеродуплексов, и полученные в результате продукты разделяли на полиакриламидном геле, и рассчитывали эффективность создания вставок/делеций.
Подтверждение принципа данного изобретения у животных в модели in vivo
Механизмы доставки
Получение AAV или лентивируса описано в других местах.
Получение состава с наночастицами: РНК примешивали в состав с наночастицами.
Гидродинамические инъекции в хвостовую вену ДНК-плазмид у мышей проводили с использованием коммерческого набора.
Cas9 и направляющие последовательности доставляли в виде вируса, наночастиц, покрытых смесью РНК, или ДНК-плазмид и вводили инъекцией испытуемым животным. Параллельному набору контрольных животных вводили инъекцией стерильный солевой раствор, Cas9 и GFP или только направляющую последовательность и GFP.
Через три недели после инъекции животных исследовали в отношении ослабления симптомов и умерщвляли. Подходящие системы органов анализировали в отношении возникновения вставок/делений. Фенотипические анализы включают уровни HDL, LDL, липидов в крови.
Анализ в отношении возникновения вставок/делений
ДНК экстрагировали из ткани с использованием коммерческих наборов; анализ вставок/делеций будут выполнять, как описано для демонстрации in vitro.
Терапевтические применения системы CRISPR-Cas являются пригодными для достижения тканеспецифичной и контролируемой по времени целенаправленной делении в связанных с заболеванием кандидатных генах. Примеры включают гены, вовлеченные в метаболизм холестерина и жирных кислот, амилоидные заболевания, доминантно-негативные заболевания, латентные вирусные инфекции среди прочих нарушений.
Примеры отдельной направляющей РНК для введения целенаправленных вставок/делений в локус гена

Примеры пар направляющих РНК для введения хромосомной микроделеции в локус гена

Пример 19. Целенаправленная интеграция или репарация генов, несущих вызывающие заболевания мутации; восстановление недостаточностей ферментов и других связанных заболеваний
План исследования
I. Идентификация и конструирование генов-мишеней
- Описывается в примере 22
II. Клонирование направляющих последовательностей и матриц для репарации в систему доставки.
- Описывается выше в примере 22
- Заявители клонировали матрицы для репарации ДНК с включением гомологичных плечей с аллелем, вызывающим заболевание, а также с матрицей для репарации дикого типа.
III. In vitro валидация на клеточных линиях
а. Трансфекция описана выше в примере 22; Cas9, направляющие РНК и матрицы для репарации совместно трансфицируют в подходящие типы клеток.
b. In vitro анализ в отношении репарации
i. Заявители собирали клетки через 72 часа после трансфекции и анализировали в отношении репарации
ii. Вкратце, заявители амплифицировали участок генома около матрицы для репарации с помощью ПЦР с использованием высокоточной полимеразы. Заявители секвенировали продукты в отношении уменьшенной частоты обнаружения мутантного аллеля.
IV. Подтверждение принципа данного изобретения у животных в модели in vivo
а. Механизмы доставки описаны выше в примерах 22 и 34.
b. In vivo анализ в отношении репарации
i. Заявители осуществляли анализ репарации, как описано в демонстрации in vitro.
V. Терапевтические применения
Система CRISPR-Cas пригодна для достижения тканеспецифичной и контролируемой по времени целенаправленной делеции в связанных с заболеванием кандидатных генах. Примеры включают гены, вовлеченные в метаболизм холестерина и жирных кислот, амилоидные заболевания, доминантно-негативные заболевания, латентные вирусные инфекции среди прочих нарушений.
Пример одной отдельной миссенс-мутации с матрицей для репарации:
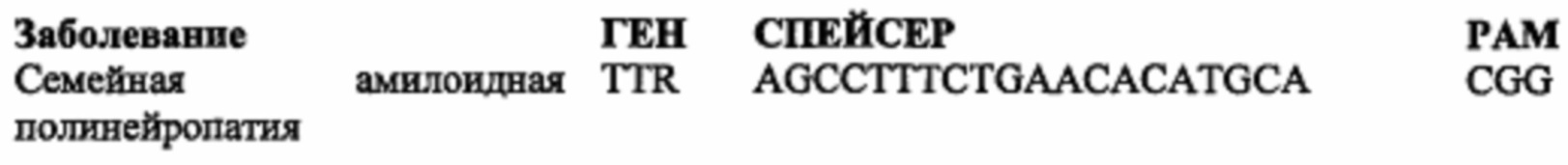

Пример 20. Терапевтическое применение системы CRISPR-Cas при глаукоме, амилоидозе и болезни Хантингтона
Глаукома: заявители сконструировали направляющие РНК для осуществления нацеливания на первый экзон гена миоциллина (MYOC). Заявители использовали аденовирусные векторы (Ad5) для упаковки и Cas9, и направляющей РНК, целенаправленно воздействующих на ген MYOC. Заявители вводили путем инъекции аденовирусные векторы в трабекулярную сеть, где находились клетки, вовлеченные в патофизиологию глаукомы. Заявители первоначально исследовали их в мышиных моделях, несущих мутированный ген MYOC, чтобы увидеть, будут ли они улучшать остроту зрения и снижать давление в глазах. При терапевтическом применении у людей используется подобная стратегия.
Амилоидоз: заявители сконструировали направляющие РНК для целенаправленного воздействия на первый экзон гена транстиретина (TTR) в печени. Заявители использовали AAV8 для упаковки Cas9, а также направляющей РНК, целенаправленно воздействующей на первый экзон гена TTR. Было показано, что AAV8 характеризуется эффективным целенаправленным воздействием в печени, и его будут вводить внутривенно. Экспрессией Cas9 можно управлять либо с использованием специфичных для печени промоторов, таких как альбуминовый промотор, либо с использованием конститутивного промотора. Промотор pol3 управляет экспрессией направляющей РНК.
В качестве альтернативы, заявители использовали гидродинамическую доставку плазмидной ДНК для нокаутирования гена TTR. Заявители доставляли плазмиду, кодирующую Cas9 и направляющую РНК, целенаправленно воздействующую на экзон 1 в TTR.
В качестве еще одного альтернативного подхода заявители вводили комбинацию РНК (мРНК для Cas9 и направляющую РНК). РНК можно упаковывать при помощи липосом, таких как Invivofectamine от Life Technologies, и доставлять внутривенно. Для снижения индуцированной РНК иммуногенности, повышения уровня экспрессия Cas9 и стабильности направляющей РНК заявители модифицировали мРНК Cas9 с помощью 5'-копирования. Заявители также вводили модифицированные нуклеотиды РНК в мРНК Cas9 и направляющую РНК для повышения их стабильности и снижения иммуногенности (например, активации TLR). Для повышения эффективности заявители вводили несколько доз вируса, ДНК или РНК.
Болезнь Хантингтона: заявители сконструировали направляющую РНК на основе аллель-специфичных мутаций в гене НТТ у пациентов. Например, у пациента, который является гетерозиготным по НТТ с расширенным CAG повтором, заявители идентифицировали нуклеотидные последовательности, уникальные для мутантного аллеля НТТ, и использовали их для конструирования направляющей РНК. Заявители убедились в том, что мутантное основание располагается в пределах последних 9 п.о. в направляющей РНК (которые, по убеждению заявителей, способны проводить различия по отдельным несовпадениям оснований ДНК между целевым сайтом и направляющей РНК).
Заявители упаковывали специфическую к мутантому аллелю НТТ направляющую РНК и Cas9 в AAV9 и доставляли в полосатое тело пациентов с болезнью Хантингтона. Вирус вводили в полосатое тело стереотаксически посредством краниотомии. Известно, что AAV9 эффективно преобразует нейроны. Заявители управляют экспрессией Cas9 с использованием нейрон-специфического промотора, такого как промотор гена синапсина I человека.
Пример 21. Терапевтическое применение системы CRISPR-Cas в HTV
Хроническая вирусная инфекция является источником значительной заболеваемости и смертности. Несмотря на то что для многих из этих вирусов существуют традиционные средства противовирусной терапии, которые эффективно целенаправленно воздействуют на различные аспекты вирусной репликации, используемые в настоящее время терапевтические воздействия обычно являются не излечивающими по своей природе вследствие "скрытого воздействия вируса". По своей природе, скрытое воздействие вируса характеризуется фазой покоя в жизненном цикле вируса без активного продуцирования вируса. Во время этого периода вирус в основном способен избегать как иммунного надзора, так и традиционных терапевтических средств, что обеспечивает создание долговременных резервуаров вируса в хозяине, из которых последующая повторная активация может давать возможность продолжающегося размножения и передачи вируса. Ключевой для скрытого воздействия вируса является возможность стабильного сохранения вирусного генома, осуществляемого за счет скрытности присутствия эписомной ДНК или провируса, которые хранят вирусный геном в цитоплазме или интегрируют его в геном хозяина, соответственно. В отсутствие эффективных вакцинаций, которые будут предотвращать первичную инфекцию, хронические вирусные инфекции, характеризующиеся скрытыми резервуарами и эпизодами литической активности, могут иметь существенные последствия: вирус папилломы человека (HPV) может приводить к раку шейки матки, вирус гепатита С (HCV) создает предрасположенность к гепатоцеллюлярной карциноме, и вирус иммунодефицита человека в конечном счете разрушает иммунную систему хозяина, приводя в результате к восприимчивости к оппортунистическим инфекциям. Вследствие этого эти инфекции требуют пожизненного применения доступных в настоящее время противовирусных терапевтических средств. Дополнительно осложняет ситуацию высокая мутабильность многих из этих вирусных геномов, которая приводит к развитию устойчивых штаммов, в отношении которых не существует эффективной терапии.
Система CRISPR-Cas представляет собой бактериальную систему адаптивного иммунитета, способную индуцировать двухнитевые разрывы ДНК (DSB) мультиплексно, специфично к последовательности, и которая была недавно воссоздана в виде систем внутри клетки млекопитающего. Было показано, что целенаправленное воздействие на ДНК одной или многочисленных направляющих РНК может приводить как к созданию вставок/делений, так и к удалениям являющихся помехой последовательностей, соответственно. Вследствие этого данная новая технология представляет средства, с помощью которых можно выполнять целенаправленный и мультиплексный мутагенез ДНК в пределах одной клетки с высокой эффективностью и специфичностью. Следовательно, доставка системы CRISPR-Cas, направленной против последовательности вирусной ДНК, может обеспечить возможность целенаправленного разрушения и удаления скрытых вирусных геномов даже в отсутствие протекающей вирусной инфекции.
В качестве примера хроническая инфекция HTV-1 представляет собой проблему для всемирного здравоохранения с инфицированными 33 миллионами людей и ежегодным возникновением 2,6 миллиона случаев инфицирования. Применение комбинированной высокоактивной антиретровирусной терапии (HAART), которая одновременно целенаправленно воздействует на различные аспекты репликации вируса, обеспечило возможность в большинстве случаев сдерживать развитие инфекции HIV до хронической, но не приводящей к смерти болезни. Без лечения прогрессирование HIV в AIDS обычно происходит в течение 9-10 лет, приводя к истощению иммунной системы хозяина и возникновению оппортунистических инфекций, обычно приводящих к. смерти вскоре после этого. На фоне скрытого воздействия вируса прерывание HAART неизменно приводит к вирусной отдаче. Более того, даже временные нарушения при терапии могут привести к отбору устойчивых штаммов HTV, не поддающихся контролю доступными средствами. Кроме того, затраты на HAART терапию являются существенными: в пределах 10000-150000 долларов США на человека в год. Вследствие этого подходы к лечению, непосредственно целенаправленно воздействующие на геном HTV, а не на процесс вирусной репликации, представляют средства, с помощью которых ликвидация скрытых резервуаров может обеспечить возможный метод лечения, позволяющий излечение.
Разработка и доставка целенаправленно воздействующей на HTV-1 системы CRISPR-Cas представляет уникальный подход, отличный от существующих средств целенаправленного мутагенеза ДНК, т.е. ZFN и TALEN, с многочисленными терапевтическими значениями. Целенаправленное разрушение и удаление генома HIV-1 с помощью опосредованного CRISPR DSB и вставки/делеции в сочетании с HAART могут обеспечить возможность одновременного предотвращения активного продуцирования вируса, а также истощения скрытых вирусных резервуаров в хозяине.
После интеграции в иммунную систему хозяина система CRISPR-Cas обеспечивает возможность создания устойчивой к HIV-1 субпопуляции, которая даже в отсутствие полной ликвидации вируса, может обеспечивать возможность поддержания и восстановления иммунной активности у хозяина. Она потенциально может предотвращать первичную инфекцию путем разрушения вирусного генома, предотвращая продуцирование и интеграцию вируса, представляя средства для "вакцинации". Мультиплексная природа системы CRISPR-Cas обеспечивает возможность целенаправленного воздействия на многие аспекты генома одновременно в пределах отдельных клеток.
Как и при HAART, формирование устойчивости вируса за счет мутаганеза сводится к минимуму из-за необходимости одновременного приобретения нескольких адаптивных мутаций. Многие штаммы HIV-1 могут подвергаться целенаправленному воздействию одновременно, что сводит к минимуму вероятность суперинфекции и предотвращает последующее возникновение новых рекомбинантных штаммов. Опосредуемая нуклеотидами, а не белком специфичность в отношении последовательности у системы CRISPR-Cas обеспечивает возможность быстрого создания терапевтических средств без необходимости существенного изменения механизма доставки.
Для выполнения этой задачи заявители создали направляющие РНК для CRISPR-Cas, которые осуществляли нацеливание на подавляющее большинство геномов HIV-1 с учетом вариантов штаммов HTV-1 для максимального охвата и эффективности. Анализы последовательности консервативности генома у подтипов и вариантов HTV-1 должен был обеспечивать возможность целенаправленного воздействия на фланкирующие консервативные участки в геноме с целью удаления лежащих между ними вирусных последовательностей или индукции мутаций со сдвигом рамки считывания, которые могут нарушать функции вирусных генов.
Заявители осуществляли доставку системы CRISPR-Cas с помощью общепринятой опосредованной аденовирусом или лентивирусом инфекции иммунной системы хозяина. В зависимости от подхода иммунные клетки хозяина могут быть а) выделены, трансдуцированы CRISPR-Cas, подвергнуты отбору и повторно введены хозяину; или b) трансдуцированы in vivo путем системной доставки системы CRISPR-Cas. Первый подход обеспечивает возможность получения популяции устойчивых иммунных клеток, тогда как второй, по всей вероятности, целенаправленно воздействует на скрытые резервуары вируса в хозяине.
Примеры потенциальных целенаправленно воздействующих на HIV-1 спейсеров, адаптированы, исходя из Mcintyre et al., которые создавали короткие шпилечные РНК (shRNA) против HIV-1, оптимизированные для максимального охвата вариантов HIV-1.

Пример 22. Целенаправленная коррекция дельтаF508 или других мутаций при муковисцидозе
Аспект настоящего изобретение предусматривает фармацевтическую композицию, которая может содержать частицы с CRISPR-Cas для генной терапии и биологически совместимый фармацевтический носитель. В соответствии с другим аспектом способ генной терапии для лечения субъекта, имеющего мутацию в гене CFTR, включает введение терапевтически эффективного количества частиц с CRISPR-Cas для генной терапии в клетки субъекта.
В данном примере демонстрируется перенос генов или доставка генов системы CRISPR-Cas в дыхательные пути нуждающегося в этом субъекта или пациента, страдающего от муковисцидоза или от связанных с муковисцидозом симптомов, с использованием частиц аденоассоциированного вируса (AAV).
План исследования Нуждающиеся во введении субъекты или пациенты: человек, не относящийся к человеку примат, собака, кошка, корова, лошадь, другие домашние животные, родственные животные. В данном исследовании изучали эффективность переноса генов системы CRISPR-Cas с помощью вектора на основе AAV. Заявители определяли уровни трансгена, достаточные для экспрессии гена, и использовали систему CRISPR-Cas, содержащую фермент Cas9, для целенаправленного воздействия на дельтаF508 или другие индуцирующие CFTR мутации.
Подвергающиеся лечению субъекты получали фармацевтически эффективное количество векторной системы на основе AAV в форме аэрозоля на легкое, доставляемое эндобронхиально при самостоятельном дыхании. Контрольные субъекты получали эквивалентное количество векторной системы на основе псевдотипированного AAV с внутренним контрольным геном. Векторную систему можно доставлять вместе с фармацевтически приемлемым или биологически совместимым фармацевтическим носителем. Через три недели или через подходящий временной интервал после введения вектора подвергающихся лечению субъектов исследовали в отношении ослабления связанных с муковисцидозом симптомов.
Заявители использовали аденовирусные частицы или частицы AAV.
Заявители клонировали конструкции со следующими генами, каждый из которых был функционально связан с одной или несколькими регуляторными последовательностями (промотор Cbh или EF1a для Cas9, промотор U6 или H1 для химерной направляющей РНК), в один или несколько векторов на основе аденовируса или AAV или в любой другой совместимый вектор: целенаправленно воздействующая на CFTR с мутацией дельта508 химерная направляющая РНК (фигура 31В), матрица для мутации дельтаF508 (фигура 31С) и кодон-оптимизированный фермент Cas9 необязательно с одним или несколькими сигналами или последовательностями ядерной локализации (NLS), например, с двумя (2) NLS.
Идентификация целевого сайта Cas9
Заявители анализировали локус генома CFTR человека и идентифицировали целевой сайт Cas9 (фигура 31А). (РАМ может содержать мотив NGG или NNAGAAW).
Стратегия репарации гена
Заявители вводили векторную систему на основе аденовируса/AAV, содержащую Cas9 (или Cas9-никазу) и направляющую РНК, вместе с векторной системой на основе аденовируса/AAV, содержащей матрицу для репарации путем гомологичной рекомбинации, содержащую остаток F508, субъекту посредством одного из обсуждаемых ранее способов доставки. Система CRISPR-Cas направлялась химерной направляющей РНК к CFTR с мутацией дельта508 и оказывала целенаправленное воздействие на специфический сайт в геномном локусе CFTR, подлежащий надрезанию или расщеплению. После расщепления матрицу для репарации вставляли в сайт расщепления посредством гомологичной рекомбинации, исправляющей делению, которая приводит к муковисцидозу или вызывает связанные с муковисцидозом симптомы. Данную стратегию для управления доставкой и обеспечения системного встраивания систем CRISPR с помощью соответствующих направляющих РНК можно использовать для целенаправленного воздействия на генные мутации, чтобы редактировать или проводить иного рода манипуляции с генами, которые вызывают метаболические заболевания и нарушения, заболевания и нарушения печени, почек и заболевания и нарушения, связанные с белками, такие как приведенные в таблице В.
Пример 23. Создание клеточной библиотеки с нокаутированными генами
В данном примере продемонстрировано, как создать библиотеку клеток, в которой каждая клетка имеет один нокаутированный ген.
Заявители получили библиотеку клеток ES, в которой каждая клетка имеет один нокаутированный ген, и в полной библиотеке клеток ES все без исключения гены будут нокаутированы. Эта библиотека применима для скрининга функции гена в клеточных процессах, а также в заболеваниях.
Для получения этой клеточной библиотеки заявители интегрировали Cas9, управляемый индуцируемым промотором (например, индуцируемым доксициклином промотором), в клетку ES. Кроме того, заявители интегрировали одну направляющую РНК, целенаправленно воздействующую на специфический ген в клетку ES. Для создания библиотеки клеток ES заявители просто смешивали ES с библиотекой генов, кодирующих направляющие РНК, которые осуществляют нацеливание на каждый ген в геноме человека. Сначала заявители вводили один сайт attB для ВхВ1 в локус AAVS1 в человеческих клетках ES. Затем заявители использовали интегразу ВхВ1 для облегчения интеграции отдельных генов направляющих РНК в сайт attB для ВхВ1 в локус AAVS1. Для облегчения интеграции каждый ген направляющей РНК размещался на плазмиде, которая несла один сайт attP. Таким образом, ВхВ1 будет рекомбинировать сайт attB в геноме с сайтом attP на плазмиде, содержащей направляющую РНК.
Для создания клеточной библиотеки заявители брали библиотеку клеток, которые имели отдельные интегрированные направляющие РНК, и индуцировали экспрессию Cas9. После индукции Cas9 опосредовала двухнитевой разрыв в сайтах, определенных направляющей РНК. Для подтверждения разнообразия этой клеточной библиотеки заявители выполняли секвенирование всего экзома для того, чтобы убедиться в том, что заявители способны наблюдать мутации в каждом отдельном подвергнутом целенаправленному воздействию гене. Данную клеточную библиотеку можно использовать для ряда применений, в том числе скрининг на основе целой библиотеки, или можно отсортировать в отдельные клоны клеток для облегчения быстрого создания клональных клеточных линий с нокаутированными отдельными генами человека.
Пример 24. Конструирование микроводорослей с использованием Cas9
Способы доставки Cas9
Способ 1: заявители доставляли Cas9 и направляющую РНК с использованием вектора, который экспрессирует Cas9 под контролем конститутивного промотора, такого как промотор Hsp70A-Rbc S2 или бета-2-тубулиновый промотор.
Способ 2: заявители доставляли Cas9 и полимеразу Т7 с использованием векторов, которые экспрессируют Cas9 и полимеразу Т7 под контролем конститутивного промотора, такого как промотор Hsp70A-Rbc S2 или бета-2-тубулиновый промотор. Направляющая РНК будет доставляться с использованием вектора, содержащего промотор Т7, управляющий экспрессией направляющей РНК.
Способ 3: заявители доставляли мРНК Cas9 и in vitro транскрибировали направляющую РНК в клетках водорослей. РНК можно транскрибировать in vitro. мРНК Cas9 будет состоять из кодирующего участка для Cas9, а также 3'UTR из Сор1, чтобы обеспечивать стабилизацию мРНК Cas9.
Для гомологичной рекомбинации заявители обеспечивали дополнительную матрицу для репарации с участием гомологичной рекомбинации.
Последовательность для кассеты, управляющей экспрессией Cas9 под контролем бета-2-тубулинового промотора, за которой следует 3' UTR Сор1.




Последовательность для кассеты, управляющей экспрессией полимеразы Т7 под контролем бета-2-тубулинового промотора, за которой следует 3' UTR Cop1:


Последовательность направляющей РНК, управляемая промотором Т7 (промотор Т7, N представляют нацеливающую последовательность):

Доставка генов
Штаммы СС-124 и СС-125 Chlamydomonas reinhardtii из Ресурсного центра Chlamydomonas (Chlamydomonas Resource Center) будут использоваться для электропорации. Протокол электропорации соответствует стандартному рекомендованному протоколу для набора GeneArt Chlamydomonas Engineering kit.
Заявители также получали линию Chlamydomonas reinhardtii, которая экспрессирует Cas9 конститутивно. Это можно выполнить при помощи pChlarny1 (линеаризованная с использованием PvuI) и отбора в отношении устойчивых к гигромицину колоний. Последовательность для pChlarny1, содержащая Cas9, приведена ниже. В данном пути для достижения нокаутирования гена специалисту необходимо просто доставить РНК для направляющей РНК. Для гомологичной рекомбинации заявители доставляли направляющую РНК, а также линеаризованную матрицу для гомологичной рекомбинации.
pChlamy1-Cas9:





Для всех модифицированных клеток Chlamydomonas reinhardtii заявители использовали ПЦР, анализ с помощью нуклеазы SURVEYOR и секвенирование ДНК для подтверждения успешной модификации.
Пример 25. Применение Cas9 для целенаправленного воздействия на разнообразные типы заболеваний
Заболевания, в которые вовлечены мутации в кодирующей последовательности белка
Целенаправленное воздействие на доминантные нарушения может оказываться путем инактивации доминантно-негативного аллеля. Заявители использовали Cas9 для целенаправленного воздействия на уникальную последовательность доминантно-негативного аллеля и введения мутации посредством NHEJ. Индуцированная NHEJ вставка/деления может быть способна к введению мутация сдвига рамки считывания в доминантно-негативном аллеле и устранению доминантно-негативного белка. Это может функционировать, если ген является гаплодостаточным (например, вызванная мутацией MYOC глаукома и болезнь Хантингтона).
Целенаправленное воздействие на рецессивные нарушения может оказываться путем репарации связанной с заболеванием мутации в обоих аллелях. Для делящихся клеток заявители использовали Cas9 для введения двухнитевых разрывов около сайта с мутацией и повышения скорости гомологичной рекомбинации с использованием экзогенной матрицы для рекомбинации. Для делящихся клеток этого можно достичь с использованием фермента с мультиплексной никазной активностью для катализа замены мутантной последовательности в обоих аллелях с помощью опосредованного NHEJ лигирования фрагмента экзогенной ДНК, несущего комплементарные "липкие" концы.
Заявители также применяли Cas9 для введения защитных мутаций (например, инактивации CCR5 для предупреждения инфекции HTV, инактивации PCSK9 для снижения уровня холестерина или введение А673Т в АРР для снижения вероятности возникновения болезни Альцгеймера).
Заболевания, в которые вовлечены некодирующие последовательности
Заявители применяли Cas9 для разрушения некодирующих последовательностей в промоторном участке, для изменения сайтов связывания транскрипционных факторов и для изменения энхансерных или репрессорных элементов. Например, Cas9 можно использовать для вырезания энхансера Klf1 для EHS1 в гемопоэтических стволовых клетках для снижения уровней BCL11a и повторной активации экспрессии гена фетального глобина в дифференцированных эритроцитах.
Заявители также применяли Cas9 для разрушения функциональных мотивов в 5' или 3' нетранслируемых участках. Например, для лечения миотонической дистрофии Cas9 можно использовать для удаления избыточных CTG повторов в гене DMPK.
Пример 26. Мультиплексная никаза
Аспекты оптимизации и информация о Cas9, подробно описанные в данной заявке, также можно использовать для создания Cas9-никаз. Заявители использовали Саз9-никазы в комбинации с парами направляющих РНК для создания двухнитевых разрывов ДНК с определенными "липкими" концами. Если используют две пары направляющих РНК, представляется возможным вырезание лежащего между ними фрагмента ДНК. Если экзогенный фрагмент ДНК расщепляется двумя парами направляющих РНК с созданием совместимых "липких" концов с геномной ДНК, то экзогенный фрагмент ДНК можно литеровать в геномную ДНК с заменой вырезаемого фрагмента. Например, это можно использовать для удаления дополнительных избыточных трехнуклеотидных повторов в гене хантинина (НТТ) для лечения болезни Хантингтона.
Если обеспечивают экзогенную ДНК, которая несет меньшее число повторов CAG, то представляется возможным получение фрагмента ДНК, который несет такие же "липкие" концы и который можно лигировать в геномный локус НТТ и заменить им вырезаемый фрагмент.

Лигирование экзогенного фрагмента ДНК в геноме не требует аппаратов для гомологичной рекомбинации и, таким образом, этот способ можно применять у клеток в постмитотическом периоде, таких как нейроны.
Пример 27. Доставка системы CMSPR
Cas9 и ее химерную направляющую РНК или комбинацию tracrRNA и crRNA можно доставлять в виде либо ДНК, либо РНК. Доставка и Cas9, и направляющей РНК в виде молекул РНК (нормальной или содержащей модификации оснований или остова) можно использовать для уменьшения количества времени, в течение которого белок Cas9 продолжает находиться в клетке. Это может снижать уровень активности в отношении нецелевого расщепления в клетке мишени. Поскольку доставка Cas9 в виде мРНК требует времени для трансляции в белок, преимущественной может быть доставка направляющей РНК через несколько часов после доставки мРНК Cas9 для максимального повышения уровня направляющей РНК, доступной для взаимодействия с белком Cas9.
В ситуациях, когда количество направляющей РНК ограничено, желательным может быть введение Cas9 в виде мРНК, а направляющей РНК в виде кассеты экспрессии ДНК с промотором, управляющим экспрессией направляющей РНК. Таким образом, количество доступной направляющей РНК будет амплифицироваться посредством транскрипции.
Для введения Cas9 (ДНК или РНК) и направляющей РНК (ДНК или РНК) в клетки-хозяева можно использовать ряд систем для доставки. Они включают применение липосом, вирусных векторов, электропорации, наночастиц, нанопроволок (Shalek et al., Nano Letters, 2012), экзосом. Липосомы по типу "молекулярного троянского коня" (Pardridge et al., Cold Spring Harb Protoc; 2010; doi: 10.1101/pdb.prot5407) можно использовать для доставки Cas9 и направляющей РНК через гематоэнцефалический барьер.
Пример 28. Терапевтические стратегии для нарушений, связанных с трехнуклеотидными повторами
Как ранее упоминалось в данной заявке, целевой полинуклеотид комплекса CRISPR может включать ряд связанных с заболеванием генов, и полинуклеотиды и некоторые из этих связанных с заболеванием генов могут принадлежать к набору генетических нарушений, называемых нарушениями, связанными с трехнуклеотидными повторами (также называемых нарушениями, связанными с дополнительными избыточными трехнуклеотидными повторами, нарушениями, связанными с дополнительными избыточными тройными повторами, или нарушениями, связанными с реитерапией кодонов).
Эти нарушения вызваны мутациями, при которых число трехнуклеотидных повторов в некоторых генах превышает нормальное стабильное пороговое значение, которое обычно может отличаться у гена. Открытие большего числа нарушений, связанных с дополнительными избыточными повторами, обеспечило возможность классификации этих нарушений на ряд категорий, исходя из лежащих в их основе подобных характеристик. Болезнь Хантингтона (HD) и спиноцеребеллярная атаксия, которые вызваны дополнительным избыточным повтором CAG в кодирующих белок частях конкретных генов, включены в категорию I. В категорию II включены заболевания или нарушения с дополнительными избыточными повторами, которые, как правило, делают их фенотипически разнообразными и включают дополнительные избыточные повторы, размер которых обычно невелик, и которые часто обнаруживаются в экзонах генов. Категория III включает нарушения или заболевания, которые характеризуются гораздо большим числом дополнительных повторов, чем любые нарушений или заболеваний из категории I или II, и обычно расположены за пределами кодирующих белок участков. Примеры заболеваний или нарушений из категории III включают без ограничения синдром ломкой Х-хромосомы, миотоническую дистрофию, две спиноцеребеллярные атаксии, ювенильную миоклоническую эпилепсию и атаксию Фридрейха.
Подобные терапевтические стратегии, такие как упоминаемые ниже для атаксии Фридрейха, можно также заимствовать, чтобы направлять на другие нарушения, связанные с трехнуклеотидными повторами или с дополнительными избыточными повторами. Например, другим заболеванием, связанным с дополнительными избыточными тройными повторами, которое можно лечить с использованием почти идентичной стратегии, является миотоническая дистрофия 1 (DM1), при которой присутствует дополнительный избыточный мотив CTG в 3' UTR. При атаксии Фридрейха заболевание является результатом дополнительных избыточных трех нуклеотидов GAA в первом интроне фратаксина (FXN). Одной терапевтической стратегией применения CRISPR является вырезание повтора GAA из первого интрона. Как полагают, дополнительный избыточный повтор GAA воздействует на структуру ДНК и приводит к мобилизации образования гетерохроматина, что выключает ген фратаксина (фигура 32А).
Преимущества, обеспечивающие конкурентноспособность по сравнению с другими терапевтическими стратегиями, перечислены ниже.
Нокдаун с использованием siRNA в этом случае не доступен, поскольку заболевание обусловлено пониженной экспрессией фратаксина. В настоящее время исследуется генная терапия с использованием вирусов. Векторы на основе HSV-1 использовали для доставки гена фратаксина в моделях на животных, и они продемонстрировали терапевтический эффект. Тем не менее,. долговременная эффективность доставки фратаксина на основе вируса подвержена нескольким проблемам: во-первых, сложно регулировать экспрессию фратаксина, чтобы она совпадала с естественными уровнями у здоровых индивидов, и во-вторых, долговременная сверхэкспрессия фратаксина приводит к гибели клеток.
Нуклеазы можно использовать для вырезания повтора GAA для восстановления здорового генотипа, но стратегии с использованием нуклеазы с доменом "цинковые пальцы" и TALEN требуют доставки двух пар высокоэффективных нуклеаз, что является сложным как в отношении доставки, так и конструирования нуклеаз (сложно достичь эффективного вырезания геномной ДНК с помощью ZFN или TALEN).
В отличие от приведенных выше стратегий, система CRISPR-Cas имеет явные преимущества. Фермент Cas9 является более эффективным и более мультиплексным, под данным утверждением подразумевается, что он одновременно может быть настроен на одну или несколько мишеней. На сегодняшний день вырезание геномной ДНК с помощью Cas9 в клетках человека обеспечивается с эффективностью >30%, и эффективность может составлять вплоть до 30%, а также она может быть улучшена в будущем. Более того, в отношении некоторых нарушений, связанных с трехнуклеотидными повторами, подобных болезни Хантингтона (HD), трехнуклеотидные повторы в кодирующем участке можно рассматривать, если существуют различия между двумя аллелями. В частности, если пациент с HD является гетерозиготным по мутантному НТТ, и существуют различия по нуклеотидам, такие как SNP между wt и мутантными аллелями НТТ, то Cas9 можно использовать для специфического целенаправленного воздействия на мутантный аллель НТТ. ZFN или TALEN не будут иметь возможности проведения различий между двумя аллелями, исходя из различий по одному основанию.
При заимствовании стратегии применения фермента CRISPR-Cas9 для направления на атаксию Фридрейха, заявители сконструировали ряд направляющих РНК, которые осуществляют нацеливание на сайты, фланкирующие дополнительные избыточные GAA, и выбирали наиболее эффективные и специфичные из них (фигура 32В).
Заявители доставляли комбинацию направляющих РНК, осуществляющих нацеливание на интрон 1 в FXN, вместе с Cas9 для опосредования вырезания участка с дополнительным избыточным GAA. AAV9 можно использовать для опосредования эффективной доставки Cas9 и в спинной мозг.
Если Alu-элемент, смежный с дополнительным избыточным GAA, считается важным, могут существовать ограничения по числу сайтов, на которые можно целенаправленно воздействовать, но заявители могут применять стратегии во избежание его нарушения.
Альтернативные стратегии
Вместо того чтобы модифицировать геном, используя Cas9, заявители могут также непосредственно активировать ген FXN с использованием ДНК-связывающего домена на основе Cas9 (лишенной нуклеазной активности) для целенаправленного воздействия активирующего транскрипцию домена на ген FXN. Возможно, заявители должны уделить внимание надежности опосредованной Cas9 искусственной активации транскрипции, чтобы обеспечить ее достаточную надежность по сравнению с другими способами (Tremblay et al., Transcription Activator-Like Effector Proteins Induce the Expression of the Frataxin Gene; Human Gene Therapy. August 2012, 23(8); 883-890.)
Пример 29. Стратегии сведения к минимуму нецелевого расщепления при использовании Cas9-никазы
Как упоминалось выше в данной заявке, Cas9 можно подвергнуть мутации для опосредования расщепления одной нити с помощью одной или нескольких из следующих мутаций: D10A, Е762А и Н840А.
Для опосредования нокаутирования гена с помощью NHEJ заявители использовали никазный вариант Cas9 вместе с двумя направляющими РНК. Нецелевое надрезание каждой отдельной направляющей РНК преимущественно может репарироваться без мутации, двухнитевые разрывы (которые могут привести к мутациям посредством NHEJ) возникают только тогда, когда целевые сайты являются смежными друг с другом. Поскольку двухнитевые разрывы, вводимые при двойном надрезании не являются "затупленными", коэкспрессия ферментов, осуществляющих процессинг концевых участков, таких как TREX1, будет повышать уровень активности NHEJ.
Следующий перечень мишеней в форме таблицы представлен для генов, вовлеченных в следующие заболевания:
болезнь Лафора - целенаправленное воздействие на GSY1 или PPP1R3C (PTG) для снижения уровня гликогена в нейронах.
Гиперхолестеринемия - целенаправленное воздействие на PCSK9.
Целевые последовательности приведены попарно (L и R) с различным числом нуклеотидов в спейсере (0-3 п.о.). Каждый спейсер можно использовать сам по себе с Cas9 дикого типа для введения двухнитевого разрыва в целевом локусе.


Альтернативные стратегии для улучшения стабильности направляющей РНК и повышения специфичности
1. Нуклеотиды в направлении 5' относительно направляющей РНК можно соединить посредством тиолэфирных связей, а не фосфоэфирных связей, как в натуральной РНК. Тиолэфирная связь может предотвращать расщепление направляющей РНК эндогенным аппаратом для разрушения РНК.
2. В качестве нуклеотидов в направляющей последовательности (20 п.о. в направлении 5') направляющей РНК можно использовать мостиковые нуклеиновые кислоты (BNA) в качестве оснований для улучшения специфичности связывания.
Пример 30. CRISPR-Cas для быстрого мультиплексного редактирования генома
Аспекты настоящего изобретения относятся к протоколам и способам, с помощью которых можно определять эффективность и специфичность модификации генов в пределах 3-4 дней после конструирования мишени, а клональные линии модифицированных клеток можно получить в пределах 2-3 недель.
Программируемые нуклеазы представляют собой эффективные технологии для опосредования изменения генома с высокой точностью. Направляемую РНК нуклеазу Cas9 из микробной системы CRISPR адаптивного иммунитета можно использовать для облегчения эффективного редактирования генома в эукариотических клетках путем простого определения 20-нуклеотидной нацеливающей последовательности в ее направляющей РНК. Заявители описывают набор протоколов для применения Cas9, чтобы облегчить эффективное редактирование генома в клетках млекопитающих и создавать клеточные линии для последующих функциональных исследований. Начиная с конструирования мишени, эффективной и специфичной модификации гена можно достичь в пределах 3-4 дней, а клональные линии модифицированных клеток можно получить в пределах 2-3 недель.
Возможность конструирования биологических систем и организмов имеет огромный потенциал для применений в фундаментальной науке, медицине и биотехнологии. Программируемые специфичные в отношении последовательности эндонуклеазы, которые облегчают точное редактирование эндогенных локусов генома, теперь дают возможность систематического формирования запросов в отношении генетических элементов и каузальных генетических изменений у широкого спектра видов, в том числе у тех, которые ранее не поддавались обработке генетическими способами. В последние годы возник ряд технологий редактирования генома, в том числе нуклеазы с доменом "цинковые пальцы" (ZFN), подобные транскрипционным активаторам эффекторные нуклеазы (TALEN) и направляемая РНК нуклеазная система CRISPR-Cas. В первых двух технологиях используется традиционная стратегия связывания эндонуклеазных каталитических доменов с модулярными ДНК-связывающими белками для индукции целенаправленных двухнитевых разрывов (DSB) ДНК в конкретных локусах генома, В отличие от этого, Cas9 представляет собой нуклеазу, направляемую небольшими РНК посредством спаривания оснований по Уотсон-Крику с целевой ДНК, представляя систему, которая является простой для конструирования эффективной и хорошо подходящей для высокопроизводительного и мультиплексного редактирования генов для ряда клеточных типов и организмов. В данном документе заявители описывают набор протоколов для применения недавно разработанной нуклеазы Cas9, чтобы облегчить эффективное редактирование генома в клетках млекопитающих и создавать клеточные линии для последующих функциональных исследований.
Подобно ZFN и TALEN Cas9 способствует редактированию генома путем стимуляции DSB в целевых локусах генома. При расщеплении Cas9 целевой локус подвергается одному из двух основных путей репарации повреждения ДНК, пути склонного к ошибкам негомологичпого соединения концов (NHEJ) или пути высокоточной репарации посредством гомологичной рекомбинации (HDR). Оба пути можно использовать для достижения желаемого результата редактирования.
NHEJ: в отсутствие матрицы для репарации в процессе NHEJ происходит повторное лигирование DSB, которое может оставлять "рубец" в виде мутаций по типу вставка/делеция. Данный способ можно использовать для достижения нокаутирования генов, поскольку вставки/делении, возникающие в пределах кодирующего экзона, могут приводить к мутациям со сдвигом рамки считывания и преждевременному стоп-кодону. Множественные DSB также можно использовать для опосредования делений большего размера в геноме.
HDR: репарация с участием гомологичной рекомбинации является альтернативным NHEJ основным путем репарации ДНК. Хотя HDR обычно происходит с более низкими частотами, чем NHEJ, его можно использовать для создания точных определенных модификаций в целевом локусе в присутствии экзогенно введенной матрицы для репарации. Матрица для репарации может присутствовать либо в виде двухнитевой ДНК, сконструированной подобно общепринятым целенаправленно воздействующим на ДНК конструкциям с гомологичными плечами, фланкирующими последовательность вставки, либо в виде однонитевых олигонуклеотидов ДНК (ssODN). Последние обеспечивают эффективный и простой способ создания небольших редакционных изменений в геноме, таких как введение однонуклеотидных мутаций для зондирования каузальных генетических изменений. В отличие от NHEJ, HDR обычно активен только в делящихся клетках, и его эффективность варьирует в зависимости от типа и состояния клетки.
Обзор CRISPR: система CRISPR-Cas, напротив, является как минимум двухкомпопентной системой, состоящей из нуклеазы Cas9 и короткой направляющей РНК. Для повторного целенаправленного воздействия Cas9 на различные локусы или одновременного редактирования нескольких генов требуется просто клонировать другой олигонуклеотид размером 20 п.о. Хотя специфичность нуклеазы Cas9 подробно еще не освещалась, простое спаривание оснований Уотсон-Крику в системе CRISPR-Cas, вероятно, является более предсказуемым, нежели у доменов ZFN или TALEN.
CRISPR-Cas типа II (короткие палиндромные повторы, регулярно расположенные группами) представляет сбой бактериальную систему адаптивного иммунитета, в которой Cas9 используется для расщепления чужеродных генетических элементов. Cas9 направляется парой некодирующих РНК, переменной crRNA и необходимой вспомогательной tracrRNA. CrRNA содержит направляющую последовательность из 20 нуклеотидов, определяющую специфичность по расположению целевой ДНК посредством спаривания оснований по Уотсон-Крику. В нативной бактериальной системе несколько crRNA совместно транскрибируются для направления Cas9 па различные мишени. В системе CRISPR-Cas, полученной из Streptococcus pyogenes, целевая ДНК должна находиться непосредственно перед 5'-NGG/NRG мотивом, смежным с протоспейсером (РАМ), которым может изменяться для других систем CRISPR.
CRISPR-Cas воспроизводят в клетках млекопитающих посредством гетерологичной экспрессии кодон-оптимизированного для человека Cas9 и необходимых компонентов РНК. Более того, crRNA и tracrRNA можно слить для создания химерной синтетической направляющей РНК (sgRNA). Cas9 можно, таким образом, перенаправить на любую представляющую интерес мишень путем изменения направляющей последовательности размером 20 нуклеотидов в sgRNA.
С учетом простоты использования и способности к мультиплексному действию Cas9 использовали для получения сконструированных эукариотических клеток, несущих специфические мутации, как посредством NHEJ, так и HDR. Кроме того, прямая инъекция sgRNA и mRNA, кодирующей Cas9, в эмбрионы обеспечила возможность быстрого получения трансгенных мышей с несколькими модифицированными аллелями; это дает перспективу для редактирования организмов, которые в ином случае не поддаются обработке генетическими способами.
Мутантная Cas9, несущая нарушение в одном из своих каталитических доменов, была сконструирована для надрезания, а не для расщепления ДНК, что обеспечивает возможность однонитевых разрывов и преимущественной репарации посредством HDR, потенциально нейтрализуя нежелательные мутации по типу вставки/делении в результате нецелевых DSB. Кроме того, мутант Cas9 с мутированными обоими расщепляющими ДНК каталитическими остатками был адаптирован для обеспечения возможности регуляции транскрипции у Е.coli, демонстрируя потенциал функционализации Cas9 для разнообразных применений. Определенные аспекты настоящего изобретения относятся к конструированию и применению Cas9 для мультиплексного редактирования клеток человека.
Заявители обеспечили кодон-оптимизированный для человека, фланкированный последовательностью ядерной локализации Cas9 для облегчения редактирования генов эукариот. Заявители описали факторы, которые следует учитывать при конструировании направляющей последовательности из 20 нуклеотидов, протоколы для быстрого конструирования и функциональной валидации sgRNA и, наконец, применение нуклеазы Cas9 для опосредования модификаций генома как на основе NHEJ, так и HDR в линиях клеток почки человеческого эмбриона (HEK-293FT) и стволовых клеток человека (HUES9). Этот протокол можно аналогичным образом применить к другим типам клеток и организмам.
Выбор мишени для sgRNA: существуют два главных фактора, которые необходимо учитывать при выборе направляющей последовательности из 20 нуклеотидов для целенаправленного воздействия на ген: 1) целевая последовательность должна находиться перед 5'-NGG РАМ для Cas9 S. pyogenes и 2) направляющие последовательности следует выбирать со сведением к минимуму нецелевой активности. Заявители обеспечили инструмент конструирования для нацеливания Cas9 в режиме онлайн, который использует вводимую представляющую интерес последовательность и идентифицирует подходящие целевые сайты. Для экспериментальной оценки нецелевых модификаций для каждой sgRNA заявители также обеспечили спрогнозированные путем вычислений нецелевые сайты для каждой предполагаемой мишени, ранжировали согласно проведенному заявителями количественному анализу специфичности в отношении эффектов идентичности, положения и распределения несовпадений при спаривании оснований.
Подробная информация по спрогнозированным путем вычислений нецелевым сайтам выглядит следующим образом:
Факторы, которые следует учитывать для активностей в отношении нецелевого расщепления: подобно другим нуклеазам, Cas9 может расщеплять нецелевые ДНК-мишени в геноме с пониженной частотой. Степень, с которой заданная направляющая последовательность проявляет нецелевую активность, зависит от комбинации факторов, включающих концентрацию фермента, термодинамические характеристики конкретной используемой направляющей последовательности и распространенности подобных последовательностей в целевом геноме. Для стандартного применения Cas9 важно учитывать пути для сведения к минимуму степени нецелевого расщепления, а также для способности выявлять наличие нецелевого расщепления.
Сведение к минимуму нецелевой активности: для применения в клеточных линиях заявители рекомендовали следующие две стадии для снижения степени нецелевой модификации генома. Во-первых, при использовании разработанного заявителями онлайн-инструмента для выбора мишени CRISPR возможно путем вычислений оценить вероятность того, что данная направляющая последовательность имеет нецелевые сайты. Эти анализы осуществляют посредством поиска методом полного перебора в геноме в отношении нецелевых последовательностей, которые представляют собой последовательности, подобные направляющей последовательности. Всестороннее экспериментальное исследование эффекта несовпадающих оснований между sgRNA и ее целевой ДНК выявило, что допустимость несовпадения является 1) зависимой от положения - 8-14 п.о. на 3' конце направляющей последовательности несовпадения являются менее допустимыми, чем у 5' оснований; 2) зависимой от количества - обычно не допускаются более 3 несовпадений; 3) зависимой от направляющей последовательности - в некоторых направляющих последовательностях несовпадения являются менее допустимыми, чем в других; и 4) зависимой от концентрации - нецелевое расщепление высоко чувствительно к количеству трансфицированной ДНК. Разработанный заявителями веб-инструмент для анализа целевых сайтов (доступный на веб-сайте genome-engineering.org/tools) объединяет эти критерии для обеспечения прогнозов вероятных нецелевых сайтов в целевом геноме. Во-вторых, заявители рекомендовали подбор количества Cas9 и плазмиды для экспрессии sgRNA для сведения к минимуму нецелевой активности.
Выявление нецелевых активностей: используя разработанный заявителями веб-инструмент для нацеливания CRISPR, возможно создать перечень наиболее вероятных нецелевых сайтов, а также праймеров, осуществляя анализ данных сайтов с помощью SURVEYOR или секвенирования. Для изогенных клонов, полученных с использованием Cas9, заявители настоятельно рекомендуют секвенирование этих кандидатных нецелевых сайтов для проверки в отношении любых нежелательных мутаций. Стоит отметить, что в сайтах, которые не включены в перечень прогнозируемых кандидатов, могут присутствовать нецелевые модификации, и полногеномное секвенирование следует проводить для полного подтверждения отсутствия нецелевых сайтов. Более того, в мультиплексных анализах, в которых несколько DSB индуцируют в одном и том же геноме, могут существовать низкие скорости событий транслокации, и их можно оценить с использованием ряда методик, таких как глубокое секвенирование.
Онлайн-инструмент обеспечивает последовательности для всех олигонуклеотидов и праймеров, необходимых для 1) получения конструкций sgRNA, 2) анализа эффективности целевой модификации и 3) оценки расщепления в потенциальных нецелевых сайтах. Стоит отметить, что вследствие того, что промотор U6 РНК полимеразы III, используемый для экспрессии sgRNA, предпочитает гуаниновый (G) нуклеотид в качестве первого основания в его транскрипте, дополнительный G прикрепляют на 5' в sgRNA в том случае, когда направляющая последовательность из 20 нуклеотидов не начинается с G.
Подходы к конструированию и доставке sgRNA: в зависимости от желаемого применения sgRNA можно доставлять либо в виде 1) ПЦР ампликонов, содержащих кассету экспрессии, либо в виде 2) экспрессирующих sgRNA плазмид. При доставке sgRNA на основе ПЦР прикрепляют последовательность специальной sgRNA на обратный ПЦР праймер, используемый для амплификации матрицы промотора U6. Полученный в результате ампликон можно совместно трансфицировать с плазмидой, содержащей Cas9 (РХ165). Этот способ является оптимальным для быстрого скрининга нескольких кандидатных sgRNA, поскольку процедуры трансфекции клеток для функционального исследования можно осуществлять всего лишь спустя часы после получения кодирующих sgRNA праймеров. Поскольку этот простой способ избегает потребности в клонировании с использованием плазмид и подтверждении последовательности, он хорошо подходит для исследования или совместной трансфекции большого количества sgRNA для создания больших библиотек нокаутных генов или для других чувствительных к масштабу применений. Следует обратить внимание, что кодирующие sgRNA праймеры имеют длину более 100 п.о. по сравнению с олигонуклеотидами длиной ~20 п.о., требующимися для доставки sgRNA с использованием плазмид.
Конструирование плазмид для экспрессии sgRNA также является простым и быстрым, включая одну стадию клонирования с парой частично комплементарных олигонуклеотидов. После гибридизации пар олигонуклеотидов полученные в результате направляющие последовательности можно вставить в плазмиду, несущую как Cas9, так и неизменяемый каркас, несущий остальную часть последовательности sgRNA (РХ330). Плазмиды для трансфекции также можно модифицировать для обеспечения возможности получения вируса для доставки in vivo.
Помимо ПЦР и способов доставки с использованием плазмид как Cas9, так и sgRNA можно вводить в клетки в виде РНК.
Конструирование матрицы для репарации: традиционно, целенаправленные модификации ДНК требовали применения донорских матриц для репарации на основе плазмид, которые содержат гомологичные плечи, фланкирующие сайт изменения. Гомологичные плечи с каждой стороны могут варьировать по длине, но, как правило, длиннее 500 п.о. Этот способ можно применять для создания модификаций большого размера, в том числе вставок репортерных генов, таких как флуоресцентные белки или маркеры, обеспечивающие устойчивость к антибиотикам. Создание и конструирование плазмид для целенаправленного воздействия было описано в других местах в данном документе.
В последнее время однонитевые олигонуклеотиды ДНК (ssODN) использовали вместо плазмид для целенаправленного воздействия для коротких модификаций в пределах определенного локуса без клонирования. Для достижения высоких эффективностей HDR ssODN содержат фланкирующие последовательности по меньшей мере из 40 п.о. с каждой стороны, которые являются гомологичными целевому участку, и которые могут быть ориентированы либо в смысловом, либо в антисмысловом направлении относительно целевого локуса.
Функциональное исследование
Анализ с помощью нуклеазы SURVEYOR: заявители выявляли мутации по типу вставки/делении либо посредством анализа с помощью нуклеазы SURVEYOR, либо посредством секвенирования ПЦР ампликона. Разработанный заявителями онлайн-инструмент для нацеливания CRISPR обеспечивает рекомендованные праймеры для обоих подходов. Тем не менее, праймеры, полученные с помощью SURVEYOR или секвенирования, также можно сконструировать вручную для амплификации представляющего интерес участка из геномной ДНК и во избежание неспецифических ампликонов с использованием Primer-BLAST от NCBI. Полученные с помощью SURVEYOR праймеры должны быть сконструированы для амплификации 300-400 п.о. (для общего ампликона 600-800 п.о.) с каждой стороны мишени Cas9, чтобы обеспечить возможность четкой визуализации соответствующих результатам расщепления полос с помощью гель-электрофореза. Для предотвращения избыточного образования димеров праймеров полученные с помощью SURVEYOR праймеры должны быть сконструированы таким образом, чтобы они, как правило, имели длину до 25 нуклеотидов с температурами плавления ~60°С; Заявители рекомендуют исследование каждой пары праймеров-кандидатов в отношении специфических ПЦР ампликонов, а также в отношении отсутствия неспецифического расщепления во время процесса расщепления нуклеазрй SURVEYOR.
HDR, опосредованная плазмидой или ssODN: HDR можно выявить с помощью ПЦР амплификации и секвенирования модифицированного участка. ПЦР праймеры для этой цели должны гибридизоваться за пределами участка, охватываемого гомологичными плечами, во избежание ложного выявления остаточной матрицы для репарации (HDR Fwd и Rev, фигура 30). Для опосредованной ssODN HDR можно использовать ПЦР праймеры, полученные с помощью SURVEYOR.
Выявление вставок/делений или HDR с помощью секвенирования: заявители выявляли целенаправленные модификации генома либо с помощью секвенирования по Сэнгеру, либо с помощью глубокого секвенирования нового поколения (NGS). Что касается первого из вышеупомянутых, геномную ДНК из модифицированного участка можно амплифицировать с использованием либо праймеров, полученных с помощью SURVEYOR, либо праймеров для HDR. Ампликоны следует субклонировать в плазмиду, такую как pUC19, для трансформации; отдельные колонии можно подвергнуть секвенированию для выявления клонального генотипа.
Заявители сконструировали используемые при секвенировании нового поколения (NGS) праймеры для более коротких ампликонов, как правило, с размером в диапазоне 100-200 п.о. Для выявления мутаций в результате NHEJ важным является конструирование праймеров по меньшей мере с 10-20 п.о. между праймирующими участками и целевым сайтом Cas9 для обеспечения возможности выявления более длинных вставок/делений. Заявители обеспечивают рекомендации для двухстадийного способа ПЦР, чтобы прикрепить адаптеры со штрих-кодами для мультиплексного глубокого секвенирования. Заявители рекомендуют платформу Illumina вследствие обычно характерных для нее низких уровней ложноположительных вставок/делений. Затем можно провести анализ в отношении нецелевой активности (описанный ранее) посредством выравнивания считанных последовательностей в программах выравнивания, таких как ClustalW, Geneious или простых скриптов для анализа последовательности.
Материалы и реактивы
Получение sgRNA
Сверхчистая, не содержащая ДНКаз/РНКаз вода (Life Technologies, кат. №10977-023)
Слитая полимераза Herculase II (Agilent Technologies, кат. №600679)
CRITICAL. Стандартная Taq полимераза, которая лишена 3'-5' экзонуклеазной корректирующей активности, характеризуется более низкой точностью и может приводить к ошибкам при амплификации. Herculase II представляет собой высокоточную полимеразу (эквивалентную по точности Pfu), которая дает высокий выход ПЦР продукта при минимальной оптимизации. Другие высокоточные полимеразы можно заменить.
Реакционный буфер для Herculase II (5х; Agilent Technologies, включенный в полимеразу)
Смесь раствора dNTP (25 мМ каждого; Enzymatics, кат. № N205L)
MgCl2 (25 мМ; ThermoScientific, кат. № R0971)
Набор для экстракции из геля QIAquick gel extraction kit (Qiagen, кат. №28704)
Набор QIAprep spin miniprep kit (Qiagen, кат. №27106)
Сверхчистый ТВЕ буфер (10Х; Life Technologies, кат. №15581-028)
LE агароза SeaKem (Lonza, кат. №50004)
Краситель ДНК SYBR Safe (10000х; Life Technologies, кат. №S33102)
Маркер длины ДНК больше 1 т.н. (Life Technologies, кат. №10787-018)
Загрузочный буфер для синего и оранжевого красителей TrackIt CyanOrange (Life Technologies, кат. №10482-028)
Рестрикционный фермент FastDigest BbsI (BpiI) (Fermentas/ThermoScientific, кат. № FD1014)
Буфер Fermentas Tango Buffer (Fermentas/ThermoScientific, кат. № BY5)
DL-дитиотреитол (DTT; Fermentas/ThermoScientific, кат. № R0862)
ДНК-лигаза Т7 (Enzymatics, кат. № L602L)
Важно: не заменять на используемую чаще лигазу Т4. Лигаза Т7 характеризуется в 1000 раз более высокой активностью на "липких" концах, чем на "тупых" концах, и более высокой общей активностью, чем коммерчески доступные концентрированные лигазы Т4.
Буфер для быстрого лигирования Т7 2Х Rapid Ligation Buffer (включен в ДНК-лигазу Т7, Enzymatics, кат. № L602L)
Полинуклеотид-киназа Т4 (New England Biolabs, кат. № M0201S)
Реакционный буфер для ДНК-лигазы Т4 (10Х; New England Biolabs, кат. № B0202S)
Аденозин-5'-трифосфат (10 мМ; New England Biolabs, кат. № P0756S)
Безопасная для плазмид АТФ-зависимая ДНКаза (Epicentre, кат. № E3101K)
Химически компетентная в отношении Stbl3 Escherichia coli (E. coli) One Shot (Life Technologies, кат. № С7373-03)
Среда SOC (New England Biolabs, кат. № B9020S)
Среда LB (Sigma, кат. № L3022)
Агаровая среда LB (Sigma, кат. № L2897)
Ампициллин, стерилизованный фильтрованием (100 мг мл-1; Sigma, кат. № А5354)
Культура клеток млекопитающего:
Клетки HEK293FT (Life Technologies, кат. № R700-07)
Среда Игла в модификации Дульбекко (DMEM, 1X, с высоким содержанием глюкозы; Life Technologies, кат. №10313-039)
Минимальная среда Игла в модификации Дульбекко (DMEM, 1X, с высоким содержанием глюкозы, без фенолового красного; Life Technologies, кат. №31053-028)
Фосфатно-буферный солевой раствор Дульбекко (DPBS, 1X; Life Technologies, кат. №14190-250)
Остальная бычья сыворотка, разбавленная и инактивированная нагреванием (Life Technologies, кат. №10438-034)
Среда с пониженным содержанием сыворотки Opti-MEM I (FBS; Life Technologies, кат. №11058-021)
Пенициллин-стрептомицин (100х; Life Technologies, кат. №15140-163)
TrypLE™ Express (1X, без фенолового красного; Life Technologies, кат. №12604-013)
Реагент для трансфекции Lipofectamine 2000 (Life Technologies, кат. №11668027)
Набор для трансфекции Amaxa SF Cell Line 4D-Nucleofector® X Kit S (32 RCT; Lonza, кат. № V4XC-2032)
Клеточная линия HUES 9 (HARVARD STEM CELL SCIENCE)
Матрикс базальной мембраны с пониженным содержанием ростовых факторов и не содержащий LDEV (Life Technologies, кат. № А1413201)
Среда mTeSR1 (Stemcell Technologies, кат. №05850)
Раствор для отслоения клеток Accutase (Stemcell Technologies, кат. №07920)
Ингибитор ROCK (Y-27632; Millipore, кат. № SCM075)
Набор для трансфекции Amaxa Р3 Primary Cell 4D-Nucleofector® X Kit S (32 RCT; Lonza кат. № V4XP-3032)
Анализ генотипирования:
Раствор для экстракции ДНК QuickExtract (Epicentre, кат. № QE09050)
ПЦР праймеры для анализа с помощью SURVEYOR, RFLP анализа или секвенирования (см. таблицу праймеров)
Слитая полимераза Herculase II (Agilent Technologies, кат. №600679)
CRITICAL. Поскольку анализ с помощью Surveyor является чувствительным к несовпадениям по одному основанию, особо важным является использование высокоточной полимеразы. Другие высокоточные полимеразы можно заменить.
Реакционный буфер для Herculase II (5х; Agilent Technologies, включенный в полимеразу)
Смесь раствора dNTP (25 мМ каждого; Enzymatics, кат. № N205L)
Набор для экстракции из геля QIAquick gel extraction kit (Qiagen, кат. №28704)
Taq буфер (10x; Genscript, кат. № В0005)
Набор для выявления мутаций с помощью SURVEYOR для стандартного гель-электрофореза (Transgenomic, кат. № 706025)
Сверхчистый ТВЕ буфер (10х; Life Technologies, кат. №15581-028)
LE агароза SeaKem (Lonza, кат. №50004)
Гели с 4-20% ТВЕ. толщиной 1,0 мм, 15 лунок (Life Technologies, кат. № ЕС62255 ВОХ)
ТВЕ буфер с высокой плотностью для образца Novex® Hi-Density ТВЕ Sample Buffer (5Х; Life Technologies, кат. № LC6678)
Краситель для окрашивания нуклеиновой кислоты в геле SYBR Gold Nucleic Acid Gel Stain (10000X; Life Technologies, кат. № S-11494)
ДНК-маркер длиной более 1 т.п.н. (Life Technologies, кат. №10787-018)
Загрузочный буфер для синего и оранжевого красителей Trackit CyanOrange (Life Technologies, кат. №10482-028)
Рестрикционный фермент FastDigest HindIII (Fennentas/ThermoScientific, кат. № FD0504)
Оборудование
Стерильные наконечники с фильтром для пипеток (Corning)
Стандартные 1,5 мл микроцентрифужные пробирки (Eppendorf, кат. №0030 125.150)
96-луночные планшеты для ПЦР Axygen (VWR, кат. № PCR-96M2-HSC)
Пробирки для ПЦР на 8-стрипов Axygen (Fischer Scientific, кат. №14-222-250)
Пробирки Falcon, полипропилен, 15 мл (BD Falcon, кат. №352097)
Пробирки Falcon, полипропилен, 50 мл (BD Falcon, кат. №352070)
Круглодонная пробирка с сетчатым фильтром для клеток, 5 мл (BD Falcon, кат. №352235)
Чашки Петри (60 мм × 15 мм; BD Biosciences, кат. №351007)
Планшет для тканевой культуры (24-лунки; BD Falcon, кат. №353047)
Планшет для тканевой культуры (96-лунок, плоскодонный; BD Falcon, кат. №353075)
Чашка для тканевой культуры (100 мм; BD Falcon, 353003)
Термопиклер на 96 лунок с программируемой функцией поэтапного изменения температуры Applied Biosystems Veriti, кат. №4375786).
Настольные микроцентрифуги 5424, 5804 (Eppendorf)
Система для гель-электрофореза (базовый источник питания PowerPac, Bio-Rad, кат. №164-5050, и лоток для геля из системы Sub-Cell GT, Bio-Rad, кат. №170-4401)
Система для электрофореза Novex XCell SureLock Mini-Cell (Life Technologies, кат. № EI0001)
Цифровая система для визуализации геля (GelDoc EZ, Bio-Rad, кат. №170-8270, и голубой лоток для образца, Bio-Rad, кат. №170-8273)
Прибор для просвечивания голубым светом и очки с оранжевым светофильтром (Safebnager 2.0; Inivitrogen, кат. № G6600)
Программное обеспечение для количественного анализа геля (Bio-Rad, ImageLab, включена в GelDoc EZ, или ImageJ с открытым исходным кодом от Национальных институтов здравоохранения, доступная на веб-сайте rsbweb.nih.gov/ij/) с помощью УФ-спектрофотометра (NanoDrop 2000 с, Thermo Scientific)
Приготовление реагента
Раствор для электрофореза на основе Tris-бората EDTA (ТВЕ). Развести ТВЕ буфер в дистиллированной воде до 1X рабочего раствора для заливки агарозных гелей и для применения в качестве буфера для электрофореза. Буфер можно хранить при комнатной температуре(18-22°С) в течение по меньшей мере 1 года.
- АТФ, 10 мМ. Разделить 10 мМ АТФ на 50 мкл аликвоты и хранить при -20°С до 1 года; избегать повторяющихся циклов замораживания-оттаивания.
- DTT, 10 мМ. Приготовить 10 мМ раствор DTT в дистиллированной воде и хранить в 20 мкл аликвотах при -70°С в течение периода до 2 лет; для каждой реакции использовать новую аликвоту, поскольку DTT легко окисляется.
- Культуральная среда D10. Для культивирования клеток HEK293FT приготовить культуральную среду D10 путем добавления к DMEM добавки 1X GlutaMAX и 10% (об./об.) фетальной бычьей сыворотки. Как указано в протоколе, эту среду также дополняют 1X пенициллином-стрептомицином. Среду D10 можно приготовить заранее и хранить при 4°С в течение периода до 1 месяца.
- Культуральная среда mTeSR1. Для культивирования эмбриональных стволовых клеток человека приготовить среду mTeSR1 путем добавления 5Х добавки (включенной в базовую среду mTeSR1) и 100 мкг/мл нормоцина.
Процедура
Конструирование нацеливающих компонентов и применение онлайн-инструмента
- Временные рамки 1 день
1) Ввести последовательность целевой геномной ДНК. Заявители обеспечили онлайн-инструмент конструирования для нацеливания Cas9, который использует вводимую представляющую интерес последовательность, идентифицирует и ранжирует подходящие целевые сайты и путем вычислений прогнозирует нецелевые сайты для каждой намеченной мишени. В качестве альтернативы, специалист может в ручном режиме выбирать направляющую последовательность путем идентификации последовательности из 20 п.о. непосредственно выше любого 5'-NGG.
2) Расположить по порядку необходимые олигонуклеотиды и праймеры, которые определены с помощью онлайн-инструмента. Если сайт выбирают вручную, олигонуклеотиды и праймеры должны быть сконструированы.
Получение экспрессионной конструкции sgRNA
3) Для создания экспрессионной конструкции sgRNA можно использовать либо протокол на основе использования либо ПЦР, либо плазмид.
(А) посредством ПЦР амплификации
- Временные рамки 2 часа
(i) Заявители готовили разведенную матрицу с U6 для ПЦР. Заявители рекомендуют использовать РХ330 в качестве матрицы для ПЦР, но любую содержащую U6 плазмиду можно подобным образом использовать в качестве матрицы для ПЦР. Заявители разводили матрицу ddH2O до концентрации 10 нг/мкг. Следует обратить внимание, что если в качестве матрицы используется плазмида или кассета, уже содержащая управляемую U6 sgRNA, следует провести экстракцию из геля, чтобы удостовериться в том, что продукт содержит только намеченную sgRNA и не содержит следовую sgRNA, перенесенную с матрицы.
(ii) Заявители готовили разведенные олигонуклеотиды для ПЦР. U6-прямой праймер и U6-sgRNA-обратный праймер разводили до конечной концентрации 10 мкМ в ddH2O (добавляли 10 мкл 100 мкМ праймера к 90 мкл ddH2O).
(iii) Реакционная смесь для ПЦР с U6-sgRNA. Заявители готовили следующие реакционные смеси для каждого U6-sgRNA-обратного праймера и мастер-микс, если необходимо:

(iv) Заявители проводили ПЦР реакцию с реакционными смесями со стадии (iii) с использованием следующих условий циклирования:

(v) По завершении реакции заявители прогоняли продукт на геле для подтверждения успешной амплификации, дающей одну полосу. Опустить 2% (вес/об.) агарозный гель в камеру с 1X ТВЕ буфером с безопасным красителем 1X SYBR. Разогнать 5 мкл PCR продукта в геле при 15 В см-1 в течение 20-30 мин. Успешно полученные ампликоны должны давать один единственный продукт из 370 п.о., а матрица должна быть невидимой. Экстракция из геля ПЦР ампликона не обязательно является необходимой.
(vi) Заявители очищали ПЦР продукт с использованием набора для очистки QIAquick PCR purification kit согласно указаниям производителя. Элюировать ДНК в 35 мкл буфера ЕВ или воды. Очищенные ПЦР продукты можно хранить при 4°С или -20°С.
(В) Клонирование sgRNA в содержащий Cas9 бицистронный вектор экспрессии ⋅ Временные рамки 3 дня
(i) Приготовить вставки олигонуклеотидов sgRNA. Заявители ресуспендировали верхнюю и нижнюю нити олигонуклеотидов для каждой структуры sgRNA в конечной концентрации 100 мкМ. Фосфорилировать и гибридизовать олигонуклеотиды следующим образом:

(ii) Гибридизовать в термоциклере с использованием следующих параметров:
37°С в течение 30 мин,
95°С в течение 5 мин,
постепенно понизить температуру до 25°С со скоростью 5°С в мин.
(iii) Заявители разводили фосфорилированные и гибридизованные олигонуклеотиды в пропорции 1:200 путем добавления 1 мкл олигонуклеотида к 199 мкл ddH2O комнатной температуры.
(iv) Клонировать олигонуклеотид sgRNA в РХ330. Заявители готовили реакционную смесь Golden Gate для каждой sgRNA. Заявители также рекомендуют приготовить не содержащий вставки отрицательный контроль только с РХ330.

(v) Инкубировать реакционную смесь Golden Gate в течение в общей сложности 1 часа:

(vi) Заявители обрабатывали реакционную смесь Golden Gate экзонуклеазой PlasmidSafe для расщепления любой остаточной линеаризованной ДНК. Эта стадия является необязательной, но очень рекомендуется.

(vii) Заявители инкубировали реакционную смесь PlasmidSafe при 37°С в течение 30 мин с последующей инактивацией при 70°С в течение 30 мин. Точка остановки: после завершения реакционную смесь можно заморозить и продолжить позже. Кольцевая ДНК должна быть стабильна в течение по меньшей мере 1 недели.
(viii) Трансформация. Заявители трансформировали обработанные PlasmidSafe плазмиды в компетентный штамм Е. coli согласно протоколу, поставляемому с клетками. Заявители рекомендуют Stb13 для быстрой трансформации. Вкратце, заявители добавляли 5 мкл продукта со стадии (vii) в 20 мкл охлажденных на льду клеток, химически компетентных в отношении Stb13. Затем их инкубировали на льду в течение 10 мин, подвергали тепловому шоку при 42°С в течение 30 с, немедленно возвращали на лед в течение 2 мин, добавляли 100 мкл среды SOC, и высевали их на чашку со средой LB, содержащую 100 мкг/мл ампициллина, и инкубируя в течение ночи при 37°С.
(ix) День 2: заявители визуально контролировали чашки в отношении роста колоний. Как правило, колонии на плашках с отрицательным контролем (лигирование только РХ330, расщепленного BbsI, без гибридизованного олигонуклеотида sgRNA) отсутствуют, и колонии на плашках для клонирования с РХ330-sgRNA присутствуют в количестве от десятков до сотен.
(х) С каждой чашки заявители отбирали 2-3 колонии для проверки правильности вставки sgRNA. Заявители использовали стерильные наконечники для пипеток, чтобы инокулировать одну колонию в 3 мл культуральной среды LB со 100 мкг/мл ампициллина. Инкубировали и встряхивали при 37°С в течение ночи.
(xi) День 3: заявители выделяли плазмидную ДНК из выращиваемых в течение ночи культур с использованием набора QiAprep Spin miniprep kit согласно инструкциям производителя.
(xii) Валидация последовательности плазмиды с CRISPR. Заявители подтверждали последовательность каждой колонии путем секвенирования от промотора U6 с использованием U6-прямого праймера. Необязательно: последовательность используемых праймеров для гена Cas9 приведена в следующей таблице праймеров.


Заявители рассматривали результаты секвенирования в сравнении с последовательностью вектора клонирования РХ330 для проверки того, что направляющая последовательность из 20 п.о. была вставлена между промотором U6 и остальной частью каркаса sgRNA. Подробности и последовательность РХ330 в виде карты в формате карты вектора, используемом в GenBank (*.gb файл), можно найти на веб-сайте crispr.genome-engineering.org.
(Необязательное) конструирование матрицы ssODN ⋅ Временные рамки 3 дня заблаговременного планирования
3) Конструирование ssODN и расположение ssODN по порядку. Либо смысловой, либо антисмысловой ssODN можно приобрести непосредственно у поставщика. Заявители рекомендовали конструирование гомологичных плечей по меньшей мере из 40 п.о. с каждой стороны и 90 п.о. для оптимальной эффективности HDR. По опыту заявителей антисмысловые олигонуклеотиды характеризуются чуть более высокими эффективностями модификации.
4) Заявители ресуспендировали и разводили ультрамеры ssODN до конечной концентрации 10 мкМ. Не объединять или гибридизовать смысловые и антисмысловые ssODN. Хранить при -20°С.
5) Для применений HDR следует учесть, что заявители рекомендуют клонировать sgRNA в плазмиду РХ330.
Функциональная валидация sgRNA: культура клеток и трансфекции ⋅ Временные рамки 3-4 дня
Система CRISPR-Cas применялась в ряде линий клеток млекопитающих. Условия могут варьировать для каждой клеточной линии. В приведенных ниже протоколах подробно описываются условия трансфекции для клеток HEK239FT. Для трансфекции с опосредованной ssODN HDR следует учесть, что набор для трансфекции Amaxa SF Cell Line Nucleofector Kit использовали для оптимальной доставки ssODN. Это описано в следующем разделе.
7) Поддержание HEK293FT. Клетки поддерживали согласно рекомендациям производителя. Вкратце, заявители культивировали клетки в среде D10 (DMEM GlutaMax, дополненная 10% фетальной бычьей сывороткой), при 37°С и 5% CO2.
8) Для пассажирования заявители удаляли среду и однократно промывали путем осторожного добавления DPBS боковой стенке сосуда для того, чтобы не сместить клетки. Заявители добавляли 2 мл TrypLE в колбу Т75 и инкубировали в течение 5 мин при 37°С. Для инактивации добавляли 10 мл теплой среды D10 и переносили в 50 мл пробирку Falcon. Заявители диссоциировали клетки путем осторожного измельчения и пересевали в новые колбы, если необходимо. Заявители, как правило, пассажировали клетки каждые 2-3 дня при индексе разведения 1:4 или 1:8, никогда не позволяя клеткам достигать больше 70% конфлюентности. Клеточные линии вновь начинали после достижения пассажа номер 15.
9) Получение клеток для трансфекции. Заявители высевали хорошо диссоциированные клетки на 24-луночные планшеты в среде D10 без антибиотиков за 16-24 часа до трансфекции при плотности посева 1,3×105 клеток на лунку и высеваемом объеме 500 мкл. Пропорциональное увеличение или уменьшение осуществлять согласно инструкциям производителя, если необходимо. Не стоит высаживать больше клеток, чем нужно для рекомендованной плотности, поскольку такие действия могут снизить эффективность трансфекции.
10) Ко дню трансфекции оптимально, чтобы клетки достигли 70-90% конфлюентности. Клетки можно трансфицировать с помощью реактива Lipofectamine 2000 или набора для трансфекции Amaxa SF Cell Line Nucleofector Kit согласно протоколам производителя.
(A) Для sgRNA, клонированных в РХ330, заявители трансфицировали 500 нг подтвержденной по последовательности плазмиды с CRISPR; при трансфекции более чем одной плазмиды смешать в эквимолярном соотношении и не более чем 500 нг в сумме.
(B) Для sgRNA, амплифицированной с помощью ПЦР, заявители смешивали следующие компоненты:

Заявители рекомендовали осуществление трансфекции в трех повторностях согласно методике для надежного количественного определения, и включали контроли трансфекции (например, плазмида GFP) для мониторинга эффективности трансфекции. Кроме того, плазмиду для клонирования РХ330 и/или ампликон sgRNA можно трансфицировать сами по себе в качестве отрицательного контроля для последующих функциональных анализов.
11) Заявители осторожно добавляли комплекс Lipofectamine к клеткам, поскольку клетки HEK293FT могут легко отделяться от планшета, и это приводит к более низкой эффективности трансфекции.
12) Заявители проверяли клетки через 24 часа после трансфекции в отношении эффективности путем оценки доли флуоресцирующих клеток в контроле (например, GFP) трансфекции с использованием флуоресцентного микроскопа. Как правило, трансфицируются более 70% клеток.
13) Заявители дополняли культуральную среду дополнительными 500 мкл теплой среды D10. Добавить D10 очень медленно по боковой стенке лунки и не использовать холодную среду, поскольку клетки легко могут отделиться.
14) Клетки инкубировали в течение периода, составляющего в сумме 48-72 часа после трансфекции, перед сбором для анализа вставок/делений. Эффективность вставок/делений заметно не повышалась после 48 часов.
(Необязательная) совместная трансфекция плазмид с CMSPR и ssODN или плазмид для целенаправленного воздействия в отношении HR ⋅ Временные рамки 3-4 дня
15) Линеаризация плазмиды для целенаправленного воздействия. Вектор для целенаправленного воздействия линеаризуют, если это возможно, путем однократного разрезания в рестрикционном сайте в каркасе вектора около одного из гомологичных плеч или на удаленном конце любого из гомологичных плеч.
16) Заявители подвергали электрофорезу небольшое количество линеаризованной плазмиды наряду с неразрезанной плазмидой в 0,8-1% агарозном геле для проверки успешной линеаризации. Линеаризованная плазмида при разделении должна идти дальше сверхспирализованной плазмиды.
17) Заявители очищали линеаризованную плазмиду с использованием набора для очистки QIAQuick PCR Purification kit.
18) Получение клеток для трансфекции. Заявители культивировали HEK293FT в колбах Т75 или Т225. Предполагалось достаточное количество клеток перед днем трансфекции. Для стрип-кюветного формата по методике Amaxa использовали 2×106 клеток на трансфекцию.
19) Получение планшетов для трансфекции. Заявители добавляли 1 мл теплой среды D10 в каждую лунку 12-луночного планшета. Планшеты помещали в инкубатор для сохранения среды теплой.
20) Нуклеофекция. Заявители трансфицировали клетки HEK293FT в соответствии с инструкциями производителя к набору для трансфекции Amaxa SF Cell Line Nucleofector 4D Kit, адаптированными в стадиях, приведенных ниже.
а. Для совместной трансфекции ssODN и CRISPR предварительно смешать следующие ДНК в пробирках для ПЦР:

b. Для совместной трансфекции плазмиды для целенаправленного воздействия посредством HDR и CRISPR предварительно смешать следующие ДНК в пробирках для ПЦР:

Что касается контролей для трансфекции, смотри предыдущий раздел. Кроме того, заявители рекомендуют трансфицироватъ ssODN или плазмиду для целенаправленного воздействия сами по себе в качестве отрицательного контроля.
21) Диссоциация на отдельные клетки. Заявители удаляли среду и однократно осторожно промывали при помощи DPBS, следя за тем, чтобы не сместить клетки. 2 мл TrypLE добавляли в колбу Т75 и инкубировали в течение 5 мин при 37°С. Для инактивации добавляли 10 мл теплой среды D10 и осторожно измельчали в 50 мл пробирке Falcon. Рекомендуется, чтобы клетки осторожно измельчали и диссоциировали на отдельные клетки. Большие скопления снижали эффективность трансфекции. Заявители брали 10 мкл аликвоту из суспензии и разводили в 90 мкл среды D10 для подсчета. Заявители производили подсчет клеток и рассчитывали количество клеток и объем суспензии, необходимый для трансфекции. Заявители, как правило, трансфицировали 2×105 клеток на процедуру с использованием стрипов Amaxa Nucleocuvette strips, и при расчетах рекомендуется учитывать на 20% больше клеток, чем требуется для того, чтобы ввести поправку на потерю объема на последующих стадиях отмеривания пипеткой. Необходимый объем переносили в новую пробирку Falcon.
23) Заявители осаждали центрифугированием новую пробирку при 200×g в течение 5 мин.
Заявители готовили раствор для трансфекции путем смешивания раствора SF и доставки S1, как рекомендовано Amaxa. Для стрип-кювег Amaxa требуется всего 20 мкл дополненного раствора SF на трансфекцию. Аналогично, при расчетах заявители рекомендовали учитывать на 20% больше объем, чем требуется.
25) Заявители полностью удаляли среду с присутствующих в виде осадка клеток со стадии 23 и осторожно ресуспендировали в подходящем объеме (20 мкл на 2×105 клеток) раствора SF, дополненного S1. Не оставлять в клетки в растворе SF в течение продолжительного периода времени.
26) 20 мкл ресуспендированных клеток добавляли пипеткой в каждую предварительно приготовленную смесь ДНК из стадии 20. Осторожно перемешать смесь с помощью пипетки и перенести в камеру для стрипов Nucleocuvette. Повторить это для каждой процедуры трансфекции.
Клетки подвергали электропорации с использованием системы Nucleofector 4D, рекомендованной Amaxa, CM-130.
28) Заявители осторожно и медленно добавляли пипеткой 100 мкл теплой среды D10 в каждую камеру для стрипов в Nucleocuvette и переносили весь объем в предварительно подогретый планшет со стадии 19. CRITICAL. Клетки являются очень хрупкими на этой стадии, и резкий отбор пипеткой может вызвать гибель клеток. Инкубировали в течение 24 часов. В этот момент эффективность трансфекции можно оценить, исходя из доли флюоресцирующих клеток в положительном контроле для трансфекции. Нуклеофекция, как правило, приводит к эффективности трансфекции более 70-80%. Заявители медленно добавляли 1 мл теплой среды D10 в каждую лунку без смещения клеток. Инкубировали клетки в течение в течение периода, составляющего в сумме 72 часа.
Культивирование и трансфекция эмбриональных стволовых клеток человека (HUES 9) ⋅ Временные рамки 3-4 дня
Поддержание линии hESC (HUES9). Заявители в установленном порядке поддерживали линию клеток HUES9 в условиях отсутствия фидера со средой mTesR1. Заявители готовили среду mTeSR1 путем добавления 5Х добавки, включенной в базовую среду, и 100 мкг/мл нормоцина. Заявители готовили 10 мкл аликвоту среды mTeSR1, дополненной, кроме того, 10 мкМ ингибитором Rock. Нанесение покрытия на планшет для тканевой культуры. Разводили холодный GelTrex в пропорции 1:100 в холодной DMEM и покрывали всю поверхность 100 мм планшета для тканевой культуры.
Помещали планшет в инкубатор по меньшей мере на 30 мин при 37°С. Отогревали пузырек с клетками при 37°С в 15 мл пробирке Falcon, добавляли 5 мл среды mTeSR1 и осаждали при 200×g в течение 5 мин. Аспирировали покрытие GelTrex и высевали ~1×106 клеток в 10 мл среды mTeSR1, содержащей ингибитор Rock. Сменяли среду на нормальную среду mTeSR1 через 24 часа после трансфекции и "подпитывали" среду ежедневно. Пассажирование клеток. Ежедневно "подпитывали" клетки свежей средой mTeSR1 и пассажировали до достижения 70% конфлюентности. Аспирировали среду mTeSR1 и однократно промывали клетки DPBS. Диссоциировали клетки путем добавления 2 мл Accutase и инкубировали при 37°С в течение 3-5 мин. Добавляли 10 мл среды mTeSR1 к отделившимся клеткам, переносили в 15 мл пробирку Falcon и осторожно ресуспендировали. Повторно высевали на планшеты с покрытием GelTrex в среду mTeSR1 с 10 мкМ ингибитором Rock. Сменяли среду на нормальную среду mTeSR1 через 24 часа после посева.
Трансфекция. Заявители рекомендовали культивирование клеток по меньшей мере в течение 1 недели после отогрева перед трансфекцией с использованием набора для трансфекции Amaxa Р3 Primary Cell 4-D Nucleofector Kit (Lonza). "Подпитывали" клетки, находящиеся в log-фазе роста, свежей средой за 2 часа до трансфекции. Диссоциировали на отдельные клетки или мелкие кластеры не более чем из 10 клеток при помощи accutase и осторожного ресуспендирования. Подсчитывали количество клеток, необходимое для нуклеофекции, и осаждали центрифугированием при 200×g в течение 5 мин. Удаляли среду полностью и ресуспендировали в рекомендованном объеме дополненного S1 раствора Р3 для нуклеофекции. Осторожно высевали подвергнутые электропорации клетки в планшеты с покрытием в присутствии 1X ингибитора Rock.
Проверяли успешность трансфекции и "подпитывали" ежедневно стандартной средой mTeSR1, начиная с момента через 24 часа после нуклеофекции. Как правило, заявители наблюдали эффективность трансфекции более 70% при использовании нуклеофекции Amaxa. Сбор ДНК. Через 48-72 часа после трансфекции диссоциировали клетки с использованием accutase и инактивировали accutase путем добавления 5-кратного объема mTeSR1. Осаждали клетки центрифугированием при 200×g в течение 5 мин. Присутствующие в виде осадка клетки можно подвергнуть непосредственной обработке для экстракции ДНК раствором QuickExtract. Рекомендуется не подвергать клетки механической диссоциации без accutase. Рекомендуется не осаждать клетки центрифугированием без инактивации accutase или со скоростью, превышающей рекомендованную; такие действия могут вызвать лизис клеток.
Выделение клональных клеточных линий при помощи FACS Временные рамки ⋅ практическое выполнение 2-3 часа; разрастание 2-3 недели
Выделение клонов можно осуществлять через 24 часа после трансфекции с помощью FACS или с помощью последовательного разведения.
54) Приготовление буфера для FACS. Клетки, для которых не требуется сортировка с с использованием флуоресцирующих красителей, можно сортировать в стандартной среде D10, дополненной 1X пенициллином/стрептиномицином. Если также требуется сортировка с использованием флуоресцирующих красителей, тогда не содержащую фенола DMEM или DPBS использовали вместо нормальной DMEM. Добавить 1X пенициллин/стрептиномицин и фильтровать через 22 мкм фильтр Steriflip.
55) Подготовка 96-луночных планшетов. Заявители добавляли 100 мкл среды D10, дополненной 1X пенициллином/стрептиномицином, на лунку и получали такое количество планшетов, которое необходимо для желаемого числа клонов.
56) Получение клеток для FACS. Заявители диссоциировали клетки путем полной аспирации среды и добавления 100 мкл TrypLE на лунку в 24-луночном планшете. Инкубировали в течение 5 мин и добавляли 400 мкл теплой среды D10.
57) Ресуспендированные клетки переносили в 15 мл пробирку Falcon и осторожно измельчали 20 раз. Рекомендована проверка под микроскопом для того, чтобы удостовериться в диссоциации на отдельные клетки.
58) Осаждали клетки центрифугированием при 200×g в течение 5 минут.
59) Заявители аспирировали среду и ресуспендировали клетки в 200 мкл среды для FACS.
60) Клетки фильтровали через 35 мкм сетчатый фильтр в пробирки с метками для FACS. Заявители рекомендовали использовать 12×75 мм пробирки BD Falcon с сетчатым фильтром для клеток в крышке. Помещали клетки на лед до сортировки.
61) Заявители сортировали отдельные клетки в 96-луночные планшеты, полученные на стадии 55. Заявители рекомендовали, чтобы в одну отдельную предусмотренную лунку на каждом планшете отсортировали 100 клеток в качестве положительного контроля.
ПРИМЕЧАНИЕ. Остальные клетки можно хранить и использовать для генотипирования при уровне численности популяции для измерения общей эффективности модификации.
62) Заявители возвращали клетки в инкубатор и обеспечивали возможность их разрастания в течение 2-3 недель, 100 мкл теплой среды D10 добавляли через 5 дней после сортировки. Заменяли 100 мкл среды каждые 3-5 дней, если необходимо.
63) Колонии подвергали осмотру в отношении "клонального" внешнего вида через 1 неделю после сортировки: округлые колонии, расходящиеся от центральной точки. Помечали лунки, которые были пустыми или могли быть засеяны с образованием двух колоний или нескольких колоний.
64) Когда клетки достигали конфлюентности более 60%, заявители готовили набор планшетов-реплик для пассажирования. 100 мкл среды D10 добавляли в каждую лунку в планшетах-репликах. Заявители непосредственно диссоциировали клетки путем интенсивного втягивания в пипетку и выталкивания клеток из пипетки 20 раз. 20% ресуспендированного объема высевали в подготовленные планшеты-реплики для сохранения клональных линий. После этого заменяли среду каждые 2-3 дня и пассажировали, соответственно.
65) Остальные 80% клеток использовали для выделения ДНК и генотипирования.
Необязательно: выделение клональных клеточных линий путем разведения. Временные рамки ⋅ практическое выполнение 2-3 часа; разрастание 2-3 недели
66) Заявители диссоциировали клетки из 24-луночных планшетов, как описано выше. Удостоверялись в диссоциации до отдельных клеток. Сетчатый фильтр для клеток можно использовать для предотвращения образования скоплений клеток.
67) Количество клеток подсчитывали при каждой процедуре. Последовательно разводили при каждой процедуре в среде D10 до конечной концентрации 0,5 клетки на 100 мкл. Для каждого 96-луночного планшета заявители рекомендовали разведение до конечного количества 60 клеток в 12 мл D10. Для надлежащего клонального разведения рекомендуется точный подсчет клеток. Клетки можно повторно подсчитать на промежуточной стадии последовательного разведения для гарантии точности.
68) Мультиканальную пипетку использовали для внесения пипеткой 100 мкл разведенных клеток в каждую лунку 96-луночного планшета.
ПРИМЕЧАНИЕ. Остальные клетки можно хранить и использовать для генотипирования при уровне численности популяции для измерения общей эффективности модификации.
69) Заявители визуально оценивали колонии в отношении "клонального" внешнего вида через ~1 неделю после посева: округлые колонии, расходящиеся от центральной точки. Помечали лунки, которые могли быть засеяны с образованием двух колоний или нескольких колоний.
70) Заявители возвращали клетки в инкубатор и обеспечивали возможность их разрастания в течение 2-3 недель. "Подпитывали" клетки, как подробно описано в предыдущем разделе, если это было необходимо.
Анализ с помощью SURVEYOR в отношении эффективности расщепления у CRISPR. Временные рамки ⋅ 5-6 часов
Перед анализом эффективности расщепления на трансфицированных клетках заявители рекомендовали провести испытание каждого нового праймера для анализа с помощью SURVEYOR на образцах отрицательного контроля (нетрансфицированных) посредством стадии расщепления нуклеазой SURVEYOR с использованием протокола, описанного ниже. Изредка, даже присутсвующие в виде одной полосы, чистые ПЦР продукты для последующего анализа с помощью SURVEYOR могут давать полосы в результате неспецифического расщепления нуклеазой SURVEYOR и потенциально создавать помехи для точного анализа вставок/делений.
71) Сбор клеток для ДНК. Диссоциировали клетки и осаждали центрифугированием при 200×g в течение 5 мин. ПРИМЕЧАНИЕ. Планшет-реплика на данной стадии необходим для сохранения трансфицированных клеточных линий.
72) Полностью аспирировали супернатанты.
73) Заявители использовали раствор для экстракции ДНК QuickExtract согласно инструкциям производителя. Заявители, как правило, использовали 50 мкл раствора для каждой лунки в 24-луночном планшете и 10 мкл для 96-луночного планшета.
74) Заявители нормализовали экстрагированную ДНК до конечной концентрации 100-200 нг/мкл при помощи ddH2O. Точка остановки: экстрагированную ДНК можно хранить при -20°С в течение нескольких месяцев.
75) Подготовка ПЦР для анализа SURVEYOR. Мастер-микс следующих компонентов с использованием праймеров для SURVEYOR был обеспечен заявителями с использованием онлайн-инструмента/инструмента на основе компьютерного алгоритма:

76) Заявители добавляли 100-200 нг матрицы нормализованной геномной ДНК со стадии 74 для каждой реакции.
77) Реакцию ПНР проводили с использованием следующих условий в циклах при не более 30 циклов амплификации:

78) Заявители разделяли 2-5 мкл продукта ПЦР в 1% геле для проверки на наличие продукта в виде одной полосы. Хотя данные условия ПЦР были разработаны для работы с большинством пар праймеров для SURVEYOR, для некоторых праймеров может требоваться дополнительная оптимизация путем корректировки концентрации матрицы, концентрации MgCl2 и/или температуры гибридизации.
79) Заявители очищали реакционные смеси для ПЦР с использованием набора для очистки QIAQuick PCR purification kit и нормализовали элюантом до 20 нг/мкл. Точка остановки: очищенный ПЦР продукт можно хранить при -20°С.
80) Образование гетеродуплекса ДНК. Реакционную смесь для гибридизации готовили следующим образом:

81) Проводили гибридизацию в реакционной смеси с использованием следующих условий:

82) Расщепление нуклеазой S SURVEYOR. Заявители готовили мастер-микс и добавляли следующие компоненты на льду к гибридизованным гетеродуплексам со стадии 81 до общего конечного объема 25 мкл:

83) Хорошо перемешивали на вихревой мешалке и осаждали центрифугированием. Инкубировали реакционную смесь при 42°С в течение 1 часа.
84) Необязательно: можно добавить 2 мкл стоп-раствора из набора SURVEYOR. Точка остановки. Расщепленный продукт можно хранить при -20°С для дальнейшего анализа.
85) Визуальная оценка реакции SURVEYOR. Продукты расщепления нуклеазой SURVEYOR можно визуально оценивать в 2% агарозном геле. Для лучшей разрешающей способности продукты можно прогнать в 4-20% градиентном полиакриламидном геле с ТВЕ. Заявители загружали 10 мкл продукта с рекомендованным загрузочным буфером и подвергали гель-электрофорезу согласно инструкциям производителя. Как правило, заявители проводили разделение до тех пор, пока краситель бромфеноловый синий не мигрировал в нижнюю часть геля. В тот же гель включали маркер длины ДНК и отрицательные контроли.
86) Заявители окрашивали гель красителем 1X SYBR Gold, разведенным в ТВЕ. Гель осторожно покачивали в течение 15 мин.
87) Заявители визуализировали гель с использованием системы для количественной визуализации без чрезмерного экспонирования полос. Отрицательные контроли должны иметь только одну полосу, соответствующую размеру продукта ПЦР, но могут могут характеризоваться полосами от неспецифического расщепления от других сайтов. Они не будут препятствовать анализу, если они отличаются по размеру от полос целевого расщепления. Сумма размеров полос целевого расщепления, обеспеченная с помощью разработанного заявителями онлайн-инструмента/инструмента на основе компьютерного алгоритма, должна быть равна размеру продукта ПЦР.
88) Оценка интенсивности расщепления. Заявители количественно определяли суммарную интенсивность каждой полосы с использованием ImageJ или другого программного обеспечения для количественного анализа геля.
89) Для каждой дорожки заявители рассчитывали долю расщепленного ПЦР продукта (ƒcut) с использованием следующей формулы: ƒcut=(b+с)/(а+b+с), где а представляет собой суммарную интенсивность для нерасщепленного продукта ПЦР, а b и с представляют собой суммарные интенсивности для каждого продукта расщепления. 90) Эффективность расщепления можно оценить с использованием следующей формулы, основанной на биномиальном распределении вероятности образования дуплексов:
91)

Секвенирование по Сэнгеру для оценки эффективности расщепления CRISPR. Временные рамки ⋅ 3 дня
Начальные стадии идентичны стадиям 71-79 в анализе с помощью SURVEYOR. Примечание: праймеры для SURVEYOR можно использовать для секвепирования по Сэнгеру, если подходящие сайты рестрикции прикреплены к прямому и обратному праймерам. Для клонирования в рекомендованный каркас pUC19 можно использовать EcoRI для прямого праймера, a HindIII для обратного праймера.
92) Расщепление ампликона. Приготовить реакционную смесь для расщепления следующим образом:


93) Расщепление каркаса pUC19. Приготовить реакционную смесь для расщепления следующим образом:

94) Заявители очищали реакционные смеси из реакций расщепления с использованием набора для очистки QIAQuick PCR purification kit. Точка остановки: очищенный ПЦР продукт можно хранить при -20°С.
95) Заявители лигировали расщепленный каркас pUC19 и ампликоны для секвенирования Сэнгеру в соотношении вектор : вставка, составляющем 1:3, следующим образом:

96) Трансформация. Заявители трансформировали плазмиды, обработанные PlasmidSafe, в компетентный штамм Е. coli согласно протоколу, поставляемому с клетками. Заявители рекомендовали Stb13 для быстрой трансформации. Вкратце, 5 мкл продукта со стадии 95 добавляли в 20 мкл охлажденных на льду химически компетентных клеток Stb13, инкубировали на льду в течение 10 мин, подвергали тепловому шоку при 42°С в течение 30 с, немедленно возвращали на лед на 2 мин, добавляли 100 мкл среды SOC и высевали па чашку со средой LB, содержащую 100 мкг/мл ампициллина. Их инкубировали в течение ночи при 37°С.
97) День 2: заявители визуально контролировали чашки в отношении роста колоний. Как правило, колонии на чашках с отрицательным контролем (лигирование только pUC19, расщепленного EcoRI-HindIII, без вставки ампликона для секвенирования по Сэнгеру) отсутствуют, и колонии на чашках для клонирования с pUC19-ампликоном для секвенирования по Сэнгеру присутствуют в количестве от десятков до сотен.
98) День 3: заявители выделяли плазмидную ДНК из выращиваемых в течение ночи культур с использованием набора QIAprep Spin miniprep kit согласно инструкциям производителя.
99) Секвенирование по Сэнгеру. Заявители подтверждали последовательность каждой колонии путем секвенирования от каркаса pUC19 с использованием pUC19-прямого праймера. Заявители рассматривали результаты секвенирования в сравнении с ожидаемой последовательностью геномной ДНК для проверки присутствия индуцированных Cas9 мутаций в результате NHEJ. % эффективности редактирования = (число модифицированных клонов)/(общее число клонов). Важно отобрать приемлемое число клонов (>24) для получения правильных значений эффективности модификации.
Генотипирование в отношении микроделеций. Временные рамки ⋅ практическое выполнение 2-3 дня; размножение 2-3 недели
100) Клетки трансфицировали, как описано выше, парой sgRNA, нацеленных на участок, подлежащий делении.
101) Через 24 часа после трансфекции клональные линии выделяли с помощью FACS или последовательного разведения, как описано выше.
102) Клетки разращивали в течение 2-3 недель.
103) Заявители отбирали ДНК из клональных линий, как описано выше, с использованием 10 мкл раствора QuickExtract и нормализованной геномной ДНК с ddH20 до конечной концентрации 50-100 нг/мкл.
104) ПЦР амплификация модифицированного участка. Реакционную смесь для ПЦР готовили следующим образом:

105) Для скрининга в отношении инверсий реакционную смесь для ПЦР готовят следующим образом:
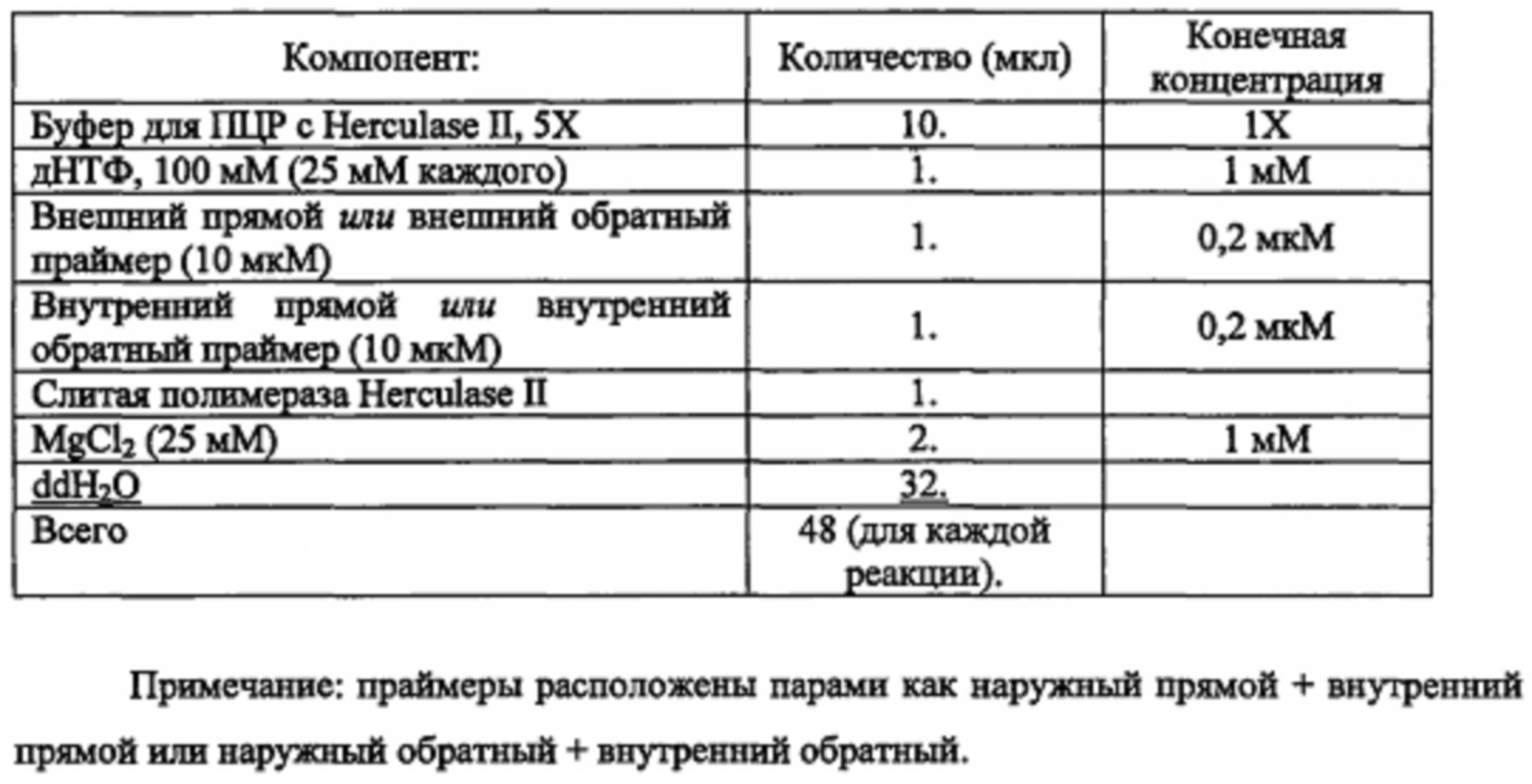
106) Заявители добавляли 100-200 нг матрицы нормализованной геномной ДНК со стадии 103 для каждой реакции.
107) Реакцию ПЦР проводили с использованием следующих условий для циклов:

108) Заявители разделяли 2-5 мкл продукта ПЦР в 1-2% геле для проверки на наличие продукта. Хотя эти условия ПЦР были разработаны для работы с большинством праймеров, для некоторых праймеров может требоваться дополнительная оптимизация путем корректировки концентрации матрицы, концентрации MgCl2 и/или температуры гибридизации.
Генотипирование для целенаправленных модификаций посредством HDR. Временные рамки ⋅ 2-3 дня, практическое выполнение 2-3 часа
109) Заявители отбирали ДНК, как описано выше с использованием раствора QuickExtract, и нормализовали геномную ДНК при помощи ТЕ до конечной концентрации 100-200 нг/мкл.
110) ПЦР амплификация модифицированного участка. Реакционную смесь для ПЦР готовили следующим образом:

111) Заявители добавляли 100-200 нг матрицы геномной ДНК со стадии 109 для каждой реакции и выполняли следующую программу.

112) Заявители прогоняли 5 мкл продукта ПЦР на 0,8-1% геле для проверки на наличие продукта в виде одной полосы. Может требоваться дополнительная оптимизация праймеров путем коррекции концентрации матрицы, концентрации MgCl2 и/или температуры гибридизации.
113) Заявители очищали реакционные смеси из реакций ПЦР с использованием набора для очистки QIAQuick PCR purification kit.
114) В одном примере HDR сайт рестрикции HindIII вводили в ген ЕМХ1. Это выявляют с помощью рестрикционного расщепления ПЦР ампликона:

i. ДНК расщепляли в течение 10 мин при 37°С:
ii. Заявители разделяли 10 мкл расщепленного продукта с загрузочным красителем в 4-20% градиентном полиакриламидном геле с ТВЕ до тех пор, пока полоса ксилол-цианола не мигрировала в нижнюю часть геля.
iii. Заявители окрашивали гель 1X красителем SYBR Gold при покачивании в течение 15 мин.
iv. Продукты расщепления визуально оценивали и количественно определяли, как описано выше, в разделе, посвященном анализу с помощью SURVEYOR. Эффективность HDR оценивали с помощью формулы: (b+с)/(а+b+с), где а представляет собой суммарную интенсивность для нерасщепленного ПЦР продукта с HDR, a b и с представляют собой суммарные интенсивности для разрезанных HindIII фрагментов.
115) В ткачестве альтернативы, очищанные ПЦР ампликоны со стадии 113 можно клонировать и генотипировать с использованием секвенирования по Сэнгеру или NGS.
Глубокое секвенирование и анализ нецелевой активности ⋅ Временные рамки 1-2 дня
Онлайн-инструмент для нацеливания CRISPR создавал кандидатные геномные нецелевые сайты для каждого идентифицированного целевого сайта. Анализ нецелевой активности в этих сайтах можно осуществлять путем анализа с помощью нуклеазы SURVEYOR, секвенирования по Сэнгеру или глубокого секвенирования нового поколения. С учетом вероятности низких или невыявляемых степеней модификации во многих из этих сайтов заявители рекомендуют глубокое секвенирование с использованием платформы Illumina Miseq для высокой чувствительности и точности. Протоколы будут изменяться с платформой для секвенирования; в данном документе заявители кратко описывали объединенный способ на основе ПЦР для прикрепления адаптеров секвенирования.
116) Конструирование праймеров для глубокого секвенирования. Праймеры для секвенирования нового поколения (NGS) конструировали для боле коротких ампликонов, как правило, с размером в диапазоне 100-200 п.о. Праймеры можно сконструировать вручную с использованием Primer-Blast от NCBI или создать с помощью онлайн-инструментов для нацеливания CRISPR (веб-сайт genome-engineering.org/tools).
117) Отбирали геномную ДНК из подвергшихся целенаправленному воздействию Cas9 клеток. Нормализовали полученную с помощью QuickExtract геномную ДНК до 100-200 нг/мкл при помощи ddH2O.
118) ПЦР для получения исходной библиотеки. С использованием праймеров для NGS со стадии 116 проводили ПЦР для получения исходной библиотеки


119) Добавляли 100-200 нг нормализованной геномной ДНК-матрицы для каждой реакции.
120) Проводили реакцию ПЦР с использованием следующих условий в циклах при не более 20 циклов амплификации:

121) Разделяли 2-5 мкл ПЦР продукта в 1% геле для проверки на наличие продукта в виде одной полосы. Как и со всеми ПЦР геномной ДНК, может потребоваться дополнительная оптимизация праймеров для NGS путем коррекции концентрации матрицы, концентрации MgCl2 и/или температуры гибридизации.
122) Очищали реакционные смеси для ПЦР с использованием набора для очистки QIAQuick PCR purification kit и нормализовали элюантом до 20 нг/мкл. Точка остановки: очищенный ПЦР продукт можно хранить при -20°С.
123) Набор для приготовления образцов ДНК Nextera XT. Следуя протоколу производителя, создавали библиотеки, готовые для секвенирования Miseq с уникальными штрих-кодами для каждого образца.
124) Анализ данных секвенирования. Анализ в отношении нецелевой активности можно проводить посредством выравнивания считанных последовательностей в программах выравнивания, таких как ClustalW, Geneious или простых скриптов для анализа последовательности.
Временные рамки
Стадии 1-2. Создание и синтез олигонуклеотидов sgRNA и ssODN: 1-5 дней, изменяются в зависимости от поставщика.
Стадии 3-5. Конструирование плазмиды с CRISPR или кассеты экспрессии для ПЦР: от 2 часов до 3 дней.
Стадии 6-53. Трансфекция в клеточные линии: 3 дня (время практического выполнения 1 час).
Стадии 54-70. Необязательное получение клональных линий: 1-3 недели, изменяются в зависимости от типа клеток.
Стадии 71-91. Функциональная валидация NHEJ с помощью SURVEYOR: 5-6 часов.
Стадии 92-124. Генотипирование посредством секвенирования по Сэнгеру или глубокого секвенирования нового поколения: 2-3 дня (время практического выполнения 3-4 часа).
Проблемные ситуации, касающиеся приведенных в данном документе примеров
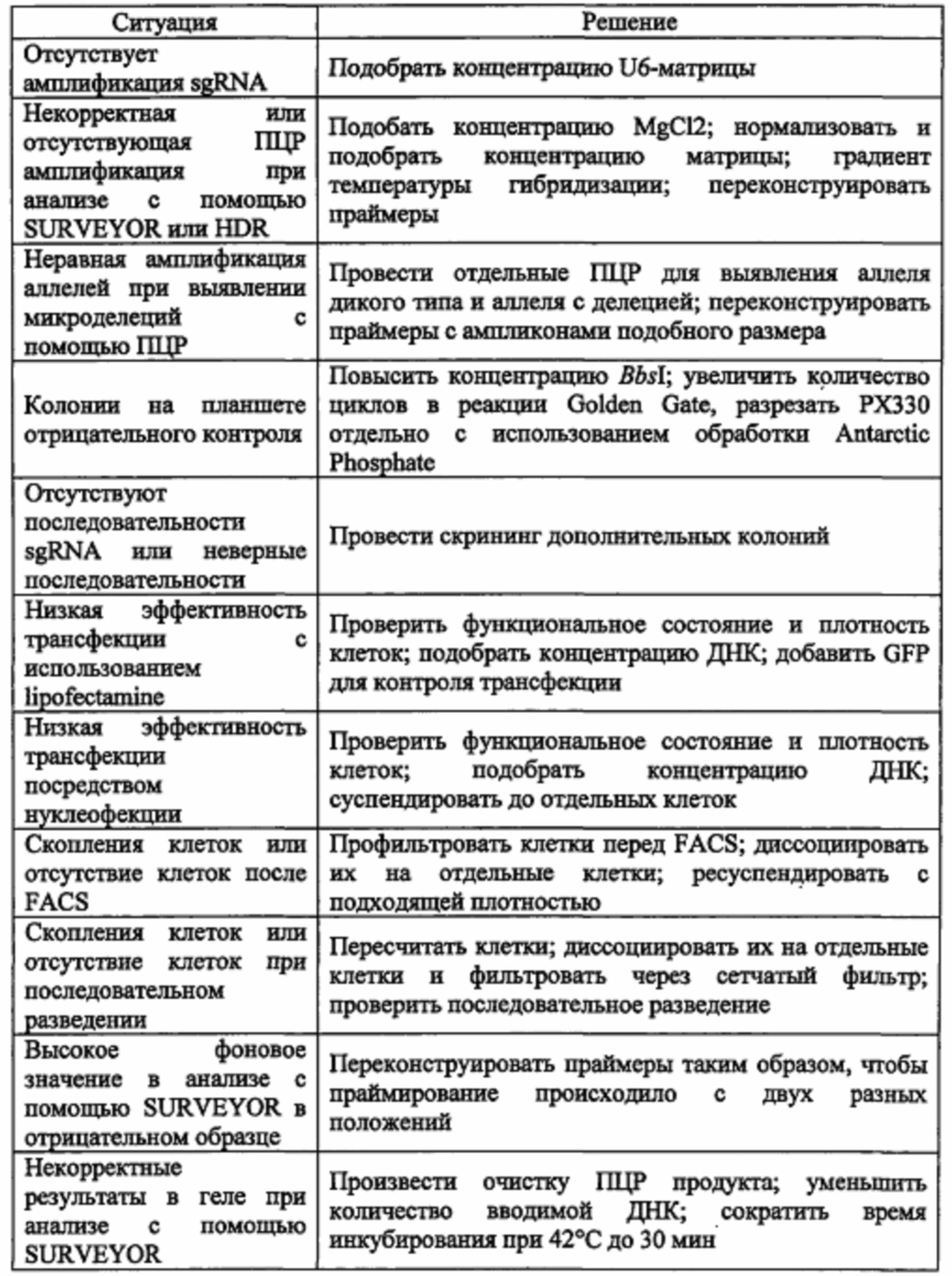

Обсуждение
Можно легко обеспечить мультиплексность CRISPR-Cas, чтобы облегчить одновременную модификацию нескольких генов и опосредован хромосомные микроделеции с высокими значениями эффективности. Заявители использовали две sgRNA для демонстрации одновременного целенаправленного воздействия на локусы GRW2B и DYRK1A человека с эффективностью до 68% в клетках HEK293FT. Подобным образом, пару sgRNA можно использовать для опосредования микроделеции, таких как вырезание экзона, которые можно генотипировать с помощью ПЦР на уровне клонов. Стоит учесть, что точное расположение соединений экзонов может изменяться. Заявители также продемонстрировали применение ssODN и вектора для целенаправленного воздействия для опосредования HDR как с использованием Cas9 дикого типа, так и никазы-мутанта Cas9 в клетках HEK 293FT и HUES9 (фигура 30). Стоит учесть, что заявители не были способны выявлять HDR в клетках HUES9 с использованием Cas9-никазы, что могло быть обусловлено низкой эффективностью или возможным различием в активности репарации в клетках HUES9. Хотя эти значения являются типичными, существует некоторая изменчивость в эффективности расщепления для заданной sgRNA, и в редких случаях определенные sgRNA могут не работать по пока еще неизвестным причинам. Заявители рекомендуют сконструировать две sgRNA для каждого локуса и исследовать их эффективность у намеченного типа клеток.
Пример 31. NLS
Модулятор транскрипции на основе Cas9: заявители собирались превратить систему CRISPR Cas9/gRNA в универсальную ДНК-связывающую систему, при помощи которой могут выполняться функции помимо расщепления ДНК. Например, путем слияния функционального домена (доменов) на каталитически неактивном Cas9 заявители придали ему новые функции, такие как активация/репрессия транскрипции, метилирование/деметилирование или модификации хроматина. Для достижения этой цели заявители получили каталитически неактивный мутант Cas9 путем замены двух остатков, необходимых для нуклеазной активности, D10 и Н840, на аланин. При мутировании этих двух остатков нуклеазная активность Cas9 устраняется при сохранении способности связываться с целевой ДНК. Функциональные домены, на которых заявители решили сфокусироваться для исследования гипотезы заявителей, представляли собой активатор транскрипции VP64 и репрессоры транскрипции SID и KRAB.
Ядерная локализация Cas9: заявители выдвинули гипотезу, что наиболее эффективный модулятор транскрипции на основе Cas9, обязательно должен быть локализован в ядре, где он будет оказывать наибольшее влияние на транскрипцию. Более того, любая остаточная Cas9 в цитоплазме может оказывать нежелательные воздействия. Заявители определили, что Cas9 дикого типа не локализуется в ядре без включения нескольких сигналов ядерной локализации (NLS) (хотя система CRISPR может не иметь одного или нескольких NLS, но преимущественно имеет по меньшей мере один или несколько NLS). Поскольку требовалось несколько последовательностей NLS, пришли к выводу о том, что сложно направить Cas9 в ядро, и любой дополнительный домен, который слит с Cas9, может нарушать ядерную локализацию. Таким образом, заявители создали четыре слитые конструкции Cas9-VP64-GFP с различными последовательностями NLS (pXRP02-pLenti2-EF1a-NLS-hSpCsn1(10A,840A)-NLS-VP64-EGFP, pXRP04-pLenti2-EF1a-NLS-hSpCsn1(10A,840A)-NLS-VP64-2A-EGFP-NLS, pXRP06-pLenti2-EF1a-NLS-EGFP-VP64-NLS-hSpCsn1(10A,840A)-NLS, pXRP08-pLenti2-EF1a-NLS-VP64-NLS-hSpCsn1(10A,840A)-NLS-VP64-EGFP-NLS). Эти конструкции клонировали в каркас на основе лентивируса с контролем экспрессии промотором EF1a человека. Элемент WPRE также добавляли для более устойчивой экспрессии белка. Каждую конструкцию трансфицировали в клетки HEK 293FT с использованием Lipofectame 2000 и визуализировали через 24 часа после трансфекции. Наилучшую ядерную локализацию получали, когда белки слияния имели последовательности NLS как на N-, так и С-конце слитого белка. Наиболее выраженная наблюдаемая ядерная локализация наблюдалась у конструкции с четырьмя NLS элементами.
Для более четкого понимания влияния NLS элементов на Cas9 заявители создали 16 слитых белков Cas9-GFP путем добавления одинаковой последовательности NLS альфа-импортина либо на N-, либо С-конец в виде тандемных повторов в количестве от нуля до трех. Каждую конструкцию трансфицировали в клетки HEK 293FT с использованием Lipofectame 2000 и визуализировали через 24 часа после трансфекции. Примечательно, что количество NLS элементов не коррелирует прямо со степенью ядерной локализации. Добавление NLS на С-конце оказывало большее влияние на ядерную локализацию, чем добавление на N-конце.
Активатор транскрипции на основе Cas9: Заявители проводили функциональное исследование белка Cas9-VP64 путем целенаправленного воздействия на локус Sox2 и количественного определения активации транскрипции с помощью RT-qPCR. Восемь целевых сайтов ДНК выбирали для охвата промотора Sox2. Каждую конструкцию трансфицировали в клетки HEK 293FT с использованием Lipofectame 2000 и через 72 часа после трансфекции общую РНК экстрагировали из клеток. 1 мкг РНК подвергали обратной транскрипции в кДНК (qScript Supermix) в 40 мкл реакционной смеси. 2 мкл продукта реакции добавляли в отдельные 20 мкл реакционной смеси для реакции qPCR в анализе TaqMan. Каждый эксперимент осуществляли в трех повторностях из биологических и методических соображений. Ни контрольная RT и ни контрольная матрица не демонстрировали амплификацию. Конструкции, которые не демонстрировали явную ядерную локализацию, pXRP02 и pXRP04, не приводили в результате к активации. Для конструкции, которая не демонстрировала явную ядерную локализацию, pXRP08, наблюдали умеренную активацию. Статистичекски значимую активацию наблюдали в случае направляющих РНК Sox2.4 и Sox2.5.
Пример 32. Данные на мышах in vivo
Материалы и реактивы
Слитая полимераза Herculase II (Agilent Technologies, кат. №600679)
10х NEBuffer 4 (NEB, кат. № B7004S)
BsaI HF (NEB, кат. № R3535S)
ДНК-лигаза Т7 (Enzymatics, кат. № L602L)
Буфер Fast Digest, 10Х (ThermoScientific, кат. № B64)
Рестриктаза NotI FastDigest (ThermoScientific, кат. № FD0594)
Щелочная фосфатаза FastAP (ThermoScientific, кат. № EF0651)
Lipofectamine2000 (Life Technologies, кат. №11668-019)
Трипсин (Life Technologies, кат. №15400054)
Щипцы №4 (Sigma, кат. № Z168777-1EA)
Щипцы №5 (Sigma, кат. № F6521-1EA)
10х сбалансированный солевой раствор Хенкса (Sigma, кат. № H4641-500ML)
Раствор пенициллина/стрептомицина (Life Technologies, кат. № Р4333)
Среда Neurobasal (Life Technologies, кат. №21103049)
Добавка В27 (Life Technologies, кат. №17504044)
L-глутамин (Life Technologies, кат. №25030081)
Глутамат (Sigma, кат. № RES5063G-A7)
β-меркаптоэтанол (Sigma, кат. № M6250-100ML)
Кроличье антитело к НА (Cell Signaling, кат. №3724S)
Набор для визуализации клеток LIVE/DEAD® Cell Imaging Kit (Life Technologies, кат. № R37601)
Шприц 30G калибра World Precision Instrument (World Precision Instruments, кат. № NANOFIL)
Стереотаксический аппарат (Kopf Instruments)
Микронасос для сверхмалых объемов UltraMicroPump3 (World Precision Instruments, кат. № UMP3-4)
Сахароза (Sigma, кат. № S7903)
Хлорид кальция (Sigma, кат. № С1016)
Ацетат магния (Sigma, кат. № М0631)
Tris-HCl (Sigma, кат. № Т5941)
EDTA (Sigma, кат. № Е6758)
NP-40 (Sigma, кат. № NP40)
Фенилметансульфонилфторид (Sigma, кат. №78830)
Хлорид магния (Sigma, кат. № М8266)
Хлорид калия (Sigma, кат. № Р9333)
β-глицерофосфат (Sigma, кат. № G9422)
Глицерин (Sigma, кат. № G9012)
Рубиновый краситель Vybrant® DyeCycle™ Ruby Stain (Life technologies, кат. № S4942)
Флуоресцентно-активированный клеточный сортер FACS Aria (Koch Institute of MIT, Кембридж, США)
Набор DNAeasy Blood & Tissue Kit (Qiagen, кат. №69504)
Процедура
Конструирование мультиплексов gRNA для применения in vivo в головном мозге
Заявители сконструировали и проводили ПЦР амплификацию gRNA, нацеленных на представителей семейств ТЕТ и DNMT (как описано в данном документе). Эффективность нацеливания оценивали на клеточной линии N2a (фигура 33). Для получения одновременной модификации нескольких генов in vivo эффективные gRNA мультиплексировали в упаковочном векторе на основе AAV (фигура 34). Для облегчения дальнейшего анализа эффективности системы заявители добавляли к системе кассету экспрессии, состоящую из слитого белка из доменов GFP-KASH под контролем промотора синапсина I человека (фигура 34). Данная модификация обеспечивала возможность дополнительного анализа эффективности системы в популяции нейронов (более подробная процедура описана в разделе Сортировка ядер и результаты in vivo).
Все 4 части системы подвергали ПЦР амплификации, применяя слитую полимеразу Herculase II с использованием следующих праймеров:
1-ый прямой праймер U6:
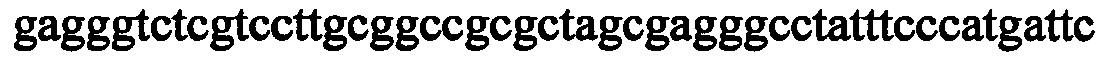
1-ый обратный праймер gRNA:

2-ой прямой праймер U6:

2-ой обратный праймер gRNA:

3-ий прямой праймер U6:

3-ый обратный праймер gRNA:

Прямой праймер hSyn_GFP-kash:

Обратный праймер hSyn_GFP-kash:

(NNNNNNNNNNNNNNNNNNNN представляет собой последовательность, обратно комплементарную целевой геномной последовательности)
Заявители использовали стратегию Golden Gate для сборки всех частей (молекулярное соотношение 1:1) системы в одностадийной реакции:


Продукт реакции Golden Gate подвергали ПЦР амплификации с использованием слитой полимеразы Herculase II и следующих праймеров:
прямой 5'

обратный 5'

Продукт ПЦР клонировали в каркас на основе AAV между последовательностями ITR с использованием сайтов рестрикции NotI:
Расщепление продукта ПЦР:

Расщепление остова на основе AAV:

Через 20 мин инкубирования при 37°С образцы очищали с использованием набора для очистки QIAQuick PCR purification kit. Стандартизированные образцы лигировали при соотношении вектор : вставка 1:3 следующим образом:

После трансформации бактерий продуктом реакции лигирования заявители подтверждали полученные клоны путем секвенирования по Сэнгеру.
Положительные клоны ДНК исследовали на клетках N2a после совместной трансфекции с конструкцией Cas9 (фигуры 35 и 36).
Создание новых конструкций Cas9 для доставки с помощью AAV
Система доставки на основе AAV, несмотря на ее характерные особенности, имеет ограничение по упаковке: для успешной доставки кассеты экспрессии in vivo она должна иметь размер < чем 4,7 т.о. Для уменьшения размера кассеты экспрессии SpCas9 и облегчения доставки заявители исследовали несколько изменений: разные промоторы, более короткий сигнал полиА и, наконец, меньший вариант Cas9 из Staphylococcus aureus (SaCas9) (фигуры 37 и 38). Все исследуемые промоторы ранее исследовались и, как описано в публикациях, являлись активными в нейронах, включая мышиный Меср2 (Gray et al., 2011), крысиный Map1b и усеченный крысиный Map1b (Liu and Fischer, 1996). Ранее было показано, что альтернативная синтетическая последовательность полиА также является функциональной (Levitt et al., 1989; Gray et al., 2011). Все клонированные конструкции экспрессировались в клетках N2a после трансфекции с использованием Lipofectamine 2000, и их исследовали при помощи вестерн-блоттинг анализа (фигура 39).
Исследование мультиплексной системы на основе AAV в первичных нейронах
Для подтверждения функциональности разработанной системы в нейронах заявители использовали первичные культуры нейронов in vitro. Нейроны коры головного мозга мыши получали согласно протоколу, опубликованному ранее Banker и Goslin (Banker and Goslin, 1988).
Клетки нейронов получали из эмбрионов на 16 день беременности. Эмбрионы извлекали из подвергнутых эвтаназии беременных самок и декапитировали, и головы помещали в охлажденный на льду HBSS. Головной мозг затем извлекали из черепов щипцами (№4 и №5) и переносили в другую сменную порцию охлажденного на льду HBSS. Дальнейшие стадии осуществляли при помощи стереоскопического микроскопа в чашке Петри, наполненной охлажденным на льду HBSS, и щипцов №5. Полушария отделяли друг от друга и ствола головного мозга и очищали от оболочек головного мозга, Гиппокампы затем очень осторожно вырезали и помещали в 15 мл коническую пробирку, заполненную охлажденным на льду HBSS. Кору, которая оставалась после вырезания гиппокампа, можно использовать для дальнейшего выделения клеток с использованием аналогичного протокола после удаления остатков ствола мозга и обонятельных луковиц. Выделенные гиппокампы промывали три раза 10 мл охлажденного на льду HBSS и диссоциировали путем 15-минутного инкубирования с трипсином в HBSS (4 мл HBSS с добавлением 10 мкл 2,5% трипсина на гиппокамп) при 37°С. После трипсинизации для удаления любых остатков трипсина гиппокампы очень осторожно промывали три раза HBSS, предварительно подогретым до 37°С, и диссоциировали в теплом HBSS. Заявители обычно диссоциировали клетки, полученные от 10-12 эмбрионов, в 1 мл HBSS с использованием 1 мл наконечников для пипетки и разводили диссоциированные клетки до 4 мл. Клетки высевали с плотностью 250 клеток/мм2 и культивировали при 37°С и 5% CO2 в течение периода до 3 недель.
HBSS
435 мл H2O
50 мл 10x сбалансированного солевого раствора Хенкса
16,5 мл 0,3 М HEPES, рН 7,3
5 мл раствора пенициллина-стрептомицина
Фильтровать (0,2 мкм) и хранить при 4°С.
Среда для посева нейронов (100 мл)
97 мл среды Neurobasal
2 мл добавки В27
1 мл раствора пенициллина-стрептомицина
250 мкл глутамина
125 мкл глутамата.
Нейроны трансдуцировали концентрированным вирусом AAV1/2 или вирусом AAV1 из фильтрованной среды клеток HEK293FT в период между 4-7 днями в культуре и выдерживали в течение по меньшей мере одной недели в культуре после трансдукции для обеспечения возможности экспрессии доставленных генов.
Управляемая AAV экспрессия в системе
Заявители подтверждали экспрессию SpCas9 и SaCas9 в культурах нейронов после доставки с помощью AAV с использованием вестерн-блоттинг анализа (фигура 42). Через одну неделю после трансдукции нейроны собирали в загрузочный буфер NuPage SDS с β-меркаптоэтанолом для денатурации белка при 95°С в течение 5 минут. Образцы разделяли в геле для SDS PAGE и переносили на мембрану из PVDF для выявления белков с помощью WB. Белки Cas9 выявляли с использованием антитела к НА.
Экспрессию Syn-GFP-kash из мультиплексной gRNA в AAV подтверждали с использованием флуоресцентной микроскопии (фигура 50).
Токсичность
Для оценки токсичности AAV с системой CRISPR заявители исследовали общую морфологию нейронов одну неделю спустя после вирусной трансдукции (фигура 45). Кроме того, заявители исследовали потенциальную токсичность сконструированной системы с использованием набора для визуализации клеток LIVE/DEAD® Cell Imaging Kit, который позволяет различать живые и мертвые клетки в культуре. Он основан на наличие внутриклеточной эстеразной активности (которая определяется по ферментативному расщеплению нефлуоресцентного кальцеина AM в интенсивно зеленый флуоресцентный кальцеин). С другой стороны, красный непроникающий в клетки компонент набора проникает только в клетки с поврежденными мембранами и связывается с ДНК, вызывая флуоресценцию у мертвых клеток. Оба флуорофора можно легко визуализировать в живых клетках с использованием флуоресцентной микроскопии. Управляемая AAV экспрессия белков Cas9 и конструкций с мультиплексными gRNA в первичных нейронах коры головного мозга хорошо переносилась и не была токсичной (фигуры 43 и 44), это указывает на то, что сконструированная система на основе AAV является подходящей для исследований in vivo.
Получение вируса
Концентрированный вирус получали согласно способам, описанным в McClure et al., 2011. Получение вируса в супернатанте происходило в клетках HEK293FT.
Хирургические операции на головном мозге
Для инъекций вирусного вектора 10-15-недельных самцов мышей C57BL/6N анестезировали смесью кетамина/ксилазина (доза кетамина 100 мг/кг и доза ксилазина 100 мг/кг) путем внутрибрюшинной инъекции. Внутрибрюшинное введение Buprenex использовали в качестве упреждающего анальгетика (1 мг/кг). Животных обездвиживали в стереотаксическом аппарате Kopf с использованием внутриушных штифтов для позиционирования и зубной планки для сохранения черепа неподвижным. С использованием ручной дрели делали отверстие (1-2 мм) в -3,0 мм позади брегмы и в 3,5 мм латерально для инъекции в участок СА1 гиппокампа. При использовании шприца от World Precision Instrument с калибром иглы 30G раствор вирусных частиц AAV в общем объеме 1 мкл инъецировали на глубину 2,5 мм. Инъекцию контролировали с помощью насоса для инъекций 'World Precision Instruments UltraMicroPump3' со скоростью потока 0,5 мкл/мин для предотвращения повреждения ткани. При завершении инъекции, иглу для инъекции извлекали медленно со скоростью 0,5 мм/мин. После инъекции кожу закрывали с использованием шовного материала 6-0 Ethilon. Животных гидратировали после операции при помощи 1 мл раствора Рингера с лактатом (подкожно) и содержали в среде с контролируемой температурой (37°С) до достижения ими восстановления активности для передвижения. Через 3 недели после хирургической операции животных подвергали эвтаназии путем глубокой анестезии с последующим удалением ткани для сортировки ядер или с перфузией 4% параформальдегидом для иммуногистохимии.
Сортировка ядер и результаты in vivo
Заявители разработали способ специфического генетического мечения ядер нейронных клеток, на которые нацеливается gRNA, с использованием GFP для флуоресцентно-активированной сортировки клеток (FACS) меченных ядер клеток и последующей обработки ДНК, РНК и ядерных белков. С этой целью разработанный заявителями вектор для мультиплексного целенаправленного воздействия конструировали для экспрессии как слитого белка из GFP и домена KASH белка ядерной мембраны (Starr DA, 2011, Current biology), так и 3 gRNA для нацеливания на специфические локусы генов, представляющие интерес (фигура 34). GFP-KASH экспрессировали под контролем промотора гена синапсина человека для специфического мечения нейронов. Слитый белок GFP-KASH имел следующие аминокислоты:

Через неделю после опосредованной AAV1/2 доставки в головной мозг наблюдали сильную' экспрессию GFP-KASH. Для FACS и последующей обработки меченных ядер гиппокампы вырезали через 3 недели после хирургического впмешательства и подвергали обработке для очистки ядер клеток с использованием стадии центрифугирования в градиенте плотности. С этой целью ткань гомогенизировали в 320 мМ сахарозе, 5 мМ CaCl, 3 мМ Mg(Ac)2, 10 мМ Tris pH7,8, 0,1 мМ EDTA, 0,1% NP40, 0,1 мМ фенилметансульфонилфториде (PMSF), 1 мМ β-меркаптоэтаноле с использованием гомогенизатора Даунса емкостью 2 мл (Sigma). Гомогенат центрифугировали в градиенте от 25% к 29% Optiprep® согласно протоколу производителя в течение 30 мин при 3500 об./мин и при 4°С. Осадок ядер ресуспендировали в 340 мМ сахарозе, 2 мМ MgCl2, 25 мМ KCl, 65 мМ глицерофосфате, 5% глицерине, 0,1 мМ PMSF, 1 мМ β-меркаптоэтаноле и добавляли красный краситель Vybrant® DyeCycle™ Ruby Stain (Life technologies) для мечения ядер клеток (обеспечивает испускание в ближней инфракрасной области спектра для ДНК). Меченные и очищенные ядра сортировали с помощью FACS с использованием флуоресцентно-активированного клеточного сортера FACS Aria и программного обеспечения BDFACS Diva. Отсортированные GFP+ и GFP- ядра в конечном итоге использовали для очистки геномной ДНК с использованием набора DNAeasy Blood & Tissue Kit (Qiagen) для анализа с помощью Surveyor подвергающихся целенаправленному воздействию участков генома. Этот же подход можно легко использовать для очистки ядерной РНК или белка от подвергающихся целенаправленному воздействию клеток для последующей обработки. Вследствие использования заявителями в данном подходе 2-векторной системы (фигура 34) ожидалось, что эффективное опосредованное Cas9 расщепление ДНК будет происходить только у небольшой субпопуляции клеток в головном мозге (клетки, которые были совместно инфицированы как вектором для мультиплексного нацеливания, так и вектором, кодирующим Cas9). Описанный в данном документе способ дает заявителям возможность специфической очистки ДНК, РНК и ядерных белков от популяции клеток, которые экспрессируют 3 представляющие интерес gRNA и, следовательно, как полагают, подвергаются опосредованному Cas9 расщеплению ДНК. При применении этого способа заявители способны визуализировать эффективное расщепление ДНК in vivo, происходящее только в небольшой субпопуляции клеток.
В сущности, здесь заявители показали целенаправленное расщепление in vivo. Более того, заявители использовали несколько подходов с несколькими разными последовательностями, на которые целенаправленное воздействие оказывалось одновременно, но независимо. Данную систему можно использовать для изучения патологических состояний головного мозга (нокаут гена, например, болезнь Паркинсона), а также для открытия области для дальнейшей разработки инструментов для редактирования генома в головном мозге. Путем замены нуклеазной активности на регуляторы транскрипции генов или эпигенетические регуляторы возможно дать ответ на весь спектр научных вопросов о роли регуляции генов и эпигенетических изменений в головном мозге не только при патологических состояниях, но также при таких физиологических процессах, как обучение и формирование памяти. Наконец, данную технологию можно применять в более сложных системах млекопитающих, таких как приматы, что позволяет преодолеть существующие на данный момент технологические ограничения.
Пример 33. Данные, полученные на модели
Специально исследовали несколько моделей заболеваний. Они включают гены CHD8, KATNAL2 и SCN2A, связанные с риском развития аутизма de novo; и ген UBE3A, связанный с синдромным аутизмом (синдром Ангельмана). Эти гены и полученные па их основе модели аутизма, конечно, являются предпочтительными, но показывают, что настоящее изобретение можно применять в отношении любого гена, и, таким образом, любая модель является возможной.
Заявители получили эти клеточные линии при использовании нуклеазы Cas9 в эмбриональных стволовых клетках человека (hESC). Линии создавали путем временной трансфекции hESC Cbh-Cas9-2A-EGFP и pU6-sgRNA. Для каждого гена конструировали две sgRNA, наиболее часто нацеливаемые на одни те же экзоны, в которых у пациентов были недавно описаны несмысловые (нокаутирующие) мутации по результатам исследований с секвенированием всего экзома пациентов с аутизмом. Плазмиды Cas9-2A-EGFP и pU6 создавали специально для этого проекта.
Пример 34. Система или протокол для получения AAV
В данном документе представлены система или протокол для получения AAV, которые были разработаны для применений в скрининге с высокой пропускной способностью и которые особенно хорошо работают с ним, но они также имеют более широкую применимость в настоящем изобретении. Манипуляция с экспрессией эндогенных генов представляет разнообразные трудности, поскольку величина экспрессии зависит от многих факторов, включая регуляторные элементы, процессинг мРНК и стабильность транскрипта. Для преодоления этой трудности заявители разработали вектор на основе аденоассоциированного вируса (AAV) для доставки. AAV имеет геном на основе ssDNA и, следовательно, является менее чувствительным к рекомбинации.
Протокол очистки концентрированного вируса AAV1/2 (серотип AAV1/2, т.е. гибридный или мозаичный капсид AAV из AAV1/AAV2) с использованием гепарина
Среда: D10 + HEPES
500 мл бутыль DMEM с высоким содержанием глюкозы + Glutamax (GIBCO)
50 мл FBS Hyclone (инактивированная нагреванием) (Thermo Fischer)
5,5 мл раствора HEPES (1 M, GIBCO)
Клетки: HEK293FT с малым числом пассажей (число пассажей <10 на момент получения вируса, произвести оттаивание новых клеток 2-4 пассажа для получения вируса, выращивать в течение 3-5 пассажей)
Реактив для трансфекции: Полиэтиленимин (PEI) "Мах"
Растворить 50 мг PEI "Max" в 50 мл стерильной воды Ultrapure H20
Довести рН до 7,1
Фильтровать с использованием фильтра в пластиковом съемном колпачке с размером отверстий 0,22 мкм
Запечатать пробирку и обернуть пленкой parafilm
Заморозить аликвоты до -20°С (для хранения, также можно использовать немедленно)
Культура клеток
Культивировать клетки HEK293FT с малым числом пассажей в D10 + HEPES
Пассажировать каждый день в соотношении от 1:2 до 1:2,5
Преимущественно не позволять клеткам достигать конфлюентности более 85%
Для Т75
- Подогреть 10 мл HBSS (-Mg2+, -Ca2+, GIBCO) + 1 мл TrypLE Express (GIBCO) на флакон до 37°С (водяная баня)
- Полностью аспирировать среду
- Осторожно добавить 10 мл теплого HBSS (чтобы полностью вымыть среду)
- Добавить 1 мл TrypLE на колбу
- Поместить колбу в инкубатор (37°С) на 1 мин
- Покачать колбу для отделения клеток
- Добавить 9 мл среды D10 + HEPES (37°С)
- Втягивать в пипетку и выталкивать клетки из пипетки 5 раз для получения суспензии отдельных клеток
- Разделить в соотношении 1:2-1:2,5 (12 мл среды для Т75) (если клетки растут медленнее или они не проявляют оптимальный рост, тогда удалить их и оттаять новую партию)
- Перенести в Т225 как только будет присутствовать достаточное количество клеток (для простоты обращения с большими количествами клеток)
Получение AAV (в масштабе 5*15 см чашек на конструкцию):
Высеять 10 миллионов клеток 21,5 мл среды в 15 см чашку
Инкубировать в течение 18-22 часов при 37°С
Трансфекция является идеальной при 80% конфлюентности
На планшет
Предварительно подогреть 22 мл среды (D10 + HEPES)
Подготовить пробирку со смесью ДНК (использовать не содержащую эндотоксина ДНК, очищенную согласно протоколу для получения больших количеств ДНК):
5,2 мкг вектора с представляющей интерес плазмидой
4,35 мкг плазмиды на основе серотипа 1 AAV
4,35 мкг плазмиды на основе серотипа 2 AAV
10,4 мкг плазмиды pDF6 (хелперные гены аденовируса) Перемешать на вихревой мешалке для смешивания
Добавить 434 мкл DMEM (без сыворотки!)
Добавить 130 мкл раствора PEI
Перемешивать па вихревой мешалке 5-10 секунд
Добавить смесь ДНК/DMEM/PEI в предварительно подогретую среду
Быстро перемешать на вихревой мешалке для смешивания
Заменить среду в 15 см чашке на смесь ДНК/DMEM/PEI
Вернуть в инкубатор с температурой 37°С
Инкубировать 48 часов перед сбором (убедиться, что среда не стала слишком кислой)
Сбор вируса:
1. осторожно аспирировать среду из 15 см чашек (преимущественно не смещая клетки)
2. Добавить 25 мл RT DPBS (Invitrogen) в каждый планшет и осторожно удалить клетки скребком для клеток. Собрать суспензию в 50 мл пробирки.
3. Осаждать клетки при 800×g в течение 10 минут.
4. Удалить супернатант
точка остановки: заморозить клеточный осадок до -80°С, если необходимо
5. ресуспендировать осадок в 150 мМ NaCl, 20 мМ Tris pH 8,0, использовать 10 мл на планшет для тканевой культуры.
6. Приготовить свежий раствор 10% дезоксихолата натрия в dH2O. Добавить 1,25 мл этого раствора на планшет для тканевой культуры для конечной концентрации 0,5%. Добавить нуклеазу бензоназу до конечной концентрации 50 единиц на мл. Тщательно перемешать пробирку.
7. Инкубировать при 37°С в течение 1 часа (водяная баня).
8. Удалить клеточный дебрис путем центрифугирования при 3000×g в течение 15 мин. Перенести в новую 50 мл пробирку и удостовериться, что весь клеточный дебрис был удален для предотвращения блокирования колонок с гепарином.
Очистка AAV1/2 на колонке с гепарином:
1. Настроить колонки HiTrap с гепарином с использованием перистальтического насоса таким образом, чтобы поток растворов через колонку составлял 1 мл в минуту. Важно удостовериться, что пузырьки воздуха не попали в колонку с гепарином.
2. Уравновесить колонку 10 мл 150 мМ NaCl, 20 мМ Tris, pH 8,0 с использованием перистальтического насоса.
3. Связывание вируса: Внести 50 мл раствора вируса на колонку и обеспечить возможность его прохождения через нее.
4. Стадия промывки 1: колонка с 20 мл 100 мМ NaCl, 20 мМ Tris, pH 8,0 (использовать перистальтический насос)
5. Стадия промывки 2: С использованием 3 мл или 5 мл шприца продолжать промывать колонку 1 мл 200 мМ NaCl, 20 мМ Tris, pH 8,0, затем промывать 1 мл 300 мМ NaCl, 20 мМ Tris, pH 8,0.
Прекратить промывание.
(приготовить шприцы с различными буферами во время 50 мин протекания раствора впруса сверху)
6. Элюирование. С использованием 5 мл шприцев и небольшого давления (скорость потока <1 мл/мин) элюировать вирус из колонки путем применения:
1,5 мл 400 мМ NaCl, 20 мМ Tris, pH 8,0
3,0 мл 450 мМ NaCl, 20 мМ Tris, pH 8,0
1,5 мл 500 мМ NaCl, 20 мМ Tris, pH 8,0
Собрать его в 15 мл центрифужную пробирку.
Концентрирование AAV1/2:
1. Стадия концентрирования 1: Концентрировать элюированный вирус с использованием 15 мл блоков для корзиночной центрифуги с фильтрующей поверхностью Amicon ultra с порогом отсечения по молекулярному весу 100000. Загрузить элюат из колонки в концентратор и центрифугировать при 2000×g в течение 2 мл (при комнатной температуре). Проверить объем сконцентрированного вируса - он должен составлять примерно 500 мкл. При необходимости центрифугировать с интервалами 1 мил до достижения правильного объема.
2. замена буфера: Добавить 1 мл стерильного DPBS в фильтрующий блок, центрифугировать с 1 мин интервалами до достижения правильного объема (500 мкл).
3. Стадия концентрирования 2: Добавить 500 мкл концентрата в фильтрующий блок 0,5 мл 100K Amicon Ultra. Центрифугировать при 6000 g в течение 2 мин. Проверить объем сконцентрированного вируса - он должен составлять примерно 100 мкл. При необходимости центрифугировать с интервалами 1 мин до достижения правильного объема.
4. Извлечение: Перевернуть фильтрующий элемент и вставить в новую пробирку для сбора. Центрифугировать при 1000 g в течение 2 мин.
Аликвотировать и заморозит до -80°С
Как правило, на один участок инъекции требуется 1 мкл, следовательно, рекомендуются небольшие аликвоты (например, 5 мкл) (во избежание замораживания-оттаивания вируса).
Определить титр устойчивых к ДНКазе I GC частиц с использованием qPCR (см отдельный протокол)
Материалы
Amicon Ultra, 0,5 мл, 100K; MILLIPORE; UFC510024
Amicon Ultra, 15 мл, 100K; MILLIPORE; UFC910024
Нуклеаза бензоназа; Sigma-Aldrich, E1014
Картридж с гепарином HiTrap; Sigma-Aldrich; 54836
Дезоксихолат натрия; Sigma-Aldrich; D5670
Протокол получения супернатанта AAV1
Среда: D10 + HEPES
500 мл бутыль DMEM с высоким содержанием глюкозы + Glutamax (Invitrogen)
50 мл FBS Hyclone (инактивированная нагреванием) (Thermo Fischer)
5,5 мл раствора HEPES (1 М, GIBCO)
Клетки: HEK293FT с малым числом пассажей (число пассажей <10 на момент получения вируса)
Произвести оттаивание новых клеток 2-4 пассажа для получения вируса, выращивать в течение 2-5 пассажей
Реактив для трансфекции: Полиэтиленимин (PEI) "Max"
Растворить 50 мг PEI "Мах" в 50 мл стерильной воды Ultrapure H20
Довести рН до 7,1
Фильтровать с использованием фильтра в пластиковом съемном колпачке с размером отверстий 0,22 мкм
Запечатать пробирку и обернуть пленкой parafilm
Заморозить аликвоты до -20°С (для хранения, также можно немедленно использовать)
Культура клеток
Культивировать HEK293FT с малым числом пассажей в D10 + HEPES. Пассажировать каждый день в соотношении от 1:2 до 1:2,5
Преимущественно позволять клеткам достигать более 85% конфлюентности
Для Т75
- Подогреть 10 мл HBSS (-Mg2+, -Са2+, GIBCO) + 1 мл TrypLE Express (GIBCO) на флакон до 37°С (водяная баня)
- Полностью аспирировать среду
- Осторожно добавить 10 мл теплого HBSS (чтобы полностью вымыть среду)
- Добавить 1 мл TrypLE на колбу
- Поместить колбу в инкубатор (37°С) на 1 мин
- Покачать колбу для отделения клеток
- Добавить 9 мл среды D10 + HEPES (37°С)
- Втягивать в пипетку и выталкивать клетки из пипетки 5 раз для получения суспензии отдельных клеток
- Разделить в соотношении 1:2-1:2,5 (12 мл среды для Т75) (если клетки растут медленнее или они не проявляют оптимальный рост, тогда удалить их и оттаять новую партию)
- Перенести в Т225 как только будет присутствовать достаточное количество клеток (для простоты обращения с большими количествами клеток)
Получение AAV (в масштабе одной 15 см чашки)
Высеять 10 миллионов клеток в 21,5 мл среды в 15 см чашку
Инкубировать в течение 18-22 часов при 37°С
Трансфекция является идеальной при 80% конфлюентности на планшет
Предварительно подогреть 22 мл среды (D10 + HEPES)
Подготовить пробирку со смесью ДНК (использовать не содержащую эндотоксина ДНК, очищенную согласно протоколу для получения больших количеств ДНК):
5,2 мкг вектора с представляющей интерес плазмидой
8,7 мкг плазмиды на основе серотипа 1 AAV
10,4 мкг плазмиды DF6 (хелперные гены аденовируса)
Перемешать на вихревой мешалке для смешивания
Добавить 434 мкл DMEM (без сыворотки!) Добавить 130 мкл раствора PEI
Перемешивать на вихревой мешалке 5-10 секунд
Добавить смесь ДНК/DMEM/PEI в предварительно подогретую среду
Быстро перемешать на вихревой мешалке для смешивания
Заменить среду в 15 см чашке на смесь ДНК/DMEM/PEI
Поместить обратно в инкубатор с температурой 37°С
Инкубировать 48 часов перед сбором (преимущественным является контроль, чтобы удостовериться в том, что среда не становится слишком кислой)
Сбор вируса:
Удалить супернатант из 15 см чашки
Фильтровать через фильтр с размером ячеек 0,45 мкм (низкая степень связывания белка). Разделить на аликвоты и заморозить до -80°С
Трансдукция (первичные культуры нейронов в 24-луночном формате, 5DIV)
Заменить полную среду neurobasal в каждой лунке с нейронами, которую нужно трансдуцировать, свежей средой neurobasal (обычно заменяют 400 мкл из 500 мкл в лунке)
Произвести оттаивание супернатанта AAV в водяной бане с температурой 37°С
Дать уравновеситься в инкубаторе в течение 30 мин
Добавить 250 мкл супернатанта AAV в каждую лунку
Инкубировать 24 часа при 37°С
Удалить среду/супернатант и заменить свежей полной средой neurobasal
Экспрессия становится видимой спустя 48 часов, насыщение приблизительно 6-7 дней после инфицирования
Конструкции для плазмиды pAAV с GOI должны иметь размер, не превышающий 4,8 т.п.о., включая обе ITRS.
Пример кодон-оптимизированной для человека последовательности (т.е. являющейся оптимизированной для экспрессии у людей): SaCas9 приведена ниже:


Пример 35. Сведение к минимуму нецелевого расщепления с использованием никазы Cas9 и двух направляющих РНК
Cas9 представляет собой направляемую РНК ДНК-нуклеазу, которая может целенаправленно воздействовать на специфические положения в геноме с помощью РНК-гида из 20 п.о. Тем не менее, направляющая последовательность может допускать некоторые несовпадения между направляющей последовательностью и последовательностью ДНК-мишени. Данная гибкость является нежелательной вследствие потенциала в отношении нецелевого расщепления, когда направляющая РНК нацеливает Cas9 на нецелевую последовательность, которая отличается небольшим числом оснований от направляющей последовательности. Для всех экспериментальных применений (целенаправленное воздействие на гены, генная инженерия сельскохозяйственных культур, терапевтические применения и т.д.) важна способность к улучшению специфичности опосредованного Cas9 целенаправленного воздействия на гены и снижению вероятности нецелевой модификации с использованием Cas9.
Заявители разработали способ применения мутанта Cas9-никазы в сочетании с двумя направляющими РНК для облегчения целенаправленных двухнитевых разрывов в геноме без нецелевых модификаций. Мутантную никазу Cas9 можно получить из нуклеазы Cas9 путем блокирования ее активности в отношении расщепления для того, чтобы вместо обеих нитей в дуплексе ДНК она расщепляла только одну нить. Никазу Cas9 можно получить путем индукции мутаций в одном или нескольких доменах нуклеазы Cas9; например, Ruvc1 или HNH. Данные мутации могут включать без ограничения мутации в каталитическом домене Cas9, например, в SpCas9 эти мутации могут находиться в положениях D10 или Н840. Данные мутации могут включать без ограничения D10A, Е762А, Н840А, N854A, N863A или D986A в SpCas9, но никазы можно получать путем индукции мутаций в соответствующих положениях у других ферментов CRISPR или ортологов Cas9. В наиболее предпочтительном варианте осуществления настоящего изобретения мутантная никаза Cas9 представляет собой никазу SpCas9 с мутацией D10A.
Это работает таким образом, что каждая направляющая РНК в комбинации с никазой Cas9 будет индуцировать целенаправленный однонитевой разрыв в присутствующей в виде дуплекса ДНК-мишени. Поскольку с помощью каждой направляющей РНК обеспечивается надрезание одной нити, окончательный результат представляет собой двухнитевой разрыв. Причина, по которой этот способ устраняет нецелевые мутации, заключается в том, что поскольку очень маловероятно, чтобы она имела нецелевой сайт, который характеризуется высокими степенями сходства в отношении обеих направляющих последовательностей (20 п.о. + 2 п.о.(РАМ) = 22 п.о. - специфичность в отношении каждого гида, а два гида означают, что любой нецелевой сайт должен будет иметь гомологичную последовательность близко к 44 п.о.). Хотя все еще существует вероятность, что отдельные гиды могут иметь нецелевые мишени, эти нецелевые мишени будут только надрезанными, которые маловероятно будут репарироваться посредством мутагенного NHEJ процесса. Таким образом, мультиплексирование надрезания двух нитей ДНК обеспечивает действенный метод введения целенаправленных двухнитевых разрывов ДНК без нецелевых мутагенных эффектов.
Заявители проводили эксперименты, включающие совместную трансфекцию клеток HEK293FT плазмидой, кодирующей никазу Cas9(D10A), а также кассеты экспрессии с ДНК для одного или нескольких гидов. Заявители трансфицировали клетки с использованием Lipofectamine 2000 и трансфицированные клетки собирали через 48 или 72 часов после трансфекций. Индуцированные двойным надрезанием NHEJ выявляли с использованием анализа с помощью нуклеазы SURVEYOR, как описано в данном документе выше (фигуры 51, 52 и 53).
Заявители дополнительно идентифицировали параметры, связанные с эффективным расщеплением мутантной никазой Cas9 в сочетании с двумя направляющими РНК, и эти параметры включают без ограничения длину "липкого" 5'-конца. Об эффективном расщеплении сообщается для "липкого" 5'-конца по меньшей мере из 26 пар оснований. В предпочтительном варианте осуществления настоящего изобретения "липкий" 5'-конец содержит по меньшей мере 30 пар оснований и, более предпочтительно, по меньшей мере 34 пары оснований. "Липкие" концы из оснований в количестве до 200 пар оснований могут быть приемлемыми для расщепления, хотя "липкие" 5'-концы менее чем из 100 пар оснований являются предпочтительными, и "липкие" 5'-концы менее чем из 50 пар оснований являются наиболее предпочтительными (фигуры 54 и 55).
Пример 36. Протоколы исследований поведения
Приподнятый крестообразный лабиринт. Приподнятый крестообразный лабиринт используют в оценке поведения, подобного тревожности, используя конфликт между врожденным страхом, который грызуны испытывают к открытым областям, и их желанием исследовать новые окружения.
Аппарат, используемый для теста в приподнятом крестообразном лабиринте, выполнен из нержавеющей стали и состоит из четырех рукавов (два открытых без стенок и два огороженных стенками высотой 15 см) длиной 35 см и шириной 5 см. Каждый рукав лабиринта прикреплен к крепким металлическим ножкам так, чтобы он был приподнят на 40 см над полом.
Мышей содержали в количестве до трех на клетку в комнате с циклом чередования 12 часов освещения/темноты с доступом к корму и воде ad libitum.
Исследование поведения осуществляли в период между 9:00 до полудня и 6:00 после полудня в ярко освещенной комнате. Все клетки, в которых содержались мыши, переносили в комнату для исследования поведения за 30 мин до начала первых испытаний.
Мышей затем извлекали из их клетки и помещали в место соединения открытых и закрытых рукавов, повернув их мордой к открытому рукаву, напротив которого находился экспериментатор, и позволяли свободно изучать лабиринт в течение 5 мин. За это время активность животных записывалась и их поведение отслеживалось. Суммарное расстояние, пройденное в открытых рукавах, время, проведенное в открытых рукавах, общее число переходов между рукавами и время ожидания до входа в открытый рукав измерялись автоматически при помощи программного обеспечения Noldus. В конце 5-минутного теста грызунов удаляли из крестообразного лабиринта и помещали в транспортировочную клетку. Помещали обратно в клетку, в которой они содержались. Приподнятый крестообразный лабиринт тщательно вычищали и вытирали насухо бумажными полотенцами перед тестированием другой мыши.
Выработка условного рефлекса в связи с переживанием чувства страха
Животные: Самцы мышей возрастом 4 недели (BL6J/57); 10 на группу
Электроболевое раздражение лап: 2 с; 0,5 мА
Протокол включал три фазы:
1. Экспозиция, которая позволяла мышам акклиматизироваться и ознакомиться с тренировочной камерой. Для непарной группы: 2 мин акклиматизации, за которой следовала подача 20-секундного звукового сигнала с последующим 80-секупдным интервалом, повторяемые шесть раз. Для парной группы период акклиматизации составлял 12 мин.
2. Тренировка; мышам подавали CS- и или US-стимулы. Период акклиматизации -4 мин. Шесть испытаний по выработке условного рефлекса, включающие звуковой сигнал (CS) и электроболевое раздражение (US) проводили периодами испытаний, которые длились 100 секунд (включая интервал между воздействиями). Для парных групп каждое испытание состояло из 20-секундного "исходного" интервала, 20-секундной подачи сигнала, 18-секундного остаточного интервала, 2-секундного электроболевого раздражения и 40-секундного интервала после электроболевого раздражения. Испытания были одинаковыми для непарных групп мышей за исключением того, что сигнал упускали.
3. Тестирование; мышей наблюдали в отношении замирания в ответ на каждый набор условий и на звуковой сигнал и остаточный интервал перед CS. Каждую фазу проводили с 24-часовыми интервалами. Мышам давали возможность свободно передвигаться в условиях тренировки (для ознакомления) и теста (изменено; плоская поверхность на сетчатую, черно-белые узоры на стенах; запах ванилина) в течение 3 мин. Все мыши получали четыре 100-секундных испытания при тестировании. Для всех мышей каждое испытание начиналось с 20-секундного интервала, и после 20-секундной подачи звукового сигнала следовал 60-секундный интервал.
Оценивание: Все процедуры оценивания выполняли, основываясь па видеозаписи. % замирающих мышей наблюдали в 5-секундных временных отрезках в каждом наборе условий воздействия в тесте и в каждом 100-секундном периоде испытаний. Замирание оценивали как 1 или 0 в каждом временном промежутке, если животное замирало по меньшей мере на 2 с.
Данные представленные на столбчатых диаграммах представляют собой средние значения для группы из 7 животных, "усы" представляют SEM. Данные на линейных диаграммах представляют процесс тренировки/тестирования во времени, и каждая точка представляет собой среднее значение для 7 животных из 6 или 4 испытаний (протокол тренировки и тестирования, соответственно). "Усы" представляют SEM.
"Открытое поле" Аппарат для теста "открытое поле" широко используется для оценки исследовательского поведения и валидирована для применения при измерении связанного с тревожностью поведения. Животных помещали в квадратную арену (40×40 см) на 10 мин, в течение которых записывалось их поведение. Оценивали общее пройденное расстояние, скорость и время, проведенное в центре арены.
Распознавание нового объекта Тест на распознавание нового объекта (NOR) является популярным тестом для исследования обучения и памяти у грызунов. Он основан на их склонности к большему взаимодействию с новым объектом, чем со знакомым объектом. В целом, животных вначале помещали в аппарат и позволяли изучать объект. После предписанного интервала животное возвращали в аппарат, которые теперь содержал знакомый объект и новый объект. Распознавание объекта отличали по большему периоду времени, затраченного на взаимодействие с новым объектом. Задача с NOR является особенно привлекательной, поскольку не требует внешней мотивации, награды или наказания, а требует небольшой тренировки или ознакомления, и его можно завершить в сравнительно короткий срок.
I Предварительная тренировка:
1. После прибытия в колонию животным давали возможность акклиматизироваться в течение по меньшей мере 3-7 дней.
2. По меньшей мере за 3 дня до начала эксперимента животных ежедневно брали в руки по меньшей мере на 3 мин в день и подвергали воздействию стандартных процедур транспортировки.
3. Ознакомление со средой, в которой проводилось тестирование: за 24 часа до эксперимента животных выпускали на арену для тестирования (40×40×35 см, прозрачные стенки) в комнате, где изучалось поведение, на 10 мин.
II Тренировка по распознаванию объекта:
1. Два 'идентичных' объекта, с которыми нужно ознакомиться (образцы), помещали в задний левый и правый углы аппарата.
2. Животных помещали посередине относительно стенки напротив тех же объектов, что предотвращает какое-либо случайное отклонение при помещении животного, при котором животное было бы преимущественно ориентировано на конкретную сторону/объект.
3. Экспериментатор записывал поведение животного с использованием камеры, оставаясь за занавеской с тем, чтобы служить в качестве сигнала для животного или вводить в исследование случайное отклонение.
4. После планируемого времени экспонирования образца-объекта (5-10 мин) животное удаляли из аппарата и возвращали в колонию на время запланированного интервала между тренировкой и тестированием. Аппарат очищают 70% этанолом после каждого животного.
III Фаза отсрочки Наиболее часто используемые интервалы между тренировкой и тестированием варьировали от 1 часа (для надежного распознавания объекта) до 24 часов.
IV Тест по распознаванию объекта:
1. Для тестирования в отношении распознавания объекта после интервала между тренировкой и тестированием один из знакомых объектов-образцов помещали в один задний угол аппарата; новый объект помещали в другой задний угол.
2. Животное помещали в аппарат, как на стадии II.2. Экспериментатор записывал поведение животного, оставаясь за занавеской.
3. После планируемого времени экспонирования (3-5 мин) животных удаляли из аппарата и возвращали в колонию. Аппарат очищали 70% этанолом после каждого животного.
Анализ данных: записанные видеозаписи использовали для последующего анализа данных: а) оценка непосредственного контакта (общая продолжительность контакта животного с объектом); b) оценка контакта в ближней зоне (животное "взаимодействует" с объектом, когда его нос находится в контакте с объектом или направлен на объект в пределах минимального расстояния 2 см). Наиболее часто используемые единицы измерения включают время со знакомым объектом в сравнении с новым объектом, оценка различения (взаимодействие с новым объектом - взаимодействие со знакомым объектом), дискриминационное отношение (взаимодействие с новым объектом/общее взаимодействие с обоими объектами). Распознавание объекта в этих единицах измерения отражается большим временем взаимодействия с новым объектом, нежели с известным, положительной оценкой различения или дискриминационным отношением выше 0,5, соответственно.
Лабиринт Барнс Кэрол Барнс разработала тест с сухопутным лабиринтом для пространственного обучения и памяти в 1979 году, в котором животные покидали ярко освещенную, открытую поверхность круглой платформы через небольшую темную углубленную камеру, расположенную под одним из 18 отверстий по периметру платформы. Хотя он был первоначально изобретен для крыс, лабиринт Барнс (ВМ) стал более популярным для оценки пространственной памяти у мышей, используя преимущество их отличных способностей искать и убегать через небольшие отверстия.
Процедура:
1. Адаптация. Мышь помещали в цилиндрическую черную стартовую камеру посередине лабиринта. По истечении 10 с камеру удаляли и включали зуммер вместе с ярким светом/ Мышь осторожно направляли к боксу-убежищу. Как только мышь оказывалась внутри бокса, зуммер и свет выключали. Мышь должна оставаться в боксе-убежище в течение 2 мин.
2. Пространственное восприятие. Мышь помещали в цилиндрическую черную стартовую камеру посередине лабиринта. Через 10 с камеру поднимали и включали зуммер и свет. Мыши позволяли исследовать лабиринт в течение 3 минут. Испытание заканчивалось, когда мышь попадала в целевой туннель, или по истечении 3 минут. Сразу после попадания мыши в туннель зуммер и свет выключали, и мыши позволяли оставаться в туннеле в течение 1 мин. Если мышь не достигала цели за 3 минуты, экспериментатор осторожно направлял ее к боксу-убежищу и оставлял мышь внутри на 1 мин. Мышь оставалась в клетке для постоянного содержания до следующего испытания. Полное испытание повторяли 4 раза с интервалами 15 минут в течение 3-4 дней.
3. Испытание на долговременную память. Через 24 часа после последнего дня тренировки проводили контрольное испытание. Целевое отверстие должно быть закрыто. Животное помещали на середину лабиринта под цилиндрическую черную стартовую камеру и через 10 с камеру удаляли, включали зуммер и свет. Мышь удаляли из лабиринта через 90 с. Контрольное испытание выполняли с целью определения того, запоминает ли животное, где расположена цель. Измеряли число неуспевающих (ошибающихся) для каждого отверстия и время ожидания и длину пути для практического достижения целевого отверстия.
Пример 37. Обновление SaCas9
На фигуре 69 показан вектор AAV-Sa-Cas9, специфичный для печени вектор AAV-Sa-Cas9 и альтернативный вектор AAV-Sa-Cas9.
На фигуре 70 показаны данные по оптимизированному вектору CMV-SaCas9-NLS-U6-sgRNA (вектор, предоставленный в последний раз); новые данные сравнивали SaCas9 с меткой на N'-конце с SaCas9 с меткой на С'-конце и показывают повышенную эффективность расщепления с использованием NLS мечения по C'-концу.
Новые мишени выбирали в экзонах 4 и 5 в гене Pcsk9 мыши (их последовательности и местоположения приведены ниже). Экзоны 4-5 лежат ниже участка N-концевого пре-домена в Pcsk9, который протеолитически расщепляется при созревании белков, и вставки/делении в этом нижележащем участке, как ожидается, приводят к деградации белков. Pcsk9 вовлечен в циклические изменения и отрицательную регуляцию LDL-рецептора, и потеря Pcsk9 должна опосредованно приводить к пониженным уровням холестерина в плазме.
SgRNA колнировали в вектор на основе AAV и тестировали в отношении активности создания вставок/делений в клеточной линии гепатоцитов мыши Hepa1-6 при трансфекции одним вектором. 500 нг вектора трансфицировали в 200000 клеток Hepa1-6 с использованием Lipofectamine 2000 и ДНК отбирали для анализа с помощью SURVEYOR, показывающего расщепление Pcsk9.
pAAV-CMV-SaCas9-NLS-U6-sgRNA(231: atctcttagataccagcate)
pAAV-CMV-SaCas9-NLS-U6-sgRNA(232: tcaatctcccgatgggcacc)
pAAV-CMV-SaCas9-NLS-U6-sgRNA(233: gcccatcgggagattgaggg)
pAAV-CMV-SaCas9-NLS-U6-sgRNA(234: acttcaacagcgtgccggag)
pAAV-CMV-SaCas9-NLS-U6-sgRNA(235: ccgctgaccacacctgccag)
pAAV-CMV-SaCas9-NLS-U6-sgRNA(236: tggcaggtgtggtcagcggc)
На фигуре 71 показано изображение результатов анализа с помощью SURVEYOR, показывающее вставки/делении, образованные в новых мишенях Pcsk9.
На фигуре 72 показана специфичность SaCas9: нецелевые сайты во всем геноме (GWOT) предсказывают на основании 2 критериев: они содержат 4 или менее несовпадающих оснований по сравнению с предполагаемой мишенью для SaCas9 и имеют наименее ограничительный РАМ для SaCas9, NNGRR. Клетки HEK 293FT трансфицировали либо SpCas9, либо SaCas9 с их соответствующими SgRNA в целевом сайте (ЕМХ1: TAGGGTTAGGGGCCCCAGGC), имеющем CGGGGT в качестве РАМ, так что он может разрезаться либо SpCas9 (CGG), либо SaCas9 (CGGGGT). ДНК из клеток собирали и анализировали с помощью технологии секвенирования Illumina в отношении вставок/делеций в целевом локусе и в 41 прогнозируемых нецелевых локусов (следуя протоколам из Hsu et al. Nature Biotech 2013, и системы для анализа данных, разработанной David Scott и Josh Weinstein).
На фигуре 73 показано, что SaCas9 может иметь более высокий уровень нецелевой активности, чем SpCas9, в определенных локусах.
Несмотря на то, что предпочтительные варианты осуществления настоящего изобретения были показаны и описаны в данном документе, для специалиста в данной области будет очевидно, что такие варианты осуществления предоставлены только в качестве примера. Многочисленные вариации, изменения и замены теперь будут очевидны для специалиста в данной области без отступления от сути настоящего изобретения. Следует понимать, что различные альтернативные варианты вариантов осуществления настоящего изобретения, раскрытые в данном документе, можно применять при практическом осуществлении настоящего изобретения.
Перечень использованной литературы
Banker G, Goslin K. Developments in neuronal cell culture. Nature. 1988 Nov 10; 336(6195): 185-6.
Bedell, V.M. et al. In vivo genome editing using a high-efficiency TALEN system. Nature 491, 114-U133 (2012).
Bhaya, D., Davison, M. & Barrangou, R. CRISPR-Cas systems in bacteria and archaea: versatile small RNAs for adaptive defense and regulation. Annu Rev Genet 45, 273-297 (2011).
Bobis-Wozowicz, S., Osiak, A., Rahman, S.H. & Cathomen, T. Targeted genome editing in pluripotent stem cells using zinc-finger nucleases. Methods 53, 339-346 (2011).
Boch, J. et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science 326, 1509-1512 (2009).
Bogenhagen, D.F. & Brown, D.D. Nucleotide sequences in Xenopus 5S DNA required for transcription termination. Cell 24, 261-270 (1981).
Bultmann, S. et al. Targeted transcriptional activation of silent oct4 pluripotency gene by combining designer TALEs and inhibition of epigenetic modifiers. Nucleic Acids Res 40, 5368-5377(2012).
Carlson, D.F. et al. Efficient TALEN-mediated gene knockout in livestock. Proc Natl Acad Sci USA 109, 17382-17387 (2012).
Chen, F.Q. et al. High-frequency genome editing using ssDNA oligonucleotides with zinc-finger nucleases. Nat Methods 8, 753-U796 (2011).
Cho, S.W., Kim, S., Kim, J.M. & Kim, J.S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nat Biotechnol 31, 230-232 (2013).
Christian, M. et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics 186, 757-761 (2010).
Cong, L. et al. Multiplex genome engineering using CRISPR-Cas systems. Science 339, 819-823 (2013).
Deltcheva, E. et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature 471, 602-607 (2011).
Deveau, H., Gameau, J.E. & Moineau, S. CRISPR-Cas system and its role in phage-bacteria interactions. Annu Rev Microbiol 64, 475-493 (2010).
Ding, Q. et al. A TALEN genome-editing system for generating human stem cell-based disease models. Cell Stem Cell 12, 238-251 (2013).
Gameau, J.E. et al. The CRISPR-Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature 468, 67-71 (2010).
Gashmas, G., Barrangou, R., Horvath, P. & Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci USA 109, E2579-2586 (2012).
Geurts, A.M. et al. Knockout Rats via Embryo Microinjection of Zinc-Finger Nucleases. Science 325, 433-433 (2009).
Gray SJ, Foti SB, Schwartz JW, Bachaboina L, Taylor-Blake B, Coleman J, Ehlers MD, Zylka MJ, McCown TJ, Samulski RJ. Optimizing promoters for recombinant adeno-associated virus-mediated gene expression in the peripheral and central nervous system using self-complementary vectors. Hum Gene Ther. 2011 Sep; 22(9): 1143-53. doi: 10.1089/hum.2010.245.
Guschin, D.Y. et al. A rapid and general assay for monitoring endogenous gene modification. Methods Mol Biol 649, 247-256 (2010).
Hasty, P., Rivera-Perez, J. & Bradley, A. The length of homology required for gene targeting in embryonic stem cells. Mol Cell Biol 11, 5586-5591 (1991).
Horvath, P. & Barrangou, R. CRISPR-Cas, the immune system of bacteria and archaea. Science 327, 167-170 (2010).
Hsu, P.D. & Zhang, F. Dissecting neural function using targeted genome engineering technologies. ACS ChemNeurosci 3, 603-610 (2012).
Hwang, W.Y. et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nat Biotechnol 31, 227-229 (2013).
Jiang, W., Bikard, D., Cox, D., Zhang, F. & Marraffini, L.A. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nat Biotechnol 31, 233-239 (2013).
Jinek, M. et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816-821 (2012).
Jinek, M. et al. RNA-programmed genome editing in human cells. eLife 2, e00471 (2013).
Kaplitt, M.G., et al., Safety and tolerability of gene therapy with an adeno-associated virus (AAV) borne GAD gene for Parkinson's disease: an open label, phase I trial. Lancet. 2007 Jun 23; 369(9579): 2097-105.
Levitt N. Briggs D. Gil A. Proudfoot N.J. Definition of an efficient synthetic poly(A) site. Genes Dev. 1989; 3: 1019-1025.
Liu D, Fischer I. Two alternative promoters direct neuron-specific expression of the rat microtubule-associated protein 1B gene. J Neurosci. 1996 Aug 15; 16(16): 5026-36.
Lopes, V.S., etc al.. Retinal gene therapy with a large MYO7A cDNA using adeno-associated virus. Gene Ther, 2013 Jan 24. dot: 10.1038/gt 2013.3. [Epub ahead of print]
Mahfouz, M.M. et al. De novo-engineered transcription activator-like effector (TALE) hybrid nuclease with novel DNA binding specificity Cre ates double-strand breaks. Proc Natl Acad Sci USA 108, 2623-2628 (2011).
Makarova, K.S. et al. Evolution and classification of the CRISPR-Cas systems. Nat Rev Microbiol 9, 467-477 (2011).
Mali, P. et al. RNA-guided human genome engineering via Cas9. Science 339, 823-826 (2013).
McClure C, Cole KL, Wuiff P, Klugmann M, Murray AJ. Production and titering of recombinant adeno-associated viral vectors. J Vis Exp. 2011 Nov 27; (57): e3348. doi: 10.3791/3348.
Michaelis, L.M., Maud "Die kinetik der invertinwirkung.". Biochem. z (1913).
Miller, J.C. et al. An improved zinc-finger nuclease architecture for highly specific genome editing. Nat Biotechnol 25, 778-785 (2007).
Miller, J.C. et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol 29, 143-148 (2011).
Moscou, M.J. & Bogdanove, A.J. A simple cipher governs DNA recognition by TAL effectors. Science 326, 1501 (2009). Porteus, M.H. & Baltimore, D. Chimeric nucleases stimulate gene targeting in human cells. Science 300, 763 (2003).
Mussolino, C. et al. A novel TALE nuclease scaffold enables high genome editing activity in combination with low toxicity. Nucleic acids research 39, 9283-9293 (2011).
Nathwani, A.C., et al., Adenovirus-associated virus vector-mediated gene transfer in hemophilia B. N Engl J Med. 2011 Dec 22; 365(25): 2357-65. doi: 10.1056/NEJMoa1108046. Epub 2011 Dec 10.
Oliveira, T.Y. et al. Translocation capture sequencing: a method for high throughput mapping of chromosomal rearrangements. J Immunol Methods 375, 176-181 (2012).
Perez, E.E. et al. Establishment of HIV-1 resistance in CD4(+) T cells by genome editing using zinc-finger nucleases. Nat Biotechnol 26, 808-816 (2008).
Qi, L.S. et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell 152, 1173-1183 (2013).
REMINGTON'S PHARMACEUTICAL SCIENCES (Mack Pub. Co., N.J. 1991)
Reyon, D. et al. FLASH assembly of TALENs for high-throughput genome editing. Nat Biotechnol 30, 460-465 (2012).
Saleh-Gohari, N. & Helleday, T. Conservative homologous recombination preferentially repairs DNA double-strand breaks in the S phase of the cell cycle in human cells. Nucleic Acids Res 32, 3683-3688 (2004).
Sander, J.D. et al. Selection-free zinc-finger-nuclease engineering by context-dependent assembly (CoDA). Nat Methods 8, 67-69 (2011).
Sanjana, N.E. et al. A transcription activator-like effector toolbox for genome engineering. Nat Profoc 7, 171-192 (2012).
Sapranauskas, R. et al. The Streptococcus thermophilus CRISPR-Cas system provides immunity in Escherichia coli. Nucleic Acids Res 39, 9275-9282 (2011).
Shen, B. et al. Generation of gene-modified mice via Cas9/RNA-mediated gene targeting. Cell Res 23, 720-723 (2013).
Smithies, O., Gregg, R.G., Boggs, S.S., Koralewski, M.A. & Kucherlapati, R.S. Insertion of DNA sequences into the human chromosomal beta-globin locus by homologous recombination. Nature 317, 230-234 (1985).
Soldner, F. et al. Generation of isogenic pluripotent stem cells differing exclusively at two early onset Parkinson point mutations. Cell 146, 318-331 (2011).
Takasu, Y. et al. Targeted mutagenesis in the silkworm Bombyx mori using zinc finger nuclease mRNA injection. Insect Biochem Molec 40, 759-765 (2010).
Tangri S, et al., Rationally engineered therapeutic proteins with reduced immunogenicity, J Immunol. 2005 Mar 15; 174(6): 3187-96.
Thomas, K.R., Folger, K.R. & Capecchi, M.R. High frequency targeting of genes to specific sites in the mammalian genome. Cell 44, 419-428 (1986).
Tuschi, T. Expanding small RNA interference. Nat Biotechnol 20, 446-448 (2002).
Umov, F.D., Rebar, E.J., Holmes, M.C., Zhang, H.S. & Gregory, P.D. Genome editing with engineered zinc finger nucleases. Nat Rev Genet 11, 636-646 (2010).
Valton, J. et al. Overcoming transcription activator-like effector (TALE) DNA binding domain sensitivity to cytosine methylation. J Biol Chem 287, 38427-38432 (2012).
Wang, H. et al. One-Step Generation of Mice Carrying Mutations in Multiple Genes by CRISPR-Cas-Mediated Genome Engineering. Cell 153, 910-918 (2013).
Watanabe, T. et al. Non-transgenic genome modifications in a hemimetabolous insect using zinc-finger and TAL effector nucleases. Nat Commun 3 (2012).
Wilson, E.B. Probable inference, the law of succession, and statistical inference. J Am Stat Assoc 22, 209-212 (1927).
Wood, A.J. et al. Targeted genome editing across species using ZFNs and TALENs. Science 333, 307 (2011).
Wu, S., Ying, G.X., Wu, Q. & Capecchi, M.R. A protocol for constructing gene targeting vectors: generating knockout mice for the cadherin family and beyond. Nat Protoc 3, 1056-1076 (2008).
Zhang, F. et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nat Biotechnol 29, 149-153 (2011).
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ZHANG, FENG
RAN, FEI
<120> DELIVERY, ENGINEERING AND OPTIMIZATION OF SYSTEMS, METHODS AND
COMPOSITIONS FOR SEQUENCE MANIPULATION AND THERAPEUTIC
APPLICATIONS
<130> 44790.00.2041
<140> 14/104,837
<141> 2013-12-12
<150> 61/847,537
<151> 2013-07-17
<150> 61/836,123
<151> 2013-06-17
<150> 61/835,931
<151> 2013-06-17
<150> 61/828,130
<151> 2013-05-28
<150> 61/819,803
<151> 2013-05-06
<150> 61/814,263
<151> 2013-04-20
<150> 61/806,375
<151> 2013-03-28
<150> 61/802,174
<151> 2013-03-15
<150> 61/791,409
<151> 2013-03-15
<150> 61/769,046
<151> 2013-02-25
<150> 61/758,468
<151> 2013-01-30
<150> 61/748,427
<151> 2013-01-02
<150> 61/736,527
<151> 2012-12-12
<160> 443
<170> PatentIn version 3.5
<210> 1
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 1
gcactgaggg cctatttccc atgattc 27
<210> 2
<211> 20
<212> DNA
<213> Homo sapiens
<400> 2
tagggttagg ggccccaggc 20
<210> 3
<211> 20
<212> DNA
<213> Homo sapiens
<400> 3
gagtccgagc agaagaagaa 20
<210> 4
<211> 20
<212> DNA
<213> Homo sapiens
<400> 4
gagtcctagc aggagaagaa 20
<210> 5
<211> 20
<212> DNA
<213> Homo sapiens
<400> 5
gagtctaagc agaagaagaa 20
<210> 6
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(44)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (47)..(60)
<223> a, c, u, g, unknown or other
<400> 6
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnnnnnnnnn nnnnggnnnn nnnnnnnnnn 60
<210> 7
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(14)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (17)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 7
nnnnnnnnnn nnnnccnnnn nnnnnnnnnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 8
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(43)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (46)..(60)
<223> a, c, u, g, unknown or other
<400> 8
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnnnnnnnnn nnnggnnnnn nnnnnnnnnn 60
<210> 9
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(15)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (18)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 9
nnnnnnnnnn nnnnnccnnn nnnnnnnnnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 10
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(42)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (45)..(60)
<223> a, c, u, g, unknown or other
<400> 10
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnnnnnnnnn nnggnnnnnn nnnnnnnnnn 60
<210> 11
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(15)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (18)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 11
nnnnnnnnnn nnnnnccnnn nnnnnnnnnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 12
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(41)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (44)..(60)
<223> a, c, u, g, unknown or other
<400> 12
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnnnnnnnnn nggnnnnnnn nnnnnnnnnn 60
<210> 13
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(17)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (20)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 13
nnnnnnnnnn nnnnnnnccn nnnnnnnnnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 14
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(40)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (43)..(60)
<223> a, c, u, g, unknown or other
<400> 14
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnnnnnnnnn ggnnnnnnnn nnnnnnnnnn 60
<210> 15
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(18)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (21)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 15
nnnnnnnnnn nnnnnnnncc nnnnnnnnnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 16
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(39)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (42)..(60)
<223> a, c, u, g, unknown or other
<400> 16
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnnnnnnnng gnnnnnnnnn nnnnnnnnnn 60
<210> 17
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(19)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (22)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 17
nnnnnnnnnn nnnnnnnnnc cnnnnnnnnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 18
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 18
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 19
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 19
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 20
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(37)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (40)..(60)
<223> a, c, u, g, unknown or other
<400> 20
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnnnnnnggn nnnnnnnnnn nnnnnnnnnn 60
<210> 21
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(21)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (24)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 21
nnnnnnnnnn nnnnnnnnnn nccnnnnnnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 22
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(36)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (39)..(60)
<223> a, c, u, g, unknown or other
<400> 22
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnnnnnggnn nnnnnnnnnn nnnnnnnnnn 60
<210> 23
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(22)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (25)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 23
nnnnnnnnnn nnnnnnnnnn nnccnnnnnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 24
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(35)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (38)..(60)
<223> a, c, u, g, unknown or other
<400> 24
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnnnnggnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 25
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(23)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (26)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 25
nnnnnnnnnn nnnnnnnnnn nnnccnnnnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 26
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(34)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (37)..(60)
<223> a, c, u, g, unknown or other
<400> 26
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnnnggnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 27
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(24)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (27)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 27
nnnnnnnnnn nnnnnnnnnn nnnnccnnnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 28
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(33)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (36)..(60)
<223> a, c, u, g, unknown or other
<400> 28
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnnggnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 29
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(25)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (28)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 29
nnnnnnnnnn nnnnnnnnnn nnnnnccnnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 30
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(32)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (35)..(60)
<223> a, c, u, g, unknown or other
<400> 30
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nnggnnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 31
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(26)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (29)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 31
nnnnnnnnnn nnnnnnnnnn nnnnnnccnn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 32
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(31)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (34)..(60)
<223> a, c, u, g, unknown or other
<400> 32
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn nggnnnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 33
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(27)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (30)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 33
nnnnnnnnnn nnnnnnnnnn nnnnnnnccn nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 34
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(30)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (33)..(60)
<223> a, c, u, g, unknown or other
<400> 34
nnnnnnnnnn nnnnnnnnnn ccnnnnnnnn ggnnnnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 35
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(28)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (31)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 35
nnnnnnnnnn nnnnnnnnnn nnnnnnnncc nnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 36
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(29)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (32)..(60)
<223> a, c, u, g, unknown or other
<400> 36
nnnnnnnnnn nnnnnnnnnn ccnnnnnnng gnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 37
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(29)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (32)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 37
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnc cnnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 38
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(28)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (31)..(60)
<223> a, c, u, g, unknown or other
<400> 38
nnnnnnnnnn nnnnnnnnnn ccnnnnnngg nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 39
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(30)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (33)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 39
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn ccnnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 40
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(27)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (30)..(60)
<223> a, c, u, g, unknown or other
<400> 40
nnnnnnnnnn nnnnnnnnnn ccnnnnnggn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 41
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(31)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (34)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 41
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nccnnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 42
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(26)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (29)..(60)
<223> a, c, u, g, unknown or other
<400> 42
nnnnnnnnnn nnnnnnnnnn ccnnnnggnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 43
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(32)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (35)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 43
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnccnnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 44
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(25)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (28)..(60)
<223> a, c, u, g, unknown or other
<400> 44
nnnnnnnnnn nnnnnnnnnn ccnnnggnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 45
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(33)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (36)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 45
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnccnnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 46
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(24)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (27)..(60)
<223> a, c, u, g, unknown or other
<400> 46
nnnnnnnnnn nnnnnnnnnn ccnnggnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 47
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(34)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (37)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 47
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnccnngg nnnnnnnnnn nnnnnnnnnn 60
<210> 48
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (23)..(23)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (26)..(60)
<223> a, c, u, g, unknown or other
<400> 48
nnnnnnnnnn nnnnnnnnnn ccnggnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 49
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(35)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (38)..(38)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (41)..(60)
<223> a, c, u, g, unknown or other
<400> 49
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnccngg nnnnnnnnnn nnnnnnnnnn 60
<210> 50
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(21)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (26)..(60)
<223> a, c, u, g, unknown or other
<400> 50
nnnnnnnnnn nnnnnnnnnn nccggnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 51
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(35)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (40)..(60)
<223> a, c, u, g, unknown or other
<400> 51
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnccggn nnnnnnnnnn nnnnnnnnnn 60
<210> 52
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(23)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (26)..(60)
<223> a, c, u, g, unknown or other
<400> 52
nnnnnnnnnn nnnnnnnnnn nnnggnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 53
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(33)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (38)..(60)
<223> a, c, u, g, unknown or other
<400> 53
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnggccnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 54
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(22)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (26)..(60)
<223> a, c, u, g, unknown or other
<400> 54
nnnnnnnnnn nnnnnnnnnn nncggnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 55
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(32)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (35)..(35)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (38)..(60)
<223> a, c, u, g, unknown or other
<400> 55
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnggnccnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 56
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(31)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (34)..(35)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (38)..(60)
<223> a, c, u, g, unknown or other
<400> 56
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nggnnccnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 57
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(30)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (33)..(35)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (38)..(60)
<223> a, c, u, g, unknown or other
<400> 57
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn ggnnnccnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 58
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(29)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (32)..(35)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (38)..(60)
<223> a, c, u, g, unknown or other
<400> 58
nnnnnnnnnn nnnnnnnnnn nnnnnnnnng gnnnnccnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 59
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(28)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (31)..(35)
<223> a, c, u, g, unknown or other
<220>
<221> modified_base
<222> (38)..(60)
<223> a, c, u, g, unknown or other
<400> 59
nnnnnnnnnn nnnnnnnnnn nnnnnnnngg nnnnnccnnn nnnnnnnnnn nnnnnnnnnn 60
<210> 60
<211> 9
<212> PRT
<213> Unknown
<220>
<223> Description of Unknown:
'LAGLIDADG' family motif peptide
<400> 60
Leu Ala Gly Leu Ile Asp Ala Asp Gly
1 5
<210> 61
<211> 12
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 61
guuuuagagc ua 12
<210> 62
<211> 7
<212> PRT
<213> Simian virus 40
<400> 62
Pro Lys Lys Lys Arg Lys Val
1 5
<210> 63
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Description of Unknown:
Nucleoplasmin bipartite NLS sequence
<400> 63
Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala Lys Lys Lys Lys
1 5 10 15
<210> 64
<211> 9
<212> PRT
<213> Unknown
<220>
<223> Description of Unknown:
C-myc NLS sequence
<400> 64
Pro Ala Ala Lys Arg Val Lys Leu Asp
1 5
<210> 65
<211> 11
<212> PRT
<213> Unknown
<220>
<223> Description of Unknown:
C-myc NLS sequence
<400> 65
Arg Gln Arg Arg Asn Glu Leu Lys Arg Ser Pro
1 5 10
<210> 66
<211> 38
<212> PRT
<213> Homo sapiens
<400> 66
Asn Gln Ser Ser Asn Phe Gly Pro Met Lys Gly Gly Asn Phe Gly Gly
1 5 10 15
Arg Ser Ser Gly Pro Tyr Gly Gly Gly Gly Gln Tyr Phe Ala Lys Pro
20 25 30
Arg Asn Gln Gly Gly Tyr
35
<210> 67
<211> 42
<212> PRT
<213> Unknown
<220>
<223> Description of Unknown:
IBB domain from importin-alpha sequence
<400> 67
Arg Met Arg Ile Glx Phe Lys Asn Lys Gly Lys Asp Thr Ala Glu Leu
1 5 10 15
Arg Arg Arg Arg Val Glu Val Ser Val Glu Leu Arg Lys Ala Lys Lys
20 25 30
Asp Glu Gln Ile Leu Lys Arg Arg Asn Val
35 40
<210> 68
<211> 8
<212> PRT
<213> Unknown
<220>
<223> Description of Unknown:
Myoma T protein sequence
<400> 68
Val Ser Arg Lys Arg Pro Arg Pro
1 5
<210> 69
<211> 8
<212> PRT
<213> Unknown
<220>
<223> Description of Unknown:
Myoma T protein sequence
<400> 69
Pro Pro Lys Lys Ala Arg Glu Asp
1 5
<210> 70
<211> 8
<212> PRT
<213> Homo sapiens
<400> 70
Pro Gln Pro Lys Lys Lys Pro Leu
1 5
<210> 71
<211> 12
<212> PRT
<213> Mus musculus
<400> 71
Ser Ala Leu Ile Lys Lys Lys Lys Lys Met Ala Pro
1 5 10
<210> 72
<211> 5
<212> PRT
<213> Influenza virus
<400> 72
Asp Arg Leu Arg Arg
1 5
<210> 73
<211> 7
<212> PRT
<213> Influenza virus
<400> 73
Pro Lys Gln Lys Lys Arg Lys
1 5
<210> 74
<211> 10
<212> PRT
<213> Hepatitus delta virus
<400> 74
Arg Lys Leu Lys Lys Lys Ile Lys Lys Leu
1 5 10
<210> 75
<211> 10
<212> PRT
<213> Mus musculus
<400> 75
Arg Glu Lys Lys Lys Phe Leu Lys Arg Arg
1 5 10
<210> 76
<211> 20
<212> PRT
<213> Homo sapiens
<400> 76
Lys Arg Lys Gly Asp Glu Val Asp Gly Val Asp Glu Val Ala Lys Lys
1 5 10 15
Lys Ser Lys Lys
20
<210> 77
<211> 17
<212> PRT
<213> Homo sapiens
<400> 77
Arg Lys Cys Leu Gln Ala Gly Met Asn Leu Glu Ala Arg Lys Thr Lys
1 5 10 15
Lys
<210> 78
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t or g
<220>
<221> modified_base
<222> (21)..(22)
<223> a, c, t, g, unknown or other
<400> 78
nnnnnnnnnn nnnnnnnnnn nnagaaw 27
<210> 79
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(12)
<223> a, c, t or g
<220>
<221> modified_base
<222> (13)..(14)
<223> a, c, t, g, unknown or other
<400> 79
nnnnnnnnnn nnnnagaaw 19
<210> 80
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t or g
<220>
<221> modified_base
<222> (21)..(22)
<223> a, c, t, g, unknown or other
<400> 80
nnnnnnnnnn nnnnnnnnnn nnagaaw 27
<210> 81
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(11)
<223> a, c, t or g
<220>
<221> modified_base
<222> (12)..(13)
<223> a, c, t, g, unknown or other
<400> 81
nnnnnnnnnn nnnagaaw 18
<210> 82
<211> 137
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polynucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 82
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcaagatt tagaaataaa tcttgcagaa 60
gctacaaaga taaggcttca tgccgaaatc aacaccctgt cattttatgg cagggtgttt 120
tcgttattta atttttt 137
<210> 83
<211> 123
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polynucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 83
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcagaaat gcagaagcta caaagataag 60
gcttcatgcc gaaatcaaca ccctgtcatt ttatggcagg gtgttttcgt tatttaattt 120
ttt 123
<210> 84
<211> 110
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polynucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 84
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcagaaat gcagaagcta caaagataag 60
gcttcatgcc gaaatcaaca ccctgtcatt ttatggcagg gtgttttttt 110
<210> 85
<211> 102
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polynucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 85
nnnnnnnnnn nnnnnnnnnn gttttagagc tagaaatagc aagttaaaat aaggctagtc 60
cgttatcaac ttgaaaaagt ggcaccgagt cggtgctttt tt 102
<210> 86
<211> 88
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 86
nnnnnnnnnn nnnnnnnnnn gttttagagc tagaaatagc aagttaaaat aaggctagtc 60
cgttatcaac ttgaaaaagt gttttttt 88
<210> 87
<211> 76
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 87
nnnnnnnnnn nnnnnnnnnn gttttagagc tagaaatagc aagttaaaat aaggctagtc 60
cgttatcatt tttttt 76
<210> 88
<211> 12
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 88
gttttagagc ta 12
<210> 89
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 89
tagcaagtta aaataaggct agtccgtttt t 31
<210> 90
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(22)
<223> a, c, t, g, unknown or other
<400> 90
nnnnnnnnnn nnnnnnnnnn nnagaaw 27
<210> 91
<211> 33
<212> DNA
<213> Homo sapiens
<400> 91
ggacatcgat gtcacctcca atgactaggg tgg 33
<210> 92
<211> 33
<212> DNA
<213> Homo sapiens
<400> 92
cattggaggt gacatcgatg tcctccccat tgg 33
<210> 93
<211> 33
<212> DNA
<213> Homo sapiens
<400> 93
ggaagggcct gagtccgagc agaagaagaa ggg 33
<210> 94
<211> 33
<212> DNA
<213> Homo sapiens
<400> 94
ggtggcgaga ggggccgaga ttgggtgttc agg 33
<210> 95
<211> 33
<212> DNA
<213> Homo sapiens
<400> 95
atgcaggagg gtggcgagag gggccgagat tgg 33
<210> 96
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 96
aaactctaga gagggcctat ttcccatgat tc 32
<210> 97
<211> 153
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 97
acctctagaa aaaaagcacc gactcggtgc cactttttca agttgataac ggactagcct 60
tattttaact tgctatgctg ttttgtttcc aaaacagcat agctctaaaa cccctagtca 120
ttggaggtga cggtgtttcg tcctttccac aag 153
<210> 98
<211> 52
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 98
taatacgact cactatagga agtgcgccac catggcccca aagaagaagc gg 52
<210> 99
<211> 60
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 99
ggtttttttt tttttttttt tttttttttt ttttcttact ttttcttttt tgcctggccg 60
<210> 100
<211> 984
<212> PRT
<213> Campylobacter jejuni
<400> 100
Met Ala Arg Ile Leu Ala Phe Asp Ile Gly Ile Ser Ser Ile Gly Trp
1 5 10 15
Ala Phe Ser Glu Asn Asp Glu Leu Lys Asp Cys Gly Val Arg Ile Phe
20 25 30
Thr Lys Val Glu Asn Pro Lys Thr Gly Glu Ser Leu Ala Leu Pro Arg
35 40 45
Arg Leu Ala Arg Ser Ala Arg Lys Arg Leu Ala Arg Arg Lys Ala Arg
50 55 60
Leu Asn His Leu Lys His Leu Ile Ala Asn Glu Phe Lys Leu Asn Tyr
65 70 75 80
Glu Asp Tyr Gln Ser Phe Asp Glu Ser Leu Ala Lys Ala Tyr Lys Gly
85 90 95
Ser Leu Ile Ser Pro Tyr Glu Leu Arg Phe Arg Ala Leu Asn Glu Leu
100 105 110
Leu Ser Lys Gln Asp Phe Ala Arg Val Ile Leu His Ile Ala Lys Arg
115 120 125
Arg Gly Tyr Asp Asp Ile Lys Asn Ser Asp Asp Lys Glu Lys Gly Ala
130 135 140
Ile Leu Lys Ala Ile Lys Gln Asn Glu Glu Lys Leu Ala Asn Tyr Gln
145 150 155 160
Ser Val Gly Glu Tyr Leu Tyr Lys Glu Tyr Phe Gln Lys Phe Lys Glu
165 170 175
Asn Ser Lys Glu Phe Thr Asn Val Arg Asn Lys Lys Glu Ser Tyr Glu
180 185 190
Arg Cys Ile Ala Gln Ser Phe Leu Lys Asp Glu Leu Lys Leu Ile Phe
195 200 205
Lys Lys Gln Arg Glu Phe Gly Phe Ser Phe Ser Lys Lys Phe Glu Glu
210 215 220
Glu Val Leu Ser Val Ala Phe Tyr Lys Arg Ala Leu Lys Asp Phe Ser
225 230 235 240
His Leu Val Gly Asn Cys Ser Phe Phe Thr Asp Glu Lys Arg Ala Pro
245 250 255
Lys Asn Ser Pro Leu Ala Phe Met Phe Val Ala Leu Thr Arg Ile Ile
260 265 270
Asn Leu Leu Asn Asn Leu Lys Asn Thr Glu Gly Ile Leu Tyr Thr Lys
275 280 285
Asp Asp Leu Asn Ala Leu Leu Asn Glu Val Leu Lys Asn Gly Thr Leu
290 295 300
Thr Tyr Lys Gln Thr Lys Lys Leu Leu Gly Leu Ser Asp Asp Tyr Glu
305 310 315 320
Phe Lys Gly Glu Lys Gly Thr Tyr Phe Ile Glu Phe Lys Lys Tyr Lys
325 330 335
Glu Phe Ile Lys Ala Leu Gly Glu His Asn Leu Ser Gln Asp Asp Leu
340 345 350
Asn Glu Ile Ala Lys Asp Ile Thr Leu Ile Lys Asp Glu Ile Lys Leu
355 360 365
Lys Lys Ala Leu Ala Lys Tyr Asp Leu Asn Gln Asn Gln Ile Asp Ser
370 375 380
Leu Ser Lys Leu Glu Phe Lys Asp His Leu Asn Ile Ser Phe Lys Ala
385 390 395 400
Leu Lys Leu Val Thr Pro Leu Met Leu Glu Gly Lys Lys Tyr Asp Glu
405 410 415
Ala Cys Asn Glu Leu Asn Leu Lys Val Ala Ile Asn Glu Asp Lys Lys
420 425 430
Asp Phe Leu Pro Ala Phe Asn Glu Thr Tyr Tyr Lys Asp Glu Val Thr
435 440 445
Asn Pro Val Val Leu Arg Ala Ile Lys Glu Tyr Arg Lys Val Leu Asn
450 455 460
Ala Leu Leu Lys Lys Tyr Gly Lys Val His Lys Ile Asn Ile Glu Leu
465 470 475 480
Ala Arg Glu Val Gly Lys Asn His Ser Gln Arg Ala Lys Ile Glu Lys
485 490 495
Glu Gln Asn Glu Asn Tyr Lys Ala Lys Lys Asp Ala Glu Leu Glu Cys
500 505 510
Glu Lys Leu Gly Leu Lys Ile Asn Ser Lys Asn Ile Leu Lys Leu Arg
515 520 525
Leu Phe Lys Glu Gln Lys Glu Phe Cys Ala Tyr Ser Gly Glu Lys Ile
530 535 540
Lys Ile Ser Asp Leu Gln Asp Glu Lys Met Leu Glu Ile Asp His Ile
545 550 555 560
Tyr Pro Tyr Ser Arg Ser Phe Asp Asp Ser Tyr Met Asn Lys Val Leu
565 570 575
Val Phe Thr Lys Gln Asn Gln Glu Lys Leu Asn Gln Thr Pro Phe Glu
580 585 590
Ala Phe Gly Asn Asp Ser Ala Lys Trp Gln Lys Ile Glu Val Leu Ala
595 600 605
Lys Asn Leu Pro Thr Lys Lys Gln Lys Arg Ile Leu Asp Lys Asn Tyr
610 615 620
Lys Asp Lys Glu Gln Lys Asn Phe Lys Asp Arg Asn Leu Asn Asp Thr
625 630 635 640
Arg Tyr Ile Ala Arg Leu Val Leu Asn Tyr Thr Lys Asp Tyr Leu Asp
645 650 655
Phe Leu Pro Leu Ser Asp Asp Glu Asn Thr Lys Leu Asn Asp Thr Gln
660 665 670
Lys Gly Ser Lys Val His Val Glu Ala Lys Ser Gly Met Leu Thr Ser
675 680 685
Ala Leu Arg His Thr Trp Gly Phe Ser Ala Lys Asp Arg Asn Asn His
690 695 700
Leu His His Ala Ile Asp Ala Val Ile Ile Ala Tyr Ala Asn Asn Ser
705 710 715 720
Ile Val Lys Ala Phe Ser Asp Phe Lys Lys Glu Gln Glu Ser Asn Ser
725 730 735
Ala Glu Leu Tyr Ala Lys Lys Ile Ser Glu Leu Asp Tyr Lys Asn Lys
740 745 750
Arg Lys Phe Phe Glu Pro Phe Ser Gly Phe Arg Gln Lys Val Leu Asp
755 760 765
Lys Ile Asp Glu Ile Phe Val Ser Lys Pro Glu Arg Lys Lys Pro Ser
770 775 780
Gly Ala Leu His Glu Glu Thr Phe Arg Lys Glu Glu Glu Phe Tyr Gln
785 790 795 800
Ser Tyr Gly Gly Lys Glu Gly Val Leu Lys Ala Leu Glu Leu Gly Lys
805 810 815
Ile Arg Lys Val Asn Gly Lys Ile Val Lys Asn Gly Asp Met Phe Arg
820 825 830
Val Asp Ile Phe Lys His Lys Lys Thr Asn Lys Phe Tyr Ala Val Pro
835 840 845
Ile Tyr Thr Met Asp Phe Ala Leu Lys Val Leu Pro Asn Lys Ala Val
850 855 860
Ala Arg Ser Lys Lys Gly Glu Ile Lys Asp Trp Ile Leu Met Asp Glu
865 870 875 880
Asn Tyr Glu Phe Cys Phe Ser Leu Tyr Lys Asp Ser Leu Ile Leu Ile
885 890 895
Gln Thr Lys Asp Met Gln Glu Pro Glu Phe Val Tyr Tyr Asn Ala Phe
900 905 910
Thr Ser Ser Thr Val Ser Leu Ile Val Ser Lys His Asp Asn Lys Phe
915 920 925
Glu Thr Leu Ser Lys Asn Gln Lys Ile Leu Phe Lys Asn Ala Asn Glu
930 935 940
Lys Glu Val Ile Ala Lys Ser Ile Gly Ile Gln Asn Leu Lys Val Phe
945 950 955 960
Glu Lys Tyr Ile Val Ser Ala Leu Gly Glu Val Thr Lys Ala Glu Phe
965 970 975
Arg Gln Arg Glu Asp Phe Lys Lys
980
<210> 101
<211> 91
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 101
tataatctca taagaaattt aaaaagggac taaaataaag agtttgcggg actctgcggg 60
gttacaatcc cctaaaaccg cttttaaaat t 91
<210> 102
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 102
attttaccat aaagaaattt aaaaagggac taaaac 36
<210> 103
<211> 95
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<400> 103
nnnnnnnnnn nnnnnnnnnn guuuuagucc cgaaagggac uaaaauaaag aguuugcggg 60
acucugcggg guuacaaucc ccuaaaaccg cuuuu 95
<210> 104
<211> 1115
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 104
Met Ser Asp Leu Val Leu Gly Leu Asp Ile Gly Ile Gly Ser Val Gly
1 5 10 15
Val Gly Ile Leu Asn Lys Val Thr Gly Glu Ile Ile His Lys Asn Ser
20 25 30
Arg Ile Phe Pro Ala Ala Gln Ala Glu Asn Asn Leu Val Arg Arg Thr
35 40 45
Asn Arg Gln Gly Arg Arg Leu Ala Arg Arg Lys Lys His Arg Arg Val
50 55 60
Arg Leu Asn Arg Leu Phe Glu Glu Ser Gly Leu Ile Thr Asp Phe Thr
65 70 75 80
Lys Ile Ser Ile Asn Leu Asn Pro Tyr Gln Leu Arg Val Lys Gly Leu
85 90 95
Thr Asp Glu Leu Ser Asn Glu Glu Leu Phe Ile Ala Leu Lys Asn Met
100 105 110
Val Lys His Arg Gly Ile Ser Tyr Leu Asp Asp Ala Ser Asp Asp Gly
115 120 125
Asn Ser Ser Val Gly Asp Tyr Ala Gln Ile Val Lys Glu Asn Ser Lys
130 135 140
Gln Leu Glu Thr Lys Thr Pro Gly Gln Ile Gln Leu Glu Arg Tyr Gln
145 150 155 160
Thr Tyr Gly Gln Leu Arg Gly Asp Phe Thr Val Glu Lys Asp Gly Lys
165 170 175
Lys His Arg Leu Ile Asn Val Phe Pro Thr Ser Ala Tyr Arg Ser Glu
180 185 190
Ala Leu Arg Ile Leu Gln Thr Gln Gln Glu Phe Asn Pro Gln Ile Thr
195 200 205
Asp Glu Phe Ile Asn Arg Tyr Leu Glu Ile Leu Thr Gly Lys Arg Lys
210 215 220
Tyr Tyr His Gly Pro Gly Asn Glu Lys Ser Arg Thr Asp Tyr Gly Arg
225 230 235 240
Tyr Arg Thr Ser Gly Glu Thr Leu Asp Asn Ile Phe Gly Ile Leu Ile
245 250 255
Gly Lys Cys Thr Phe Tyr Pro Asp Glu Phe Arg Ala Ala Lys Ala Ser
260 265 270
Tyr Thr Ala Gln Glu Phe Asn Leu Leu Asn Asp Leu Asn Asn Leu Thr
275 280 285
Val Pro Thr Glu Thr Lys Lys Leu Ser Lys Glu Gln Lys Asn Gln Ile
290 295 300
Ile Asn Tyr Val Lys Asn Glu Lys Ala Met Gly Pro Ala Lys Leu Phe
305 310 315 320
Lys Tyr Ile Ala Lys Leu Leu Ser Cys Asp Val Ala Asp Ile Lys Gly
325 330 335
Tyr Arg Ile Asp Lys Ser Gly Lys Ala Glu Ile His Thr Phe Glu Ala
340 345 350
Tyr Arg Lys Met Lys Thr Leu Glu Thr Leu Asp Ile Glu Gln Met Asp
355 360 365
Arg Glu Thr Leu Asp Lys Leu Ala Tyr Val Leu Thr Leu Asn Thr Glu
370 375 380
Arg Glu Gly Ile Gln Glu Ala Leu Glu His Glu Phe Ala Asp Gly Ser
385 390 395 400
Phe Ser Gln Lys Gln Val Asp Glu Leu Val Gln Phe Arg Lys Ala Asn
405 410 415
Ser Ser Ile Phe Gly Lys Gly Trp His Asn Phe Ser Val Lys Leu Met
420 425 430
Met Glu Leu Ile Pro Glu Leu Tyr Glu Thr Ser Glu Glu Gln Met Thr
435 440 445
Ile Leu Thr Arg Leu Gly Lys Gln Lys Thr Thr Ser Ser Ser Asn Lys
450 455 460
Thr Lys Tyr Ile Asp Glu Lys Leu Leu Thr Glu Glu Ile Tyr Asn Pro
465 470 475 480
Val Val Ala Lys Ser Val Arg Gln Ala Ile Lys Ile Val Asn Ala Ala
485 490 495
Ile Lys Glu Tyr Gly Asp Phe Asp Asn Ile Val Ile Glu Met Ala Arg
500 505 510
Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met
515 520 525
Lys Arg Ile Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys
530 535 540
Glu His Pro Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu
545 550 555 560
Tyr Tyr Leu Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp
565 570 575
Ile Asn Arg Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser
580 585 590
Phe Leu Lys Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp
595 600 605
Lys Asn Arg Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys
610 615 620
Lys Met Lys Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr
625 630 635 640
Gln Arg Lys Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser
645 650 655
Glu Leu Asp Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg
660 665 670
Gln Ile Thr Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr
675 680 685
Lys Tyr Asp Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr
690 695 700
Leu Lys Ser Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr
705 710 715 720
Lys Val Arg Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu
725 730 735
Asn Ala Val Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu
740 745 750
Ser Glu Phe Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met
755 760 765
Ile Ala Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe
770 775 780
Phe Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala
785 790 795 800
Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr
805 810 815
Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys
820 825 830
Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln
835 840 845
Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser Asp
850 855 860
Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr Gly Gly
865 870 875 880
Phe Asp Ser Pro Thr Val Ala Tyr Ser Val Leu Val Val Ala Lys Val
885 890 895
Glu Lys Gly Lys Ser Lys Lys Leu Lys Ser Val Lys Glu Leu Leu Gly
900 905 910
Ile Thr Ile Met Glu Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe
915 920 925
Leu Glu Ala Lys Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys
930 935 940
Leu Pro Lys Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met
945 950 955 960
Leu Ala Ser Ala Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro
965 970 975
Ser Lys Tyr Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu
980 985 990
Lys Gly Ser Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln
995 1000 1005
His Lys His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe
1010 1015 1020
Ser Lys Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu
1025 1030 1035
Ser Ala Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala
1040 1045 1050
Glu Asn Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro
1055 1060 1065
Ala Ala Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr
1070 1075 1080
Thr Ser Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser
1085 1090 1095
Ile Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly
1100 1105 1110
Gly Asp
1115
<210> 105
<211> 1374
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 105
Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Thr Asn
755 760 765
Glu Asp Asp Glu Lys Lys Ala Ile Gln Lys Ile Gln Lys Ala Asn Lys
770 775 780
Asp Glu Lys Asp Ala Ala Met Leu Lys Ala Ala Asn Gln Tyr Asn Gly
785 790 795 800
Lys Ala Glu Leu Pro His Ser Val Phe His Gly His Lys Gln Leu Ala
805 810 815
Thr Lys Ile Arg Leu Trp His Gln Gln Gly Glu Arg Cys Leu Tyr Thr
820 825 830
Gly Lys Thr Ile Ser Ile His Asp Leu Ile Asn Asn Ser Asn Gln Phe
835 840 845
Glu Val Asp His Ile Leu Pro Leu Ser Ile Thr Phe Asp Asp Ser Leu
850 855 860
Ala Asn Lys Val Leu Val Tyr Ala Thr Ala Asn Gln Glu Lys Gly Gln
865 870 875 880
Arg Thr Pro Tyr Gln Ala Leu Asp Ser Met Asp Asp Ala Trp Ser Phe
885 890 895
Arg Glu Leu Lys Ala Phe Val Arg Glu Ser Lys Thr Leu Ser Asn Lys
900 905 910
Lys Lys Glu Tyr Leu Leu Thr Glu Glu Asp Ile Ser Lys Phe Asp Val
915 920 925
Arg Lys Lys Phe Ile Glu Arg Asn Leu Val Asp Thr Arg Tyr Ala Ser
930 935 940
Arg Val Val Leu Asn Ala Leu Gln Glu His Phe Arg Ala His Lys Ile
945 950 955 960
Asp Thr Lys Val Ser Val Val Arg Gly Gln Phe Thr Ser Gln Leu Arg
965 970 975
Arg His Trp Gly Ile Glu Lys Thr Arg Asp Thr Tyr His His His Ala
980 985 990
Val Asp Ala Leu Ile Ile Ala Ala Ser Ser Gln Leu Asn Leu Trp Lys
995 1000 1005
Lys Gln Lys Asn Thr Leu Val Ser Tyr Ser Glu Asp Gln Leu Leu
1010 1015 1020
Asp Ile Glu Thr Gly Glu Leu Ile Ser Asp Asp Glu Tyr Lys Glu
1025 1030 1035
Ser Val Phe Lys Ala Pro Tyr Gln His Phe Val Asp Thr Leu Lys
1040 1045 1050
Ser Lys Glu Phe Glu Asp Ser Ile Leu Phe Ser Tyr Gln Val Asp
1055 1060 1065
Ser Lys Phe Asn Arg Lys Ile Ser Asp Ala Thr Ile Tyr Ala Thr
1070 1075 1080
Arg Gln Ala Lys Val Gly Lys Asp Lys Ala Asp Glu Thr Tyr Val
1085 1090 1095
Leu Gly Lys Ile Lys Asp Ile Tyr Thr Gln Asp Gly Tyr Asp Ala
1100 1105 1110
Phe Met Lys Ile Tyr Lys Lys Asp Lys Ser Lys Phe Leu Met Tyr
1115 1120 1125
Arg His Asp Pro Gln Thr Phe Glu Lys Val Ile Glu Pro Ile Leu
1130 1135 1140
Glu Asn Tyr Pro Asn Lys Gln Ile Asn Glu Lys Gly Lys Glu Val
1145 1150 1155
Pro Cys Asn Pro Phe Leu Lys Tyr Lys Glu Glu His Gly Tyr Ile
1160 1165 1170
Arg Lys Tyr Ser Lys Lys Gly Asn Gly Pro Glu Ile Lys Ser Leu
1175 1180 1185
Lys Tyr Tyr Asp Ser Lys Leu Gly Asn His Ile Asp Ile Thr Pro
1190 1195 1200
Lys Asp Ser Asn Asn Lys Val Val Leu Gln Ser Val Ser Pro Trp
1205 1210 1215
Arg Ala Asp Val Tyr Phe Asn Lys Thr Thr Gly Lys Tyr Glu Ile
1220 1225 1230
Leu Gly Leu Lys Tyr Ala Asp Leu Gln Phe Glu Lys Gly Thr Gly
1235 1240 1245
Thr Tyr Lys Ile Ser Gln Glu Lys Tyr Asn Asp Ile Lys Lys Lys
1250 1255 1260
Glu Gly Val Asp Ser Asp Ser Glu Phe Lys Phe Thr Leu Tyr Lys
1265 1270 1275
Asn Asp Leu Leu Leu Val Lys Asp Thr Glu Thr Lys Glu Gln Gln
1280 1285 1290
Leu Phe Arg Phe Leu Ser Arg Thr Met Pro Lys Gln Lys His Tyr
1295 1300 1305
Val Glu Leu Lys Pro Tyr Asp Lys Gln Lys Phe Glu Gly Gly Glu
1310 1315 1320
Ala Leu Ile Lys Val Leu Gly Asn Val Ala Asn Ser Gly Gln Cys
1325 1330 1335
Lys Lys Gly Leu Gly Lys Ser Asn Ile Ser Ile Tyr Lys Val Arg
1340 1345 1350
Thr Asp Val Leu Gly Asn Gln His Ile Ile Lys Asn Glu Gly Asp
1355 1360 1365
Lys Pro Lys Leu Asp Phe
1370
<210> 106
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 106
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 107
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 107
Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
1 5 10 15
<210> 108
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 108
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly
1 5 10 15
Gly Ser
<210> 109
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 109
gccaaattgg acgaccctcg cgg 23
<210> 110
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 110
cgaggagacc cccgtttcgg tgg 23
<210> 111
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 111
cccgccgccg ccgtggctcg agg 23
<210> 112
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 112
tgagctctac gagatccaca agg 23
<210> 113
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 113
ctcaaaattc ataccggttg tgg 23
<210> 114
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 114
cgttaaacaa caaccggact tgg 23
<210> 115
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 115
ttcaccccgc ggcgctgaat ggg 23
<210> 116
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 116
accactacca gtccgtccac agg 23
<210> 117
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 117
agcctttctg aacacatgca cgg 23
<210> 118
<211> 39
<212> DNA
<213> Homo sapiens
<400> 118
cctgccatca atgtggccat gcatgtgttc agaaaggct 39
<210> 119
<211> 39
<212> DNA
<213> Homo sapiens
<400> 119
cctgccatca atgtggccgt gcatgtgttc agaaaggct 39
<210> 120
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 120
cactgcttaa gcctcgctcg agg 23
<210> 121
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 121
tcaccagcaa tattcgctcg agg 23
<210> 122
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 122
caccagcaat attccgctcg agg 23
<210> 123
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 123
tagcaacaga catacgctcg agg 23
<210> 124
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 124
gggcagtagt aatacgctcg agg 23
<210> 125
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 125
ccaattccca tacattattg tac 23
<210> 126
<211> 4677
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polynucleotide
<400> 126
tctttcttgc gctatgacac ttccagcaaa aggtagggcg ggctgcgaga cggcttcccg 60
gcgctgcatg caacaccgat gatgcttcga ccccccgaag ctccttcggg gctgcatggg 120
cgctccgatg ccgctccagg gcgagcgctg tttaaatagc caggcccccg attgcaaaga 180
cattatagcg agctaccaaa gccatattca aacacctaga tcactaccac ttctacacag 240
gccactcgag cttgtgatcg cactccgcta agggggcgcc tcttcctctt cgtttcagtc 300
acaacccgca aacatgtacc catacgatgt tccagattac gcttcgccga agaaaaagcg 360
caaggtcgaa gcgtccgaca agaagtacag catcggcctg gacatcggca ccaactctgt 420
gggctgggcc gtgatcaccg acgagtacaa ggtgcccagc aagaaattca aggtgctggg 480
caacaccgac cggcacagca tcaagaagaa cctgatcgga gccctgctgt tcgacagcgg 540
cgaaacagcc gaggccaccc ggctgaagag aaccgccaga agaagataca ccagacggaa 600
gaaccggatc tgctatctgc aagagatctt cagcaacgag atggccaagg tggacgacag 660
cttcttccac agactggaag agtccttcct ggtggaagag gataagaagc acgagcggca 720
ccccatcttc ggcaacatcg tggacgaggt ggcctaccac gagaagtacc ccaccatcta 780
ccacctgaga aagaaactgg tggacagcac cgacaaggcc gacctgcggc tgatctatct 840
ggccctggcc cacatgatca agttccgggg ccacttcctg atcgagggcg acctgaaccc 900
cgacaacagc gacgtggaca agctgttcat ccagctggtg cagacctaca accagctgtt 960
cgaggaaaac cccatcaacg ccagcggcgt ggacgccaag gccatcctgt ctgccagact 1020
gagcaagagc agacggctgg aaaatctgat cgcccagctg cccggcgaga agaagaatgg 1080
cctgttcggc aacctgattg ccctgagcct gggcctgacc cccaacttca agagcaactt 1140
cgacctggcc gaggatgcca aactgcagct gagcaaggac acctacgacg acgacctgga 1200
caacctgctg gcccagatcg gcgaccagta cgccgacctg tttctggccg ccaagaacct 1260
gtccgacgcc atcctgctga gcgacatcct gagagtgaac accgagatca ccaaggcccc 1320
cctgagcgcc tctatgatca agagatacga cgagcaccac caggacctga ccctgctgaa 1380
agctctcgtg cggcagcagc tgcctgagaa gtacaaagag attttcttcg accagagcaa 1440
gaacggctac gccggctaca ttgacggcgg agccagccag gaagagttct acaagttcat 1500
caagcccatc ctggaaaaga tggacggcac cgaggaactg ctcgtgaagc tgaacagaga 1560
ggacctgctg cggaagcagc ggaccttcga caacggcagc atcccccacc agatccacct 1620
gggagagctg cacgccattc tgcggcggca ggaagatttt tacccattcc tgaaggacaa 1680
ccgggaaaag atcgagaaga tcctgacctt ccgcatcccc tactacgtgg gccctctggc 1740
caggggaaac agcagattcg cctggatgac cagaaagagc gaggaaacca tcaccccctg 1800
gaacttcgag gaagtggtgg acaagggcgc ttccgcccag agcttcatcg agcggatgac 1860
caacttcgat aagaacctgc ccaacgagaa ggtgctgccc aagcacagcc tgctgtacga 1920
gtacttcacc gtgtataacg agctgaccaa agtgaaatac gtgaccgagg gaatgagaaa 1980
gcccgccttc ctgagcggcg agcagaaaaa ggccatcgtg gacctgctgt tcaagaccaa 2040
ccggaaagtg accgtgaagc agctgaaaga ggactacttc aagaaaatcg agtgcttcga 2100
ctccgtggaa atctccggcg tggaagatcg gttcaacgcc tccctgggca cataccacga 2160
tctgctgaaa attatcaagg acaaggactt cctggacaat gaggaaaacg aggacattct 2220
ggaagatatc gtgctgaccc tgacactgtt tgaggacaga gagatgatcg aggaacggct 2280
gaaaacctat gcccacctgt tcgacgacaa agtgatgaag cagctgaagc ggcggagata 2340
caccggctgg ggcaggctga gccggaagct gatcaacggc atccgggaca agcagtccgg 2400
caagacaatc ctggatttcc tgaagtccga cggcttcgcc aacagaaact tcatgcagct 2460
gatccacgac gacagcctga cctttaaaga ggacatccag aaagcccagg tgtccggcca 2520
gggcgatagc ctgcacgagc acattgccaa tctggccggc agccccgcca ttaagaaggg 2580
catcctgcag acagtgaagg tggtggacga gctcgtgaaa gtgatgggcc ggcacaagcc 2640
cgagaacatc gtgatcgaaa tggccagaga gaaccagacc acccagaagg gacagaagaa 2700
cagccgcgag agaatgaagc ggatcgaaga gggcatcaaa gagctgggca gccagatcct 2760
gaaagaacac cccgtggaaa acacccagct gcagaacgag aagctgtacc tgtactacct 2820
gcagaatggg cgggatatgt acgtggacca ggaactggac atcaaccggc tgtccgacta 2880
cgatgtggac catatcgtgc ctcagagctt tctgaaggac gactccatcg acaacaaggt 2940
gctgaccaga agcgacaaga accggggcaa gagcgacaac gtgccctccg aagaggtcgt 3000
gaagaagatg aagaactact ggcggcagct gctgaacgcc aagctgatta cccagagaaa 3060
gttcgacaat ctgaccaagg ccgagagagg cggcctgagc gaactggata aggccggctt 3120
catcaagaga cagctggtgg aaacccggca gatcacaaag cacgtggcac agatcctgga 3180
ctcccggatg aacactaagt acgacgagaa tgacaagctg atccgggaag tgaaagtgat 3240
caccctgaag tccaagctgg tgtccgattt ccggaaggat ttccagtttt acaaagtgcg 3300
cgagatcaac aactaccacc acgcccacga cgcctacctg aacgccgtcg tgggaaccgc 3360
cctgatcaaa aagtacccta agctggaaag cgagttcgtg tacggcgact acaaggtgta 3420
cgacgtgcgg aagatgatcg ccaagagcga gcaggaaatc ggcaaggcta ccgccaagta 3480
cttcttctac agcaacatca tgaacttttt caagaccgag attaccctgg ccaacggcga 3540
gatccggaag cggcctctga tcgagacaaa cggcgaaacc ggggagatcg tgtgggataa 3600
gggccgggat tttgccaccg tgcggaaagt gctgagcatg ccccaagtga atatcgtgaa 3660
aaagaccgag gtgcagacag gcggcttcag caaagagtct atcctgccca agaggaacag 3720
cgataagctg atcgccagaa agaaggactg ggaccctaag aagtacggcg gcttcgacag 3780
ccccaccgtg gcctattctg tgctggtggt ggccaaagtg gaaaagggca agtccaagaa 3840
actgaagagt gtgaaagagc tgctggggat caccatcatg gaaagaagca gcttcgagaa 3900
gaatcccatc gactttctgg aagccaaggg ctacaaagaa gtgaaaaagg acctgatcat 3960
caagctgcct aagtactccc tgttcgagct ggaaaacggc cggaagagaa tgctggcctc 4020
tgccggcgaa ctgcagaagg gaaacgaact ggccctgccc tccaaatatg tgaacttcct 4080
gtacctggcc agccactatg agaagctgaa gggctccccc gaggataatg agcagaaaca 4140
gctgtttgtg gaacagcaca agcactacct ggacgagatc atcgagcaga tcagcgagtt 4200
ctccaagaga gtgatcctgg ccgacgctaa tctggacaaa gtgctgtccg cctacaacaa 4260
gcaccgggat aagcccatca gagagcaggc cgagaatatc atccacctgt ttaccctgac 4320
caatctggga gcccctgccg ccttcaagta ctttgacacc accatcgacc ggaagaggta 4380
caccagcacc aaagaggtgc tggacgccac cctgatccac cagagcatca ccggcctgta 4440
cgagacacgg atcgacctgt ctcagctggg aggcgacagc cccaagaaga agagaaaggt 4500
ggaggccagc taaggatccg gcaagactgg ccccgcttgg caacgcaaca gtgagcccct 4560
ccctagtgtg tttggggatg tgactatgta ttcgtgtgtt ggccaacggg tcaacccgaa 4620
cagattgata cccgccttgg catttcctgt cagaatgtaa cgtcagttga tggtact 4677
<210> 127
<211> 3150
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polynucleotide
<400> 127
tctttcttgc gctatgacac ttccagcaaa aggtagggcg ggctgcgaga cggcttcccg 60
gcgctgcatg caacaccgat gatgcttcga ccccccgaag ctccttcggg gctgcatggg 120
cgctccgatg ccgctccagg gcgagcgctg tttaaatagc caggcccccg attgcaaaga 180
cattatagcg agctaccaaa gccatattca aacacctaga tcactaccac ttctacacag 240
gccactcgag cttgtgatcg cactccgcta agggggcgcc tcttcctctt cgtttcagtc 300
acaacccgca aacatgccta agaagaagag gaaggttaac acgattaaca tcgctaagaa 360
cgacttctct gacatcgaac tggctgctat cccgttcaac actctggctg accattacgg 420
tgagcgttta gctcgcgaac agttggccct tgagcatgag tcttacgaga tgggtgaagc 480
acgcttccgc aagatgtttg agcgtcaact taaagctggt gaggttgcgg ataacgctgc 540
cgccaagcct ctcatcacta ccctactccc taagatgatt gcacgcatca acgactggtt 600
tgaggaagtg aaagctaagc gcggcaagcg cccgacagcc ttccagttcc tgcaagaaat 660
caagccggaa gccgtagcgt acatcaccat taagaccact ctggcttgcc taaccagtgc 720
tgacaataca accgttcagg ctgtagcaag cgcaatcggt cgggccattg aggacgaggc 780
tcgcttcggt cgtatccgtg accttgaagc taagcacttc aagaaaaacg ttgaggaaca 840
actcaacaag cgcgtagggc acgtctacaa gaaagcattt atgcaagttg tcgaggctga 900
catgctctct aagggtctac tcggtggcga ggcgtggtct tcgtggcata aggaagactc 960
tattcatgta ggagtacgct gcatcgagat gctcattgag tcaaccggaa tggttagctt 1020
acaccgccaa aatgctggcg tagtaggtca agactctgag actatcgaac tcgcacctga 1080
atacgctgag gctatcgcaa cccgtgcagg tgcgctggct ggcatctctc cgatgttcca 1140
accttgcgta gttcctccta agccgtggac tggcattact ggtggtggct attgggctaa 1200
cggtcgtcgt cctctggcgc tggtgcgtac tcacagtaag aaagcactga tgcgctacga 1260
agacgtttac atgcctgagg tgtacaaagc gattaacatt gcgcaaaaca ccgcatggaa 1320
aatcaacaag aaagtcctag cggtcgccaa cgtaatcacc aagtggaagc attgtccggt 1380
cgaggacatc cctgcgattg agcgtgaaga actcccgatg aaaccggaag acatcgacat 1440
gaatcctgag gctctcaccg cgtggaaacg tgctgccgct gctgtgtacc gcaaggacaa 1500
ggctcgcaag tctcgccgta tcagccttga gttcatgctt gagcaagcca ataagtttgc 1560
taaccataag gccatctggt tcccttacaa catggactgg cgcggtcgtg tttacgctgt 1620
gtcaatgttc aacccgcaag gtaacgatat gaccaaagga ctgcttacgc tggcgaaagg 1680
taaaccaatc ggtaaggaag gttactactg gctgaaaatc cacggtgcaa actgtgcggg 1740
tgtcgacaag gttccgttcc ctgagcgcat caagttcatt gaggaaaacc acgagaacat 1800
catggcttgc gctaagtctc cactggagaa cacttggtgg gctgagcaag attctccgtt 1860
ctgcttcctt gcgttctgct ttgagtacgc tggggtacag caccacggcc tgagctataa 1920
ctgctccctt ccgctggcgt ttgacgggtc ttgctctggc atccagcact tctccgcgat 1980
gctccgagat gaggtaggtg gtcgcgcggt taacttgctt cctagtgaaa ccgttcagga 2040
catctacggg attgttgcta agaaagtcaa cgagattcta caagcagacg caatcaatgg 2100
gaccgataac gaagtagtta ccgtgaccga tgagaacact ggtgaaatct ctgagaaagt 2160
caagctgggc actaaggcac tggctggtca atggctggct tacggtgtta ctcgcagtgt 2220
gactaagcgt tcagtcatga cgctggctta cgggtccaaa gagttcggct tccgtcaaca 2280
agtgctggaa gataccattc agccagctat tgattccggc aagggtctga tgttcactca 2340
gccgaatcag gctgctggat acatggctaa gctgatttgg gaatctgtga gcgtgacggt 2400
ggtagctgcg gttgaagcaa tgaactggct taagtctgct gctaagctgc tggctgctga 2460
ggtcaaagat aagaagactg gagagattct tcgcaagcgt tgcgctgtgc attgggtaac 2520
tcctgatggt ttccctgtgt ggcaggaata caagaagcct attcagacgc gcttgaacct 2580
gatgttcctc ggtcagttcc gcttacagcc taccattaac accaacaaag atagcgagat 2640
tgatgcacac aaacaggagt ctggtatcgc tcctaacttt gtacacagcc aagacggtag 2700
ccaccttcgt aagactgtag tgtgggcaca cgagaagtac ggaatcgaat cttttgcact 2760
gattcacgac tccttcggta cgattccggc tgacgctgcg aacctgttca aagcagtgcg 2820
cgaaactatg gttgacacat atgagtcttg tgatgtactg gctgatttct acgaccagtt 2880
cgctgaccag ttgcacgagt ctcaattgga caaaatgcca gcacttccgg ctaaaggtaa 2940
cttgaacctc cgtgacatct tagagtcgga cttcgcgttc gcgtaaggat ccggcaagac 3000
tggccccgct tggcaacgca acagtgagcc cctccctagt gtgtttgggg atgtgactat 3060
gtattcgtgt gttggccaac gggtcaaccc gaacagattg atacccgcct tggcatttcc 3120
tgtcagaatg taacgtcagt tgatggtact 3150
<210> 128
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polynucleotide
<220>
<221> modified_base
<222> (23)..(42)
<223> a, c, t, g, unknown or other
<400> 128
gaaattaata cgactcacta tannnnnnnn nnnnnnnnnn nngttttaga gctagaaata 60
gcaagttaaa ataaggctag tccgttatca acttgaaaaa gtggcaccga gtcggtgctt 120
ttttt 125
<210> 129
<211> 8452
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polynucleotide
<400> 129
tgcggtattt cacaccgcat caggtggcac ttttcgggga aatgtgcgcg gaacccctat 60
ttgtttattt ttctaaatac attcaaatat gtatccgctc atgagattat caaaaaggat 120
cttcacctag atccttttaa attaaaaatg aagttttaaa tcaatctaaa gtatatatga 180
gtaaacttgg tctgacagtt accaatgctt aatcagtgag gcacctatct cagcgatctg 240
tctatttcgt tcatccatag ttgcctgact ccccgtcgtg tagataacta cgatacggga 300
gggcttacca tctggcccca gtgctgcaat gataccgcga gacccacgct caccggctcc 360
agatttatca gcaataaacc agccagccgg aagggccgag cgcagaagtg gtcctgcaac 420
tttatccgcc tccatccagt ctattaattg ttgccgggaa gctagagtaa gtagttcgcc 480
agttaatagt ttgcgcaacg ttgttgccat tgctacaggc atcgtggtgt cacgctcgtc 540
gtttggtatg gcttcattca gctccggttc ccaacgatca aggcgagtta catgatcccc 600
catgttgtgc aaaaaagcgg ttagctcctt cggtcctccg atcgttgtca gaagtaagtt 660
ggccgcagtg ttatcactca tggttatggc agcactgcat aattctctta ctgtcatgcc 720
atccgtaaga tgcttttctg tgactggtga gtactcaacc aagtcattct gagaatagtg 780
tatgcggcga ccgagttgct cttgcccggc gtcaatacgg gataataccg cgccacatag 840
cagaacttta aaagtgctca tcattggaaa acgttcttcg gggcgaaaac tctcaaggat 900
cttaccgctg ttgagatcca gttcgatgta acccactcgt gcacccaact gatcttcagc 960
atcttttact ttcaccagcg tttctgggtg agcaaaaaca ggaaggcaaa atgccgcaaa 1020
aaagggaata agggcgacac ggaaatgttg aatactcata ctcttccttt ttcaatatta 1080
ttgaagcatt tatcagggtt attgtctcat gaccaaaatc ccttaacgtg agttttcgtt 1140
ccactgagcg tcagaccccg tagaaaagat caaaggatct tcttgagatc ctttttttct 1200
gcgcgtaatc tgctgcttgc aaacaaaaaa accaccgcta ccagcggtgg tttgtttgcc 1260
ggatcaagag ctaccaactc tttttccgaa ggtaactggc ttcagcagag cgcagatacc 1320
aaatactgtt cttctagtgt agccgtagtt aggccaccac ttcaagaact ctgtagcacc 1380
gcctacatac ctcgctctgc taatcctgtt accagtggct gttgccagtg gcgataagtc 1440
gtgtcttacc gggttggact caagacgata gttaccggat aaggcgcagc ggtcgggctg 1500
aacggggggt tcgtgcacac agcccagctt ggagcgaacg acctacaccg aactgagata 1560
cctacagcgt gagctatgag aaagcgccac gcttcccgaa gggagaaagg cggacaggta 1620
tccggtaagc ggcagggtcg gaacaggaga gcgcacgagg gagcttccag ggggaaacgc 1680
ctggtatctt tatagtcctg tcgggtttcg ccacctctga cttgagcgtc gatttttgtg 1740
atgctcgtca ggggggcgga gcctatggaa aaacgccagc aacgcggcct ttttacggtt 1800
cctggccttt tgctggcctt ttgctcacat gttctttcct gcgttatccc ctgattctgt 1860
ggataaccgt attaccgcct ttgagtgagc tgataccgct cgccgcagcc gaacgaccga 1920
gcgcagcgag tcagtgagcg aggaagcggt cgctgaggct tgacatgatt ggtgcgtatg 1980
tttgtatgaa gctacaggac tgatttggcg ggctatgagg gcgggggaag ctctggaagg 2040
gccgcgatgg ggcgcgcggc gtccagaagg cgccatacgg cccgctggcg gcacccatcc 2100
ggtataaaag cccgcgaccc cgaacggtga cctccacttt cagcgacaaa cgagcactta 2160
tacatacgcg actattctgc cgctatacat aaccactcag ctagcttaag atcccatcaa 2220
gcttgcatgc cgggcgcgcc agaaggagcg cagccaaacc aggatgatgt ttgatggggt 2280
atttgagcac ttgcaaccct tatccggaag ccccctggcc cacaaaggct aggcgccaat 2340
gcaagcagtt cgcatgcagc ccctggagcg gtgccctcct gataaaccgg ccagggggcc 2400
tatgttcttt acttttttac aagagaagtc actcaacatc ttaaaatggc caggtgagtc 2460
gacgagcaag cccggcggat caggcagcgt gcttgcagat ttgacttgca acgcccgcat 2520
tgtgtcgacg aaggcttttg gctcctctgt cgctgtctca agcagcatct aaccctgcgt 2580
cgccgtttcc atttgcagga gattcgaggt accatgtacc catacgatgt tccagattac 2640
gcttcgccga agaaaaagcg caaggtcgaa gcgtccgaca agaagtacag catcggcctg 2700
gacatcggca ccaactctgt gggctgggcc gtgatcaccg acgagtacaa ggtgcccagc 2760
aagaaattca aggtgctggg caacaccgac cggcacagca tcaagaagaa cctgatcgga 2820
gccctgctgt tcgacagcgg cgaaacagcc gaggccaccc ggctgaagag aaccgccaga 2880
agaagataca ccagacggaa gaaccggatc tgctatctgc aagagatctt cagcaacgag 2940
atggccaagg tggacgacag cttcttccac agactggaag agtccttcct ggtggaagag 3000
gataagaagc acgagcggca ccccatcttc ggcaacatcg tggacgaggt ggcctaccac 3060
gagaagtacc ccaccatcta ccacctgaga aagaaactgg tggacagcac cgacaaggcc 3120
gacctgcggc tgatctatct ggccctggcc cacatgatca agttccgggg ccacttcctg 3180
atcgagggcg acctgaaccc cgacaacagc gacgtggaca agctgttcat ccagctggtg 3240
cagacctaca accagctgtt cgaggaaaac cccatcaacg ccagcggcgt ggacgccaag 3300
gccatcctgt ctgccagact gagcaagagc agacggctgg aaaatctgat cgcccagctg 3360
cccggcgaga agaagaatgg cctgttcggc aacctgattg ccctgagcct gggcctgacc 3420
cccaacttca agagcaactt cgacctggcc gaggatgcca aactgcagct gagcaaggac 3480
acctacgacg acgacctgga caacctgctg gcccagatcg gcgaccagta cgccgacctg 3540
tttctggccg ccaagaacct gtccgacgcc atcctgctga gcgacatcct gagagtgaac 3600
accgagatca ccaaggcccc cctgagcgcc tctatgatca agagatacga cgagcaccac 3660
caggacctga ccctgctgaa agctctcgtg cggcagcagc tgcctgagaa gtacaaagag 3720
attttcttcg accagagcaa gaacggctac gccggctaca ttgacggcgg agccagccag 3780
gaagagttct acaagttcat caagcccatc ctggaaaaga tggacggcac cgaggaactg 3840
ctcgtgaagc tgaacagaga ggacctgctg cggaagcagc ggaccttcga caacggcagc 3900
atcccccacc agatccacct gggagagctg cacgccattc tgcggcggca ggaagatttt 3960
tacccattcc tgaaggacaa ccgggaaaag atcgagaaga tcctgacctt ccgcatcccc 4020
tactacgtgg gccctctggc caggggaaac agcagattcg cctggatgac cagaaagagc 4080
gaggaaacca tcaccccctg gaacttcgag gaagtggtgg acaagggcgc ttccgcccag 4140
agcttcatcg agcggatgac caacttcgat aagaacctgc ccaacgagaa ggtgctgccc 4200
aagcacagcc tgctgtacga gtacttcacc gtgtataacg agctgaccaa agtgaaatac 4260
gtgaccgagg gaatgagaaa gcccgccttc ctgagcggcg agcagaaaaa ggccatcgtg 4320
gacctgctgt tcaagaccaa ccggaaagtg accgtgaagc agctgaaaga ggactacttc 4380
aagaaaatcg agtgcttcga ctccgtggaa atctccggcg tggaagatcg gttcaacgcc 4440
tccctgggca cataccacga tctgctgaaa attatcaagg acaaggactt cctggacaat 4500
gaggaaaacg aggacattct ggaagatatc gtgctgaccc tgacactgtt tgaggacaga 4560
gagatgatcg aggaacggct gaaaacctat gcccacctgt tcgacgacaa agtgatgaag 4620
cagctgaagc ggcggagata caccggctgg ggcaggctga gccggaagct gatcaacggc 4680
atccgggaca agcagtccgg caagacaatc ctggatttcc tgaagtccga cggcttcgcc 4740
aacagaaact tcatgcagct gatccacgac gacagcctga cctttaaaga ggacatccag 4800
aaagcccagg tgtccggcca gggcgatagc ctgcacgagc acattgccaa tctggccggc 4860
agccccgcca ttaagaaggg catcctgcag acagtgaagg tggtggacga gctcgtgaaa 4920
gtgatgggcc ggcacaagcc cgagaacatc gtgatcgaaa tggccagaga gaaccagacc 4980
acccagaagg gacagaagaa cagccgcgag agaatgaagc ggatcgaaga gggcatcaaa 5040
gagctgggca gccagatcct gaaagaacac cccgtggaaa acacccagct gcagaacgag 5100
aagctgtacc tgtactacct gcagaatggg cgggatatgt acgtggacca ggaactggac 5160
atcaaccggc tgtccgacta cgatgtggac catatcgtgc ctcagagctt tctgaaggac 5220
gactccatcg acaacaaggt gctgaccaga agcgacaaga accggggcaa gagcgacaac 5280
gtgccctccg aagaggtcgt gaagaagatg aagaactact ggcggcagct gctgaacgcc 5340
aagctgatta cccagagaaa gttcgacaat ctgaccaagg ccgagagagg cggcctgagc 5400
gaactggata aggccggctt catcaagaga cagctggtgg aaacccggca gatcacaaag 5460
cacgtggcac agatcctgga ctcccggatg aacactaagt acgacgagaa tgacaagctg 5520
atccgggaag tgaaagtgat caccctgaag tccaagctgg tgtccgattt ccggaaggat 5580
ttccagtttt acaaagtgcg cgagatcaac aactaccacc acgcccacga cgcctacctg 5640
aacgccgtcg tgggaaccgc cctgatcaaa aagtacccta agctggaaag cgagttcgtg 5700
tacggcgact acaaggtgta cgacgtgcgg aagatgatcg ccaagagcga gcaggaaatc 5760
ggcaaggcta ccgccaagta cttcttctac agcaacatca tgaacttttt caagaccgag 5820
attaccctgg ccaacggcga gatccggaag cggcctctga tcgagacaaa cggcgaaacc 5880
ggggagatcg tgtgggataa gggccgggat tttgccaccg tgcggaaagt gctgagcatg 5940
ccccaagtga atatcgtgaa aaagaccgag gtgcagacag gcggcttcag caaagagtct 6000
atcctgccca agaggaacag cgataagctg atcgccagaa agaaggactg ggaccctaag 6060
aagtacggcg gcttcgacag ccccaccgtg gcctattctg tgctggtggt ggccaaagtg 6120
gaaaagggca agtccaagaa actgaagagt gtgaaagagc tgctggggat caccatcatg 6180
gaaagaagca gcttcgagaa gaatcccatc gactttctgg aagccaaggg ctacaaagaa 6240
gtgaaaaagg acctgatcat caagctgcct aagtactccc tgttcgagct ggaaaacggc 6300
cggaagagaa tgctggcctc tgccggcgaa ctgcagaagg gaaacgaact ggccctgccc 6360
tccaaatatg tgaacttcct gtacctggcc agccactatg agaagctgaa gggctccccc 6420
gaggataatg agcagaaaca gctgtttgtg gaacagcaca agcactacct ggacgagatc 6480
atcgagcaga tcagcgagtt ctccaagaga gtgatcctgg ccgacgctaa tctggacaaa 6540
gtgctgtccg cctacaacaa gcaccgggat aagcccatca gagagcaggc cgagaatatc 6600
atccacctgt ttaccctgac caatctggga gcccctgccg ccttcaagta ctttgacacc 6660
accatcgacc ggaagaggta caccagcacc aaagaggtgc tggacgccac cctgatccac 6720
cagagcatca ccggcctgta cgagacacgg atcgacctgt ctcagctggg aggcgacagc 6780
cccaagaaga agagaaaggt ggaggccagc taacatatga ttcgaatgtc tttcttgcgc 6840
tatgacactt ccagcaaaag gtagggcggg ctgcgagacg gcttcccggc gctgcatgca 6900
acaccgatga tgcttcgacc ccccgaagct ccttcggggc tgcatgggcg ctccgatgcc 6960
gctccagggc gagcgctgtt taaatagcca ggcccccgat tgcaaagaca ttatagcgag 7020
ctaccaaagc catattcaaa cacctagatc actaccactt ctacacaggc cactcgagct 7080
tgtgatcgca ctccgctaag ggggcgcctc ttcctcttcg tttcagtcac aacccgcaaa 7140
catgacacaa gaatccctgt tacttctcga ccgtattgat tcggatgatt cctacgcgag 7200
cctgcggaac gaccaggaat tctgggaggt gagtcgacga gcaagcccgg cggatcaggc 7260
agcgtgcttg cagatttgac ttgcaacgcc cgcattgtgt cgacgaaggc ttttggctcc 7320
tctgtcgctg tctcaagcag catctaaccc tgcgtcgccg tttccatttg cagccgctgg 7380
cccgccgagc cctggaggag ctcgggctgc cggtgccgcc ggtgctgcgg gtgcccggcg 7440
agagcaccaa ccccgtactg gtcggcgagc ccggcccggt gatcaagctg ttcggcgagc 7500
actggtgcgg tccggagagc ctcgcgtcgg agtcggaggc gtacgcggtc ctggcggacg 7560
ccccggtgcc ggtgccccgc ctcctcggcc gcggcgagct gcggcccggc accggagcct 7620
ggccgtggcc ctacctggtg atgagccgga tgaccggcac cacctggcgg tccgcgatgg 7680
acggcacgac cgaccggaac gcgctgctcg ccctggcccg cgaactcggc cgggtgctcg 7740
gccggctgca cagggtgccg ctgaccggga acaccgtgct caccccccat tccgaggtct 7800
tcccggaact gctgcgggaa cgccgcgcgg cgaccgtcga ggaccaccgc gggtggggct 7860
acctctcgcc ccggctgctg gaccgcctgg aggactggct gccggacgtg gacacgctgc 7920
tggccggccg cgaaccccgg ttcgtccacg gcgacctgca cgggaccaac atcttcgtgg 7980
acctggccgc gaccgaggtc accgggatcg tcgacttcac cgacgtctat gcgggagact 8040
cccgctacag cctggtgcaa ctgcatctca acgccttccg gggcgaccgc gagatcctgg 8100
ccgcgctgct cgacggggcg cagtggaagc ggaccgagga cttcgcccgc gaactgctcg 8160
ccttcacctt cctgcacgac ttcgaggtgt tcgaggagac cccgctggat ctctccggct 8220
tcaccgatcc ggaggaactg gcgcagttcc tctgggggcc gccggacacc gcccccggcg 8280
cctgataagg atccggcaag actggccccg cttggcaacg caacagtgag cccctcccta 8340
gtgtgtttgg ggatgtgact atgtattcgt gtgttggcca acgggtcaac ccgaacagat 8400
tgatacccgc cttggcattt cctgtcagaa tgtaacgtca gttgatggta ct 8452
<210> 130
<211> 27
<212> DNA
<213> Homo sapiens
<400> 130
ccgtgccggg cggggagacc gccatgg 27
<210> 131
<211> 22
<212> DNA
<213> Homo sapiens
<400> 131
ggcccggctg tggctgagga gc 22
<210> 132
<211> 20
<212> DNA
<213> Homo sapiens
<400> 132
cggtctcccg cccggcacgg 20
<210> 133
<211> 26
<212> DNA
<213> Homo sapiens
<400> 133
gctcctcagc cacagccggg ccgggt 26
<210> 134
<211> 12
<212> DNA
<213> Homo sapiens
<400> 134
cgaccctgga aa 12
<210> 135
<211> 14
<212> DNA
<213> Homo sapiens
<400> 135
ccccgccgcc accc 14
<210> 136
<211> 18
<212> DNA
<213> Homo sapiens
<400> 136
tttccagggt cgccatgg 18
<210> 137
<211> 10
<212> DNA
<213> Homo sapiens
<400> 137
ggcggcgggg 10
<210> 138
<211> 20
<212> DNA
<213> Homo sapiens
<400> 138
acccttgtta gccacctccc 20
<210> 139
<211> 20
<212> DNA
<213> Homo sapiens
<400> 139
gaacgcagtg ctcttcgaag 20
<210> 140
<211> 20
<212> DNA
<213> Homo sapiens
<400> 140
ctcacgccct gctccgtgta 20
<210> 141
<211> 20
<212> DNA
<213> Homo sapiens
<400> 141
ggcgacaact acttcctggt 20
<210> 142
<211> 20
<212> DNA
<213> Homo sapiens
<400> 142
ctcacgccct gctccgtgta 20
<210> 143
<211> 20
<212> DNA
<213> Homo sapiens
<400> 143
gggcgacaac tacttcctgg 20
<210> 144
<211> 20
<212> DNA
<213> Homo sapiens
<400> 144
cctcttcagg gccggggtgg 20
<210> 145
<211> 20
<212> DNA
<213> Homo sapiens
<400> 145
gaggacccag gtggaactgc 20
<210> 146
<211> 20
<212> DNA
<213> Homo sapiens
<400> 146
tcagctccag gcggtcctgg 20
<210> 147
<211> 20
<212> DNA
<213> Homo sapiens
<400> 147
agcagcagca gcagtggcag 20
<210> 148
<211> 20
<212> DNA
<213> Homo sapiens
<400> 148
tgggcaccgt cagctccagg 20
<210> 149
<211> 20
<212> DNA
<213> Homo sapiens
<400> 149
cagcagtggc agcggccacc 20
<210> 150
<211> 20
<212> DNA
<213> Homo sapiens
<400> 150
acctctcccc tggccctcat 20
<210> 151
<211> 20
<212> DNA
<213> Homo sapiens
<400> 151
ccaggaccgc ctggagctga 20
<210> 152
<211> 20
<212> DNA
<213> Homo sapiens
<400> 152
ccgtcagctc caggcggtcc 20
<210> 153
<211> 20
<212> DNA
<213> Homo sapiens
<400> 153
agcagcagca gcagtggcag 20
<210> 154
<211> 20
<212> DNA
<213> Homo sapiens
<400> 154
atgtgccaag caaagcctca 20
<210> 155
<211> 20
<212> DNA
<213> Homo sapiens
<400> 155
ttcggtcatg cccgtggatg 20
<210> 156
<211> 20
<212> DNA
<213> Homo sapiens
<400> 156
gtcgttgaaa ttcatcgtac 20
<210> 157
<211> 20
<212> DNA
<213> Homo sapiens
<400> 157
accacctgtg aagagtttcc 20
<210> 158
<211> 20
<212> DNA
<213> Homo sapiens
<400> 158
cgtcgttgaa attcatcgta 20
<210> 159
<211> 20
<212> DNA
<213> Homo sapiens
<400> 159
accacctgtg aagagtttcc 20
<210> 160
<211> 20
<212> DNA
<213> Mus musculus
<400> 160
gaacgcagtg cttttcgagg 20
<210> 161
<211> 20
<212> DNA
<213> Mus musculus
<400> 161
acccttgttg gccacctccc 20
<210> 162
<211> 20
<212> DNA
<213> Mus musculus
<400> 162
ggtgacaact actatctggt 20
<210> 163
<211> 20
<212> DNA
<213> Mus musculus
<400> 163
ctcacaccct gctccgtgta 20
<210> 164
<211> 20
<212> DNA
<213> Mus musculus
<400> 164
gggtgacaac tactatctgg 20
<210> 165
<211> 20
<212> DNA
<213> Mus musculus
<400> 165
ctcacaccct gctccgtgta 20
<210> 166
<211> 20
<212> DNA
<213> Mus musculus
<400> 166
cgagaacgca gtgcttttcg 20
<210> 167
<211> 20
<212> DNA
<213> Mus musculus
<400> 167
acccttgttg gccacctccc 20
<210> 168
<211> 20
<212> DNA
<213> Mus musculus
<400> 168
atgagccaag caaatcctca 20
<210> 169
<211> 20
<212> DNA
<213> Mus musculus
<400> 169
ttccgtcatg cccgtggaca 20
<210> 170
<211> 20
<212> DNA
<213> Mus musculus
<400> 170
cttcgttgaa aaccattgta 20
<210> 171
<211> 20
<212> DNA
<213> Mus musculus
<400> 171
ccacctctga agagtttcct 20
<210> 172
<211> 20
<212> DNA
<213> Mus musculus
<400> 172
cttcgttgaa aaccattgta 20
<210> 173
<211> 20
<212> DNA
<213> Mus musculus
<400> 173
accacctctg aagagtttcc 20
<210> 174
<211> 20
<212> DNA
<213> Mus musculus
<400> 174
cttccactca ctctgcgatt 20
<210> 175
<211> 20
<212> DNA
<213> Mus musculus
<400> 175
accatgtctc agtgtcaagc 20
<210> 176
<211> 20
<212> DNA
<213> Mus musculus
<400> 176
ggcggcaaca gcggcaacag 20
<210> 177
<211> 20
<212> DNA
<213> Mus musculus
<400> 177
actgctctgc gtggctgcgg 20
<210> 178
<211> 20
<212> DNA
<213> Mus musculus
<400> 178
ccgcagccac gcagagcagt 20
<210> 179
<211> 20
<212> DNA
<213> Mus musculus
<400> 179
gcacctctcc tcgccccgat 20
<210> 180
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 180
gagggcctat ttcccatgat tcc 23
<210> 181
<211> 126
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<220>
<221> modified_base
<222> (84)..(102)
<223> a, c, t, g, unknown or other
<400> 181
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacnnnnnnn nnnnnnnnnn nnccggtgtt tcgtcctttc 120
cacaag 126
<210> 182
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<220>
<221> modified_base
<222> (6)..(24)
<223> a, c, t, g, unknown or other
<400> 182
caccgnnnnn nnnnnnnnnn nnnn 24
<210> 183
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<220>
<221> modified_base
<222> (5)..(23)
<223> a, c, t, g, unknown or other
<400> 183
aaacnnnnnn nnnnnnnnnn nnnc 24
<210> 184
<211> 126
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 184
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacccctagt cattggaggt gaccggtgtt tcgtcctttc 120
cacaag 126
<210> 185
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 185
caccgtcacc tccaatgact aggg 24
<210> 186
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 186
aaacccctag tcattggagg tgac 24
<210> 187
<211> 192
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 187
cagaagaaga agggctccca tcacatcaac cggtggcgca ttgccacgaa gcaggccaat 60
ggggaggaca tcgatgtcac ctccaatgac aagcttgcta gcggtgggca accacaaacc 120
cacgagggca gagtgctgct tgctgctggc caggcccctg cgtgggccca agctggactc 180
tggccactcc ct 192
<210> 188
<211> 192
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 188
agggagtggc cagagtccag cttgggccca cgcaggggcc tggccagcag caagcagcac 60
tctgccctcg tgggtttgtg gttgcccacc gctagcaagc ttgtcattgg aggtgacatc 120
gatgtcctcc ccattggcct gcttcgtggc aatgcgccac cggttgatgt gatgggagcc 180
cttcttcttc tg 192
<210> 189
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 189
ccatcccctt ctgtgaatgt 20
<210> 190
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 190
ggagattgga gacacggaga 20
<210> 191
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 191
ggctccctgg gttcaaagta 20
<210> 192
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 192
agaggggtct ggatgtcgta a 21
<210> 193
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 193
cgccagggtt ttcccagtca cgac 24
<210> 194
<211> 51
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 194
gagggtctcg tccttgcggc cgcgctagcg agggcctatt tcccatgatt c 51
<210> 195
<211> 133
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<220>
<221> modified_base
<222> (95)..(114)
<223> a, c, t, g, unknown or other
<400> 195
ctcggtctcg gtaaaaaagc accgactcgg tgccactttt tcaagttgat aacggactag 60
ccttatttta acttgctatt tctagctcta aaacnnnnnn nnnnnnnnnn nnnnggtgtt 120
tcgtcctttc cac 133
<210> 196
<211> 41
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 196
gagggtctct ttaccggtga gggcctattt cccatgattc c 41
<210> 197
<211> 133
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<220>
<221> modified_base
<222> (95)..(114)
<223> a, c, t, g, unknown or other
<400> 197
ctcggtctcc tcaaaaaagc accgactcgg tgccactttt tcaagttgat aacggactag 60
ccttatttta acttgctatt tctagctcta aaacnnnnnn nnnnnnnnnn nnnnggtgtt 120
tcgtcctttc cac 133
<210> 198
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 198
gagggtctct ttgagctcga gggcctattt cccatgattc 40
<210> 199
<211> 133
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<220>
<221> modified_base
<222> (96)..(115)
<223> a, c, t, g, unknown or other
<400> 199
ctcggtctcg cgtaaaaaag caccgactcg gtgccacttt ttcaagttga taacggacta 60
gccttatttt aacttgctat ttctagctct aaaacnnnnn nnnnnnnnnn nnnnnggtgt 120
ttcgtccttt cca 133
<210> 200
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 200
gagggtctct tacgcgtgtg tctagac 27
<210> 201
<211> 98
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 201
ctcggtctca aggacaggga agggagcagt ggttcacgcc tgtaatccca gcaatttggg 60
aggccaaggt gggtagatca cctgagatta ggagttgc 98
<210> 202
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 202
cctgtccttg cggccgcgct agcgagggcc 30
<210> 203
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 203
cacgcggccg caaggacagg gaagggagca g 31
<210> 204
<211> 327
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 204
Met Val Ser Lys Gly Glu Glu Leu Phe Thr Gly Val Val Pro Ile Leu
1 5 10 15
Val Glu Leu Asp Gly Asp Val Asn Gly His Lys Phe Ser Val Ser Gly
20 25 30
Glu Gly Glu Gly Asp Ala Thr Tyr Gly Lys Leu Thr Leu Lys Phe Ile
35 40 45
Cys Thr Thr Gly Lys Leu Pro Val Pro Trp Pro Thr Leu Val Thr Thr
50 55 60
Leu Thr Tyr Gly Val Gln Cys Phe Ser Arg Tyr Pro Asp His Met Lys
65 70 75 80
Gln His Asp Phe Phe Lys Ser Ala Met Pro Glu Gly Tyr Val Gln Glu
85 90 95
Arg Thr Ile Phe Phe Lys Asp Asp Gly Asn Tyr Lys Thr Arg Ala Glu
100 105 110
Val Lys Phe Glu Gly Asp Thr Leu Val Asn Arg Ile Glu Leu Lys Gly
115 120 125
Ile Asp Phe Lys Glu Asp Gly Asn Ile Leu Gly His Lys Leu Glu Tyr
130 135 140
Asn Tyr Asn Ser His Asn Val Tyr Ile Met Ala Asp Lys Gln Lys Asn
145 150 155 160
Gly Ile Lys Val Asn Phe Lys Ile Arg His Asn Ile Glu Asp Gly Ser
165 170 175
Val Gln Leu Ala Asp His Tyr Gln Gln Asn Thr Pro Ile Gly Asp Gly
180 185 190
Pro Val Leu Leu Pro Asp Asn His Tyr Leu Ser Thr Gln Ser Ala Leu
195 200 205
Ser Lys Asp Pro Asn Glu Lys Arg Asp His Met Val Leu Leu Glu Phe
210 215 220
Val Thr Ala Ala Gly Ile Thr Leu Gly Met Asp Glu Leu Tyr Lys Ser
225 230 235 240
Gly Leu Arg Ser Arg Glu Glu Glu Glu Glu Thr Asp Ser Arg Met Pro
245 250 255
His Leu Asp Ser Pro Gly Ser Ser Gln Pro Arg Arg Ser Phe Leu Ser
260 265 270
Arg Val Ile Arg Ala Ala Leu Pro Leu Gln Leu Leu Leu Leu Leu Leu
275 280 285
Leu Leu Leu Ala Cys Leu Leu Pro Ala Ser Glu Asp Asp Tyr Ser Cys
290 295 300
Thr Gln Ala Asn Asn Phe Ala Arg Ser Phe Tyr Pro Met Leu Arg Tyr
305 310 315 320
Thr Asn Gly Pro Pro Pro Thr
325
<210> 205
<211> 3243
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polynucleotide
<400> 205
accggtgcca ccatgtaccc atacgatgtt ccagattacg cttcgccgaa gaaaaagcgc 60
aaggtcgaag cgtccatgaa aaggaactac attctggggc tggacatcgg gattacaagc 120
gtggggtatg ggattattga ctatgaaaca agggacgtga tcgacgcagg cgtcagactg 180
ttcaaggagg ccaacgtgga aaacaatgag ggacggagaa gcaagagggg agccaggcgc 240
ctgaaacgac ggagaaggca cagaatccag agggtgaaga aactgctgtt cgattacaac 300
ctgctgaccg accattctga gctgagtgga attaatcctt atgaagccag ggtgaaaggc 360
ctgagtcaga agctgtcaga ggaagagttt tccgcagctc tgctgcacct ggctaagcgc 420
cgaggagtgc ataacgtcaa tgaggtggaa gaggacaccg gcaacgagct gtctacaaag 480
gaacagatct cacgcaatag caaagctctg gaagagaagt atgtcgcaga gctgcagctg 540
gaacggctga agaaagatgg cgaggtgaga gggtcaatta ataggttcaa gacaagcgac 600
tacgtcaaag aagccaagca gctgctgaaa gtgcagaagg cttaccacca gctggatcag 660
agcttcatcg atacttatat cgacctgctg gagactcgga gaacctacta tgagggacca 720
ggagaaggga gccccttcgg atggaaagac atcaaggaat ggtacgagat gctgatggga 780
cattgcacct attttccaga agagctgaga agcgtcaagt acgcttataa cgcagatctg 840
tacaacgccc tgaatgacct gaacaacctg gtcatcacca gggatgaaaa cgagaaactg 900
gaatactatg agaagttcca gatcatcgaa aacgtgttta agcagaagaa aaagcctaca 960
ctgaaacaga ttgctaagga gatcctggtc aacgaagagg acatcaaggg ctaccgggtg 1020
acaagcactg gaaaaccaga gttcaccaat ctgaaagtgt atcacgatat taaggacatc 1080
acagcacgga aagaaatcat tgagaacgcc gaactgctgg atcagattgc taagatcctg 1140
actatctacc agagctccga ggacatccag gaagagctga ctaacctgaa cagcgagctg 1200
acccaggaag agatcgaaca gattagtaat ctgaaggggt acaccggaac acacaacctg 1260
tccctgaaag ctatcaatct gattctggat gagctgtggc atacaaacga caatcagatt 1320
gcaatcttta accggctgaa gctggtccca aaaaaggtgg acctgagtca gcagaaagag 1380
atcccaacca cactggtgga cgatttcatt ctgtcacccg tggtcaagcg gagcttcatc 1440
cagagcatca aagtgatcaa cgccatcatc aagaagtacg gcctgcccaa tgatatcatt 1500
atcgagctgg ctagggagaa gaacagcaag gacgcacaga agatgatcaa tgagatgcag 1560
aaacgaaacc ggcagaccaa tgaacgcatt gaagagatta tccgaactac cgggaaagag 1620
aacgcaaagt acctgattga aaaaatcaag ctgcacgata tgcaggaggg aaagtgtctg 1680
tattctctgg aggccatccc cctggaggac ctgctgaaca atccattcaa ctacgaggtc 1740
gatcatatta tccccagaag cgtgtccttc gacaattcct ttaacaacaa ggtgctggtc 1800
aagcaggaag agaactctaa aaagggcaat aggactcctt tccagtacct gtctagttca 1860
gattccaaga tctcttacga aacctttaaa aagcacattc tgaatctggc caaaggaaag 1920
ggccgcatca gcaagaccaa aaaggagtac ctgctggaag agcgggacat caacagattc 1980
tccgtccaga aggattttat taaccggaat ctggtggaca caagatacgc tactcgcggc 2040
ctgatgaatc tgctgcgatc ctatttccgg gtgaacaatc tggatgtgaa agtcaagtcc 2100
atcaacggcg ggttcacatc ttttctgagg cgcaaatgga agtttaaaaa ggagcgcaac 2160
aaagggtaca agcaccatgc cgaagatgct ctgattatcg caaatgccga cttcatcttt 2220
aaggagtgga aaaagctgga caaagccaag aaagtgatgg agaaccagat gttcgaagag 2280
aagcaggccg aatctatgcc cgaaatcgag acagaacagg agtacaagga gattttcatc 2340
actcctcacc agatcaagca tatcaaggat ttcaaggact acaagtactc tcaccgggtg 2400
gataaaaagc ccaacagaga gctgatcaat gacaccctgt atagtacaag aaaagacgat 2460
aaggggaata ccctgattgt gaacaatctg aacggactgt acgacaaaga taatgacaag 2520
ctgaaaaagc tgatcaacaa aagtcccgag aagctgctga tgtaccacca tgatcctcag 2580
acatatcaga aactgaagct gattatggag cagtacggcg acgagaagaa cccactgtat 2640
aagtactatg aagagactgg gaactacctg accaagtata gcaaaaagga taatggcccc 2700
gtgatcaaga agatcaagta ctatgggaac aagctgaatg cccatctgga catcacagac 2760
gattacccta acagtcgcaa caaggtggtc aagctgtcac tgaagccata cagattcgat 2820
gtctatctgg acaacggcgt gtataaattt gtgactgtca agaatctgga tgtcatcaaa 2880
aaggagaact actatgaagt gaatagcaag tgctacgaag aggctaaaaa gctgaaaaag 2940
attagcaacc aggcagagtt catcgcctcc ttttacaaca acgacctgat taagatcaat 3000
ggcgaactgt atagggtcat cggggtgaac aatgatctgc tgaaccgcat tgaagtgaat 3060
atgattgaca tcacttaccg agagtatctg gaaaacatga atgataagcg cccccctcga 3120
attatcaaaa caattgcctc taagactcag agtatcaaaa agtactcaac cgacattctg 3180
ggaaacctgt atgaggtgaa gagcaaaaag caccctcaga ttatcaaaaa gggctaagaa 3240
ttc 3243
<210> 206
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 206
atctcttaga taccagcatc 20
<210> 207
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 207
tcaatctccc gatgggcacc 20
<210> 208
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 208
gcccatcggg agattgaggg 20
<210> 209
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 209
acttcaacag cgtgccggag 20
<210> 210
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 210
ccgctgacca cacctgccag 20
<210> 211
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 211
tggcaggtgt ggtcagcggc 20
<210> 212
<211> 102
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polynucleotide
<400> 212
gttttagagc tatgctgttt tgaatggtcc caaaacggaa gggcctgagt ccgagcagaa 60
gaagaagttt tagagctatg ctgttttgaa tggtcccaaa ac 102
<210> 213
<211> 100
<212> DNA
<213> Homo sapiens
<400> 213
cggaggacaa agtacaaacg gcagaagctg gaggaggaag ggcctgagtc cgagcagaag 60
aagaagggct cccatcacat caaccggtgg cgcattgcca 100
<210> 214
<211> 50
<212> DNA
<213> Homo sapiens
<400> 214
agctggagga ggaagggcct gagtccgagc agaagaagaa gggctcccac 50
<210> 215
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 215
gaguccgagc agaagaagaa guuuuagagc 30
<210> 216
<211> 49
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 216
agctggagga ggaagggcct gagtccgagc agaagagaag ggctcccac 49
<210> 217
<211> 53
<212> DNA
<213> Homo sapiens
<400> 217
ctggaggagg aagggcctga gtccgagcag aagaagaagg gctcccatca cat 53
<210> 218
<211> 52
<212> DNA
<213> Homo sapiens
<400> 218
ctggaggagg aagggcctga gtccgagcag aagagaaggg ctcccatcac at 52
<210> 219
<211> 54
<212> DNA
<213> Homo sapiens
<400> 219
ctggaggagg aagggcctga gtccgagcag aagaaagaag ggctcccatc acat 54
<210> 220
<211> 50
<212> DNA
<213> Homo sapiens
<400> 220
ctggaggagg aagggcctga gtccgagcag aagaagggct cccatcacat 50
<210> 221
<211> 47
<212> DNA
<213> Homo sapiens
<400> 221
ctggaggagg aagggcctga gcccgagcag aagggctccc atcacat 47
<210> 222
<211> 48
<212> DNA
<213> Homo sapiens
<400> 222
ctggaggagg aagggcctga gtccgagcag aagaagaagg gctcccat 48
<210> 223
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 223
gaguccgagc agaagaagau 20
<210> 224
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 224
gaguccgagc agaagaagua 20
<210> 225
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 225
gaguccgagc agaagaacaa 20
<210> 226
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 226
gaguccgagc agaagaugaa 20
<210> 227
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 227
gaguccgagc agaaguagaa 20
<210> 228
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 228
gaguccgagc agaugaagaa 20
<210> 229
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 229
gaguccgagc acaagaagaa 20
<210> 230
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 230
gaguccgagg agaagaagaa 20
<210> 231
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 231
gaguccgugc agaagaagaa 20
<210> 232
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 232
gagucggagc agaagaagaa 20
<210> 233
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 233
gagaccgagc agaagaagaa 20
<210> 234
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 234
aatgacaagc ttgctagcgg tggg 24
<210> 235
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 235
aaaacggaag ggcctgagtc cgagcagaag aagaagttt 39
<210> 236
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 236
aaacaggggc cgagattggg tgttcagggc agaggtttt 39
<210> 237
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 237
aaaacggaag ggcctgagtc cgagcagaag aagaagtt 38
<210> 238
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 238
aacggaggga ggggcacaga tgagaaactc agggttttag 40
<210> 239
<211> 38
<212> DNA
<213> Homo sapiens
<400> 239
agcccttctt cttctgctcg gactcaggcc cttcctcc 38
<210> 240
<211> 40
<212> DNA
<213> Homo sapiens
<400> 240
cagggaggga ggggcacaga tgagaaactc aggaggcccc 40
<210> 241
<211> 80
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 241
ggcaatgcgc caccggttga tgtgatggga gcccttctag gaggccccca gagcagccac 60
tggggcctca acactcaggc 80
<210> 242
<211> 33
<212> DNA
<213> Homo sapiens
<400> 242
catcgatgtc ctccccattg gcctgcttcg tgg 33
<210> 243
<211> 33
<212> DNA
<213> Homo sapiens
<400> 243
ttcgtggcaa tgcgccaccg gttgatgtga tgg 33
<210> 244
<211> 33
<212> DNA
<213> Homo sapiens
<400> 244
tcgtggcaat gcgccaccgg ttgatgtgat ggg 33
<210> 245
<211> 33
<212> DNA
<213> Homo sapiens
<400> 245
tccagcttct gccgtttgta ctttgtcctc cgg 33
<210> 246
<211> 33
<212> DNA
<213> Homo sapiens
<400> 246
ggagggaggg gcacagatga gaaactcagg agg 33
<210> 247
<211> 33
<212> DNA
<213> Homo sapiens
<400> 247
aggggccgag attgggtgtt cagggcagag agg 33
<210> 248
<211> 33
<212> DNA
<213> Mus musculus
<400> 248
caagcactga gtgccattag ctaaatgcat agg 33
<210> 249
<211> 33
<212> DNA
<213> Mus musculus
<400> 249
aatgcatagg gtaccaccca caggtgccag ggg 33
<210> 250
<211> 33
<212> DNA
<213> Mus musculus
<400> 250
acacacatgg gaaagcctct gggccaggaa agg 33
<210> 251
<211> 37
<212> DNA
<213> Homo sapiens
<400> 251
ggaggaggta gtatacagaa acacagagaa gtagaat 37
<210> 252
<211> 37
<212> DNA
<213> Homo sapiens
<400> 252
agaatgtaga ggagtcacag aaactcagca ctagaaa 37
<210> 253
<211> 98
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 253
ggacgaaaca ccggaaccat tcaaaacagc atagcaagtt aaaataaggc tagtccgtta 60
tcaacttgaa aaagtggcac cgagtcggtg cttttttt 98
<210> 254
<211> 186
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polynucleotide
<400> 254
ggacgaaaca ccggtagtat taagtattgt tttatggctg ataaatttct ttgaatttct 60
ccttgattat ttgttataaa agttataaaa taatcttgtt ggaaccattc aaaacagcat 120
agcaagttaa aataaggcta gtccgttatc aacttgaaaa agtggcaccg agtcggtgct 180
tttttt 186
<210> 255
<211> 95
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 255
gggttttaga gctatgctgt tttgaatggt cccaaaacgg gtcttcgaga agacgtttta 60
gagctatgct gttttgaatg gtcccaaaac ttttt 95
<210> 256
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (5)..(34)
<223> a, c, t, g, unknown or other
<400> 256
aaacnnnnnn nnnnnnnnnn nnnnnnnnnn nnnngt 36
<210> 257
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (7)..(36)
<223> a, c, t, g, unknown or other
<400> 257
taaaacnnnn nnnnnnnnnn nnnnnnnnnn nnnnnn 36
<210> 258
<211> 84
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 258
gtggaaagga cgaaacaccg ggtcttcgag aagacctgtt ttagagctag aaatagcaag 60
ttaaaataag gctagtccgt tttt 84
<210> 259
<211> 46
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(19)
<223> a, c, u, g, unknown or other
<400> 259
nnnnnnnnnn nnnnnnnnng uuauuguacu cucaagauuu auuuuu 46
<210> 260
<211> 91
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 260
guuacuuaaa ucuugcagaa gcuacaaaga uaaggcuuca ugccgaaauc aacacccugu 60
cauuuuaugg caggguguuu ucguuauuua a 91
<210> 261
<211> 70
<212> DNA
<213> Homo sapiens
<400> 261
ttttctagtg ctgagtttct gtgactcctc tacattctac ttctctgtgt ttctgtatac 60
tacctcctcc 70
<210> 262
<211> 122
<212> DNA
<213> Homo sapiens
<400> 262
ggaggaaggg cctgagtccg agcagaagaa gaagggctcc catcacatca accggtggcg 60
cattgccacg aagcaggcca atggggagga catcgatgtc acctccaatg actagggtgg 120
gc 122
<210> 263
<211> 48
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(32)
<223> a, c, u, g, unknown or other
<400> 263
acnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnguuuuaga gcuaugcu 48
<210> 264
<211> 67
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<400> 264
agcauagcaa guuaaaauaa ggctaguccg uuaucaacuu gaaaaagugg caccgagucg 60
gugcuuu 67
<210> 265
<211> 62
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<400> 265
nnnnnnnnnn nnnnnnnnnn guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cg 62
<210> 266
<211> 73
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 266
tgaatggtcc caaaacggaa gggcctgagt ccgagcagaa gaagaagttt tagagctatg 60
ctgttttgaa tgg 73
<210> 267
<211> 69
<212> DNA
<213> Homo sapiens
<400> 267
ctggtcttcc acctctctgc cctgaacacc caatctcggc ccctctcgcc accctcctgc 60
atttctgtt 69
<210> 268
<211> 138
<212> DNA
<213> Mus musculus
<400> 268
acccaagcac tgagtgccat tagctaaatg catagggtac cacccacagg tgccaggggc 60
ctttcccaaa gttcccagcc ccttctccaa cctttcctgg cccagaggct ttcccatgtg 120
tgtggctgga ccctttga 138
<210> 269
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 269
aaaaccaccc ttctctctgg c 21
<210> 270
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 270
ggagattgga gacacggaga g 21
<210> 271
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 271
ctggaaagcc aatgcctgac 20
<210> 272
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 272
ggcagcaaac tccttgtcct 20
<210> 273
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 273
gtgctttgca gaggcctacc 20
<210> 274
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 274
cctggagcgc atgcagtagt 20
<210> 275
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 275
accttctgtg tttccaccat tc 22
<210> 276
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 276
ttggggagtg cacagacttc 20
<210> 277
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
probe
<400> 277
tagctctaaa acttcttctt ctgctcggac 30
<210> 278
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
probe
<400> 278
ctagccttat tttaacttgc tatgctgttt 30
<210> 279
<211> 99
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<400> 279
nnnnnnnnnn nnnnnnnnnn guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcuuu 99
<210> 280
<211> 12
<212> DNA
<213> Homo sapiens
<400> 280
tagcgggtaa gc 12
<210> 281
<211> 12
<212> DNA
<213> Homo sapiens
<400> 281
tcggtgacat gt 12
<210> 282
<211> 12
<212> DNA
<213> Homo sapiens
<400> 282
actccccgta gg 12
<210> 283
<211> 12
<212> DNA
<213> Homo sapiens
<400> 283
actgcgtgtt aa 12
<210> 284
<211> 12
<212> DNA
<213> Homo sapiens
<400> 284
acgtcgcctg at 12
<210> 285
<211> 12
<212> DNA
<213> Homo sapiens
<400> 285
taggtcgacc ag 12
<210> 286
<211> 12
<212> DNA
<213> Homo sapiens
<400> 286
ggcgttaatg at 12
<210> 287
<211> 12
<212> DNA
<213> Homo sapiens
<400> 287
tgtcgcatgt ta 12
<210> 288
<211> 12
<212> DNA
<213> Homo sapiens
<400> 288
atggaaacgc at 12
<210> 289
<211> 12
<212> DNA
<213> Homo sapiens
<400> 289
gccgaattcc tc 12
<210> 290
<211> 12
<212> DNA
<213> Homo sapiens
<400> 290
gcatggtacg ga 12
<210> 291
<211> 12
<212> DNA
<213> Homo sapiens
<400> 291
cggtactctt ac 12
<210> 292
<211> 12
<212> DNA
<213> Homo sapiens
<400> 292
gcctgtgccg ta 12
<210> 293
<211> 12
<212> DNA
<213> Homo sapiens
<400> 293
tacggtaagt cg 12
<210> 294
<211> 12
<212> DNA
<213> Homo sapiens
<400> 294
cacgaaatta cc 12
<210> 295
<211> 12
<212> DNA
<213> Homo sapiens
<400> 295
aaccaagata cg 12
<210> 296
<211> 12
<212> DNA
<213> Homo sapiens
<400> 296
gagtcgatac gc 12
<210> 297
<211> 12
<212> DNA
<213> Homo sapiens
<400> 297
gtctcacgat cg 12
<210> 298
<211> 12
<212> DNA
<213> Homo sapiens
<400> 298
tcgtcgggtg ca 12
<210> 299
<211> 12
<212> DNA
<213> Homo sapiens
<400> 299
actccgtagt ga 12
<210> 300
<211> 12
<212> DNA
<213> Homo sapiens
<400> 300
caggacgtcc gt 12
<210> 301
<211> 12
<212> DNA
<213> Homo sapiens
<400> 301
tcgtatccct ac 12
<210> 302
<211> 12
<212> DNA
<213> Homo sapiens
<400> 302
tttcaaggcc gg 12
<210> 303
<211> 12
<212> DNA
<213> Homo sapiens
<400> 303
cgccggtgga at 12
<210> 304
<211> 12
<212> DNA
<213> Homo sapiens
<400> 304
gaacccgtcc ta 12
<210> 305
<211> 12
<212> DNA
<213> Homo sapiens
<400> 305
gattcatcag cg 12
<210> 306
<211> 12
<212> DNA
<213> Homo sapiens
<400> 306
acaccggtct tc 12
<210> 307
<211> 12
<212> DNA
<213> Homo sapiens
<400> 307
atcgtgccct aa 12
<210> 308
<211> 12
<212> DNA
<213> Homo sapiens
<400> 308
gcgtcaatgt tc 12
<210> 309
<211> 12
<212> DNA
<213> Homo sapiens
<400> 309
ctccgtatct cg 12
<210> 310
<211> 12
<212> DNA
<213> Homo sapiens
<400> 310
ccgattcctt cg 12
<210> 311
<211> 12
<212> DNA
<213> Homo sapiens
<400> 311
tgcgcctcca gt 12
<210> 312
<211> 12
<212> DNA
<213> Homo sapiens
<400> 312
taacgtcgga gc 12
<210> 313
<211> 12
<212> DNA
<213> Homo sapiens
<400> 313
aaggtcgccc at 12
<210> 314
<211> 12
<212> DNA
<213> Homo sapiens
<400> 314
gtcggggact at 12
<210> 315
<211> 12
<212> DNA
<213> Homo sapiens
<400> 315
ttcgagcgat tt 12
<210> 316
<211> 12
<212> DNA
<213> Homo sapiens
<400> 316
tgagtcgtcg ag 12
<210> 317
<211> 12
<212> DNA
<213> Homo sapiens
<400> 317
tttacgcaga gg 12
<210> 318
<211> 12
<212> DNA
<213> Homo sapiens
<400> 318
aggaagtatc gc 12
<210> 319
<211> 12
<212> DNA
<213> Homo sapiens
<400> 319
actcgatacc at 12
<210> 320
<211> 12
<212> DNA
<213> Homo sapiens
<400> 320
cgctacatag ca 12
<210> 321
<211> 12
<212> DNA
<213> Homo sapiens
<400> 321
ttcataaccg gc 12
<210> 322
<211> 12
<212> DNA
<213> Homo sapiens
<400> 322
ccaaacggtt aa 12
<210> 323
<211> 12
<212> DNA
<213> Homo sapiens
<400> 323
cgattccttc gt 12
<210> 324
<211> 12
<212> DNA
<213> Homo sapiens
<400> 324
cgtcatgaat aa 12
<210> 325
<211> 12
<212> DNA
<213> Homo sapiens
<400> 325
agtggcgatg ac 12
<210> 326
<211> 12
<212> DNA
<213> Homo sapiens
<400> 326
cccctacggc ac 12
<210> 327
<211> 12
<212> DNA
<213> Homo sapiens
<400> 327
gccaacccgc ac 12
<210> 328
<211> 12
<212> DNA
<213> Homo sapiens
<400> 328
tgggacaccg gt 12
<210> 329
<211> 12
<212> DNA
<213> Homo sapiens
<400> 329
ttgactgcgg cg 12
<210> 330
<211> 12
<212> DNA
<213> Homo sapiens
<400> 330
actatgcgta gg 12
<210> 331
<211> 12
<212> DNA
<213> Homo sapiens
<400> 331
tcacccaaag cg 12
<210> 332
<211> 12
<212> DNA
<213> Homo sapiens
<400> 332
gcaggacgtc cg 12
<210> 333
<211> 12
<212> DNA
<213> Homo sapiens
<400> 333
acaccgaaaa cg 12
<210> 334
<211> 12
<212> DNA
<213> Homo sapiens
<400> 334
cggtgtattg ag 12
<210> 335
<211> 12
<212> DNA
<213> Homo sapiens
<400> 335
cacgaggtat gc 12
<210> 336
<211> 12
<212> DNA
<213> Homo sapiens
<400> 336
taaagcgacc cg 12
<210> 337
<211> 12
<212> DNA
<213> Homo sapiens
<400> 337
cttagtcggc ca 12
<210> 338
<211> 12
<212> DNA
<213> Homo sapiens
<400> 338
cgaaaacgtg gc 12
<210> 339
<211> 12
<212> DNA
<213> Homo sapiens
<400> 339
cgtgccctga ac 12
<210> 340
<211> 12
<212> DNA
<213> Homo sapiens
<400> 340
tttaccatcg aa 12
<210> 341
<211> 12
<212> DNA
<213> Homo sapiens
<400> 341
cgtagccatg tt 12
<210> 342
<211> 12
<212> DNA
<213> Homo sapiens
<400> 342
cccaaacggt ta 12
<210> 343
<211> 12
<212> DNA
<213> Homo sapiens
<400> 343
gcgttatcag aa 12
<210> 344
<211> 12
<212> DNA
<213> Homo sapiens
<400> 344
tcgatggtaa ac 12
<210> 345
<211> 12
<212> DNA
<213> Homo sapiens
<400> 345
cgactttttg ca 12
<210> 346
<211> 12
<212> DNA
<213> Homo sapiens
<400> 346
tcgacgactc ac 12
<210> 347
<211> 12
<212> DNA
<213> Homo sapiens
<400> 347
acgcgtcaga ta 12
<210> 348
<211> 12
<212> DNA
<213> Homo sapiens
<400> 348
cgtacggcac ag 12
<210> 349
<211> 12
<212> DNA
<213> Homo sapiens
<400> 349
ctatgccgtg ca 12
<210> 350
<211> 12
<212> DNA
<213> Homo sapiens
<400> 350
cgcgtcagat at 12
<210> 351
<211> 12
<212> DNA
<213> Homo sapiens
<400> 351
aagatcggta gc 12
<210> 352
<211> 12
<212> DNA
<213> Homo sapiens
<400> 352
cttcgcaagg ag 12
<210> 353
<211> 12
<212> DNA
<213> Homo sapiens
<400> 353
gtcgtggact ac 12
<210> 354
<211> 12
<212> DNA
<213> Homo sapiens
<400> 354
ggtcgtcatc aa 12
<210> 355
<211> 12
<212> DNA
<213> Homo sapiens
<400> 355
gttaacagcg tg 12
<210> 356
<211> 12
<212> DNA
<213> Homo sapiens
<400> 356
tagctaaccg tt 12
<210> 357
<211> 12
<212> DNA
<213> Homo sapiens
<400> 357
agtaaaggcg ct 12
<210> 358
<211> 12
<212> DNA
<213> Homo sapiens
<400> 358
ggtaatttcg tg 12
<210> 359
<211> 15
<212> DNA
<213> Homo sapiens
<400> 359
cagaagaaga agggc 15
<210> 360
<211> 51
<212> DNA
<213> Homo sapiens
<400> 360
ccaatgggga ggacatcgat gtcacctcca atgactaggg tggtgggcaa c 51
<210> 361
<211> 15
<212> DNA
<213> Homo sapiens
<400> 361
ctctggccac tccct 15
<210> 362
<211> 52
<212> DNA
<213> Homo sapiens
<400> 362
acatcgatgt cacctccaat gacaagcttg ctagcggtgg gcaaccacaa ac 52
<210> 363
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (6)..(25)
<223> a, c, t, g, unknown or other
<400> 363
caccgnnnnn nnnnnnnnnn nnnnn 25
<210> 364
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (5)..(24)
<223> a, c, t, g, unknown or other
<400> 364
aaacnnnnnn nnnnnnnnnn nnnnc 25
<210> 365
<211> 54
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 365
aacaccgggt cttcgagaag acctgtttta gagctagaaa tagcaagtta aaat 54
<210> 366
<211> 54
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 366
caaaacgggt cttcgagaag acgttttaga gctatgctgt tttgaatggt ccca 54
<210> 367
<211> 4104
<212> DNA
<213> Homo sapiens
<220>
<221> CDS
<222> (1)..(4104)
<400> 367
atg gac aag aag tac agc atc ggc ctg gac atc ggc acc aac tct gtg 48
Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
ggc tgg gcc gtg atc acc gac gag tac aag gtg ccc agc aag aaa ttc 96
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
aag gtg ctg ggc aac acc gac cgg cac agc atc aag aag aac ctg atc 144
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
gga gcc ctg ctg ttc gac agc ggc gaa aca gcc gag gcc acc cgg ctg 192
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
aag aga acc gcc aga aga aga tac acc aga cgg aag aac cgg atc tgc 240
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
tat ctg caa gag atc ttc agc aac gag atg gcc aag gtg gac gac agc 288
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
ttc ttc cac aga ctg gaa gag tcc ttc ctg gtg gaa gag gat aag aag 336
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
cac gag cgg cac ccc atc ttc ggc aac atc gtg gac gag gtg gcc tac 384
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
cac gag aag tac ccc acc atc tac cac ctg aga aag aaa ctg gtg gac 432
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
agc acc gac aag gcc gac ctg cgg ctg atc tat ctg gcc ctg gcc cac 480
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
atg atc aag ttc cgg ggc cac ttc ctg atc gag ggc gac ctg aac ccc 528
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
gac aac agc gac gtg gac aag ctg ttc atc cag ctg gtg cag acc tac 576
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
aac cag ctg ttc gag gaa aac ccc atc aac gcc agc ggc gtg gac gcc 624
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
aag gcc atc ctg tct gcc aga ctg agc aag agc aga cgg ctg gaa aat 672
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
ctg atc gcc cag ctg ccc ggc gag aag aag aat ggc ctg ttc ggc aac 720
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
ctg att gcc ctg agc ctg ggc ctg acc ccc aac ttc aag agc aac ttc 768
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
gac ctg gcc gag gat gcc aaa ctg cag ctg agc aag gac acc tac gac 816
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
gac gac ctg gac aac ctg ctg gcc cag atc ggc gac cag tac gcc gac 864
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
ctg ttt ctg gcc gcc aag aac ctg tcc gac gcc atc ctg ctg agc gac 912
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
atc ctg aga gtg aac acc gag atc acc aag gcc ccc ctg agc gcc tct 960
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
atg atc aag aga tac gac gag cac cac cag gac ctg acc ctg ctg aaa 1008
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
gct ctc gtg cgg cag cag ctg cct gag aag tac aaa gag att ttc ttc 1056
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
gac cag agc aag aac ggc tac gcc ggc tac att gac ggc gga gcc agc 1104
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
cag gaa gag ttc tac aag ttc atc aag ccc atc ctg gaa aag atg gac 1152
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
ggc acc gag gaa ctg ctc gtg aag ctg aac aga gag gac ctg ctg cgg 1200
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
aag cag cgg acc ttc gac aac ggc agc atc ccc cac cag atc cac ctg 1248
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
gga gag ctg cac gcc att ctg cgg cgg cag gaa gat ttt tac cca ttc 1296
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
ctg aag gac aac cgg gaa aag atc gag aag atc ctg acc ttc cgc atc 1344
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
ccc tac tac gtg ggc cct ctg gcc agg gga aac agc aga ttc gcc tgg 1392
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
atg acc aga aag agc gag gaa acc atc acc ccc tgg aac ttc gag gaa 1440
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
gtg gtg gac aag ggc gct tcc gcc cag agc ttc atc gag cgg atg acc 1488
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
aac ttc gat aag aac ctg ccc aac gag aag gtg ctg ccc aag cac agc 1536
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
ctg ctg tac gag tac ttc acc gtg tat aac gag ctg acc aaa gtg aaa 1584
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
tac gtg acc gag gga atg aga aag ccc gcc ttc ctg agc ggc gag cag 1632
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
aaa aag gcc atc gtg gac ctg ctg ttc aag acc aac cgg aaa gtg acc 1680
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
gtg aag cag ctg aaa gag gac tac ttc aag aaa atc gag tgc ttc gac 1728
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
tcc gtg gaa atc tcc ggc gtg gaa gat cgg ttc aac gcc tcc ctg ggc 1776
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
aca tac cac gat ctg ctg aaa att atc aag gac aag gac ttc ctg gac 1824
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
aat gag gaa aac gag gac att ctg gaa gat atc gtg ctg acc ctg aca 1872
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
ctg ttt gag gac aga gag atg atc gag gaa cgg ctg aaa acc tat gcc 1920
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
cac ctg ttc gac gac aaa gtg atg aag cag ctg aag cgg cgg aga tac 1968
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
acc ggc tgg ggc agg ctg agc cgg aag ctg atc aac ggc atc cgg gac 2016
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
aag cag tcc ggc aag aca atc ctg gat ttc ctg aag tcc gac ggc ttc 2064
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
gcc aac aga aac ttc atg cag ctg atc cac gac gac agc ctg acc ttt 2112
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
aaa gag gac atc cag aaa gcc cag gtg tcc ggc cag ggc gat agc ctg 2160
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
cac gag cac att gcc aat ctg gcc ggc agc ccc gcc att aag aag ggc 2208
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
atc ctg cag aca gtg aag gtg gtg gac gag ctc gtg aaa gtg atg ggc 2256
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
cgg cac aag ccc gag aac atc gtg atc gcc atg gcc aga gag aac cag 2304
Arg His Lys Pro Glu Asn Ile Val Ile Ala Met Ala Arg Glu Asn Gln
755 760 765
acc acc cag aag gga cag aag aac agc cgc gag aga atg aag cgg atc 2352
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
gaa gag ggc atc aaa gag ctg ggc agc cag atc ctg aaa gaa cac ccc 2400
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
gtg gaa aac acc cag ctg cag aac gag aag ctg tac ctg tac tac ctg 2448
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
cag aat ggg cgg gat atg tac gtg gac cag gaa ctg gac atc aac cgg 2496
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
ctg tcc gac tac gat gtg gac gcc atc gtg cct cag agc ttt ctg aag 2544
Leu Ser Asp Tyr Asp Val Asp Ala Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
gac gac tcc atc gac gcc aag gtg ctg acc aga agc gac aag gcc cgg 2592
Asp Asp Ser Ile Asp Ala Lys Val Leu Thr Arg Ser Asp Lys Ala Arg
850 855 860
ggc aag agc gac aac gtg ccc tcc gaa gag gtc gtg aag aag atg aag 2640
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
aac tac tgg cgg cag ctg ctg aac gcc aag ctg att acc cag aga aag 2688
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
ttc gac aat ctg acc aag gcc gag aga ggc ggc ctg agc gaa ctg gat 2736
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
aag gcc ggc ttc atc aag aga cag ctg gtg gaa acc cgg cag atc aca 2784
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
aag cac gtg gca cag atc ctg gac tcc cgg atg aac act aag tac gac 2832
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
gag aat gac aag ctg atc cgg gaa gtg aaa gtg atc acc ctg aag tcc 2880
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
aag ctg gtg tcc gat ttc cgg aag gat ttc cag ttt tac aaa gtg cgc 2928
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
gag atc aac aac tac cac cac gcc cac gcc gcc tac ctg aac gcc gtc 2976
Glu Ile Asn Asn Tyr His His Ala His Ala Ala Tyr Leu Asn Ala Val
980 985 990
gtg gga acc gcc ctg atc aaa aag tac cct aag ctg gaa agc gag ttc 3024
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
gtg tac ggc gac tac aag gtg tac gac gtg cgg aag atg atc gcc 3069
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala
1010 1015 1020
aag agc gag cag gaa atc ggc aag gct acc gcc aag tac ttc ttc 3114
Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe
1025 1030 1035
tac agc aac atc atg aac ttt ttc aag acc gag att acc ctg gcc 3159
Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala
1040 1045 1050
aac ggc gag atc cgg aag cgg cct ctg atc gag aca aac ggc gaa 3204
Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu
1055 1060 1065
acc ggg gag atc gtg tgg gat aag ggc cgg gat ttt gcc acc gtg 3249
Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val
1070 1075 1080
cgg aaa gtg ctg agc atg ccc caa gtg aat atc gtg aaa aag acc 3294
Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr
1085 1090 1095
gag gtg cag aca ggc ggc ttc agc aaa gag tct atc ctg ccc aag 3339
Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys
1100 1105 1110
agg aac agc gat aag ctg atc gcc aga aag aag gac tgg gac cct 3384
Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro
1115 1120 1125
aag aag tac ggc ggc ttc gac agc ccc acc gtg gcc tat tct gtg 3429
Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val
1130 1135 1140
ctg gtg gtg gcc aaa gtg gaa aag ggc aag tcc aag aaa ctg aag 3474
Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys
1145 1150 1155
agt gtg aaa gag ctg ctg ggg atc acc atc atg gaa aga agc agc 3519
Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser
1160 1165 1170
ttc gag aag aat ccc atc gac ttt ctg gaa gcc aag ggc tac aaa 3564
Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys
1175 1180 1185
gaa gtg aaa aag gac ctg atc atc aag ctg cct aag tac tcc ctg 3609
Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1190 1195 1200
ttc gag ctg gaa aac ggc cgg aag aga atg ctg gcc tct gcc ggc 3654
Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly
1205 1210 1215
gaa ctg cag aag gga aac gaa ctg gcc ctg ccc tcc aaa tat gtg 3699
Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val
1220 1225 1230
aac ttc ctg tac ctg gcc agc cac tat gag aag ctg aag ggc tcc 3744
Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
ccc gag gat aat gag cag aaa cag ctg ttt gtg gaa cag cac aag 3789
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys
1250 1255 1260
cac tac ctg gac gag atc atc gag cag atc agc gag ttc tcc aag 3834
His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys
1265 1270 1275
aga gtg atc ctg gcc gac gct aat ctg gac aaa gtg ctg tcc gcc 3879
Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala
1280 1285 1290
tac aac aag cac cgg gat aag ccc atc aga gag cag gcc gag aat 3924
Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn
1295 1300 1305
atc atc cac ctg ttt acc ctg acc aat ctg gga gcc cct gcc gcc 3969
Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala
1310 1315 1320
ttc aag tac ttt gac acc acc atc gac cgg aag agg tac acc agc 4014
Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser
1325 1330 1335
acc aaa gag gtg ctg gac gcc acc ctg atc cac cag agc atc acc 4059
Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr
1340 1345 1350
ggc ctg tac gag aca cgg atc gac ctg tct cag ctg gga ggc gac 4104
Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp
1355 1360 1365
<210> 368
<211> 1368
<212> PRT
<213> Homo sapiens
<400> 368
Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Ala Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Asp Ala Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Ala Lys Val Leu Thr Arg Ser Asp Lys Ala Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Ala Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala
1010 1015 1020
Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe
1025 1030 1035
Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala
1040 1045 1050
Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu
1055 1060 1065
Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val
1070 1075 1080
Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr
1085 1090 1095
Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys
1100 1105 1110
Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro
1115 1120 1125
Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val
1130 1135 1140
Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys
1145 1150 1155
Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser
1160 1165 1170
Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys
1175 1180 1185
Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1190 1195 1200
Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly
1205 1210 1215
Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val
1220 1225 1230
Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys
1250 1255 1260
His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys
1265 1270 1275
Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala
1280 1285 1290
Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn
1295 1300 1305
Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala
1310 1315 1320
Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser
1325 1330 1335
Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr
1340 1345 1350
Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp
1355 1360 1365
<210> 369
<211> 26
<212> DNA
<213> Homo sapiens
<400> 369
tagggttagg ggccccaggc cggggt 26
<210> 370
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
6xHis tag
<400> 370
His His His His His His
1 5
<210> 371
<211> 57
<212> DNA
<213> Homo sapiens
<220>
<221> CDS
<222> (1)..(57)
<400> 371
ggc acc att aaa gaa aat atc att ggt gtt tcc tat gat gaa tat aga 48
Gly Thr Ile Lys Glu Asn Ile Ile Gly Val Ser Tyr Asp Glu Tyr Arg
1 5 10 15
tac aga agc 57
Tyr Arg Ser
<210> 372
<211> 19
<212> PRT
<213> Homo sapiens
<400> 372
Gly Thr Ile Lys Glu Asn Ile Ile Gly Val Ser Tyr Asp Glu Tyr Arg
1 5 10 15
Tyr Arg Ser
<210> 373
<211> 48
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> CDS
<222> (1)..(48)
<400> 373
att aaa gaa aat atc att ggc ttt gtt tcc tat gat gaa tat aga tac 48
Ile Lys Glu Asn Ile Ile Gly Phe Val Ser Tyr Asp Glu Tyr Arg Tyr
1 5 10 15
<210> 374
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 374
Ile Lys Glu Asn Ile Ile Gly Phe Val Ser Tyr Asp Glu Tyr Arg Tyr
1 5 10 15
<210> 375
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(48)
<223> a, c, t, g, unknown or other
<400> 375
ccnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnngg 50
<210> 376
<211> 46
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(44)
<223> a, c, t, g, unknown or other
<400> 376
ccnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnngg 46
<210> 377
<211> 42
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(40)
<223> a, c, t, g, unknown or other
<400> 377
ccnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn gg 42
<210> 378
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(36)
<223> a, c, t, g, unknown or other
<400> 378
ccnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnngg 38
<210> 379
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(32)
<223> a, c, t, g, unknown or other
<400> 379
ccnnnnnnnn nnnnnnnnnn nnnnnnnnnn nngg 34
<210> 380
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(28)
<223> a, c, t, g, unknown or other
<400> 380
ccnnnnnnnn nnnnnnnnnn nnnnnnnngg 30
<210> 381
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(24)
<223> a, c, t, g, unknown or other
<400> 381
ccnnnnnnnn nnnnnnnnnn nnnngg 26
<210> 382
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(20)
<223> a, c, t, g, unknown or other
<400> 382
ccnnnnnnnn nnnnnnnnnn gg 22
<210> 383
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(16)
<223> a, c, t, g, unknown or other
<400> 383
ccnnnnnnnn nnnnnngg 18
<210> 384
<211> 16
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(14)
<223> a, c, t, g, unknown or other
<400> 384
ccnnnnnnnn nnnngg 16
<210> 385
<211> 15
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(13)
<223> a, c, t, g, unknown or other
<400> 385
ccnnnnnnnn nnngg 15
<210> 386
<211> 14
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(12)
<223> a, c, t, g, unknown or other
<400> 386
ccnnnnnnnn nngg 14
<210> 387
<211> 13
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(11)
<223> a, c, t, g, unknown or other
<400> 387
ccnnnnnnnn ngg 13
<210> 388
<211> 12
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(10)
<223> a, c, t, g, unknown or other
<400> 388
ccnnnnnnnn gg 12
<210> 389
<211> 11
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(9)
<223> a, c, t, g, unknown or other
<400> 389
ccnnnnnnng g 11
<210> 390
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(8)
<223> a, c, t, g, unknown or other
<400> 390
ccnnnnnngg 10
<210> 391
<211> 12
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (3)..(10)
<223> a, c, t, g, unknown or other
<400> 391
ggnnnnnnnn cc 12
<210> 392
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 392
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aactcacatc aaccggtggc gcaggtgttt cgtcctttcc 120
acaag 125
<210> 393
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 393
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aactcacatc aaccggtggc gcaggtgttt cgtcctttcc 120
acaag 125
<210> 394
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 394
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgaggaca aagtacaaac ggcggtgttt cgtcctttcc 120
acaag 125
<210> 395
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 395
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgaggaca aagtacaaac ggcggtgttt cgtcctttcc 120
acaag 125
<210> 396
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 396
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgtggcgc attgccacga agcggtgttt cgtcctttcc 120
acaag 125
<210> 397
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 397
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aaccgagggc agagtgctgc ttgggtgttt cgtcctttcc 120
acaag 125
<210> 398
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 398
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgagtccg agcagaagaa gaaggtgttt cgtcctttcc 120
acaag 125
<210> 399
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 399
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgaggaca aagtacaaac ggcggtgttt cgtcctttcc 120
acaag 125
<210> 400
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 400
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacagcagaa gaagaagggc tccggtgttt cgtcctttcc 120
acaag 125
<210> 401
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 401
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aactcacatc aaccggtggc gcaggtgttt cgtcctttcc 120
acaag 125
<210> 402
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 402
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacccctggc ccaggtgaag gtgggtgttt cgtcctttcc 120
acaag 125
<210> 403
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 403
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aactccctcc ctggcccagg tgaggtgttt cgtcctttcc 120
acaag 125
<210> 404
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 404
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgaaccgg aggacaaagt acaggtgttt cgtcctttcc 120
acaag 125
<210> 405
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 405
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacaggtgaa ggtgtggttc cagggtgttt cgtcctttcc 120
acaag 125
<210> 406
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 406
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacggtgaag gtgtggttcc agaggtgttt cgtcctttcc 120
acaag 125
<210> 407
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 407
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgaaccgg aggacaaagt acaggtgttt cgtcctttcc 120
acaag 125
<210> 408
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 408
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacccctggc ccaggtgaag gtgggtgttt cgtcctttcc 120
acaag 125
<210> 409
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 409
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacaggtgaa ggtgtggttc cagggtgttt cgtcctttcc 120
acaag 125
<210> 410
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 410
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgaggaca aagtacaaac ggcggtgttt cgtcctttcc 120
acaag 125
<210> 411
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 411
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgggaggg aggggcacag atgggtgttt cgtcctttcc 120
acaag 125
<210> 412
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 412
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aaccaccttc acctgggcca gggggtgttt cgtcctttcc 120
acaag 125
<210> 413
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 413
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacaccctag tcattggagg tgaggtgttt cgtcctttcc 120
acaag 125
<210> 414
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 414
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aaccagagca gccactgggg cctggtgttt cgtcctttcc 120
acaag 125
<210> 415
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 415
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aaccaccttc acctgggcca gggggtgttt cgtcctttcc 120
acaag 125
<210> 416
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 416
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacccccatt ggcctgcttc gtgggtgttt cgtcctttcc 120
acaag 125
<210> 417
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 417
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacattggcc tgcttcgtgg caaggtgttt cgtcctttcc 120
acaag 125
<210> 418
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 418
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aactcctcct ccagcttctg ccgggtgttt cgtcctttcc 120
acaag 125
<210> 419
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 419
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aaccctccag cttctgccgt ttgggtgttt cgtcctttcc 120
acaag 125
<210> 420
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 420
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacattggcc tgcttcgtgg caaggtgttt cgtcctttcc 120
acaag 125
<210> 421
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 421
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgcagcaa gcagcactct gccggtgttt cgtcctttcc 120
acaag 125
<210> 422
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 422
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacttcttct tctgctcgga ctcggtgttt cgtcctttcc 120
acaag 125
<210> 423
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 423
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacaccggag gacaaagtac aaaggtgttt cgtcctttcc 120
acaag 125
<210> 424
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 424
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aactcttctt ctgctcggac tcaggtgttt cgtcctttcc 120
acaag 125
<210> 425
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 425
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgttgatg tgatgggagc cctggtgttt cgtcctttcc 120
acaag 125
<210> 426
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 426
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgggccag ggagggaggg gcaggtgttt cgtcctttcc 120
acaag 125
<210> 427
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 427
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgggaggg aggggcacag atgggtgttt cgtcctttcc 120
acaag 125
<210> 428
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 428
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacccggttc tggaaccaca cctggtgttt cgtcctttcc 120
acaag 125
<210> 429
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 429
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aactcacctg ggccagggag ggaggtgttt cgtcctttcc 120
acaag 125
<210> 430
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 430
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aactcacctg ggccagggag ggaggtgttt cgtcctttcc 120
acaag 125
<210> 431
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 431
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgttctgg aaccacacct tcaggtgttt cgtcctttcc 120
acaag 125
<210> 432
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 432
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgggaggg aggggcacag atgggtgttt cgtcctttcc 120
acaag 125
<210> 433
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 433
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgggccag ggagggaggg gcaggtgttt cgtcctttcc 120
acaag 125
<210> 434
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 434
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgttctgg aaccacacct tcaggtgttt cgtcctttcc 120
acaag 125
<210> 435
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 435
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacaggtgaa ggtgtggttc cagggtgttt cgtcctttcc 120
acaag 125
<210> 436
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 436
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgaaccgg aggacaaagt acaggtgttt cgtcctttcc 120
acaag 125
<210> 437
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 437
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aaccaaaccc acgagggcag agtggtgttt cgtcctttcc 120
acaag 125
<210> 438
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 438
aaaaaaagca ccgactcggt gccacttttt caagttgata acggactagc cttattttaa 60
cttgctattt ctagctctaa aacgagtttc tcatctgtgc cccggtgttt cgtcctttcc 120
acaag 125
<210> 439
<211> 684
<212> DNA
<213> Homo sapiens
<220>
<221> CDS
<222> (1)..(123)
<220>
<221> CDS
<222> (127)..(159)
<220>
<221> CDS
<222> (163)..(399)
<220>
<221> CDS
<222> (403)..(684)
<400> 439
aaa acc acc ctt ctc tct ggc cca ctg tgt cct ctt cct gcc ctg cca 48
Lys Thr Thr Leu Leu Ser Gly Pro Leu Cys Pro Leu Pro Ala Leu Pro
1 5 10 15
tcc cct tct gtg aat gtt aga ccc atg gga gca gct ggt cag agg gga 96
Ser Pro Ser Val Asn Val Arg Pro Met Gly Ala Ala Gly Gln Arg Gly
20 25 30
ccc cgg cct ggg gcc cct aac cct atg tag cct cag tct tcc cat cag 144
Pro Arg Pro Gly Ala Pro Asn Pro Met Pro Gln Ser Ser His Gln
35 40 45
gct ctc agc tca gcc tga gtg ttg agg ccc cag tgg ctg ctc tgg ggg 192
Ala Leu Ser Ser Ala Val Leu Arg Pro Gln Trp Leu Leu Trp Gly
50 55 60
cct cct gag ttt ctc atc tgt gcc cct ccc tcc ctg gcc cag gtg aag 240
Pro Pro Glu Phe Leu Ile Cys Ala Pro Pro Ser Leu Ala Gln Val Lys
65 70 75
gtg tgg ttc cag aac cgg agg aca aag tac aaa cgg cag aag ctg gag 288
Val Trp Phe Gln Asn Arg Arg Thr Lys Tyr Lys Arg Gln Lys Leu Glu
80 85 90
gag gaa ggg cct gag tcc gag cag aag aag aag ggc tcc cat cac atc 336
Glu Glu Gly Pro Glu Ser Glu Gln Lys Lys Lys Gly Ser His His Ile
95 100 105 110
aac cgg tgg cgc att gcc acg aag cag gcc aat ggg gag gac atc gat 384
Asn Arg Trp Arg Ile Ala Thr Lys Gln Ala Asn Gly Glu Asp Ile Asp
115 120 125
gtc acc tcc aat gac tag ggt ggg caa cca caa acc cac gag ggc aga 432
Val Thr Ser Asn Asp Gly Gly Gln Pro Gln Thr His Glu Gly Arg
130 135 140
gtg ctg ctt gct gct ggc cag gcc cct gcg tgg gcc caa gct gga ctc 480
Val Leu Leu Ala Ala Gly Gln Ala Pro Ala Trp Ala Gln Ala Gly Leu
145 150 155
tgg cca ctc cct ggc cag gct ttg ggg agg cct gga gtc atg gcc cca 528
Trp Pro Leu Pro Gly Gln Ala Leu Gly Arg Pro Gly Val Met Ala Pro
160 165 170
cag ggc ttg aag ccc ggg gcc gcc att gac aga ggg aca agc aat ggg 576
Gln Gly Leu Lys Pro Gly Ala Ala Ile Asp Arg Gly Thr Ser Asn Gly
175 180 185
ctg gct gag gcc tgg gac cac ttg gcc ttc tcc tcg gag agc ctg cct 624
Leu Ala Glu Ala Trp Asp His Leu Ala Phe Ser Ser Glu Ser Leu Pro
190 195 200 205
gcc tgg gcg ggc ccg ccc gcc acc gca gcc tcc cag ctg ctc tcc gtg 672
Ala Trp Ala Gly Pro Pro Ala Thr Ala Ala Ser Gln Leu Leu Ser Val
210 215 220
tct cca atc tcc 684
Ser Pro Ile Ser
225
<210> 440
<211> 41
<212> PRT
<213> Homo sapiens
<400> 440
Lys Thr Thr Leu Leu Ser Gly Pro Leu Cys Pro Leu Pro Ala Leu Pro
1 5 10 15
Ser Pro Ser Val Asn Val Arg Pro Met Gly Ala Ala Gly Gln Arg Gly
20 25 30
Pro Arg Pro Gly Ala Pro Asn Pro Met
35 40
<210> 441
<211> 11
<212> PRT
<213> Homo sapiens
<400> 441
Pro Gln Ser Ser His Gln Ala Leu Ser Ser Ala
1 5 10
<210> 442
<211> 79
<212> PRT
<213> Homo sapiens
<400> 442
Val Leu Arg Pro Gln Trp Leu Leu Trp Gly Pro Pro Glu Phe Leu Ile
1 5 10 15
Cys Ala Pro Pro Ser Leu Ala Gln Val Lys Val Trp Phe Gln Asn Arg
20 25 30
Arg Thr Lys Tyr Lys Arg Gln Lys Leu Glu Glu Glu Gly Pro Glu Ser
35 40 45
Glu Gln Lys Lys Lys Gly Ser His His Ile Asn Arg Trp Arg Ile Ala
50 55 60
Thr Lys Gln Ala Asn Gly Glu Asp Ile Asp Val Thr Ser Asn Asp
65 70 75
<210> 443
<211> 94
<212> PRT
<213> Homo sapiens
<400> 443
Gly Gly Gln Pro Gln Thr His Glu Gly Arg Val Leu Leu Ala Ala Gly
1 5 10 15
Gln Ala Pro Ala Trp Ala Gln Ala Gly Leu Trp Pro Leu Pro Gly Gln
20 25 30
Ala Leu Gly Arg Pro Gly Val Met Ala Pro Gln Gly Leu Lys Pro Gly
35 40 45
Ala Ala Ile Asp Arg Gly Thr Ser Asn Gly Leu Ala Glu Ala Trp Asp
50 55 60
His Leu Ala Phe Ser Ser Glu Ser Leu Pro Ala Trp Ala Gly Pro Pro
65 70 75 80
Ala Thr Ala Ala Ser Gln Leu Leu Ser Val Ser Pro Ile Ser
85 90
<---
Реферат
Настоящее изобретение относится к биотехнологии. Предложены способы модификации эукариотического организма путем манипуляции с целевой последовательностью в представляющем интерес локусе генома посредством использования системы CRISPR-Cas, а также способ лечения или подавления состояния, вызванного дефектом в целевой последовательности, и композиция, содержащая компоненты вышеупомянутой системы. Данное изобретение обеспечивает возможность изменения геномных локусов эукариотической клетки за счет использования нуклеазы Cas9, слитой с одной или несколькими последовательностями ядерной локализации (NLS). 6 н. и 15 з.п. ф-лы, 73 ил., 35 табл., 37 пр.

Комментарии