Локусы функциональности fad2 и соответствующие специфичные к участку-мишени связывающие белки, способные индуцировать направленные разрывы - RU2656159C2
Код документа: RU2656159C2
Чертежи


























































































































































































































Описание
ПЕРЕКРЕСТНАЯ ССЫЛКА НА РОДСТВЕННЫЕ ЗАЯВКИ
По настоящей заявке испрашивается приоритет по временной патентной заявке США № 61/697886, поданной 7 сентября 2012 года, содержание которой включено в настоящее описание в качестве ссылки в полном объеме.
ОБЛАСТЬ ИЗОБРЕТЕНИЯ
Настоящее изобретение относится, главным образом, к композициям и способам для применения в технологии рекомбинантных растений (например, для получения трансгенного растения). Более конкретно, настоящее изобретения относится к клеткам растений и к растениям, включающим локусы в их геномах, которые можно использовать для сайт-специфического внесения любой представляющей интерес нуклеиновой кислоты.
УРОВЕНЬ ТЕХНИКИ
Многие растения генетически трансформируют экзогенными нуклеиновыми кислотами (например, трансгенами) для внесения желаемых признаков, например, для повышения сельскохозяйственной ценности. Примеры повышения сельскохозяйственной ценности, которое может быть достигнуто с помощью генетической трансформации, включат: улучшение питательных качеств, увеличение урожая, устойчивость к паразитам или заболеваниям, устойчивость к засухе и стрессовым факторам, улучшение садоводческих качеств (например, улучшение пигментации и/или роста), устойчивость к гербицидам, продуцирование полезных для промышленности соединений и/или материалов в растении и/или продуцирование фармацевтических веществ. Внесение клонированных генов в клетки растений и восстановление стабильных фертильных трансгенных растений можно использовать для осуществления генетической модификации растения, стабильной на протяжении множества поколений, и, тем самым, для получения возможности создания способами генной инженерии сельскохозяйственной культуры.
В способах генетической трансформации и получения трансгенного растения экзогенную ДНК, как правило, случайным образом вносят в ядерную или пластидную ДНК эукариотической клетки растений, после чего проводят выделение клеток, содержащих встроенную экзогенную ДНК, и последующую регенерацию стабильно трансформированного растения. Трансгенные растения, как правило, получают с помощью технологии опосредуемой Agrobacterium трансформации. Успех этих способов способствовал разработке других способов внесения представляющей интерес молекулы нуклеиновой кислоты в геном растения, таких как опосредуемый PEG захват ДНК в протопласты, бомбардировка микроснарядами и опосредуемая кремниевой нитью трансформация.
Однако во всех из этих способов трансформации растений экзогенные нуклеиновые кислоты, встраивающиеся в геном растения, встраиваются в геном клетки растения случайным образом и в непрогнозируемом числе копий. Terada et al. (2002) Nat Biotechnol 20(10):1030; Terada et al. (2007) Plant Physiol 144(2):846; D'Halluin et al. (2008) Plant Biotechnology J. 6(1):93. Например, трансгены часто встраиваются в форме повторов последовательности либо целого трансгена, либо его частей. Такая картина комплексного встраивания часто неблагоприятно влияет на уровень экспрессии встроенной нуклеиновой кислоты (например, посредством разрушения транскрибируемой РНК с помощью механизмов посттрансляционного сайленсинга генов, или вследствие индукции метилирования встроенной ДНК). Также на уровень экспрессии встроенной нуклеиновой кислоты часто влияет расположение участка встраивания. Более того, встраивание экзогенной ДНК может оказывать эффект разрушения на область генома, где происходит встраивание, и, тем самым, может влиять и нарушать нормальную функцию этой области-мишени, вызывая нежелательные побочные эффекты. Комбинация факторов, включая указанные выше, приводит к широкому варьированию уровня экспрессии трансгена или экзогенной ДНК (и общего агрономического качества) между различными клетками растений и линиями растений, даже теми из них, которые созданы одними и теми же способами. Поскольку встраивание является случайным, практикующий специалист не может контролировать эти эффекты, когда он или она пытается получить новое растение с желаемыми характеристиками.
Указанные выше соображения указывают на то, что, когда исследуют эффекты внесения конкретной нуклеиновой кислоты в растение, необходимо получить и проанализировать большое количество линий трансгенных растений, чтобы достигнуть значимых результатов. Аналогично, при получении трансгенного растения, содержащего конкретную встроенную нуклеиновую кислоту, чтобы получить трансгенное растение с желаемым фенотипом, должна быть создана большая группа независимо полученных линий трансгенных растений для возможности селекции линии растений с оптимальной экспрессией нуклеиновой кислоты и с минимальными побочными эффектами на общий фенотип и функциональность трансгенного растения или с их отсутствием. Эти практические соображения приобретают дополнительную важность в случае трансгенных растений, созданных путем встраивания множества экзогенных нуклеиновых кислот (т.е. стэкинга генов). В таких растениях явления, такие как посттрансляционный сайленсинг генов, могут усиливаться.
В попытках контроля встраивания трансгена в растения было разработано несколько способов. См., например, Kumar and Fladung (2001) Trends Plant Sci. 6:155-9. В основе этих способов лежит основанное на гомологичной рекомбинации встраивание трансгена, которое успешно использовали как в прокариотах, так и в низших эукариотах. Paszkowski et al. (1988) EMBO J. 7:4021-6. Однако до недавнего времени в растениях преобладающий механизм встраивания трансгенов был основан на незаконной рекомбинации, которая вовлекает малую гомологию между рекомбинирующими цепями ДНК. Основной проблемой в этом случае, таким образом, является обнаружение и селективное получение редких событий гомологичной рекомбинации, которые маскируются значительно более эффективными событиями встраивания вследствие незаконной рекомбинации. Более того, даже если осуществлено селективное получение и обнаружение заданных событий гомологичной рекомбинации, событие должно быть нацелено в желаемую область генома хозяина для достижения максимальной пользы этой стратегии.
Например, предполагаемой пользой направленной генетической трансформации является уменьшение вариабельности экспрессии трансгена от события к событию, по сравнению с событиями трансформации, которые достигнуты случайным встраиванием. Следующей предполагаемой пользой является значительное снижение количества событий, требуемых для скрининга внесенных нуклеиновых кислот, сортировки конструкций трансформации и достижения событий, которые приводят к желаемым общим характеристикам полученного трансгенного растения. Критическим фактором, требуемым для реализации этой пользы, является идентификация конкретных положений в геноме, где функциональность трансгена является постоянной и, если возможно, где неблагоприятные эффекты растения-хозяина устранены или минимизированы.
Недавно были описаны способы и композиции для направленного расщепления геномной ДНК. Такие события направленного расщепления можно использовать, например, для индукции направленного мутагенеза, для индукции направленных делеций в последовательностях ДНК клеток, и для облегчения направленной рекомбинации и встраивания в заданном хромосомном локусе. См., например, Umov et al. (2010) Nature 435(7042):646-51; публикации патентов США 20030232410; 20050208489; 20050026157; 20050064474; 20060188987; 20090263900; 20090117617; 20100047805; 20110207221; 20110301073; 2011089775; 20110239315; 20110145940 и международную публикацию WO 2007/014275, содержание которых включено в качестве ссылок в полном объеме для всех целей. Расщепление можно проводить с использованием специфических нуклеаз, таких как модифицированные способами инженерии нуклеазы с цинковыми пальцами (ZFN), подобные активаторам транскрипции эффекторные нуклеазы (TALEN), или с использованием системы CRISPR/Cas с модифицированной способами инженерии crRNA/tracr РНК ("единая РНК-гид") для обеспечения специфического расщепления. В публикации патента США № 20080182332 описано применение неканонических нуклеаз с цинковыми пальцами (ZFN) для направленной модификации геномов растений; в публикации патента США № 20090205083 описана опосредуемая ZFN направленная модификация локуса EPSPS растений; в публикации патента США № 20100199389 описана направленная модификация локуса Zp15 растений и в публикации патента США № 20110167521 описана направленная модификация генов растений, вовлеченных в биосинтез жирных кислот. Кроме того, в Moehle et al. (2007) Proc. Natl Acad, Sci. USA 104(9):3055-3060 описано использование сконструированных ZFN для направленного добавления гена в конкретный локус. В публикации патента США 20110041195 описаны способы получения гомозиготных диплоидных организмов.
Однако остается потребность в композициях и способах для модификации и/или модулирования экспрессии генов FAD2 в растениях, включая получение растений с направленными вставками желаемых трансгенов в локус FAD2.
КРАТКОЕ ОПИСАНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
Настоящее изобретение относится к композициям и способам для модулирования экспрессии генов FAD2 (например, в растениях, водорослях и грибах) и к применению этих локусов в качестве участков для направленного введения представляющей интерес последовательности нуклеиновой кислоты (например, экзогенной последовательности нуклеиновой кислоты) в клетку-хозяина. В некоторых вариантах осуществления клетка-хозяин может содержать один или несколько геномов с одной или несколькими последовательностями FAD2 (например, гомологи или паралоги), любые или все из которых могут быть селективно модифицированы и/или разрушены. В конкретных примерах настоящее изобретение относится к генам FAD2A, FAD2A'', FAD2C и FAD2C', а также к соответствующим гомологам или паралогам, в Brassica napus (т.е. в линии B. napus DH12075) и к их применению в качестве локусов для направленного встраивания представляющей интерес последовательности нуклеиновой кислоты. Как описано в настоящем описании, хотя гены FAD2 вовлечены в биосинтез жирных кислот в хозяине, их модификация или разрушение (например, вследствие встраивания экзогенной нуклеиновой кислоты в кодирующую последовательность FAD2) неожиданно могут не иметь неблагоприятных эффектов или могут иметь минимальные неблагоприятные эффекты на полученный организм хозяина.
Также в настоящем описании описано применение одного или нескольких конкретных локусов FAD2 в тандеме с полипептидом, способным осуществлять расщепление и/или встраивание конкретных последовательностей нуклеиновой кислоты в локусы FAD2. Примеры применения локусов FAD2 в тандеме с полипептидом, способным осуществлять расщепление и/или встраивание локусов FAD2, включают полипептид, выбранный из группы, состоящей из белков с цинковыми пальцами, мегануклеаз, TAL-доменов, TALEN, РНК-направляемых CRISPR-Cas9, рекомбиназ, лейциновых молний, CRISPr/Cas и других полипептидов, известных в данной области. Конкретные примеры включают химерный ("слитый") белок, содержащий полипептид с сайт-специфическим ДНК-связывающим доменом и полипептид с доменом расщепления (например, нуклеаза), такой как белок ZFN, содержащий полипептид с цинковыми пальцами и полипептид нуклеазы Fok1. Например, в настоящем описании описана демонстрация эффективности и специфичности in vitro и in vivo конкретных ZFN, сконструированных для связывания и индукции двухцепочечных разрывов в FAD2A, FAD2A', FAD2C, FAD2C' и в их комбинациях без расщепления соответствующих гомологов или паралогов. В некоторых вариантах осуществления конкретные локусы FAD2 можно использовать с любым из указанных выше полипептидов для обеспечения сайт-специфического встраивания представляющей интерес нуклеиновой кислоты, которая впоследствии экспрессируется в хозяине, оказывая минимальное неблагоприятное влияние на агрономическую функциональность хозяина.
В некоторых аспектах в рамках настоящего изобретения описаны полипептиды, содержащие ДНК-связывающий домен, который специфически связывается с геном FAD2. В некоторых вариантах осуществления такой полипептид также может содержать нуклеазный домен (домен расщепления) или половинный домен (например, ZFN, рекомбиназа, транспозаза или хоминг-эндонуклеаза, включая хоминг-эндонуклеазу с модифицированным ДНК-связывающим доменом, TAL-доменами, TALEN, РНК-направляемыми CRISP-Cas9), и/или лигазный домен, так что полипептид может индуцировать направленный двухцепочечный разрыв и/или способствовать рекомбинации представляющей интерес нуклеиновой кислоты в области разрыва. В конкретных вариантах осуществления ДНК-связывающий домен, который нацелен на локус FAD2, может представлять собой расщепляющий ДНК функциональный домен. В некоторых вариантах осуществления указанные выше полипептиды можно использовать для внесения экзогенной нуклеиновой кислоты в геном организма хозяина (например, виды растений или животных) в одном или нескольких локусах FAD2. В определенных вариантах осуществления ДНК-связывающие домены содержат белок с цинковыми пальцами, который имеет один или несколько цинковых пальцев (например, 2, 3, 4, 5, 6, 7, 8, 9 или более цинковых пальцев) и который может быть модифицированным способами инженерии (не является встречающимся в природе) так, чтобы он связывался с любой последовательностью в гене FAD2. Любой из белков с цинковыми пальцами, описанных в настоящем описании, может связываться с участком-мишенью в кодирующей последовательности гена-мишени или в соседних последовательностях (например, промоторы или другие элементы экспрессии). В определенных вариантах осуществления белок с цинковыми пальцами связывается с участком-мишенью в гене FAD2, например, как показано в таблице 5. Распознаваемые спиральные области иллюстративных связывающих FAD2 цинковых пальцев представлены в таблице 4. Один или несколько связывающих доменов в виде цинковых пальцев, являющихся компонентами белка с цинковыми пальцами, могут представлять собой канонические (C2H2) цинковые пальцы или неканонические (например, C3H) цинковые пальцы (например, N-концевой и/или C-концевой цинковый палец может представлять собой неканонический палец).
Также в настоящем описании описаны способы разрушения или редактирования гена FAD2. Кроме того в настоящем описании описаны генетически модифицированные организмы-хозяева (например, трансгенные растения), получаемые способами согласно вариантам осуществления изобретения. В конкретных примерах трансгенный организм, получаемый способом согласно одному варианту осуществления изобретения, может представлять собой, но не ограничивается ими, водоросль, гриб, однодольное растение, двудольное растение и т.д.
Гены FAD2, описанные в настоящем описании, могут включать гены, встречающиеся в любом растении, водоросли или грибе, которые могут иметь один или несколько генов FAD2.
Указанные выше и другие признаки станут более понятными из представленного ниже подробного описания нескольких вариантов осуществления, которое предоставлено с отсылкой на прилагаемые чертежи.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
На фиг. 1, панели с A по E, представлено выравнивание последовательностей гена FAD2 (SEQ ID NO: 5-8), проведенное с использованием AlignX®.
На фиг. 2 представлена схема, на которой показано филогенетическое дерево последовательностей генов FAD2, полученное с использованием Jalview v 2.3 на основе расстояний, полученных методом ближайшего связывания.
На фиг. 3 представлена карта плазмиды pDAB104010, которая является репрезентативной экспрессирующей кассетой для нуклеаз с цинковыми пальцами. План этой конструкции сходен с другими экспрессирующими кассетами для ZFN, где домены цинковых пальцев 24828 и 24829 заменены альтернативными доменами цинковых пальцев, которые описаны выше.
На фиг. 4 представлен иллюстративный график с множеством кривых, демонстрирующий количество считанных последовательностей с делециями в заданном участке ZFN на 10000 считанных последовательностей. На оси X на графике представлено количество удаленных оснований, на оси Y представлено количество считанных последовательностей, и на оси Z представлено кодируемое цветом обозначение образца, как описано справа на графике. Конкретный представленный пример представляет собой пример локуса 1 семейства генов FAD2, которое содержит 3 участка-мишени для ZFN: A, B и C, с четырьмя представителями семейства генов и двумя контрольными трансфекциями, оцениваемыми в качестве контрольных образцов A и B. Линии, приведенные сверху вниз (от A-контроль_FADA' сверху легенды до C_образец_FAD2C снизу легенды) представлены на графике от наиболее близкой к обозначенной оси X (A_контроль_FADA') до наиболее удаленной от обозначенной оси X (C_образец_FAD2C).
На фиг. 5, панели A и B, представлено нацеливание ZFN на гены FAD2. На фиг. 5A представлен график, на котором представлены данные нацеливания ZFN на локус 4 семейства генов FAD2. Локус содержит два участка ZFN и две необходимые контрольных трансфекции. На фиг. 5B представлен конкретный контекст последовательности (SEQ ID NO: 471-480), окружающий участок-мишень для ZFN, где обозначены FAD2A и C, содержащие тринуклеотидные повторы C, T и G, что приводит к наблюдаемому увеличению однонуклеотидных делеций при секвенировании локусов FAD2A и C.
На фиг. 6 представлена карта плазмиды pDA000130.
На фиг. 7 представлена карта плазмиды pDAS000031.
На фиг. 8 представлена схема, демонстрирующая участки связывания праймеров и зонда на трансгене-мишени для анализа в целях оценки числа копий трансгена.
На фиг. 9 представлен файл Sequencher, демонстрирующий домен ZFN, распознающий ДНК FAD2A (bc12075_Fad2a-r272a2 и bc12075_Fad2a-278a2) и участки связывания специфических праймеров для ZFN (FAD2A.UnE.F1 и FAD2A.UnE.R1) и эндогенных праймеров (FAD2A/2C.RB.UnE.F1 и FAD2A/2C.RB.UnE.R1).
На фиг. 10 представлена схема, демонстрирующая участки связывания праймеров на эндогенной и трансгенной мишени, используемых для обнаружения встраивания трансгенов FAD2A с помощью полной HDR.
На фиг. 11 представлена схема, где участки рестрикции эндонуклеазой pn1 встречаются в полностью отредактированном локусе FAD2A, и где связываются зонды для саузерн-блоттинга FAD2a 5', hph и FAD2A 3'.
На фиг. 12 представлено расположение и размер фрагментов Kpn1, зондов FAD2A 5', hph, FAD2A 3' и ожидаемый исход анализа с использованием саузерн-блоттинга для растений, в которых произошло встраивание ETIP в локус FAD2A посредством HDR.
На фиг. 13 представлены репрезентативные данные оценки числа копий с помощью q-ПЦР. В левом столбце представлены данные, полученные для известного растения T0 с одной случайной вставкой, которое использовали в качестве калибровочного образца, относительно которого все другие образцы "нормализовывали". В правом столбце представлено известное трансгенное растение T0 с 5 вставками трансгенов. Число встраиваемых копий для обоих растений определяли с использованием саузерн-анализа. В остальных столбцах предоставлены оценки числа копий для предполагаемых трансгенных растений. Столбцы обозначены как; контроль с 1 копией, 310420, 311819, 311821, 311822, 311823, 311824, 311827, 312524, 312525, 312526, 312527, 312529, 312530, 312532, 313810, 313811, 313905, 313941, 313942, 313944 и контроль с 5 копиями. Столбцы можно использовать для определения оцененного числа копий каждого трансгенного растения. При использовании программного обеспечения для оценки числа копий, растения дикого типа, нетрансформированные растения и контроли только с плазмидой не дают результата о числе копий, поскольку они не обладают Cq как для мишени hph, так и для мишени HMG 7/7.
На фиг. 14 представлена карта плазмиды pDAS000129.
На фиг. 15 представлена схема встраивания pDAS000129 в локус FAD2A.
На фиг. 16 представлена карта плазмиды pDAS000097.
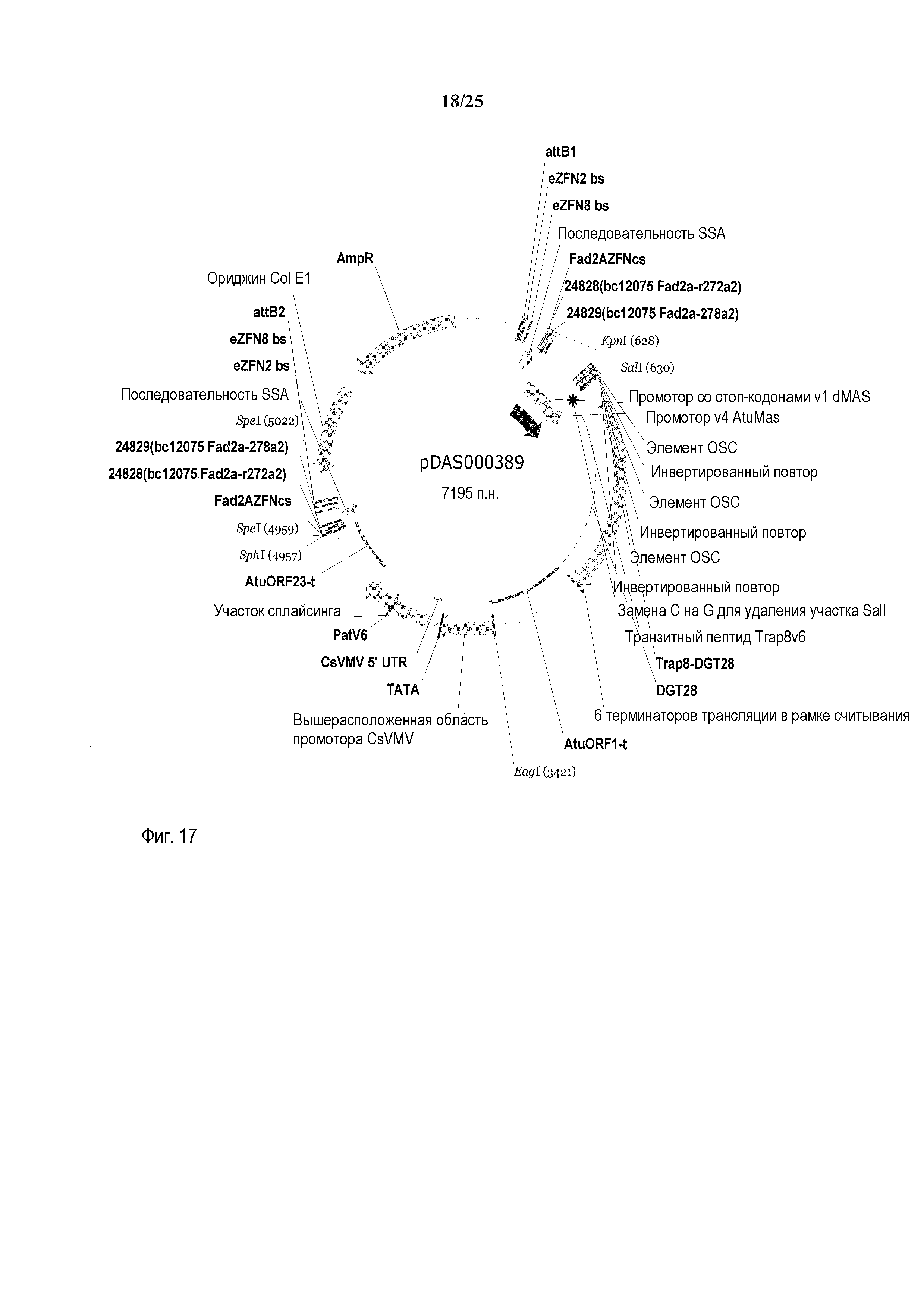
На фиг. 17 представлена карта плазмиды pDAS000389.
На фиг. 18 представлена карта плазмиды pDAS000391.
На фиг. 19 представлена карта плазмиды pDAS000392.
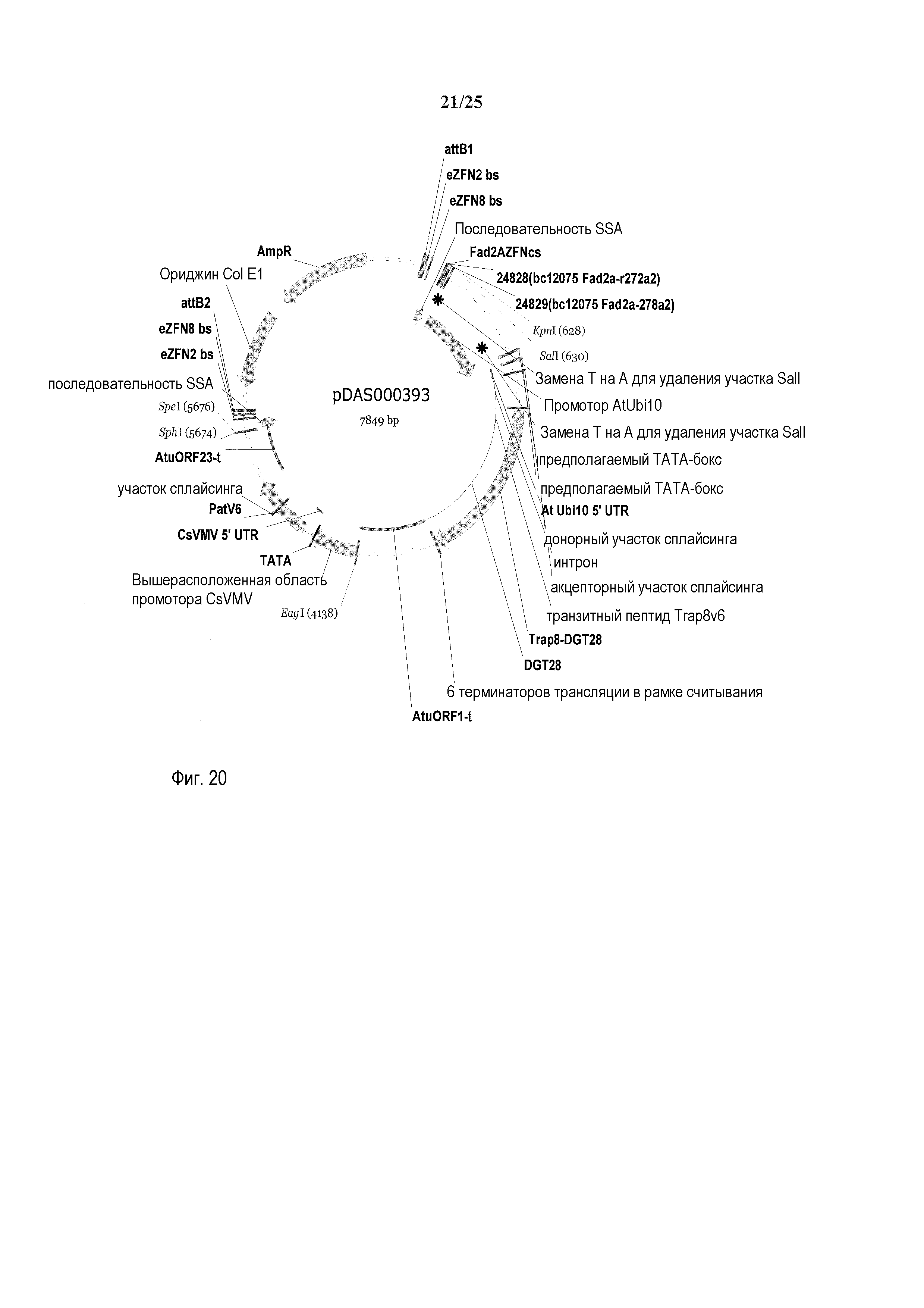
На фиг. 20 представлена карта плазмиды pDAS000393.
На фиг. 21 представлена карта плазмиды pDAS000394.
На фиг. 22 представлена карта плазмиды pDAS000395.
На фиг. 23 представлена карта плазмиды pDAS000396.
На фиг. 24 представлена карта плазмиды pDAS000397.
ПОСЛЕДОВАТЕЛЬНОСТИ
Последовательности нуклеиновых кислот представлены с использованием стандартных буквенных сокращений для нуклеотидных оснований, как определено в 37 C.F.R. § 1.822. Показана только одна цепь каждой последовательности нуклеиновой кислоты, однако понятно, что комплементарная цепь охватывается при любом указании на представленную цепь.
ПОДРОБНОЕ ОПИСАНИЕ
I. Обзор нескольких вариантов осуществления
Варианты осуществления изобретения демонстрируют подход для направленного встраивания экзогенных нуклеиновых кислот (например, трансгенов) в геном хозяина без значительного неблагоприятного влияния на другие фенотипы хозяина, помимо тех из них, на которые воздействует встроенная нуклеиновая кислота. Некоторые варианты осуществления можно использовать для "стэкинга" множества нуклеиновых кислот в одном геноме хозяина. Такой подход требует разработки и внедрения четырех взаимосвязанных технологий: технологии нацеливания, позволяющие внесение двухцепочечных разрывов в конкретные положения в геномной ДНК (см., например, Puchta et al. (1993) Nucleic Acids Res. 21:5034-40; Siebert and Puchta (2002) Plant Cell 14:1121-31; D'Halluin et al. (2008) Plant Biotechnol. J. 6(1):93-102; Cai et al. (2009) Plant Mol. Biol. 69(6):699-709; ShuHa et al. (2009) Nature 459(7245):437-41); Shan et al. (2103) Nature Biotechnol. 31:686-680; Le et al. (2013) Nature Biotechnol 31: 688-691; Nekrasov et al. (2013) Nature Biotechnol. 31: 691-693, Ainely et al. (2013) Plant Biotechnol. J. (On Line 19 Aug); технологии доставки, позволяющие доставку оптимизированной экзогенной (донорной) нуклеиновой кислоты (Bibikova et al. (2003) Science 300(5620):764); технологии встраивания, вовлекающие модификацию генов хозяина (расположенных в каскадах либо гомологичной рекомбинации, либо NHEJ), чтобы увеличить частоты HDR или NHEJ для направленного встраивания донорной ДНК; аналитические инструменты для увеличения количества и охарактеризации событий направленного встраивания; и определенные положения в геноме желаемого хозяина ("локусы функциональности"), которые генетически хорошо определены и которые поддерживают стабильную экспрессию генов на протяжении поколений без значительного неблагоприятного влияния на трансформированный организм хозяина. Также см. патентные публикации США 20030232410; 20050208489; 20050026157; 20050064474; 20060188987; 20090263900; 20090117617; 20100047805; 20110207221; 20110301073; 2011089775; 20110239315; 20110145940; 20080182332; 20090205083; 20100199389; 20110167521. Например, в растениях локус функциональности представляет собой локус, в котором отрицательное влияние на агрономические или качественные свойства трансгенного растения, когда трансген встроен в локус, являются незначительными или отсутствуют.
В вариантах осуществления, описанных в настоящем описании, используется неожиданное открытие, что гены FAD2 растений являются локусами функциональности для направленного встраивания экзогенных нуклеиновых кислот (например, ген(ы); некодирующие последовательности ДНК, такие как сконструированные способами инженерии подушки для встраивания (ELP) (заявка США 12/011735) и сконструированная способами инженерии платформа для встраивания трансгена (ETIP) (находящаяся на рассмотрении заявка США № 61/697882); и элемент(ы) трансформации растений). Характер повсеместной распространенности локусов FAD2 в растениях и данные о том, что изменение или нокаут FAD2 в каноле, кукурузе, подсолнечнике, пшенице, хлопке и сое не приводит к ухудшению агрономических или качественных признаков, идентифицируют локусы FAD2 в качестве широкого класса локусов функциональности среди коммерчески значимых видов растений.
В некоторых вариантах осуществления используется сайт-специфическое расщепление двухцепочечной ДНК в локусе FAD2, например, путем доставки и экспрессии специфичного к участку-мишени белка, распознающего и расщепляющего ДНК. В конкретных примерах такой специфичный к FAD2 распознающий и расщепляющий ДНК белок может представлять собой, например, но не ограничиваясь этим, ZFN; TALEN; РНК-направляемые CRISPR-Cas9, рекомбиназу (например, рекомбиназы Cre, Hin, RecA, Tre и FLP); мегануклеазу и созданный способами инженерии белок, происходящий из любого из вышеуказанных белков или их эквивалентов. На расщепление можно влиять с использованием системы CRISPR Cas с модифицированной способами инженерии crRNA-tracr РНК ("единая РНК-гид") для направления на специфическое расщепление. В некоторых вариантах осуществления такой двухцепочечный разрыв можно репарировать путем встраивания донорной нуклеиновой кислоты в участок расщепления в локусе функциональности FAD2, например, с помощью направляемой гомологией репарации (HDR) или связывания негомологичных концов (NHEJ).
Настоящее описание иллюстрирует применимость локусов FAD2 в качестве локусов функциональности, например, путем описания локуса FAD2A, 2A', 2C или 2C в каноле (Brassica napus), и соответствующих специфичных к FAD2 ZFN, которые можно использовать для встраивания экзогенной нуклеиновой кислоты в локус FAD2A, 2A', 2C или 2C.
Варианты осуществления настоящего изобретения направлены на множество нерешенных проблем в данной области. Например, селективность подхода направленного встраивания, описанного в настоящем описании, может снижать или устранять необходимость в многократных испытаниях в полевых условиях, требуемых для устранения нежелательных трансгенных событий, которые являются дорогостоящими вследствие вовлеченных ресурсов и обременительных нормативных требований в этой области. Более того, подходы направленного встраивания ДНК, описанные в настоящем описании, могут быть особенно полезными в процессе стэкинга трансгенов.
Хотя нативную нуклеотидную последовательность в эндогенном локусе FAD2 можно использовать для прямого нацеливания представляющей интерес нуклеиновой кислоты, в некоторых вариантах осуществления нуклеиновая кислота может быть сначала нацелена по меньшей мере на один локус FAD2 хозяина так, чтобы встраивание других молекул представляющих интерес нуклеиновой кислоты облегчалось. В других примерах можно использовать нуклеотидные последовательности, которые не являются гомологичными нативным последовательностям организма хозяина (например, по существу случайным образом полученные последовательности нуклеиновой кислоты), которые фланкируют участок распознавания ДНК (например, участки распознавания цинковыми пальцами).
II. Термины
Как используют в настоящей заявке, включая формулу изобретения, термины в единственном числе и формы единственного числа включают множественное число упоминаемых объектов, если контекст явно не указывает на иное. Таким образом, например, указание на "растение" также относится к множеству растений. Более того, в зависимости от контекста, использование термина "растение" также может относиться к генетически сходному или идентичному потомству этого растения. Аналогично, термин "нуклеиновая кислота" может относиться к множеству копий молекулы нуклеиновой кислоты. Аналогично, термин "зонд" может относиться к множеству сходных или идентичных молекул зондов.
Числовые диапазоны включают числа, определяющие диапазон, и прямо включают каждую целочисленную и нецелочисленную долю в определенном диапазоне. Если нет иных указаний, все технические и научные термины, используемые в настоящем описании, обладают тем же значением, которое обычно подразумевает специалист в данной области.
Чтобы упросить обзор различных вариантов осуществления, описанных в настоящем описании, предоставляется следующее пояснение конкретных терминов:
Выделенный: "выделенный" биологический компонент (такой как нуклеиновая кислота или белок) по существу отделен, получен отдельно или очищен от других биологических компонентов в клетке организма, в котором компонент встречается в природе (т.е. другие хромосомные и внехромосомные ДНК и РНК и белки), с осуществлением химического или функционального изменения компонента (например, нуклеиновая кислота может быть выделена из хромосомы посредством разрушения химических связей, соединяющих нуклеиновую кислоту с остальной ДНК в хромосоме). Молекулы нуклеиновой кислоты и белки, которые являются "выделенными", включают молекулы нуклеиновой кислоты и белки, очищенные стандартными способами очистки. Также термин охватывает нуклеиновые кислоты и белки, полученные посредством рекомбинантной экспрессии в клетке-хозяине, а также химически синтезированные молекулы нуклеиновой кислоты, белки и пептиды.
Скрещивание: как используют в рамках изобретения в отношении растений, термин "скрещивание" или "скрещенный" относится к слиянию гамет посредством опыления с образованием потомства (например, клеток, семян и растений). Этот термин охватывает как половое скрещивание (т.е. опыление одного растения другим), так и селфинг (т.е. самоопыление, например, с использованием пыльцы и яйцеклетки одного и того же растения).
Обратное скрещивание: способы обратного скрещивания можно использовать для введения последовательности нуклеиновой кислоты в растения. Этот способ широко используется в течение десятилетий для введения новых признаков в растения. N. Jensen, Ed. Plant Breeding Methodology, John Wiley & Sons, Inc., 1988. В типичном протоколе обратного скрещивания исходный представляющий интерес сорт (рекуррентный родитель) скрещивают со вторым сортом (нерекуррентный родитель), который содержит представляющую интерес последовательность нуклеиновой кислоты, подлежащую переносу. Затем потомство, полученное с помощью этого скрещивания, вновь скрещивают с рекуррентным родителем, и процесс повторяют до тех пор, пока не получат растение, где по существу все из желаемых морфологических и физиологических характеристик рекуррентного родительского растения воспроизведены в преобразованном растении, в дополнение к перенесенной последовательности нуклеиновой кислоты из нерекуррентного родителя.
Интрогрессия: Как используют в рамках изобретения, термин "интрогрессия" относится к переносу аллеля (или модифицированного аллеля, содержащего экзогенную нуклеиновую кислоту) в генетический фон конкретного локуса. В некоторых вариантах осуществления интрогрессия конкретной формы аллеля в локусе может происходить путем передачи аллеля по меньшей мере одному потомку посредством полового скрещивания между двумя родителями одного и того же вида, где по меньшей мере один из родителей имеет конкретную форму аллеля в его геноме. Потомство, содержащее конкретную форму аллеля, можно повторно скрещивать с линией, имеющей желаемый генетический фон. Потомство обратного скрещивания можно выбирать по конкретной форме аллеля, чтобы получить новый сорт, где конкретная форма аллеля является фиксированной в генетическом фоне. В некоторых вариантах осуществления интргрессия конкретной формы аллеля может происходить посредством рекомбинации двух донорных геномов (например, в слитом протопласте), где по меньшей мере один из донорных геномов имеет конкретную форму аллеля в его геноме. Интрогрессия может вовлекать передачу конкретной формы аллеля, которая может представлять собой, например, но не ограничиваясь этим, разрушенный или модифицированный аллель, трансген; PTU и ELP.
Зародышевая плазма: как используют в рамках изобретения, термин "зародышевая плазма" относится к генетическому материалу отдельного растения, группы растений (например, линия, сорт и семейство растений), и клона, происходящего из растения или группы растений. Зародышевая плазма может быть частью организма или клетки, или она может быть отдельной (например, выделенной) от организма или клетки. Как правило, зародышевая плазма обеспечивает генетический материал с конкретным молекулярным составом, который является основной наследуемых качеств растения. Как используют в рамках изобретения, "зародышевая плазма" относится к клеткам конкретного растения; семени; ткани конкретного растения (например, ткань, из которой можно выращивать новые растения); и происходящим не из семян частям конкретного растения (например, листья, стебель, пыльца и клетки). Как используют в рамках изобретения, термин "зародышевая плазма" является синонимом "генетического материала" и его можно использовать для обозначения семени (или другого растительного материала), посредством которого растение может быть размножено. "Банк зародышевой плазмы" может относиться к организованной коллекции различных семян или другого генетического материала (где каждый генотип является уникально идентифицированным), из которого можно культивировать известный сорт и из которого можно получать новый сорт.
Ген: как используют в рамках изобретения, термин "ген" (или "генетический элемент") может относиться к наследуемой геномной последовательности ДНК, имеющей функциональную значимость. Ген может представлять собой нативную нуклеиновую кислоту или нуклеиновую кислоту, которая была встроена в геном. Термин "ген" также можно использовать для обозначения, например, но не ограничиваясь этим, кДНК и/или мРНК, кодируемой наследуемой последовательностью геномной ДНК.
Молекула нуклеиновой кислоты: как используют в рамках изобретения, термин "молекула нуклеиновой кислоты" может относиться к полимерной форме нуклеотидов (т.е. рибонуклеотиды, дезоксирибонуклеотиды и/или модифицированная форма любого из вышеуказанных). Этот термин включает как смысловую, так и антисмысловую цепи РНК, кДНК, геномной ДНК, и их синтетические формы и смешанные полимеры. Термин включает любую топологическую конформацию, включая одноцепочечную, двухцепочечную, частично дуплексную, триплексную, шпилечную, кольцевую и замкнутую конформации. Молекула нуклеиновой кислоты может включать любой или оба типа из встречающихся в природе и модифицированных нуклеотидов. Такие нуклеотиды могут быть связаны вместе встречающимся в природе и/или не встречающимися в природе нуклеотидными связями.
Молекулы нуклеиновой кислоты могут быть модифицированы химически или биохимически, или они могут содержать дериватизированные нуклеотидные основания, как будет хорошо понятно специалистам в данной области. Такие модификации включают, например, но не ограничиваясь ими: метки; метилирование; замена одного или нескольких встречающихся в природе нуклеотидов аналогом; и межнуклеотидные модификации (например, незаряженные связи, например, метилфосфонаты, фосфотриэфиры, фосфорамидаты и карбаматы; заряженные связи, например, фосфоротиоаты и фосфородитиоаты; выступающие группы, например, пептиды; интеркаляторы, например, акридиновые и псораленовые хелаторы; алкилаторы; и модифицированные связи, например, альфа-аномерные нуклеиновые кислоты).
Экзогенный: "экзогенная" молекула представляет собой молекулу, которая не является нативной для определенной системы (например, зародышевая плазма, сорт, элитный сорт и/или растение) в отношении нуклеотидной последовательности и/или положения полинуклеотида в геноме (т.е. локуса) (и в отношении аминокислотной последовательности и/или клеточной локализации полипептида). В вариантах осуществления экзогенные или гетерологичные полинуклеотиды или полипептиды могут представлять собой молекулы, которые искусственным образом предоставлены биологической системе (например, клетка растения, ген растения, конкретный вид или сорт растений, и/или хромосома растения) и они не являются нативными для конкретной биологической системы. Таким образом, обозначение нуклеиновой кислоты как "экзогенная" может указывать на то, что нуклеиновая кислота произошла из источника, отличного от природного источника, или оно может указывать на то, что нуклеиновая кислота имеет неприродную конфигурацию, генетическое положение или расположение элементов.
Напротив, например, "нативная" или "эндогенная" нуклеиновая кислота представляет собой нуклеиновую кислоту (например, ген), которая не содержит элемент нуклеиновой кислоты, отличный от элементов, обычно присутствующих в хромосоме или другом генетическом материале, в котором нуклеиновая кислота в норме встречается в природе. Транскрипт эндогенного гена кодируется нуклеотидной последовательностью в ее природном хромосомном локусе и не предоставляется искусственным образом клетке.
Функционально связанный: первая последовательность нуклеиновой кислоты функционально связана со второй последовательностью нуклеиновой кислоты, когда первая последовательность нуклеиновой кислоты находится в функциональной взаимосвязи со второй последовательностью нуклеиновой кислоты. Например, промотор функционально связан с кодирующей последовательностью, когда промотор влияет на транскрипцию или экспрессию кодирующей последовательности. При рекомбинантной продукции функционально связанные последовательности нуклеиновой кислоты, как правило, являются непрерывными, и, когда необходимо связать две кодирующих белок области, находятся в одной рамке считывания. Однако элементы не должны быть соседними, чтобы быть функционально связанными.
Промотор: промотор представляет собой область в ДНК, которая обычно расположена выше (в направлении 5'-области) нуклеиновой кислоты, которая усиливает транскрипцию нуклеиновой кислоты. Промоторы обеспечивают надлежащую активацию или регрессию нуклеиновой кислоты(кислот), с которой они функционально связаны. Промотор содержит определенные последовательности, которые распознаются факторами транскрипции. Эти факторы связываются с промоторными последовательностями ДНК и обеспечивают привлечение РНК-полимеразы - фермента, который синтезирует РНК с кодирующей области нуклеиновой кислоты. Трансформированный: вектор "трансформирует или "трансдуцирует" клетку, когда он переносит молекулы нуклеиновой кислоты в клетку. Клетка является "трансформированной" молекулой нуклеиновой кислоты, когда молекула нуклеиновой кислоты становится стабильно реплицируемой клеткой, либо путем включения молекулы нуклеиновой кислоты в геном клетки, либо путем эписомальной репликации. Как используют в рамках изобретения, термин "трансформация" охватывает все способы, посредством которых молекулу нуклеиновой кислоты можно вводить в клетку. Примеры включают, но не ограничиваются ими: трансфекцию вирусными векторами; трансформацию плазмидными векторами; электропорацию (Fromm et al. (1986) Nature 319:791-3); липофекцию (Feigner et al. (1987) Proc. Natl. Acad. Sci. USA 84:7413-7); микроинъекцию (Mueller et al. (1978) Cell 15:579-85); опосредуемый Agrobacterium перенос (Fraley et al. (1983) Proc. Natl. Acad. Sci. USA 80:4803-7); прямой захват ДНК и бомбардировку микроснарядами (Klein et al. (1987) Nature 327:70).
Введенный: как используют в рамках изобретения, термин "введенный" при указании на перенос экзогенной нуклеиновой кислоты в клетку относится к включению нуклеиновой кислоты в клетку с использованием любой методологии, доступной в данной области. Этот термин охватывает способы введения нуклеиновых кислот, включающие, например, но не ограничивающиеся ими, трансфекцию; трансформацию и трансдукцию.
Трансген: как используют в рамках изобретения, термин "трансген" относится к экзогенной нуклеиновой кислоте, кодирующей представляющую интерес последовательность. Например, трансген может кодировать полезное для промышленности или фармацевтики соединение или продукт экспрессии, который обеспечивает желаемый сельскохозяйственный признак (например, устойчивость к гербицидам и устойчивость к паразитам). В следующем примере трансген может представлять собой антисмысловую нуклеиновую кислоту, где экспрессия антисмысловой нуклеиновой кислоты ингибирует экспрессию последовательности нуклеиновой кислоты-мишени. Трансген может содержать регуляторные последовательности, функционально связанные с трансгеном (например, промотор). В некоторых вариантах осуществления представляющая интерес молекула нуклеиновой кислоты, подлежащая введению с помощью сайт-специфического нацеливания в локус FAD2, представляет собой трансген. Однако в других вариантах осуществления представляющая интерес молекула нуклеиновой кислоты может представлять собой PTU, ELP, ΕΤIΡ или эндогенную последовательность нуклеиновой кислоты (например, когда являются желательными дополнительные экзогенные геномные копии эндогенной последовательности нуклеиновой кислоты).
Элементы также могут включать ДНК, которая кодирует структурную РНК, такую как кшРНК (shRNA). Такая РНК может модифицировать экзогенные или эндогенные гены, включая, но не ограничиваясь ими, влияние на перенос или сообщение устойчивости к гербицидам.
Рекомбинантный: как используют в рамках изобретения, термин "рекомбинантный" относится к материалу (например, нуклеиновая кислота, ген, полинуклеотид и/или полипептид), который изменен посредством вмешательства человека. Например, расположение частей или элементов рекомбинантной молекулы может не быть нативным расположением и/или первичная последовательность рекомбинантной молекулы может быть изменена относительно ее нативной последовательности, например, для оптимизации ее экспрессии и/или активности. Материал может быть изменен так, чтобы получить рекомбинантный материал, находящийся в его природном окружении или состоянии, или извлеченный из его природного окружения или состояния. В качестве одного примера, открытая рамка считывания нуклеиновой кислоты является рекомбинантной, если нуклеотидная последовательность открытой рамки считывания удалена из ее природного контекста и клонирована в любой тип искусственной нуклеиновой кислоты (например, вектор). Протоколы и реагенты для получения рекомбинантных молекул (например, рекомбинантных нуклеиновых кислот) распространены и являются стандартными в данной области. Термин "рекомбинантный" также может относиться в настоящем описании к клетке или организму, которые содержат рекомбинантный материал (например, растение и/или клетка растения, которые содержат рекомбинантную нуклеиновую кислоту). В некоторых примерах рекомбинантный организм представляет собой трансгенный организм.
Вектор: как используют в рамках изобретения, термин "вектор" относится к полинуклеотиду или другой молекуле, которые способны переносить по меньшей мере один сегмент нуклеиновой кислоты в клетку. Вектор необязательно может содержать компоненты/элементы, которые опосредуют поддержание вектора и обеспечивают его предполагаемое применение (например, последовательности, необходимые для репликации, гены, сообщающие устойчивость к лекарственному средству или антибиотику, участок множественного клонирования и/или функционально связанные промоторные/энхансерные элементы, которые обеспечивают экспрессию клонированного гена). Векторы могут происходить, например, из плазмид, бактериофагов или вирусов растений или животных. "Клонирующий вектор", "челночный вектор" или "субклонирующий вектор", как правило, включает функционально связанные элементы, облегчающие стадии клонирования или субклонирования (например, участок множественного клонирования, содержащий множество участков эндонуклеаз рестрикции).
Экспрессирующий вектор: термин "экспрессирующий вектор", как используют в рамках изобретения, относится к вектору, содержащему функционально связанные полинуклеотидные последовательности, которые могут способствовать экспрессии кодирующей последовательности в конкретном организме-хозяине. Например, бактериальный экспрессирующий вектор может способствовать экспрессии кодирующей последовательности в бактерии. Аналогично, экспрессирующий вектор растений может способствовать экспрессии кодирующей последовательности в клетке растения. Полинуклеотидные последовательности, которые способствуют экспрессии в прокариотах, могут включать, например, но не ограничиваясь ими, промотор; оператор и участок связывания рибосом. Эукариотические экспрессирующие векторы (например, экспрессирующий вектор растений) содержат промоторы, энхансеры, сигналы терминации и полиаденилирования (и другие последовательности), которые, как правило, отличаются от последовательностей, используемых в прокариотических экспрессирующих векторах.
Идентичность последовательностей: термин "идентичность последовательностей" или "идентичность", как используют в рамках изобретения в контексте двух последовательностей нуклеиновых кислот или полипептидов, относится к остаткам в двух последовательностях, которые являются одинаковыми при выравнивании на максимальное соответствие на протяжении указанного окна сравнения. Величину идентичности последовательности можно определять путем сравнения двух оптимально выровненных последовательностей (например, последовательности нуклеиновых кислот и аминокислотные последовательности) на протяжении окна сравнения, где часть последовательности в окне сравнения может содержать вставки или делеции (т.е. пропуски) по сравнению с эталонной последовательностью (которая не содержит вставок или делеций) для оптимального выравнивания двух последовательностей. Идентичность последовательностей вычисляют в качестве процента путем определения количества положений, в которых идентичный нуклеотидный или аминокислотный остаток встречается в обеих последовательностях с получением количества совпавших положений, деления количества совпавших положений на общее количество положений в окне сравнения, и умножения результата на 100 с получением процента идентичности последовательностей.
Способы выравнивания последовательностей для сравнения хорошо известны в данной области. Различные программы и алгоритмы выравнивания описаны, например, в: Smith and Waterman (1981) Adv. Appl. Math. 2:482; Needleman and Wunsch (1970) J. Mol. Biol. 48:443; Pearson and Lipman (1988) Proc. Natl. Acad. Sci. U.S.A. 85:2444; Higgins and Sharp (1988) Gene 73:237-44; Higgins and Sharp (1989) CABIOS 5:151-3; Corpet et al. (1988) Nucleic Acids Res. 16:10881-90; Huang et al. (1992) Comp. Appl. Biosci. 8:155-65; Pearson et al. (1994) Methods Mol. Biol. 24:307-31; Tatiana et al. (1999) FEMS Microbiol. Lett. 174:247-50. Подробное описание способов выравнивания последовательностей и вычисления гомологии может быть найдено, например, в Altschul et al. (1990) J. Mol. Biol. 215:403-10.
Для выравнивания последовательностей можно использовать Basic Local Alignment Search Tool (BLAST™; Altschul et al. (1990)) от National Center for Biotechnology Information (NCBI) и он доступен из нескольких источников, в том числе National Center for Biotechnology Information (Bethesda, MD), и через Интернет, для применения совместно с несколькими программами анализа последовательностей. Описание того, как определять идентичность последовательностей с использованием этой программы, доступно через Интернет в разделе "help" для BLAST™. Для сравнений последовательностей нуклеиновых кислот можно использовать функцию "Blast 2 sequences" программы BLAST™ (Blastn) с параметрами по умолчанию. Последовательности нуклеиновых кислот с более высоким сходством с эталонными последовательностями будут демонстрировать более высокую процентную идентичность при оценке этим способом.
Как используют в рамках изобретения, термин "по существу идентичный" может относиться к нуклеотидным последовательностям, которые являются более чем на 80% идентичными. Например, по существу идентичная нуклеотидная последовательность может быть по меньшей мере на 85%, по меньшей мере на 86%; по меньшей мере на 87%; по меньшей мере на 88%; по меньшей мере на 89%; по меньшей мере на 90%; по меньшей мере на 91%; по меньшей мере на 92%; по меньшей мере на 93%; по меньшей мере на 94%; по меньшей мере на 95%; по меньшей мере на 96%; по меньшей мере на 97%; по меньшей мере на 98%; по меньшей мере на 99%; или по меньшей мере на 99,5% идентичной эталонной последовательности.
Локус: как используют в рамках изобретения, термин "локус" относится к положению в геноме, которое соответствует поддающейся измерению характеристике (например, признаку). В некоторых вариантах осуществления представляющий особый интерес локус представляет собой геномное положение гена FAD2, где разрушение гена снижает или устраняет экспрессию мРНК, транскрибируемой с гена дикого типа. Локус может можно определять с помощью зонда, который гибридизуется с уникальной последовательностью, содержащейся в локусе, в ходе либо саузерн-гибридизации, либо ПЦР.
Маркер: как используют в рамках изобретения, "маркер" относится к гену или нуклеотидной последовательности, которые можно использовать для идентификации растений, которые, вероятно, имеют конкретный аллель и/или проявляют конкретный признак или фенотип. Маркер может быть описан как варьирование в данном геномном локусе. Генетический маркер может представлять собой короткую последовательность ДНК, такую как последовательность, окружающая изменение одной пары оснований (однонуклеотидный полиморфизм или "SNP"), или длинную последовательность, например, микросателлитный/простой повтор последовательности ("SSR"). "Маркерный аллель" относится к версии маркера, которая присутствует в конкретном растении. Термин "маркер", как используют в рамках изобретения" может относиться к клонированному сегменту хромосомной ДНК растения (например, сегмент, содержащий локус FAD2 или модифицированный и/или разрушенный локус FAD2), и также или альтернативно может относиться к молекуле ДНК, которая комплементарна клонированному сегменту хромосомной ДНК растения. Как известно специалистам в данной области, процесс получения дополнительной непрерывной нуклеотидной последовательности для включения в маркер можно повторять практически неограниченно (ограничиваясь только длиной хромосомы), тем самым идентифицируя дополнительные маркеры вдоль хромосомы. Любые и все из описанных выше типов маркеров можно использовать в некоторых вариантах осуществления настоящего изобретения.
В некоторых вариантах осуществления присутствие трансгена или маркера (которые характеризуются последовательностью-"мишенью") в зародышевой плазме можно выявлять с использованием зонда нуклеиновой кислоты; например, олигонуклеотида. Зонд может представлять собой молекулу ДНК или молекулу РНК. Олигонуклеотидный зонд можно получать синтетически или путем клонирования. Пригодные клонирующие векторы хорошо известны специалистам в данной области. Зонды РНК можно синтезировать способами, известными в данной области, например, с использованием матричной молекулы ДНК.
Олигонуклеотидный зонд может быть меченым или немеченым. Существует широкое множество способов мечения молекул нуклеиновой кислоты, включая, например, но не ограничиваясь ими: радиоактивное мечение посредством ник-трансляции; использование случайных праймеров; достраивание терминальной дезокситрансферазой и т.п., где используемые нуклеотиды являются мечеными, например, радиоактивным32P. Другие метки, которые можно использовать, включают, например, но не ограничиваясь ими: флуорофоры; ферменты; субстраты ферментов; кофакторы ферментов и ингибиторы ферментов. Альтернативно использование метки, которая обеспечивает поддающийся выявлению сигнал, сама по себе или вместе с другими реакционно-способными агентами, можно заменять лигандами, с которыми связываются рецепторы, где рецепторы являются мечеными (например, указанными выше метками), чтобы обеспечить поддающиеся обнаружению сигналы, либо самостоятельно, либо вместе с другими реагентами. См., например, Leary et al. (1983) Proc. Natl. Acad. Sci. USA 80:4045-9.
Зонд может представлять собой точную копию маркера, подлежащего обнаружению. Зонд также может представлять собой молекулу нуклеиновой кислоты, содержащую или состоящую из, нуклеотидной последовательности, которая по существу идентична клонированному сегменту хромосомной ДНК, содержащему трансген или маркер, подлежащие обнаружению. Кроме того, зонд может содержать дополнительные последовательности нуклеиновой кислоты, например, промоторы; сигналы транскрипции и/или векторные последовательности.
Зонд может содержать всю или часть нуклеотидной последовательности-мишени и дополнительную смежную нуклеотидную последовательность из генома растений. Это обозначается в настоящем описании как "смежный зонд". Дополнительная смежная нуклеотидная последовательность обозначается как "вышерасположенная" или "нижерасположенная" относительно исходной мишени, в зависимости от того, находится ли смежная нуклеотидная последовательность из хромосомы растений с 5'-стороны или с 3'-стороны исходного маркера, как хорошо понятно. Зонд также может содержать нуклеотидную последовательность, которая не является смежной с последовательностью исходной мишени; этот зонд обозначают в настоящем описании как "несмежный зонд". Последовательность несмежного зонда может быть расположена достаточно близко к последовательности исходной мишени на хромосоме, так чтобы несмежный зонд был сцеплен с исходным маркером или трансгеном.
В некоторых вариантах осуществления зонд представляет собой молекулу нуклеиновой кислоты, которая "специфически гибридизуется" или "специфически комплементарна" точной копии мишени, подлежащей обнаружению. "Специфически гибридизующийся" и "специфически комплементарный" представляют собой термины, которые указывают на достаточную степень комплементарности, чтобы между молекулой нуклеиновой кислоты и мишенью происходило стабильное и специфическое связывание. Молекула нуклеиновой кислоты не должна быть на 100% комплементарной ее последовательности-мишени, чтобы быть специфически гибридизующейся. Молекула нуклеиновой кислоты является специфически гибридизующейся, когда существует достаточная степень комплементарности, чтобы предотвращалось неспецифическое связывание нуклеиновой кислоты с не являющимися мишенью последовательностями в условиях, когда является желательным специфическое связывание, например, в жестких условиях гибридизации.
Условия гибридизации, обеспечивающие конкретные степени жесткости, варьируют в зависимости от типа выбранного способа гибридизации и композиции и длины гибридизующихся последовательностей нуклеиновых кислот. Как правило, жесткость гибридизации определяется температурой гибридизации и ионной силой (особенно концентрацией Na+ и/или Mg++) буфера для гибридизации, хотя также на жесткость влияет количество раз промывания. Вычисления, касающиеся условий гибридизации, требуемых для достижения конкретных степеней жесткости, известны средним специалистам в данной области, и рассмотрены, например, в Sambrook et al. (ed.) Molecular Cloning: A Laboratory Manual, 2nd ed., vol. 1-3, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, 1989, chapters 9 and 11; и Hames and Higgins (eds.) Nucleic Acid Hybridization, IRL Press, Oxford, 1985. Дальнейшая детальная инструкция и руководство в отношении гибридизации нуклеиновых кислот могут быть найдены, например, в Tijssen, "Overview of principles of hybridization and the strategy of nucleic acid probe assays", Laboratory Techniques in Biochemistry and Molecular Biology-Hybridization with Nucleic Acid Probes, Part I, Chapter 2, Elsevier, NY, 1993; и Ausubel et al., Eds., Current Protocols in Molecular Biology, Chapter 2, Greene Publishing and Wiley-Interscience, NY, 1995.
Как используют в рамках изобретения, "жесткие условия" охватывают условия, при которых гибридизация происходит, только если существует менее 25% несоответствий гибридизующейся молекулой и ДНК-мишенью. "Жесткие условия" включают другие конкретные уровни жесткости. Таким образом, как используют в рамках изобретения, условия "умеренной жесткости" представляют собой условия, в которых молекулы с более чем 25% несоответствием последовательностей не гибридизуются; условия средней жесткости представляют собой условия, в которых молекулы с более чем 15% несоответствий не гибридизуются и условия "высокой жесткости" представляют собой условия, в которых последовательности с более чем 10% несоответствий не гибридизуются. Условия "очень высокой жесткости" представляют собой условия, при которых последовательности с более чем 6% несоответствий не гибридизуются.
В конкретных вариантах осуществления жесткие условия представляют собой гибридизацию при 65°C в 6x солевом натрий-цитратном (SSC) буфере, 5x растворе Денхардта, 0,5% SDS, и 100 мкг расщепленной ДНК семенников лосося, за которой следуют последовательные промывания в течение 15-30 при 65°C в 2x буфере SSC и 0,5% SDS, а затем в 1x буфере SSC и 0,5% SDS, и, наконец, в 0,2x буфере SSC и 0,5% SDS.
(Не)равновесие по сцеплению: как используют в рамках изобретения, термин "равновесие по сцеплению" относится к ситуации, где маркер и вторая нуклеиновая кислота (например, трансген, PTU и второй маркер) независимо сегрегируют; т.е. маркер и вторая нуклеиновая кислота распределяются среди потомков случайным образом. Нуклеиновые кислоты, которые демонстрируют равновесие по сцеплению, считают несцепленными (независимо от того, лежат ли они на одной и той же хромосоме). Как используют в рамках изобретения, термин "неравновесие по сцеплению" относится к ситуации, где маркер и вторая нуклеиновая кислота сегрегируют неслучайным образом; т.е. маркеры имеют частоту рекомбинации менее 50% (и, таким образом, по определению разделены менее чем на 50 cM на одной и той же группе сцепления). В некоторых примерах нуклеиновые кислоты, которые демонстрируют неравновесие по сцеплению, считают сцепленными.
Сцепленный, прочно сцепленный и очень прочно сцепленный: как используют в рамках изобретения, сцепление между маркером и второй нуклеиновой кислотой (например, трансген, PTU и второй маркер) может относиться к явлению, когда нуклеиновые кислоты на хромосоме демонстрируют поддающуюся измерению вероятность передачи вместе в следующее поколение. Таким образом, сцепление одного маркера со второй нуклеиновой кислотой можно измерять и/или выражать в качестве частоты рекомбинаций. Чем ближе два гена или маркера находятся друг к другу, тем ближе эта вероятность к "1". Таким образом, термин "сцепленный" может относиться к одному или нескольким генам или маркерам, которые передаются вместе с геном с вероятностью более 0,5 (которая ожидается вследствие независимого расхождения, где маркеры/гены расположены на различных хромосомах). Когда присутствие гена (например, трансгена) приводит к фенотипу у особи, маркеры, которые сцеплены с геном, можно называть сцепленными с фенотипом. Таким образом, термин "сцепленный" может относиться к взаимосвязи между маркером и геном, или между маркером и фенотипом.
Относительное генетическое расстояние (определяемое по частотам кроссинговера и измеряемое в сантиморганах (cM)), как правило, пропорционально физическому расстоянию (измеряемому в парах оснований), на которое два сцепленных маркера или гена отделены друг от друга на хромосоме. Один сантиморган определяют как расстояние между двумя генетическими маркерами, которое демонстрирует частоту рекомбинации 1% (т.е. событие кроссинговера происходит между двумя маркерами один раз каждые 100 делений клеток). Как правило, чем ближе один маркер к другому маркеру или гену (независимо от того, измеряют ли расстояние между ними в значениях генетического расстояния или физического расстояния), тем более прочно они сцеплены. Поскольку хромосомное расстояние приблизительно пропорционально частоте событий рекомбинации между признаками, существует приблизительное физическое расстояние, которое коррелирует с частотой рекомбинации. Эта корреляция является общедоступной и ее можно без труда определить для основных культурных растений (Helentjaris and Burr (eds.) (1989) Development and Application of Molecular Markers to Problems in Plant Genetics. Cold Spring Harbor Laboratory, Cold Spring Harbor, NY; Gresshoff (ed.) (1994) Plant Genome Analysis. CRC Press, Boca Raton, FL; Lander et al. (1987) Genomics 1: 174-81; Tanksley et al. (1 88) "Molecular mapping of plant chromosomes", Chromosome Structure and Function. Gustafson and Appels (eds.) Plenum Press, NY, pp. 157-73) и многих других организмов. Например, 1 cM соответствует приблизительно 2,5-3,0 т.п.н. в дрожжах, приблизительно 140 т.п.н. в арабидопсисе, приблизительно 400 т.п.н. в подсолнечнике, и приблизительно 350 т.п.н. в эвкалипте.
Термин "сцепленный" в рамках настоящего изобретения может относиться к одной или нескольким нуклеиновым кислотам, которые демонстрируют частоту рекомбинации менее 50% (т.е. менее 50 cM). Например, "сцепленные" нуклеиновые кислоты могут рекомбинировать с частотой приблизительно 45% или менее, приблизительно 40% или менее, приблизительно 35% или менее, приблизительно 30% или менее, приблизительно 25% или менее, приблизительно 20% или менее, приблизительно 15% или менее, и приблизительно 10% или менее. Физические расстояния между такими нуклеиновыми кислотами на одной и той же хромосоме (ожидается, что нуклеиновые кислоты на различных хромосомах находится в равновесии по сцеплению), которые соответствуют указанным выше частотам рекомбинации, зависят от генома хозяина и могут быть без труда вычислены, как указано выше.
Как используют в рамках изобретения, термин "прочно сцепленный" может относиться к одной или нескольким нуклеиновым кислотам, которые демонстрируют частоту рекомбинации приблизительно 20% или менее (т.е. приблизительно 20 cM или менее). Например, "прочно сцепленные" нуклеиновые кислоты могут рекомбинировать с частотой 22% или менее, приблизительно 18% или менее, приблизительно 16% или менее, приблизительно 14% или менее, приблизительно 12% или менее, приблизительно 10% или менее, приблизительно 8% или менее, приблизительно 6% или менее, приблизительно 4% или менее и приблизительно 2% или менее.
Как используют в рамках изобретения, термин "чрезвычайно прочно сцепленный" может относиться к одной или нескольким нуклеиновым кислотам, которые демонстрируют частоту рекомбинации приблизительно 10% или менее (т.е. приблизительно 10 cM или менее). Например, "чрезвычайно прочно сцепленные" нуклеиновые кислоты могут рекомбинировать с частотой 11% или менее, приблизительно 9% или менее, приблизительно 8% или менее, приблизительно 7% или менее, приблизительно 6% или менее, приблизительно 5% или менее, приблизительно 4% или менее, приблизительно 3% или менее, приблизительно 2% или менее и приблизительно 1% или менее.
Чем ближе конкретная нуклеиновая кислота находится к гену, который кодирует полипептид, который вносит вклад в конкретный фенотип (независимо от того, измеряется ли расстояние в значениях генетического или физического расстояния), тем более прочно сцеплена конкретная нуклеиновая кислота с фенотипом. Ввиду вышесказанного, будет понятно, что нуклеиновые кислоты, сцепленные с конкретным геном или фенотипом, включают нуклеиновые кислоты, которые прочно сцеплены и которые чрезвычайно прочно сцеплены, с геном или фенотипом. В некоторых вариантах осуществления чем ближе конкретная нуклеиновая кислота находится к локусу FAD2 (например, модифицированному или разорванному локусу FAD2), независимо от того, измеряется ли расстояние в значениях генетического или физического расстояния, тем более прочно сцеплена конкретная нуклеиновая кислота с фенотипом любого признака, сообщаемого экзогенной нуклеиновой кислотой, встроенной в локус FAD2 (или с фенотипом FAD2 дикого типа в случае немодифицированного локуса). Таким образом, генетические маркеры, которые сцеплены, прочно сцеплены и/или чрезвычайно прочно сцеплены с локусом FAD2, содержащим встроенную экзогенную нуклеиновую кислоту, могут быть полезными в программе MAS для идентификации организмов (например, растений и сортов растений), содержащих встроенную нуклеиновую кислоту, для идентификации организмов, имеющих фенотип, сообщаемый встроенной нуклеиновой кислотой, и для выведения такой встроенной нуклеиновой кислоты и/или фенотипа, сообщаемого встроенной нуклеиновой кислотой, в других совместимых организмах.
Разведение с помощью маркера: как используют в рамках изобретения, термин "разведение с помощью маркера" может относиться к подходу для прямого разведения растений по одному или нескольким признаку(ам) (например, полигенному признаку). В современной практике растениеводы-селекционеры предпринимают попытки идентифицировать легко выявляемые признаки, такие как цвет цветков, внешний вид оболочки семян или варианты изоферментов, которые сцеплены с агрономически желаемым признаком. Затем растениеводы-селекционеры наблюдают за агрономическим признаком в сегрегирующих популяциях для разведения путем наблюдения за сегрегацией легко выявляемого признака. Однако для применения в разведении растений доступно очень мало этих взаимосвязей сцепления между представляющими интерес признаками и легко выявляемыми признаками. В некоторых вариантах осуществления изобретения разведение с помощью маркера включает идентификацию одного или нескольких генетических маркеров (например, SNP, изофермент и/или маркеры SSR), которые сцеплены с локусом FAD2, в который встроена экзогенная нуклеиновая кислота, обеспечивающая представляющий интерес признак, и наблюдение за представляющим интерес признаком в сегрегирущей популяции для разведения путем наблюдения за сегрегацией одного или нескольких генетических маркеров. В некоторых примерах сегрегацию одного или нескольких генетических маркеров можно определять с использованием зонда для одного или нескольких генетических маркеров путем анализа генетического образца из растения-потомка в отношении присутствия одного или нескольких генетических маркеров. Разведение с помощью маркера обеспечивает времяемкий и экономичный процесс усовершенствования сортов растений.
Признак или фенотип: термины "признак" и "фенотип" используют в настоящем описании взаимозаменяемо. Для целей настоящего описания, представляющие особый интерес признаки включают агрономически важные признаки, которые могут экспрессироваться, например, в сколькохозяйственной культуре, и продукцию продуктов экспрессии трансгенов после события направленного встраивания. Термин "молекулярный фенотип" может относиться к фенотипу, который поддается обнаружению на уровне популяции (одной или нескольких) молекул. В некоторых примерах молекулярный фенотип может обнаруживаться на молекулярном уровне. Поддающиеся обнаружению молекулы фенотипа могут представлять собой нуклеиновые кислоты (например, геномная ДНК или РНК); белки и/или метаболиты. Например, молекулярный фенотип может представлять собой профиль экспрессии одного или нескольких продуктов генов (например, на конкретной стадии развития растения или в ответ на условия или стрессовые воздействия окружающей среды).
Локус количественного признака: признаки, которые постоянно варьируют вследствие генетических (аддитивные, доминантные и эпистатические) и внешних влияний обычно называют "количественными признаками". Количественные признаки можно отличить от "качественных" или "дискретных" признаков, исходя из двух факторов: внешние влияния на экспрессию генов, которые обеспечивают непрерывное распределение фенотипов, и сложный профиль сегрегации, обеспечиваемый полигенным наследованием. Идентификация одной или нескольких областей генома, сцепленных с экспрессией количественного признака, определяет такие области как локусы количественных признаков ("QTL").
Растение: как используют в рамках изобретения, термин "растение" может относиться к целому растению, культуре клеток или тканей, полученной из растений, и/или любой части любого из вышеуказанных. Таким образом, термин "растение" охватывает, например, но не ограничиваясь ими, целые растения; компоненты и/или органы растений (например, листья, стебли и корни); ткань растений; семена и клетку растений. Клетка растений может представлять собой, например, но не ограничиваясь ими, клетку в растении и/или клетку растения, клетку, выделенную из растения и клетку, полученную посредством культивирования клетки, выделенной из растения.
"Трансгенное растение" представляет собой растение, содержащее по меньшей мере в одной из его клеток экзогенный полинуклеотид. Термин "трансгенный" используют в настоящем описании для обозначения любой клетки, клеточной линии, каллюса, ткани, части растения или растения, генотип которых изменен присутствием экзогенной нуклеиновой кислоты. Таким образом, этот термин охватывает трансгенные организмы и клетки, которые исходно изменены так, чтобы они содержали экзогенный полинуклеотид, и организмы и клетки, полученные посредством скрещиваний и бесполого размножения исходного трансгенного организма или клетки. Термин "трансгенный", как используют в рамках изобретения, не охватывает геномные (хромосомные или внехромосомные) изменения, внесенные общепринятыми способами разведения растений (например, скрещивание только нетрансгенных организмов) или посредством встречающихся в природе событий (например, случайное перекрестное оплодотворение, нерекомбинантная вирусная инфекция, нерекомбинантная бактериальная трансформация, нерекомбинантная транспозиция и спонтанная мутация).
"Линия", "сорт" или "штамм" растений представляет собой группу отдельных растений, имеющих одно происхождение. Растения линии, как правило, являются инбредными до некоторой степени и, как правило, являются гомозиготными и гомогенными в большинстве генетических локусов (например, локус FAD2). "Сублиния" может относиться к инбредной подгруппе потомков общего предшественника, которые генетически отличаются от других аналогично инбредных подгрупп, происходящих от того же предшественника. В некоторых вариантах осуществления "сублинию" можно получать посредством инбридинга семян из отдельного растения подсолнечника, выбранных из поколения F3-F5 до тех пор, пока остаточные сегрегирующие локусы не будут гомозиготными среди большинства или всех локусов.
"Связывающий белок" представляет собой белок, который способен связываться с другой молекулой. Связывающий белок может связываться, например, с молекулой ДНК (ДНК-связывающий белок), молекулой РНК (РНК-связывающий белок) и/или с молекулой белка (белок-связывающий белок). В случае белок-связывающего белка, он может связываться сам с собой (с образованием гомодимеров, гомотримеров и т.д.) и/или он может связываться с одной или несколькими молекулами другого белка или белков. Связывающий белок может иметь более одного типа активности связывания. Например, белки с цинковыми пальцами имеют активность связывания ДНК, активность связывания РНК и активность связывания белка.
"ДНК-связывающий белок с цинковыми пальцами" (или связывающий домен) представляет собой белок или домен в более крупном белке, который связывает ДНК специфическим для последовательности образом через один или несколько цинковых пальцев, которые представляют собой области аминокислотной последовательности в связывающем домене, структура которого стабилизирована координационной связью с ионом цинка. Термин "ДНК-связывающий белок с цинковыми пальцами" часто сокращенно обозначают как белок с цинковыми пальцами или ZFP.
"ДНК-связывающий домен TALE" или "TALE" представляет собой полипептид, содержащий один или несколько доменов/элементов с повторами TALE. Домены с повторами вовлечены в связывание TALE с распознаваемой им последовательностью ДНК-мишенью. Единичный "элемент повтора" (также обозначаемый как "повтор"), как правило, имеет длину 33-35 аминокислот и проявляет по меньшей мере некоторую гомологию последовательности с другими последовательностями повтора TALE во встречающемся в природе белке TALE.
Связывающие домены с цинковыми пальцами и TALE можно "конструировать способами инженерии" так, чтобы они связывались с заданной нуклеотидной последовательностью, например, посредством модификации способами инженерии (изменения одной или нескольких аминокислот) распознающей спиральной области встречающегося в природе белка с цинковыми пальцами или TALE. Таким образом, модифицированные способами инженерии ДНК-связывающие белки (цинковые пальцы или TALE) представляют собой белки, которые не являются встречающимися в природе. Неограничивающие примеры способов модификации способами инженерии ДНК-связывающих белков являются моделирование и селекция. Смоделированный ДНК-связывающий белок представляет собой белок, который не встречается в природе, конструкция/композиция которого в основном является результатом рациональных критериев. Рациональные критерии для конструирования включают использование правил замены и компьютерных алгоритмов для обработки информации в базе данных, хранящей информацию о существующих конструкциях ZFP и/или TALE и данные о связывании. См., например, патенты США 6140081; 6453242 и 6534261; также см. WO 98/53058; WO 98/53059; WO 98/53060; WO 02/016536 и WO 03/016496 и публикацию США № 20110301073.
"Отобранный путем селекции" белок с цинковыми пальцами или TALE представляет собой белок, не встречающийся в природе, получение которого обычно является результатом эмпирического процесса, такого как фаговый дисплей, ловушка взаимодействий или гибридная селекция. См. например, US 5789538; US 5925523; US 6007988; US 6013453; US 6200759; WO 95/19431; WO 96/06166; WO 98/53057; WO 98/54311; WO 00/27878; WO 01/60970 WO 01/88197, WO 02/099084 и публикацию США № 20110301073.
"Расщепление" относится к разрыву ковалентного остова молекулы ДНК. Расщепление можно инициировать различными способами, включая, но не ограничиваясь ими, ферментативный или химический гидролиз фосфодиэфирной связи. Возможно как одноцепочечное расщепление, так и двухцепочечное расщепление, и двухцепочечное расщепление может происходить в результате двух отдельных событий одноцепочечного расщепления. Расщепление ДНК может приводить к образованию либо тупых концов, либо совмещающихся концов. В определенных вариантах осуществления слитые полипептиды используют для направленного расщепления двухцепочечной ДНК.
"Половинный домен расщепления" представляет собой полипептидную последовательность, которая, совместно со вторым полипептидом (либо идентичным, либо отличающимся) формирует комплекс, обладающий активностью расщепления (предпочтительно, активностью двухцепочечного расщепления). Термины "первый и второй половинные домены расщепления" "+ и - половинные домены расщепления" и "правый и левый половинные домены расщепления" используют взаимозаменяемо для обозначения пар половинных доменов расщепления, которые димеризуются.
"Модифицированный способами инженерии половинный домен расщепления" представляет собой половинный домен расщепления, который модифицирован так, чтобы он обязательно формировал гетеродимеры с другим половинным доменом расщепления (например, другой модифицированный способами инженерии половинный домен расщепления). Также см. публикации патентов США № 2005/0064474, 20070218528, 2008/0131962 и 2011/0201055, включенные в настоящее описание в качестве ссылок в полном объеме.
Средства для внесения двухцепочечного разрыва ДНК: как используют в рамках изобретения, термин "средства для внесения двухцепочечного разрыва ДНК" подразумевает отсылку на специальные условия составления формулы изобретения, утвержденные Конгрессом в 35 U.S.C. § 112, шестой абзац. В частности, "средства для внесения двухцепочечного разрыва ДНК" относятся к молекулярной структуре, которая способна расщеплять обе цепи двухцепочечной молекулы ДНК. Такие структуры включают полипептидные домены, содержащиеся во многих известных нуклеазных белках, например, нуклеазный домен Fok1; каталитический домен выбирают из группы, состоящей из белков Mme1, колицина-E7 (CEA7_ECOLX), колицина-E9, APFL, EndA, Endo I (END1_ECOLI), Endo G человека (NUCG_HUMAN), Endo G животных семейства бычьи (NUCG_BOVIN), R.HinPII, 1-Bas1, 1-Bmo1, 1-Hmu1, 1-Tev1, 1-Tev11, 1-Tev111, 1-Two1, R.Msp1, R.Mva1, NucA, NucM, Vvn, Vvn_CLS, стафилококковой нуклеазы (NUC_STAAU), стафилококковой нуклеазы (NUC_STAHY), микрококковой нуклеазы (NUC__SHIFL), эндонуклеазы yncB, эндодезоксирибонуклеазы I (ENRN_BPT7), метназы, Nb.BsrDI, BsrDI A, Nt.BspD61 (R.BspD61, большая субъединица), ss.BspD61 (R.BspD61, малая субъединица), R.PIe1, Mly1, Alw1, Mva12691, Bsr1, Bsm1, Nb.BtsCI, NtBtsCI, R1.Bts1, R2.Bts1, субъединицы 1 BbvCI, субъединцы 2 BbvCI, альфа-субъединицы BpulOI, бета-субъединицы BpulOI, Bmr1, Bfi1, 1-Cre1, hExo1 (EX01JHUMAN), Exo1 дрожжей (EX01_YEAST), Exo1 E.coli, TREX2 человека, TREX1 мыши, TREX1 человека, TREX1 животных семейства бычьи, TREX1 крысы, DNA2 человека, DNA2 дрожжей (DNA2_YEAST).
Средства для репарации двухцепочечного разрыва ДНК: как используют в рамках изобретения, термин "средства для репарации двухцепочечного разрыва ДНК" подразумевает отсылку на специальные условия составления формулы изобретения, утвержденные Конгрессом в 35 U.S.C. § 112, шестой абзац. В частности, "средства для репарации двухцепочечного разрыва ДНК" относятся к молекулярной структуре, которая способна облегчать/катализировать соединение концов двухцепочечных молекул молекулы, например, путем соединения концов, образованных в результате расщепления одной двухцепочечной молекулы ДНК, или путем связывания одного конца, образованного в результате расщепления одной двухцепочечной молекулы ДНК, с концом экзогенной двухцепочечной молекулы ДНК. Такие структуры включают полипептидные домены, содержащиеся во многих известных лигазных белках, например, в рекомбиназе Cre. В некоторых примерах одна и та же молекулярная структура может служить в качестве как средства для образования двухцепочечного разрыва ДНК, так и в качестве средства для репарации двухцепочечного разрыва ДНК, где одна и также структура способствует как расщеплению, так и репарации двухцепочечных молекул ДНК (например, рекомбиназа Hin).
Индукция сайт-специфических двухцепочечных разрывов в геноме индуцирует каскад репарации ДНК в растительной клетке-хозяине, который устраняет двухцепочечный разрыв с помощью направляемой гомологией репарации (HDR) или репарации связывания негомологичных концов (NHEJ). В научной литературе описано, что в растениях точное встраивание гена или донорной ДНК в нативные геномные или в предварительно модифицированные способами инженерии положения вовлекает входящую донорную конструкцию(и) ДНК, которая содержит различные количества последовательностей, гомологичных последовательностям, фланкирующим заданный двухцепочечный разрыв. Встраивание таких доноров в конкретный заданный локус предположительно основано на каскаде HDR. Подход HDR в качестве исключительной основы для нацеливания генов в растениях может иметь ограничения вследствие сообщений, что каскад репарации HDR не является преобладающим каскадом репарации ДНК по сравнению с NHEJ. В опубликованной научной литературе по использованию в растениях специфичных к мишеням разрывов ДНК (ZFN, TALeN или сконструированные способами инженерии мегануклеазы и т.д.) каскад NHEJ описан в качестве способа внесения специфических точковых мутаций (инсерций или делеций) в геном. В настоящем описании авторы сообщают, что сайт-специфические двухцепочечные разрывы (индуцируемые ZFN, TALeN и т.д.) в присутствии различных конструкций донорной ДНК с областями гомологии от 0 до <10 п.н. могут быть специфически внесены в намеченное положение через каскад репарации NHEJ в растениях. Множество различных конструкций донорной ДНК с гомологией от нулевой до малой 1-10 п.н., варьирующих от линейных до кольцевых, от одноцепочечных до двухцепочечных, может быть нацелено в конкретные положения с использованием каскада NHEJ. Нацеливание в геном растений донорной ДНК на основе NHEJ может быть основано на "связывании липких концов", где направленный двухцепочечный разрыв в геноме вносят с помощью Fok1 (или других доменов эндонуклеаз типа II) и на донорных конструкциях ДНК для NHEJ находятся соответствующие липкие концы. Донорную ДНК с липкими концами можно доставлять в клетку в качестве линейной донорной ДНК с заданными выступающими концами. Альтернативным подходом является формирование липких концов донорной ДНК in vivo путем совместной доставки ZFN для мишени в хозяине и кольцевой донорной молекулы ДНК, которая содержит по меньшей мере один участок распознавания ZFN, который идентичен участку распознавания в мишени. Экспрессия по меньшей мере одного ZFN приводит к расщеплению геномной ДНК хозяина (нативной или предварительно модифицированной способами инженерии) и кольцевой донорной ДНК с образованием липких концов, которые связываются с использованием каскада репарации NHEJ хозяина.
Являются возможными один или несколько участков расщепления ZFN на донорной молекуле (один участок расщепления ZFN для линеаризации всей донорной молекулы, 2 одинаковых участка ZFN для высвобождения меньшего фрагмента донорной ДНК, или 2 различных участка ZFN для высвобождения фрагмента из донора и соответствующего фрагмента из геномной ДНК хозяина (замена ДНК).
Таким образом, донорный полинуклеотид может представлять собой ДНК или РНК, может быть одноцепочечным и/или двухцепочечным и его можно вводить в клетку в линейной или в кольцевой форме. См., например, публикации патентов США № 20100047805 и 20110207221. Определенные варианты осуществления настоящего изобретения также могут включать композиции линейной экзогенной (донорной) нуклеиновой кислоты(кислот), содержащие эти нуклеиновые кислоты, и способы получения и применения этих линейных донорных молекул. В определенных вариантах осуществления линейная донорная молекула стабильно присутствует в клетке, в которую она введена. В других вариантах осуществления линейная донорная молекула модифицирована так, чтобы она была устойчивой к расщеплению экзонуклеазами, например, путем помещения одной или нескольких фосфоротиоатных связей между одной или несколькими парами оснований на концах донорной молекулы. Линейная экзогенная нуклеиновая кислота также может включать одноцепочечную специфическую ДНК.
III. Локусы функциональности FAD2
Локусы, обозначаемые как FAD2 (десатураза жирных кислот 2) включены в QTL, вовлеченные в наследование комплексного полигенного признака содержания жирных кислот в растениях. FAD2 кодирует фермент, ответственный за десатурацию олеиновой кислоты (18:1) до линолеиновой кислоты (C18:2). Tanhuanpaa et al. (1998) Mol. Breed. 4:543-50; Schierholt et al. (2001) Crop Sci. 41:1444-9.
В каскаде биосинтеза растительных масел десатуразы жирных кислот (FAD) играют ключевую роль при биосинтезе липидов растений, и их активность в значительной степени влияет на состав жирных кислот. FAD широко распространены в растениях, и анализ экспрессии показал, что мРНК FAD продуцируется в избытке. Более того, гены FAD экспрессируются в различных тканях и типах клеток, а также в субклеточных компартментах, включающих пластиды и эндоплазматическую сеть.
Композиция жирных кислот в растениях и функциональность масел, производимых из них, во многих применениях определяются относительными концентрациями основных составляющих их жирных кислот: олеиновой, линолевой и линоленовой (C18:3). Концентрации этих жирных кислот в основном регулируются функцией ферментов FAD2 и FAD3. Олеиновая кислота конвертируется в растениях в линолеиновую кислоту и линоленовую кислоту по схеме:

Гены FAD2 идентифицировали в большинстве видов растений и водорослей, включая, но не ограничиваясь ими, кукурузу, сою, хлопок, арабидопсис, пшеницу, кормовые травы, рис, подсолнечник и канолу, и модификация экспрессии FAD2 приводит к измененным профилям жирных кислот в таких организмах. Более того, растения, содержащие модифицированные гены FAD2, коммерционализированы, и было показано, что разрушение гена FAD2 способно улучшить питательные и функциональные свойства масла, продуцируемого растением-хозяином, без агрономического ухудшения растения-хозяина. Например, сорта канолы и подсолнечника, которые коммерционализированы под торговым названием Nexera® (Dow AgroSciences, LLC), характеризуются более высоким содержанием олеиновой кислоты, более низким содержанием линолевой кислоты и более низким содержанием линоленовой кислоты (и более низким содержанием насыщенных жирных кислот) по сравнению с профилями канолы и подсолнечника дикого типа.
Преобладающим видом канолы, выращиваемым в Европе, Северной Америке и Австралии является Brassica napus, при этом считается, что полиплоидные виды Brassica появились в результате гибридизации B. oleracea (имеющего диплоидный геном C) и B. rapa (имеющего диплоидный геном A). Цитогенетическое исследование показало, что геномы AA и CC демонстрируют некоторую степень родства, являясь частично гомологичными друг другу. Как геном A, так и геном C, имеют высокий процент гомологичных или паралогичных генов. Таким образом, полагают, что геномы AA и CC происходят из общего генома-предшественника. Prakash and Hinata (180) Opera Botanica 55:1-57. Хотя геномы обоих видов-предшественников технически классифицируют как диплоидные, эти геномы содержат высокий процент областей, которые являются дубликатами друг друга. Song et al. (1991) Theor. Appl. Genet. 82:296-304. Детальный анализ RFLP органелл и ядра показал, что геном AA B. rapa привнес десять хромосом в B. napus, в то время как B. oleracea привнес девять хромосом из его генома CC в качестве материнского донора. Song et al. (1992) Genome 35:992-1001. Посредством ряда удвоений генома в обоих геномах-предшественниках, а также вследствие высокого процента сходства между геномами A, B и C, появилось несколько копий генов FAD2 и FAD3. На практике, этот факт делает разведение конолы с модифицированными и/или разрушенными копиями этих генов для достижения конкретного профиля жирных кислот особенно трудным.
Известные функциональные копии генов FAD2 в каноле расположены в группе сцепления N4 генома A. Scheffler et al. (1997) TAG 94(5):583-91; Schierholt et al. (2000) TAG 101(5-6):897-901. Позднее была установлена ассоциация признака высокого содержания олеиновой кислоты с модифицированным и разрушенным геном FAD2, который расположен в геноме A. Публикация патентной заявки США № US 2006/0248611 A1; Hu et al. (2006) "Identification and Mapping of FAD2 and FAD3 Mutations and Development of Allele-specific Markers for High Oleic and Low Linolenic Acid Contents in Canola (Brassica napus L.)", Plant & Animal Genomes XIV Conference, January 14-18, 2006, San Diego, CA. Инактивация аллеля FAD2 обеспечивает контроль содержания олеиновой кислоты посредством уменьшения десатурации олеиновой кислоты до линолеиновой кислоты. Этот признак высокого содержания олеиновой кислоты и fad2 был идентифицирован в сорте B. napus (DMS100), который имеет характерное содержание олеиновой кислоты приблизительно 77%. См., заявку США № 10/545100. Кроме того, недавно была установлена локализация генов FAD2 на хромосоме A5, и было предположено, что они ответственны за высокое содержание C18:1. См., Yang et al., "Brassica napus genome" Theor Appl Genet (2012 125:715-729. Кроме того, были разработаны генетические маркеры для облегчения интрогрессии FAD2 и признака высокого содержания олеиновой кислоты в канолу.
Локусы FAD2 можно модифицировать и/или разрушать в растении без неблагоприятного влияния на ценность растения, и, для многих целей, с фактическим повышением его ценности, включая изменение экспрессии FAD2, изменение содержания/соотношения масел и/или встраивание и экспрессию желаемых трансгенов. Более того, ввиду повсеместного характера локусов FAD в растениях, локусы FAD2 можно модифицировать или разрушать без вреда по меньшей мере для некоторых целей во многих видах, включая, например, но не ограничиваясь ими: канолу; сою; кукурузу; пшеницу; кормовые травы; brassica sp.; рис, томаты, ячмень; рожь; сорго; хлопок и подсолнечник, а также грибы и водоросли. Варианты осуществления изобретения включают локусы FAD2 и их применение в качестве локусов функциональности для встраивания экзогенных нуклеиновых кислот. Например, локус FAD2 проявляет по меньшей мере один из нескольких признаков, которые определены в качестве желательных в контексте его применения в качестве локуса функциональности, в том числе, например, но не ограничиваясь этим, то, что уровень экспрессии в ходе жизненного цикла организма-хозяина является приблизительно постоянным и, неожиданно, что встраивание донорной ДНК в локус FAD2 не индуцирует ухудшения качества или пригодности хозяина.
В некоторых вариантах осуществления настоящего изобретения по меньшей мере один локус FAD2 (например, локус FAD2A, FAD2A', FAD2C и/или FAD2С') используют в качестве участка-мишени для сайт-специфического встраивания экзогенной нуклеиновой кислоты (например, нуклеиновая кислота, содержащая нуклеотидную последовательность, кодирующую представляющий интерес полипептид). В конкретных вариантах осуществления встраивание экзогенной нуклеиновой кислоты приводит к модифицированному локусу. Например, встраивание экзогенной нуклеиновой кислоты может модифицировать локус так, чтобы образовался разрушенный (т.е. инактивированный) ген FAD2.
В некоторых вариантах осуществления локус FAD2 может содержать нуклеотидную последовательность, которая специфически гибридизуется с последовательностью, комплементарной нуклеотидной последовательности, выбранной из группы, состоящей из SEQ ID NO: 22-26, SEQ ID NO: 28-33 и SEQ ID NO: 35-38. Например, локус FAD2 может содержать нуклеотидную последовательность, выбранную из группы, состоящей из SEQ ID NO: 22-26, SEQ ID NO: 28-33 и SEQ ID NO: 35-38. В некоторых вариантах осуществления локус FAD2 может содержать нуклеотидную последовательность, которая по существу идентична нуклеотидной последовательности, выбранной из группы, состоящей из SEQ ID NO: 22-26, SEQ ID NO: 28-33 и SEQ ID NO: 35-38. Например, в некоторых вариантах осуществления локус FAD2 представляет собой гомолог FAD2 (например, ортолог или паралог), который содержит нуклеотидную последовательность, которая по меньшей мере приблизительно на 85% идентична нуклеотидной последовательности, выбранной из группы, состоящей из SEQ ID NO: 22-26, SEQ ID NO: 28-33 и SEQ ID NO: 35-38. Гомолог FAD2 может содержать нуклеотидную последовательность, которая, например, но не ограничиваясь этим: по меньшей мере на 80%; по меньшей мере на 85%; по меньшей мере приблизительно на 90%; по меньшей мере приблизительно на 91%; по меньшей мере приблизительно на 92%; по меньшей мере приблизительно на 93%; по меньшей мере приблизительно на 94%; по меньшей мере приблизительно на 95%; по меньшей мере приблизительно на 96%; по меньшей мере приблизительно на 97%; по меньшей мере приблизительно на 98%; по меньшей мере приблизительно на 99%; по меньшей мере приблизительно на 99,5%; 99,6%, 99,7%, 99,8% и/или по меньшей мере приблизительно на 99,9% идентична нуклеотидной последовательности, выбранной из группы, состоящей из SEQ ID NO: 22-26, SEQ ID NO: 28-33 и SEQ ID NO: 35-38. Такой гомолог FAD2 можно без труда идентифицировать и выделять из любого полного или неполного генома, свободно доступного специалистам в данной области для различных организмов.
IV. Направленное встраивание нуклеиновой кислоты в локус FAD2
Сайт-специфическое встраивание экзогенной нуклеиновой кислоты в локус FAD2 можно проводить любым способом, известным специалистам в данной области. В некоторых вариантах осуществления встраивание экзогенной нуклеиновой кислоты в локус FAD2 включает контактирование клетки (например, выделенной клетки или клетки в ткани или организме) с молекулой нуклеиновой кислоты, содержащей экзогенную нуклеиновую кислоту. Например, такая молекула нуклеиновой кислоты может содержать нуклеотидные последовательности, фланкирующие экзогенную нуклеиновую кислоту, которые облегчают гомологичную рекомбинацию между молекулой нуклеиновой кислоты и по меньшей мере одним локусом FAD2. В конкретных примерах нуклеотидные последовательности, фланкирующие экзогенную нуклеиновую кислоту, которые облегчают гомологичную рекомбинацию, могут быть комплементарными эндогенным нуклеотидам локуса FAD2. В конкретных примерах нуклеотидные последовательности, фланкирующие экзогенную нуклеиновую кислоту, которые способствуют гомологичной рекомбинации, могут быть комплементарными ранее встроенным экзогенным нуклеотидам. В некоторых вариантах осуществления множество экзогенных нуклеиновых кислот можно встраивать в один локус FAD2, как например, при стэкинге генов.
В некоторых вариантах осуществления встраивание нуклеиновой кислоты в локус FAD2 может облегчаться (например, катализироваться) эндогенным клеточным аппаратом клетки-хозяина, таким как, например, но не ограничиваясь этим, эндогенная ДНК и эндогенные ферменты рекомбиназы. В некоторых вариантах осуществления встраивание нуклеиновой кислоты в локус FAD2 может облегчаться с помощью одного или нескольких факторов (например, полипептидов), которые предоставлены клетке-хозяину. Например, полипептиды нуклеазы(нуклеаз), рекомбиназы(рекомбиназ) и/или лигазы могут быть предоставлены (либо независимо, либо в качестве части химерного полипептида) путем контактирования полипептидов с клеткой-хозяином или путем экспрессии полипептидов в клетке-хозяине. Таким образом, в некоторых примерах нуклеиновую кислоту, содержащую нуклеотидную последовательность, кодирующую по меньшей мере один полипептид нуклеазы, рекомбиназы и/или лигазы, можно вводить в клетку-хозяина либо одновременно, либо последовательно с нуклеиновой кислотой, подлежащей сайт-специфическому встраиванию в локус FAD2, где по меньшей мере один полипептид нуклеазы, рекомбиназы и/или лигазы экспрессируется с нуклеотидной последовательности в клетке-хозяине.
A. ДНК-связывающие полипептиды
В некоторых вариантах осуществления сайт-специфическое встраивание можно проводить с использованием факторов, которые способны распознавать и связываться с конкретными нуклеотидными последовательностями, например, в геноме организма хозяина. Например, многие белки содержат полипептидные домены, которые способны распознавать и связывать ДНК сайт-специфическим образом. Последовательность ДНК, которую распознает ДНК-связывающий полипептид, может быть обозначена как последовательность-"мишень". Полипептидные домены, которые способны распознавать и связывать ДНК сайт-специфическим образом, как правило, правильно сворачиваются и функционируют независимо, связывая ДНК сайт-специфическим образом, даже при экспрессии в полипептиде, отличном от белка, из которого домен был первоначально выделен. Аналогично, последовательности-мишени для распознавания и связывания ДНК-связывающими полипептидами, как правило, могут распознаваться и связываться такими полипептидами, даже когда они присутствуют в крупных структурах ДНК (например, в хромосоме), в частности, когда участок, где расположена последовательность-мишень, представляет собой участок, о котором известно, что он является доступным для растворимых клеточных белков (например, ген).
В то время как ДНК-связывающие полипептиды, идентифицированные в белках, которые существуют в природе, как правило, связываются с определенной нуклеотидной последовательностью или мотивом (например, консенсусная последовательность распознавания), в данной области существуют и известны способы модификации многих таких ДНК-связывающих полипептидов так, чтобы они распознавали отличающуюся нуклеотидную последовательность или мотив ДНК-связывающих полипептидов, которые включают, например, но не ограничиваясь ими: ДНК-связывающие домены с цинковыми пальцами; лейциновые молнии; ДНК-связывающие домены UPA; GAL4; TAL; LexA; репрессор Tet; LacR и рецептор стероидных гормонов.
В некоторых примерах ДНК-связывающий полипептид представляет собой цинковый палец. Индивидуальные мотивы в виде цинковых пальцев можно конструировать для нацеливания и специфического связывания с любым из большого диапазона участков. Канонические полипептиды с цинковыми пальцами Cys2His2(а также неканонические Cys3His) связывают ДНК путем встраивания α-спирали в большую бороздку двойной спирали ДНК-мишени. Распознавание ДНК цинковым пальцем является модульным; каждый палец контактирует в основном с тремя последовательно расположенными парами оснований в мишени, и распознавание опосредуют несколько ключевых остатков в полипептиде. Путем включения нескольких ДНК-связывающих доменов с цинковыми пальцами в нацеливающую эндонуклеазу, специфичность связывания ДНК у нацеливающей эндонуклеазы может быть дополнительно увеличена (и, таким образом, также может быть увеличена специфичность любых сообщаемых ей эффектов регуляции генов). См., например, Urnov et al. (2005) Nature 435:646-51. Таким образом, один или несколько ДНК-связывающих полипептидов с цинковыми пальцами можно модифицировать способами инженерии и использовать таким образом, чтобы нацеливающая эндонуклеаза, введенная в клетку-хозяина, взаимодействовала с последовательностью ДНК, которая является уникальной в геноме клетки-хозяина.
Предпочтительно, белок с цинковыми пальцами не является встречающимся в природе, а является сконструированным для связывания выбранного участка-мишени. См., например, Beerli et al. (2002) Nature Biotechnol. 20:135-141; Pabo et al. (2001) Ann. Rev. Biochem. 70:313-340; Isalan et al. (2001) Nature Biotechnol. 19:656-660; Segal et al. (2001) Curr. Opin. Biotechnol. 12:632-637; Choo et al. (2000) Curr. Opin. Struct. Biol. 10:411-416; патенты США № 6453242; 6534261; 6599692; 6503717; 6689558; 7030215; 6794136; 7067317; 7262054; 7070934; 7361635; 7253273; и публикации патентов США № 2005/0064474; 2007/0218528; 2005/0267061, все из которых включены в настоящее описание в качестве ссылок в полном объеме.
Сконструированный связывающий домен с цинковыми пальцами может иметь новую специфичность связывания по сравнению со встречающимся в природе белком с цинковыми пальцами. Способы конструирования включают, но не ограничиваются ими, рациональное конструирование и различные типы селекции. Рациональное конструирование включает, например, использование баз данных, содержащих триплетные (или квадруплетные) нуклеотидные последовательности и индивидуальные аминокислотные последовательности цинковых пальцев, в которых каждая триплетная или квадруплетная нуклеотидная последовательность ассоциирована с одной или несколькими аминокислотными последовательностями цинковых пальцев, которые связывают конкретную триплетную или квадруплетную последовательность. См., например, патенты США того же заявителя 6453242 и 6534261, включенные в настоящее описание в качестве ссылок в полном объеме.
Иллюстративные способы селекции, включая фаговый дисплей и двухгибридные системы, описаны в патентах США 5789538; 5925523; 6007988; 6013453; 6410248; 6140466; 6200759 и 6242568; а также в WO 98/37186; WO 98/53057; WO 00/27878; WO 01/88197 и GB 2338237. Кроме того, усиление специфичности связывания у связывающих доменов с цинковыми пальцами описано, например, в WO 02/077227 того же заявителя.
Кроме того, как описано в этих и других источниках литературы, домены цинковых пальцев и/или белки с несколькими цинковыми пальцами могут быть связаны вместе с использованием любых подходящих линкерных последовательностей, включая, например, линкеры длиной 5 или более аминокислот. Также см. патенты США № 6479626; 6903185 и 7153949 для иллюстративных линкерных последовательностей длиной 6 или более аминокислот. Белки, описанные в настоящем описании, могут включать любую комбинацию подходящих линкеров между отдельными цинковыми пальцами белка.
Выбор участков-мишеней; ZFP и способы моделирования и конструирования слитых белков (и полинуклеотидов, кодирующих их) известны специалистам в данной области и подробно описаны в патентах США № 61400815; 789538; 6453242; 6534261; 5925523; 6007988; 6013453; 6200759; WO 95/19431; WO 96/06166; WO 98/53057; WO 98/54311; WO 00/27878; WO 01/60970 WO 01/88197; WO 02/099084; WO 98/53058; WO 98/53059; WO 98/53060; WO 02/016536 и WO 03/016496.
Кроме того, как описано в этих и других источниках литературы, домены цинковых пальцев и/или белки с несколькими цинковыми пальцами могут быть связаны вместе с использованием любых подходящих линкерных последовательностей, включая, например, линкеры длиной 5 или более аминокислот. Также см. патенты США № 6479626; 6903185 и 7153949 для иллюстративных линкерных последовательностей длиной 6 или более аминокислот. Белки, описанные в настоящем описании, могут включать любую комбинацию подходящих линкеров между отдельными цинковыми пальцами белка.
В некоторых примерах ДНК-связывающий полипептид представляет собой ДНК-связывающий домен из GAL4. GAL4 представляет собой модульный трансактиватор в Saccharomyces cerevisiae, однако он также функционирует в качестве трансактиватора во многих других организмах. См., например, Sadowski et al. (1988) Nature 335:563-4. В этой регуляторной системе экспрессия генов, кодирующих ферменты метаболического пути галактозы в S. cerevisiae, строго регулируются доступным источником углерода. Johnston (1987) Microbiol. Rev. 51: 458-76. Контроль транскрипции этих метаболических ферментов опосредуется взаимодействием между положительным регуляторным белком, GAL4 и симметричной последовательностью ДНК размером 17 п.н., с которой GAL4 специфически связывается (UAS).
Нативный GAL4 включает 881 аминокислотный остаток с молекулярной массой 99 кДа. GAL4 содержит функционально автономные домены, совокупная активность которых обеспечивает активность GAL4 in vivo. Ma and Ptashne (1987) Cell 48:847-53); Brent and Ptashne (1985) Cell 43(3 Pt 2):729-36. N-концевые 65 аминокислот GAL4 содержат ДНК-связывающий домен GAL4. Keegan et al. (1986) Science 231: 699-704; Johnston (1987) Nature 328:353-5. Специфическое связывание последовательности требует присутствия двухвалентного катиона, координируемого 6 остатками Cys, присутствующими в ДНК-связывающем домене. Скоординированный катионсодержащий домен взаимодействует и распознает консервативный триплет CCG на каждом конце UAS из 17 п.н. посредством прямых контактов с большой бороздкой спирали ДНК. Marmorstein et al. (1992) Nature 356:408-14. Функция связывания ДНК у белка обеспечивает расположение C-концевых доменов активации транскрипции вблизи промотора, так чтобы активирующие домены могли контролировать транскрипцию.
Дополнительные ДНК-связывающие полипептиды, которые можно использовать в определенных вариантах осуществления, включают, например, но не ограничиваясь ими, связывающую последовательность из AVRBS3-индуцируемого гена; консенсусную связывающую последовательность из AVRBS3-индуцируемого гена или синтетическую связывающую последовательность, сконструированную из них (например, ДНК-связывающий домен UPA); TAL; LexA (см., например, Brent & Ptashne (1985), выше); LacR (см., например, Labow et al. (1990) Mol. Cell. Biol. 10:3343-56; Baim et al. (1991) Proc. Natl. Acad. Sci. USA 88(12):5072-6); рецептор стероидных гормонов (Ellliston et al. (1990) J. Biol. Chem. 265:11517-121); репрессор Tet (патент США 6271341) и мутантный репрессор Tet, который связывается с последовательностью оператора tet в присутствии, но не в отсутствие тетрациклина (Tc); ДНК-связывающий домен NF-κΒ; и компоненты регуляторной системы, описанной в Wang et al. (1994) Proc. Natl. Acad. Sci. USA 91(17):8180-4, в которой используется слияние GAL4, рецептора гормона и VP16.
В определенных вариантах осуществления ДНК-связывающий домен одной или нескольких нуклеаз, используемых в способах и композициях, описанных в настоящем описании, содержит встречающийся в природе или сконструированный (не встречающийся в природе) ДНК-связывающий домен эффектора TAL. См., например, публикацию патента США № 20110301073, включенную в настоящее описание в качестве ссылки в полном объеме. Известно, что патогенные для растений бактерии рода Xanthomonas вызывают множество заболеваний в важных сельскохозяйственных культурах. Патогенность Xanthomonas зависит от консервативной системы секреции типа III (T3S), которая инъецирует более 25 различных эффекторных белков в растительную клетку. Среди этих инъецируемых белков находятся эффекторы, подобные активаторам транскрипции (TAL), которые имитируют активаторы транскрипции растений и манипулируют транскриптомом растений (см. Kay et al. (2007) Science 318:648-651). Эти белки содержат ДНК-связывающий домен и домен активации транскрипции. Одним из наиболее хорошо охарактеризованных эффекторов TAL является AvrBs3 из Xanthomonas campestgris pv. Vesicatoria (см. Bonas et al. (1989) Mol Gen Genet 218: 127-136 и WO2010079430). Эффекторы TAL содержат централизованный домен из тандемных повторов, причем каждый повтор содержит приблизительно 34 аминокислоты, которые являются ключевыми для специфичности этих белков в отношении связывания ДНК. Кроме того, они содержат последовательность ядерной локализации и кислый домен активации транскрипции (для обзора см. Schornack S, et al. (2006) J Plant Physiol 163(3): 256-272). Кроме того, в фитопатогенных бактериях Ralstonia solanacearum было выявлено два гена, обозначаемых brg11 и hpx17, которые гомологичны семейству AvrBs3 Xanthomonas в R. solanacearum биоваре 1 штамма GMI1000 и в биоваре 4 штамма RS1000 (См. Heuer et al. (2007) Appl and Envir Micro 73(13): 4379-4384). Эти гены на 98,9% идентичны друг другу по нуклеотидной последовательности, но отличаются делецией 1575 п.н. в домене повтора hpx17. Однако продукты обоих генов имеют менее чем 40% идентичность последовательности с белками семейства AvrBs3 Xanthomonas. См., например, патенты США № 8420782 и 8440431 и публикацию патента США № 20110301073.
В других вариантах осуществления нуклеаза содержит систему CRISPR/Cas. Локус CRISPR (кластеризованные регулярно расположенные короткие палиндромные повторы), который кодирует РНК-компоненты системы, и локус cas (ассоциированный с CRISPR), который кодирует белки (Jansen et al., 2002. Mol. Microbiol. 43: 1565-1575; Makarova el al., 2002. Nucleic Acids Res. 30: 482-496; Makarova et al., 2006. Biol. Direct 1: 7; Haft et al., 2005. PLoS Comput. Biol. 1: e60) формируют последовательности генов системы нуклеаз CRISPR/Cas. Локусы CRISPR в микробных хозяевах содержат комбинацию ассоциированных с CRISPR (Cas) генов, а также некодирующие РНК-элементы, способные программировать специфичность опосредуемого CRISPR расщепления нуклеиновых кислот.
CRISPR типа II представляет собой одну из наиболее охарактеризованных систем, и она осуществляет направленный разрыв двухцепочечной ДНК за четыре последовательных стадии. Во-первых, с локуса CRISPR транскрибируются две некодирующих РНК: матрица пре-crRNA и tracrRNA. Во-вторых, tracrRNA гибридизуется с областями повторов пре-crRNA и опосредует процессинг пре-crRNA в зрелые crRNA, содержащие индивидуальные спейсерные последовательности. В-третьих, зрелый комплекс crRNA:tracrRNA направляет Cas9 к ДНК-мишени через образование пар по принципу Уотсона-Крика между спейсером на crRNA и протоспейсером на ДНК-мишени рядом с соседним с протоспейсером мотивом (PAM), что является дополнительным требованием для распознавания мишени. Наконец, Cas9 опосредует расщепление ДНК-мишени, создавая двухцепочечный разрыв в протоспейсере. Активность Cas-системы CRISPR включает три стадии: (i) встраивание чужеродных последовательностей ДНК в матрицу CRISPR для предупреждения будущих атак в процессе, называемом "адаптацией", (ii) экспрессия соответствующих белков, а также экспрессия и процессинг матрицы, за которой следует (iii) РНК-опосредуемая интерференция в отношении чужеродной нуклеиновой кислоты. Таким образом, в бактериальной клетке несколько так называемых белков "Cas" вовлечено в природную функцию системы CRISPR/Cas и участвуют в таких функциях, как встраивание чужеродной ДНК и т.д.
В определенных вариантах осуществления белок Cas может представлять собой "функциональное производное" встречающегося в природе белка Cas. "Функциональное производное" полипептида с нативной последовательностью представляет собой соединение, обладающее качественным биологическим свойством, являющимся общим с полипептидом с нативной последовательностью. "Функциональные производные" включают, но не ограничиваются ими, фрагменты полипептида с нативной последовательностью и производные полипептида с нативной последовательностью и их фрагменты при условии, что они обладают общей биологической активностью с соответствующим полипептидом с нативной последовательностью. Биологическая активность, предусматриваемая в рамках настоящего изобретения, представляет собой способность функционального производного гидролизовать ДНК-субстрат на фрагменты. Термин "производное" охватывает как варианты аминокислотной последовательности полипептида, так и ковалентные модификации, и его слитые конструкции. Подходящие производные полипептида Cas или его фрагмента включают, но не ограничиваются ими, мутанты, слитые конструкции, ковалентные модификации белка Cas или его фрагмента. Белок Cas, который включает белок Cas или его фрагмент, а также производные белка Cas или его фрагмента, можно получать из клетки или синтезировать химически или путем комбинирования этих двух действий. Клетка может представлять собой клетку, которая в естественным путем продуцирует белок Cas, или клетку, которая естественным путем продуцирует белок Cas и модифицирована способами генной инженерии для продуцирования эндогенного белка Cas на более высоком уровне экспрессии или для продуцирования белка Cas с экзогенно введенной нуклеиновой кислоты, которая кодирует Cas, который является таким же или отличается от эндогенного Cas. В некоторых случаях клетка не продуцирует естественным образом белок Cas и она модифицирована способами генной инженерии для продуцирования белка Cas.
В конкретных вариантах осуществления ДНК-связывающий полипептид специфически распознает и связывает нуклеотидную последовательность-мишень, содержащуюся в геномной нуклеиновой кислоте организма хозяина. В некоторых примерах в геноме хозяина может быть найдено любое количество отдельных копий нуклеотидной последовательности-мишени. Нуклеотидная последовательность-мишень может быть редкой в геноме организма (например, в геноме может существовать менее чем приблизительно 10, приблизительно 9, приблизительно 8, приблизительно 7, приблизительно 6, приблизительно 5, приблизительно 4, приблизительно 3, приблизительно 2 или приблизительно 1 копия(ий) последовательности-мишени). Например, нуклеотидная последовательность-мишень может быть расположена в уникальном участке в геноме организма. Нуклеотидные последовательности-мишени могут быть, например, но не ограничиваясь этим, случайным образом распределены в геноме относительно друг друга; расположены в различных группах сцепления в геноме; расположены в одной и той же группе сцепления; расположены на различных хромосомах; расположены на одной и той же хромосоме; расположены в геноме в участках, которые экспрессируются в сходных условиях в организме (например, под контролем одних и тех же или по существу функционально идентичных регуляторных факторов); и расположены близко друг к другу в геноме (например, последовательности-мишени могут содержаться в нуклеиновых кислотах, встроенных в качестве конкатемеров в геномные локусы).
B. Нацеливающие эндонуклеазы
В конкретных вариантах осуществления ДНК-связывающий полипептид, который специфически распознает и связывает нуклеотидную последовательность-мишень, может содержаться в химерном полипептиде, так чтобы сообщать химерному полипептиду специфическое связывание с последовательностью-мишенью. Например, такой химерный полипептид может содержать, например, но не ограничиваясь ими, полипептиды нуклеазы, рекомбиназы и/или лигазы, описанные выше. Химерные полипептиды, содержащие ДНК-связывающий полипептид и полипептид нуклеазы, рекомбиназы и/или лигазы, также может содержать другие функциональные полипептидные мотивы и/или домены, такие как, например, но не ограничиваясь ими: спейсерная последовательность, расположенная между функциональными полипептидами в химерном белке; лидерный пептид; пептид, который нацеливает слитый белок на органеллу (например, ядро); полипептиды, которые расщепляются клеточным ферментом; пептидные метки (например, Myc, His и т.д.); и другие аминокислотные последовательности, которые не препятствуют функции химерного полипептида.
Функциональные полипептиды (например, ДНК-связывающие полипептиды и нуклеазные полипептиды) в химерном полипептиде могут быть функционально связаны. В некоторых вариантах осуществления функциональные полипептиды химерного полипептида могут быть функционально связанными посредством их экспрессии с одного полинуклеотида, кодирующего по меньшей мере функциональные полипептиды, лигированные друг с другом в рамке считывания, так чтобы получился химерный ген, кодирующий химерный белок. В альтернативных вариантах осуществления функциональные полипептиды химерного полипептида могут быть функционально связаны другими способами, такими как сшивание независимо экспрессированных полипептидов.
В некоторых вариантах осуществления ДНК-связывающий полипептид, который специфически распознает и связывает нуклеотидную последовательность-мишень, может содержаться в природном выделенном белке (или его мутанте), где природный выделенный белок или его мутант также содержит нуклеазный полипептид (и также может содержать полипептид рекомбиназы и/или лигазы). Примеры таких выделенных белков включают TALEN, рекомбиназы (например, рекомбиназа Cre, Hin, Tre и FLP), РНК-контролируемые CRISPR-Cas9, и мегануклеазы.
Как используют в рамках изобретения, термин "нацеливающая эндонуклеаза" относится к природным или сконструированным выделенным белкам или их мутантам, которые содержат ДНК-связывающий полипептид и нуклеазный полипептид, а также к химерным полипептидам, содержащим ДНК-связывающий полипептид и нуклеазу. В определенных вариантах осуществления можно использовать любую нацеливающую эндонуклеазу, содержащую ДНК-связывающий полипептид, который специфически распознает и связывает нуклеотидную последовательность-мишень, содержащуюся в локусе FAD2 (например, либо поскольку последовательность-мишень содержится в нативной последовательности в локусе, либо поскольку последовательность-мишень введена в локус, например, посредством рекомбинации).
Некоторые примеры химерных полипептидов, которые могут быть полезными в конкретных вариантах осуществления изобретения, включают, но не ограничиваются ими, комбинации следующих полипептидов: ДНК-связывающие полипептиды с цинковыми пальцами; нуклеазный полипептид Fok1; домены TALE; лейциновые молнии; ДНК-связывающие мотивы факторов транскрипции и домены распознавания и/или расщепления ДНК, выделенные из, например, но не ограничиваясь этим, TALEN, рекомбиназы (например, рекомбиназы Cre, Hin, RecA, Tre и FLP), РНК-контролируемой CRISPR-Cas9, мегануклеазы; и другие белки, известные в данной области. Конкретные примеры включают химерный белок, содержащий сайт-специфический ДНК-связывающий полипептид и нуклеазный полипептид. Химерные полипептиды можно модифицировать способами инженерии, известными специалистам в данной области, для изменения последовательности распознавания ДНК-связывающего полипептида, содержащегося в химерном полипептиде, так чтобы химерный полипептид был нацелен на конкретную представляющую интерес нуклеотидную последовательность.
В определенных вариантах осуществления химерный полипептид содержит ДНК-связывающий домен (например, цинковый палец, эффекторный домен TAL и т.д.) и нуклеазный домен (домен расщепления). Домен расщепления может быть гетерологичным ДНК-связывающему домену, как например, ДНК-связывающий домен с цинковыми пальцами и домен расщепления из нуклеазы, или ДНК-связывающий домен TALEN и домен расщепления, или ДНК-связывающий домен мегануклеазы и домен расщепления из другой нуклеазы. Гетерологичные домены можно получать из любой эндонуклеазы или экзонуклеазы. Иллюстративные эндонуклеазы, из которых может происходить домен расщепления, включают, но не ограничиваются ими, эндонуклеазы рестрикции и хоминг-эндонуклеазы. См., например, 2002-2003 Catalogue, New England Biolabs, Beverly, MA; и Belfort et al. (1997) Nucleic Acids Res. 25:3379-3388. Дополнительные ферменты, которые расщепляют ДНК, известны (например, нуклеаза SI; нуклеаза фасоли золотистой; ДНК-аза I поджелудочной железы; микрококковая нуклеаза; эндонуклеаза HO дрожжей; также см. Linn et al. (eds.) Nucleases, Cold Spring Harbor Laboratory Press, 1993). Один или несколько этих ферментов (или их функциональных фрагментов) можно использовать в качестве источника доменов расщепления и половинных доменов расщепления.
Аналогично, половинный домен расщепления может происходить из любой нуклеазы или ее части, как указано выше, которая требует димеризации для активности расщепления. Как правило, если слитые белки содержат половинные домены расщепления, для расщепления требуется два слитых белка. Альтернативно можно использовать один белок, содержащий два половинных домена расщепления. Два половинных домена расщепления могут происходить из одной и той же эндонуклеазы (или ее функциональных фрагментов), или каждый половинный домен расщепления может происходить из отличающейся эндонуклеазы (или ее функциональных фрагментов). Кроме того, участки-мишени для двух слитых белков предпочтительно располагаются друг относительно друга так, что связывание двух слитых белков с их соответствующими участками располагает половинные домены в такой пространственной ориентации друг относительно друга, которая позволяет половинным доменам расщепления формировать функциональный домен расщепления, например, путем димеризации. Таким образом, в определенных вариантах осуществления ближайшие края участков-мишеней разделены 5-8 нуклеотидами или 15-18 нуклеотидами. Однако между двумя участками-мишенями может располагаться любое целое число нуклеотидов или пар нуклеотидов (например, от 2 до 50 пар нуклеотидов или более). Как правило, участок расщепления лежит между участками-мишенями.
Эндонуклеазы рестрикции (ферменты рестрикции) присутствуют во многих видах и способны к специфическому к последовательности связыванию с ДНК (в участке распознавания), и расщеплению ДНК в или вблизи участка связывания, например, так чтобы одна или несколько экзогенных последовательностей (доноры/трансгены) встраивалась в или вблизи участков связывания (мишеней). Определенные ферменты рестрикции (например, типа IIS) расщепляют ДНК в участках, удаленных от участка распознавания, и имеют отдельные домены связывания и расщепления. Например, фермент FokI типа IIS катализирует двухцепочечное расщепление ДНК на расстоянии 9 нуклеотидов от его участка распознавания на одной цепи и на расстоянии 13 нуклеотидов от его участка распознавания на другой цепи. См., например, патенты США 5356802; 5436150 и 5487994; а также Li et al. (1992) Proc. Natl. Acad. Sci. USA 89:4275-4279; Li et al. (1993) Proc. Natl. Acad. Sci. USA 90:2764-2768; Kim et al. (1994a) Proc. Natl. Acad. Sci. USA 91:883-887; Kim et al. (1994b) J. Biol. Chem. 269:31.978-31.982. Таким образом, в одном варианте осуществления слитые белки содержат домен расщепления (или половинный домен расщепления) из по меньшей мере одного фермента рестрикции типа IIS и одного или нескольких связывающих доменов с цинковыми пальцами, которые могут быть модифицированными способами инженерии или могут не быть модифицированными.
Иллюстративным ферментом рестрикции типа IIS, домен расщепления которого является отделимым от домена связывания, является FokI. Этот конкретный фермент активен в качестве димера. Bitinaite et al. (1998) Proc. Natl Acad. Sci. USA 95: 10.570-10.575. Таким образом, для целей настоящего изобретения часть фермента FokI, используемая в описанных слитых белках, считается половинным доменом расщепления. Таким образом, для направленного двухцепочечного расщепления и/или направленной замены клеточных последовательностей с использованием слитых конструкций цинковый палец-fokI, можно использовать два слитых белка, каждый из которых содержит половинный домен расщепления FokI, чтобы восстановить каталитически активный домен расщепления. Альтернативно также можно использовать одну полипептидную молекулу, содержащую ДНК-связывающий домен и два половинных домена расщепления FokI.
Домен расщепления или половинный домен расщепления может представлять собой любую часть белка, которая сохраняет активность расщепления или которая сохраняет способность к мультимеризации (например, димеризации) с образованием функционального домена расщепления.
Иллюстративные ферменты рестрикции типа IIS описаны в публикации патента США № 20070134796, включенной в настоящее описание в полном объеме. Дополнительные ферменты рестрикции также содержат разделимые домены связывания и расщепления, и они охватываются настоящем изобретением. См., например, Roberts et al. (2003) Nucleic Acids Res. 31:418-420.
В определенных вариантах осуществления домен расщепления содержит один или несколько модифицированных способами инженерии половинных доменов расщепления (также обозначаемых как мутанты домена димеризации), которые минимизируют или предотвращают гомодимеризацию, как описано, например, в публикациях патентов США № 20050064474; 20060188987 и 20080131962, содержание всех из которых включено в настоящее описание в качестве ссылок в полном объеме. Все из аминокислотных остатков в положениях 446, 447, 479, 483, 484, 486, 487, 490, 491, 496, 498, 499, 500, 531, 534, 537 и 538 FokI являются мишенями для влияния на димеризацию половинных доменов расщепления FokI.
Иллюстративные модифицированные способами инженерии половинные домены FokI, которые обязательно образуют гетеродимеры, включают пару, в которой первый половинный домен расщепления включает мутации в аминокислотных остатках в положениях 490 и 538 FokI и второй половинный домен расщепления включает мутации в аминокислотных остатках 486 и 499.
Таким образом, в одном варианте осуществления мутация в положении 490 представляет собой замену Glu (E) на Lys (K); мутация в положении 538 представляет собой замену Iso (I) на Lys (K); мутация в положении 486 представляет собой замену Gln (Q) на Glu (E); и мутация в положении 499 представляет собой замену Iso (I) на Lys (K). В частности, модифицированные способами инженерии половинные домены расщепления, описанные в настоящем описании, получали путем внесения мутаций в положения 490 (E→K) и 53 (I→K) в одном половинном домене расщепления с получением модифицированного способами инженерии половинного домена, обозначаемого как "E490K:I538K", и путем мутации положений 486 (Q→E) и 499 (I→L) в другом половинном домене расщепления с получением модифицированного способами инженерии половинного домена, обозначаемого как "Q486E:I499L". Модифицированные способами инженерии половинные домены, описанные в настоящем описании, представляют собой обязательно гетеродимерные мутанты, в которых аберрантное расщепление минимизировано или устранено. См., например, публикацию патента США № 2008/0131962, содержание которой включено в настоящее описание в качестве ссылки в полном объеме для любых целей.
В определенных вариантах осуществления модифицированный способами инженерии половинный домен расщепления содержит мутации в положениях 486, 499 и 496 (пронумерованы относительно FokI дикого типа), например, мутации, которые заменяют остаток Gln (Q) дикого типа в положении 486 на остаток Glu (E), остаток Iso (I) дикого типа в положении 499 на остаток Leu (L) и остаток Asn (N) дикого типа в положении 496 на остаток Asp (D) или Glu (E) (также обозначаемые как домены "ELD" и "ELE", соответственно). В других вариантах осуществления модифицированный способами инженерии половинный домен расщепления содержит мутации в положениях 490, 538 и 537 (пронумерованы относительно FokI дикого типа), например, мутации, которые заменяют остаток Glu (E) дикого типа в положении 490 на остаток Lys (K), остаток Iso (I) дикого типа в положении 538 на остаток Lys (K), и остаток His (H) дикого типа в положении 537 на остаток Lys (K) или остаток Arg (R) (также обозначаемые как домены "KKK" и "KKR", соответственно). В других вариантах осуществления модифицированный способами инженерии половинный домен расщепления содержит мутации в положениях 490 и 537 (пронумерованы относительно FokI дикого типа), например, мутации, которые заменяют остаток Glu (E) дикого типа в положении 490 на остаток Lys (K) и остаток His (H) дикого типа в положении 537 на остаток Lys (K) или остаток Arg (R) (также обозначаемые как домены "KIK" и "KIR", соответственно). (См. публикацию патента США № 20110201055). Модифицированные способами инженерии половинные домены расщепления, описанные в настоящем описании, можно получать с использованием любого подходящего способа, например, сайт-направленного мутагенеза половинных доменов расщепления дикого типа (FokI), как описано в публикациях патентов США № 20050064474; 20080131962 и 20110201055.
Альтернативно нуклеазы можно собирать in vivo в участке-мишени нуклеиновой кислоты с использованием так называемой технологии "разделенного фермента" (см., например, публикацию патента США № 20090068164). Компоненты таких разделенных ферментов можно экспрессировать либо на отдельных экспрессируюющих конструкциях, либо их можно связывать в одну открытую рамку считывания, где индивидуальные компоненты разделены, например, саморасщепляющимся пептидом 2A или последовательностью IRES. Компоненты могут представлять собой индивидуальные связывающие домены цинковых пальцев или домены связывающего нуклеиновую кислоту домена мегануклеазы.
C. Нуклеазы с цинковыми пальцами
В конкретных вариантах осуществления химерный полипептид представляет собой специально сконструированную нуклеазу с цинковыми пальцами (ZFN), которая может быть сконструирована для обеспечения направленного сайт-специфического разрыва двухцепочечной ДНК, в которую может быть встроена экзогенная нуклеиновая кислота или донорная ДНК (см. публикацию патента США 20100257638 того же заявителя, включенную в настоящее описание в качестве ссылки). ZFN представляют собой химерные полипептиды, содержащие домен неспецифического расщепления из эндонуклеазы рестрикции (например, FokI) и полипептид ДНК-связывающего домена с цинковыми пальцами. См., например, Huang et al. (1996) J. Protein Chem. 15:481-9; Kim et al. (1997a) Proc, Natl. Acad. Sci. USA 94:3616-20; Kim et al. (1996) Proc. Natl. Acad. Sci. USA 93:1156-60; Kim et al. (1994) Proc Natl. Acad. Sci. USA 91:883-7; Kim et al. (1997b) Proc. Natl. Acad. Sci. USA 94:12875-9; Kim et al. (1997c) Gene 203:43-9; Kim et al. (1998) Biol. Chem. 379:489-95; Nahon and Raveh (1998) Nucleic Acids Res. 26:1233-9; Smith et al. (1999) Nucleic Acids Res. 27:674-81. В некоторых вариантах осуществления ZFN содержат ДНК-связывающие домены с неканоническими цинковыми пальцами (см. публикацию патента США 20080182332 того же заявителя, включенную в настоящее описание в качестве ссылки). Эндонуклеаза рестрикции FokI должна димеризоваться через нуклеазный домен, чтобы расщепить ДНК и внести двухцепочечный разрыв. Следовательно, для ZFN, содержащих нуклеазный домен из такой эндонуклеазы, также требуется димеризация нуклеазного домена, чтобы расщепить ДНК-мишень. Mani et al. (2005) Biochem. Biophys. Res. Commun. 334:1191-7; Smith et al. (2000) Nucleic Acids Res. 28:3361-9. Димеризации ZFN могут способствовать два соседних противоположно ориентированных ДНК-связывающих участка. Там же.
Приспособляемость и специфичность системы ZFN обеспечивает уровень контроля, ранее не достижимый с помощью известных стратегий опосредуемого рекомбиназой редактивования генов. В качестве одного примера, ZFN можно без труда модифицировать способами инженерии так, например, чтобы она распознавала определенные последовательности нуклеиновой кислоты. Wu et al. (2007) Cell. Mol. Life Sci. 64:2933-44 (cм. публикации патентов США 20090205083, 20110189775, 20110167521 и 20100199389, включенные в настоящее описание в качестве ссылок в полном объеме). Рандомизация кодонов для остатков распознавания цинковых пальцев позволяет селекцию новых пальцев, которые обладают высокой аффинностью в отношении произвольно выбранных последовательностей ДНК. Более того, цинковые пальцы представляют собой природные ДНК-связывающие молекулы, и было показано, что модифицированные способами инженерии цинковые пальцы действуют в живых клетках на их намеченные мишени. Таким образом, нуклеазы на основе цинковых пальцев могут быть нацелены на конкретные, но произвольные, участки распознавания.
В конкретных примерах способ сайт-специфического встраивания экзогенной нуклеиновой кислоты по меньшей мере в один локус функциональности FAD2 хозяина включает введение в клетку-хозяина ZFN, где ZFN распознает и связывает нуклеотидную последовательность-мишень, где нуклеотидная последовательность-мишень содержится по меньшей мере в одном локусе FAD2 хозяина. В определенных примерах нуклеотидная последовательность-мишень не содержится в геноме хозяина в каком-либо положении, отличном от по меньшей мере одного локуса FAD2. Например, ДНК-связывающий полипептид ZFN можно модифицировать способами инженерии так, чтобы он распознавал и связывался с нуклеотидной последовательностью-мишенью, идентифицированной по меньшей мере в одном локусе FAD2 (например, путем секвенирования локуса FAD2). Способ сайт-специфического встраивания экзогенной нуклеиновой кислоты по меньшей мере в один локус функциональности FAD2 хозяина, который включает введение в клетку хозяина ZFN, также может включать введение в клетку экзогенной нуклеиновой кислоты, где встраивание путем рекомбинации экзогенной нуклеиновой кислоты в нуклеиновую кислоту хозяина, содержащего по меньшей мере один локус FAD2, облегчается сайт-специфическим распознаванием и связыванием ZFN с последовательностью-мишенью (и последующим расщеплением нуклеиновой кислоты, содержащей локус FAD2).
V. Экзогенные нуклеиновые кислоты для встраивания в локус FAD2
Варианты осуществления изобретения могут включать одну или несколько нуклеиновых кислот, выбранных из группы, состоящей из: экзогенной нуклеиновой кислоты для сайт-специфического встраивания по меньшей мере в один локус FAD2, например, но не ограничиваясь этим, PTU, ELP, ΕΤIΡ или ORF; нуклеиновой кислоты, содержащей нуклеотидную последовательность, кодирующую нацеливающую эндонуклеазу; и вектор, содержащий по меньшей мере любую одну или обе из указанных выше. Таким образом, конкретные нуклеиновые кислоты для применения в некоторых вариантах осуществления включают нуклеотидные последовательности, кодирующие полипептид, структурные нуклеотидные последовательности и/или участки распознавания и связывания ДНК-связывающего полипептида.
A. Экзогенные молекулы нуклеиновых кислот для сайт-специфического встраивания
Как отмечалось выше, встраивание экзогенной последовательности (также называемой "донорной последовательностью" или "донором" или "трансгеном") предусматривается, например, для экспрессии полипептида, коррекции мутантного гена или увеличенной экспрессии гена дикого типа. Будет хорошо понятно, что донорная последовательность, как правило, не является идентичной геномной последовательности, в которую ее помещают. Донорная последовательность может содержать негомологичную последовательность, фланкированную двумя областями гомологии для обеспечения эффективной HDR в представляющем интерес положении. Кроме того, донорные последовательности могут содержать векторную молекулу, содержащую последовательности, которые не являются гомологичными представляющей интерес области в клеточном хроматине. Донорная молекула может содержать несколько прерывающихся областей гомологии с клеточным хроматином. Например, для направленного встраивания последовательностей, обычно не присутствующих в представляющей интерес области, указанные последовательности могут присутствовать в донорной молекуле нуклеиновой кислоты и могут фланкироваться областями гомологии с последовательностью в представляющей интерес области.
Донорный полинуклеотид может представлять собой ДНК или РНК, может быть одноцепочечным или двухцепочечным, и может быть введен в клетку в линейной или кольцевой форме. См., например, публикации патентов США № 20100047805, 20110281361, 20110207221 и заявку США № 13/889162. В случае введения в линейной форме, концы донорной последовательности могут быть защищены (например, от деградации экзонуклеазой) способами, известными специалистам в данной области. Например, один или несколько дидезоксинуклеотидных остатков добавляют на 3'-конец линейной молекулы и/или с одним или обоими концами лигируют самокомплементарные олигонуклеотиды. См., например, Chang et al. (1987) Proc. Natl. Acad. Sci. USA 84:4959-4963; Nehls et al. (1996) Science 272:886-889. Дополнительные способы защиты экзогенных полинуклеотидов от деградации включают, но не ограничиваются ими, присоединение концевой аминогруппы(аминогрупп) и использование модифицированных межнуклеотидных связей, например, таких как фосфоротиоаты, фосфорамидаты и остатки O-метилрибозы или дезоксирибозы.
Полинуклеотид можно вводить в клетку в качестве части молекулы вектора, имеющей дополнительные последовательности, например, такие как ориджины репликации, промоторы и гены, кодирующие устойчивость к антибиотикам. Более того, донорные полинуклеотиды можно вводить в качестве голой нуклеиновой кислоты, в качестве нуклеиновой кислоты в комплексе с агентом, таким как липосома или полоксамер, или их можно доставлять с помощью вирусов (например, аденовирус, AAV, вирус герпеса, ретровирус, лентивирус и лентивирус с дефектом интегразы (IDLV)).
Донор обычно встраивают так, чтобы его экспрессия контролировалась эндогенным промотором в участке связывания, а именно, промотором, который контролирует экспрессию эндогенного гена, в который встраивают донор (например, FAD2). Однако будет понятно, что донор может содержать промотор и/или энхансер, например конститутивный промотор или индуцибельный или тканеспецифический промотор.
Более того, хотя это и не требуется для экспрессии, экзогенные последовательности также могут включать последовательности контроля транскрипции или трансляции, например, промоторы, энхансеры, инсуляторы, участки внутренней посадки рибосомы, последовательности, кодирующие пептиды 2A, и/или сигналы полиаденилирования.
Экзогенные нуклеиновые кислоты, которые можно встраивать сайт-специфическим образом по меньшей мере в один локус FAD2, так чтобы модифицировать локус FAD2, в вариантах осуществления включают, например, но не ограничиваясь ими, нуклеиновые кислоты, содержащие нуклеотидную последовательность, кодирующую представляющий интерес полипептид; нуклеиновые кислоты, содержащие агрономический ген; нуклеиновые кислоты, содержащие нуклеотидную последовательность, кодирующую молекулу РНК-i; или нуклеиновые кислоты, которые разрушают ген FAD2.
В некоторых вариантах осуществления экзогенная нуклеиновая кислота встраивается в локус FAD2 так, что локус FAD2 модифицируется, где нуклеиновая кислота содержит агрономический ген или нуклеотидную последовательность, кодирующую представляющий интерес полипептид, так что агрономический ген или нуклеотидная последовательность экспрессируются в хозяине с локуса FAD2. В некоторых примерах представляющий интерес полипептид (например, чужеродный белок) экспрессируется с нуклеотидной последовательности, кодирующей представляющий интерес полипептид, в коммерческих количествах. В таких примерах представляющий интерес полипептид можно экстрагировать из клетки-хозяина, ткани или биомассы. В некоторых вариантах осуществления хозяином является растение, и растительный материал, предоставляемый для коммерческого продуцирования представляющего интерес полипептида, может представлять собой растение, часть растения, ткань растения или клетку растения. В некоторых примерах часть растения может представлять собой семя растения. Экстракцию белка из растительной биомассы можно проводить известными способами, которые рассмотрены, например, в Heney and Orr (1981) Anal. Biochem. 114:92-6.
Аналогично, агрономические гены можно экспрессировать в трансформированных клетках растений, растениях и/или их потомстве. Например, согласно конкретным вариантам осуществления растение можно модифицировать способами генной инженерии для экспрессии различных представляющих агрономический интерес фенотипов по меньшей мере с одного локуса FAD2.
В некоторых вариантах осуществления нуклеиновые кислоты, содержащие агрономический ген или нуклеотидную последовательность, кодирующую представляющий интерес полипептид, могут включать, например, но не ограничиваясь ими: ген, который сообщает устойчивость к паразитам или заболеванию (см., например, Jones et al. (1994) Science 266:789 (клонирование гена томата Cf-9 для устойчивости к Cladosporium fiilvum); Martin et al. (1993) Science 262:1432; Mindrinos et al. (1994) Cell 78:1089 (ген RSP2 для устойчивости к Pseudomonas syringae); международную публикацию патента PCT № WO 96/30517 (устойчивость к нематодам кист сои); международную публикацию патента PCT № WO 93/19181); ген, который кодирует белок Bacillus thuringiensis, его производное или смоделированный на его основе синтетический полипептид (см., например, Geiser et al. (1986) Gene 48:109 (клонирование и нуклеотидная последовательность гена δ-эндотоксина Bt; более того, молекулы ДНК, кодирующие гены δ-эндотоксина, могут быть приобретены от American Type Culture Collection (Manassas, VA), например, под номерами доступа ATCC № 40098; 67136; 31995 и 31998)); ген, который кодирует лектин (см., например, Van Damme et al. (1994) Plant Molec. Biol. 24:25 (нуклеотидные последовательности нескольких генов маннозосвязывающих лектинов Clivia miniata)); ген, который кодирует витамин-связывающий белок, например, авидин (см. международную публикацию патента PCT № US93/06487 (применение авидина и гомологов авидина в качестве ларвицидов против паразитов насекомых)); ген, который кодирует ингибитор фермента, например, ингибитор протеазы или ингибитор амилазы (см., например, Abe et al. (1987) J. Biol. Chem. 262:16793 (нуклеотидная последовательность ингибитора цистеиновой протеазы риса); Huub et al. (1993) Plant Molec. Biol. 21: 985 (нуклеотидная последовательность кДНК, кодирующей ингибитор протеаз табака I); Sumitani et al. (1993) Biosci. Biotech. Biochem. 57:1243 (нуклеотидная последовательность ингибитора альфа-амилазы Streptomyces nitrosporeus) и патент США 5494813); ген, кодирующий специфичный к насекомым гормон или феромон, например, экдистероид или ювенильный гормон, их вариант, миметик на их основе или их антагонист или агонист (см., например, Hammock et al. (1990) Nature 344:458 (бакуловирусная экспрессия клонированного ювенильного гормона эстеразы, инактивация ювенильного гормона)); ген, кодирующий специфичный к насекомым пептид или нейропептид, который при экспрессии нарушает физиологию пораженного паразита (см., например, Regan (1994) J. Biol. Chem. 269:9 (клонирование для экспрессии приводит к ДНК, кодирующей рецептор диуретического гормона насекомых); Pratt et al. (1989) Biochem. Biophys. Res. Comm. 163:1243 (аллостатин в Diploptera puntata); и патент США 5266317 (гены, кодирующие специфические к насекомым паралитические нейротоксины)); ген, кодирующий специфический для насекомых яд, продуцируемый в природе змеями, пчелами или другим организмом (см., например, Pang et al. (1992) Gene 116:165 (гетерологичная экспрессия в растениях гена, кодирующего инсектотоксический пептид скорпиона)); ген, кодирующий фермент, ответственный за избыточное накопление монотерпенов, сесквитерпенов, стероидов, гидроксамовой кислоты, фенилпропаноидного производного или другой молекулы с инсектицидной активностью; ген, кодирующий фермент, вовлеченный в модификацию, в том числе в пострансляционную модификацию, биологически активной молекулы, например, a гликолитического фермента, протеолитического фермента, липолитического фермента, нуклеазы, циклазы, трансаминазы, эстеразы, гидролазы, фосфатазы, киназы, фосфорилазы, полимеразы, эластазы, хитиназы или глюканазы, как природных, так и синтетических (см., например, международную публикацию патента PCT № WO 93/02197 (нуклеотидная последовательность гена каллазы); более того, молекулы ДНК, содержащие кодирующие хитиназу последовательности, можно получать, например, от ATCC, под номерами доступа № 39637 и 67152; Kramer et al. (1993) Insect Biochem. Molec. Biol. 23:691 (нуклеотидная последовательность кДНК, кодирующей хитиназу бражника табака); и Kawalleck et al. (1993) Plant Molec. Biol. 21: 673 (нуклеотидная последовательность гена полиубиквитина петрушки ubi4-2)); ген, кодирующий молекулу, которая стимулирует передачу сигнала (см., например, Botella et al. (1994) Plant Molec. Biol. 24:757 (нуклеотидные последовательности для клонов кДНК кальмодулина фасоли золотистой); и Griess et al. (1994) Plant Physiol. 104:1467 (нуклеотидная последовательность клона кДНК кальмодулина кукурузы)); ген, который кодирует пептид гидрофобного момента (см., например, международную публикацию патента PCT № WO 95/16776 (пептидные производные тахиплесина, который ингибирует грибковые патогены растений); и международную публикацию патента PCT № WO 95/18855 (синтетические противомикробные пептиды, которые сообщают устойчивость к заболеваниям)); ген, который кодирует мембранную пермеазу, формирователь канала или блокатор канала (см., например, Jaynes et al. (1993) Plant Sci 89:43 (гетерологичная экспрессия аналога литического пептида цекропин-β для сообщения трансгенным растениям табака устойчивости к Pseudomonas solanacearum)); ген, который кодирует белок вирусной инвазии или комплексный токсин, происходящий из него (См., например, Beachy et al. (1990) Ann. rev. Phytopathol. 28:451); ген, который кодирует специфичное к насекомым антитело или иммунотоксин, происходящий из него (cм., например, Taylor et al., Abstract #497, Seventh Int'l Symposium on Molecular Plant-Microbe Interactions (Echnburgh, Scotland) (1994) (ферментативная инактивация трансгенного табака путем продуцирования одноцепочечных фрагментов антител)); ген, кодирующий специфичное к вирусу антитело (см., например, Tavladoraki etal. (1993) Nature 366:469 (трансгенные растения, экспрессирующие рекомбинантные гены антител, защищены от вирусной атаки)); ген, кодирующий останавливающий развитие белок, продуцируемый в природе патогеном или паразитом (См., например, Lamb et al. (92) Bio/Technology 10:1436 (грибковые эндо α-1,4-D-полигалактуроназы способствуют колонизации грибков и высвобождению питательных веществ растений путем солюбилизации гомо-α-1,4-D-галактуроназы клеточной стенки растений); Toubart et al. (1992) Plant J. 2:367 (клонирование и охарактеризация гена, который кодирует белок, ингибирующий эндополигалактуроназу)); ген, кодирующий останавливающий развитие белок, продуцируемый в природе растением (см., например, Logemann et al. (1992) Bio/Technology 10:305 (трансгенные растения, экспрессирующие ген, инактивирующий рибосомы, ячменя, обладают увеличенной устойчивостью к грибковым заболеваниям)).
В некоторых вариантах осуществления нуклеиновые кислоты, содержащие агрономический ген или нуклеотидную последовательность, кодирующую представляющий интерес полипептид, также и/или альтернативно могут включать, например, но не ограничиваясь этим: гены, которые сообщаю устойчивость к гербициду, такому как гербицид, который ингибирует конус нарастания или меристему, например, имидазолинону или сульфонилмочевине (иллюстративные гены в этой категории включают мутантные ферменты ALS и AHAS, как описано, например, Lee et al. (1988) EMBO J. 7:1241, и Miki et al. (1990) Theor. Appl. Genet. 80:449, соответственно); устойчивость к глифосату, сообщаемую, например, мутантными генами 5-енолпирувилшикимат-3-фосфатсинтазы (EPSP) (посредством введения рекомбинантных нуклеиновых кислот и/или различных форм мутагенеза нативных генов EPSP in vivo (включая, но не ограничиваясь ими, CP4, DMMG и DGT-28); гены aroA и гены глифосатацетилтрансферазы (GAT), соответственно); гены устойчивости к другим фосфоносоединениям, такие как гены глюфосинатфосфинотрицинацетилтрансферазы (PAT) из видов Streptomyces, включая Streptomyces hygroscopicus и Streptomyces viridichromogenes); и гены устойчивости к пиридинокси- или феноксипропионовым кислотам и циклогексонам (гены, кодирующие ингибитор ACC-азы). См., например, патенты США 4940835 и 6248876 (нуклеотидные последовательности форм EPSP, которые могут сообщать растению устойчивость к глифосату). Молекулу DNA, кодирующую мутантный ген aroA, можно получать под номером доступа ATCC 39256. Также см. патент США № 4769061 (нуклеотидная последовательность мутантного гена aroA). В патентной заявке Европы № 0333033 и в патенте США № 4975374 описаны нуклеотидные последовательности генов глутаминсинтетазы, которые могут сообщать устойчивость к гербицидам, таким как L-фосфинотрицин. Нуклеотидные последовательности иллюстративных генов PAT представлены в заявке Европы № 0 242 246 и DeGreef et al. (1989) Bio/Technology 7:61 (получение трансгенных растений, которые экспрессируют химерные гены bar, кодирующие активность PAT). Примеры генов, сообщающих устойчивость к феноксипропионовым кислотам и циклогексонам, таким как сетоксидим и галоксифоп, включают гены Accl-S1, Accl-S2 and Accl-S3, описанные Marshall et al. (1992) Theor. Appl. Genet. 83:435. Гены GAT, способные сообщать устойчивость к глифосату, описаны, например, в WO 2005012515. Гены, сообщающие устойчивость к 2,4-D-феноксипропионовой кислоте и пиридилоксиауксиновым гербицидам, описаны, например, в WO 2005107437 и WO 2007053482.
Нуклеиновые кислоты, содержащие агрономический ген, или нуклеотидная последовательность, кодирующая представляющий интерес полипептид, также могут включать, например, не ограничиваясь ими: ген, сообщающий устойчивость к гербициду, который ингибирует фотосинтез, такому как триазин (гены psbA и gs+) или бензонитрилу (ген нитрилазы). См., например, Przibila et al. (1991) Plant Cell 3:169 (трансформация Chlamydomonas плазмидами, кодирующими мутантные гены psbA). Нуклеотидные последовательности для генов нитрилазы описаны в патенте США 4810648, и молекулы ДНК, содержащие эти гены, доступны под номерами доступа ATCC № 53435; 67441 и 67442. Также см. Hayes et al. (1992) Biochem. J. 285:173 (клонирование и экспрессия ДНК, кодирующей глутатион-S-трансферазу).
В некоторых вариантах осуществления нуклеиновые кислоты, содержащие агрономический ген или нуклеотидную последовательность, кодирующую представляющий интерес полипептид, также и/или альтернативно могут включать гены, которые сообщают или вносят вклад в дополнительный ценный признак, например, но не ограничиваясь этим: модифицированный метаболизм жирных кислот, например, путем трансформации растения антисмысловым геном стеарил-ACP-десатуразы для увеличения содержания стеариновой кислоты в растении (см., например, Knultzon et al. (1992) Proc. Natl. Acad. Sci. U.S.A. 89:2624); снижение содержания фитатов, например, введение гена, кодирующего фитазу, может усиливать разрушение фитатов, увеличивая содержание свободного фосфата в трансформированном растении (см., например, Van Hartingsveldt et al. (1993) Gene 127:87 (нуклеотидная последовательность гена фитазы Aspergillus niger); можно вводить ген для снижения содержания фитатов в кукурузе, например, это можно осуществлять путем клонирования, а затем повторного введения ДНК, ассоциированной с единичным аллелем, который может быть ответственен за мутанты кукурузы, характерзиующиеся низкими уровнями фитиновой кислоты (см. Raboy et al. (1990) Maydica 35:383)); и модифицированный углеводный состав, обеспечиваемый, например, путем трансформации растений геном, кодирующим фермент, который изменяет характер ветвления крахмала (см., например, Shiroza et al. (1988) J. Bacteol. 170:810 (нуклеотидная последовательность мутантного гена фруктозилтрансфераза Streptococcus); Steinmetz et al. (1985) Mol. Gen. Genet. 20:220 (ген левансукразы); Pen et al. (1992) Bio/Technology 10:292 (α-амилаза); Elliot et al. (1993) Plant Molec. Biol. 21: 515 (нуклеотидные последовательности генов инвертазы томата); Sogaard et al. (1993) J. Biol. Chem. 268:22480 (ген α-амилазы ячменя); и Fisher et al. (1993) Plant Physiol. 102:1045 (фермент II ветвления крахмала эндосперма кукурузы)).
В некоторых вариантах осуществления экзогенная нуклеиновая кислота встраивается в локус FAD2, модифицируя локус FAD2, где нуклеиновая кислота содержит PTU или ELP, чтобы, например, способствовать последующему сайт-специфическому встраиванию второй экзогенной нуклеиновой кислоты в участке PTU или ELP. Также см. заявку США № 13/889162.
Обеспечение опосредуемого эндонуклеазой встраивания представляющей интерес молекулы нуклеиновой кислоты в геном растений посредством направленного встраивания требует доставки нацеливающих эндонуклеаз или кодирующих нацеливающие эндонуклеазы молекул нуклеиновой кислоты с последующей экспрессией функционального нацеливающего белка эндонуклеазы в хозяине. Экзогенная нуклеиновая кислота также предпочтительно присутствует в клетке хозяине в то время, когда доставляют и экспрессируют в ней нацеливающую эндонуклеазу, так что функциональный нацеливающий белок эндонуклеазы индуцирует двухцепочечные разрывы в участке(ах)-мишени по меньшей мере в одном локусе FAD2, которые затем репарируются, например, посредством опосредуемого гомологией встраивания экзогенной нуклеиновой кислоты в этот локус. Специалисту в данной области будет понятно, что экспрессия функционального нацеливающего белка эндонуклеазы может быть достигнута несколькими способами, включая, но не ограничиваясь ими, трансгенез нацеливающей кодирующей эндонуклеазу конструкции и временную экспрессию нацеливающей кодирующей эндонуклеазу конструкции. В обоих из этих случаев экспрессию функционального нацеливающего белка эндонуклеазы и доставку экзогенной нуклеиновой кислоты в клетку-хозяина можно осуществлять одновременно, чтобы обеспечить направленное встраивание в локус FAD2.
Конкретным преимуществом, достигаемым в вариантах осуществления с использованием ZFN в качестве нацеливающих эндонуклеаз, является то, что необходимость в димеризации доменов расщепления химерных нуклеаз с цинковыми пальцами обеспечивает высокий уровень специфичности к последовательности, и, таким образом, расщепления. Поскольку каждый набор из трех пальцев связывает девять последовательно расположенных пар оснований, двум химерным нуклеазам для эффективности требуется мишень размером 18 п.н., если каждый домен цинкового пальца обладает абсолютной специфичностью. Согласно прогнозам, любая данная последовательность этой длины является уникальной в одном геноме (предполагая приблизительно 109 п.н.). Bibikova et al. (2001) Mol. Cell. Biol. 21(1):289-97; Wu et al. (2007), выше. Более того, дополнительные пальцы могут обеспечить увеличение специфичности, Beerli et al. (1998) Proc. Natl. Acad. Sci. USA 95: 14628-33; Kim and Pabo (1998) Proc. Natl. Acad. Sci. USA 95:2812-7; Liu et al. (1997) Proc. Natl. Acad. Sci. USA 94:5525-30, так что количество цинковых пальцев в каждом ДНК-связывающем домене может быть увеличено для обеспечения еще большей специфичности. Например, специфичность может быть далее увеличена с использованием пары ZFN с 4-, 5-, 6- или более пальцами, которая распознает последовательность из 24 п.н. Urnov et al. (2005) Nature 435:646-51. Таким образом, ZFN можно использовать так, чтобы распознаваемая последовательность при введении в геном растения-хозяина была уникальной в геноме.
B. Молекулы нуклеиновой кислоты, содержащие нуклеотидную последовательность, кодирующую нацеливающую эндонуклеазу
В некоторых вариантах осуществления нуклеотидную последовательность, кодирующую нацеливающую эндонуклеазу, можно модифицировать способами инженерии путем манипулирования (например, лигирования) нативными нуклеотидными последовательностями, кодирующими полипептиды, содержащиеся в нацеливающей эндонуклеазе. Например, нуклеотидную последовательность гена, кодирующего белок, содержащий ДНК-связывающий полипептид, можно исследовать для идентификации нуклеотидной последовательности гена, которая соответствует ДНК-связывающему полипептиду, и эту нуклеотидную последовательность можно использовать в качестве элемента нуклеотидной последовательности, кодирующей нацеливающую эндонуклеазу, содержащую ДНК-связывающий полипептид. Альтернативно аминокислотную последовательность нацеливающей эндонуклеазы можно использовать для установления нуклеотидной последовательности, кодирующей нацеливающую эндонуклеазу, например, исходя из вырожденности генетического кода.
В иллюстративных молекулах нуклеиновой кислоты, содержащих нуклеотидную последовательность, кодирующую нацеливающую эндонуклеазу, последний кодон первой полинуклеотидной последовательности, кодирующей полипептид нуклеазы, и первый кодон второй полинуклеотидной последовательности, кодирующей ДНК-связывающий полипептид, могут быть разделены любым количеством нуклеотидных триплетов, например, без кодирования интрона или "стоп-кодона". Аналогично, последний кодон нуклеотидной последовательности, кодирующей первую полинуклеотидную последовательность, кодирующую ДНК-связывающий полипептид, и первый кодон второй полинуклеотидной последовательности, кодирующей полипептид нуклеазы, могут быть разделены любым количеством нуклеотидных триплетов. В этих и других вариантах осуществления последний кодон последней из (т.е. самый 3' в последовательности нуклеиновой кислоты) первой полинуклеотидной последовательности, кодирующей полипептид нуклеазы, и второй полинуклеотидной последовательности, кодирующей ДНК-связывающий полипептид, может быть слит в рамке считывания с первым кодоном следующего полинуклеотида, кодирующего последовательность, непосредственно соседним или отделенным от них не более чем короткой пептидной последовательностью, такой как последовательность, кодируемая синтетическим нуклеотидным линкером (например, нуклеотидный линкер, который можно использовать для обеспечения слияния). Примеры таких следующих полинуклеотидных последовательностей включают, например, но не ограничиваются ими, метки, нацеливающие пептиды и участки ферментативного расщепления. Аналогично, первый кодон наиболее 5' (в последовательности нуклеиновой кислоты) из первой и второй полинуклеотидной последовательности может быть слит в рамке считывания с последним кодоном следующего полинуклеотида, кодирующего последовательность, непосредственно соседним или отделенным от них не более чем короткой пептидной последовательностью.
Последовательность, разделяющая полинуклеотидные последовательности, кодирующие функциональные полипептиды в нацеливающей эндонуклеазе (например, ДНК-связывающий полипептид и полипептид нуклеазы) может, например, включать любую последовательность, так чтобы было вероятно, что кодируемая аминокислотная последовательность не изменит значительно трансляцию нацеливающей эндонуклеазы. Вследствие автономной природы известных полипептидов нуклеаз и известных ДНК-связывающих полипептидов, встроенные последовательности не препятствуют, например, соответствующим функциям этих структур.
C. Векторы и экспрессирующие конструкции
В некоторых вариантах осуществления по меньшей мере одну молекулу(ы) нуклеиновой кислоты, содержащую по меньшей мере одну экзогенную полинуклеотидную последовательность, кодирующую представляющий интерес полипептид и/или нацеливающую эндонуклеазу, можно вводить в клетку, ткань или организм для экспрессии в них. Например, молекулу нуклеиновой кислоты, содержащую полинуклеотидную последовательность, кодирующую нацеливающую эндонуклеазу, которая специфически распознает нуклеотидную последовательность, содержащуюся по меньшей мере в одном локусе FAD2, можно вводить в клетку для экспрессии нацеливающей эндонуклеазы, и молекулу нуклеиновой кислоты, содержащую полинуклеотидную последовательность, кодирующую представляющий интерес полипептид, можно вводить в клетку, так чтобы полинуклеотидная последовательность, кодирующая представляющий интерес полипептид, встраивалась по меньшей мере в один локус FAD2, например, путем гомологичной рекомбинации, с последующим внесением двухцепочечного разрыва в локус экспрессируемой нацеливающей эндонуклеазой, и чтобы с встроенной полинуклеотидной последовательности экспрессировался представляющий интерес полипептид.
В некоторых вариантах осуществления молекула нуклеиновой кислоты, такая как одна из указанных выше молекул, может представлять собой, например, векторную систему, включающую, например, но не ограничивающуюся этим, линейную плазмиду или замкнутую кольцевую плазмиду. В конкретных примерах вектор может представлять собой экспрессирующий вектор. Последовательности нуклеиновых кислот согласно конкретным вариантам осуществления можно, например, встраивать в вектор, так чтобы последовательность нуклеиновой кислоты была функционально связана с одной или несколькими регулирующими последовательностями. Для этой цели доступно множество векторов, и выбор конкретного вектора может зависеть, например, от размера нуклеиновой кислоты, подлежащей встраиванию в вектор, конкретной клетки-хозяина, подлежащей трансформации вектором, и/или количества любого кодируемого полипептида, экспрессия которого является желательной. Вектор, как правило, содержит различные компоненты, тип которых зависит от функции вектора (например, амплификация ДНК или экспрессия ДНК) и конкретной клетки(ок)-хозяина, с которой вектор совместим.
В некоторых вариантах осуществления регуляторная последовательность, функционально связанная с одной или несколькими кодирующей последовательностью(ями), может представлять собой промоторную последовательность, которая функционирует в клетке-хозяине, такой как бактериальная клетка, клетка водорослей, клетка грибов или клетка растений, в которой молекула нуклеиновой кислоты подлежит амплификации или экспрессии. Некоторые варианты осуществления могут включать вектор для трансформации растений, который содержит нуклеотидную последовательность, содержащую по меньшей мере одну регуляторную последовательность, функционально связанную с одной или несколькими нуклеотидной последовательностью(ями), кодирующей представляющий интерес полипептид или нацеливающую эндонуклеазу, где одна или несколько нуклеотидная последовательность(ей) могут экспрессироваться под контролем регуляторной последовательности(ей), в клетке, ткани или организме растений, продуцируя представляющий интерес полипептид или нацеливающую эндонуклеазу.
Промоторы, пригодные для применения в молекулах нуклеиновой кислоты согласно некоторым вариантам осуществления, включают промоторы, которые являются индуцибельными, тканеспецифическими, вирусными, синтетическими или конститутивными, все из которых хорошо известны в данной области. Неограничивающие примеры промоторов, которые могут быть пригодными в вариантах осуществления изобретения, описаны в: патентах США № 6437217 (промотор RS81 кукурузы); 5641876 (промотор актина риса); 6426446 (промотор RS324 кукурузы); 6429362 (промотор PR-1 кукурузы); 6232526 (промотор A3 кукурузы); 6177611 (конститутивные промоторы кукурузы); 5322938, 5352605, 5359142 и 5530196 (промотор 35S); 6433252 (промотор олеосина L3); 6429357 (промотор 2 актина риса и интрон 2 актина риса); 6294714 (индуцируемые светом промоторы); 6140078 (индуцируемые солью промоторы); 6252138 (индуцируемые патогенами промоторы); 6175060 (индуцируемые дефицитом фосфора промоторы); 6388170 (двунаправленные промоторы); 6635806 (промотор гамма-коиксина); 5447858 (промотор белка теплового шока сои); и патентная заявка США с серийным номером № 09/757089 (промотор альдолазы хлоропластов кукурузы).
Дополнительные иллюстративные промоторы включают промотор нопалинсинтазы (NOS) (Ebert et al. (1987) Proc. Natl. Acad. Sci. USA 84(16):5745-9); промотор октопинсинтазы (OCS) (который находится на индуцирующих опухоль плазмидах Agrobacterium tumefaciens); (промоторы колимовируса, такие как промотор 19S вируса мозаики цветной капусты (CaMV) (Lawton et al. (1987) Plant Mol. Biol. 9:315-24); промотор 35S CaMV (Odell et al. (1985) Nature 313:810-2; промотор 35S вируса мозаики норичника Walker et al (1987) Proc. Natl. Acad. Sci. USA 84(19):6624-8); промотор сахарозосинтазы (Yang and Russell (1990) Proc. Natl. Acad. Sci. USA 87:4144-8); комплексный промотор R-генов (Chandler et al. (1989) Plant Cell 1:1175-83); промотор гена белка, связывающего хлорофилл a/b; CaMV35S (патенты США № 5322938, 5352605, 5359142 и 5530196); FMV35S (патенты США № 6051753 и 5378619); промотор PC1SV (патент США № 5850019); промотор SCP1 (патент США № 6677503); и промоторы AGRtu.nos (номер доступа GenBank № V00087; Depicker et al. (1982) J. Mol. Appl. Genet. 1:561-73; Bevan et al. (1983) Nature 304:184-7).
В конкретных вариантах осуществления молекулы нуклеиновой кислоты могут содержать тканеспецифический промотор. Тканеспецифический промотор представляет собой нуклеотидную последовательность, которая обеспечивает более высокий уровень транскрипции функционально связанной нуклеотидной последовательности в ткани, в отношении которой промотор является специфическим, относительно других тканей организма. Примеры тканеспецифических промоторов включают, но не ограничиваются ими: специфичные к тапетуму промоторы; специфичные к пыльнику промоторы; специфичные к пыльце промоторы (см., например, патент США № 7141424, и международную публикацию PCT № WO 99/042587); специфичные к семяпочке промоторы; (см., например, патентную заявку США № 2001/047525 A1); специфичные к плодам промоторы (см., например, патенты США № 4943674 и 5753475); и специфичные к семенам промоторы (см., например, патенты США № 5420034 и 5608152). В некоторых вариантах осуществления можно использовать промотор, специфичный к стадии развития (например, промотор, активный на более поздней стадии развития).
Дополнительные регуляторные последовательности, которые в некоторых вариантах осуществления могут быть функционально связаны с молекулой нуклеиновой кислоты, включают 5'-UTR, расположенные между промоторной последовательностью и кодирующей последовательностью, которая функционирует в качестве лидерной последовательности трансляции. Лидерная последовательность трансляции присутствует в полностью процессированной мРНК, и она может влиять на процессинг первичного транскрипта и/или стабильность РНК. Примеры лидерных последовательностей трансляции включают лидерные последовательности белка теплового шока кукурузы и петунии (патент США № 5362865), лидерные последовательности белков оболочки вирусов растений, лидерные последовательности рибулозо-1,5-бифосфаткарбоксилазы/оксигеназы растений и другие. См., например, Turner and Foster (1995) Molecular Biotech. 3(3):225-36. Неограничивающие примеры 5'-UTR происходят из: GmHsp (патент США № 5659122); PhDNA (патент США № 5362865); AtAnt1; TEV (Carrington and Freed (1990) J. Virol. 64:1590-7); и AGRtunos (номер доступа GenBank № V00087; и Bevan et al. (1983), выше).
Дополнительные регуляторные последовательности, которые в некоторых вариантах осуществления могут быть функционально связаны с молекулой нуклеиновой кислоты, также включают 3'-нетранслируемые последовательности, 3'-области терминации транскрипции, или области полиаденилирования. Они представляют собой генетические элементы, расположенные ниже нуклеотидной последовательности, и включают полинуклеотиды, которые обеспечивают сигнал полиаденилирования и/или другие регуляторные сигналы, способные влиять на транскрипцию или процессинг мРНК.
Сигнал полиаденилирования функционирует в растениях, обеспечивая присоединение полиаденилатных нуклеотидов к 3'-концу мРНК-предшественника. Последовательность полиаденилирования может происходить из различных генов растений или из генов T-ДНК. Неограничивающим примером 3'-области терминации транскрипции является 3'-область нопалинссинтазы (nos 3'; Fraley et al. (1983) Proc. Natl. Acad. Sci. USA 80:4803-7). Пример применения различных 3'-нетранслируемых областей предоставлен в Ingelbrecht et al. (1989) Plant Cell 1:671-80. Неограничивающие примеры сигналов полиаденилирования включают сигнал из гена RbcS2 Pisum sativum (Ps.RbcS2-E9; Coruzzi et al. (1984) EMBO J. 3:1671-9) и AGRhmos (номер доступа GenBank № E01312).
Дополнительная информация, касающаяся регуляторных последовательностей, которые могут быть пригодными в конкретных вариантах осуществления, описана, например, в Goeddel (1990) "Gene Expression Technology", Methods Enzymol. 185, Academic Press, San Diego, CA.
Рекомбинантная молекула нуклеиновой кислоты или вектор могут содержать селективный маркер, который сообщает поддающийся селекции фенотип трансформированной клетке, такой как растительная клетка. Селективные маркеры также можно использовать для селекции клеток или организмов, которые содержат молекулу нуклеиновой кислоты, содержащую селективный маркер. Маркер может кодировать устойчивость к биоцидам, устойчивость к антибиотикам (например, канамицин, генетицин (G418), блеомицин и гигромицин) или устойчивость к гербицидам (например, глифосат). Примеры селективных маркеров включают, но не ограничиваются ими: ген neo, который сообщает устойчивость к канамицину и селекцию по которому можно проводить с использованием, например, канамицина и G418; ген bar, который сообщает устойчивость к биалафосу; мутантный ген EPSP-синтазы, который сообщает устойчивость к глифосату; ген нитрилазы, который сообщает устойчивость к бромксиснилу; мутантный ген ацетолактатсинтазы (ALS), который сообщает устойчивость к имидазолинону или сульфонилмочевине; и ген DHFR устойчивости к метотрексату. Доступно множество селективных маркеров, которые сообщают устойчивость химическим веществам, включая, например, но не ограничиваясь ими, ампициллин; блеомицин; хлорамфеникол; гентамицин; гигромицин; канамицин; линкомицин; метотрексат; фосфинорицин; пуромицин; спектиномицин; рифампицин; стрептомицин и тетрациклин. Примеры таких селективных маркеров проиллюстрированы, например, в патентах США 5550318; 5633435; 5780708 и 6118047.
Молекула нуклеиновой кислоты или вектор также или альтернативно могут включать поддающийся скринингу маркер. Поддающиеся скринингу маркеры можно использовать для мониторинга экспрессии. Иллюстративные поддающиеся скринингу маркеры включают ген β-глюкуронидазы или uidA (GUS), который кодирует фермент, для которого известны различные хромогенные субстраты (Jefferson et al. (1987) Plant Mol. Biol. Rep. 5:387-405); ген R-локуса, который кодирует продукт, который регулирует продукцию антоцианиновых пигментов (красный цвет) в тканях растений (Dellaporta et al. (1988) "Molecular cloning of the maize R-nj allele by transposon tagging with Ac." In 18th Stadler Genetics Symposium. P. Gustafson and R. Appels, eds., Plenum, NY (pp. 263-82); ген β-лактамазы (Sutcliffe et al. (1978) Proc. Natl. Acad. Sci. USA 75:3737-41); ген, который кодирует фермент, для которого известны различные хромогенные субстраты (например, PADAC, хромогенный цефалоспорин); ген люциферазы (Ow et al. (1986) Science 234:856-9); ген xylE, который кодирует катехолдиоксигеназу, которая конвертирует хромогенные катехолы (Zukowski et al. (1983) Gene 46(2-3):247-55); ген амилазы (Ikatu et al. (1990) Bio/Technol. 8:241-2); ген тирозиназы, который кодирует фермент, способный окислять тирозин до DOPA и допахинона, которые в свою очередь конденсируются до меланина (Katz et al. (1983) J. Gen. Microbiol. 129:2703-14); и ген α-галактозидазы.
Все из нуклеотидных последовательностей, которые кодируют, например, конкретный представляющий интерес полипептид или конкретную нацеливающую эндонуклеазу, будут сразу узнаны специалистами в данной области. Вырожденность генетического кода обеспечивает конечное количество кодирующих последовательностей для конкретной аминокислотной последовательности. Выбор конкретной последовательности для кодирования полипептида в соответствии с вариантами осуществления изобретения проводится по усмотрению практикующего специалиста. В различных применениях могут быть желательными различные кодирующие последовательности.
В некоторых вариантах осуществления может быть желательной модификация нуклеотидов нуклеиновой кислоты, например, для усиления экспрессии полинуклеотидной последовательности, содержащейся в нуклеиновой кислоте, в конкретном хозяине. Генетический код является избыточным с наличием 64 возможных кодонов, однако большинство организмов предпочтительно использует подгруппу этих кодонов. Кодоны, которые используются наиболее часто в виде, называют оптимальными кодонами, и кодоны, не используемые очень часто, классифицируют как редкие или низко используемые кодоны. Zhang et al. (1991) Gene 105:61-72. Кодоны могут быть заменены так, чтобы они отражали предпочтительное использование кодонов у конкретного хозяина, в процессе, иногда называемом "оптимизацией кодонов". Оптимизированные кодирующие последовательности, содержащие кодоны, предпочтительные для конкретного прокариотического или эукариотического хозяина, можно получать, например, для увеличения скорости трансляции или для продукции рекомбинантных РНК-транскриптов, имеющих желаемые свойства (например, более длительное время полужизни по сравнению с транскриптами, продуцируемыми с неоптимизированной последовательности).
В вариантах осуществления нуклеиновые кислоты можно вводить в клетки-хозяева любым способом, известным специалистам в данной области, включая, например, но не ограничиваясь ими: трансформацию протопластов (см., например, патент США 5508184); опосредуемый обезвоживанием/ингибированием захват ДНК (см., например, Potrykus et al. (1985) Mol. Gen. Genet. 199:183-8); электропорацию (см., например, патент США 5384253); встряхивание с волокнами карбида кремния (см., например, патенты США 5302523 и 5464765); опосредуемую Agrobacterium трансформацию (см., например, патенты США 5563055, 5591616, 5693512, 5824877, 5981840 и 6384301); и ускорение покрытых посредством ДНК частиц (см., например, патенты США 5015580, 5550318, 5538880, 6160208, 6399861 и 6403865). С использованием способов, таких как эти, можно стабильно трансформировать клетки практически любого вида. В некоторых вариантах осуществления трансформирующая ДНК встраивается в геном клетки-хозяина. В случае многоклеточных видов, трансгенные клетки могут регенерировать в трансгенный организм. Любые из этих способов можно использовать для получения трансгенного растения, например, содержащего одну или несколько последовательностей нуклеиновых кислот по изобретению в геноме трансгенного растения.
Наиболее широко используемый способ введения экспрессирующего вектора в растения основан на природной системе трансформации Agrobacterium. A. tumefaciens и A. rhizogenes представляют собой патогенные для растений бактерии почвы, которые генетически трансформируют клетки растений. Плазмиды Ti и Ri α. tumefaciens и A. rhizogenes, соответственно, содержат гены, ответственные за генетическую трансформацию растения. Плазмиды Ti (индуцирующие опухоль) содержат большой сегмент, известный как T-ДНК, который переносится в трансформированные растения. Другой сегмент плазмиды Ti, область vir, ответственен за перенос T-ДНК. Область T-ДНК ограничивается левой и правой границами, каждая из которых состоит из концевых повторяющихся нуклеотидных последовательностей. В некоторых модифицированных бинарных векторах индуцирующие опухоль гены удалены, и функции области vir используются для переноса чужеродной ДНК, ограниченной последовательностями границы T-ДНК. T-область также может содержать, например, селективный маркер для эффективного выделения трансгенных растений и клеток, и участок множественного клонирования для встраивания переносимых последовательностей, таких как нуклеиновая кислота, кодирующая слитый белок по изобретению.
Таким образом, в некоторых вариантах осуществления вектор для трансформации растений происходит из плазмиды Ti A. tumefaciens (см., например, патенты США № 4536475, 4693977, 4886937 и 5501967; и патент Европы EP 0 122 791) или плазмиды Ri A. rhizogenes. Дополнительные векторы для трансформации растений включают, например, но не ограничиваясь ими, векторы, описанные Herrera-Estrella et al. (1983) Nature 303:209-13; Bevan et al. (1983), выше; Klee et al. (1985) Bio Technol. 3: 637-42; и в патенте Европы EP 0 120 516, и векторы, происходящие из любого из указанных выше. Другие бактерии, такие как Sinorhizobium, Rhizobium и Mesorhizobium, которые в природе взаимодействуют с растениями, можно модифицировать, чтобы они опосредовали перенос генов в ряд разнообразных растений. Эти ассоциированные с растениями симбиотические бактерии можно преобразовывать в компетентные для переноса генов путем введения как разоруженной плазмиды Ti, так и подходящего бинарного вектора.
После предоставления экзогенной ДНК клеткам-реципиентам, трансформированные клетки, как правило, идентифицируют для дальнейшего культивирования и регенерации растений. Для улучшения способности идентифицировать трансформированные клетки, может быть желательным использование поддающегося селекции или поддающегося скринингу маркерного гена, как описано ранее, вместе с вектором, используемым для получения трансформанта. В случае, когда используют селективный маркер, трансформированные клетки идентифицируют в популяции потенциально трансформированных клеток путем воздействия на клетки селективного агента или агентов. В случае, когда используют поддающийся скринингу маркер, клетки можно подвергать скринингу в отношении желаемого признака маркерного гена.
Клетки, которые выживают после воздействия селективного агента, или клетки, которые были оценены как положительные в скрининговом анализе, можно культивировать в средах, которые способствуют регенерации растений. В некоторых вариантах осуществления любые подходящие среды для культивирования тканей растений (например, среды MS и N6) можно модифицировать путем включения дополнительных веществ, таких как регуляторы роста. Ткань можно поддерживать на базовой среде с регуляторами роста до тех пор, пока не будет доступно достаточно ткани для начала попытки регенерации растений, или, после повторяющихся раундов селекции вручную, до тех пор, пока морфология ткани не будет пригодной для регенерации (например, по меньшей мере в течение 2 недель), а затем ее можно переносить на среды, способствующие образованию побегов. Культуры переносят периодически до тех пор, пока не произойдет достаточное образование побегов. После образования побегов их переносят в среды, способствующие образованию корней. После достаточного образования корней растения переносят в почву для дальнейшего роста и созревания.
Для подтверждения присутствия представляющей интерес молекулы нуклеиновой кислоты (например, нуклеотидной последовательности, кодирующей полипептид, содержащий по меньшей мере один слитый белок по изобретению) в регенерирующем растении, можно проводить множество анализов. Такие анализы включают, например: молекулярно-биологические анализы, такие как саузерн- и нозерн-блоттинг, ПЦР и секвенирование нуклеиновых кислот; биохимические анализы, такие как выявление присутствия белкового продукта, например, с помощью иммунологических способов (ELISA и/или вестерн-блоттинг) или по ферментативной функции; анализы частей растений, такие как анализы листьев или корней; и анализы фенотипа целого регенерировавшего растения.
События встраивания можно анализировать, например, с помощью амплификации способом ПЦР с использованием, например, олигонуклеотидных праймеров, которые являются специфичными к представляющей интерес нуклеотидной последовательности. Подразумевают, что генотипирование способом ПЦР включает, но не ограничивается этим, амплификацию с помощью полимеразной цепной реакции (ПЦР) геномной ДНК, происходящей из выделенной ткани растения-хозяина, для которой спрогнозировано, что она содержит представляющую интерес молекулу нуклеиновой кислоты, встроенную в геном, за которой следует стандартное клонирование и анализ последовательности продуктов амплификации с помощью ПЦР. Способы генотипирования с помощью ПЦР описаны (см., например, Rios, G. et al. (2002) Plant J. 32:243-53) и их можно использовать для геномной ДНК, происходящей из любого вида растений или типа ткани, включая культуры клеток.
Трансгенное растение, полученное с помощью способов зависимой от Agrobacterium трансформации, как правило, содержит от одной до множества копий рекомбинантной ДНК. Одну последовательность рекомбинантной ДНК называют "трансгенным событием" или "событием встраивания". Такие трансгенные растения являются гомозиготными по встроенной последовательности ДНК. В некоторых вариантах осуществления трансгенное растение, являющееся гомозиготным в отношении трансгена, можно получать путем полового скрещивания (самоопыления) независимого сегрегантного трансгенного растения, которое содержит одну последовательность экзогенного гена, с самим собой, например, растением F0, с получением семян F1. Одна четверть образовавшихся семян F1 будет гомозиготной в отношении трансгена. Проращивание семян F1 приведет к растениям, которые можно исследовать в отношении гетерозиготности, как правило, с использованием анализа SNP или анализа способом концевой амплификации, который позволяет различить гетерозиготы и гомозиготы (т.е. анализ зиготности).
В дополнение к прямой трансформации растения или клетки растения молекулой нуклеиновой кислоты, в конкретных вариантах осуществления трансгенные растения можно получать путем скрещивания первого растения, имеющего по меньшей мере одно трансгенное событие, со вторым растением, лишенным такого события. Например, нуклеиновую кислоту, содержащую по меньшей мере один модифицированный локус FAD2, где экзогенная нуклеиновая кислота встроена сайт-специфическим образом, можно вводить в первую линию растений, которая поддается трансформации, с получением трансгенного растения, которое можно скрещивать со второй линией растений для интрогрессии по меньшей мере одного модифицированного локуса FAD2 (и, таким образом, экзогенной нуклеиновой кислоты) во вторую линию растений.
Для подтверждения присутствия представляющей интерес молекулы нуклеиновой кислоты в регенерировавших растениях можно проводить множество анализов. Такие анализы включают, например: молекулярно-биологические анализы, такие как саузерн- и нозерн-блоттинг и ПЦР; биохимические анализы, такие как выявление присутствия белкового продукта, например, с помощью иммунологических способов (ELISA и/или вестерн-блоттинг) или по ферментативной функции; анализы частей растений, такие как анализы листьев или корней; и анализ фенотипа целого регенерировавшего растения.
Скрининг событий направленного встраивания можно проводить, например, с помощью амплификации способом ПЦР с использованием, например, олигонуклеотидных праймеров, специфичных к представляющим интерес молекулам нуклеиновой кислоты. Подразумевают, что генотипирование способом ПЦР включает, но не ограничивается ими, амплификацию с помощью полимеразной цепной реакции (ПЦР) геномной ДНК, выделенной из ткани каллюса растения-хозяина, для которой спрогнозировано, что она содержит представляющую интерес молекулу нуклеиновой кислоты, встроенную в геном, с последующим стандартным клонированием и анализом последовательности продуктов амплификации способом ПЦР. Способы генотипирования с помощью ПЦР подробно описаны (например, Rios, G. et al. (2002) Plant J. 32:243-53) и их можно использовать для геномной ДНК, происходящей из любого вида растений или типа тканей, включая культуры клеток. Комбинации олигонуклеотидных праймеров, которые связываются как с последовательностью-мишенью, так и с введенной последовательностью, можно использовать в реакциях амплификации способом ПЦР последовательно или в мультиплексном формате. Являются возможными олигонуклеотидные праймеры, сконструированные для отжига с участком-мишенью, введенными последовательностями нуклеиновой кислоты и/или их комбинацией. Таким образом, стратегии ПЦР-генотипирования могут включать (но не ограничиваются ими) амплификацию конкретных последовательностей в геноме растения, амплификацию множества конкретных последовательностей в геном растения, амплификацию неспецифических последовательностей в геноме растения или их комбинацию. Специалист в данной области может установить дополнительные комбинации праймеров и реакций амплификации для исследования генома. Например, можно конструировать набор прямых и обратных олигонуклеотидных праймеров для отжига с последовательностью(ями) нуклеиновой кислоты, специфичной к мишени, вне границ введенной последовательности нуклеиновой кислоты.
Прямые и обратные олигонуклеотидные праймеры можно конструировать для специфического отжига с представляющей интерес введенной молекулой нуклеиновой кислоты, например, в последовательности, соответствующей кодирующей области в представляющей интерес молекуле нуклеиновой кислоты, или в других частях представляющей интерес молекулы нуклеиновой кислоты. Эти праймеры можно использовать совместно с праймерами, описанными выше. Олигонуклеотидные праймеры можно синтезировать в зависимости от желаемой последовательности, и они являются коммерчески доступными (например, от Integrated DNA Technologies, Inc., Coralville, IA). После амплификации может следовать клонирование и секвенирование, или прямой анализ последовательности продуктов амплификации. Специалист в данной области может предусмотреть альтернативные способы анализа продуктов амплификации, образовавшихся в ходе генотипирования способом ПЦР. В одном варианте осуществления в амплификациях способом ПЦР используют олигонуклеотидные праймеры, специфичные к гену-мишени.
VI. Трансгенные растения и материалы растений, содержащие нуклеиновую кислоту, встроенную в локус функциональности FAD2
В некоторых вариантах осуществления предусматривается трансгенное растение, где растение содержит клетку растения, содержащую по меньшей мере один модифицированный (например, с разрушением и/или направленным встраиванием экзогенной последовательности) локус FAD2. В конкретных вариантах осуществления такое растение можно получать путем трансформации ткани растения или клетки растения и регенерации целого растения. В следующих вариантах осуществления такое растение можно получать путем введения экзогенной нуклеиновой кислоты по меньшей мере в один локус FAD2 сайт-специфическим образом или путем интрогрессии модифицированного локуса FAD2 в зародышевую плазму. Также предусматриваются материалы растений, содержащие такую клетку растений. Такой материал растений можно получать из растения, содержащего клетку растения.
Трансгенное растение или материал растения, содержащий клетку растения, содержащую по меньшей мере один модифицированный локус FAD2, в некоторых вариантах осуществления могут проявлять одну или несколько из следующих характеристик: экспрессия нацеливающей эндонуклеазы в клетке растения; экспрессия представляющего интерес полипептида в клетке растения (или в его пластиде); экспрессия нацеливающей эндонуклеазы в ядре клетки растения; локализация нацеливающей эндонуклеазы в клетке растения; встраивание в локус FAD2 в геноме клетки растения; встраивание нуклеотидной последовательности, кодирующей представляющий интерес полипептид или агрономический ген, в локус FAD2 в геноме клетки растения; и/или присутствие РНК-транскрипта, соответствующего кодирующей последовательности, встроенной в локус FAD2 в геноме клетки растения. Такое растение, кроме того, может иметь один или нескольких желаемых признаков, включая, например, но не ограничиваясь ими, признаки, являющиеся следствием экспрессии эндогенной или трансгенной нуклеотидной последовательности, экспрессия которой регулируется представляющим интерес полипептидом или агрономическим геном, встроенным в локус FAD2 генома клетки растения; устойчивость к насекомым, другим паразитам и вызывающим заболевание агентам; устойчивость к гербицидам; увеличенную стабильность, выход или срок хранения; устойчивость к внешним факторам; продуцирование фармацевтического средства; продуцирование промышленного продукта; и усиление питательности.
Трансгенное растение по изобретению может представлять собой любое растение, которое может быть трансформировано нуклеиновой кислотой, которая затем встраивается по меньшей мере в один локус FAD2, в соответствии со способами, описанными в настоящем описании. Таким образом, растение может быть двудольным или однодольным. Неограничивающие примеры двудольных растений, пригодных в способах по настоящему изобретению, включают арабидопсис, люцерну, боб, брокколи, капусту, канолу, морковь, цветную капусту, сельдерей, китайскую капусту, хлопок, огурец, баклажан, салат-латук, дыню, горох, перец, арахис, картофель, тыкву, хрен, рапс, шпинат, сою, тыкву крупноплодную, сахарную свеклу, подсолнечник, табак, томат и арбуз. Неограничивающие примеры однодольных растений, пригодных в способах по настоящему изобретению, включают кукурузу, ячмень, лук, рис, сорго, пшеницу, рожь, щетинник, сахарный тростник, овес, тритикале, просо прутьевидное и газонную траву. Трансгенные растения по изобретению можно использовать или культивировать любым способом.
Некоторые варианты осуществления также относятся к товарным продуктам, полученным из трансгенных растений по изобретению. Товарные продукты включают, но не ограничиваются ими: пищевые продукты, муку, масла или молотые или цельные зерна или семена растения, содержащего одну или несколько нуклеотидных последовательностей, встроенных по меньшей мере в один локус FAD2. Обнаружение одной или нескольких таких нуклеотидных последовательностей в одном или нескольких товарах или товарных продуктах также фактически является свидетельством того, что товар или товарный продукт по меньшей мере частично получен из трансгенного растения, полученного в соответствии с вариантом осуществления изобретения. В некоторых вариантах осуществления трансгенное растение или семя, содержащие клетку растения, содержащую по меньшей мере один модифицированный локус FAD2, могут содержать по меньшей мере одно другое трансгенное событие в их геноме, включая, но не ограничиваясь этим: трансгенное событие, с которого транскрибируется молекула РНК-i; ген, кодирующий инсектицидный белок (например, инсектицидный белок Bacillus thuringiensis); ген устойчивости к гербициду (например, ген, обеспечивающий устойчивость к глифосату); и ген, обеспечивающий желаемый фенотип в трансгенном растении (например, увеличенный выход, измененный метаболизм жирных кислот или восстановление цитоплазматической мужской стерильности).
Трансгенное растение, содержащее клетку растения, содержащую по меньшей мере один модифицированный локус FAD2, может иметь один или несколько желательных признаков. Такие признаки могут включать, например: устойчивость к насекомым, другим паразитам и вызывающим заболевание агентам; устойчивость к гербицидам; увеличенную стабильность, выход или срок хранения; устойчивость к внешним факторам; продуцирование фармацевтических средств; продуцирование промышленного продукта и усиление питательности. Желаемые признаки могут сообщаться одной или несколькими молекулами нуклеиновой кислоты, встроенными путем направленной рекомбинации в локус FAD2, которые экспресируются в растении, проявляя желаемые признаки. Таким образом, в некоторых вариантах осуществления желаемый признак может быть следствием присутствия трансгена(ов) в растении, который введен в геном растения в участке по меньшей мере одного модифицированного локуса FAD2. В дополнительном варианте осуществления желаемый признак может быть достигнут посредством традиционного разведения, причем этот признак может сообщаться одной или несколькими молекулами нуклеиновой кислоты, встроенными путем направленной рекомбинации по меньшей мере в один модифицированный локус FAD2.
Трансгенные растения по изобретению можно использовать или культивировать для любых целей, где является желательным присутствие по меньшей мере одного модифицированного локуса FAD2. Таким образом, растение можно модифицировать способами инженерии, среди прочего, чтобы оно имело один или несколько желаемых признаков, путем трансформации молекулами нуклеиновой кислоты, которые впоследствии встраиваются сайт-специфическим образом по меньшей мере в один локус FAD2 по изобретению, и высевать и культивировать любым способом, известным специалистам в данной области.
VII. Разведение с помощью маркеров трансгенных растений, содержащих нуклеиновую кислоту, встроенную в локус функциональности FAD2
Предусматриваются молекулярные маркеры, которые сцеплены (например, прочно сцеплены) с fad2 в Brasicca spp. Например, идентифицированы сегменты ДНК, содержащие последовательности, вовлеченные в признак HO (fad2). Эти сегменты расположены вблизи и между маркерами, которые сцеплены (например, прочно сцеплены) с мутантными аллелями в геномной группе сцепления. Таким образом, предусматриваются молекулы нуклеиновой кислоты, содержащие мутантный ген FAD2, имеющий инактивирующую мутацию. Идентифицированные сегменты и их маркеры включены в настоящее изобретение, частично, вследствие их положения в группах сцепления в геноме B. napus. Например, FAD2 и молекулярные маркеры, сцепленные с ним, могут быть расположены в группах сцепления N5 и N1.
Все ссылки, включая публикации, патенты и патентные заявки, цитированные в настоящем описании, включены в настоящее описание в качестве ссылок, до той степени, чтобы они не были противоречащими настоящему изобретению, и, таким образом, они включены в той же степени, как если было отдельно и конкретно указано, что каждая ссылка включена в качестве ссылки и была представлена в настоящем описании в полном объеме Ссылки, рассмотренные в настоящем описании, предоставлены только для их раскрытия до даты подачи настоящей заявки. Ничто в настоящем документе не следует истолковывать как допущение того, что это изобретение дает право на противопоставление факта создания изобретения с более ранним приоритетом. Представленные ниже примеры предоставлены для иллюстрации определенных конкретных признаков и/или вариантов осуществления. Примеры не следует истолковывать как ограничивающие раскрытие конкретных проиллюстрированных признаков или вариантов осуществления.
ПРИМЕРЫ
ПРИМЕР 1: ИДЕНТИФИКАЦИЯ ПОСЛЕДОВАТЕЛЬНОСТЕЙ-МИШЕНЕЙ В FAD2 ИЗ БИБЛИОТЕКИ БАКТЕРИАЛЬНЫХ ИСКУССТВЕННЫХ ХРОМОСОМ
КОНСТРУИРОВАНИЕ БИБЛИОТЕКИ BAC
Библиотеку бактериальных искусственных хромосом (BAC) получали от коммерческого поставщика (Amplicon Express, Pullman, WA). Библиотека BAC включала 110592 клонов BAC, содержащих высокомолекулярные фрагменты геномной ДНК (гДНК), выделенные из Brassica napus L. сорта DH10275. гДНК расщепляли ферментом рестрикции либо ΒamΗΙ, либо HindIII. Выделенные фрагменты гДНК размером приблизительно 135 т.п.н. лигировали в вектор pCC1BAC (Epicentre, Madison, WI) и трансформировали в Escherichia coli штамма DH10B (Invitrogen). Библиотека BAC состояла из равного количества клонов BAC, которые были сконструированы с использованием двух различных ферментов рестрикции. По существу, сконструированная с помощью HindIII библиотека BAC содержалась в 144 индивидуальных 384-луночных планшетах. Аналогично, сконструированная с помощью BamHI библиотека BAC содержалась в 144 индивидуальных 384-луночных планшетах. Всего 110592 клонов BAC было выделено и распределено в 288 индивидуальных 384-луночных планшетах. Каждый из 288 индивидуальных 384-луночных планшетов был предоставлен поставщиком в качестве единичного экстракта ДНК для быстрого скрининга на основе ПЦР. Полученная библиотека BAC охватывает приблизительно 15 м.п.н. гДНК, что соответствует 12-кратному охвату генома Brassica napus L. сорта DH10275 (согласно оценке геном Brassica napus L. составляет приблизительно 1,132 м.п.н., как описано в Johnston et al. (2005) Annals of Botany 95:229-235).
АНАЛИЗ ПОСЛЕДОВАТЕЛЬНОСТИ КОДИРУЮЩИХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ FAD2, ВЫДЕЛЕННЫХ ИЗ БИБЛИОТЕКИ BAC
Сконструированную библиотеку BAC использовали для выделения кодирующих последовательностей гена FAD2. Эксперименты по секвенированию проводили для идентификации конкретных последовательностей генов для четырех паралогов гена FAD2 из Brassica napus L. сорта DH10275.
Последовательность гена FAD2 первоначально идентифицировали в модельном виде Arabidopsis thaliana. Последовательность гена приведена в Genbank под обозначением локуса: At3g12120. Ранее была описана сравнительная геномная взаимосвязь между модельным видом растений Arabidopsis thaliana и диплоидным Brassica rapa, одним из предшественников тетраплоидного Brassica napus. (Schranz et al. (2006) Trends in Plant Science 11(11):535-542). В отношении конкретно гена FAD2 сравнительный анализ спрогнозировал, что в диплоидном геноме Brassica может встречаться 3-4 копии гена. Дополнительные исследования по генетическому картированию были осуществлены Scheffler et al. (1997) Theoretical and Applied Genetics 94; 583-591. Результаты этих исследований по генетическому картированию показали, что в Brassica napus присутствует четыре копии гена FAD2.
Анализ путем секвенирования библиотеки BAC, которая была сконструирована из B. napus L. сорта DH12075, привел к выделению четырех последовательностей BAC (SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3 и SEQ ID NO: 4), в которых были определены кодирующие последовательности для генов FAD2A (SEQ ID NO: 5), FAD2-1 (SEQ ID NO: 6), FAD2-2 (SEQ ID NO: 7) и FAD2-3 (SEQ ID NO: 8). Последовательности генов FAD2A, FAD2-1, FAD2-2 и FAD2-3 были идентифицированы и генетически картированы. Анализ последовательностей четырех генов FAD2 проводили с использованием программы для выравнивания последовательностей и дерева, полученного по методу ближайшего связывания, с использованием процента идентичности. Выравнивание последовательностей проводили с помощью программы AlignX® из компьютерной программы Vector NTI Advance 11.0 (Life Technologies, Carlsbad, CA), и оно представлено на фиг. 1. В AlignX® используется модифицированный алгоритм Clustal W для проведения множественного выравнивания либо белковых последовательностей, либо последовательностей нуклеиновых кислот, для сравнения сходства и для проведения аннотирования.
Дерево, полученное по методу ближайшего связывания, создавали с помощью программного обеспечения Jalview v2.3®, и оно представлено на фиг. 2. (Waterhouse et al. (2009) Bioinformatics 25 (9) 1189-1191). Как показано на фиг. 2, анализ выделенных последовательностей показал, что последовательности FAD2A и FAD2-3 обладали высокими уровнями сходства последовательностей и что, аналогично, FAD2-1 и FAD2-2 обладали высокими уровнями сходства последовательностей. Эти четыре последовательности могут быть подразделены на два клада, где FAD2A и FAD2-3 составляют первый клад, и FAD2-1 и FAD2-2 составляют второй клад.
Далее, вновь выделенные последовательности FAD2 из Brassica napus использовали для проведения BLAST геномных библиотек, выделенных из геномной библиотеки BAC Brassica rapa и получения данных о геномной последовательности Brassica oleracea с помощью метода стохастических геномных фрагментов. Как Brassica rapa, и так и Brassica oleracea, являются диплоидными предшественники Brassica napus, который является амфиплоидным видом (геном AC, n=19). Brassica napus произошел в результате недавнего события гибридизации между Brassica rapa (субгеном A, n=10) и Brassica oleracea (субгеном C, n=9). Диплоидные последовательности-предшественники сравнивали с четырьмя различными кодирующими последовательностями FAD2, выделенными из Brassica napus, с использованием анализа BLASTn. Этот анализ последовательностей идентифицировал специфические аннотированные последовательности генов из Brassica rapa и Brassica oleracea, которые обладали наибольшим сходством последовательностей с вновь выявленными последовательностями FAD2 Brassica napus. В таблице 1 приведена вновь идентифицированная кодирующая последовательность FAD2, и соответствующий номер доступа эталонной последовательности предшественника, и организм-источник.

Гены FAD2 существуют в геноме Brassica napus в качестве двух копий каждого гена на субгеном. Одна копия каждого гена расположена на субгеноме A, и, аналогично, одна копия каждого гена расположена на субгеноме C. Новые соглашения о наименованиях описаны для указания на то, на каком субгеноме расположен каждый ген. Более высокие уровни сходства последовательностей между четырьмя различными кодирующими последовательностями FAD2, выделенными из библиотеки геномной ДНК BAC Brassica napus, и данные о последовательностях-предшественниках указывают на то, что FAD2-3 является дубликатом последовательности FAD2 из субгенома C, и он может быть переобозначен как FAD2C; FAD2-1 является дубликатом последовательности FAD2 из субгенома A и, таким образом, он может быть обозначен как FAD2A'; и, наконец, FAD2-2 является второй копией, которая является дубликатом последовательности FAD2 субгенома С, и он может быть обозначен как FAD2C.
СКРИНИНГ НА ОСНОВЕ ПЦР
Для скрининга упомянутой выше библиотеки BAC была сконструирована группа праймеров для ПЦР. Праймеры были сконструированы либо в качестве универсальных праймеров, которые амплифицируют всех представителей данного семейства генов, либо в качестве специфических генных праймеров для направленной амплификации аллеля. Праймеры для ПЦР были сконструированы так, чтобы они имели длину 20 п.н. (+/- 1 п.н.) и имели содержание G/C 50% (+/- 8%). В таблице 2 приведены праймеры, которые были сконструированы и синтезированы. Клоны библиотеки BAC были объединены и подвергнуты скринингу с помощью полимеразной цепной реакции (ПЦР).

Для полимеразных цепных реакций (ПЦР) использовали два различных набора условий. Первая серия реакций ПЦР включала: 1X буфер для ПЦР (содержащий dNTP); 1,5 мМ MgCl2; 200 мкМ ДНК-полимеразы Immolase® с 0,25 Е (Bioline, London, Великобритания); 250 нМ каждого праймера и приблизительно 5-10 нг ДНК-матрицы. Вторую серию реакций ПЦР разрабатывали для амплификации геномной ДНК, и она включала: 5-10 нг геномной ДНК, 1X буфер для ПЦР, 2 мМ dNTP, 0,4-мкМ прямой и обратный праймер и 0,25 Е ДНК-полимеразы Immolase® (Bioline, London, Великобритания). Реагенты для амплификации объединяли в конечном объеме 13 мкл и амплифицировали с использованием термоциклера MJ PTC200® (BioRad, Hercules, CA) или ABI 9700 Gene Amp System® (Life Technologies, Carlsbad, CA). Скрининг на основе ПЦР в специальных планшетах проводили с использованием подхода 4-мерного скрининга на основе системы скрининга, описанной Bryan et al (Scottish Crops Research Institute annual report: 2001-2002) с описанными выше условиями ПЦР. После скрининга на основе ПЦР объединенных библиотек BAC, амплифицированный продукт ПЦР секвенировали с использованием способа прямого секвенирования Сэнгера. Амплифицированные продукты очищали этанолом, ацетатом натрия и EDTA в соответствии с протоколом BigDye® v3.1 (Applied Biosystems) и проводили электрофорез на автоматической платформе для капиллярного электрофореза ABI3730x1®.
После скрининга на основе ПЦР и подтверждающего секвенирования по методу Сэнгера идентифицировали набор планшетов, который содержал различных представителей семейства генов FAD2. Было идентифицировано всего четыре уникальных последовательности паралогов гена FAD2 (таблица 2). Всего два планшета на каждую последовательность паралога гена FAD2 было выбрано для проведения скрининга планшета в целях идентификации конкретной лунки и клона в планшете, которые содержали ген FAD2 (таблица 3). Конкретные лунки идентифицировали для обоих планшетов и для каждого из представителей семейства генов FAD2 выбирали индивидуальный клон.

Единичный клон BAC для каждого идентифицированного представителя семейства генов FAD далее анализировали секвенированием. ДНК выделяли для клона BAC и подготавливали для секвенирования с использованием набора Large Construct kit® (Qiagen, Valencia, CA) в соответствии с инструкциями изготовителя. Экстрагированную ДНК BAC подготавливали для секвенирования с использованием GS-FLX Titanium Technology® (Roche, Indianapolis, IΝ) в соответствии с инструкциями изготовителя. Реакции секвенирования проводили с использованием физически секторированного планшета GS-FLX TI Pico-titer plate®, где BAC были объединены парами для оптимального получения данных. BAC объединяли парами, причем ген FAD2 объединяли в пару с геном FAD3. Все полученные данные о последовательности объединяли в Newbler v2.0.01.14® (454 Life Sciences, Branford, CT). Собранные контиги оценивали вручную в отношении присутствия соответствующего гена FAD с использованием Sequencher v3.7® (GeneCodes, Ann Arbor, MI).
После идентификации и полной охарактеризации полной геномной последовательности всех четырех генов FAD2, конструировали нуклеазы с цинковыми пальцами, чтобы они связывались с последовательностями каждого конкретного представителя семейства генов.
ПРИМЕР 2: КОНСТРУИРОВАНИЕ СВЯЗЫВАЮЩИХ ДОМЕНОВ С ЦИНКОВЫМИ ПАЛЬЦАМИ, СПЕЦИФИЧНЫХ К ГЕНАМ FAD2
Новые белки с цинковыми пальцами, направленные против последовательностей ДНК, кодирующих различные функциональные последовательности локуса генов FAD2, конструировали, по существу как описано ранее. См., например, Urnov et al. (2005) Nature 435:646-651. Иллюстративная последовательность-мишень и распознающие спирали представлены в таблице 4 (модели распознающих спиральных областей) и в таблице 5 (участки-мишени). В таблице 5 нуклеотиды в участке-мишени, которые контактируют с распознающими спиралями ZFP, указаны прописными буквами; неконтактирующие нуклеотиды указаны строчными буквами.
Участки-мишени нуклеазы с цинковыми пальцами (ZFN) конструировали так, чтобы они связывали пять участков-мишеней FAD2A. Конструкции цинковых пальцев для FAD2A включали в экспрессирующие векторы для цинковых пальцев, кодирующие белок, имеющий по меньшей мере один палец со структурой CCHC. См., публикацию патента США № 2008/0182332. В частности, последний палец в каждом белке имел остов CCHC для распознающей спирали. Неканонические последовательности, кодирующие цинковые пальцы, подвергали слиянию с нуклеазным доменом фермента рестрикции FokI типа IIS (аминокислоты 384-579 последовательности Wah et al., (1998) Proc. Natl. Acad. Sci. USA 95:10564-10569) через линкер ZC из четырех аминокислот и сигнал ядерной локализации opaque-2, происходящий из Zea mays, с получением нуклеаз с цинковыми пальцами (ZFN) для FAD2A. Экспрессия слитых белков контролировалась относительно сильным конститутивным промотором, таким как промотор, происходящий из промотора вируса мозаики прожилок маниоки (CsVMV) и фланкировались 3'-нетранслируемой областью ORF23 Agrobacterium tumefaciens (3'UTR v1 AtuORF23). Между двумя слитыми белками нуклеаз с цинковыми пальцами, которые были клонированы в конструкцию, добавляли самогидролизующуюся нуклеотидную последовательность, кодирующую 2A, из вируса Thosea asigna (Szymczak et al., 2004). Иллюстративные векторы или плазмиды описаны в таблице 5, ниже.
Активность расщепления у оптимальных нуклеаз с цинковыми пальцами для FAD2 подтверждали с использованием системы на основе почкования дрожжей, для которой ранее было показано, что она идентифицирует активные нуклеазы. См., например, публикацию патента США № 20090111119; Doyon et al. (2008) Nat Biotechnol. 26:702-708; Geurts et al. (2009) Science 325:433. Цинковые пальцы для различных функциональных доменов выбирали для применения in-vivo. Среди многочисленных ZFN, которые были сконструированы, продуцированы и исследованы в отношении связывания с предполагаемыми геномными полинуклеотидными участками-мишенями FAD, одиннадцать ZFN было идентифицировано в качестве имеющих активность in vivo на высоких уровнях, и выбрано для дальнейшего экспериментирования. Эти ZFN были охарактеризованы как способные эффективно связывать и расщеплять уникальные геномные полинуклеотидные участки-мишени FAD2 в растениях.






ПРИМЕР 3: ОЦЕНКА РАСЩЕПЛЕНИЯ НУКЛЕАЗАМИ С ЦИНКОВЫМИ ПАЛЬЦАМИ ГЕНОВ FAD2
СБОРКА КОНСТРУКЦИИ
Плазмидные векторы, содержащие конструкции для экспрессии иллюстративных нуклеаз с цинковыми пальцами ZFN, которые были идентифицированы с использованием анализа на дрожжах, как описано в примере 2, моделировали и получали с использованием навыков и способов, широко известных в данной области. Каждую последовательность, кодирующую цинковый палец, подвергали слиянию с последовательностью, кодирующей сигнал ядерной локализации opaque-2 (Maddaloni et al. (1989) Nuc. Acids Res. 17(18):7532), который был расположен выше нуклеазы с цинковыми пальцами.
Далее формировали пару из слитой последовательности сигнал ядерной локализации opaque-2 :: нуклеаза с цинковыми пальцами с комплементарной слитой последовательностью сигнал ядерной локализации opaque-2 :: нуклеаза с цинковыми пальцами. По существу, каждая конструкция включала одну открытую рамку считывания, состоящую из двух слитых последовательностей сигнал ядерной локализации opaque-2 :: нуклеаза с цинковыми пальцами, разделенных последовательностью 2A из вируса Thosea asigna (Mattion et al. (1996) J. Virol. 70:8124-8127). Экспрессия слитых белков контролировалась относительно сильным конститутивным промотором, таким как промотор, происходящий из промотора вируса мозаики прожилок маниоки (CsVMV), и фланкировалась 3'-нетранслируемой областью ORF23 Agrobacterium tumefaciens (3'UTR AtuORF23).
Векторы собирали с использованием IΝ-FUSION™ Advantage Technology (Clontech, Mountain View, CA). Эндонуклеазы рестрикции получали от New England BioLabs (NEB; Ipswich, MA) и для лигирования ДНК использовали ДНК-лигазу T4 (Invitrogen). Получение плазмид проводили с использованием набора NUCLEOSPIN® Plasmid Kit (Macherey-Nagel Inc., Bethlehem, PA) или набора Plasmid Midi Kit (Qiagen) в соответствии с инструкциями поставщиков. Фрагменты ДНК выделяли с использованием набора QIAquick Gel Extraction Kit™ (Qiagen) после электрофореза в агарозном Tris-ацетатном геле. Колонии всех собранных плазмид сначала подвергали скринингу с помощью рестрикционного расщепления ДНК Miniprep. Плазмидную ДНК выбранных клонов секвенировали в коммерческой организации, осуществляющей секвенирование (Eurofins MWG Operon, Huntsville, AL). Данные о последовательности собирали и анализировали с использованием программного обеспечения SEQUENCHER™ (Gene Codes Corp., Ann Arbor, MI). Перед доставкой в протопласты B. napus, плазмидную ДНК получали из культур E. coli с использованием Pure Yield Plasmid Maxiprep System® (Promega Corporation, Madison, WI) или Plasmid Maxi Kit® (Qiagen, Valencia, CA) в соответствии с инструкциями поставщиков.
Полученные одиннадцать плазмидных конструкций; pDAB104008 (содержащая конструкции ZFN24845 и ZFN24844), pDAB104009 (содержащая конструкции ZFN24820 и ZFN24821), pDAB104010 (содержащая конструкции ZFN24828 и ZFN24829) (фиг. 3), pDAB104003 (содержащая конструкции ZFN24810 и ZFN24811), pDAB104011 (содержащая конструкции ZFN24832 и ZFN24833), pDAB104002 (содержащая конструкции ZFN24794 и ZFN24795), pDAB104006 (содержащая конструкции ZFN24796 и ZFN24797), pDAB104004 (содержащая конструкции ZFN24814 и ZFN24815), pDAB104001 (содержащая конструкции ZFN24800 и ZFN24801), pDAB104005 (содержащая конструкции ZFN24818 и ZFN24819) и DAB104007 (содержащая конструкции ZFN24836 и ZFN24837) подтверждали посредством расщепления ферментами рестрикции и секвенирования ДНК. В таблице 6 приведены различные конструкции и конкретная последовательность FAD2A, для расщепления и связывания которой была сконструирована каждая ZFN.


ПОЛУЧЕНИЕ ДНК ДЛЯ ТРАНСФЕКЦИИ
Плазмидную ДНК описанных выше векторов стерилизовали путем осаждения и промывания в 100% (об./об.) этаноле и сушили в вытяжном шкафу с ламинарным потоком. Осадок ДНК суспендировали в 30 мкл стерильной дважды дистиллированной воды в конечной концентрации 0,7 г/л для трансфекции в клетки протопластов, как описано ниже. Получение плазмидной ДНК проводили так, чтобы получить сверхскрученную плазмидную ДНК для временной трансфекции и линеаризованную плазмидную ДНК для стабильной трансфекции. Добавление ДНК-носителя (например, ДНК спермы рыб) к трансформирующей плазмиде не требовалось для временной трансфекции протопластов клеток. Для временных исследований использовали приблизительно 30 мкг плазмидной ДНК на 106 протопластов на трансформацию.
ТРАНСФЕКЦИЯ
Трансфекцию Brassica napus L. сорта DH10275 проводили, как описано в Spangenberg et al., (1986) Plant Physiology 66: 1-8, составы сред описаны в Spangenberg G. and Protrykus I. (1995) Polyethylene Glycol-Mediated Direct Gene Transfer in Tobacco Protoplasts., Gene Transfer to Plants. (Protrykus I. and Spangenberg G. Eds.) Springer-Verlag, Berlin. Семена Brassica napus подвергали поверхностной стерилизации в 70% этаноле. Семена погружали в 12 мл 70% раствора этанола и перемешивали путем осторожного качания смеси в течение 10 минут. 70% раствор этанола удаляли путем декантации раствора и заменяли раствором для стерилизации семян, состоявшим из 1% масс./об. гипохлорита кальция и 0,1% об./об. Tween-20. Семена погружали в раствор для стерилизации семян и перемешивали путем осторожного качания смеси в течение 25 минут. Раствор для стерилизации семян декантировали и стерилизованные семена промывали три раза в 50 мл стерильной воды. Наконец, семена переносили на стерильный 80-мм фильтрующий бумажный диск Whatman filter paper disc® (Fisher-Scientific, St. Louis, MO), который был помещен в чашку Петри, и семена слегка насыщали стерильной водой. Чашку Петри накрывали с помощью Parafilm® (Fisher-Scientific, St. Louis, MO) и планшеты инкубировали при 25°C в условиях полной темноты в течение от одних до двух суток. После того, как наблюдали появление признаков прорастания семян, проростки переносили в чашку Петри, содержавшую затвердевшую среду GEM, для стимуляции дальнейшего прорастания семян. Проростки инкубировали на среде GEM при 25°C в течение от четырех до пяти суток.
Объем жидкой среды PS (приблизительно 10 мл) декантировали в стерильную чашку Петри. С использованием стерильных щипцов и скальпеля, надземную часть проростков в возрасте от четырех до пяти суток на стадии роста и развития 4 листков, удаляли и отбрасывали. Было определено, что сегменты гипоктиля длиной 20-40 мм образуют наиболее высокую популяцию небольших богатых цитоплазмой протопластов. Сегменты гипоктиля вырезали в асептических условиях и переносили в жидкую среду PS. Вырезанные сегменты гипоктиля группировали вместе и нарезали поперечно на сегменты размером 5-10 мм. Далее сегменты гипоктиля переносили в свежую среду PS и инкубировали при комнатной температуре в течение 1 часа. Подвергнутые плазмолизу гипоктили переносили в чашку Петри, содержавшую раствор фермента. Обращали внимание на то, чтобы погрузить в раствор все сегменты гипоктиля. Чашки Петри накрывали Parafilm® и инкубировали в течение от шестнадцати до восемнадцати часов при 20-22°C при осторожном качании.
Из сегментов гипоктиля высвобождали протопласты клеток. Продукты расщепления гипоктиля в течение ночи осторожно встряхивали для высвобождения протопластов в раствор фермента. Чашку Петри наклоняли немного, чтобы облегчить перенос расщепляющей суспензии раствора фермента и растительного дебриса. С использованием 10-мл пипетки расщепляющую суспензию переносили в стерилизованный элемент для фильтрации протопластов (фильтр с размером сита 100 микрометров) для дальнейшего отделения протопластов от растительного дебриса. По фильтрационному элементу постукивали осторожно для высвобождения избытка жидкости, который остался в сите. Суспензию протопластов объемом приблизительно от 8 до 9 мл осторожно перемешивали и распределяли в 14-мл стерильные пластмассовые центрифужные пробирки с круглым дном. На каждую суспензию наслаивали 1,5 мл раствора W5. Раствор W5 осторожно распределяли над суспензией протопластов под углом и распределяли капельно с минимальным встряхиванием. Добавление раствора W5 к суспензии протопластов приводило к образованию обогащенной протопластами поверхности контакта. Этот слой поверхности контакта собирали с использованием пипетки. Далее собранные протопласты переносили в новую 14-мл центрифужную пробирку и осторожно перемешивали. Выход полученных протопластов определяли с использованием счетной камеры для определения количества протопластов на миллилитр. Способ повторяли, где ткань листьев расщепляли с получением протопластов мезофилла.
Далее, добавляли раствор W5 до объема 10 мл и протопласты осаждали при 70g, а затем раствор W5 удаляли. Оставшуюся суспензию протопластов ресупендировали путем осторожного качания. Каждую пробирку, содержавшую суспензию протопластов, заполняли 5 мл раствора W5 и инкубировали при комнатной температуре в течение от одного до четырех часов. Суспензии протопластов осаждали при 70g и весь раствор W5 удаляли. Далее в каждую из осажденных суспензий протопластов, содержавших выделенные протопласты, добавляли 300 мкл буфера для трансформации. В каждой из пробирок к суспензиям протопластов добавляли 10 мкг плазмидной ДНК. Плазмидная ДНК включала конструкции нуклеаз с цинковыми пальцами, описанные выше (например, pDAB104010). Далее к суспензии протопластов добавляли 300 мкл предварительно нагретого раствора PEG 4000 и проводили осторожное постукивание по пробиркам. Суспензиям протопластов и смеси для трансформации позволяли инкубироваться при комнатной температуре в течение пятнадцати минут без какого-либо встряхивания. В каждую пробирку добавляли дополнительные 10 мл раствора W5 последовательными аликвотами объемом 1 мл, 1 мл, 1 мл, 2 мл, 2 мл и 3 мл с осторожным переворачиванием пробирок между добавлениями раствора W5 в каждом случае. Протопласты осаждали путем центрифугирования в центрифуге при 70g. Весь раствор W5 удаляли, оставляя чистую суспензию протопластов.
Далее к осажденным протопластам клеток добавляли 0,5 мл среды K3 и клетки ресуспендировали. Ресуспендированные протопласты клеток помещали в центр чашки Петри и добавляли 5 мл K3 и 0,6 мл агарозы Sea Plaque™ (Cambrex, East Rutherford, NJ) в концентрации 1:1. Чашки Петри встряхивали одним осторожным вращающим движением и оставляли инкубироваться в течение 20-30 минут при комнатной температуре. Чашки Петри накрывали Parafilm® и протопласты культивировали в течение двадцати четырех часов в полной темноте. После инкубации в темноте чашки Петри культивировали в течение шести суток при тусклом освещении (белые трубки Osram L36 W/21 Lumilux с 5 мкМоль м-2 с-1). После стадии культивирования использовали стерильный шпатель для разделения содержавших агарозу протопластов на квадранты. Отделенные квадранты помещали в 250-мл пластмассовую емкость для культивирования, содержавшую 20 мл среды A, и инкубировали на ротационном устройстве для встряхивания при 80 об/мин и смещении 1,25 см при 24°C при постоянном тусклом освещении в течение 14 суток, а затем анализировали для определения уровня активности каждой конструкции нуклеазы с цинковыми пальцами.
ВЫДЕЛЕНИЕ ГЕНОМНОЙ ДНК ИЗ ПРОТОПЛАСТОВ КАНОЛЫ
Трансфицированные протопласты предоставляли в индивидуальных 1,5- или 2,0-мл микроцентрифужных пробирках. Клетки осаждали на дне пробирки в буферном растворе. Экстракцию ДНК проводили путем быстрого замораживания клеток в жидком азоте с последующей лиофилизацией клеток в течение приблизительно 48 часов в Labconco Freezone 4.5® (Labconco, Kansas City, MO) при -40°C и давлении приблизительно 133×10-3 мбар. Лиофилизированные клетки подвергали экстракции ДНК с использованием набора для растений DNeasy® (QIAGEN, Carlsbad, CA) в соответствии с инструкцией изготовителя, за исключением того, что разрушение ткани не требовалось и протопласты клеток добавляли непосредственно в лизирующий буфер.
ИССЛЕДОВАНИЕ ZFN ДЛЯ FAD2A В ОТНОШЕНИИ РАСЩЕПЛЕНИЯ ПОСЛЕДОВАТЕЛЬНОСТИ ГЕНОМНОЙ ДНК В ПРОТОПЛАСТАХ КАНОЛЫ
Смоделированные участки-мишени для ZFN в локусе FAD2A кластеризовали, так чтобы сконструировать множество пар ZFN для перекрывания участков-мишеней. Кластеризация участков-мишеней для ZFN обеспечила конструирование праймеров для ПЦР, которые амплифицируют окружающую геномную последовательность всех представителей семейства генов FAD2A в пределах окна размером 100 п.н., чтобы охватить все перекрывающиеся участки-мишени для ZFN. По существу, для оценки целостности участка-мишени для ZFN в трансфицированных протопластах можно использовать технологию короткого считывания последовательностей Illumina. Кроме того, необходимо, чтобы конструкции праймеров для ПЦР включали конкретные нуклеотидные основания, которые позволят соотнести данные считывания последовательностей с конкретным представителем семейства FAD2A. Таким образом, требуется, чтобы все праймеры для ПЦР связывались на удалении 5-10 нуклеотидов от участка-мишени для расщепления посредством ZFN, поскольку известно, что активность негомологичного соединения концов (NHEJ) вызывает небольшие делеции, которые могут удалять участок затравки, ингибировать амплификацию и, таким образом, исказить оценку активности NHEJ.
Праймеры конструировали так, чтобы они связывались со всеми локусами-мишенями для ZFN из семейств генов FAD2A (таблица 7), и их эмпирически исследовали в отношении амплификации всех представителей семейства с помощью секвенирования продуктов амплификации способом ПЦР по методу Сэнгера. В нескольких случаях нельзя было разработать праймеры, которые различают всех представителей семейства генов (таблица 8), однако во всех случаях последовательности гена-мишени FAD2A, можно было различить. После конструирования праймеров для ПЦР в праймеры для ПЦР включали специализированные последовательности ДНК штрихового кода, которые использовали для различения различных локусов-мишеней для ZFN и идентификации конкретных считанных последовательностей для трансфекции и ZFN (таблицы 7 и 8).


Эффективность амплификации сконструированных праймеров для ПЦР в отношении семейств генов FAD2 представлена в таблице 8. "X" указывает на специфичность обнаружения копий генов, серым цветом и "+" указано, что в конкретном рассматриваемом локусе считанные последовательности, сконструированные с помощью двух праймеров, было невозможно различить.

После экстракции ДНК протопластов канолы, трансфицированных ZFN (несколькими ZFN), проводили амплификацию способом ПЦР локусов-мишеней для ZFN, чтобы получить необходимые специфичные к локусам молекулы ДНК в правильном формате для технологии секвенирования на основе IIlumina посредством синтеза. Каждый анализ оптимизировали для функционирования на 25 нг исходной ДНК (приблизительно 12500 клеточных эквивалентов генома Brassica napus). Проводили множество реакций на образец для обеспечения охвата, требуемого для оценки эффективности и специфичности NHEJ на соответствующем уровне: приблизительно шестнадцать реакций ПЦР, эквивалентных 200000 копиям генома Brassica napus, полученным из индивидуальных протопластов. Основные смеси для амплификации способом ПЦР получали для всех образцов, подлежащих исследованию с помощью одного и того же анализа, и анализировали одну реакцию, проведенную в трех экземплярах, с использованием способа количественной ПЦР, который использовали для определения оптимального количества циклов для проведения исследования на заданной ткани, чтобы гарантировать, что амплификация ПЦР не будет ограничиваться реагентом и все еще будет находиться на стадии экспоненциальной амплификации. Экспериментирование с необходимыми отрицательными контрольными реакциями проводили в 96-луночном формате с использованием термоциклера MX3000P thermocycler® (Stratagene, LaJolla, CA). Из данных, полученных с помощью платформы количественной ПЦР, относительное увеличение флуоресценции наносили на график от цикла к циклу и определяли количество циклов на анализ, которое обеспечивало достаточную амплификацию, одновременно не позволяя реакции стать ограниченной реагентом, в попытках снизить избыточное циклирование и амплификацию общих транскриптов или молекул. Неиспользованную основную смесь, остававшуюся на льду до тех пор, пока не завершился анализ способом количественной ПЦР и не было определено количество циклов, распределяли аликвотами в желаемое количество реакционных пробирок (приблизительно 16 на анализ ZFN) и проводили реакцию ПЦР.
После амплификации образцы для одного локуса ZFN объединяли вместе и 200 мкл объеденного продукта на ZFN очищали с использованием набора MinElute PCR purification kit® (Qiagen) в соответствии с инструкциями изготовителя. Для обеспечения секвенирования образца с использованием технологии короткого считывания Illumina требовалось связывание дополнительных парных концевых праймеров путем амплификации с полученными фрагментами. Это обеспечивали амплификацией способом ПЦР с использованием праймеров, которые являются частично комплементарными последовательности, добавленной в первом раунде амплификации, но также содержат требуемую парную концевую последовательность. Оптимальное количество циклов проводимых ПЦР, которое добавит парные концевые последовательности к матрице без избыточной амплификации общих фрагментов, вновь определяли с использованием подвергания образца количественному анализу циклов ПЦР, как описано ранее.
После амплификации способом ПЦР полученный продукт очищали с использованием колонки MinElute column® (Qiagen) в соответствии с инструкциями изготовителя и разделяли на 2,5% агарозном геле. Фрагменты ДНК, визуализированные с использованием Syber® Safe (Life Technologies, Carlsbad, CA), в качестве полос правильного размера подвергали экстракции из геля для удаления каких-либо остаточных образовавшихся в ходе ПЦР димеров праймеров или других случайных фрагментов, ДНК экстрагировали из среза геля с использованием набора MinElute gel extraction kit® (Qiagen) в соответствии с инструкциями изготовителя. После завершения экстракции из геля проводили дополнительную очистку ДНК с использованием магнитных гранул AMPure magnetic beads® (Beckman-Coulter, Brea, CA) с соотношением ДНК и гранул 1:1,7. Затем ДНК оценивали в отношении концентрации с использованием набора для количественного определения библиотеки на основе количественной ПЦР, чтобы провести секвенирование с помощью Illumina (KAPA) с разведением 1/40000 и 1/80000 и с проведением реакции в трех экземплярах. Исходя из результатов количественной ПЦР ДНК разбавляли до стандартной концентрации 2 нМ и все библиотеки объединяли для секвенирования. Образцы приготавливали для секвенирования с использованием набора cBot cluster generation kit® (Illumina, San Diego, CA) и их секвенировали на Illumina GA2x® со считыванием в секвенировании с парными концами по 100 п.н. в соответствии с инструкциями изготовителя.
СПОСОБ АНАЛИЗА ДАННЫХ ДЛЯ ОБНАРУЖЕНИЯ НЕГОМОЛОГИЧНОГО СОЕДИНЕНИЯ КОНЦОВ В УЧАСТКАХ-МИШЕНЯХ ЦИНКОВЫХ ПАЛЬЦЕВ
После завершения реакции секвенирования и первичного сбора данных, проведенного с использованием биоинформатической системы Illumina для основного анализа, проводили полный анализ для идентификации удаленных оснований в участке-мишени для ZFN в каждом случае. Был разработан специализированный сценарий PERL для компьютерного извлечения и сортировки штриховых кодов из последовательностей ДНК по перечню входящих последовательностей. Штриховой код должен был совпадать с эталонной последовательностью с приемлемым показателем Phred более 30 для снижения неправильного сопоставления считанных последовательностей. После сортировки считанных последовательностей на две различных группы штриховых кодов, которые были использованы, все последовательности пропускали через фильтр качества. Фильтр качества был вторым, специально разработанным сценарием PERL. Считанные данные о последовательностях исключали, если существовало более трех оснований, обозначенных как "N", или если срединный показатель Phred составлял менее 20, или если 3 последовательно расположенных основания имели показатель Phred менее 20, или если считанная последовательность имела длину короче 40 п.н. Оставшиеся последовательности объединяли, где обе из считанных парных последовательностей были доступны, с использованием пакета программ NextGENe® (SoftGenetics, State College, PA). Затем оставшиеся объединенные считанные последовательности сводили к коллекции уникальных считанных последовательностей с использованием третьего специализированного сценария PERL с количеством избыточных последовательностей, которые были идентифицированы, записанным на конце оставшегося идентификатора последовательности. Затем уникальные считанные последовательности выравнивали с эталонной последовательностью FAD2 с использованием программного обеспечения NextGENe®, которое создавало файл выравнивания с пропусками FASTA.
С использованием файла с пропусками FASTA проводили преобразование положения оснований с пропусками во входной эталонный сигнал с использованием четвертого специализированного сценария PERL. Это обеспечило идентификацию оснований, которые различают различных представителей семейства генов (варьирование либо гомологичных, либо паралогичных последовательностей между различными представителями семейства генов) в собранных данных. После проведения преобразования нумерации оснований стало возможным получить отчет о гаплотипе для каждой из уникальных считанных последовательностей и отнести считанные последовательности к конкретным представителям семейства генов. После группировки считанных последовательностей по генам было идентифицировано окно из 10 п.н., и было оценено, что оно окружало участок-мишень для ZFN. Регистрировали количество последовательностей с делециями на ген вместе с количеством отсутствующих оснований.
Затем данные графически отображались в качестве графика с множеством кривых с количеством последовательностей с делецией с 1 по 10 основания на участок-мишень для ZFN на 10000 считанных последовательностей (фиг. 4). Этот анализ проводили для всех трансфекций ZFN вместе с контрольными трансфекциями. В нескольких случаях повторы в нативной последовательности ДНК приводили к увеличению ошибок секвенирования в участке-мишени для ZFN, такую ошибку обычно можно увидеть в качестве увеличения распространенности делеций единичных оснований, которые были сообщены для всех образцов, как для трансфицированных ZFN, так и для контролей (фиг. 5).
Из этих результатов наиболее высокий уровень активности ZFN в участке-мишени FAD2 при определении по наибольшей активности NHEJ, был идентифицирован в локусе E. ZFN, которые кодировались на плазмиде pDAB104010 (т.е. ZFN24828 и 24829), отбирали для нацеливания в растениях на сконструированную способами инженерии платформу для встраивания трансгена (ETIP), учитывая их характеристики значительной активности расщепления геномной ДНК и минимальной активности вне мишени.
ПРИМЕР 4: КОНСТРУКЦИИ ДНК ДЛЯ СКОНСТРУИРОВАННОЙ СПОСОБАМИ ИНЖЕНЕРИИ ПЛАТФОРМЫ ДЛЯ ВСТРАИВАНИЯ ТРАНСГЕНА (ETIP) В ЛИНИЯХ РАСТЕНИЙ КАНОЛЫ
Конструкции плазмидных векторов, описанные ниже, конструировали с использованием способов и технологий, широко известных специалистам в данной области. Использование конкретных реагентов и способов, описанных в этом разделе, хорошо известно специалистам в данной области, и они могут быть без труда заменены другими реагентами и способами для достижения желаемой цели конструирования конструкций плазмидных векторов. Эндонуклеазы рестрикции приобретали от New England BioLabs (NEB; Ipswich, MA). Лигирование осуществляли с помощью ДНК-лигазы T4 (Invitrogen, Carlsbad, CA). Реакции Gateway проводили с использованием смеси ферментов GATEWAY® LR CLONASE® (Invitrogen) для сбора одного исходного вектора в единый конечный вектор. Реакции IN-FUSION™ проводили с использованием технологии IN-FUSION™ Advantage Technology (Clontech, Mountain View, CA) для сбора одного исходного вектора в единый конечный вектор. Получение плазмид проводили с использованием набора NUCLEOSPEN® Plasmid Kit (Macherey-Nagel Inc., Bethlehem, PA) или Plasmid Midi Kit® (Qiagen) в соответствии с инструкциями поставщиков. Фрагменты ДНК выделяли с использованием набора QIAquick Gel Extraction Kit™ (Qiagen) после электрофореза в агарозном Tris-ацетатном геле. Колонии всех собранных плазмид первоначально подвергали скринингу посредством рестрикционного расщепления ДНК Miniprep. Плазмидную ДНК выбранных клонов секвенировала коммерческая организация, осуществляющая секвенирование (Eurofins MWG Operon, Huntsville, AL). Данные о последовательности собирали и анализировали с использованием программного обеспечения SEQUENCHER™ (Gene Codes Corp., Ann Arbor, MI).
ВЕКТОРЫ ДЛЯ ПРЯМОЙ ДОСТАВКИ ДЛЯ ПРИЦЕЛЬНОГО ВСТРАИВАНИЯ ETIP В ЛОКУС FAD2A КАНОЛЫ
Для конструирования содержащих ΕTIΡ векторов pDAS000130 (фиг. 6, вставка T-цепи в качестве SEQ ID NO: 61) для специфического встраивания в ген FAD2A B. napus использовали стандартные способы клонирования. Эту конструкцию конструировали для доставки в протопласты канолы конструкции нуклеазы с цинковыми пальцами pDAB104010. Конструкция нуклеазы с цинковыми пальцами расщепляет локус FAD2A, а затем конструкция pDAS000130 встраивается в геном конолы посредством механизма направляемой гомологией репарации или механизма репарации путем негомологичного соединения концов. ETIP включает четыре экспрессирующих кассеты (две неполных), разделенных дополнительными последовательностями распознавания ZFN и сконструированной способами инженерии подушкой для встраивания (ELP), содержащей другие последовательности распознавания ZFN. Дополнительные последовательности распознавания ZFN являются уникальными, и они были сконструированы для нацеливания встраивания полинуклеотидных последовательностей во вставки трансгенов ETIP и ELP. Аналогично, последовательности распознавания ZFN можно использовать для вырезания полинуклеотидных последовательностей. Первая экспрессирующая ген кассета представляла собой неполную экспрессирующую кассету dsRED, и она содержала промотор, 5'-нетранслируемую область и интрон из гена полиубиквитина 10 (промотор AtUbi) Arabidopsis thaliana (Callis, et al., (1990) J. Biol. Chem., 265: 12486-12493), за которыми следовали 210 п.н. гена dsRed из рифовых кораллов Discosoma sp. (Clontech, Mountain View, CA), кодон-оптимизированного для экспрессии в двудольных растениях (dsRED (оптимизированный для двудольных растений) экзон 1), за которыми следовали интрон из тиоредуктаза-подобного гена Arabidopsis thaliana (интрон 1 из тиоредуктазы At: номер доступа №: NC_00374) и 3'-нетранслируемая область, содержащая терминатор транскрипции и участок полиаденилирования гена Viviparous-1 (Vp1) Zea mays (терминатор Zmlip: Paek et al., (1998) Molecules and Cells , 8(3): 336-342). Вторая экспрессирующая кассета содержала промотор 19S, включая 5'-UTR из вируса мозаики цветной капусты (CaMV 19S: Cook and Penon (1990) Plant Molecular Biology 14(3): 391-405), за которым следовал ген hph из E. coli, кодон-оптимизированный для экспрессии в двудольных растениях (hph(HygR): Kaster et al., (1983) Nucleic Acids Research 11(19): 6895-691 1) и 3-'UTR, содержащая терминатор транскрипции и участок полиаденилирования открытой рамки считывания 1 A. tumefaciens pTi15955 (терминатор At-ORF1: Barker et al., (1983) Plant Molecular Biology 2(6): 335-50). Третья экспрессирующая кассета представляла собой неполную экспрессирующую кассету PAT, и она содержала первый интрон из 4-кумарил-CoA-синтазы Arabidopsis (интрон #2 4-кумарил-CoA-синтазы v: номер доступа № At3g21320/NC003074), за которым следовали последние 256 п.н. синтетической оптимизированной для растений версии гена фосфинотрицинацетилтрансферазы, выделенной из Streptomyces viridochromogenes, которые кодируют белок, который сообщает устойчивость к ингибиторам глутаминсинтетазы, включающим фосфинотрицин, глюфосинат и биалафос (3'-конец PAT(v6): Wohlleben et al., (1988) Gene 70(1): 25-37). Эта кассета терминировалась 3'-UTR, содержащей терминатор транскрипции и участки полиаденилирования открытой рамки считывания 23 pTi15955 A tumefaciens (терминатор AtuORF23: Barker et al., (1983), Plant Molecular Biology 2(6): 335-50). Четвертая экспрессирующая кассета представляла собой кассету с геном ipt, и она содержала укороченную версию промотора размером 588 п.н. и 5'-UTR из гена MYB32 ДНК-связывающего белка Arabidopsis (U26933) (промотор AtMYB32(T): Li et al., (1999) Plant Physiology 121:313), за которыми следовал ген изопентилтрансферазы (ipt) из A. tumefaciens и терминатор 35s, содержащий терминатор транскрипции и участки полиаденилирования из вируса мозаики цветной капусты (терминатор CaMV 35S: Chenault et al., (1993) Plant Physiology 101 (4): 1395-1396). Для доставки в FAD2A, каждый конец последовательности ETIP был фланкирован 1 т.п.н. геномной последовательности FAD2A с каждой стороны от положения двухцепоченого разрыва, индуцируемого доставкой ZFN, кодируемой в pDAB104010, в гене FAD2A B. napus.
Последовательность ETIP синтезировала коммерческая организация, осуществляющая синтез генов (GeneArt, Life Technologies). Сегменты размером 1 т.п.н. геномной последовательности FAD2A амплифицировали с геномной ДНК, очищенной из ткани листьев B. napus DH12075, с использованием набора Qiagen DNeasy plant mini kit® (Qiagen, Hilden) в соответствии с инструкциями, предоставленными поставщиком. Последовательности FAD2A размером 1 т.п.н. лигировали в вектор ΕΤIΡ с использованием лигазы T4 (NEB, Ipswich, MA). Колонии всех собранных плазмид первоначально подвергали скринингу путем рестрикционного расщепления ДНК Miniprep. Эндонуклеазы рестрикции получали от New England BioLabs (NEB, Ipswich, MA) и Promega (Promega Corporation, WI). Получение плазмид проводили с использованием набора QIAprep Spin Miniprep Kit® (Qiagen) или системы Pure Yield Plasmid Maxiprep System® (Promega Corporation, WI) в соответствии с инструкциями поставщиков. Плазмидную ДНК выбранных клонов секвенировали с использованием секвенирования по методу Сэнгера ABI и протокола секвенирования Big Dye Terminator v3.1 cycle sequencing protocol® (Applied Biosystems, Life Technologies). Данные о последовательностях собирали и анализировали с использованием программного обеспечения SEQUENCHER™ (Gene Codes Corp., Arm Arbor, MI).
КОНТРОЛЬНЫЕ ВЕКТОРЫ
Контрольный вектор использовали для разработки способа сортировки клеток на основе активированной флуоресценцией сортировки клеток (FACS). Для конструирования контрольного вектора pDAS000031 (фиг. 7: вставка T-цепи в качестве SEQ ID NO: 62), включающего две экспрессирующих кассеты для генов, использовали стандартные способы клонирования. Первая экспрессирующая кассета для гена содержала промотор вируса мозаики цветной капусты 19s (промотор CaMV 19S; Shillito, et al., (1985) Bio/Technology 3; 1099-1103) :: ген устойчивости к гигромицину (hph(HygR); патент США № 4727028) :: и 3'-нетранслируемую область открытой рамки считывания 1 Agrobacterium tumefaciens (терминатор AtORF1; Huang et al., (1990) J. Bacteriol. 1990 172:1814-1822). Вторая экспрессирующая кассета для гена содержала промотор убиквитина 10 Arabidopsis thaliana (промотор AtUbi10; Callis, et al., (1990) J. Biol Chem., 265: 12486-12493):: dsRED (dsRED(D); патент США № 6852849) и интрон из Arabidopsis (интрон #1; GenBank: AB025639.1) :: 3'-нетранслируемую область открытой рамки считывания 23 Agrobacterium tumefaciens (терминатор AtORF23; патент США № 5428147) в качестве слитой в рамке считывания конструкции с транс-ориентацией (например, ориентация "голова к голове"). Плазмидный вектор собирали с использованием технологии IΝ-FUSION™ Advantage Technology (Clontech, Mountain View, CA).
ПРИМЕР 5: ПОЛУЧЕНИЕ ЛИНИЙ РАСТЕНИЙ КАНОЛЫ С ETIP
ТРАНСФОРМАЦИЯ BRASSICA NAPUS
В сайт-специфическом встраивании в FAD2A задействовано использование конструкции ETIP (pDAS000130) в сопровождении нуклеазы с цинковыми пальцами (pDAB104010) и контрольной конструкции DS-Red (pDAS000031), описанной в примере 4. Бинарные векторы трансформировали в штамм Agrobacterium tumefaciens GV3101:PM90. Трансформацию клеток протопластов Brassica napus осуществляли с использованием протокола трансфекции, описанного в примере 3, с некоторой модификацией.
Модификации протокола включали использование альгината натрия вместо агарозы Sea Plaque™. Эксперименты по трансфекции, в которых как конструкцию нуклеазы с цинковыми пальцами pDAB104010, так и конструкцию ETIP pDAS000130 совместно доставляли в клетки протопластов Brassica napus, осуществляли в концентрациях ДНК, составляющих молярное соотношение 5:1 и 12:1, где в случае pDAS000130 имел концентрацию 27,8 мкг плазмидной ДНК и pDAB104010 имел концентрацию 2,2 мкг плазмидной ДНК. Контрольные плазмидные конструкции трансформировали в концентрациях 30 мкг плазмидной ДНК.
Дополнительные модификации протокола включали восстановление целых растений из трансформированных протопластов клеток в среде, содержавшей 1,5 мг/мл гигромицина. Восстановление целых растений требовало, чтобы среду A заменяли каждые две недели и проводили мониторинг роста происходящих из протопластов колоний. После вырастания происходящих из протопластов колоний до размера приблизительно 2-3 мм в диаметре колонии переносили в индивидуальные ячейки планшета с 12 ячейками Costar® (Fisher Scientific, St. Louis, MO), содержавшие застывшую среду MS morpho. Планшеты инкубировали в течение от одной до двух недель при 24°C при непрерывном тусклом освещении до тех пор, пока каллюсы не пролиферировали до размера 8-10 мм в диаметре.
После достижения протопластами клеток диаметра 1-2 см протопласты клеток переносили в индивидуальные 250-мл емкости для культивирования, содержавшие среду MS morpho. Емкости инкубировали при 24°C в условиях 16 ч на свету (белые трубки Osram L36 W/21 Lumilux с 20 мкМоль м-2 с-1) и 8 ч в темноте. В течение от одной до двух недель становилось видно множество побегов. Побеги переносили в 250-мл емкости для культивирования, содержавшие среду MS, после того, как они достигали длины 3-4 см. 250-мл емкости для культивирования инкубировали при 24°C в условиях 16 ч на свету (белые трубки Osram L36 W/21 Lumilux с 20 мкМоль м-2 с-1) и 8 ч в темноте. Побеги поддерживали в емкостях для культивирования до тех пор, пока они не развивались в проростки, и в это время их переносили в теплицу для выращивания до зрелого состояния.
ПРИМЕР 6: МОЛЕКУЛЯРНОЕ ПОДТВЕРЖДЕНИЕ ВСТРАИВАНИЯ T-ДНК, СОДЕРЖАЩИХ ETIP, В КАНОЛЕ
Геномную ДНК экстрагировали из ткани листьев всех предполагаемых трансгенных растений с использованием набора DNeasy Plant Mini Kit™ (Qiagen) в соответствии с инструкциями изготовителя, за исключением того, что ткань извлекали в 80 мкл буфера AE. Тридцать миллиграмм ткани молодых листьев из регенерировавших растений быстро замораживали в жидком азоте, а затем растирали в порошок.
Молекулярную охарактеризацию локуса FAD2A проводили с использованием трех независимых анализов. Анализы были разработаны и оптимизированы с использованием следующих контролей: охарактеризованные трансгенные события, содержащие один случайным образом встроенный трансген, охарактеризованное трансгенное событие с пятью случайным образом встроенными трансгенными, растения канолы дикого типа сорта DH12075 и контрольные реакции без матрицы. Результаты трех описанных ниже молекулярных анализов рассматривали вместе для получения доказательства встраивания ΕΤIΡ в FAD2A посредством HDR.
ИДЕНТИФИКАЦИЯ ВСТРАИВАНИЯ ТРАНСГЕНА С ПОМОЩЬЮ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ В РЕАЛЬНОМ ВРЕМЕНИ
Четыре реплики для каждого растения анализировали с использованием праймеров, специфичных к гену-мишени hph (также описанному как hpt) (SEQ ID NO: 63, hpt F791 5' CTTACATGCTTAGGATCGGACTTG 3'; SEQ ID NO: 64, hpt R909 5' AGTTCCAGCACCAGATCTAACG 3'; SEQ ID NO: 65, hpt Taqman 872 5' CCCTGAGCCCAAGCAGCATCATCG 3' FAM) (фиг. 8) и эталонному гену, кодирующему белок группы высокой подвижности I/Y (HMG I/Y) (SEQ ID NO: 66, F 5' CGGAGAGGGCGTGGAAGG 3'; SEQ ID NO: 67, R 5' TTCGATTTGCTACAGCGTCAAC 3'; SEQ TD NO: 68, зонд 5' AGGCACCATCGCAGGCTTCGCT 3' HEX). Реакционные смеси амплифицировали с использованием следующих условий: 95°C в течение 10 минут, затем 40 циклов из 95°C в течение 30 секунд, 60°C в течение 1 минуты, причем данные амплификации получали в конце каждой стадии отжига. Количество копий вычисляли с использованием способа ACq, где ACq=Cq(ген-мишень) - Cq(эталонный ген). Livak, K.J. и T.D. Schmittgen, Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods, 2001. 25(4): p. 402-8. Растения с амплификацией hph и HMG I/Y и количеством копий 0,5 или более считали трансгенными, в то время как растения с количеством копий ≥0,5 и ≤1,2 оценивали как предположительно имеющие одну копию. Амплификацию проводили на системе для детекции BioRad CFX96 Touch™ Real-Time PCR Detection System с FastStart Universal Probe Master (ROX) (Roche, Basel, Швейцария).
ОБНАРУЖЕНИЕ РАЗРУШЕННОГО УЧАСТКА ZFN В FAD2A
Каждое растение анализировали в отношении присутствия или отсутствия эндогенной мишени в исследовании разрушенного локуса, которое является основным анализом. Анализ представляет собой анализ SYBR® Green I qPCR и в моноплексном формате, но при этом каждую реакцию проводят одновременно на одном и том же планшете для ПЦР, он нацелен на эндогенный локус (FAD2A/2C.RB.UnE.F1, SEQ ID NO: 69, 5' CTTCCACTCCTTCCTCCTCGT*C 3' и FAD2A 2C.RB.UnE.R1, 5' SEQ ID NO: 70, GCGTCCCAAAGGGTTGTTGA*G 3') и локус ZFN (локус, с которым связывается ZFN pDAB104010 и расщепляет геном) (FAD2A.UnE.F1, SEQ ID NO: 71, 5' TCTCTACTGGGCCTGCCAGGG*C 3' и FAD2A.UnE.R1, SEQ ID NO: 72, 5' CCCCGAGACGTTGAAGGCTAAGTACAA*A 3') (фиг. 9). Обе пары праймеров амплифицировали с использованием следующих условий: 98°C в течение 30 секунд, затем 35 циклов (98°C в течение 10 секунд, 65°C в течение 20 секунд, 72°C в течение 90 секунд), за которыми следуют 95°C в течение 10 секунд, а затем анализ плавления от 50°C до 95°C с приращением 0,5°C в течение 0,05 секунд и считыванием планшета при каждом приращении. Условия реакции приведены в таблице 9.
Растения, которые имели амплификацию эндогенной мишени, однако не имели амплификации мишени ZFN, оценивали как положительные в испытании разрушения локуса, и их считали имеющими разрушенный локус ZFN. Этот анализ считали положительным, когда участок связывания ZFN на обоих аллелях в локусе FAD2A был разрушен.
ОБНАРУЖЕНИЕ СПОСОБОМ ПЦР ВСТРАИВАНИЯ ТРАНСГЕНА В FAD2A ПОСРЕДСТВОМ НАПРАВЛЯЕМОЙ ГОМОЛОГИЕЙ РЕПАРАЦИИ
Каждое предполагаемое трансформированное растение анализировали с использованием конечного результата, достигаемого с праймерами для ПЦР, сконструированными для амплификации трансгена-мишени hph (hph_ExoDigPC_F1, SEQ ID NO: 73, 5' TTGCGCTGACGGATTCTACAAGGA 3' и hph_ExoDigPC_R1, SEQ ID NO: 74, 5' TCCATCAGTCCAAACAGCAGCAGA 3'), эндогенным локусом FAD2A (FAD2A.Out.F1, SEQ ID NO: 75, 5' CATAGCAGTCTCACGTCCTGGT*C 3' и FAD2A.Out.Rvs3, SEQ ID NO: 76, 5' GGAAGCTAAGCCATTACACTGTTCA*G 3'), областью, охватывающей 5'-конец трансгена, встроенного в локус FAD2A посредством HDR и область выше трансгена в локусе FAD2A (FAD2A.Out.F1, SEQ ID NO: 77, 5' CATAGCAGTCTCACGTCCTGGT*C 3' и QA520, SEQ ID NO: 78, 5' CCTGATCCGTTGACCTGCAG 3') и областью, охватывающей 3' конце любого трансгена, встроенного в локус FAD2A посредством HDR и область ниже трансгена в локусе FAD2A (QA558, SEQ ID NO: 79, 5' GTGTGAGGTGGCTAGGCATC 3' и FAD2A.Out.Rvs3, SEQ ID NO: 80, 5' GGAAGCTAAGCCATTACACTGTTCA*G 3') (фиг. 3). Все пары праймеров амплифицировали с использованием следующих условий: 98°C в течение 30 секунд, затем 35 циклов (98°C в течение 10 секунд, 65°C в течение 20 секунд, 72°C в течение 90 секунд). Условия и реагенты реакции описаны в таблице 10.
Амплификация участка 5'-трансген-геном, фланкирующий мишень, и/или амплификация участка 3'-трансген-геном, фланкирующий мишень, указывала на предполагаемое событие встраивания. Необходимо отметить, что вследствие наличия участков гомологии FAD2A размером приблизительно 1000 п.н. в кассете pDAS000130 (содержащей полинуклеотидные последовательности со 100% идентичностью последовательности с областями FAD2A непосредственно выше участка расщепления ZFN), в реакциях ПЦР происходила ложно-положительная амплификация продукта ПЦР вследствие химеризма ПЦР из-за амплификации событий встраивания вне мишени ETIP. Амплификация мишени hph подтвердила, что произошло встраивание трансгена. Амплификация мишени FAD2A указывает на то, что локус FAD2A является интактным или содержит только частичную вставку. Вследствие размера ETIP (11462 п.н. для кассет ETIP или 13472 п.н., включая гомологичные участки FAD2A и кассеты ETIP) ожидается, что праймеры для FAD2A не будут амплфицировать продукт, когда интактный ETIP встроен в локус FAD2A.
ОБНАРУЖЕНИЕ С ПОМОЩЬЮ САУЗЕРН-АНАЛИЗА РЕДАКТИРОВАНИЯ FAD2A
Растения, которые имели либо амплификацию продукта в виде участка 5'-трансген-геном, фланкирующий мишень, и/либо амплификацию участка 3'-трансген-геном, фланкирующий мишень, или отсутствие амплификации локуса-мишени ZFN, или оба из них, подвергали саузерн-анализу для обнаружения встраивания трансгена в локус FAD2A. Геномную ДНК из 5 г ткани листьев с использованием модифицированного способа CTAB (Maguire, T.L., G.G. Collins, and M. Sedgley A modified CTAB DNA extraction procedure for plants belonging to the family proteaceae. Plant Molecular Biology Reporter, 1994. 12(2): p. 106-109). Далее 12 мкг геномной ДНК расщепляли Kpn1-F (New England BioLabs) и фрагменты расщепления разделяли электрофорезом на 0,8% агарозном геле, а затем переносили на мембрану с использованием стандартного протокола саузерн-блоттинга. Праймеры для 5'-области-мишени FAD2A (F, SEQ ID NO: 81, 5' AGAGAGGAGACAGAGAGAGAGT 3' и R, SEQ ID NO: 82, 5' AGACAGCATCAAGATTTCACACA 3'), 3'-области мишени FAD2A (F, SEQ ID NO:83, 5' CAACGGCGAGCGTAATCTTAG 3' и R, SEQ ID NO: 84, 5' GTTCCCTGGAATTGCTGATAGG 3') и hph (F, SEQ ID NO:85, 5' TGTTGGTGGAAGAGGATACG 3' и R, SEQ ID NO: 86, 5' ATCAGCAGCAGCGATAGC 3') использовали для получения зондов для обнаружения присутствия ETIP в локусе FAD2A с использованием системы DIG Easy Hyb System® (Roche, South San Francisco, CA) в соответствии с инструкциями изготовителя (фиг. 11). Гибридизацию проводили при 42°C для 5'-области FAD2A, 45°C для 3'-области FAD2A и 42°C для обнаружения hph.
Связанную с мембраной геномную ДНК исследовали с помощью зондов в конкретном порядке; сначала исследовали с помощью зондов 5'-последовательности FAD2A, затем исследовали с помощью зондов 3'-последовательности FAD2A, и, наконец, исследовали с помощью зондов последовательности hph (фиг. 12). Обоснование для этого является следующим. Первый зонд (FAD2A 5') представляет собой диагностический зонд, и, если ETIP встроился в FAD2A посредством полной HDR, на мембране будет виден фрагмент размером 5321 п.н. Полученный размер полосы легко дифференцировать в процессе электрофореза, и он будет расположен вблизи фрагментов размером 5148 п.н. в меченном DIG маркере молекулярной массы ДНК Roche DNA Molecular Weight Marker III® (Roche, Indianapolis, IN). Второй зонд мембраны представляет собой зонд FAD2A 3' и растение после редактирования будет иметь фрагмент размером 22433 п.н., в то время как растение без редактирования будет иметь фрагмент размером 16468 п.н. Тот же фрагмент размером 22433 п.н., идентифицированный с помощью зонда FAD2A 3', также должен связываться и идентифицироваться зондом hph. Эти фрагменты трудно различить на геле, поскольку они являются чрезвычайно крупными и может быть трудно определить какое-либо отличие между фрагментом, находящимся выше или ниже наиболее крупного фрагмента размером 21226 п.н. в меченном DIG маркере молекулярной массы ДНК Roche DNA Molecular Weight Marker III®. По существу, эти зонды обеспечивают доказательства, которые могут подтвердить идентификацию встраивания ETIP в FAD2A через направляемую гомологией репарацию (HDR) путем визуализации фрагмента размером 5 т.п.н. с использованием зонда FAD2A 5'. Фермент рестрикции Kpn1 был единственной подходящей эндонуклеазой рестрикции для применения в этом анализе, поскольку участки Kpn1 встречались в одном локусе расщепления кассеты ETIP, и они присутствовали в двух участках локуса ZFN для FAD2A. Один участок был расположен выше, а второй участок был расположен ниже участков гомологии FAD2A. Кроме того, Kpn1 не является чувствительной к метилированию и доступна в качестве рекомбинантного фермента с повышенной точностью (New England Biolabs).
Результаты молекулярного анализа и саузерн-анализа
После трансфекции, культивирования и селекции трансгенные растения переносили в почву. В этом процессе 139 растений выжило и для них проводили взятие образцов тканей для экстракции и анализа гДНК. Все 139 растений анализировали для оценки количества копий. Среди этих 139 растений, 56 были положительными в отношении ΕΤIΡ и 11 из 56 положительных растений имели предполагаемое встраивание одной копии (фиг. 13) (таблица 11). Из 56 растений, которые были положительными в отношении встраивания ETIP, амплификация 5'-геномной фланкирующей трансген последовательности FAD2A происходила в 7 растениях. Амплификация 3'-геномной фланкирующей трансген последовательности FAD2A не происходила ни в одном из 56 растений, которые были положительными по встраиванию ETIP. Кроме того, среди 56 растений, которые были положительными по встраиванию трансгена, 11 растений были положительными в исследовании с помощью q-ПЦР разрушения локуса. Четырнадцать растений, которые были положительными по амплификации FAD2A 5' геномной фланкирующей трансген последовательности и/или положительными в исследовании с помощью q-ПЦР разрушения локуса, подвергали саузерн-анализу с 3 зондами, описанными выше. Среди 14 растений, для которых проводили саузерн-анализ, все растения продемонстрировали частичное встраивание в локус FAD2A, однако ни одно из этих растений не продемонстрировало доказательств полноразмерного встраивания ETIP в локус FAD2A посредством HDR, когда проводили обработку зондом FAD2A 5', зондом FAD2A 3' и зондом hph. Не было выявлено полос, которые оказались i) большими, чем WT, и ii) идентичными полосам, выявленным для этих образцов, когда проводили обработку зондом FAD2A 3' (таблица 11).
РЕЗУЛЬТАТЫ ДЛЯ ТРАНСГЕННОЙ КАНОЛЫ С ETIP, ТРАНСФОРМИРОВАННОЙ PDAS000130 И PDAB104010
Трансгенные события Brassica napus, которые получены путем трансформации pDAS000130 и DAB104010, приводят к встраиванию единичной копии полноразмерной вставки T-цепи полинуклеотидной последовательности ETIP из pDAS000130 в локус FAD2A. От трех до четырех событий полностью охарактеризовывают и подтверждают, что они содержат встроенный ETIP. Подтверждение осуществляют способом амплификации ПЦР внутри-снаружи, и далее подтверждают с помощью саузерн-блоттинга. Выбранные растения с событиями T0 выращивают до стадии развития T1. Растения T1 повторно подвергают скринингу для определения зиготности встроенной T-цепи. Подвергнутые скринингу события распределяют на категории в качестве гомозиготных, гемизиготных или нулевых.
Гомозиготные события используют для получения протопластов посредством ранее описанного способа. Затем протопласты котрансформируют по меньшей мере одной нуклеазой с цинковыми пальцами, которая сконструирована для нацеливания на участок связывания, который включен в последовательность ETEP, и донорной плазмидой, которая обладает гомологией с конкретными областями ETIP, где донор встраивается в ETIP посредством механизма HDR. Аналогично, протопласты впоследствии котрансформируют по меньшей мере одной нуклеазой с цинковыми пальцами, которая сконструирована для нацеливания на участок связывания, который включен в последовательность ETIP, и донорной плазмидой, которая не обладает гомологией с конкретными областями ETIP, где донор встраивается в ETIP посредством механизма негомологичного соединения концов. ZFN (несколько ZFN) расщепляет(ют) локус ETIP и донорная плазмида встраивается в геном клеток Brassica napus посредством направляемой гомологией репарации или негомологичного соединения концов.
В результате встраивания донорной плазмиды, частичный трансген DS-red репарируется до полноразмерного трансгена DS-red. Экспрессию теперь полностью функционального трансгена DS-red используют для сортировки протопластов клеток способом FACS. Предполагаемые трансгенные растения сортируют с использованием способа FACS, описанного в примере 7, и выделенные протопласты регенерируют в зрелые растения. Встраивание донорной плазмиды подтверждают в растениях с нацеливанием на ETIP с использованием способов молекулярного подтверждения. По существу, локус ETIP служит в качестве сайт-специфического локуса для направленного встраивания гена донорной полинуклеотидной последовательности.
ПРИМЕР 7: СОРТИРОВКА НА ОСНОВЕ FACS КЛЕТОК ПРОТОПЛАСТОВ
Протопласты Brassica napus, которые были трансфицированы контрольной конструкцией DS-Red pDAS000031, сортировали с помощью опосредуемой FACS сортировки клеток с использованием сортера BD Biosciences Influx-Cell sorter™ (San Jose, CA). Протопласты клеток выделяли и трансфицировали, как описано в примере 3. После трансфекции клеток pDAS000031, клетки сортировали с использованием сортера FACS с условиями, описанными в таблице 12.
Протопласты, которые экспрессировали трансген DS-red, сортировали и выделяли. Выделенные с помощью FACS протопласты подсчитывали с использованием сортера. Приблизительно от 1×105 до 1,8×105 клеток помещали в лунку 24-луночного микропланшета для титрования на первые сутки после выделения с помощью FACS. Клетки переносили в гранульную культуру на от 5 до 20 суток. Исследовали сходные условия, где приблизительно 1×104 клеток помещали в ячейку микропланшета для титрования с 2 или 4 ячейками на вторые сутки после выделения с помощью FACS. Различные условия, которые исследовали, привели к выделению клеток с жизнеспособностью 95-98% от всех выделенных протопластов клеток. Отсортированные с помощью FACS протопласты клеток переносили в гранульную культуру на 3-20 суток. Отсортированные с помощью FACS протопласты клеток регенерировали в растения на среде, которая содержала 1,5 мг/мл гигромицина, с использованием описанного выше протокола. Было подтверждено, что предполагаемые трансгенные растения содержат интактную вставку T-цепи из pDAS000031 с помощью протоколов молекулярного подтверждения.
Способ сортировки FACS прямо применим для скрининга любой флуоресцентной последовательности трансгена, и его используют для выделения части протопластов клеток Brassica napus, на которую нацелился флуоресценоный трансген через опосредуемую гомологией репарацию в конкретном участке области ETIP в геномном локусе.
ПРИМЕР 8: НАПРАВЛЕННОЕ ВСТРАИВАНИЕ И РАЗРУШЕНИЕ ДЕСАТУРАЗЫ ОМЕГА-3 ЖИРНЫХ КИСЛОТ (FAD2) Brassica napus ПОСРЕДСТВОМ НАПРАВЛЯЕМОЙ ГОМОЛОГИЕЙ РЕПАРАЦИИ
ВЫБОР ДОМЕНОВ С ЦИНКОВЫМИ ПАЛЬЦАМИ, СПЕЦИФИЧНЫХ К FAD2A
Транскрибируемые области гомологичных генов FAD2 идентифицировали и охарактеризовывали нуклеазы с цинковыми пальцами, которые были сконструированы для связывания и расщепления этих участков для опосредуемого NHEJ нацеливания донорной последовательности. Белки с цинковыми пальцами (ZFP), направленные против последовательностей ДНК из гомологов последовательностей FAD2, конструировали и исследовали, как описано выше. Из ZFN, демонстрирующих активность в отношении мишени, был отобран один белок с цинковыми пальцами, который расщепляет мишень FAD2 с высокой эффективностью: ZFP 24828-2A-24829 распознает SEQ ID NO: 35 5'- agGCCCAGtAGAGAGGCCaggcgaagta -3' и SEQ ID NO: 36 5' -ccAGGGCTGCGTCCTAACCGgcgtctgg -3'. Было показано, что этот ZFN специфически связывается и расщепляет геномный локус FAD2A.
МОДЕЛИРОВАНИЕ И КОНСТРУИРОВАНИЕ "ДОНОРНЫХ" ВЕКТОРОВ ДЛЯ КОНТРОЛИРУЕМОЙ HDR РЕПАРАЦИИ ДНК
Для встраивания донорной последовательности посредством HDR конструировали единый вектор. Этот вектор кодировал экспрессирущую кассету с геном устойчивости к гигромицину (hph или hpt). Экспрессирующая кассета с геном устойчивости к гигромицину включала промотор 19S, включающий 5'-UTR из вируса мозаики цветной капусты (CaMV) (Cook and Penon Plant Molecular Biology 1990 14(3), 391-405), за которым следует ген гигромицинфосфотрансферазы (hph) (Kaster et al Nucleic Acids Research 1983 11 (19), 6895-6911). Ген hph был кодон-оптимизирован для экспрессии в двудольных растениях, и он фланкировался 3'-UTR, содержащей терминатор транскрипции и участок полиаденилирования открытой рамки считывания 1 (ORF1) A. tumefaciens pTi15955 (Barker et al., Plant Molecular Biology 1983, 2(6), 335-50). Кассеты синтезировала коммерческая организация, осуществляющая синтез генов (GeneArt, Life Technologies, Regensberg, Германия). Фланкирующие последовательности FAD2A присоединяли выше и ниже экспрессирующей кассеты для гена. Кассету устойчивости к гигромицину клонировали в конкретные участки фермента рестрикции каждого вектора, что приводило к "донорным" векторам: pDAS000129 (донор гена устойчивости к гигромицину со сплайсингом: SEQ ID NO: 87, фиг. 14).
Колонии собранных плазмид первоначально подвергали скринингу путем расщепления эндонуклеазой рестрикции ДНК, очищенной из E.coli, культивированных в течение ночи. Эндонуклеазы рестрикции получали от NEW ENGLAND BIOLABS™ (NEB, Ipswich, MA) и PROMEGA™ (Promega Corporation, WI). Получение плазмид проводили с использованием набора QIAPREP SPIN MINIPREP KIT™ (Qiagen, Hilden, Germany) или системы PURE YIELD PLASMID MAXIPREP SYSTEM™ (Promega Corporation, WI) в соответствии с инструкциями поставщиков. После подтверждения полученных фрагментов рестрикции с помощью агарозного гель-электрофореза, плазмидную ДНК выбранных клонов секвенировали с использованием секвенирования методом Сэнгера ABI и протокола циклического секвенирования BIG DYE TERMINATOR V3.1™ (Applied Biosystems, Life Technologies). Данные о последовательности собирали и анализировали с использованием программного обеспечения SEQUENCHER™ (Gene Codes, Ann Arbor, MI).
ТРАНСФОРМАЦИЯ BRASSICA NAPUS
Опосредуемая PEG трансфекция протопластов и регенерация ETIP, нацеленных на FAD2a и ZFN для FAD2a (точные события)
Происходящие из мезофилла протопласты выделяли из трехнедельных стерильных культур побегов Brassica napus (DH10275). Соответствующие семена проращивали. Семена подвергали стерилизации поверхности с использованием 70% этанола в течение 1 минуты путем осторожного встряхивания, а затем 3-4 промываний в стерильной дважды дистиллированной воде, а затем стерилизовали с использованием 20% отбеливателя и 10 мкл Tween 20™; семена обрабатывали отбеливателем на устройстве для встряхивания (настольное ротационное устройство для встряхивания при приблизительно 100 об/мин) в течение 15 минут, а затем проводили 3-4 промывания в стерильной дважды дистиллированной воде, семена тщательно переносили на стерильную фильтровальную бумагу для удаления избытка жидкости и высевали на среду для проращивания семян (½ концентрация витаминов MS/B5+1% сахароза+0,8% агар; pH 5,8, и наливали 50-60 мл среды на чашку Петри (15×100 мм), которую помещали под небольшим углом с использованием подставки); в каждую чашку помещали приблизительно 50 семян. Чашки инкубировали вертикально при 22°C при освещении 16 ч/с (20 мкмоль м-2 с-1) в течение 6 суток. Сегменты гипоктиля размером 0,5 см вырезали из проростков в возрасте шести суток и культивировали на среде для индукции побегов (витамины MS/B5+3% сахароза+500 мг/л MES+BAP (13 мкМ)+Zeatin (5 мкМ)+нитрат серебра (5 мг/л)+0,8% агар (pH 5,8) и перелитая в стерильные чашки Петри 100×20 мм), в каждую чашку помещали приблизительно 20 эксплантатов. Меристемы побегов, которые появились через 3-4 недели, переносили в среду для удлинения побегов (витамины MS/B5+2% сахароза+500 мг/л MES+BAP (2 мкМ)+GA-3 (0,1 мкМ)+0,8% агар (pH 5,8) и перелитая в 250-мл емкости для культивирования) и культуры поддерживали в этой среде в течение 4 недель с одним раундом субкультивирования между ними. Затем побеги высотой 2-3 см переносили в среду для инициации корней (1/2 концентрация витаминов MS/B5+1% сахароза+500 мг/л MES+IBα (2,5 мкМ)+0,6% агар (pH 5,8) и перелитая в 700-мл емкости для культивирования) для развития корней. Укорененные побеги субкультивировали в свежей среде для инициации корней с интервалами 3-4 недели в качестве черенков стеблей в течение двух-трех раундов перед применением. Культуры поддерживали при 22°C в условиях освещения 16 ч/с (30 мкмоль м-2 с-1).
Выделение и очистка протопластов
Выращенные in vitro растения Brassica napus DH12075 использовали в качестве источника эксплантатов для выделения протопластов мезофилла. От 3-го до 4-го верхние полностью раскрытые листья ростков в возрасте от 3 до 4 недель нарезали на небольшие полоски (от 0,5 до 1 мм) с помощью острого скальпеля для выделения протопластов. Ферментативное расщепление проводили обработкой 250-500 мг материала листьев 25 мл буфера для расщепления (1,2% (масс./об.) целлюлаза "Onozuka" R10® и 0,2% (масс./об.) Macerozyme® R10, растворенные в среде K4 (Spangenberg et al. 1998)). Чашку Петри, содержавшую материал листьев и буфер для расщепления, накрывали Parafilm™ и инкубировали при комнатной температуре в течение от 12 до 15 ч в темноте. После инкубации в течение ночи продукты расщепления фильтровали через клеточное сито BD® (размер ячеек 70 мкм). Суспензию протопластов (5-6 мл) собирали в 14-мл круглодонную пробирку, и на нее наслаивали 1 мл промывочного раствора буфера W5 (154 мМ NaCl, 125 мМ CaCl2, 5 мМ KCl и 5 мМ глюкоза; pH 5,8 Menczel et al. 1981) и центрифугировали при 400 об/мин в течение 10 мин. После центрифугирования протопласты, которые плавали на поверхности контакта, отбирали и промывали центрифугированием с использованием 10 мл буфера W5 при 400 об/мин в течение 10 мин. После последнего промывания выделенные протопласты ресуспендировали при плотности 1×106 протопластов на мл буфера и инкубировали в течение 1 часа перед трансфекциями.
Оценка выхода и жизнеспособности протопластов
Выход протопластов оценивали с использованием гемоцитометра в соответствии с Sambrook and Russel, 2006 и жизнеспособность исследовали с использованием красителя голубой Эванс (400 мг/л, растворенный в 0,5 M манните) в соответствии с Huang et al. 1996 с небольшими модификациями.
Опосредуемая PEG4000 стабильная доставка ДНК
Плазмидную ДНК содержащего ETIP вектора pDAS000129 и вектора для ZFN (pDAB104010), нацеленных на локус FAD2A, выделяли из культур E. coli с использованием Pure Yield Plasmid Maxiprep System™ (Promega Corporation, WI) или Plasmid Maxi Kit™ (Qiagen, Hilden) в соответствии с инструкциями поставщиков. Плазмидную ДНК растворяли при плотности 0,7 мкг на мл стерильной дважды дистиллированной воды. Использовали всего тридцать микрограмм (30 мкг) плазмидной ДНК (5:1 моль/л pDAS000129 и pDAB104010) на один миллион протопластов (жизнеспособность >95), суспендированных в 100 мкл буфера для трансформации (15 мМ MgCl2, 0,1% (масс./об.) морфолиноэтансульфоновая кислота (MES) и 0,5 M маннит; pH 5,8), а затем 150 мкл раствора PEG (40% (масс./об.) PEG 4000 в 0,4 M манните и 0,1 M Ca(NO3)2 (pH 6-7) Spangenberg and Potrykus (1995). Контрольные трансформации включали всего тридцать микрограмм (30 мкг) плазмидной ДНК либо pDAS000129, либо pDAB104010. После инкубации в течение 10-15 минут при комнатной температуре капельно добавляли 5 мл буфера W5 и протопласты осторожно смешивали и в суспензию протопластов добавляли дополнительные 5 мл буфера W5 в виде медленного потока. Протопласты осторожно перемешивали и центрифугировали при 400 об/мин в течение 10 мин и суспернатант W5 осторожно удаляли, оставляя протопласты в форме осадка. Затем трансфицированные протопласты инкубировали в 1 мл буфера W5 при комнатной температуре, после чего их погружали в культуры гранульного типа. Образцы протопластов котрансфицировали конструкцией pDAS000129 и pDAB104010, обладающей жизнеспособностью клеток в диапазоне 60-80%, сразу после трансфекции. Трансфицированные протопласты погружали либо способом с агарозой sea plaque, либо способом с альгинатом натрия.
Культивирование происходящих из мезофилла протопластов для выделения жизнеспособных микрокаллюсов
Перед погружением трансфицированные протопласты центрифугировали при 400 об/мин в течение 10 мин и буфер W5 осторожно удаляли. Затем протопласты ресуспендировали в 0,5 мл среды K3 (Spangenberg et al. 1998). Точно 0,5 мл перенесенной суспензии протопластов (приблизительно 5×105 протопластов) помещали в 6-см чашку Петри и в нее добавляли 4,5 мл предварительно нагретой (расплавленной в микроволновой печи и инкубированной на водяной бане при 40-45°C) смеси 1:1 сред K3:H (Spangenberg et al. 1998), содержавшей 0,6% агарозу Sea Plaque™. Агарозу и суспензию протопластов осторожно перемешивали и позволяли им осесть. После застывания (через 20-30 мин), чашки накрывали Parafilm® и протопласты культивировали в течение 24 ч в темноте при 24°C, а затем в течение 6 суток при постоянном тусклом освещении (5-10 мкмоль м-2 с-1), когда происходило первое и множественное деление клеток. Через 6 суток протопласты, погруженные в агарозу, разрезали на четыре квадранта и помещали в 100 мл среды A (Spangenberg et al. 1998) в 700-мл емкость для культивирования. Жидкую среду A дополняли 1,5 мг/л гигромицина. Культуры инкубировали на ротационном устройстве для встряхивания при 80-100 об/мин при 24°C с постоянным тусклым освещением. Устойчивые колонии появлялись через 5-6 недель и 3-4 недели после посева протопластов в случае способов с агарозой sea-plaque и альгинатом натрия, соответственно. Микрокаллюсы размером 2-3 мм в диаметре переносили в среду B1 (витамины MS/MS+3,5% сахароза+500 мг/л MES+BAP (5 мкМ) +NAA (мкМ)+2,4-D (мкМ)+1,5 мг/л гигромицина+0,7% агароза типа I (pH 6,0) и перелитая в стерильную чашку Петри размером 100×20 мм) путем осторожного разрушения агарозных гранул. Полученные таким образом микрокаллюсы ресуспендировали в достаточном количестве жидкости A (50 мл жидкости A использовали на один мл объема осажденных клеток (SCV: его измеряли после переноса всех высвободившихся микрокаллюсов в стерильную 50- или 15-мл пробирку falcon и позволения осесть в течение 5 мин)). После единообразного перемешивания микрокаллюсов, 0,5 мл микрокаллюсов, суспендированных в жидкой среде A, переносили в планшеты с B1 и с использованием 1-2 мл дополнительной жидкой среды A микрокаллюсы распределяли единообразно в среде B1 и избыток жидкой среды A осторожно извлекали из каждого планшета. Планшеты накрывали с использованием ленты из микропористого материала, которая усиливала созревание зародышей.
Способ с альгинатом натрия
Перед погружением трансфицированные протопласты центрифугировали при 400 об/мин в течение 10 мин и буфер W5 осторожно удаляли. Затем протопласты ресуспендировали в 1,0 мл 0,5 M маннита и инкубировали на льду. К нему добавляли равный объем 1,0% альгината натрия и осторожно перемешивали. Суспензию протопластов инкубировали на льду, после чего погружали. Формирующий гранулы раствор (0,4 M маннит+50 мМ CaCl2 (pH 5,8)) переносили в стерильный планшет с шестью ячейками (3-4 мл на ячейку) с использованием серологической пипетки. Точно 1,0 мл суспензии протопластов капельно добавляли с использованием 1-мл пипетки в формирующий гранулы раствор и каждый трансфицированный образец (приблизительно 5×105 протопластов) погружали в каждую ячейку. Суспензию протопластов инкубировали в течение 1-2 часов при комнатной температуре для формирования гранул альгината натрия. После периода инкубации формирующий гранулы раствор осторожно выделяли и заменяли 4-5 мл смеси 1:2 среды K3+H:A (Spangenberg et al. 1998), дополненной 1,5 мг/л гигромицина. Протопласты культивировали в течение 3-4 недель в темноте при 22°C в устройстве для встряхивания (50 об/мин). Через 3-4 недели устойчивые микрокаллюсы (0,5-1,0 мм) высвобождали путем обработки буфером для деполимеризации (0,3 M маннит+20 мМ цитрат натрия (pH 5,8)). После удаления жидкой среды в каждую ячейку, содержавшую культуры гранульного типа, добавляли 3-4 мл буфера для деполимеризации и инкубировали при комнатной температуре в течение 2 часов. С использованием стерильных щипцов гранулы осторожно перемешивали для усиления эффективного высвобождения микрокаллюсов. С использованием стерильной 1,0-мл пипетки осторожно удаляли смешанный гелеобразующий агент, высвободившийся в буфер для деполимеризации. Микрокаллюсы промывали дважды с использованием 5 мл жидкой среды A и микрокаллюсы ресуспендировали в достаточном количестве в жидкости A (50 мл жидкости A использовали на один мл объема осажденных клеток (SCV: его измеряли после переноса всех высвободившихся микрокаллюсов в стерильную 50- или 15-мл пробирку falcon и позволения осесть в течение 5 мин)). После единообразного перемешивания микрокаллюсов, 0,5 мл микрокаллюсов, суспендированных в жидкой среде A, переносили в планшеты со средой B1 (витамины MS/MS+3,5% сахароза+500 мг/л MES+BAP (5 мкМ)+NAA (5 мкМ)+2,4-D (5 мкМ)+1,5 мг/л гигромицина+0,7% агарозы типа I (pH 6,0) и перелитая в стерильную чашку Петри 100×20 мм) и с использованием 1-2 мл дополнительной жидкой среды A микрокаллюсы распределяли единообразно в среде B1 и избыток жидкой среды A осторожно извлекали из каждого планшета. Планшеты накрывали с использованием ленты из микропористого материала, которая усиливала созревание зародышей. Культуры поддерживали при 22°C при освещении 16 ч/с (30 мкмоль м-2 с-1).
ВЫДЕЛЕНИЕ ГЕНОМНОЙ ДНК ИЗ ПРОТОПЛАСТОВ МЕЗОФИЛЛА
Трансфицированные протопласты переносили из 3-см чашку PETRI™ в 2-мл микроцентрифужную пробирку. Клетки осаждали центрифугированием при 70g и супернатант удаляли. Для максимального выделения трансфицированных протопластов чашку PETRI™ промывали три раза 1 мл промывочного буфера. Каждое промывание проводили вращательным движением в чашке PETRI™ в течение 1 минуты с последующим переносом жидкости в ту же 2-мл микроцентрифужную пробирку. В конце каждого промывания клетки осаждали путем центрифугирования при 70g и супернатант удаляли. Осажденные протопласты быстро замораживали в жидком азоте, а затем проводили лиофилизацию в течение 24 ч в LABCONCO FREEZONE 4.5® (Labconco, Kansas City, MO) при -40°C и под давлением 133×10-3 мбар. Лиофилизированные клетки подвергали экстракции ДНК с использованием набора DNEASY® PLANT DNA EXTRACTION MINI KIT (Qiagen) в соответствии с инструкциями изготовителя, за исключением того, что разрушение ткани не требовалось, и клетки протопластов добавляли непосредственно в лизирующий буфер.
ВЫДЕЛЕНИЕ ГЕНОМНОЙ ДНК ИЗ ТКАНИ КАЛЛЮСА
Индивидуаульные каллюсы быстро замораживали в жидком азоте, после чего проводили лиофилизацию в течение 24 ч в LABCONCO FREEZONE 4.5® (Labconco, Kansas City, MO) при -40°C и под давлением 133×10-3 мбар. Лиофилизирвоанные каллюсы подвергали экстракции ДНК с использованием набора DNEASY® PLANT DNA EXTRACTION MAXI kit (Qiagen, Hilden, Германия) в соответствии с инструкциями изготовителя.
ОБНАРУЖЕНИЕ ВСТРАИВАНИЯ ГЕНА В FAD2A ПОСРЕДСТВОМ НАПРАВЛЯЕМОЙ ГОМОЛОГИЕЙ РЕПАРАЦИИ В ПРОЛИФЕРИРОВАВШЕМ КАЛЛЮСЕ
Геномную ДНК экстрагировали из объединенных фракций протопластов (один миллион протопластов на объединенную фракцию), в которую за двадцать четыре часа до этого была доставлена донорная ДНК, кодирующая функциональную репортерную кассету HGH (pDAS000129), ДНК ZFN (pDAB104010) или смесь донорной ДНК и ДНК ZFN. Количества ДНК, доставляемой для трансформации, описаны выше. Продукты ПЦР клонировали в плазмидные векторы. Редактирование генома происходит независимо в каждой клетке, обеспечивая множество различных событий встраивания, путем клонирования плазмидного вектора редактирование генома в каждом случае можно точно отсеквенировать. Несколько клонов секвенировали на автоматизированной платформе для капиллярного электрофореза ABI3730XL®. Анализ последовательностей генов проводили с использованием SEQUENCHER SOFTWARE V5.0™ (GeneCodes, Ann Arbor, MI).
Доказательства встраивания гена в локус FAD2A посредством направляемой гомологией репарации предоставляли путем амплификации как кассеты 5' и 3' FAD2A, так и областей соединения, из геномной ДНК, экстрагированной из протопластов, с использованием праймеров. описанных в таблице 13. Не наблюдали амплификации в случае протопластов, в которые доставляли плазмиду ZFN или донорную плазмиду отдельно. Все последовательности области соединения указывали на встраивание кассеты hgh в локус Fad2A посредством опосредуемого HDR каскада репарации. Наблюдали делеции различной длины из любого или из обоих из генома и кассеты, а также вставку последовательностей, происходящих из основных цепей векторов (либо из донора, либо из ZFN), встраиваемых между геномом и кассетой (фиг. 15).

ПРИМЕР 9: НАПРАВЛЕННОЕ ВСТРАИВАНИЕ И РАЗРУШЕНИЕ ДЕСАТУРАЗЫ ОМЕГА-3 ЖИРНЫХ КИСЛОТ (FAD2) Brassica napus ПОСРЕДСТВОМ НЕГОМОЛОГИЧНОГО СОЕДИНЕНИЯ КОНЦОВ
ОТБОР СВЯЗЫВАЮЩИХ ДОМЕНОВ С ЦИНКОВЫМИ ПАЛЬЦАМИ, СПЕЦИФИЧНЫХ К FAD2A
Транскрибируемые области гомологичных генов FAD2 идентифицировали и охарактеризовывали нуклеазы с цинковыми пальцами, которые были сконструированы для связывания и расщепления этих участков для опосредуемого NHEJ нацеливания донорной последовательности. Белки с цинковыми пальцами (ZFP), направленные против последовательностей ДНК из гомологов последовательностей FAD2, конструировали и исследовали, как описано выше. Из ZFN, демонстрирующих активность в отношении мишени, был отобран один белок с цинковыми пальцами, который расщепляет мишень FAD2 с высокой эффективностью: ZFP 24828-2A-24829 распознает SEQ ID NO: 35 5'- agGCCCAGtAGAGAGGCCaggcgaagta -3' и SEQ ID NO: 36 5' -ccAGGGCTGCGTCCTAACCGgcgtctgg -3'. Было показано, что этот ZFN специфически связывается и расщепляет геномный локус FAD2A. Плазмидную конструкцию pDAB104010, которая описана выше, конструировали и использовали в экспериментах по трансформации.
МОДЕЛИРОВАНИЕ И КОНСТРУИРОВАНИЕ ЭКСПРЕССИРУЮЩИХ ВЕКТОРОВ, КОДИРУЮЩИХ НУКЛЕАЗЫ С ЦИНКОВЫМИ ПАЛЬЦАМИ, И ДОНОРНЫХ ВЕКТОРОВ, СПЕЦИФИЧНЫХ К FAD2A
Для встраивания донорной последовательности посредством NHEJ конструировали единый вектор. Этот вектор кодировал экспрессирующую кассету с репортерным геном dsRED. Экспрессирующая кассета с репортерным геном dsRED включала промотор убиквитина 10 Arabidopsis thalinana (Callis, et al., 1990, J. Biol. Chem., 265:12486-12493), за которым следовал ген dsRED (Dietrich et al. (2002) Biotechniques 2(2):286-293). Ген dsRED был кодон-оптимизированным для экспрессии в двудольных растениях и фланкировался 3'UTR, содержащей терминатор транскрипции и участок полиаденилирования открытой рамки считывания 23 (ORF23) A. tumefaciens pTi15955 (Barker et al., Plant Molecular Biology 1983, 2(6), 335-50). Кассета с селективным маркером включала промотор CsVMV, слитый с трансгеном pat. Трансген pat терминировался открытой рамкой считывания 1 (ORF1) A. tumefaciens pTi15955 (Barker et al., Plant Molecular Biology 1983, 2(6), 335-50). Кассету устойчивости с dsRED клонировали в определенные участки ферментов рестрикции каждого вектора с получением "донорного" вектора: pDAS000097 (SEQ ID NO: 88, фиг. 16). Донорный вектор pDAS00097 сконструирован для доставки в качестве линейной ДНК или кольцевой ДНК в клетку растения, и он встраивается в локус FAD2A при расщеплении геномного локуса FAD2A посредством ZFN pDAB104010. Опосредуемое линейной ДНК встраивание является результатом встраивания линеаризованной плазмиды pDAS000097 в клетку растения во время трансформации. Плазмиду можно линеаризовать путем расщепления в уникальном участке фермента рестрикции. Опосредуемое кольцевой ДНК встраивание является результатом встраивания замкнутой плазмиды pDAS000097 в растения в ходе трансформации. pDAS000097 модифицирован так, чтобы он содержал участок связывания с цинковыми пальцами, который может расщепляться нуклеазой с цинковыми пальцами ZFP 24828-2A-24829. Кольцевая плазмида pDAS000097 расщепляется в клетке растения кодируемой pDAB104010 нуклеазой с цинковыми пальцами, и кассета с геном dsRED встраивается в геномный локус FAD2A.
Колонии собранных плазмид первоначально подвергали скринингу путем расщепления эндонуклеазой рестрикции ДНК, очищенной из E.coli, культивированных в течение ночи. Эндонуклеазы рестрикции получали от NEW ENGLAND BIOLABS™ (NEB, Ipswich, MA) и PROMEGA™ (Promega Corporation, WI). Получение плазмид проводили с использованием набора QIAPREP SPIN MINIPREP KIT™ (Qiagen, Hilden, Germany) или системы PURE YIELD PLASMID MAXIPREP SYSTEM™ (Promega Corporation, WI) в соответствии с инструкциями поставщиков. После подтверждения полученных фрагментов рестрикции с помощью агарозного гель-электрофореза, плазмидную ДНК выбранных клонов секвенировали с использованием секвенирования методом Сэнгера ABI и протокола циклического секвенирования BIG DYE TERMINATOR V3.1™ (Applied Biosystems, Life Technologies). Данные о последовательности собирали и анализировали с использованием программного обеспечения SEQUENCHER™ (Gene Codes, Ann Arbor, MI).
ТРАНСФОРМАЦИЯ BRASSICA NAPUS
Происходящие из мезофилла протопласты выделяют и получают из растений Brassica napus (DH10275), как описано выше. Протопласты трансформируют очищенной плазмидной ДНК. Аликвоты донорной ДНК и плазмидной ДНК ZFN приготавливают в трех молярных соотношениях: 1:1 (30 мкг каждой плазмиды), 5:1 (донорная плазмида к плазмиде ZFN всего до 30 мкг плазмидной ДНК) и 10:1 (донорная плазмида к плазмиде ZFN всего до 30 мкг плазмидной ДНК). Кроме того, приготавливают аликвоты только донора или только ZFN (30 мкг) в качестве контролей. Количества ДНК, доставленной в протопласты B. napus опосредуемой PEG4000 трансформацией, обобщенно представлены в таблице 14. Трансформированные клетки протопластов культивируют, как описано ранее, где среда для селекции представляет собой среду для селекции с глюфосинатом, и предполагаемые трансформанты оценивают с помощью анализа qPCR в отношении вставок трансгенов.
ОБНАРУЖЕНИЕ ВСТРАИВАНИЯ ГЕНА В FAD2A В ПРОТОПЛАСТАХ ПОСРЕДСТВОМ НЕГОМОЛОГИЧНОГО СОЕДИНЕНИЯ КОНЦОВ
Геномную ДНК экстрагируют из объединенных фракций протопластов (один миллион протопластов на объединенную фракцию), в которую за двадцать четыре часа до этого была доставлена донорная ДНК, кодирующая функциональную репортерную кассету dsRFP (pDAS000097), ДНК ZFN (pDAB104010) или смесь донорной ДНК и ДНК ZFN. Количества ДНК, доставляемой для трансформации, описаны выше. Продукты ПЦР клонируют в плазмидные векторы. Редактирование генома происходит независимо в каждой клетке, обеспечивая множество различных событий встраивания, путем клонирования плазмидного вектора редактирование генома в каждом случае можно точно отсеквенировать. Несколько клонов секвенируют на автоматизированной платформе для капиллярного электрофореза ABI3730XL®. Анализ последовательностей генов проводят с использованием SEQUENCHER SOFTWARE V5.0™ (GeneCodes, Ann Arbor, MI).
Доказательства встраивания гена в локус FAD2A посредством редактирования или сплайсинга предоставляют путем амплификации областей соединения как кассеты 5', так и 3' FAD2A, из геномной ДНК, экстрагированной из протопластов. Не наблюдают амплификации в случае протопластов, в которые доставляли плазмиду ZFN или донорную плазмиду отдельно. Все последовательности области соединения указывают на встраивание кассеты dsRED в локус Fad2A посредством опосредуемого NHEJ каскада репарации. Наблюдают делеции различной длины из любого или из обоих из генома и кассеты, а также вставку последовательностей, происходящих из основных цепей векторов (либо из донора, либо из ZFN), встраиваемых между геномом и кассетой.
ОБНАРУЖЕНИЕ ВСТАВКИ ГЕНА В FAD2A ПОСРЕДСТВОМ НЕГОМОЛОГИЧНОГО СОЕДИНЕНИЯ КОНЦОВ В ТКАНИ КАЛЛЮСА, РЕГЕНЕРИРОВАВШЕЙ ИЗ ПРОТОПЛАСТОВ
Дальнейшие доказательства сплайсинга и редактирования локуса FAD2A получали из ткани каллюса, регенерировавшей из протопластов после селекции, в которые доставляли донорную ДНК, кодирующую кассету dsRED (pDAS000097), только ДНК ZFN (pDAB104010) или донорную ДНК и ДНК ZFN. ДНК экстрагируют приблизительно из 80 каллюсов для каждого соотношения.
Встраивание кассеты dsRED в геном B. napus подтверждают с помощью TAQMAN™ qPCR с использованием праймеров и зондов, специфичных к донорной вставке и геномным фланкирующим последовательностям. Относительное количественное определение проводят способом 2-∆∆Ct(Livak and Schmittgen, 2001), который обеспечивает оценку количества копий кассеты dsRED, встроенных в геном. Доказательства опосредуемого NHEJ сплайсинга и редактирования FAD2A получают путем проведения анализов ПЦР с одним праймером, специфичным к FAD2A, и вторым праймером, специфичным либо к промотору, либо к терминатору кассеты dsRED. Продукты ПЦР очищают из геля с использованием набора QIAQUICK MFNIELUTE PCR PURIFICATION KIT™ (Qiagen) и секвенируют с использованием способа прямого секвенирования Сэнгера. Продукты секвенирования очищают с помощью этанола, ацетата натрия и EDTA в соответствии с протоколом BIGDYE® v3.1 (Applied Biosystems) и секвенируют и анализируют, как описано выше.
Определяют количества каллюсов, содержащих донорную кассету, в каждом эксперименте. Доказательства встраивания донорного гена в локус FAD2A путем редактирования и/или сплайсинга получают путем амплификации ПЦР областей, охватывающих участки разрезания ZFN и как 5', так и 3' области соединения кассет FAD2A-dsRED. Амплификация способом ПЦР геномной ДНК, выделенной из ткани каллюса, восстановившейся из контрольных протопластов, которые были трансформированы только плазмидой dsRED (pDAS000097) или только плазмидой ZFN (pDAB104010), не приводят к продуцированию продуктов амплификации способом ПЦР.
Ампликоны ПЦР, продуцированные в результате амплификации 5' и 3' областей соединений кассет FAD2A-dsRED, очищают из агарозного геля и секвенируют для подтверждения специфичности встраивания в геномный локус FAD2A. Результаты анализа с помощью секвенирования продуктов ПЦР указывают на то, что каждый отдельный каллюс, образованный из индивидуально трансформированного протопласта, обеспечивает только один продукт амплификации способом ПЦР и не содержит клеток смешанных генотипов.
Обнаружение встраивания гена в FAD2A путем негомологичного соединения концов в растениях
ДНК экстрагируют из растений, которые регенерировали из протопластов, и переносят в среду для посева. Согласно оценке, большинство восстановившихся растений содержат только 1-2 копии кассеты dsRED, кодируемой донорной ДНК. Растения анализируют с помощью того же набора анализов, которые описаны для ткани каллюса, а также с помощью анализов для определения того, встроилась ли кассета в локус FAD2A.
Частоту сплайсинга в мишени, где кассету dsRED встраивают в локус FAD2A, определяют с использованием анализов ПЦР, описанных выше. Полученные полосы ампликонов секвенируют для определения фланкирующих последовательностей. Кроме того, растения подвергают скринингу в отношении встраиваний вне мишени для определения частоты встраивания dsRED в участках, отличных от FAD2A.
ПРИМЕР 10: НАПРАВЛЕННОЕ ВСТРАИВАНИЕ В ЛОКУС ДЕСАТУРАЗЫ ОМЕГА-3 ЖИРНЫХ КИСЛОТ (FAD2) Brassica napus АГРОНОМИЧЕСКИ ВАЖНОГО ГЕНА
Конструкции, содержащие трансген DGT-28 (международная патентная заявка № WO/2013/116700, включенная в настоящее описание в качестве ссылки), которые сообщают устойчивость к гербициду глифосату, моделируют и конструируют для встраивания в геномные локусы FAD2A Brassica napus. Иллюстративные донорные конструкции включают pDAS000389 (фиг. 17, SEQ ID NO: 89) для встраивания по типу NHEJ в локус FAD2A, pDAS000391 (фиг. 18, SEQ ID NO: 90) для встраивания по типу NHEJ в локус FAD2A, pDAS000392 (фиг. 19, SEQ FD NO:91) для встраивания по типу NHEJ в локус FAD2A, pDAS000393 (фиг. 20, SEQ ID NO: 92) для встраивания по типу NHEJ в локус FAD2A, pDAS000394 (фиг. 21, SEQ ID NO: 93) для встраивания по типу HDR в участок ΕΤIΡ локуса FAD2A, pDAS000395 (фиг. 22, SEQ ID NO: 208) для встраивания по типу HDR в участок ΕΤIΡ локуса FAD2A, pDAS000396 (фиг. 23, SEQ ID NO: 209) для встраивания по типу HDR в локус FAD2A, и pDAS000397 (фиг. 24, SEQ ID NO: 210) для встраивания по типу HDR в локус FAD2A. Эти конструкции и ассоциированные с ними конструкции нуклеаз с цинковыми пальцами (например, pDAB104010) трансформируют в клетки Brassica napus, как описано ранее. Трансформанты идентифицируют и подтверждают с помощью молекулярных анализов для подтверждения, как описано выше. Встроенные в хромосому FAD2A конструкции, содержащие встроенный трансген dgt-28, выделяют. Встраивание трансгесна dgt-28 в локус FAD2A иллюстрируется опосредуемым NHEJ встраиванием и опосредуемым HDR встраиванием. Встраивание в локус FAD2A может быть направлено в эндогенную последовательность FAD2A или в ранее описанный ETIP (pDAS000130), который стабильно встроен в локус FAD2A. Встраивание в локус FAD2A путем опосредуемого NHEJ механизма можно проводить с использованием линеаризованных конструкций донорной ДНК или кольцевых конструкций донорной ДНК. События трансформации DGT-28 в Brassica napus осуществляют и исследуют в отношении устойчивой экспрессии DGT-28 и последующей устойчивости к гербициду глифосату.
Хотя определенные иллюстративные варианты осуществления описаны в настоящем описании, специалистам в данной области будет известно и понятно, что можно проводить множество дополнений, удалений или модификаций иллюстративных вариантов осуществления без отклонения от объема нижеследующей формулы изобретения. Кроме того, признаки одного варианта осуществления можно комбинировать с признаками другого варианта осуществления.
Реферат
Изобретение относится к области биохимии, в частности к способу интегрирования интересующей последовательности нуклеиновой кислоты в ген FAD2 клетки, включающему сайт-специфическое расщепление гена FAD2 клетки растения с использованием нуклеазы с цинковыми пальцами. Также раскрыта сайт-специфическая нуклеаза с цинковыми пальцами для применения в модифицировании гена FAD2. Изобретение позволяет эффективно интегрировать интересующую последовательность нуклеиновой кислоты в клетку растения. 2 н. и 5 з.п. ф-лы, 24 ил., 14 табл., 10 пр.

Комментарии