Прямой захват, амплификация и секвенирование днк-мишени с использованием иммобилизированных праймеров - RU2565550C2
Код документа: RU2565550C2
Чертежи



































Описание
Перекрестная ссылка
Данная заявка претендует на приоритет предварительной заявки на патент США номер 61/386390, поданной 24 сентября 2010 года, и номер 61/485062, поданной 11 мая 2011 года, каковые заявки полностью включены в настоящий документ во всей полноте
Права правительства
Данное изобретение выполнено при поддержке Правительства на основании контракта HG000205, полученного от Национального Института Здоровья. Правительство имеет определенные права на данное изобретение.
Уровень техники
Во многих способах секвенирования, в частности, в способах ре-секвенирования (т.е., способах, в которых локус повторно секвенируется), мишень вначале захватывается, а потом секвенируется. Было разработано несколько методологий направленного захвата и внедрено в высокоэффективные системы секвенирования. В частности, для захвата ДНК-мишени могут применяться методы, основанные на гибридизации, использующие шарики или микроматрицы, и технологии, базирующиеся на растворах, использующих молекулярные инверсионные зонды или геномные циркуляризационные олигонуклеотиды. Захваченная ДНК затем готовится для секвенирования. Для приготовления обогащенного образца ДНК часто используются сложные молекулярно-биологические протоколы и в определенных случаях получение библиотеки секвенирования включает много ферментных реакций, этапов очистки и отбора по размеру при помощи гель-электрофореза. Процесс подготовки образца для секвенирования захваченной ДНК-мишени может быть трудоемким и последующие манипуляции с образцом могут вызвать отклонения в составе ДНК и повысить уровень ошибки секвенирования.
Сущность изобретения
Здесь предоставляются способы для захвата и амплификации фрагмента нуклеиновой кислоты, например, фрагмента генома или кДНК, приготовленной из РНК. Также предоставляются наборы для осуществления способа на практике. В определенных вариантах воплощения способ включает: а) получение субстрата, содержащего первую популяцию связанных с поверхностью олигонуклеотидов, и вторую популяцию связанных с поверхностью олигонуклеотидов, где члены первой и второй популяций связанных с поверхностью олигонуклеотидов пространственно не направлены на субстрат; б) гибридизацию первого члена первой популяции связанных с поверхностью олигонуклеотидов с олигонуклеотидом отбора, включающим участок, который гибридизируется с первым членом, и участок, который содержит геномную последовательность, в) удлинение первого члена первой популяции связанных с поверхностью олигонуклеотидов для получения связанного с подложкой праймера отбора, который содержит последовательность, комплементарную геномной последовательности; г) гибридизацию связанного с подложкой праймера отбора с фрагментом нуклеиновой кислоты (например, с геномным фрагментом или кДНК), содержащим геномную последовательность; д) удлинение связанного с подложкой праймера отбора для получения продукта удлинения, который содержит последовательность, которая фланкирует геномную последовательность, например, в геноме; е) амплификацию продукта удлинения на субстрате, например, путем мостиковой ПЦР с использованием неудлиненных членов первой и второй популяций связанных с поверхностью олигонуклеотидов для получения продукта ПЦР.
В определенных вариантах воплощения способ включает: а) получение субстрата, содержащего первую популяцию связанных с поверхностью олигонуклеотидов и вторую популяцию связанных с поверхностью олигонуклеотидов, где первая и вторая популяции связанных с поверхностью олигонуклеотидов пространственно не направлены на субстрат; б) гибридизацию первого члена первой популяции связанных с поверхностью олигонуклеотидов с олигонуклеотидом отбора, содержащим участок, который гибридизируется с первым членом, и участок, который содержит геномную последовательность; в) удлинение первого члена первой популяции связанных с поверхностью олигонуклеотидов для получения связанного с подложкой праймера отбора, который содержит последовательность, которая комплементарна геномной последовательности; г) гибридизацию связанного с подложкой праймера отбора с фрагментом нуклеиновой кислоты, содержащим геномную последовательность; д) удлинение связанного с подложкой праймера отбора для получения продукта удлинения, который содержит последовательность, которая фланкирует геномную последовательность, например, в геноме; и е) амплификацию продукта удлинения, например, используя мостиковую ПЦР на субстрате для получения продукта ПЦР.
В зависимости от того, как выполняется способ, адаптер может быть лигирован либо к геномному фрагменту перед гибридизацией, либо к продукту удлинения после того, как связанный с подложкой праймер отбора будет удлинен. Дистальный адаптер может гибридизироваться со связанным с поверхностью олигонуклеотидом (который сам по себе может быть продуктом удлинения, получаемым путем матричного удлинения второй популяции связанных с поверхностью олигонуклеотидов), тем самым обеспечивая проведение мостиковой ПЦР. Праймер отбора может также содержать связывающий сайт праймера секвенирования, который может быть применен для секвенирования продукта ПЦР.
Описываемый выше способ находит применение в способах ре-секвенирования, в которых доступна последовательность референсного локуса, и тот же локус следует ре-секвенировать во множестве тестовых образцов. В этом применении создается олигонуклеотид отбора для гибридизации с олигонуклеотидом на субстрате, и участок, который фланкирует локус, который следует ресеквенировать. Этот локус захватывается на субстрате и затем амплифицируется перед секвенированием. Например, единичный локус или множественные различные локусы (например, до 10, 50, 100, 200 или 1000 или более локусов) могут быть захвачены из образца, который получен от одного индивидуума или от множества индивидуумов (например, до 10, 50, 100, 200 или 1000 или более индивидуумов).
В определенных вариантах воплощения способ включает: а) получение субстрата, содержащего первую популяцию связанных с поверхностью олигонуклеотидов и вторую популяцию связанных с поверхностью олигонуклеотидов, где первая и вторая популяции связанных с поверхностью олигонуклеотидов случайным образом рассеяны на субстрате и пространственно не направлены; б) гибридизацию первого члена первой популяции связанных с поверхностью олигонуклеотидов с олигонуклеотидом отбора, содержащим участок, который гибридизируется с первым членом, и участок, который содержит геномную последовательность; в) удлинение первого члена первой популяции связанных с поверхностью олигонуклеотидов для получения связанного с подложкой праймера отбора, который содержит последовательность, комплементарную геномной последовательности; г) гибридизацию связанного с подложкой праймера отбора с пришитым к адаптеру фрагментом (например, с пришитым к адаптеру геномным фрагментом), содержащим геномную последовательность; д) удлинение связанного с подложкой праймера отбора для получения продукта, содержащего последовательность, которая фланкирует геномную последовательность (например, в геноме), и последовательность адаптера, лигированного с адаптером геномного фрагмента; и е) амплификацию продукта с использованием мостиковой ПЦР для получения продукта ПЦР.
В альтернативных вариантах воплощения способ может включать: а) получение субстрата, содержащего первую популяцию связанных с поверхностью олигонуклеотидов и вторую популяцию связанных с поверхностью олигонуклеотидов, где первая и вторая популяции связанных с поверхностью олигонуклеотидов случайным образом рассеяны на субстрате и пространственно не направлены; б) гибридизацию первого члена первой популяции связанных с поверхностью олигонуклеотидов с олигонуклеотидом отбора, содержащим участок, который гибридизируется с первым членом, и участок, который содержит геномную последовательность; в) удлинение первого члена первой популяции связанных с поверхностью олигонуклеотидов для получения связанного с подложкой праймера отбора, который содержит последовательность, комплементарную геномной последовательности; г) удлинение связанного с подложкой праймера отбора для получения продукта, который содержит последовательность, которая фланкирует геномную последовательность; д) лигирование двуцепочечного адаптера с продуктом для получения продукта, модифицированного адаптером; и е) амплификацию продукта, модифицированного адаптером, с использованием мостиковой ПЦР для получения продукта ПЦР.
В конкретных случаях способ далее может включать: i. лигирование геномных фрагментов с адаптером, который содержит сайт для праймера секвенирования и нуклеотидную последовательность, такую же, как и вторые связанные с поверхностью олигонуклеотиды, ii. гибридизацию дотированных с адаптером геномных фрагментов с первым членом первой популяции связанных с поверхностью олигонуклеотидов, ii. удлинение первого члена первой популяции связанных с поверхностью олигонуклеотидов, с которыми гибридизируется лигированный с адаптером фрагмент; и iv. гибридизацию содержащего адаптер конца продукта удлинения со вторым связанным с подложкой полинуклеотидом, тем самым создавая мостик и способствуя мостиковой ПЦР.
Краткое описание фигур
Определенные аспекты следующего детального описания лучше всего воспринимаются, если их читать вместе с сопутствующими иллюстрациями. Подчеркивается, что исходя из общепринятой практики, различные характеристики иллюстрации не предназначены для масштабирования. С другой стороны, направления различных характеристик произвольно расширены или уменьшены для ясности. Иллюстрации включают следующие фигуры:
Фиг.1. Обзор одного из вариантов воплощения рассматриваемого способа, называемого «OS-Seq» (олигонуклеотид-селективного секвенирования). (a) OS-Seq является способом направленного ре-секвенирования, который органично интегрирован в платформу Illumina NGS (Next Generation Sequencing - секвенирование нового поколения). Для этого способа нужны мишень-специфические олигонуклеотиды, библиотека секвенирования и набор Illumina для создания кластеров. Захват мишеней, процессинг и секвенирование выполняются на системе NGS. Данные, получаемые от каждого праймера-зонда, являются намеченнымии специфическими для цепи. Здесь показан медианный профиль покрытия для OS-Seq-366. (b) Процессинг OS-Seq включает три этапа: гибридизацию, удлинение, опосредованное ДНК-полимеразой, и денатурацию ДНК. Этап 1; Используются мишень-специфические олигонуклеотиды для преобразования праймеров проточной ячейки в праймеры-зонды. В системе секвенирования Illumina два типа праймеров (названные С и D) иммобилизированы на проточной ячейке со спаренными концами. В OS-Seq подгруппа D праймеров модифицируется до праймеров-зондов с использованием комплексной библиотеки олигонуклеотидов. Олигонуклеотиды имеют последовательности, которые гибридизируются с праймерами проточной ячейки D типа. Гибридизированные олигонуклеотиды затем используются в качестве матрицы для ДНК полимеразы, и D праймеры удлиняются. После денатурации мишень-специфические праймеры-зонды случайным образом иммобилизируются на проточной ячейке. Этап 2: Геномные мишени в одноадаптерной библиотеке захватываются с применением праймеров-зондов. Подготовка образца для секвенирования для Illumina включает добавление специфических ДНК адаптеров к фрагментам геномной ДНК. Эти адаптеры содержат в себе сайты для секвенирующих праймеров и для иммобилизированных праймеров проточной ячейки. В OS-Seq мы используем модифицированный адаптер для приготовления одноадапторных библиотек из геномной ДНК. Мишени в одноадаптерной библиотеке захватываются во время гибридизации в условиях высокого нагрева своими комплементарными праймерами-зондами. Захваченные фрагменты одноадаптерной библиотеки используются в качестве матрицы для ДНК полимеразы, и праймеры-зонды удлиняются. Денатурация высвобождает ДНК матрицы из иммобилизированных мишеней. Этап 3: Иммобилизированные мишени становятся совместимыми с секвенированием Illumina. При секвенировании Illumina требуется твердофазная амплификация фрагментов иммобилизированной библиотеки секвенирования с использованием праймеров С и D. В OS-Seq во время гибридизации в условиях низкого нагрева хвосты одиночных адаптеров иммобилизованных мишеней гибридизируются с праймерами С типа на поверхности проточной ячейки, что стабилизирует мостиковую структуру. 3' концы иммобилизированных мишеней и С праймеры удлиняются с использованием ДНК полимеразы. После денатурации формируются два комплементарных иммобилизированных фрагмента библиотеки секвенирования, содержащие полные примирующие сайты С и D, и они совместимы с твердофазной амплификацией. После этих трех этапов OS-Seq иммобилизированные мишени структурно идентичны по отношению к стандартной библиотеке со спаренными концами Illumina, и они амплифицируются и далее обрабатываются с использованием стандартных наборов и протоколов Illumina. Принципы данного способа могут быть применены и с другими секвенирующими платформами, (с) Показан профиль покрытия вдоль гена KRAS из анализа OS-Seq-366. На оси x представлены положения оснований по отношению к началу экзона 1 и указаны экзоны KRAS. (d) Оценка однородности выходов у праймер-зонд нуклеотидов, синтезированных на колонке и на матрице. Однородность захвата сравнивалась между олигонуклеотидами, синтезированными на колонке (синим цветом, n=366), и олигонуклеотидами, синтезированными на матрице (красным цветом, n=11742). На оси x олигонуклеотиды рассортированы по выходу захваченных последовательностей, на оси y - нормализованный выход праймера-зонда. Для расчета нормализованного выхода каждый выход олигонуклеотида делился на средний выход от всех олигонуклеотидов.
Фиг.2: Подготовка библиотеки секвенирования для OS-Seq. Общая схема фрагментации геномной ДНК, восстановления концов, наращивание А-«хвоста» (добавление липких 3'-А концов ДНК). В приготовлении библиотек OS-Seq использовалось лигирование адаптера и ПЦР.
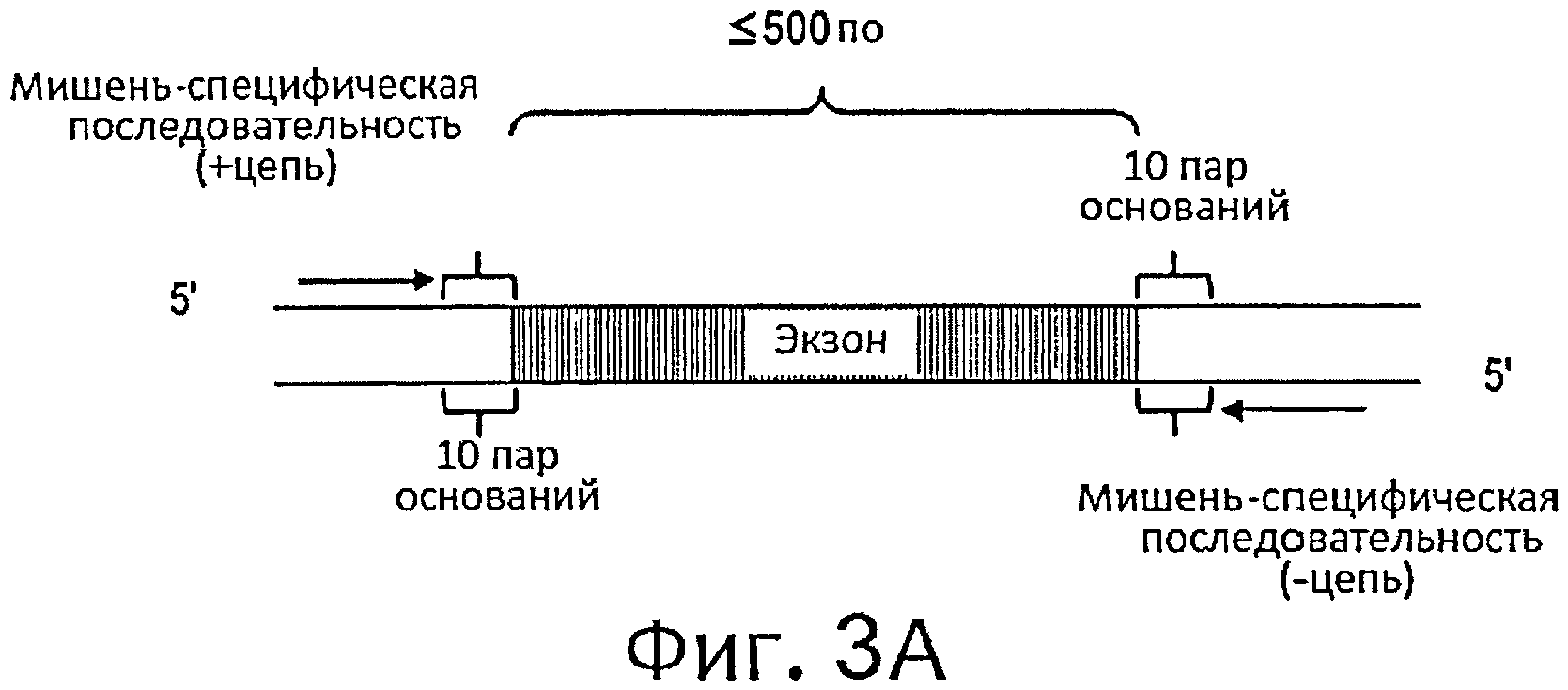
Фиг.3. Разработка стратегий для OS-Seq. (а) Праймеры-зонды были помещены на расстоянии 10 оснований от экзона или (b) покрывали каждые 500 оснований внутри больших экзонов.
Фиг.4. Генерирование OS-Seq олигонуклеотидов. Колоночный синтез дал в результате большое количество зрелых 101-мерных OS-Seq олигонуклеотидов, которые были готовы для применения в анализе. Микроматричный синтез был применен для наработки пулов с высоким содержанием олигонуклеотидов. Олигонуклеотиды - предшественники были амплифицированы с использованием праймеров, которые вводили дополнительные последовательности в олигонуклеотиды. Было применено вырезание урацила для отщепления сайта амплификации праймера от кодирующих цепей OS-seq олигонуклеотидов.
Фиг.5. Структуры олигонуклеотидных компонентов в OS-Seq. (а) Зрелые 101-мерные OS-Seq олигонуклеотиды содержали мишень-специфический сайт и последовательности, кодирующие секвенирующий праймер 2 и праймер проточной ячейки «D». (b) Олигонуклеотиды, полученные путем микроматричного синтеза, были амплифицированы с использованием праймеров, которые вводили Урацил на 5' конце OS-Seq олигонуклеотида и дополнительные активные сайты для секвенирования. (с) Адаптер для OS-Seq содержал выступающий конец - Т (липкий конец ДНК) для лигирования липкого конца ДНК с геномными фрагментами, содержащими А-«хвост». В дополнение, в адаптере двуцепочечной ДНК (дцДНК) присутствовала индексирующая последовательность наряду с праймером «С» проточной ячейки.
Фиг.6. Описания распределений размера вставки, встречающихся в данных OS-Seq. Фрагментирование геномной ДНК дает фрагменты между 200 и 2кб. Приготовление библиотеки секвенирования добавляет одинаковый адаптер к концам фрагментов. ПЦР амплификация искажает распределение размера фрагмента. Сайты-мишени случайным образом распределены среди фрагментов одноадаптерной библиотеки. Фрагменты библиотеки были иммобилизированы на проточной ячейке и расстояние между праймером-зондом и адаптером определяло размер вставки геномной ДНК. Для амплификации иммобилизированной ДНК-мишени применена мостиковая ПЦР (в общем, твердофазная ПЦР предпочтительно амплифицирует более короткие фрагменты). После кластерной амплификации и процессинга иммобилизированные фрагменты секвенируются с использованием двух сайтов. Считывание 1 начинается с геномной ДНК и Считывание 2 начинается с синтетических праймеров-зондов. Считывание 1 используется для оценки последовательности геномной ДНК из данных OS-Seq.
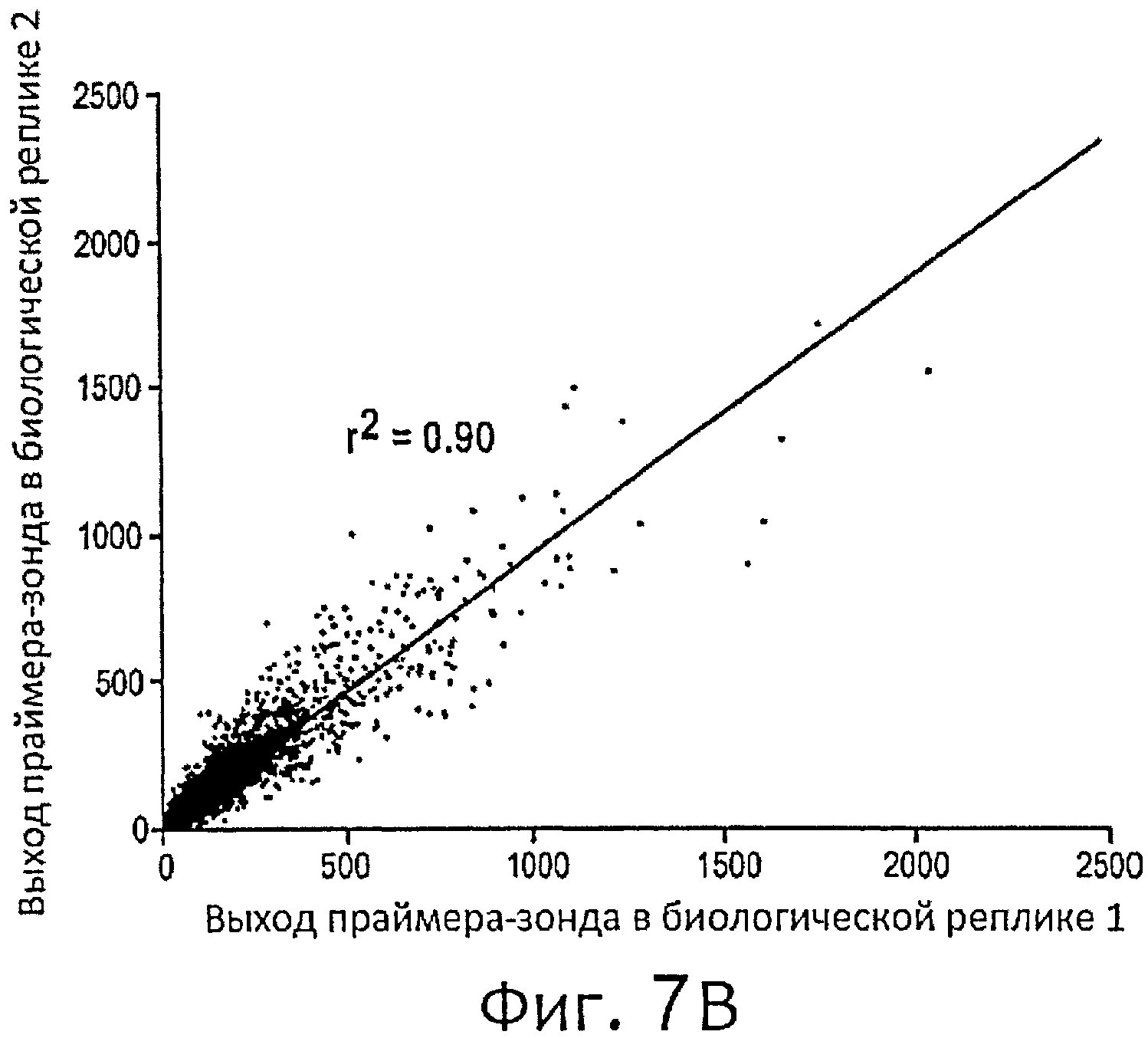
Фиг.7. Воспроизводимость OS-Seq. (а) Техническая воспроизводимость OS-Seq. Были проанализированы две идентичные библиотеки с использованием OS-Seq. Итоги секвенирования отдельных праймеров-зондов сравнивались среди технических реплик. (b) Биологическая воспроизводимость OS-Seq. Были приготовлены две различные библиотеки геномной ДНК с использованием индексированных адаптеров. Библиотеки были проанализировали в одном и том же эксперименте OS-Seq. На фигуре итоги специфического захвата праймера-зонда сравниваются среди двух независимых биологических реплик.
Фиг.8А-В. Влияние содержания ГЦ на выход таргетинга. Для анализа воздействия содержания ГЦ на эффективность праймеров-зондов мы определяли содержание ГЦ у каждой мишень-специфической последовательности праймера-зонда. Мы классифицировали праймеры-зонды, которые потерпели неудачу (захватили 0 мишеней). Пропорции потерпевших неудачу праймеров-зондов сравнивались между категориями, различными по процентному содержанию ГЦ. Ось Х представляет процентный состав рассортированных по ГЦ категорий, и ось У показывает пропорцию потерпевших неудачу праймеров-зондов в каждой категории содержания ГЦ.
Фиг.9А-В. Сравнение хода работ по процессингу между OS-Seq и стохастическими способами создания библиотек.
Определения
Если только в этом документе не определено иным образом, все технические и научные термины, используемые в данном документе, имеют то же значение, что и обыкновенно понимается обычным специалистом в области техники, к которой относится данное изобретение. Хотя на практики или при тестировании настоящего изобретения могут использоваться любые способы и материалы, схожие или эквивалентные тем, которые описаны в данном документе, здесь приводятся предпочтительные способы и материалы.
Все патенты и публикации, включая все последовательности, раскрываемые в таких патентах и публикациях, упоминаемых здесь, явным образом включены в данный документ посредством ссылки.
Числовые диапазоны охватывают числа, определяющие диапазон. Если не указано иным образом, нуклеиновые кислоты пишутся слева направо в ориентации от 5' до 3'; аминокислотные последовательности пишутся слева направо, соответственно, в ориентации от амино до карбокси.
Предоставленные здесь заголовки не являются ограничениями различных аспектов или вариантов воплощения изобретения. Соответственно, термины, определенные непосредственно ниже, более полно определяются посредством ссылки на описание в целом.
Если не определено иным образом, все технические и научные термины, используемые в данном документе, имеют то же значение, что и обыкновенно понимается обычным специалистом в области техники, к которой относится данное изобретение. Singleton, et al., DICTIONARY OF MICROBIOLOGY AND MOLECULAR BIOLOGY, 2D ED., John Wiley and Sons, New York (1994), и Hale & Markham, THE HARPER COLLINS DICTIONARY OF BIOLOGY, Harper Perennial, N.Y. (1991) предоставляют специалисту общее значение многих терминов, используемых в данном документе. Тем не менее, некоторые термины определятся ниже для полной ясности и удобства ссылки.
Термин «образец» в данном контексте относится к материалу или смеси материалов, как правило, хотя не обязательно, в жидкой форме, содержащему один или более искомых анализируемых материалов Используемые здесь анализируемые образцы нуклеиновой кислоты могут быть комплексом, в этом они содержат множественные различные молекулы, которые содержат последовательности. Фрагментированная геномная ДНК или кДНК, полученная из мРНК млекопитающих (например, мыши или человека), являются типичными представителями комплексных образцов. Комплексные образцы могут иметь более чем 104, 105, 106 или 107 различных молекул нуклеиновой кислоты. Мишень ДНК может происходить из любого источника, такого, как геномная ДНК, кДНК (или РНК) или искусственных ДНК конструктов. Любой образец, содержащий нуклеиновую кислоту, например, геномную ДНК, приготовленную из клеток культуры тканей, образца ткани или фиксированных образцов ткани, заключенных в парафин (FRET), могут быть использованы здесь.
Термин «нуклеотид», как подразумевается, включает те функциональные группы, которые содержат не только известные пуриновые и пиримидиновые основания, но также и другие гетероциклические основания, которые подверглись модификации. Такие модификации включают метилированные пурины или пиримидины, ацилированные пурины или пиримидины, алкилированные рибозы или другие гетероциклы. В дополнение, термин «нуклеотид» включает те функциональные группы, которые содержат гаптен или флуоресцентные метки, и могут содержать не только обычные сахара рибозу и дезоксирибозу, но также и другие сахара. Модифицированные нуклеозиды или нуклеотиды также включают модификации сахарной функциональной группы, например, когда одна или более из гидроксильных групп заменены атомами галогенов или алифатическими группами, функционализированы в качестве эфиров, аминов или подобных.
Термин «нуклеиновая кислота» и «полинуклеотид» в данном документе используются взаимозаменяемо для описания полимера любой длины, например, большего чем, приблизительно, 2 основания, большего чем, приблизительно, 10 оснований, большего чем, приблизительно, 100 оснований, большего чем, приблизительно, 500 оснований, большего чем 1000 оснований, вплоть до, приблизительно, 10000 или более оснований, состоящего из нуклеотидов, например, дезоксирибонуклеотидов или рибонуклеотидов, и может быть получен ферментативным путем или синтетически (например, ПНА (пептид-нуклеиновая кислота), как описано в U.S. Patent No. 5948902 и в цитированных там ссылках), который может гибридизироваться со встречающимися в природе нуклеиновыми кислотами в соответствии с последовательностями аналогично тому, как это происходит с двумя встречающимися в природе нуклеиновыми кислотами, например, может участвовать во взаимодействиях образования пар оснований Уотсона-Крика. Встречающиеся в природе нуклеотиды включают гуанин, цитозин, аденин и тимин (соответственно, Г, Ц, А и Т).
Термин «образец нуклеиновой кислоты» в данном контексте обозначает образец, содержащий нуклеиновые кислоты.
Термин «полинуклеотид-мишень» в данном контексте относится к искомому изучаемому полинуклеотиду. В определенных вариантах воплощения полинуклеотид-мишень содержит одну или более искомых изучаемых последовательностей.
Термин «олигонуклеотид» в данном контексте обозначает одноцепочечный мультимер нуклеотида от 2 до 200 нуклеотидов, до 500 нуклеотидов в длину. Олигонуклеотиды могут быть синтетическими, или могут быть получены ферментативным путем и, в некоторых вариантах воплощения, составляют от 30 до 150 нуклеотидов в длину. Олигонуклеотиды могут содержать рибонуклеотидные мономеры (т.е., могут быть олигорибонуклеотидами) или дезоксирибонуклеотидные мономеры. Олигонуклеотид может быть в длину, например, от 10 до 20, от 11 до 30, от 31 до 40, от 41 до 50, 51-60, от 61 до 70, от 71 до 80, от 80 до 100, от 100 до 150 или от 150 до 200 нуклеотидов.
Термин «гибридизация» относится к процессу, при помощи которого цепь нуклеиновой кислоты соединяется с комплементарной цепью путем спаривания оснований, как это известно в данной области техники. Нуклеиновая кислота, как считается, является «селективно гибридизируемой» с последовательностями референсной нуклеиновой кислоты, если две нуклеиновые кислоты специфически гибридизируются одна с другой в условиях гибридизации и отмывания от умеренно жестких до жестких. Условия гибридизации от умеренно жестких до жестких известны (см., например, Ausubel, et al., Short Protocols in Molecular Biology, 3rd ed., Wiley & Sons 1995 и Sambrook et al., Molecular Cloning: A Laboratory Manual, Third Edition, 2001 Cold Spring Harbor, N.Y.). Один пример жеских условий гибридизации включает гибридизацию при, приблизительно, 42°C в 50% формамиде, 5Х SSC (раствор цитрата и хлорида натрия), 5Х растворе Денхардта, 0,5% SDS (додецил сульфат натрия) и 100 мкг/мл денатурированной ДНК-носителя с последующим двукратным отмыванием в 2Х SSC и 0,5% SDS при комнатной температуре и еще два раза в 0,1 Х SSC и 0,5% SDS при 42°С.
Термин «двойная спираль» или «соединенный в виде двойной спирали» в данном контексте описывает два комплементарных полинуклеотида, спаренных по основаниям, т.е., сгибридизированных вместе.
Термин «амплификация» в данном контексте относится к созданию одной или более копий нуклеиновой кислоты-мишени с использованием в качестве матрицы нуклеиновой кислоты-мишени.
Термины «установление», «измерение», «определение», «оценка», «испытание» и «анализирование» в данном документе используются взаимозаменяемо для отсылки к любой форме измерения и включают определение того, присутствует ли этот элемент или нет. Эти термины включают оба - количественные и/или качественные определения. Оценка может быть относительной или абсолютной. «Оценка наличия» включает определение присутствующего количества чего-либо, наряду с определением того, присутствует ли это или отсутствует.
Термин «применение» имеет свое традиционное значение и, как таковой, означает использование, например, ввод в действие, способа или композиции для достижения цели. Например, если для создания файла используется программа, программа выполняется для создания файла, файл обычно является продуктом программы. В другом примере, если используется компьютерный файл, к нему обычно получают доступ, его читают и информацию, хранящуюся в файле, используют для достижения цели. Сходным образом, если используется уникальный идентификатор, например, штрих-код, этот уникальный идентификатор обычно считывается для идентификации, например, объекта или файла, связанного с уникальным идентификатором.
В данном контексте, термин «Tm» относится к температуре плавления двойной спирали олигонуклеотидов, при которой половина двойных спиралей остается загибридизированными и половина двойных спиралей диссоциируют на одиночные цепи. Tm двойной спирали олигонуклеотиднов может быть определена экспериментально или предсказана путем использования следующей формулы Tm=81,5+16,6(log10[Na+])+0,41 (фракция Г+Ц) - (60/N), где N - длина цепи и [Na+] менее чем 1 М. См. Sambrook and Russell (2001; Molecular Cloning: A Laboratory Manual, 3rd ed., Cold Spring Harbor Press, Cold Spring Harbor N.Y., ch. 10). Существуют другие формулы для предсказания Tm двойных спиралей олигонуклеотидов, и одна формула может быть более или менее подходящей для данного условия или набора условий.
Термин «свободный в растворе» в данном контексте описывает молекулу, такую, как полинуклеотид, которая не связана с другой молекулой или не привязана к другой молекуле.
Термин «денатурация» в данном контексте относится к разделению двойной спирали нуклеиновой кислоты на две одиночные цепи.
Термин «геномная последовательность» в данном контексте относится к последовательности, которая имеет место в геноме. Поскольку РНК транскрибируются из генома, этот термин охватывает последовательность, существующую в ядерном геноме организма, наряду с последовательностями, которые присутствуют в кДНК копиях РНК (например, мРНК), транскрибируемых из такого генома.
Термин «геномный фрагмент» в данном контексте относится к участку генома, например, генома животного или растения, такого как геном человека, обезьяны, крысы, рыбы или насекомого, или растения. Геномный фрагмент может быть и может не быть дотированным с адаптером. Геномный фрагмент может быть адаптер-лигированным (в этом случае он имеет адаптер, дотированный с одним или с обоими концами фрагмента, с, по меньшей мере, 5' концом молекулы) или может не быть адаптер-лигированным.
В определенных случаях олигонуклеотид, используемый в описываемом в данном документе способе, может быть сконструирован с использованием референсного геномного участка, т.е., геномного участка с известной нуклеотидной последовательностью, например, хромосомного участка, последовательность которого депонирована, например, в базе данных Genbank Национального Центра Биотехнологической Информации или в других базах данных. Такой олигонуклеотид может быть применен при анализе, который использует образец, содержащий тестируемый геном, где тестируемый геном содержит связывающий сайт для олигонуклеотида.
Термин «дотирование» в данном контексте относится к катализируемому ферментами соединению концевогонуклеотида на 5' конце первой молекулы ДНК с концевым нуклеотидом на 3' конце второй молекулы ДНК.
Термин «адаптер» относится как к двуцепочечным, так и к одноцепочечным молекулам.
«Множество» содержит, по меньшей мере, 2 члена. В определенных случаях множество может иметь, по меньшей мере, 10, по меньшей мере, 100, по меньшей мере, 100, по меньшей мере, 10000, по меньшей мере, 100000, по меньшей мере, 106, по меньшей мере, 107, по меньшей мере, 108 или, по меньшей мере, 109 или более членов.
Если две нуклеиновые кислоты «комплементарны», каждое основание одной из нуклеиновых кислот образует пару с соответствующими нуклеотидами в другой нуклеиновой кислоте. Термин «комплементарный» и «полностью комплементарный» в данном документе используются в качестве синонимов.
«Сайт связывания праймера» относится к сайту, с которым праймер гибридизируется в олигонуклеотиде или в его комплементарной цепи.
Термин «разделение» в данном контексте относится как к физическому разделению двух элементов (например, по размеру, аффинности, т.п.), так и деградации одного элемента, при этом другой элемент остается интактным.
Термин «секвенирование» в данном контексте относится к способу, при помощи которого определяют тип, по меньшей мере, 10 последовательных нуклеотидов (например, тип, по меньшей мере, 20, по меньшей мере, 50, по меньшей мере, 100 или, по меньшей мере, 200 или более последовательных нуклеотидов) полинуклеотида.
Термин «пространственно не направленный» в контексте субстрата, содержащего связанную с поверхностью популяцию олигонуклеотидов, которые пространственно не направленны, относится к субстрату, содержащему поверхность, содержащую различные олигонуклеотидные молекулы, которые не расположены в определенном порядке или положении по отношению одна к другой, т.е. находятся в случайных положениях или случайным образом рассеяны по отношению одна к другой. Не требуется, чтобы такой субстрат был плоским, и в некоторых случаях он может быть в форме шариков. Субстраты, содержащие пространственно или оптически направленные популяции единичного олигонуклеотида (например, микроматрицы или кодированные шарики, т.п.) исключены из этого определения. Субстрат, содержащий первую популяцию связанных с поверхностью олигонуклеотидов и вторую популяцию связанных с поверхностью олигонуклеотидов, где первая и вторая популяции связанных с поверхностью олигонуклеотидов пространственно не направлены, относится к субстрату, содержащему, по меньшей мере, две популяции различных олигонуклеотидов, которые случайным образом распределены по субстрату. Субстрат может быть, например, плоским или в форме шариков.
Термин «адаптер-лигированный» в данном контексте относится к нуклеиновой кислоте, которая была лигирована с адаптером. Адаптер может быть лигирован к 5' концу или к 3' концу молекулы нуклеиновой кислоты.
Термин «удлинение» в данном контексте относится к удлинению праймера путем добавления нуклеотидов с использованием полимеразы. Если праймер, который отжигается к нуклеиновой кислоте, удлиняется, нуклеиновая кислота действует в качестве матрицы для реакции удлинения.
Термин «мостиковая ПЦР» относится к твердофазной полимеразной цепной реакции, при которой праймеры, которые удлиняются в реакции, присоединяются к субстрату своими 5' концами. Во время амплификации ампликоны формируют мостик между присоединенными праймерами. Мостиковая ПЦР (которая также может называться «кластерной ПЦР») используется в платформе Illumina Solexa. Мостиковая ПЦР и платформа Illumina Solexa, в основном, описаны в разнообразных публикациях например, Gudmundsson et al (Nat. Genet. 2009 41:1122-6), Out et al (Hum. Mutat. 2009 30:1703-12) и Turner (Nat. Methods 2009 6:315-6), US patent 7115400, и публикации заявок №№. US20080160580 и US20080286795.
Термин «штрих-код последовательность» в данном контексте относится к уникальной последовательности нуклеотидов, которая используется для идентификации и/или прослеживания источника полинуклеотида в реакции. Штрих-код последовательность может быть на 5'-конце или на 3'-конце олигонуклеотида. Штрих-код последовательности могут широко варьировать по размеру и составу, и последующие ссылки предоставляют руководство для отбора наборов штрих-код последовательностей для конкретных вариантов воплощения: Brenner, U.S. Pat. No. 5635400; Brenner et al, Proc. Natl. Acad. Sci., 97: 1665-1670 (2000); Shoemaker et al, Nature Genetics, 14: 450-456 (1996); Morris et al, European patent publication 0799897A1; Wallace, U.S. Pat. No. 5981179; и подобные. В конкретных вариантах воплощения штрих-код последовательность может иметь длину в диапазоне от 4 до 36 нуклеотидов, или от 6 до 30 нуклеотидов, или от 8 до 20 нуклеотидов.
Другие определения терминов могут появляться по всему описанию.
Подробное описание примерных вариантов воплощения
Определенные признаки рассматриваемого способа, описаны с отсылкой на Фиг.1, которая иллюстрирует вариант воплощения, в котором перед гибридизацией фрагмента с субстратом к фрагменту лигируют адаптеры. В альтернативных вариантах воплощения адаптер может быть добавлен позже по протоколу. Этот способ, в общем, включает получение субстрата, который содержит, по меньшей мере, два связанных с поверхностью олигонуклеотида с различающейся последовательностью, которые пространственно рассеяны один от другого. Такие субстраты в настоящее время применяются в технологии секвенирования Solexa Illumina и описаны в разнообразных ссылках, например, US patent no. 7115400 и публикация №№ US20080160580 и US20080286795, которые включены в данное раскрытие посредством ссылки. Некоторые из вариантов воплощения, изложенные ниже, могут описывать применение способа для изолирования фрагментов генома. Эти варианты воплощения могут быть легко адаптированы к другим типам последовательностей, например, кДНК или синтетической ДНК.
В определенных вариантах воплощения первый член первой популяции связанных с поверхностью олигонуклеотидов гибридизируется с олигонуклеотидом отбора, который содержит а) участок, который гибридизируется с первым членом, и участок, сайт праймера секвенирования и б) участок, который содержит геномную последовательность-мишень. Количество олигонуклеотида отбора, используемого на этом этапе, может быть оптимизировано таким образом, что достаточное количество олигонуклеотидов первой популяции остаются негибридизированными с олигонуклеотидом отбора и доступными для использования на этапе мостиковой ПЦР, который происходит позже по протоколу. Первый член первой популяции связанных с поверхностью олигонуклеотидов удлиняется для получения двойной спирали, которая содержит связанный с подложкой праймер отбора, который содержит последовательность, комплементарную. геномной последовательности-мишени. Олигонуклеотид отбора удаляется денатурацией, чтобы оставить удлиненный, связанный с подложкой праймер отбора. Удлиненный, связанный с подложкой праймер отбора затем гибридизируется с адаптер-лигированным геномным фрагментом (который может быть получен путем фрагментирования геномной ДНК, химическим, физическим путем или путем использования фермента, и затем лигированием адаптеров с концами полученных в результате фрагментов), содержащим геномную последовательность-мишень, последовательность, которая фланкирует геномную последовательность-мишень, и адаптерную последовательность на 5' конце одной из цепей или обеих цепей. Связанный с подложкой праймер отбора удлиняется для получения продукта, который содержит последовательность, которая фланкирует геномную последовательность в геноме и последовательность адаптера из адаптер-лигированного геномного фрагмента.
В некоторых вариантах воплощения адаптер из адаптер-лигированного геномного фрагмента может гибридизироваться со второй популяцией связанных с поверхностью олигонуклеотидов. Однако, в определенных случаях, перед амплификацией вторую популяцию связанных с поверхностью олигонуклеотидов можно гибридизировать для модификации олоигонуклеотида, содержащего а) участок, который гибридизируется со вторым членом, и участок, который содержит адаптерную последовательность. Количество модифицирующего олигонуклеотида, используемого на этом этапе, может быть оптимизировано таким образом, что гибридизируется достаточное количество молекул - продуктов. Второй член второй популяции связанных с поверхностью олигонуклеотидов может быть удлинен для получения двойной спирали, которая содержит связанный с подложкой праймер адаптера, который включает последовательность, комплементарную адаптерной последовательности. Модифицирующий олигонуклеотид удаляется путем денатурации, чтобы оставить связанный с подложкой праймер адаптера. Продукт затем можно амплифицировать путем мостиковой ПЦР.
Как проиллюстрировано на Фиг.1b, продукт амплифицируется как первым неудлиненным связанным с поверхностью олигонуклеотидом, так и вторым связанным с поверхностью олигонуклеотидом для получения продукта ПЦР. В определенных случаях геномным фрагментом является адаптер-лигированный геномный фрагмент, включающий адаптер с 5' конца. В этих случаях члены второй популяции связанных с поверхностью олигонуклеотидов гибридизируются для дополнения адаптера. В альтернативных вариантах воплощения адаптер может быть дотированным с продуктом удлинения, тем самым помещая адаптер, гибридизирующийся со второй популяцией связанных с поверхностью олигонуклеотидов, на 3' конец продукта удлинения. В других вариантах воплощения амплификацию проводят, используя: а) неудлиненные члены первой популяции связанных с поверхностью олигонуклеотидов; и б) связанные с подложкой праймеры, полученные с помощью: i. гибридизации членов второй популяции связанных с поверхностью олигонуклеотидов с олигонуклеотидом, содержащим участок, который гибридизируется с членами второй популяции связанных с поверхностью олигонуклеотидов, и участок, который комплементарный адаптеру; и ii. удлинение членов второй популяции связанных с поверхностью олигонуклеотидов для получения связанных с подложкой праймеров, гибридизирующихся с 5' концом продукта удлинения.
В некоторых вариантах воплощения геномный фрагмент является адаптер-лигированным геномным фрагментом, включающим адаптер 5' конца, где удлинение дает в результате продукт удлинения, который содержит на своем 3' конце последовательность, комплементарную адаптеру, и где во время мостиковой ПЦР члены второй популяции связанных с поверхностью олигонуклеотидов гибридизируются с последовательностью, которая комплементарна адаптеру. В этом варианте воплощения адаптер 5' конца содержит связывающий сайт для праймера секвенирования на конце, который лигирован с геномным фрагментом.
В других вариантах воплощения способ содержит между этапами е) и f) дотирование адаптера с 3' концом продукта удлинения, и где члены второй популяции связанных с поверхностью олигонуклеотидов гибридизируются с адаптером во время мостиковой ПЦР. В этих вариантах воплощения адаптер содержит связывающий сайт для праймера секвенирования на конце, который лигирован с геномным фрагментом.
В некоторых вариантах воплощения вторая популяции связанных с поверхностью олигонуклеотидов получена путем: i. гибридизации членов исходной второй популяции связанных с поверхностью олигонуклеотидов с олигонуклеотидом, содержащим участок, который гибридизируется с членами второй популяции связанных с поверхностью олигонуклеотидов, и участок, который комплементарный последовательности геномного фрагмента; и ii. удлинение членов исходной второй популяции связанных с поверхностью олигонуклеотидов для получения второй популяции связанных с поверхностью олигонуклеотидов.
В некоторых вариантах воплощения вторая популяции связанных с поверхностью олигонуклеотидов может быть получена путем лигирования олигонуклеотида, содержащего участок, комплементарный последовательности указанного фрагмента нуклеиновой кислоты, с исходной второй популяцией связанных с поверхностью олигонуклеотидов для получения указанной второй популяции связанных с поверхностью олигонуклеотидов. Этому лигированию может способствовать мостиковый нуклеотид, формирующий мостик между двумя олигонуклеотидами, подвергающимися лигированию. Другими словами, путем процесса, основанного на лигировании, может быть введен модифицирующий олигонуклеотид, при этом мостиковый олигонуклеотид используется для направления модификации оригинального олигонуклеотида твердой подложки с целью создания связанного с подложкой праймера адаптера. Сходным образом, может быть создан связанный с подложкой праймер адаптера с использованием подобного мостикового олигонуклеотида для создания удлинения праймера, необходимого для модификации мишени.
В некоторых случаях олигонуклеотид отбора содержит связывающий сайт для праймера секвенирования между указанным участком, который гибридизируется с указанным первым членом указанного участка, который содержит указанную геномную последовательность.
В некоторых вариантах воплощения данный способ может далее содержать секвенирование первой цепи ПЦР продукта для получения, по меньшей мере, части нуклеотидной последовательности от последовательности, которая фланкирует геномную последовательность. Этот способ может далее включать секвенирование второй цепи ПЦР продукта для получения, по меньшей мере, части нуклеотидной последовательности от последовательности, которая фланкирует геномную последовательность.
В конкретных вариантах воплощения данный способ может включать фрагментирование генома млекопитающих для получения фрагментированного генома, необязательно добавляя адаптеры к фрагментированному геному, и наложение фрагментированного генома на субстрат. Это фрагментирование производится физически, химически или с использованием рестрикционного фермента. Фрагментирование выполняется, например, путем обработки ультразвуком или гидродинамической фрагментацией.
В конкретных случаях гибридизация может быть проведена путем приготовления множества фрагментированных геномов из множества различных индивидуумов, собирания в пул множества фрагментированных геномов для получения пула, нанесение пула фрагментированных геномов на субстрат и получения ПЦР продуктов, которые включают последовательность, которая фланкирует геномную последовательность в различных индивидуумах. Эти варианты воплощения могут далее содержать секвенирование, по меньшей мере, первой цепи ПЦР продуктов для получения, по меньшей мере, части нуклеотидной последовательности от последовательности, которая фланкирует геномную последовательность в различных индивидуумах. В конкретных случаях перед собиранием в пул различные адаптеры дотируются с фрагментированными геномами от различных индивидуумов, где адаптер содержит «штрих-код» последовательность, которая позволяет идентифицировать источник лигированного с адаптером геномного фрагмента после секвенирования ПЦР продуктов.
В некоторых вариантах воплощения данный способ содержит: дотирование адаптера с фрагментированной геномной ДНК от первого субъекта с использованием первого адаптера, который содержит первую «штрих-код» последовательность, для получения первого продукта; дотирование адаптера с фрагментированной геномной ДНК от второго субъекта с использованием второго адаптера, который содержит вторую «штрих-код» последовательность для получения второго продукта; комбинирование первого и второго продуктов для получения смешанной матрицы, и выполнение способа пункта 1 с использованием смешанной матрицы для предоставления первого и второго ПЦР продукта, каждый из которых содержит «штрих-код» последовательность. Смешанная матрица в некоторых случаях может включать фрагментированную геномную ДНК от, по меньшей мере, 1000 субъектов.
В некоторых вариантах воплощения данный способ может задействовать i. лигирование геномных фрагментов с адаптером, содержащим сайт для праймера секвенирования и нуклеотидную последовательность, которая такая же, как и вторые связанные с поверхностью олигонуклеотиды, ii. гибридизацию дотированных с адаптером геномных фрагментом с первым членом первой популяции связанных с поверхностью олигонуклеотидов, iii. удлинение первого члена первой популяции связанных с поверхностью олигонуклеотидов, с которыми гибридизируется адаптер-летированный фрагмент; и iv. гибридизацию адаптер-содержащего конца продукта удлинения со вторым связанным с подложкой полинуклеотидом, тем самым создавая мостик и способствуя мостиковой ПЦР.
Также предоставляется система. В определенных случаях система может содержать: а) субстрат, включающий первую популяцию связанных с поверхностью олигонуклеотидов и вторую популяцию связанных с поверхностью олигонуклеотидов, где первая и вторая популяции связанных с поверхностью олигонуклеотидов пространственно не направлены на субстрат; б) олигонуклеотид отбора, который содержит участок, который гибридизируется с первым членом первой популяции, и участок, который содержит геномную последовательность; в) адаптер; и г) инструкции для проведения способа пункта 1. ПЦР продукт может быть секвенирован, например, с использованием платформы Solexa Illumina или другого твердофазного способа секвенирования, для получения, по меньшей мере, части нуклеотидной последовательности от последовательности, которая фланкирует геномную последовательность-мишень.
В конкретных вариантах воплощения данный способ может использовать последовательности штрих-кода, которые позволяют установить источник последовательности, которая фланкирует геномную последовательность-мишень. В этих вариантах воплощения из адаптер адаптер-лигированного геномного фрагмента может содержать «штрих-код» последовательность, что позволяет идентифицировать источник адаптер-лигированного геномного фрагмента после секвенирования ПЦР продукта. В конкретных вариантах воплощения этот способ содержит лигирование адаптера с фрагментированной геномной ДНК от первого субъекта (каковой субъект может быть включен в пул первых субъектов) с использованием первого адаптера, который содержит первую «штрих-код» последовательность для получения первого продукта; лигирование адаптера с фрагментированной геномной ДНК от второго субъекта (каковой субъект может быть включен в пул вторых субъектов) с использованием второго адаптера, который содержит вторую «штрих-код» последовательность для получения второго продукта; комбинирование первого и второго продуктов для получения смешанной матрицы; и выполнение вышеописанного способа, используя смешанную матрицу для предоставления первого и второго ПЦР продуктов, каждый из которых содержит «штрих-код» последовательность. В вышеописанном способе используемые адаптеры имеют часть, которая имеет одну и ту же последовательность, и которая гибридизируется со связанным с поверхностью олигонуклеотидом, и часть, которая имеет отличную нуклеотидную последовательность, которая содержит «штрих-код» последовательность.
Предоставляется второй способ амплификации избранной последовательности. Принцип этого способа подобен принципу способа, описанного выше, кроме того, что а) геномный фрагмент, который гибридизируется со связанным с подложкой праймером-отбора не лигирован с адаптером; и б) адаптеры после того, как связанный с подложкой праймер отбора удлиняется. Лигирование адаптера, продукт может быть применен в мостиковой ПЦР реакции, как обсуждается выше. Как в альтернативном варианте воплощения, описанном выше, амплификацию проводят, используя: а) неудлиненные члены первой популяции связанных с поверхностью олигонуклеотидов; и) связанные с подложкой праймеры, которые приготовлены путем: i. гибридизации членов второй популяции связанных с поверхностью олигонуклеотидов с олигонуклеотидом, содержащим участок, который гибридизируется с членами второй популяции связанных с поверхностью олигонуклеотидов, и участок, который комплементарный последовательности адаптера; и ii. удлинение членов второй популяции связанных с поверхностью олигонуклеотидов для получения связанных с подложкой праймеров. Как и со способом, описанным выше, ПЦР продукт может быть секвенирован для получения, по меньшей мере, части нуклеотидной последовательности от последовательности, которая фланкирует геномную последовательность.
В альтернативном варианте воплощения геномные фрагменты могут быть лигированы с адаптером, который не только содержит сайт связывания праймера секвенирования, но также и последовательность, которая является такой же, как и вторая популяция связанных с поверхностью олигонуклеотидов. Как показано, когда удлиненная первая популяция связанных с поверхностью олигонуклеотидов (которая обычно производится при высокой температуре, т.е., по меньшей мере, при 90°С) гибридизируется с дотированными с адаптером фрагментами и удлиняется, продукт удлинения содержит последовательность, которая гибридизируется со второй популяцией связанных с поверхностью олигонуклеотидов (которая обычно производится при более низкой температуре, например, ниже, чем 60°С, например, ниже, чем 55°С), тем самым способствуя амплификации геномных фрагментов с использованием первого и второго связанных с поверхностью олигонуклеотидов. Этот способ проиллюстрирован на Фиг.14.
В конкретных вариантах воплощения олигонуклеотиды первой популяции присутствуют в молярном избытке в, по меньшей мере, 5Х, 10Х, 20Х, 50Х или 100Х, 500Х, 1,000Х, 2000Х, 10000Х, 50000Х по отношению к количеству олигонуклеотида отбора, наложенного на субстрат. В одном варианте воплощения молярный избыток может быть в диапазоне от 5Х до 50000Х молярного избытка, например, от 100Х до 5000Х молярного избытка.
В определенных вариантах воплощения субстрат может входить в контакт со множеством различных селекционных олигонуклеотидов, каждый содержит участок, который гибридизируется с членами первой популяции связанных с поверхностью олигонуклеотидов (участок, который имеет одну и ту же нуклеотидную последовательность в различных олигонуклеотидах отбора), и участок, который содержит геномную последовательность. Геномная последовательность каждого из олигонуклеотидов отбора различна, тем самым позволяя захватывать, амплифицировать и секвенировать несколько геномных участков на субстрате.
Наборы
Также в настоящем раскрытии предоставляются наборы для осуществления на практике рассматриваемого способа, как это описано выше. В определенных вариантах воплощения рассматриваемый набор может содержать а) субстрат, включающий первую популяцию связанных с поверхностью олигонуклеотидов и вторую популяцию связанных с поверхностью олигонуклеотидов, где первая и вторая популяции связанных с поверхностью олигонуклеотидов пространственно не направлены на субстрат и б) олигонуклеотид отбора, который содержит участок, который гибридизируется с первым членом первой популяции, и участок, который содержит геномную последовательность. Набор может также содержать другие реагенты, описанные выше и ниже, которые могут быть использованы в способе, например, адаптеры, лигаза, гибридизационные буферы, т.п.
В дополнение к вышеуказанным компонентам рассматриваемый набор обычно далее включает инструкции для использования компонентов набора для осуществления на практике рассматриваемого способа. Инструкции для осуществления на практике рассматриваемого способа, в общем, записаны на подходящей для записи среде. Например, инструкции могут быть напечатаны на субстрате, таком, как бумага или пластик, т.п. Как таковые, инструкции могут присутствовать в наборе в качестве вкладыша в пакет, на этикетке контейнера набора или компонентов того (т.е., связанные с упаковкой или внутренней упаковкой), и т.п. В других вариантах воплощения инструкции присутствуют в файле электронного хранения данных, присутствующем на среде хранения, подходящей для компьютерного считывания, например CD-ROM, дискете, т.п. В еще других вариантах воплощения действующие инструкции не присутствуют в наборе, однако предоставляются средства для получения инструкций из отдаленного источника, например, через интернет. Примером этого варианта воплощения является набор, который включает интернет-адрес, где можно просмотреть инструкции и/или откуда инструкции можно загрузить. Так же, как и с инструкциями, эти средства для получения инструкций записаны на подходящем субстрате. Другие требуемые компоненты будут включать соответствующие компьютерные программы и/или компьютерные скрипты для проведения модификаций предыдущих программ, уже инсталлированных на секвенаторе.
В дополнение к инструкциям наборы также могут включать один или более контрольных смесей аналитов, например, два или более контрольных аналита для использования с целью тестирования набора.
Для дальнейшей иллюстрации настоящего изобретения предоставляются следующие конкретные примеры с пониманием того, что они предлагаются для иллюстрации настоящего изобретения, и ни в коем случае не как ограничения его объема.
Раскрытия предварительных заявок на патент США сер. номера 61/386390, поданной 24 сентября 2010 года, и 61/485062, поданной 11 мая 2011 года, включая все фигуры, примеры, детальное описание и олигонуклеотидные последовательности включены в настоящую заявку во всей полноте.
ПРИМЕРЫ
Ниже представлен новый подход для выполнения направленного секвенирования ДНК. Способ основан на модификации исходной площадки праймеров (т.е. площадки, содержащей, по меньшей мере, два праймера, которые случайным образом распределены) на твердофазной подложке для того, чтобы служить устройством для захвата ДНК-мишени, тем самым делая возможным прямое секвенирование захваченной ДНК и без значительных манипуляций с образцом. Этот способ делает возможным полную интеграцию захвата ДНК-мишени и постановку секвенирования на соответствующей струйной платформой. Этот подход использует универсальную площадку праймеров на твердофазной подложке в качестве субстрата для захвата ДНК, при этом сохраняя его потенциал для секвенирования. Данный способ может использовать необработанную, природную ДНК в качестве матрицы для секвенирования. Секвенирование с использованием этого способа не обязательно зависит от лабораторного оборудования. Более того, по сравнению с другими способами, избегаются многие отклонения, которые получаются при обработке образца, и значительно более малые образцы могут быть проанализированы за меньшее время и с меньшей стоимостью. Способ может быть использован для анализирования одноцепочечных и двуцепочечных матриц. Способность к анализированию одноцепочечных ДНК матриц может быть важной для некоторых применений секвенирования, которые используют фиксированные в формалине и заключенные в парафин образцы из патологических архивов. Сходным образом, позволяя проводить секвенирование одноцепочечных ДНК матриц, способ не требует сложных этапов экстрагирования нуклеиновой кислоты и дорогого оборудования для фрагментирования, разработанного с целью сохранения двуцепочечной структуры ДНК. Скорее, образец может быть приготовлен путем лизиса и тепловой фрагментации, что является недорогим и эффективным. Анализ секвенирования с прямым захватом не ограничивается человеческой геномной ДНК, но также могут анализироваться другие субстраты нуклеиновой кислоты, такие, как бактериальная и вирусная ДНК и РНК. Также могут захватываться и секвенироваться транскриптомы, некодирующие и миРНК. В дополнение, может изучаться захват и секвенирование нуклеотидной последовательности, других генетических и эпигенетиеских свойств, таких, как метилирование ДНК, большие геномные перестановки и экспрессия генов. Способ также может быть использован для отбора из популяции синтетической ДНК.
В общем, секвенирование считалось процессом, при котором образец ДНК структурно модифицируется для облегчения анализа на системе секвенирования. Описываемый ниже способ модифицирует систему секвенирования и, поэтому, нет необходимости модифицировать и в значительной степени готовить образец. Путем функционализирования исходной площадки праймеров при использовании библиотеки синтетических ДНК олигонуклеотидов гены-мишени из необработанных образцов могут быть анализированы напрямую. Для снижения неспецифического захвата специфические компоненты ДНК, которые предоставляют последовательности, используемые в образовании мостиковой структуры, вносятся последовательно, и площадка праймеров сама модифицируется. Приготовление библиотеки секвенирования для всех типов секвенаторов зависит от добавления специфических двуцепочечных адаптерных последовательностей в ДНК матрице. Поскольку олигонуклеотиды захвата служили в качестве адаптеров, иммобилизированных на твердой подложке, приготовление библиотеки для анализа требует только добавления единичного адаптера. Это существенно укорачивает процессинг образца и не требует ни клональной амплификации, ни разделения по размеру, основанного на гель-электрофорезе. В определенных случаях на твердой подложке к захваченной матрице может быть добавлен второй адаптер. Определенные варианты воплощения способа позволят использовать в качестве матрицы секвенирования необработанную ДНК.
Несколько текущих способов проведения высокоэффективного ре-секвенирования включают захват ДНК-мишени и секвенирование как отдельные способы. Это в определенном случае может приводить к множественным проблемам, включая i) значительные трудоемкие и занимающие много времени манипуляции с ДНК материалом, ii) ошибки - побочные эффекты сложных экспериментальных протоколов, iii) отклонения, создаваемые процессом отбора и молекулярной амплификации, и iv) требования к большому количеству исходного материала. Предполагается, что описываемый ниже способ, элиминирует источник многих из этих проблем, так как он задействует мало, или вовсе не задействует предварительных манипуляций с образцом, и является полностью автоматизированным и высоко масштабируемым.
Как доказательство правильности концепции были секвенированы все экзоны 10 раковых генов в человеческом геноме для демонстрации того, что анализ является воспроизводимым и может быть использован для захвата и секвенирования специфических участков генома. Эта технология анализа была продемонстрирована с анализатором Illumina Genome Analyzer, однако отметьте, что этот подход широко применим к любому секвенатору, использующему твердофазную подложку.
Описываемые ниже способы, некоторые из принципов, которые проиллюстрированы на Фигуре 1, могут быть использованы для эффективного захвата любой последовательности ДНК-мишени и позволяют проводить прямое секвенирование захваченных геномных фрагментов. Образец геномной ДНК может быть приготовлен для секвенирования простым этапом теплового фрагментирования и весь анализ может быть полностью автоматизирован и проводиться на твердой подложке. Захват и последующие реакции могут быть опосредованы струйной системой.
Дополнительный вариант воплощения предоставляет способ, который позволяет проводить приготовление ДНК фрагментов для секвенирования на твердой подложке путем использования фрагментированной ДНК в качестве матрицы и добавления адаптеров секвенирования к захваченным ДНК фрагментам с использованием струйных систем. Как доказательство правильности концепции для разработки этих подходов был использован ДНК секвенатор Illumina следующего поколения. Представлены результаты от интегрированного захвата и реакции подготовки секвенирования с использованием модификации площадки праймеров и 366 сайтов-мишеней человеческого генома. За исключением 25-минутной тепловой фрагментации все этапы могут быть выполнены на твердофазной подложке проточной ячейки Illumina.
Описываемые ниже данные демонстрируют надежность анализа и применимость универсальной площадки праймеров и струйной системы в качестве субстрата захвата. Были идентифицированы уникальные параметры модификации площадок праймеров, которые позволяют способу устойчиво работать, В дополнение к сложным эукариотическим геномам способ может быть применен для захвата геномов микробов и других организмов, вирусной ДНК и РНК, транскриптом от различных источников, наряду с синтетической ДНК. Более того, концепция «программирования» нативной площадки праймеров, иммобилизированной на твердой подложке струйной системы и выполнение конкретных применений, внедрено и валидизировано.
Материалы и методы
Образцы геномной ДНК. Была получена геномная ДНК NA18507 из института Кориэль (Coriell Institute). Свежезамороженные образцы тканей были поучены от пациента с колоректальным раком. Материал пациента был получен с формой информированного согласия от Стэндфордского Центра Рака (Stanford Cancer Center), и исследование было одобрено Экспертным советом организации Школы медицины Стэндфордского университета. Были приготовлены замороженные срезы ткани, проведено окрашивание гематоксилин-эозином, и был определен опухолевый состав каждого образца путем патологического исследования. Образцы, представляющие опухолевые и нормальные ткани, были отпрепарированы от зон, где клеточный состав был, соответственно, на 90% опухолевым или чисто нормальным. Геномная ДНК экстрагировалась с использованием набора E.Z.N.A SQ DNA/RNA Protein Kit (Omega Bio-Tek, Norcross, GA). Были использованы стандартные протоколы для приготовления ДНК, матричной гибридизации и сканирования для анализа образцов с использованием матриц SNP 6.0 (Affymetrix, Santa Clara, CA). Анализ данных проводился с использованием консольного программного обеспечения Genotyping Console и алгоритма Birdseed V2 (Affymetrix). Совместно с изучаемыми образцами были проанализированы тринадцать дополнительных наборов данных микроматрицы с целью оценки качества вызовов SNP (однонуклеотидный полиморфизм). Матричные данные SNP 6.0 фильтровались с использованием значения вероятности 0,01.
Выбор мишени и компьютерное моделирование олигонуклеотида OS-Seq. В качестве референсного набора данных по полиморфизму использовали конструкцию CCDS (проект консенсусных кодирующих последовательностей) выпуск 20090902, конструкции человеческого генома NCBI 37 - hg19 и dbSNP Конструкцию с ID 131. Для отбора генов была использована аннотированная база данных GeneRanker где выбрали 344 раковых генов, ранжированных по важности. Для того чтобы найти мишень-специфические последовательности олигонуклеотидов, указания границ экзонов для генов - кандидатов были взяты из CCDS. У большинства интересующих экзонов (менее 500 пар оснований), 40-мерными мишень-специфическими последовательностями были 10 оснований снаружи от 5' конца границы экзона (Фиг.3а). Нацелианиена обе цепи экзонов осуществлялось с использованием индивидуальных праймеров-зондов. OS-Seq-366 покрывал только фланги экзонов. В анализе OS-Seq-11k экзоны, содержащие больше чем 500 пар оснований, обрабатывались мостиковыми мишень-специфическими последовательностями до тех пор, пока не был покрыт полный экзонный участок (Фиг.3b). Для повышения специфичности попадания у OS-Seq-11k мы использовали Repbase для идентификации и элиминирования олигонуклеотидных последовательностей, которые были направлены на высокоповторяющиеся последовательности.
Синтез олигонуклеотидов. Для синтеза олигонуклеотидов были применены две стратегии. Для OS-Seq-366 мы создали 366 101-мерные олигонуклеотиды (Фиг.5а), которые были затем синтезированы на колонке (Stanford Genome Technology Center, Stanford, CA) (Фиг.4а). Олигонуклеотиды были количественно оценены и собраны в пул в эквимолярной концентрации. Для OS-Seq-11k, был использован микроматричный синтез in-situ (LC Sciences, Houston) для синтеза 11742 олигонуклеотидов - предшественников (Фиг.5b). Последовательности мишень-специфических олигонуклеотидов представлены в Таблице 2 ниже.


Амплификация олигонуклеотидов, синтезированных при помощи микроматрицы. Были использованы три под-пула по 25 мкл предшественников 80-мерных олигонуклеотидов (587, 638 и 415 нМ) (Фиг.5b). Для амплификации предшественника, низкой концентрации олигонуклеотидов был использован метод ПЦР (Фиг.4b). Под-пулы олигонуклеотидов, синтезированные при помощи микроматрицы, были разбавлены, до 10 фМ/олиго и использованы в качестве матрицы для ПЦР амплификации. ПЦР выполнялась с использованием Taq ДНК полимеразы (NEB), и дНТФ (1 мМ дАТФ, 1 мМ дЦТФ, 1 мМ цГТФ, 500 нМ дТТФ и 500 нМ дУТФ) в стандартных условиях реакции. После денатурации при 95°C в течение 30 сек. было выполнено 20 циклов амплификации (95°C, 30 сек.; 55°C, 30 сек.; 68°C, 30 сек.). Амплификационный Праймер 1 содержал урацил на 3' конце, в то время, как Амплификационный Праймер 2 включал дополнительные функциональные последовательности (Фиг.5b). Амплифицированные олигонуклеотиды были очищены для удаления избытка праймера (Fermentas), затем обработаны с использованием 0,1 Ед./мкл урацил-ДНК-вырезающей смесью (Uracil DNA-excision Mix) (Epicentre, Madison, WI) при 37°C в течение 45 для отделения универсального амплификационного праймера и расщепления зрелых 101-мерных кодирующих цепей олигонуклеотидов. Олигонуклеотидам требуется, чтобы 5' концы были функциональными и свободными для точного удлинения мишень-специфического сайта во время иммобилизации праймера-зонда. После инактивации ферментов способом теплового шока (65°C, 10 мин.) препараты олигонуклеотидов были очищены (Fermentas). Наконец, мы провели количественное определение трех под-пулов олигонуклеотидов и создали единственный пул с эквимолярной концентрацией каждого под-пула.
Получение OS-Seq праймеров-зондов путем модификации площадки праймеров проточной ячейки. В системе анализатора Illumina Genome Analyzer Их (Illumina, San Diego) твердофазная подложка (т.е., проточная ячейка) имеет два праймера («C» и «D»), которые случайным образом иммобилизированы на полиакриламидном слое при исключительно высокой плотности. Для экспериментов OS-Seq был специфически модифицирован под-набор «D» праймеров с использованием кластерной станции Illumina Cluster station. Перед модификацией праймеров в NGS 133 нМ пулы олигонуклеотидов были денатурированы нагреванием при 95°C в течение 5 мин. Мы использовали тепловой шок (95°C в течение 5 мин.) для высвобождения кодирующей цепи OS-Seq олигонуклеотидов. Дополнительная очистка цепи не требовалась, так как вторая цепь неактивна в проточной ячейке и она вымывается после гибридизации. Денатурированные олигонуклеотиды были разбавлены 4х гибридизационным буфером (20х SSC, 0,2% Tween-20). Полученные в результате 100 нМ олигонуклеотиды были использованы в экспериментах по модификации проточной ячейки. По 30 мкл смеси олигонуклеотидов было внесено в каждую дорожку проточной ячейки. Во время быстрого линейного уменьшения температуры (от 96°C до 40°C в течение 18 минут) олигонуклеотиды специфически отжигались на иммобилизированный праймер «D». Затем была использована ДНК-полимераза для удлинения праймера «D» с подвергшимися отжигу олигонуклеотидами в качестве матрицы. После удлинения первоначальная олигонуклеотидная матрица была денатурирована от удлиненного праймера «D» и отмыта от твердофазной подложки. Для этапов удлинения, отмывания и денатурации использовались стандартные реагенты Illumina v4. Модификация праймера «D» вызывала иммобилизацию праймеров-зондов.
Получение библиотеки секвенирования. Мы намечаем в общих чертах общую схему фрагментации геномной ДНК, восстановления концов, наращивание А-«хвоста», дотирования адаптера и ПЦР, используемых в получении библиотеки секвенирования OS-Seq на Фиг.2. Мы использовали 1 мкг геномной ДНК из NA18507 и моментально замороженный образец колоректального рака в качестве начального материала. Геномная ДНК была фрагментирована с использованием Covaris E210R (Covaris, Woburn, MA) для получения фрагмента среднего размера в 500 пар оснований (рабочий цикл 5%, интенсивность 3200 циклов на импульс и 80 секунд). В случайным образом фрагментированной ДНК были восстановлены концы с использованием 0,25 ед. большого фрагмента Кленова (New England Biolabs, Ipswich, MA), 7,5 ед. Т4 ДНК полимеразы (NEB), по 400 мкМ каждого дНТФ (NEB), 25 ед. Т4 полинуклеотид киназы (NEB) и Т4 ДНК-лигазного буфера с АТФ (NEB) в 50 мкл реакционном объеме при комнатной температуре в течение 45 минут. После восстановления концов аденины были добавлены к 3' концам матричной ДНК с использованием 3,2 ед. Taq ДНК-полимеразы (NEB), 100 мкМ дАТФ (Invitrogen) и Taq буфера с 1.5 мМ MgCl2 в 80 мкл реакции при 72°C в течение 15 мин. Перед лигированием адаптера реакции были очищены с использованием набора ПЦР очистки (Fermentas).
Была разработана система указателей для OS-Seq. Адаптеры библиотеки секвенирования содержат необязательную указательную последовательность из 6 оснований, праймер секвенирования сайта 1 и 12-мерную последовательность для праймера гибридизации «C» (Таблица 2 выше. Фиг.5c). Было разработано 16 намеченных указательных адаптеров. Адаптерные олигонуклеотиды были синтезированы в Стэндфордском Центре Геномной Технологии (Stanford Genome Technology Center). Перед лигирование адаптерные олигонуклеотиды отжигались во время быстрого линейного уменьшения температуры. Для направленного ре-секвенирования NA18507 мы использовали как синплексный адаптер, наряду с мультиплексным адаптером с меткой 'AACCTG'. Для индексирования подходящего образца нормальной опухолевой ткани мы использовали штрих-код 'TGCTAA' для нормальной ткани, в то время как опухолевый образец метился 'AGGTCA'. Адаптеры - двуцепочечные ДНК - с выступающим липким концом Т были лигированы с матрицами с А-«хвостом» с использованием 2000 ед. Т4 ДНК лигазы (NEB) и Т4 ДНК лигазного буфера при комнатной температуре в течение 1 часа. После лигирования адаптера реакционные смеси были очищены с использованием набора для ПЦР очистки (Fermentas), и библиотеки были амплифицированы с использованием ПЦР. Было приготовлено 50 млк реакции 1 ед. ДНК-полимеразы Phusion Hot Start (Finnzymes, Finland), 1 мкМ библиотеки праймера амплификации (Дополнительная Таблица 1), буфер Phusion HF и по 200 мкМ каждого дНТФ (NEB). Реакции были денатурированы при 98°C в течение 30 сек. После этого было выполнено 22 цикла ПЦР (98°C в течение 10 сек., 65°C в течение 30 сек. и 72°C в течение 30 сек.) с последующими 72°C в течение 7 мин. и 4°C. Затем продукты PCR реакции были очищены с использованием набора для ПЦР очистки (Fermentas) и количественно определены. Мультиплексированные библиотеки были собраны в пул в равных концентрациях.
Захват мишеней с использованием праймеров-зондов. Мишени захватывались на проточной ячейке с использованием праймеров-зондов OS-Seq (Фиг.1b и олигонуклеотидные последовательности ниже). Мы вводили 30 мкл библиотек геномного секвенирования (30-42 нг/мкл) в проточную ячейку. ДНК-мишень гибридизировали с праймерами-зондами путем инкубирования библиотек секвенирования в проточной ячейке при 65°C в течение 20 часов. Во время гибридизации геномной ДНК библиотек и последующего удлинения проточная ячейка выдерживалась при постоянной температуре 65°C. Для этапов проведения гибридизации праймера-зонда и удлинения была использована кластерная станция Illumina Cluster Station. Перед гибридизацией с праймерами-зондами 22,5 мкл библиотеки секвенирования (40-56,6 нг/мкл) было денатурировано при 95°C в течение 5 мин. После теплового шока библиотеки геномной ДНК были разбавлены до общего объема 30 мкл с использованием 4х гибридизационного буфера. Окончательные концентрации ДНК библиотеки секвенирования находилась в диапазоне от 30 до 41,7 нг/мкл. Из-за высокой концентрации библиотеки секвенирования удерживался минимальный гибридизационный объем. Поэтому была разработана специализированная программа кластерной станции для обеспечения воспроизводимой гибридизации в малом объеме. Последующие удлинение, отмывание и денатурация выполнялись с использованием реагентов Illuming v4.
Процессинг и секвенирование в проточной ячейке. После захвата мишеней температура в проточной ячейке понижалась до 40°C в течение 30 минут, чтобы дать возможность 12 основаниям на 3' конце захваченных фрагментов библиотеки геномной ДНК гибридизироваться с праймером «C» (Фиг.1b и олигонуклеотидные последовательности ниже). При образовании мостика фрагменты библиотеки и праймер «C» удлинялись с использованием ДНК полимеразы для завершения и репликации захваченного фрагмента ДНК. Потом проводилась мостиковая ПЦР для создания клонально амплифицированных кластеров секвенирования. Образцы секвенировались с использованием циклов со спаренными концами - 40 на 40 (OS-Seq-366) или 60 на 60 (OS-Seq-11k) на геномном анализаторе Illumina Genome Analyzer IIх с использованием стандартной версии 4 реагентов секвенирования и рецептов (Illumina). Анализ изображений и вызов оснований выполняли с использованием программного обеспечения SCS 2.8 и RTA 2.8 (Illumina).
Анализ последовательностей и детекция вариантов. Считанные последовательности выравнивали по отношению к человеческому геному версии человеческий геном конструкция NCBI 37 - hg19 с использованием программы для выравнивания Burrows-Wheeler Aligner (BWA)19. После выравнивания считывания на мишени (Считывание 1) были определены как находящиеся в пределах 1 кб от 5' конца праймера-зонда. Считывания за пределами мишени были определены как выравнивающиеся снаружи(вне) 1 кб от 5' конца праймера-зонда или картирующиеся на другой хромосоме, отличной от расположения ассоциированного праймера-зонда. Для демультиплексирования индексированных дорожек мы использовали perl-скрипт для создания указателя меток из 7 оснований с использованием файлов вызова оснований. Этот указательный файл и другой perl-скрипт использовались для демультиплексирования либо комбинированного файла вызова оснований (так, чтобы могли создаваться отдельные файлы fastq для дальнейшего процессинга), либо выравненного файла.
Для элиминирования любых последовательностей синтетического праймера-зонда для вызова вариантов, было применено фильтрования размера вставки у подходящих пар. Размер вставки определялся путем сравнения выравнивания считываний спаренных последовательностей. Для вызова вариантов требовалось, чтобы экстрагированные последовательности имели размер вставки более, чем [40 + длина Считывания 1]. После фильтрования размера вставки проводился вызов вариантов с использованием инструментов SAMtools и BCFtools. Выполнялось наложение последовательностей по отношение к человеческому геному (hg19) с использованием mpileup SAMtools с пределом качества картирования, равным 50. Обзор BCFtools использовался для генотипирования положений оснований и данные фильтровались с использованием vcfutils.pl, фильтра вариантов perl-скрипта, предоставляемого в пакете SAMtools. Условия vcfutils varFilter были следующие: i) покрытие, равное 10 или более, ii) удаление фильтра отклонения цепи (поскольку OS-Seq является способом специфического для цепи захвата), iii) вынуждающего скрипт выдавать как референсные, так и нереференсные положения. Референсные и нереференсные вызовы использовались для сравнения с данными матрицы Affymetrix SNP 6.0. Генотипированные положения фильтровались для получения балла качества типа Phred свыше 50. Мы использовали BEDtools intersectBed для определения участков-мишенней для каждого праймера-зонда и комбинаций, где зонды частично совпадают в своих мишенях.
Сравнение вариантов. Для оценки качества экстрагированных вариантов вызовы вариантов данных NA 18507 сравнивали с вызовами от вариантов, идентифицированных из полного анализа геномной последовательности3 и данными генотипирования Hapmap (www.hapmap.org). Сравнения данных OS-Seq и данных матрицы Affymetrix SNP 6.0 были сделаны с использованием perl-скриптов. Для аннотирования SNP использовался dbSNP131.









Результаты
Данный раздел описывает новый подход для направленного секвенирования, названный Олигонуклеотид-селективное секвенирование (OS-Seq), который разрешает многие из ограничений, наблюдаемых в подходах направленного секвенирования. Концептуально отличный от других способов, OS-Seq является интегрированным подходом, при котором как захват, так и секвенирование геномных мишеней выполняется на твердофазной подложке NGS, такой, как проточная ячейка Illumina (Фиг.1а). Для приготовления OS-Seq из геномной ДНК готовится одноадаптерная библиотека секвенирования, и синтезируются мишень-специфические олигонуклеотиды, которые используются для конструирования праймеров-зондов на проточной ячейке. Затем иммобилизированные праймеры-зонды на проточной ячейке используются для захвата одиночных молекул - мишеней из одноадаптерной библиотеки геномной ДНК.
Процессинг OS-Seq включает три этапа, где система секвенирования Illumina модифицируется с включением мишень-специфических праймеров-зондов, мишени захватываются из одноадаптерной библиотеки и иммобилизированные фрагменты завершаются для секвенирования (Фиг.1b). Для приготовления субстрата захвата мы молекулярно перепроектировали проточную ячейку Illumina путем модификации подмножества существующих площадок праймеров с тем, чтобы они превратились в мишень-специфические праймеры-зонды. Для создания этих праймеров-зондов мы гибридизировали 3' универсальную последовательность сложного пула олигонуклеотидов с их комплементарными молекулами на проточной ячейке и удлинили иммобилизированный праймер с использованием реакции удлинения ДНК полимеразой. Результатом явился набор расположенных случайным образом мишень-специфических праймеров-зондов, фиксированных на поверхности проточной ячейки. Во время высокотемпературной инкубации при 65°C праймеры-зонды специфически гибридизируются с комплементарными последовательностями мишеней в одноадаптерной библиотеке геномной ДНК; после гибридизации праймеры-зонды затем функционируют как праймеры для другой реакции удлинения ДНК полимеразой. Этап удлинения эффективно захватывает последовательность-мишень. После удлинения выполняется этап денатурации и последующей низкотемпературной гибридизацией при 40°C для стабилизации адаптера библиотеки секвенирования с его комплементарным компонентом на проточной ячейке, что создает мостиковую структуру. Третья реакция удлинения ДНК полимеразой встраивает дополнительную последовательность на 3' конце, создавая две молекулы, способные к твердофазной амплификации. После трех этапов, специфических для OS-Seq, захваченные молекулы поддаются мостиковой амплификации, процессингу и секвенированию с использованием стандартного протокола системы секвенирования Illumina NGS. Детальное описание молекулярно-биологических этапов в OS-Seq приводится выше и, соответственно, для OS-Seq модифицируются программы кластерной станции Illumina для OS-Seq.
В качестве демонстрации для доказательства принципа было разработано два анализа захвата. Первый, были сконструированы 366 праймеров-зондов OS-Seq для фланкирования экзонов 10 раковых генов (OS-Seq-366) (Фиг.3). Этот анализ был предназначен для тестирования способа OS-Seq, а не для покрытия определенного экзона. Мы синтезировали OS-Seq-366 олигонуклеотиды с использованием способов, основанных на колонках. Второй, для демонстрации масштабируемости мы сконструировали и синтезировали 11742 праймера-зонда для захвата экзонов 344 раковых генов (OS-Seq-11k). Для улучшенного покрытия экзонов эти праймеры-зонды избегали повторов и перекрывали все большие экзоны. Для высокоэффективной наработки OS-Seq-11k мы синтезировали олигонуклеотиды на программируемой микроматрице. Эти синтезированные на матрице олигонуклеотиды требовали амплификации для процессинга и для получения достаточного материала для OS-Seq (Фиг.4). После процессинга OS-Seq олигонуклеотиды содержат мишень-специфический 40-мер, комплементарный 5' концу участка-мишени (Фиг.5). Эти олигонуклеотиды также содержат последовательность, необходимую для отжига праймера секвенирования со спаренными концами и для гибридизации иммобилизированнного праймера к площадке иммобилизированнного праймера на проточной ячейке.
Для оценки выполнения захвата анализами OS-Seq-366 и OS-Seq11k была приготовлена ДНК от предварительно секвенированного представителя Йоруба (NA 18507). В трех анализах таргетинга проводилось секвенирование спаренных концов. Первое считывание (Считывание 1) получено от геномной ДНК-мишени, в то время, как второе считывание (Считывание 2) происходит от синтетических мишень-специфических праймеров-зондов (Фиг.1а). OS-Seq-366 проводилась на одной дорожке пробега GAIIx. Каждый образец OS-Seq-11k разгоняли на равноценных 1,3 дорожках, на основе нашей схемы индексирования. Мы разработали схему индексирования с использованием адаптеров с уникальной «штрих-код» последовательностью (Фиг.5c) для метки образцов. Штрих-коды были получены от первых семи оснований Считывания 1. В целом, 87,6% считываний OS-Seq-366 и 91,3% считываний OS-Seq-11k, содержащих правильные штрих-коды, картировались со ссылкой на человеческий геном (Таблица 1). По сравнению с этим, могли быть картированы со ссылкой на человеческий геном 58% считываний, полученных с использованием ранее докладывавшегося способа гибридной селекции.
Для оценки общего покрытия каждого праймера-зонда мы определяли количество считываний, происходящих из данных Считывания 1, которые попадали в 1 кб от 3' конца праймера-зонда. OS-Seq праймеров-зондов является специфичным к цепи и захватывает только 5' концы ДНК мишеней (Фиг.6). В качестве примера, средний профиль покрытия всех праймеров-зондов в OS-Seq-366 (Фиг.1а) иллюстрирует, как захватывается последовательность в пределах 1 кб ниже праймера-зонда по направлению считывания. В целом, было обнаружено смещение в отношении более малых размеров вставки, для OS-Seq-366 50% направленных считываний картировалось в пределах 283 оснований от праймеров-зондов. В обоих анализах были идентифицированы дополнительные считывания за пределами 1 кб интервала, удаленные вплоть до 1,7 кб. Считывания последовательность за пределами 1 кб представляет собой конец хвоста распределения захвата от любого данного праймера-зонда и составляло менее, чем 0,15% всех данных последовательностей как от OS-Seq-366, так и от OS-Seq-11k. Также наблюдалось, что характеристики распределения покрытия коррелировали с размером фрагмента, введенного при создании библиотеки, и ограничений размера, присущего образованию мостиков и твердофазной ПЦР (Фиг.6). Также может повышать покрытие вдоль мишени введение более высокой молярной концентрации библиотики с одинаковым адаптером, дополнительные дорожки секвенирования или использование более длинных считываний.
Считывания на мишени были определены как последовательности Считывания 1, картируемые в пределах 1 кб от праймера-зонда. Используя эти критерии покрытия мишени, на мишени находилось 86,9% считываний по 40 оснований в OS-Seq-366 и 93,3% считываний по 53 основания в OS-Seq-11k (Таблица 1). OS-Seq-11k продемонстрировал улучшенную специфичность с учетом усилий по усовершенствованию схемы компьютерного моделирования праймеров-зондов. В частности, для отбора праймера-зонда для OS-Seq-11k при помощи компьютерного моделирования был использован фильтр экранирования повторов, что в результате привело к меньшему количеству считываний вне мишени. В сравнение, 89% считываний по 76 оснований и 50% считываний по 36 оснований, картированных вблизи зонда в опубликованном способе отбора гибридов, свидетельствуют об одинаковой специфичности в отношении мишени у обоих способов, и данные склоняются к тому, что направление в сторону более длинных считываний может улучшить специфичность по отношению к мишени в OS-Seq. Специфичность OS-Seq по отношению к экзону также была сходная с таковой у опубликованного способа отбора гибридов. С использованием OS-Seq-11K мы наблюдали, что 42,7% считываний картировалось в пределах экзознов (Таблица 1), в то время, как в технологии захвата с отбором гибридов сообщалось о 42% считываний, картированных на экзонах.
В качестве примера типичного профиля покрытия гена мы представляем данные для захваченной последовательности гена KRAS на Фиг.1c. Экзоны - мишени секвенировались при многократном покрытии по отношению к прилегающим участкам, находящимся вне мишени. Как ранее отмечалось, OS-Seq-366 был создан для фланкирования экзонов и он не перекрывал большие участки. Средне кратное покрытие для экзонов представлено в Таблице 1, и детальные разбивки по классам покрытия (т.е. 10Х, 20Х) - в Таблице 2. В целом, 83,9% оснований экзонов в OS-Seq-366 были покрыты, по меньшей мере, одним считыванием, при этом в данном пилотном исследовании преднамеренно не было нацеливания на оставшуюся часть нацеливания. Сходным образом, среди трех образцов, проанализированных с OS-Seq-11k, от 94 до 95,6% оснований экзонов были покрыты, по меньшей мере, одним считыванием. По сравнению с OS-Seq-366, анализ OS-Seq-11k продемонстрировал повышенное покрытие последовательностей в экзонах благодаря улучшению дизайна праймера-зонда по сравнению с дизайном OS-Seq-366, конкретно, OS-Seq-11k создавал перекрвающиеся праймеры-зонды для экзонов, имеющих больше 500 оснований.
Также в анализах оценивалось единообразие выбора мишени путем группирования данных Считывания 1 по сопутствующим им праймерам-зондам и подсчета выравниваний с мишенью. Праймеры-зонды OS-Seq были рассортированы на основе наблюдаемых результатов захвата, и распределения по OS-Seq-366 и OS-Seq-11k представлены в виде покрытий на Фиг.1d. В OS-Seq-366 наблюдалось, что 100% праймеров-зондов имели результирующий выход, минимум, в считывании одной последовательности, и выход от 89,6% праймеров-зондов был в 10-кратном диапазоне. Сходным образом, для OS-Seq-11k 95,7% праймеров-зондов имели результирующий выход захватов, минимум, одной последовательности, и 54% праймеров-зондов имели выход в 10-кратном диапазоне. Олигонуклеотиды OS-Seq-366 были синтезированы на колонке, и их количество определялось отдельно перед собиранием в пул, что обеспечивает то, что каждая мишень-специфическая последовательность была в эквимолярной концентрации на этапе конструирования праймера-зонда. Большая вариабельность в результирующих выходах праймера-зонда для OS-Seq-11k, скорее всего, объясняется ошибками амплификации, возникшими во время ПЦР олигонуклеотидов, синтезированных при помощи микроматрицы, которые использовались для создания праймера-зонда.
Техническая воспроизводимость OS-Seq оценивалась путем сравнения выхода последовательностей индивидуальных праймеров-зондов в результате анализа OS-Seq-11k (Фиг.7). Мультиплексированные библиотеки (NA 18507, нормальные и опухолевые) собирали в пул и проводили захват и секвенирование на двух независимых дорожках Illumina GAIIx. Выходы последовательностей каждого индивидуального праймера-зонда сравнивали среди технических реплик и рассчитывали коэффициент корреляции: R2=0,986. Для оценки биологической воспроизводимости две различные мультиплексированные библиотеки секвенирований прогоняли на одной и той же дорожке. Коэффециент корреляции биологических реплик равнялся R2=0,90. Высокая воспроизводимость OS-Seq, очевидно, связана с присущей применению системы NGS автоматизацией, способность выполнять этапы захвата и секвенирования в одной реакции и отсутствием необходимости в применении ПЦР после захвата.
Для оценки выполнения вызова вариантов анализов OS-Seq-366 и OS-Seq-11k были проведены анализы, анализ направленного секвенирования на NA 18507, представителя Йоруба, у которого было ранее проведено полное секвенирования генома. Для вызова SNV каким-либо из анализов OS-Seq мы анализировали только положения на мишени с баллами качества генотипа выше 50 и, минимум, 10Х покрытия (Таблица 1). Для данных OS-Seq-366 и OS-Seq-11k всего выполняли эти критерии, соответственно, 191 кб и 1541 кб. Из этих намеченных позиций высокого качества мы вызывали 105 SNV из OS-Seq-336 и 985 SNV из OS-Seq-11k (Таблица 1). Мы получили и опубликованные SNV NA18507 и другие доложенные SNP, которые встречаются в тех же участках высокого качества. В сравнении, ранее докладывались 97% OS-Seq-366 и 95,7% OS-Seq-11k (Таблица 1). Для OS-Seq-366 и OS-Seq-11k чувствительность детекции вариантов, основанная на доложенных SNP, была, соответственно, 0,97 и 0,95 (Таблица 3 ниже).
Анализ OS-Seq-11k также был применен к геномной ДНК, полученной от соответствующих опухолевых пар нормальная ткань - колоректальная карцинома. Используя те же идентифицированные критерии качества и покрытия NA18507, было идентифицировано 871 SNV из нормального образца и 727 из опухоли (Таблица 4). Для сравнения, эти два образца были генотипированы при помощи матрицы Affymetrix SNP 6.0 array. В соответствии с предыдущими анализами, точность генотипирования с использованием матриц Affymetrix SNP 6.0 и алгоритма Birdseed является высокой, так как среднийй уровень успешных вызовов для SNPs равен 99,47% и вызванные SNP имеют 99,74% соответствия с генотипами НарМар от других платформ. При сравнивании SNVOS-Seq с SNP Affymetrix наблюдалось высокое соответствие, равное 99,8% для нормальной ткани и 99,5% для опухоли. Путем фильтрования вариантов нормальной ткани и учета новых раково-специфических вариантов, где покрытие было больше 40, была идентифицирована и валидизирована четкая патогенная несмысловая мутация SMAD4 (S144*). Этот ген часто мутирует при колоректальном раке и является геном, запускающим рак прямой кишки.
Была исследована эффективность захвата индивидуальных праймеров-зондов в анализах OS-Seq-366 и OS-Seq-11k, и была оценена производительность каждого праймера-зонда. Уникальной характеристикой OS-Seq является то, что захваченные последовательности генома при секвенировании со спаренными концами могут совпадать со своими соответствующими праймерами-зондами. Считывание 1 происходит с 3' конца захваченной мишени и Считывание 2 начинается на синтетической последовательности праймера-зонда OS-Seq. Таким образом. Считывание 1 всегда представляет захваченную последовательность геномной ДНК, в то время как Считывание 2 функционально служит в качестве молекулярного штрих-кода для индивидуального праймера-зонда. Это дает возможность идентификации точного праймера-зонда OS-Seq, который обусловливает направленность и способствует оценке выполнения индивидуальных праймеров-зондов. Например, мы наблюдали сильную взаимосвязь между содержанием праймера-зонда ГЦ и выходом последовательности-мишени (данные не показаны). Исключительно низкое содержание ГЦ (менее 20%) или высокое содержание ГЦ (>70%) было связано с увеличивающейся неспособностью праймера-зонда захватывать свою мишень (Фиг.8). Считается, что способность напрямую оценивать выполнение захвата будет важным показателем контроля качества праймера-зонда.
Технология OS-Seq была разработана для хорошо налаженного и масштабированного направленного секвенирования. Будучи отходом от традиционных способов захвата при обогащении мишени перед секвенированием, OS-Seq интегрирует захват и секвенирования ДНК-мишени через гибридизацию и отбор на твердофазной подложке системы NGS. Данное исследование доказательства принципа показывает, что анализ OS-Seq эффективно и воспроизводимо захватывает участки-мишени генома с хорошим единообразием и высокой специфичностью. Анализ вариантов референсного генома NA18507 продемонстрировал высокую специфичность и низкий уровень ложных открытый для определения SNV. Направленное секвенирование подходящих образцов нормальной и опухолевой ткани колоректального рака продемонстрировало применимость OS-Seq к высокоэффективному генетическому анализу раковых геномов.
Технология OS-Seq дает возможность создавать индивидуализированные направленные анализы ре-секвенирования. Конструирование и получение олигонуклеотидов праймера-зонда является относительно простым, и участи-мишени могут быть легко отобраны путем использования сбалансированных ГЦ и неповторяющейся последовательности. Могут быть использованы ресурсы программируемого синтеза при помощи микроматрицы для создания индивидуализированных и сложных олигонуклеотидных библиотек en masse. Сходным образом, могут быть использованы способы традиционного синтеза для создания индивидуализированных анализов для меньших наборов генов-мишенней. В то время, как наш самый большой направленный анализ покрывал экзоны и прилегающую последовательность 344 генов, мы считаем, что OS-Seq может быть в значительной степени масштабирован для большего содержания мишеней. Из данных OS-Seq-366 мы установили, что в гибридизациолнной смеси в проточной ячейке существовал более, чем 2000-кратный избыток праймеров-зондов по сравнению с фрагментами мишени. Во время 20-часовой гибридизации мы устанавливаем, что 4,9% всех потенциальных мишеней в библиотеке были захвачены для секвенирования. Мы также протестировали то, что концентрация олигонуклеотидов может быть увеличена, по меньшей мере, 10-кратно, и концентрация библиотеки секвенирования может быть увеличена 5-кратно (данные не показаны) без подвергания риску образование кластеров.
Приготовление образца OS-Seq является простым: оно может быть выполнено в течение одного дня и легко автоматизируется (Фиг.9). В отношении трудозатрат, использование OS-Seq выигрывает в сравнении с выполнением эксперимента секвенирования способом стохастических геномных фрагментов. Из-за того, что оставшиеся адаптеры не гибридизируются в проточной ячейке по время захвата, библиотеки OS-Seq могут использовать ДНК фрагменты варьирующих размеров без необходимости в очистке узкого диапазона размеров способами физического разделения. К 5' концу фрагментов геномной ДНК требуется только добавление одинаковых адаптеров. Конструкция одного адаптера также легко приспосабливается под индексирование с введением молекулярного штрих-кода. Эта характеристика позволяет проводить прямое мультиплексирование образца матрицы секвенирования и имеет много потенциальных применений. Например, анализ соответствующих образцов нормальной и опухолевой ткани происходит в одной и той же реакции захвата, что может снижать отклонения.
С учетом возрастающего интереса к «персонализированной медицине» существует четкая потребность в разработке быстрых и простых подходов к ре-секвенированию человеческого генома. Это включает анализ вариантов зародышевой линии и соматических мутаций, обнаруживаемых в раковых геномах. Как практический и эффективный подход к направленному секвенированию, OS-Seq особенно полезен для исследований трансляции и клинической диагностики путем предоставления возможности высокоэффективного анализа генов - кандидатов и идентификации участков-мишеней, на которых может осуществляться клиническое воздействие.
Для описываемого выше способа был использован анализатор Illumina Genome Analyzer. Однако ожидается, что эта система будет широко применима к любой параллельной платформе секвенирования.
Реферат
Изобретение относится к биотехнологии, а именно к способу захвата и амплификации избранной последовательности и набору для его осуществления. Способ включает получение субстрата, представленного твердофазной подложкой, содержащего первую и вторую популяции связанных с поверхностью олигонуклеотидов, где данные олигонуклеотиды распределены случайным образом относительно друг друга на субстрате. Гибридизируют первый член первой популяции с синтетическим олигонуклеотидом отбора, содержащим участок, который гибридизируется с первым членом, и участок, который содержит геномную последовательность. Удлиняют первый член первой популяции для получения дуплекса, который содержит связанный с подложкой праймер отбора, содержащий последовательность, комплементарную геномной последовательности. Удаляют олигонуклеотид отбора путем денатурации, оставив удлиненный, связанный с подложкой праймер отбора. Гибридизируют связанный с подложкой праймер отбора с фрагментированным геномом, содержащим фрагмент нуклеиновой кислоты, включающий геномную последовательность. Удлиняют связанный с подложкой праймер отбора для получения продукта удлинения, который содержит последовательность, комплементарную фланкирующей последовательности, где фланкирующая последовательность фланкирует геномную последовательность. Амплифицируют геномный продукт на субстрате с помощью мостиковой ПЦР с использованием неудлиненных членов первой и второй популяции связанных с поверхностью олигонуклеотидов, для получения продукта ПЦР. Предложенное изобретение позволяет осуществить обогащение мишени непосредственно на субстрате, �
Формула
а) получение субстрата, представленного твердофазной подложкой, содержащего первую популяцию связанных с поверхностью олигонуклеотидов, и вторую популяцию связанных с поверхностью олигонуклеотидов, где члены указанной первой и второй популяции связанных с поверхностью олигонуклеотидов распределены случайным образом относительно друг друга на указанном субстрате;
б) гибридизацию первого члена указанной первой популяции связанных с поверхностью олигонуклеотидов с синтетическим олигонуклеотидом отбора, содержащим участок, который гибридизируется с указанным первым членом, и участок, который содержит геномную последовательность,
в) удлинение указанного первого члена указанной первой популяции связанных с поверхностью олигонуклеотидов для получения дуплекса, который содержит связанный с подложкой праймер отбора, который содержит последовательность, комплементарную указанной геномной последовательности, и удаление олигонуклеотида отбора путем денатурации, оставив удлиненный, связанный с подложкой праймер отбора;
г) гибридизацию указанного связанного с подложкой праймера отбора с фрагментированным геномом, содержащим фрагмент нуклеиновой кислоты, включающий указанную геномную последовательность;
д) удлинение указанного связанного с подложкой праймера отбора для получения продукта удлинения, который содержит последовательность, комплементарную фланкирующей последовательности, где фланкирующая последовательность фланкирует указанную геномную последовательность;
е) амплификацию указанного геномного продукта на указанном субстрате с помощью мостиковой ПЦР с использованием неудлиненных членов указанных первой и второй популяции связанных с поверхностью олигонуклеотидов для получения продукта ПЦР,
где указанный фрагмент нуклеиновой кислоты является лигированным с адаптером геномным фрагментом, включающим 5′ концевой адаптер, и тем, что указанное удлинение производит продукт удлинения, который содержит на своем 3′ конце последовательность, которая комплементарна указанному адаптеру, и тем, что члены указанной второй популяции указанных связанных с поверхностью олигонуклеотидов гибридизируются с указанной последовательностью, которая комплементарна указанному адаптеру, во время указанной мостиковой ПЦР; или
где указанный способ содержит, между этапами д) и е), лигирование адаптера к 3′ концу указанного продукта удлинения, и где члены указанной второй популяции указанных связанных с поверхностью олигонуклеотидов гибридизируются с указанным адаптером во время указанной амплификации; или
где указанная вторая популяция связанных с поверхностью олигонуклеотидов сделана путем:
i. гибридизации членов исходной второй популяции связанных с поверхностью олигонуклеотидов с олигонуклеотидом, включающим участок, который гибридизируется с членами указанной второй популяции связанных с поверхностью олигонуклеотидов, и участок, комплементарный последовательности указанного фрагмента нуклеиновой кислоты; и
ii. удлинение указанных членов указанной исходной второй популяции связанных с поверхностью олигонуклеотидов для получения указанной второй популяции связанных с поверхностью олигонуклеотидов.
лигирование с адаптером фрагментированной геномной ДНК от первого субъекта, с использованием первого адаптера, который содержит первую штрих-код-последовательность, для получения первого продукта;
лигирование с адаптером фрагментированной геномной ДНК от второго субъекта, с использованием второго адаптера, который содержит вторую штрих-код-последовательность, для получения второго продукта;
комбинирование указанных первого и второго продуктов для получения смешанной матрицы; и
выполнение способа пункта 1 с использованием указанной смешанной матрицы для получения первого и второго продукта ПЦР, каждый из которых содержит указанную штрих-код-последовательность.
i. лигирование фрагментов нуклеиновой кислоты с адаптером, который содержит сайт для праймера секвенирования и нуклеотидную последовательность, такую же, как и вторые связанные с поверхностью олигонуклеотиды,
ii. гибридизацию лигированных с адаптером геномных фрагментов с первым членом первой популяции связанных с поверхностью олигонуклеотидов,
iii. удлинение первого члена первой популяции связанных с поверхностью олигонуклеотидов, с которыми гибридизируется лигированный с адаптером фрагмент; и
iv. гибридизацию содержащего адаптер конца продукта удлинения со вторым связанным с подложкой полинуклеотидом, тем самым создавая мостик и способствуя мостиковой ПЦР.
а) субстрат, представленный твердофазной подложкой, содержащий первую популяцию связанных с поверхностью олигонуклеотидов, и вторую популяцию связанных с поверхностью олигонуклеотидов, где первая и вторая популяции связанных с поверхностью олигонуклеотидов распределены случайным образом относительно друг друга на субстрате;
б) синтетический олигонуклеотид отбора, который содержит участок, который гибридизируется с первым членом первой популяции, и участок, который содержит геномную последовательность;
в) адаптер; и
г) инструкции для выполнения способа пункта 1.

Комментарии