Улавливание и характеристика совместно локализованного хроматина - RU2478716C2
Код документа: RU2478716C2
Чертежи
















Описание
ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
Настоящее изобретение относится к анализу частоты взаимодействий двух или более нуклеотидных последовательностей в ядерном пространстве.
УРОВЕНЬ ТЕХНИКИ
Целью исследований архитектуры ядра клеток млекопитающих является понимание того, как ДНК длиной 2 метра укладывается в ядре размером 10 мкм, обеспечивая при этом точную экспрессию генов, которые определяют тип клетки, и каким образом она точно реплицируется во время каждого клеточного цикла. Значительный прогресс в данной области был достигнут в результате микроскопических исследований, которые выявили, что геномы распределены в ядерном пространстве не случайно. Например, плотно упакованный гетерохроматин расположен отдельно от более открытого эухроматина, и хромосомы занимают разные территории в ядерном пространстве. Существует сложная взаимосвязь между положением в ядре и транскрипционной активностью. Хотя транскрипция происходит во всем внутреннем пространстве ядра, активные гены, которые образуют кластеры на хромосомах, преимущественно локализованы на краю или на внешней стороне территории своей хромосомы. Отдельные гены могут перемещаться при изменениях их транскрипционного состояния, как было измерено относительно сравнительно крупных ориентиров в ядре, таких как территории хромосом, центромеры или периферия ядра. Кроме того, активно транскрибируемые гены, находящиеся на расстоянии десятков миллионов оснований друг от друга на хромосоме, могут находиться рядом в ядре, как было недавно показано посредством флуоресцентной гибридизации in situ (FISH) для локуса β-глобина и нескольких других выбранных генов. Кроме транскрипции геномная организация связана с координацией репликации, рекомбинации и вероятности транслокации локусов (что может приводить к злокачественным перерождениям) и установкой и переустановкой эпигенетических программ. На основании указанных наблюдений полагают, что архитектурная организация ДНК в клеточном ядре вносит основной вклад в функционирование генома.
Разработаны различные анализы, позволяющие проникнуть в пространственную организацию геномных локусов in vivo. Разработан один анализ, называемый «RNA-TRAP» (Carter et al. (2002) Nat. Genet. 32, 623), который заключается в направлении пероксидазы хрена (HRP) к мишени - образующимся РНК-транскриптам с последующим количественным анализом катализируемого HRP отложения биотина в области хроматина.
Другой анализ, который был разработан, назван способом улавливания конформации хромосом (chromosome conformation capture, 3C), которая является средством исследования структурной организации области генома. Способ 3C заключается в количественном ПЦР-анализе частот поперечного связывания между двумя данными рестрикционными фрагментами ДНК, что является мерой их близости в ядерном пространстве (см. фиг.1). Исходно разработанный для анализа конформации хромосом дрожжей (Dekker et al., 2002), указанный способ был адаптирован для исследования взаимосвязи между экспрессией генов и укладкой хроматина в сложных кластерах генов млекопитающих (см., например, Tolhuis et al., 2002; Palstra et al., 2003; and Drissen et al., 2004). Коротко, способ 3C заключается в поперечном сшивании клеток формальдегидом in vivo и расщеплении хроматина в ядре ферментом рестрикции с последующим лигированием фрагментов ДНК, которые были поперечно сшиты в один комплекс. Затем продукты лигирования количественно оценивают в ПЦР. Стадия ПЦР-амплификации требует наличия информации о последовательности для каждого из фрагментов ДНК, которые необходимо амплифицировать. Таким образом, способ 3C обеспечивает меру частоты взаимодействия между выбранными фрагментами ДНК.
Существует большая потребность в высокопроизводительном способе, посредством которого можно систематически проводить объективный скрининг целого генома в отношении локусов ДНК, которые контактируют друг с другом в ядерном пространстве.
В настоящем изобретении предпринята попытка усовершенствовать способ 3C.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Способ 3C, который применяют в настоящее время, дает возможность анализировать только ограниченное количество выбранных взаимодействий ДНК-ДНК вследствие ограничений стадии ПЦР-амплификации, которая требует знания информации о конкретной последовательности для каждого анализируемого фрагмента. Кроме того, отбор фрагментов рестрикции в качестве кандидатов для взаимодействий ДНК дальнего действия требует значительного количества предварительных сведений (например, положение гиперчувствительных сайтов) о представляющем интерес локусе, которые обычно не доступны. Учитывая функциональную значимость многих ДНК-ДНК-взаимодействий дальнего действия, описанных до настоящего времени, возможность случайным образом проводить скрининг элементов ДНК, которые за счет образования петли приближаются к представляющей интерес последовательности - такой как генный промотор, энхансер, инсулятор, сайленсер, начало репликации или MAR/SAR - или к представляющей интерес области генома - такой как область с высокой плотностью генов или область с низкой плотностью генов или повторяющийся элемент - может значительно облегчить картирование последовательностей в регуляторной сети.
Настоящее изобретение относится к способу 4C [т.е. улавливанию и характеристике совместно локализованного хроматина «capture and characterise co-localised chromatin), который обеспечивает высокопроизводительный анализ частоты взаимодействий двух или более нуклеотидных последовательностей в ядерном пространстве.
Способ 4C (улавливание и характеристика совместно локализованного хроматина) является модифицированным вариантом способа 3C, который обеспечивает возможность объективного широкого поиска в геноме фрагментов ДНК, которые взаимодействуют с выбранным локусом. Коротко, анализ 3C осуществляют как обычно, но минуя стадию ПЦР. Матрица 3C содержит «приманку» (например, выбранный фрагмент рестрикции, который охватывает представляющий интерес ген), лигированную со многими разными представляющими интерес нуклеотидными последовательностями (представляющими собой геномное окружение данного гена). Матрицу расщепляют другим, вторым ферментом рестрикции и лигируют. Преимущественно амплифицируют одну или несколько представляющих интерес нуклеотидных последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью, используя по меньшей мере один (предпочтительно по меньшей мере два) олигонуклеотидный праймер, при этом по меньшей мере один праймер гибридизуется с последовательностью ДНК, которая фланкирует представляющие интерес нуклеотидные последовательности. Обычно это дает картину фрагментов ПЦР, которая является высоко воспроизводимой в независимых реакциях амплификации и специфичной для данной ткани. В одном варианте используют HindIII и DpnII в качестве первого и второго ферментов рестрикции. Затем амплифицированные фрагменты можно пометить и необязательно гибридизовать с чипом, обычно против контрольного образца, содержащего геномную ДНК, расщепленную такой же комбинацией ферментов рестрикции.
В одном предпочтительном варианте осуществления настоящего изобретения лигированные фрагменты, которые расщепляют вторым ферментом рестрикции, затем повторно лигируют с образованием небольших колец ДНК.
Следовательно, способ 3C был модифицирован так, чтобы амплифицировать все представляющие интерес нуклеотидные последовательности, которые взаимодействуют с нуклеотидной последовательностью-мишенью. На практике это означает, что вместо осуществления реакции амплификации с праймерами, которые являются специфичными по отношению к фрагментам, которые хотят проанализировать, амплификацию осуществляют, используя олигонуклеотидный праймер(ры), который гибридизуется с последовательностью ДНК, которая фланкирует представляющие интерес нуклеотидные последовательности. Преимуществом является то, что 4C не смещен в сторону конструирования праймеров ПЦР, которые включены в стадию ПЦР-амплификации и поэтому могут быть использованы для поиска во всем геноме взаимодействующих элементов ДНК.
СУЩНОСТЬ АСПЕКТОВ НАСТОЯЩЕГО ИЗОБРЕТЕНИЯ
Аспекты настоящего изобретения представлены в прилагаемой формуле изобретения.
В первом аспекте предлагается способ анализа частоты взаимодействия нуклеотидной последовательности-мишени с одной или несколькими представляющими интерес нуклеотидными последовательностями (например, одним или несколькими геномными локусами), включающий стадии (a) получения образца поперечно сшитой ДНК; (b) расщепления поперечно сшитой ДНК первым ферментом рестрикции; (c) лигирования поперечно сшитых нуклеотидных последовательностей; (d) удаления поперечных сшивок; (e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции; (f) лигирования одной или нескольких последовательностей ДНК, имеющих известный состав нуклеотидов, с доступным сайтом(ами) расщепления вторым ферментом рестрикции, который фланкирует одну или несколько представляющих интерес нуклеотидных последовательностей; (g) амплификации одной или нескольких представляющих интерес нуклеотидных последовательностей с использованием по меньшей мере двух олигонуклеотидных праймеров, причем каждый праймер гибридизуется с последовательностями ДНК, которые фланкируют представляющие интерес нуклеотидные последовательности; (h) гибридизации амплифицированной последовательности(ей) с чипом; и (i) определения частоты взаимодействия между последовательностями ДНК.
Во втором аспекте предлагается способ анализа частоты взаимодействия нуклеотидной последовательности-мишени с одной или несколькими нуклеотидными последовательностями (например, одним или несколькими геномными локусами), включающий стадии (a) получения образца поперечно сшитой ДНК; (b) расщепления поперечно сшитой ДНК первым ферментом рестрикции; (c) лигирования поперечно сшитых нуклеотидных последовательностей; (d) удаления поперечных сшивок; (e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции; (f) образования кольцевых нуклеотидных последовательностей; (g) амплификации одной или нескольких нуклеотидных последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью; (h) необязательно гибридизации амплифицированных последовательностей с чипом; и (i) определения частоты взаимодействия между последовательностями ДНК.
В третьем аспекте предлагается кольцевая нуклеотидная последовательность, содержащая первую и вторую нуклеотидные последовательности, в которой концы первой и второй нуклеотидных последовательностей разделены разными сайтами узнавания ферментами рестрикции и в которой указанная первая нуклеотидная последовательность является нуклеотидной последовательностью-мишенью, а указанная вторая нуклеотидная последовательность может быть получена поперечным сшиванием геномной ДНК.
В четвертом аспекте предлагается способ получения кольцевой нуклеотидной последовательности, включающий стадии (a) получения образца поперечно сшитой ДНК; (b) расщепления поперечно сшитой ДНК первым ферментом рестрикции; (c) лигирования поперечно сшитых нуклеотидных последовательностей; (d) удаления поперечных сшивок; (e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции; и (f) образования кольцевых нуклеотидных последовательностей.
В пятом аспекте предлагается способ анализа частоты взаимодействия нуклеотидной последовательности-мишени с одной или несколькими нуклеотидными последовательностями (например, одним или несколькими геномными локусами), включающий применение кольцевой нуклеотидной последовательности.
В шестом аспекте предлагается чип с зондами, иммобилизованными на подложке, содержащий один или несколько зондов, которые гибридизуются или способны гибридизоваться с кольцевой нуклеотидной последовательностью.
В седьмом аспекте предлагается набор зондов с последовательностями, комплементарными последовательностям нуклеиновой кислоты, граничащей с каждым из первых сайтов узнавания рестрикционными ферментами первого фермента рестрикции в геномной ДНК.
В восьмом аспекте предлагается способ получения набора зондов, включающий стадии (a) идентификации каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК; (b) конструирования зондов, которые способны гибридизоваться с последовательностями, граничащими с каждым из первых сайтов узнавания ферментом рестрикции в геномной ДНК; (c) синтеза зондов; и (d) объединения зондов с образованием набора зондов или по существу набора зондов.
В девятом аспекте предлагается набор зондов или по существу набор зондов, который получен или может быть получен способом, описанным в данной публикации.
В десятом аспекте предлагается чип, содержащий матрицу зондов, или по существу набор зондов, описанных в данной публикации.
В одиннадцатом аспекте предлагается чип, содержащий набор зондов согласно настоящему описанию.
В двенадцатом аспекте предлагается способ получения чипа, включающий в себя стадию иммобилизации на твердой подложке по существу матрицы зондов или по существу набора зондов, описанных в данной публикации.
В тринадцатом аспекте предлагается способ получения чипа, включающий в себя стадию иммобилизации на твердой подложке матрицы зондов или набора зондов, описанных в данной публикации.
В четырнадцатом аспекте предлагается чип, который получен или может быть получен способом, описанным в данной публикации.
В пятнадцатом аспекте предлагается способ идентификации одного или нескольких ДНК-ДНК-взаимодействий, которые являются показателем конкретного патологического состояния, включающий в себя стадию осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения, при этом на стадии (a) образец поперечно сшитой ДНК получают из пораженной заболеванием и не пораженной заболеванием клетки, и при этом разница между частотой взаимодействия между последовательностями ДНК из пораженной заболеванием клетки и не пораженной заболеванием клетки свидетельствует о том, что ДНК-ДНК-взаимодействие является показателем конкретного патологического состояния.
В шестнадцатом аспекте предлагается способ диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия, включающий в себя стадию осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения, при этом стадия (a) заключается в получении образца поперечно сшитой ДНК от субъекта; и стадия (i) заключается в сравнении частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в непораженном контроле; при этом различие между значением, полученным для контроля, и значением, полученным для субъекта, является показателем того, что субъект имеет данное заболевание или синдром, или является показателем того, что субъект будет иметь данное заболевание или синдром.
В семнадцатом аспекте предлагается способ диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия, включающий в себя стадию осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения, при этом стадия (a) заключается в получении образца поперечно сшитой ДНК от субъекта; и при этом указанный способ включает в себя дополнительную стадию (j) идентификации одного или нескольких локусов, которые были подвергнуты перестройке в геноме, которая связана с заболеванием.
В восемнадцатом аспекте предлагается способ анализа для идентификации одного или нескольких агентов, которые модулируют ДНК-ДНК-взаимодействие, включающий в себя стадии (a) контактирования образца с одним или несколькими агентами; и (b) осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения, при этом стадия (a) заключается в получении поперечно сшитой ДНК из образца; при этом разница между (i) частотой взаимодействия между последовательностями ДНК в присутствии агента и (ii) частотой взаимодействия между последовательностями ДНК в отсутствие агента являются показателем того, что агент модулирует ДНК-ДНК-взаимодействие.
В девятнадцатом аспекте предлагается способ определения положения сбалансированного и/или несбалансированного точечного разрыва (например, в случае транслокации), включающий в себя стадию (a) осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения; и (b) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле; при этом переход от низкой к высокой частоте ДНК-ДНК-взаимодействия в образце по сравнению с контролем является показателем положения точки разрыва.
В двадцатом аспекте предлагается способ определения положения сбалансированной и/или несбалансированной инверсии, включающий в себя стадии (a) осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения; и (b) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле; при этом обратная картина частот ДНК-ДНК-взаимодействий для образца по сравнению с контролем является показателем инверсии.
В двадцать первом аспекте предлагается способ определения положения делеции, включающий в себя стадии (a) осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения; (b) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле; при этом снижение частоты ДНК-ДНК-взаимодействия в образце по сравнению с контролем является показателем делеции.
В двадцать втором аспекте предлагается способ определения положения дупликации, включающий в себя стадии (a) осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения; и (b) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле; при этом увеличение или уменьшение частоты ДНК-ДНК-взаимодействия в случае образца от субъекта по сравнению с контролем является показателем дупликации или инсерции.
В двадцать третьем аспекте предлагается агент, который получен или может быть получен с применением способа анализа, описанного в данной публикации.
В двадцать четвертом аспекте предлагается применение кольцевой нуклеотидной последовательности для идентификация одного или нескольких ДНК-ДНК-взаимодействий в образце.
В двадцать пятом аспекте предлагается применение кольцевой нуклеотидной последовательности для диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия.
В двадцать шестом аспекте предлагается применение матрицы зондов или набора зондов, описанных в данной публикации, для идентификации одного или нескольких ДНК-ДНК-взаимодействий в образце.
В двадцать седьмом аспекте предлагается применение матрицы зондов или набора зондов, описанных в данной публикации, для диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия.
В двадцать восьмом аспекте предлагается применение чипа, описанного в данной публикации, для идентификация одного или нескольких ДНК-ДНК-взаимодействий в образце.
В двадцать девятом аспекте предлагается применение чипа, описанного в данной публикации, для диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия.
В тридцатом аспекте предлагается способ, матрица зондов, набор зондов, способ, чип, способ анализа, агент или применение, которые по существу описаны в данной публикации со ссылками на примеры и фигуры.
ПРЕДПОЧТИТЕЛЬНЫЕ ВАРИАНТЫ ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
Предпочтительно реакция лигирования на стадии (f) приводит к образованию колец ДНК.
Предпочтительно нуклеотидная последовательность-мишень выбрана из группы, состоящей из геномной перестройки, промотора, энхансера, сайленсера, инсулятора, связанной с матриксом области, области регуляции локуса, транскрипционной единицы, начала репликации, горячей точки рекомбинации, точки разрыва при транслокации, центромеры, теломеры, области с высокой плотностью генов, области с низкой плотностью генов, повторяющегося элемента и сайта интеграции (вируса).
Предпочтительно нуклеотидной последовательностью-мишенью является нуклеотидная последовательность, которая связана или вызывает заболевание, или расположена на расстоянии вплоть до 15 млн.п.о. или более на линейной ДНК-матрице от локуса, который ассоциирован с заболеванием или вызывает заболевание.
Предпочтительно нуклеотидная последовательность-мишень выбрана из группы, состоящей из AML1, MLL, MYC, BCL, BCR, ABL1, IGH, LYL1, TAL1, TAL2, LMO2, TCRα/δ, TCRβ и HOX или других локусов, связанных с заболеванием, которые описаны в «Catalogue of Unbalanced Chromosome Aberrations in Man» 2nd edition. Albert Schinzel. Berlin: Walter de Gxuyter, 2001. ISBN.3-11-011607-3.
Предпочтительно первым ферментом рестрикции является фермент рестрикции, который узнает сайт узнавания, состоящий из 6-8 п.о.
Предпочтительно первый фермент рестрикции выбран из группы, состоящей из BglII, HindIII, EcoRI, BamHI, SpeI, PstI и NdeI.
Предпочтительно вторым ферментом рестрикции является фермент рестрикции, который узнает сайт узнавания нуклеотидной последовательности, состоящий из 4 или 5 п.о.
Предпочтительно второй сайт узнавания ферментом рестрикции расположен на расстоянии более чем примерно 350 п.о. от первого сайта рестрикции в нуклеотидной последовательности-мишени.
Предпочтительно нуклеотидная последовательность является меченой.
Предпочтительно последовательности зондов комплементарны последовательности нуклеиновой кислоты, граничащей с каждой стороны с каждым из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
Предпочтительно последовательности зондов комплементарны последовательности нуклеиновой кислоты, которая расположена в пределах менее чем 300 пар оснований от каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
Предпочтительно зонды комплементарны последовательности, которая расположена в пределах менее чем 300 п.о. от каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
Предпочтительно зонды комплементарны последовательности, которая расположена в пределах от 200 до 300 п.о. от каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
Предпочтительно зонды комплементарны последовательности, которая расположена в пределах от 100 до 200 п.о. или от 0 до 100 п.о. от каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
Предпочтительно два или более зондов способны гибридизоваться с последовательностью, граничащей с каждым первым сайтом узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
Предпочтительно зонды перекрываются или частично перекрываются.
Предпочтительно перекрывание составляет менее 10 нуклеотидов.
Предпочтительно последовательность зонда соответствует всей или части последовательности между каждым из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции и каждым из первых ближайших вторых сайтов узнавания рестрикционными ферментами для второго фермента рестрикции.
Предпочтительно каждый зонд является по меньшей мере 25-мером.
Предпочтительно каждый зонд является 25-60-мером.
Предпочтительно зонды являются продуктами ПЦР-амплификации.
Предпочтительно чип содержит примерно 300000-400000 зондов.
Предпочтительно чип содержит примерно 385000 или более зондов, предпочтительно примерно 750000 зондов, более предпочтительно 6×750000 зондов.
Предпочтительно чип содержит или представляет полный геном данного вида при более низком разрешении.
Предпочтительно чип содержит один из каждых 2, 3, 4, 5, 6, 7, 8, 9 или 10 зондов, которые располагаются по порядку на линейной хромосомной матрице.
Предпочтительно переход от более низкой к более высокой частоте взаимодействия является показателем положения сбалансированного и/или несбалансированного точечного разрыва.
Предпочтительно обратная картина частоты ДНК-ДНК-взаимодействия в образце от субъекта по сравнению с контролем является показателем сбалансированной и/или несбалансированной инверсии.
Предпочтительно уменьшение частоты ДНК-ДНК-взаимодействия в случае образца от субъекта по сравнению с контролем в сочетании с увеличением частоты ДНК-ДНК-взаимодействия для более отдаленных областей является показателем сбалансированной и/или несбалансированной делеции.
Предпочтительно увеличение или уменьшение частоты ДНК-ДНК-взаимодействия в случае образца от субъекта по сравнению с контролем является показателем сбалансированной и/или несбалансированной дупликации или инсерции.
Предпочтительно перед осуществлением указанного способа используют спектральное кариотипирование и/или FISH.
Предпочтительно заболевание является наследственным заболеванием.
Предпочтительно заболевание представляет собой злокачественную опухоль.
Предпочтительно две или более амплифицированных последовательностей метят по-разному.
Предпочтительно две или более амплифицированных последовательностей метят идентично в том случае, когда последовательности находятся на разных хромосомах.
Предпочтительно две или более амплифицированных последовательностей метят идентично в том случае, когда последовательности находятся на одной и той же хромосоме на расстоянии, которое является достаточно большим, чтобы было минимальное перекрывание между сигналами ДНК-ДНК-взаимодействия.
Предпочтительно диагностика или прогноз являются пренатальной диагностикой или прогнозом.
ПРЕИМУЩЕСТВА
Настоящее изобретение имеет ряд преимуществ. Указанные преимущества будут понятны из следующего описания.
В качестве примера настоящее изобретение имеет преимущество, так как помимо прочего относится к коммерчески применимым нуклеотидным последовательностям, способам, зондам и чипам.
В качестве следующего примера настоящее изобретение имеет преимущество, так как оно относится к высокопроизводительному анализу частоты взаимодействия двух или более нуклеотидных последовательностей в ядерном пространстве.
В качестве следующего примера настоящее изобретение имеет преимущество, так как при использовании обычного способа 3C каждое отдельное ДНК-ДНК-взаимодействие необходимо анализировать в отдельной особой реакции ПЦР с использованием уникальной пары праймеров. Поэтому высокопроизводительный анализ возможен только в том случае, если ПЦР автоматизирована, но расходы на такое большое количество праймеров будут слишком велики. Соответственно высокопроизводительный (широко охватывающий геном) анализ ДНК-ДНК-взаимодействий не осуществим в случае обычного способа 3C. В отличие от этого, настоящее изобретение дает возможность одновременного скрининга тысяч ДНК-ДНК-взаимодействий. Высокопроизводительный анализ ДНК-ДНК-взаимодействий согласно настоящему изобретению существенно увеличит масштаб и разрешающую способность анализа.
В качестве следующего примера настоящее изобретение имеет преимущество, так как при использовании обычного способа 3C скрининг смещен к таким последовательностям ДНК, для которых сконструированы, упорядочены и включены в анализ олигонуклеотидные праймеры. Выбор таких олигонуклеотидных праймеров обычно основан на данных, касающихся, например, положения (отдаленных) энхансеров и/или других регуляторных элементов/гиперчувствительных сайтов, которые, как полагают, будут поперечно связываться с исследуемой нуклеотидной последовательностью. Таким образом, обычный 3C смещен в сторону конструирования ПЦР-праймеров, которые включают на стадии ПЦР-амплификации, тогда как 4C не смещен и может быть использован для поиска во всем геноме в отношении взаимодействующих элементов ДНК. Это связано с тем, что амплификация поперечно сшитых последовательностей в 4C не основана на рассчитанных данных о последовательностях, которые поперечно связываются с исследуемой нуклеотидной последовательностью. Точнее, в одном варианте осуществления 4C последовательности, которые перекрестно связываются с первой нуклеотидной последовательностью (мишенью), могут быть амплифицированы с использованием ПЦР-праймеров, которые гибридизуются с данной нуклеотидной последовательностью. Таким образом, настоящее изобретение обеспечивает объективный широко охватывающий геном скрининг ДНК-ДНК-взаимодействий.
В качестве следующего примера настоящее изобретение имеет преимущество, так как использование обычного способа 3C обеспечивает возможность только избирательной амплификации одного ДНК-ДНК-взаимодействия. Это не информативно при гибридизации с чипом. Способ был усовершенствован так, что все фрагменты, которые взаимодействуют с первой нуклеотидной последовательностью (мишенью) теперь амплифицируют, например, избирательно амплифицируют.
В качестве следующего примера настоящее изобретение имеет преимущество, так как способ 4C можно применять для выявления сбалансированных или несбалансированных генетических аберраций - таких как все типы транслокаций, делеций, инверсий, дупликаций и других генетических перестроек - в нуклеиновой кислоте, например, в хромосомах. Способом 4C (в котором измеряют близость фрагментов ДНК) даже можно определять предрасположенность субъекта к возникновению некоторых транслокаций, делеций, инверсий, дупликаций и других перестроек в геноме (например, сбалансированных или несбалансированных транслокаций, делеций, инверсий, дупликаций и других перестроек в геноме). Преимущество по сравнению с имеющимся в настоящее время способом заключается в том, что он не требует знания точного положения изменения, так как разрешающая способность способа 4C является такой, что его можно применять для выявления перестроек даже тогда, когда «4C-приманка» (которую определяют по сайтам узнавания первым и вторым ферментами рестрикции, которые анализируют) расположена далеко (например, до одного миллиона оснований или еще больше) от изменения. Другое преимущество заключается в том, что способ 4C позволяет точно картировать изменения, так как его можно использовать для определения двух (первых) сайтов рестрикции, между которыми происходит изменение. Другое преимущество состоит в том, что клетки не надо культивировать перед фиксацией. Таким образом, также можно анализировать, например, твердые опухоли в отношении геномных перестроек.
В качестве следующего примера настоящее изобретение имеет преимущество, так как способом 4C также можно выявлять изменения (например, перестройки) в предзлокачественном состоянии, т.е. до того, как все клетки будут содержать указанные изменения. Таким образом, способ можно применять не только для диагностики заболевания, но также для прогнозирования заболевания.
В качестве следующего примера конструкция чипа согласно настоящему изобретению особенно предпочтительна по сравнению с существующими мозаичными геномными чипами - такими как мозаичные геномные чипы Nimblegen - так как конструкция позволяет представлять намного большую часть генома на одном чипе. В качестве примера в случае узнавания ферментом рестрикции гекса-нуклеотидной последовательности будет достаточно 3 чипа примерно с 385000 зондами каждый, чтобы охватить, например, полный геном человека или мыши. В случае узнавания ферментом рестрикции более чем 6 п.о. можно использовать один чип примерно с 385000 зондами, чтобы охватить, например, полный геном человека или мыши. Преимущества конструкции чипа заключаются в том, что (1) каждый зонд является информативным, так как анализирует независимое событие лигирования, что значительно облегчает интерпретацию результатов; и (2) можно осуществить большое представление генома в виде пятен на одном чипе, что является эффективным с точки зрения затрат.
Способ 4C предпочтительно можно использовать для тонкого картирования плохо охарактеризованных перестроек, исходно выявленных цитогенетическими способами (световая микроскопия, FISH, SKY и т.д.).
Способ 4C предпочтительно можно использовать для одновременного скрининга на одном чипе в отношении комбинаций перестроек, которые произошли вблизи нескольких локусов.
КРАТКОЕ ОПИСАНИЕ ФИГУР
Фиг.1
Принцип способа 3C
Фиг.2
(a) Принцип варианта способа 4C. Анализ 3C осуществляют как обычно, например, используя HindIII (H) в качестве фермента рестрикции. После удаления поперечных сшивок смесь ДНК будет содержать первую нуклеотидную последовательность (мишень), лигированную со многими разными фрагментами. Указанные фрагменты будут амплифицированы и помечены с использованием способов амплификации - таких как инвертированная ПЦР - например, на DpnII-кольцах, используя праймеры, специфичные для первой нуклеотидной последовательности (мишени). Меченые продукты амплификации могут быть гибридизованы с чипами, как описано в данной публикации. В качестве примеров приведены HindIII и DpnII, но также могут быть использованы другие комбинации ферментов рестрикции - например, рестриктазы, узнающие 6 или 8 нуклеотидов и рестриктазы, узнающие 4 или 5 нуклеотидов, (b) результаты ПЦР после разделения продуктов гель-электрофорезом из двух независимых образцов фетальной печени (L1, L2) и головного мозга (B1, B2), (c) схематичное представление положения зондов на микроматрице. Зонды конструировали в пределах 100 п.о. HindIII-сайтов. Таким образом, каждый зонд позволяет проанализировать одного возможного партнера при лигировании.
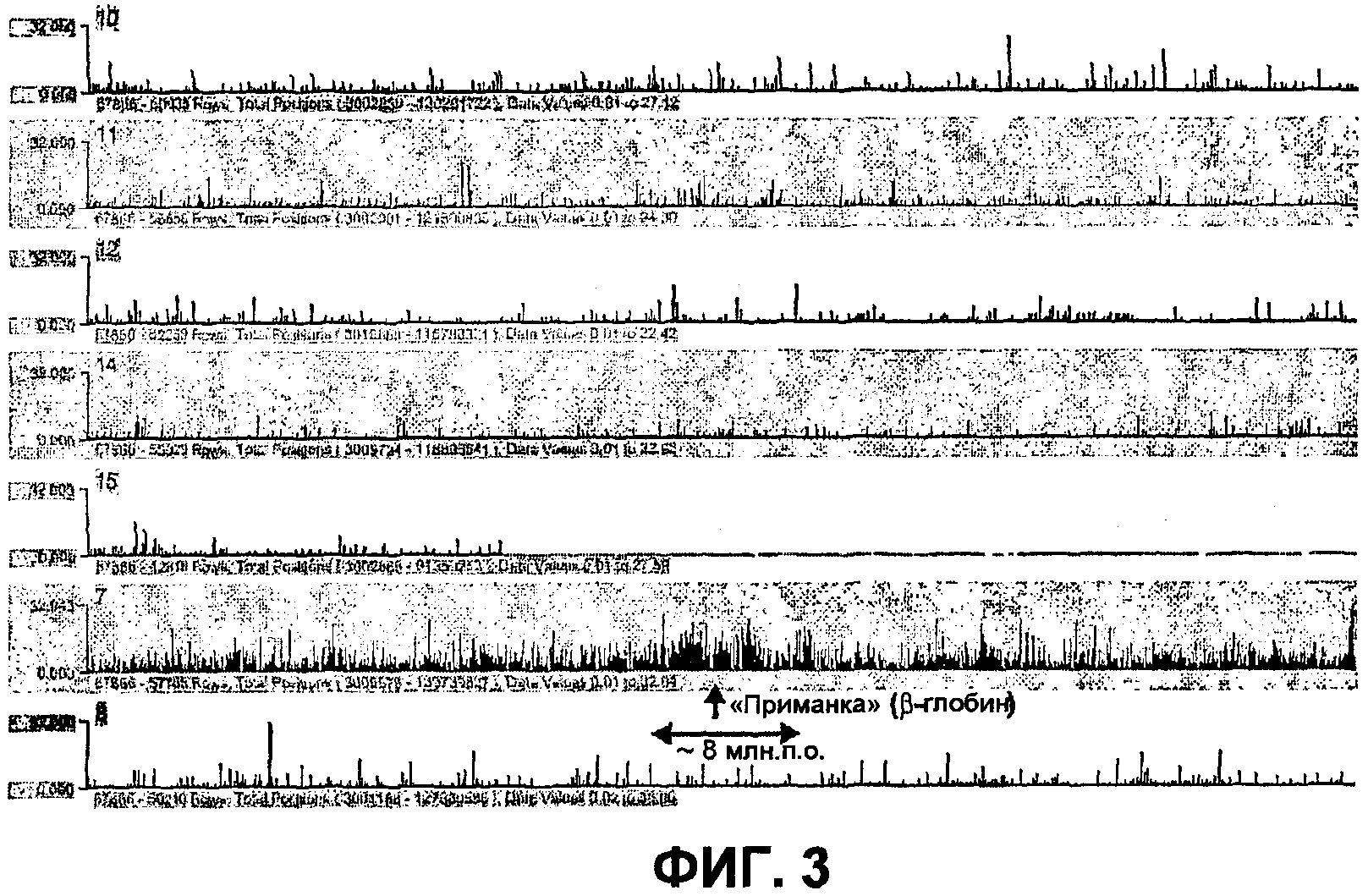
Фиг.3
Способ 4C выявляет геномное окружение гена β-глобина (хромосома 7). Показаны необработанные отношения (сигналы 4C для HS2-β-глобина, деленные на сигналы, полученные для контрольного образца) для зондов, локализованных в областях генома размером ~35 млн.п.о. в хромосомах мыши 10, 11, 12, 14, 15, 7 и 8 (сверху вниз; показанные области находятся на одинаковом расстоянии от каждой соответствующей центромеры). Следует обратить внимание на большой кластер сильных сигналов вокруг «приманки» (глобина) в хромосоме 7 (ряд 6), который показывает, что способ 4C выявляет геномные фрагменты, находящиеся вблизи линейной хромосомной матрицы (в соответствии с тем фактом, что частоты взаимодействия обратно пропорциональны расстоянию между сайтами в геноме). Следует обратить внимание, что область, связанная в цис-положении вокруг «приманки», где наблюдаются высокие интенсивности сигнала, является большой (>5 млн.п.о.), свидетельствуя, например, что транслокации могут быть выявлены даже в случае «приманок», находящихся на расстоянии более 1 млн.п.о. от точки разрыва.
Фиг.4
Способ 4C выявляет геномное окружение Rad23A (хромосома 8). Показаны необработанные отношения (сигналы 4C для Rad23A, деленные на сигнал, полученный для контрольного образца) для зондов, локализованных в геномных областях ~15 млн.п.о. или более в хромосомах мыши 10, 11, 12, 14, 15, 7 и 8 (сверху вниз; показанные области находятся на одинаковом расстоянии от каждой соответствующей центромеры). Следует обратить внимание на большой кластер сильных сигналов вокруг «приманки» (Rad23A) в хромосоме 8 (ряд 7), который показывает, что способ 4C выявляет геномные фрагменты, находящиеся вблизи линейной хромосомной матрицы (в соответствии с тем фактом, что частоты взаимодействия обратно пропорциональны расстоянию между сайтами в геноме). Следует обратить внимание, что область, связанная в цис-положении вокруг «приманки», где наблюдаются высокие интенсивности сигнала, является большой (>5 млн.п.о.), свидетельствуя, например, что транслокации могут быть выявлены даже в случае «приманок», находящихся на расстоянии более 1 млн.п.о. от точки разрыва.
Фиг.5
4C-взаимодействия β-глобина в хромосоме 7 (~135 млн.п.о.) для транскрибирующей ткани (фетальная печень) и нетранскрибирующей ткани (фетальный головной мозг) (анализируемые способом скользящих средних). Следует обратить внимание, что взаимодействия дальнего действия с β-глобином в разных тканях отличаются (вероятно в зависимости от транскрипционного статуса гена). Независимо от ткани сильные сигналы 4C определяют границы большой области (>5 млн.п.о.) вокруг приманки.
Фиг.6
Uros и Eraf взаимодействуют с β-глобином в клетках фетальной печени. Способ 4C показал, что два гена, Eraf и Uros, взаимодействуют через >30 млн.п.о. с локусом β-глобина, расположенным на расстоянии ~30 млн.п.о. Два указанных взаимодействия ранее обнаружены другим способом (флуоресцентной гибридизацией in situ), который описан в Osborne et al., Nature Genetics 36, 1065 (2004). Данный пример показывает, что взаимодействия дальнего действия, выявленные способом 4C, могут быть подтверждены FISH и правильно отражают близость в ядре.
Фиг.7
Способ 4C точно идентифицирует переходы между неродственными геномными областями, которые связаны в цис-положении. Для данных экспериментов использовали трансгенных мышей, которые содержат кассету регуляторной области локуса (LCR) β-глобина человека (~20 т.п.о.), встроенную (посредством гомологичной рекомбинации) в локус Rad23A в хромосоме 8 мыши. Способ 4C осуществляли на фетальной печени E14.5 трансгенных мышей, которые были гомозиготными по данной инсерции. HindIII-фрагмент в интегрирующей кассете (HS2) использовали в качестве «4C-приманки». Данные показывают, что способ 4C точно определяет оба конца трансгенной кассеты (нижний ряд: 4C-сигналы давали только зонды в LCR человека (~20 т.п.о.), но не зонды в остальной части последовательности β-глобина человека ~380 т.п.о.) и четко выявляет положение интеграции в мышиной хромосоме 8 (верхняя панель: следует сравнить сигналы на хромосоме 8 (в отношении положения интеграции, см. стрелку) с сигналами на 6 других мышиных хромосомах) (изображены полные хромосомы). Данный пример показывает, что способ 4C можно применять для выявления положения в геноме эктопически интегрированных фрагментов ДНК (вируса, трансгена и т.д.). Пример показывает, что можно точно идентифицировать перемещения между неродственными областями генома, которые связаны в цис-положении, что может быть использовано для идентификации точек разрыва в геноме и партнеров при транслокации.
Фиг.8
Способ 4C дает воспроизводимые данные, так как профили для HS2 и β-глобина очень сходны. Проводили четыре биологически независимых эксперимента 4C на фетальной печени E14.5 с использованием либо гена β-глобина β-основного (2 верхних ряда), либо β-глобина-HS2 (два нижних ряда) в качестве приманки. Указанные приманки находятся на расстоянии ~40 т.п.о. друг от друга на линейной хромосомной матрице, но, как было показано ранее, расположены рядом в ядерном пространстве (Tolhuis et al., Molecular Cell 10, 1453 (2002)). Изображена область ~5 млн.п.о. на мышиной хромосоме 7, которая находится на расстоянии 20-20 млн.п.о. от локуса β-глобина. Данные показывают высокую воспроизводимость в независимых экспериментах и показывают, что два фрагмента, близко расположенные в ядерном пространстве, имеют общих взаимодействующих партнеров, локализованных в другом месте генома.
Фиг.9
Способ 4C применяли для измерения частот ДНК-ДНК-взаимодействия с последовательностью X (на хромосоме A) в клетках здорового человека (вверху) и пациента с транслокацией (A;B) (внизу). Интенсивности сигналов, представляющих частоты ДНК-ДНК-взаимодействий (ось Y) отложены на графике для зондов, расположенных по порядку на линейных хромосомных матрицах (ось X). В нормальных клетках выявлены частые ДНК-ДНК-взаимодействия на хромосоме A вокруг последовательности X. В клетках пациента наблюдается 50% снижение частоты взаимодействия для зондов на хромосоме A, расположенных с другой стороны от точки разрыва (BP) (сравните серую кривую (пациент) с черной кривой (здоровый человек). Кроме того, транслокация переносит часть хромосомы B в тесную физическую близость с последовательностью X, и частые ДНК-ДНК-взаимодействия после этого наблюдаются для указанной области на хромосоме B. Внезапный переход от низкой к высокой частоте взаимодействия на данной хромосоме служит признаком положения точки разрыва.
Фиг.10
(Сбалансированная) инверсия (инверсии) может быть выявлена способом 4C. Обратные картины частот ДНК-ДНК-взаимодействия (измеренных способом 4C в виде интенсивностей сигнала гибридизации) наблюдаются у больного (сплошная кривая) по сравнению со здоровым (пунктирная кривая) субъектом, что свидетельствует о наличии и размере инверсии.
Фиг.11
Выявление гетерозиготной делеции (делеций) способом 4C. Зонды с уменьшенной частотой ДНК-ДНК-взаимодействий (измеренной способом 4C в виде интенсивностей сигнала гибридизации) у больных (серая кривая) по сравнению со здоровыми (черная кривая) субъектами, показывают положение и размер делетированной области. Остаточные сигналы гибридизации в делетированной области больного субъекта возникают из-за интактного аллеля (гетерозиготная делеция). Делеция обычно сопровождается увеличением интенсивностей сигнала для зондов, расположенных непосредственно за пределами делетированной области (следует обратить внимание, что серая кривая проходит выше черной кривой справа от делеции), так как указанные области становятся физически ближе к последовательности 4C (к приманке).
Фиг.12
Дупликация, выявленная способом 4C. Зонды с увеличенными сигналами гибридизации у пациента (сплошная кривая) по сравнению со здоровым человеком (пунктирная кривая) указывают положение и размер дупликации. Дупликация, которую выявляют способом 4C, обычно сопровождается уменьшенными сигналами гибридизации у больных по сравнению со здоровыми субъектами для зондов вне дуплицированной области (дупликация увеличивает расстояние, отделяющее их сайт в геноме от последовательности 4C).
Фиг.13
Взаимодействия дальнего действия с β-глобином, выявленные способом 4C. a) Необработанные отношения сигналов гибридизации 4C по сравнению с контролем, показывающие взаимодействия β-глобина HS2 с хромосомой 7 и двумя неродственными хромосомами (8 и 14). b-c) Необработанные данные для двух независимых образцов фетальной печени (сверху, красным) и фетального головного мозга (снизу, синим) отложены на графике вдоль двух разных областей длиной 1-2 млн.п.о. на хромосоме 7. Высоко воспроизводимые кластеры взаимодействий наблюдаются либо в двух образца фетальной печени (b), либо двух образцах головного мозга (c). d-e) Данные скользящих средних для тех же областей. Уровень ложных обнаружений устанавливали 5% (пунктирная линия). f) Схематичное представление областей взаимодействия с активным (фетальная печень, вверху) и неактивным (фетальный головной мозг, внизу) β-глобином на хромосоме 7.
Фиг.14
Активный и неактивный β-глобин взаимодействуют с активными и неактивными областями хромосомы, соответственно. a) Сравнение между взаимодействиями дальнего действия β-глобина в фетальной печени (скользящие средние 4C, вверху), анализом экспрессии на микроматрице в фетальной печени (log-масштаб, в середине) и положением генов (внизу), отложенным на графике вдоль области длиной 4 млн.п.о., которая содержит ген Uros (на расстоянии ~30 млн.п.о. от β-глобина), показывающие, что активный β-глобин преимущественно взаимодействует с другими активно транскрибируемыми генами. b) Такое же сравнение в фетальном головном мозге вокруг кластера генов OR, расположенного на расстоянии ~38 млн.п.о. от глобина, показывающее, что неактивный β-глобин преимущественно взаимодействует с неактивными областями. c) Характеристика областей, взаимодействующих с β-глобином в фетальной печени (слева) и головном мозге (справа) с точки зрения содержания и активности генов.
Фиг.15
Повсеместно экспрессируемый Rad23A взаимодействует с очень сходными активными областями в фетальной печени и головном мозге. a) Схематичное представление областей на хромосоме 8, взаимодействующих с активным Rad23A в фетальной печени (вверху, красным) и головном мозге (внизу, синим). b) Сравнение между взаимодействиями дальнего действия Rad23A (скользящие средние 4C) и анализом экспрессии на микроматрице (log-масштаб) в фетальной печени (две панели вверху), взаимодействиями дальнего действия Rad23A (скользящие средние 4C) и анализом экспрессии на микроматрице (log-масштаб) в фетальном головном мозге (панели 3 и 4) и положением генов (нижняя панель), отложенным на графике вдоль области длиной 3 млн.п.о. на хромосоме 8. c) Характеристика областей, взаимодействующих с Rad23A в фетальной печени (слева) и головном мозге (справа) с точки зрения содержания и активности генов.
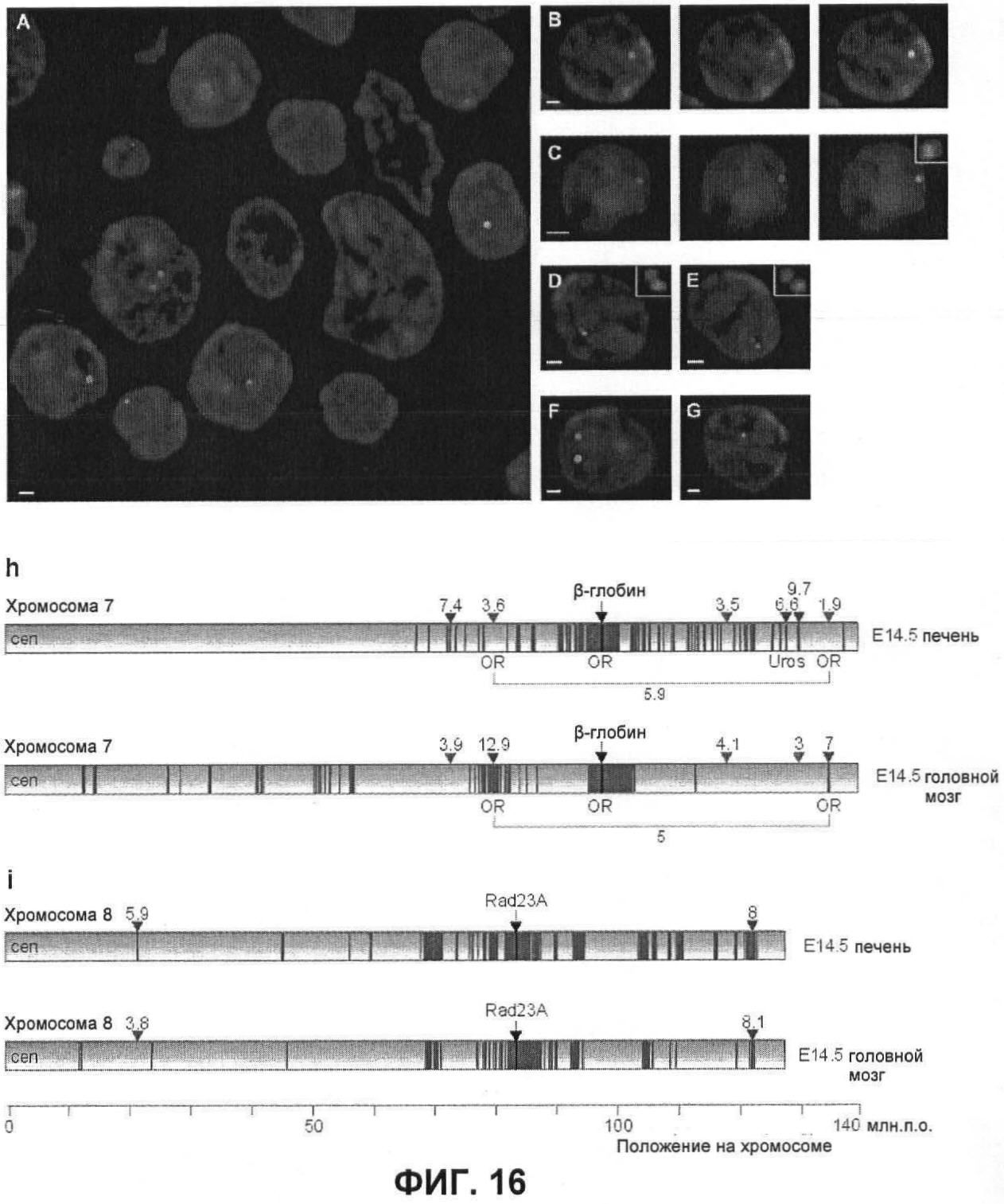
Фиг.16
Крио-FISH подтверждает, что способ 4C точно идентифицирует взаимодействующие области. a) Пример части (200 нм) среза замороженной ткани, показывающий более 10 ядер, некоторые из которых содержат локус β-глобина (зеленый) и/или Uros (красный). Из-за того, что получали срезы, многие ядра не содержат сигналов указанных двух локусов. b-d) Примеры полностью (b) и частично (c) перекрывающихся сигналов и контактирующих сигналов (d), все из которых считались позитивными сигналами взаимодействия. e-g) примеры ядер, содержащих неконтактирующие аллели (e-f) и ядра, содержащего только β-глобин (g), все из которых считали негативными в отношении взаимодействия. h-i) Схематичное представление результатов крио-FISH. Проценты взаимодействия с β-глобином (h) и Rad23A (i) указаны над хромосомами для областей, идентифицированных как позитивные (красная стрелка) и идентифицированных как негативные (синяя стрелка) способом 4C. Одни и те же BAC использовали для двух тканей. Частоты взаимодействия, измеренные с использованием крио-FISH, между двумя отдаленными кластерами генов OR в фетальной печени и головном мозге, указаны под хромосомами.
Фиг.17
Анализ 4C HS2 и β-основного дает очень сходные результаты. (a) Необработанные данные 4C для четырех независимых образцов печени E14.5 показывают очень сходную картину взаимодействия с HS2 (вверху) и β-основным (внизу). (b) Существует большое перекрывание между зондами, оцениваемыми как позитивные в отношении взаимодействия в эксперименте с HS-2, и зондами, которые оценивали как позитивные в отношении взаимодействия в эксперименте с β-основным.
Фиг.18
Сравнение между взаимодействиями в цис- и транс-положениях. (a) Необработанные данные 4C для двух независимых экспериментов, показывающие взаимодействия β-глобина с областью, идентифицированной как позитивная в цис-положении (хромосома 7, вверху), и областью в транс-положении, содержащей локус α-глобина (хромосома 11, внизу). (b) Необработанные данные 4C для двух независимых экспериментов, показывающие взаимодействия Rad23A с областью, идентифицированной как позитивная в цис-положении (хромосома 8, вверху), и областью в транс-положении, которая выходит на первое место при оценке согласно наибольшему значению скользящих средних. Ни одна из областей в транс-положении не удовлетворяла жестким условиям, которые позволяют идентифицировать далеко расположенные взаимодействующие области в цис-положении.
Фиг.19
Области, которые взаимодействуют с β-глобином, также часто контактируют друг с другом. Две области (находящиеся на расстоянии почти 60 млн.п.о. друг от друга), содержащие активно транскрибируемые гены и идентифицированные способом 4C как взаимодействующие с β-глобином в фетальной печени, имели частоту совместной локализации, как показано в крио-FISH, составляющую 5,5%, что значительно выше, чем фоновая частота совместной локализации.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
СПОСОБ 3C
Способ 3C подробно описан в Dekker et al. (2002), Tolhuis et al. (2002), Palstra et al. (2003), Splinter et al. (2004) и Drissen et al. (2004). Коротко, 3C осуществляют посредством расщепления поперечно сшитой ДНК первым фермент рестрикции с последующим лигированием при очень низких концентрациях ДНК. В указанных условиях преимущественно происходит лигирование поперечно сшитых фрагментов, которое является внутримолекулярным, по сравнению с лигированием случайных фрагментов, которое является межмолекулярным. Поперечные сшивки затем снова удаляют и отдельные продукты лигирования выявляют и количественно оценивают в полимеразной цепной реакции (ПЦР), используя специфичные по отношению к локусу праймеры. Частоту поперечного сшивания (X) двух специфичных локусов определяют в реакциях количественной ПЦР, используя контрольные и поперечно сшитые матрицы, и X выражают в виде отношения количества продукта, полученного с использованием поперечно сшитой матрицы, и продукта, полученного с использованием контрольной матрицы.
Согласно настоящему изобретению матрицу 3C получают, используя способы, описанные в Splinter et al., (2004) Methods Enzymol. 375, 493-507 (т.е. фиксацию формальдегидом, расщепление (первым) ферментом рестрикции, повторное лигирование поперечно сшитых фрагментов ДНК и очистку ДНК). Коротко, образец - такой как клетки, ткани и ядра - фиксируют, используя поперечно сшивающий агент - такой как формальдегид. Затем осуществляют расщепление первым ферментом рестрикции, так что расщепляют ДНК в ситуации поперечно сшитого ядра. Затем осуществляют внутримолекулярное лигирование при низких концентрациях ДНК (например, около 3,7 нг/мкл), что создает преимущества для лигирования между поперечно сшитыми фрагментами ДНК (т.е. для внутримолекулярного лигирования) по сравнению с лигированием между поперечно не сшитыми фрагментами ДНК (т.е. для межмолекулярного или случайного лигирования). Затем поперечные сшивки удаляют, и ДНК может быть очищена. Матрица 3C, которую получают, содержит фрагменты рестрикции, которые лигированы, так как они исходно находятся близко друг к другу в ядерном пространстве.
Так как первый фермент рестрикции используют для расщепления ДНК перед стадией внутримолекулярного лигирования, то сайт узнавания ферментом для первого фермента рестрикции будет разделять первую нуклеотидную последовательность (мишень) и нуклеотидную последовательность, которая была лигирована. Соответственно, первый сайт узнавания расположен между первой нуклеотидной последовательностью (мишенью) и лигированной нуклеотидной последовательностью (т.е. лигированной второй последовательностью).
НУКЛЕОТИДНАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
Настоящее изобретение относится к применению нуклеотидных последовательностей (например, матриц 3C, матриц 4C, ДНК-матриц, матриц для амплификации, фрагментов ДНК и геномных ДНК), которые могут быть доступны в базах данных.
Нуклеотидная последовательность может представлять собой ДНК или РНК геномного, синтетического или рекомбинантного происхождения, например кДНК. Например, рекомбинантные нуклеотидные последовательности могут быть получены с использованием способов ПЦР-клонирования. Способы будут заключаться в получении пары праймеров, фланкирующих область последовательности, которую требуется клонировать, приведении праймеров в контакт с мРНК или кДНК, полученной, например, из клеток млекопитающих (например, клеток животных или человека) клеток других организмов, отличных от млекопитающих, осуществлении полимеразной цепной реакции (ПЦР) в условиях, которые вызывают амплификацию требуемой области, выделении амплифицированного фрагмента (например, очистке реакционной смеси в агарозном геле) и извлечении амплифицированной ДНК. Праймеры могут быть сконструированы так, чтобы они содержали подходящие сайты узнавания ферментами рестрикции для того, чтобы амплифицированную ДНК можно было клонировать в подходящем клонирующем векторе.
Нуклеотидная последовательность может быть двунитевой или однонитевой, представляющей собой либо смысловую, либо антисмысловую нить, либо их комбинации.
Для некоторых аспектов предпочтительно, чтобы нуклеотидная последовательность представляла собой однонитевую ДНК - такую как однонитевые праймеры и зонды.
Для некоторых аспектов предпочтительно, чтобы нуклеотидная последовательность представляла собой двунитевую ДНК - такую как двунитевые матрицы 3C и 4C.
Для некоторых аспектов предпочтительно, чтобы нуклеотидная последовательность представляла собой геномную ДНК - такую как один или несколько геномных локусов.
Для некоторых аспектов предпочтительно, чтобы нуклеотидная последовательность представляла собой хромосомную ДНК.
Нуклеотидная последовательность может содержать первую нуклеотидную последовательность (мишень) и/или вторую нуклеотидную последовательность.
Первый и второй сайты узнавания ферментами рестрикции будут отличаться друг от друга и обычно будут встречаться только один раз в нуклеотидной последовательности.
В одном аспекте предлагается кольцевая нуклеотидная последовательность, содержащая первую нуклеотидная последовательность и (например, лигированную) вторую нуклеотидную последовательность, которые отделены друг от друга (например, разделены или разделены на части) первым и вторым сайтами узнавания ферментами рестрикции, при этом указанная первая нуклеотидная последовательность представляет собой нуклеотидную последовательность-мишень, а указанную вторую нуклеотидную последовательность можно получить поперечным сшиванием геномной ДНК (например, in vivo или in vitro). Первый и второй сайты узнавания ферментами рестрикции будут отличаться друг от друга и обычно будут встречаться только один раз в нуклеотидной последовательности.
В следующем аспекте предлагается кольцевая нуклеотидная последовательность, содержащая первую нуклеотидную последовательность и (например, лигированную) вторую нуклеотидную последовательность, которые отделены друг от друга (например, разделены или разделены на части) первым и вторым сайтами узнавания ферментами рестрикции, при этом указанная первая нуклеотидная последовательность представляет собой нуклеотидную последовательность-мишень, и указанные первая и вторая нуклеотидные последовательности можно получить способом, включающим в себя стадии (a) поперечного сшивания геномной ДНК (например, in vivo или in vitro); (b) расщепления поперечно сшитой ДНК первым ферментом рестрикции; (c) лигирования поперечно сшитых нуклеотидных последовательностей; (d) удаления поперечных сшивок; и (e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции, чтобы получить кольцевые нуклеотидные последовательности.
Предпочтительно вторая нуклеотидная последовательность делит на части (например, делит пополам) первую нуклеотидную последовательность (мишень). Таким образом, нуклеотидная последовательность содержит вторую нуклеотидную последовательность, которая делит первую нуклеотидную последовательность (мишень) на две части или два фрагмента - например, на две части или два фрагмента примерно одинакового размера. Обычно части или фрагменты будут иметь длину по меньшей мере примерно 16 нуклеотидов.
ПЕРВАЯ НУКЛЕОТИДНАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
Первая нуклеотидная последовательность представляет собой нуклеотидную последовательность-мишень.
В используемом в данном описании смысле термин «нуклеотидная последовательность-мишень» относится к последовательности, которую используют в качестве последовательности-приманки, чтобы идентифицировать одну или несколько последовательностей, с которыми она образует поперечные сшивки (например, одну или несколько представляющих интерес нуклеотидных последовательностей или одну или несколько последовательностей с неизвестным составом нуклеотидов в последовательности).
Нуклеотидная последовательность-мишень является известной последовательностью.
Поперечные сшивки являются показателем того, что нуклеотидная последовательность-мишень и поперечно сшитая с ней последовательность исходно находились рядом в ядерном пространстве. Определяя частоту, с которой последовательности располагаются рядом друг с другом, можно, например, понять конформацию хромосом и областей хромосом в пространстве ядра (например, in vivo или in vitro). Кроме того, можно понять сложную структурную организацию в геноме, например, когда энхансеры или другие элементы регуляции транскрипции взаимодействуют с отдаленными промоторами, расположенными в цис-положении или даже в транс-положении. Кроме того, можно понять положение данной области генома относительно нуклеотидных последовательностей, имеющихся в той же хромосоме (в цис-положении), а также нуклеотидных последовательностей в других хромосомах (в транс-положении). Таким образом, можно картировать нуклеотидные последовательности на разных хромосомах, которые часто имеют общие места в ядерном пространстве. Кроме того, можно выявлять даже сбалансированные и/или несбалансированные генетические аберрации - такие как сбалансированные и/или несбалансированные транслокации, делеции, инверсии, дупликации и другие геномные перестройки (например, делеции или транслокации в одной или нескольких хромосомах). В этом отношении, генетические аберрации приводят к изменениям ДНК-ДНК-взаимодействий, которые могут быть определены в положении, в котором происходит изменение.
Первая нуклеотидная последовательность (мишень) согласно настоящему изобретению может представлять собой любую последовательность, в которой требуется определить частоту взаимодействий в ядерном пространстве с одной или несколькими другими последовательностями.
В одном варианте первая нуклеотидная последовательность (мишень) будет иметь длину примерно более 350 п.о., так как второй фермент рестрикции выбирают так, чтобы он разрезал первую нуклеотидная последовательность (мишень) на расстоянии примерно 350 п.о. или более от первого сайта рестрикции. Это может минимизировать искажения при образовании кольца вследствие топологических ограничений (Rippe et al. (2001) Trends in Biochem. Sciences 26, 733-40).
Соответственно, первая нуклеотидная последовательность (мишень) после амплификации содержит по меньшей мере примерно 32 п.о. вследствие того факта, что минимальная длина по меньшей мере двух праймеров амплификации, используемых для того, чтобы амплифицировать вторую нуклеотидную последовательность, составляет примерно 16 оснований в каждом случае.
В предпочтительном варианте первая нуклеотидная последовательность (мишень) может содержать полностью или частично (например, фрагмент) или располагаться рядом (например, в непосредственной близости), с промотором, энхансером, сайленсером, инсулятором, областью связывания с матриксом, областью регуляции локуса, транскрипционной единицей, началом репликации, горячей точкой рекомбинации, точкой разрыва при транслокации, центромерой, теломерой, областью с высокой плотностью генов, областью с низкой плотностью генов, повторяющимся элементом, сайтом интеграции (вируса), нуклеотидной последовательностью, делеции и/или мутации в которой связаны с эффектом (например, заболеванием, физиологическим, функциональным или структурным эффектом - таким как SNP (полиморфизм одиночных нуклеотидов), или нуклеотидной последовательностью(ями), содержащей такие делеции и/или мутации, или любой последовательностью, в которой требуется определить частоту взаимодействий в ядерном пространстве с другими последовательностями.
Как указано выше, первая нуклеотидная последовательность (мишень) может содержать полностью или частично (например, фрагмент) или располагаться рядом (например, в непосредственной близости) с нуклеотидной последовательностью, генетические аберрации в которой - такие как делеции и/или мутации - связаны с эффектом (например, заболеванием). Поэтому согласно данному варианту осуществления изобретения первая нуклеотидная последовательность (мишень) может представлять собой нуклеотидную последовательность (например, ген или локус), граничащую с геномной областью (на физической матрице ДНК) или находящуюся в геномной области, изменения в которой связаны или коррелируют с заболеванием - таким как наследственное или врожденное заболевание. Другими словами, первая нуклеотидная последовательность (мишень) может представлять собой или может быть выбрана на основе связи с клиническим фенотипом. В предпочтительном варианте изменения представляют собой изменения в одной или нескольких хромосомах, и заболевание может является, например, следствием одной или нескольких делеций, одной или нескольких транслокаций, одной или нескольких дупликаций и/или одной или нескольких инверсий и т.д. в такой последовательности. Неограничивающими примерами таких генов/локусов являются AML1, MLL, MYC, BCL, BCR, ABL1, локусы иммуноглобулинов, LYL1, TAL1, TAL2, LMO2, TCRα/δ, TCRβ, HOX и другие локусы при различных лимфобластных лейкозах.
Другие примеры описаны в электронных базах данных, таких как
http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=cancerchromosomes
http://cgap.nci.nih.gov/Chromosomes/Mitelman
http://www.progenetix.net/progenetix/P 14603437/ideogram.html
http://www.changbioscience.com/cytogenetics/cyto1.pl?query=47,xy
http://www.possum.net.au/
http://www.lmdatabases.com/
http://www.wiley.com/legacy/products/subject/life/borgaonkar/index.html
http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=OMIM
http://www.sanger.ac.uk/PostGenomics/decipher/
http://agserver01.azn.nl:8080/ecaruca/ecaruca.jsp
Другие примеры описаны в «Catalogue of Unbalanced Chromosome Aberrations in Man» 2nd edition. Albert Schinzel. Berlin: Walter de Gruyter, 2001. ISBN 3-11-011607-3.
В одном варианте термин «граничащая» означает «непосредственно граничащая», так что между расположенными рядом последовательностями нет промежуточных нуклеотидов.
В другом варианте термин «граничащая» в контексте последовательности нуклеиновой кислоты и первого сайта узнавания ферментом рестрикции означает «непосредственно граничащая», так что между последовательностью нуклеиновой кислоты и первым сайтом узнавания ферментом рестрикции нет промежуточных нуклеотидов.
ВТОРАЯ НУКЛЕОТИДНАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
Вторая нуклеотидная последовательность может быть получена, ее получают, идентифицируют или могут идентифицировать с помощью поперечного сшивания геномной ДНК (например, in vivo или in vitro).
Вторая нуклеотидная последовательность (например, представляющая интерес нуклеотидная последовательность) становится лигированной с первой нуклеотидной последовательностью (мишенью) после обработки образца поперечно сшивающим агентом и расщепления/лигирования поперечно сшитых фрагментов ДНК. Такие последовательности поперечно сшиваются с первой нуклеотидной последовательностью (мишенью), так как они исходно находятся рядом в ядерном пространстве, и подвергаются лигированию с первой нуклеотидной последовательностью (мишенью), так как условия лигирования благоприятствуют лигированию между поперечно сшитыми фрагментами ДНК (внутримолекулярному) по сравнению со случайными событиями лигирования.
Заболевания, основанные на изменениях - таких как транслокации, делеции, инверсии, дупликации и другие геномные перестройки, - обычно вызваны аномальными ДНК-ДНК-взаимодействиями. Способ 4C измеряет частоту ДНК-ДНК-взаимодействий, которая главным образом является функцией расстояния между сайтами в геноме, т.е. частота ДНК-ДНК-взаимодействий обратно пропорциональна линейному расстоянию (в тысячах оснований) между двумя локусами ДНК, присутствующими в одной и той же физической матрице ДНК (Dekker et al., 2002). Таким образом, изменение(ия), которое приводит к образованию новых и/или физически отличающихся матриц ДНК, сопровождается измененными ДНК-ДНК-взаимодействиями и может быть измерено способом 4C.
Соответственно, вторая нуклеотидная последовательность содержит по меньшей мере 40 пар оснований.
Поперечно сшивающие агенты - такие как формальдегид - могут быть использованы для поперечного сшивания белков с другими соседними белками и нуклеиновой кислотой. Таким образом, две или более нуклеотидных последовательностей могут быть поперечно сшиты только посредством белков, связанных с (одной из) указанными нуклеотидными последовательностями. Согласно настоящему изобретению также могут быть использованы другие поперечно сшивающие агенты, отличные от формальдегида, включая такие поперечно сшивающие агенты, которые непосредственно поперечно сшивают нуклеотидные последовательности. Примеры агентов, которые поперечно сшивают ДНК, включают, без ограничения, УФ-излучение, митомицин C, азотистый иприт, мелфалан, диэпоксид 1,3-бутадиена, цис-диаминдихлорплатину (II) и циклофосфамид. Соответственно, поперечно сшивающий агент будет образовывать поперечные сшивки, которые образуют мостики в случае относительно коротких расстояний - например, около 2Ǻ - таким образом отбирая близкие взаимодействия, которые могут быть обратимы.
Поперечное сшивание может быть осуществлено, например, посредством инкубации клеток в 2% формальдегиде при комнатной температуре - например, посредством инкубации 1×107 клеток в 10 мл DMEM - 10% FCS с добавлением 2% формальдегида в течение 10 мин при комнатной температуре.
ПЕРВЫЙ ФЕРМЕНТ РЕСТРИКЦИИ
В используемом в данном описании смысле термин «первый фермент рестрикции» относится к первому ферменту рестрикции, который используют для расщепления поперечно сшитой ДНК.
Первый фермент рестрикции будет выбран в зависимости от типа анализируемой последовательности-мишени (например, локуса). Желательно, чтобы были осуществлены предварительные эксперименты, чтобы оптимизировать условия расщепления.
Первый фермент рестрикции может быть выбран из ферментов рестрикции, узнающих последовательности по меньшей мере из 6 п.о. или более в ДНК.
Ферменты рестрикции, которые узнают последовательности ДНК длиной 6 п.о., включают, без ограничения, AclI, HindIII, SspI, BspLU11I, AgeI, MluI, SpeI, BglII, Eco47III, StuI, SсaI, ClaI, AvaIII, VspI, MfeI, PmaCI, PvuII, NdeI, NcoI, SmaI, SacII, AvrII, PvuI, XmaIII, SplI, XhoI, PstI, AflII, EcoRI, AatII, SacI, EcoRV, SphI, NaeI, BsePI, NheI, BamHI, NarI, ApaI, KpnI, SnaI, SalI, ApaLI, HpaI, SnaBI, BspHI, BspMII, NruI, XbaI, BclI, MstI, BalI, Bsp1407I, PsiI, AsuII и AhaIII.
Ферменты рестрикции, которые узнают последовательности ДНК длиной более 6 п.о., включают, без ограничения, BbvC I, AscI, AsiS I, Fse I, Not I, Pac I, Pme I, Sbf I, SgrA I, Swa I, Sap I, Cci NI, FspA I, Mss I, Sgf I, Smi I, Srf I и Sse8387 I.
Для некоторых аспектов настоящего изобретения в случае ферментов рестрикции, узнающих последовательности длиной 6 п.о., предпочтительными являются BglII, HindIII или EcoRI.
Термин «первый сайт узнавания ферментом рестрикции» относится к сайту в нуклеотидной последовательности, который распознается и расщепляется первым ферментом рестрикции.
ВТОРОЙ ФЕРМЕНТ РЕСТРИКЦИИ
В используемом в данном описании смысле термин «второй фермент рестрикции» относится ко второму ферменту рестрикции, который используют после расщепления первым ферментом рестрикции, лигирования поперечно сшитой ДНК удаления поперечных сшивок и (необязательно) очистки ДНК. В одном варианте второй фермент рестрикции используют для получения заданных концов ДНК в представляющих интерес нуклеотидных последовательностях, которые обеспечивают возможность лигирования последовательностей с известным нуклеотидным составом со вторыми сайтами узнавания ферментами рестрикции, которые фланкируют представляющие интерес нуклеотидные последовательности.
В одном варианте лигирование последовательностей с известным нуклеотидным составом со вторыми сайтами узнавания ферментом рестрикции, которые фланкируют (например, находятся с каждой стороны и на каждом конце) представляющие интерес нуклеотидные последовательности, заключается в лигировании в условиях разбавления, чтобы создать преимущества для внутримолекулярного лигирования между вторыми сайтами узнавания ферментом рестрикции, которые фланкируют нуклеотидные последовательности-мишени, и связанными представляющими интерес нуклеотидными последовательностям. Такое лигирование эффективно приводит к образованию колец ДНК, в которых известные нуклеотидные последовательности-мишени фланкируют неизвестные представляющие интерес последовательности.
В другом варианте лигирование последовательностей с известным составом нуклеотидов со вторыми сайтами узнавания ферментом рестрикции, которые фланкируют (например, находятся с каждой стороны или на конце) представляющие интерес нуклеотидные последовательности, заключается в добавлении уникальных последовательностей ДНК с известным составом нуклеотидов с последующим лигированием в условиях, которые создают преимущества для внутримолекулярного лигирования между вторыми сайтами узнавания ферментом рестрикции, которые фланкируют представляющие интерес нуклеотидные последовательности, и введенными уникальными последовательностями ДНК с известным составом нуклеотидов.
В одном варианте второй фермент рестрикции выбран так, что вторые сайты узнавания ферментом рестрикции находятся в пределах примерно 350 п.о. (например, 350-400 п.о.) от первого сайта узнавания.
В другом варианте второй фермент рестрикции выбран так, что такой же сайт узнавания вторым ферментом рестрикции вероятно будет располагаться в лигированной нуклеотидной последовательности (т.е. лигированной поперечно сшитой последовательности). Так как концы первой нуклеотидной последовательности (мишени) и лигированной нуклеотидной последовательности могут быть совместимо липкими (или тупыми) концами, то последовательности также могут быть лигированы с образованием колец ДНК. Соответственно, за стадией расщепления следует лигирование в условиях разбавления, чтобы создать преимущества для внутримолекулярных взаимодействий и необязательного образования колец ДНК посредством совместимых концов.
Предпочтительно второй сайт узнавания ферментом рестрикции представляет собой сайт узнавания нуклеотидной последовательности из 4 или 5 п.о. Ферменты, которые узнают последовательности ДНК длиной 4 или 5 п.о., включают, без ограничения, TspEI, MaeII, AluI, NlaIII, HpaII, FnuDII, MaeI, DpnI, MboI, HhaI, HaeIII, RsaI, TaqI, CviRI, MseI, Sth132I, AciI, DpnII, Sau3AI и MnlI.
В предпочтительном варианте вторым ферментом рестрикции является NlaIII и/или DpnII.
Термин «второй сайт узнавания ферментом рестрикции» относится к сайту в нуклеотидной последовательности, который распознается и расщепляется вторым ферментом рестрикции.
После расщепления вторым ферментом рестрикции осуществляют следующую реакцию лигирования. В одном варианте указанная реакция лигирования связывает последовательности ДНК с известным составом нуклеотидной последовательности со вторым сайтом расщепления ферментом рестрикции одной или нескольких последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью.
ТРЕТИЙ ФЕРМЕНТ РЕСТРИКЦИИ
В используемом в данном описании смысле термин «третий фермент рестрикции» относится к третьему ферменту рестрикции, который необязательно можно использовать после стадии рестрикции вторым ферментом, чтобы линеаризовать кольцевую ДНК перед амплификацией.
Предпочтительно третий фермент рестрикции представляет собой фермент, который распознает сайт узнавания, состоящий из 6 п.о. или большего количества нуклеотидов.
Предпочтительно третий фермент рестрикции расщепляет первую нуклеотидную последовательность (мишень) между сайтами узнавания первым и вторым ферментами рестрикции. Как будет понятно специалисту, желательно, чтобы третий фермент рестрикции не расщеплял первую нуклеотидную последовательность (мишень) слишком близко к сайтам узнавания первым и вторым ферментами рестрикции, чтобы праймеры для амплификация не могли гибридизоваться дальше. Соответственно, предпочтительно, чтобы сайт узнавания третьим ферментом рестрикции располагался по меньшей мере на таком же расстоянии от сайтов узнавания первым и вторым ферментами рестрикции, как длина используемого праймера, так чтобы праймер(ры) для амплификации еще могли гибридизоваться.
В предпочтительном варианте третьим ферментом рестрикции является фермент, который узнает последовательность ДНК из 6 п.о.
Термин «третий сайт узнавания ферментом рестрикции» относится к сайту в нуклеотидной последовательности, который распознается и расщепляется третьим ферментом рестрикции.
САЙТ УЗНАВАНИЯ
Эндонуклеазы рестрикции являются ферментами, которые расщепляют сахаро-фосфатный остов ДНК. В большинстве случаев на практике данный фермент рестрикции разрезает обе нити ДНК-дуплекса в пределах участка, состоящего всего из нескольких оснований. Субстратами для ферментов рестрикции являются последовательности двунитевой ДНК, называемые сайтами/последовательностями узнавания.
Длина сайтов узнавания для рестрикции варьирует в зависимости от фермента рестрикции, который используют. Длина последовательности узнавания определяет то, как часто фермент будет разрезать последовательность ДНК.
В качестве примера ряд ферментов рестрикции узнают последовательность ДНК из 4 п.о. Последовательности и ферменты, которые узнают последовательность ДНК из 4 п.о., включают, без ограничения, AATT (TspEI), ACGT (MaeII), AGCT (AluI), CATG (NlaIII), CCGG (HpaII), CGCG (FnuDII), CTAG (MaeI), GATC (DpnI, DpnII, Sau3AI и MboI), GCGC (HhaI), GGCC (HaeIII), GTAC (RsaI), TCGA (TaqI), TGCA (CviRI), TTAA (MseI), CCCG (Sth132I), CCGC (AciI) и CCTC (MnlI).
В качестве следующего примера, ряд ферментов рестрикции узнают последовательность ДНК из 6 п.о. Последовательности и ферменты, которые узнают последовательность ДНК из 6 пар оснований, включают, без ограничения, AACGTT (AclI), AAGCTT (HindIII), AATATT (SspI), ACATGT (BspLU11I), ACCGGT (AgeI), ACGCGT (MlnI), ACTAGT (SpeI), AGATCT (BglII), AGCGCT (Eco47III), AGGCCT (StuI), AGTACT (ScaI), ATCGAT (ClaI), ATGCAT (AvaIII), ATTAAT (VspI), CAATTG (MfeI), CACGTG (PmaCI), CAGCTG (PvuII), CATATG (NdeI), CCATGG (NcoI), CCCGGG (SmaI), CCGCGG (SacII), CCTAGG (AvrII), CGATCG (PvuI), CGGCCG (XmaIII), CGTACG (SplI), CTCGAG (XhoI), CTGCAG (PstI), CTTAAG (AflII), GAATTC (EcoRI), GACGTC (AatII), GAGCTC (SacI), GATATC (EcoRV), GCATGC (SphI), GCCGGC (NaeI), GCGCGC (BsePI), GCTAGC (NheI), GGATCC (BamHI), GGCGCC (NarI), GGGCCC (ApaI), GGTACC (KpnI), GTATAC (SnaI), GTCGAC (SalI), GTGCAC (ApaLI), GTTAAC (HpaI), TACGTA (SnaBI), TCATGA (BspHI), TCCGGA (BspMII), TCGCGA (NruI), TCTAGA (XbaI), TGATCA (BclI), TGCGCA (MstI), TGGCCA (BalI), TGTACA (Bspl407I), TTATAA (PsiI), TTCGAA (AsuII) и TTTAAA(AhaIII).
В качестве следующего примера ряд ферментов рестрикции узнают последовательность ДНК из 7 п.о. Последовательности и ферменты, которые узнают последовательность ДНК из 7 п.о., включают, без ограничения, CCTNAGG (SauI), GCTNAGC (EspI), GGTNACC (BstEII) и TCCNGGA (PfoI).
В качестве следующего примера ряд ферментов рестрикции узнают последовательность ДНК из 8 п.о. Последовательности и ферменты, которые узнают последовательность ДНК из 8 п.о., включают, без ограничения, ATTTAAAT (SwaI), CCTGCAGG (Sse8387I), CGCCGGCG (Sse232I), CGTCGACG (SgrDI), GCCCGGGC (SrfI), GCGATCGC (SgfI), GCGGCCGC (NotI), GGCCGGCC (FseI), GGCGCGCC (AscI), GTTTAAAC (PmeI) и TTAATTAA (PacI).
Ряд сайтов для таких ферментов содержат последовательность CG, которая может быть метилирована in vivo. Некоторые ферменты рестрикции чувствительны к такому метилированию и не будут расщеплять метилированную последовательность, например, HpaII не будет расщеплять последовательность CCmGG, тогда как его изошизомер MspI не чувствителен к такой модификации и будет расщеплять метилированную последовательность. Соответственно, в некоторых случаях не используют ферменты, чувствительные к метилированию у эукариот.
В одном варианте сайт узнавания является сайтом расщепления.
В одном варианте сайт узнавания ферментом рестрикции является сайтом расщепления ферментом рестрикции.
ОБРАЗОВАНИЕ КОЛЬЦЕВЫХ МОЛЕКУЛ
Согласно одному варианту осуществления настоящего изобретения материал для 4C готовят, создавая кольца ДНК расщеплением матрицы 3C вторым ферментом рестрикции с последующим лигированием.
Предпочтительно выбирают такой второй фермент рестрикции, который разрезает первую нуклеотидную последовательность (мишень) на расстоянии не более чем примерно 350 п.о. (например, 350-400 п.о.) от первого сайта рестрикции. Преимущественно это минимизирует искажения при образовании кольца вследствие топологических ограничений (Rippe et al. (2001) Trends in Biochem. Sciences 26, 733-40). Предпочтительно второй фермент рестрикции является часто расщепляющей рестриктазой, узнающей сайт узнавания ферментом рестрикции длиной 4 или 5 п.о. Таким образом, можно получить более мелкие фрагменты рестрикции для одинаковой эффективности амплификации всех лигированных фрагментов во время амплификации.
Перед расщеплением вторым ферментом рестрикции и лигированием ДНК-матрица будет содержать один сайт узнавания вторым ферментом в первой нуклеотидной последовательности (мишени), расположенный на расстоянии более чем примерно 350-400 п.о. от первого сайта узнавания и другой сайт узнавания вторым ферментом рестрикции, расположенный в нуклеотидной последовательности, которая была лигирована (т.е. во второй нуклеотидной последовательности).
Предпочтительно стадию расщепления вторым ферментом рестрикции осуществляют в течение периода, превышающего 1 час, до периода в течение ночи, с последующей инактивацией фермента нагреванием.
Предпочтительно ДНК в указанной реакционной смеси является очищенной с использованием обычных способов/наборов, которые известны в данной области.
После стадии расщепления вторым ферментом рестрикции сайт рестрикции вторым ферментом будет находиться на расстоянии более чем 350-400 п.о. от первого сайта рестрикции в первой нуклеотидной последовательности (мишени), а другой сайт рестрикции вторым ферментом будет находиться в лигированной нуклеотидной последовательности (т.е. во второй нуклеотидной последовательности). Так как концы первой нуклеотидной последовательности (мишени) и лигированной нуклеотидной последовательности имеют совместимые концы, то последовательности могут быть лигированы с образованием кольцевой ДНК.
Затем после стадии расщепления следует лигирование в условиях разбавления, которые создают преимущества для внутримолекулярных взаимодействий и образования колец ДНК посредством совместимых концов.
Предпочтительно реакцию лигирования осуществляют при концентрации ДНК примерно 1-5 нг/мкл.
Предпочтительно реакцию лигирования осуществляют в течение более чем 1 часа (например, в течение 2, 3, 4 или более часов) примерно при 16-25°C.
Таким образом, после реакции лигирования может быть получена кольцевая ДНК. Кольцевая ДНК будет содержать сайты узнавания по меньшей мере для второго фермента рестрикции или для первого и второго ферментов рестрикции. В кольцевой ДНК, содержащей первую нуклеотидную последовательность (мишень), сайт узнавания первым ферментом рестрикции и сайт узнавания вторым ферментом рестрикции будут определять концы первой нуклеотидной последовательности (мишени) и лигированной нуклеотидной последовательности (т.е. второй нуклеотидной последовательности). Соответственно первая нуклеотидная последовательность (мишень) и лигированная нуклеотидная последовательность отделены друг от друга (например, разделены) сайтом узнавания первым ферментом рестрикции и сайтом узнавания вторым ферментом рестрикции.
АМПЛИФИКАЦИЯ
Могут быть осуществлены одна или несколько реакций амплификации, чтобы амплифицировать ДНК-матрицу 4C.
ДНК-амплификация может быть осуществлена с использованием нескольких разных способов, которые известны в данной области. Например, ДНК может быть амплифицирована с использованием полимеразной цепной реакции (Saiki et al., 1988); опосредованной лигированием ПЦР, амплификации с использованием репликазы Qb (Cahill, Foster and Mahan, 1991; Chetverin and Spirin, 1995; Katanaev, Kurnasov and Spirin, 1995); лигазной цепной реакции (LCR) (Landegren et al., 1988; Barany, 1991); самоподдерживающуюся репликацию последовательностей (Fahy, Kwoh and Gingeras, 1991) и амплификацию с замещением цепей (Walker et al., 1992).
Предпочтительно ДНК амплифицируют, используя ПЦР. «ПЦР» относится к способу K. B. Mullis, патенты США №№ 4683195, 4683202 и 4965188, в которых описан способ увеличения концентрации участка нуклеотидной последовательности в смеси геномной ДНК без клонирования или очистки.
В одном варианте используют инвертированную ПЦР. Инвертированная ПЦР (и-ПЦР) (описанная Ochman et al. (1988) Genetics 120(3), 621-3) является способом быстрой амплификации in vitro последовательностей ДНК, которые фланкируют область известной последовательности. В способе используют полимеразную цепную реакцию (ПЦР), но при этом праймеры ориентированы в обратном направлении по сравнению с обычной ориентацией. Матрицей для обратных праймеров является фрагмент рестрикции, который был лигирован сам с собой с образованием кольца. Инвертированная ПЦР имеет много применений в молекулярной генетике, например, для амплификации и идентификации последовательностей, фланкирующих мобильные генетические элементы. Чтобы повысить эффективность и воспроизводимость амплификации, предпочтительно, чтобы кольца ДНК были линеаризованы перед амплификацией с использованием третьего фермента рестрикции. Предпочтительно используют третий фермент рестрикции, который разрезает по 6 п.о. или более. Предпочтительно третий фермент рестрикции разрезает первую нуклеотидную последовательность (мишень) между сайтами рестрикции первым и вторым ферментами.
Расщепление матрицы 3C вторым ферментом рестрикции, необязательное образование колец, лигирование (например, лигирование в условиях разбавления) и необязательная линеаризация колец, содержащих первую нуклеотидную последовательность (мишень), дают ДНК-матрицу для амплификации («ДНК-матрицу 4C»).
Для стадии амплификации используют по меньшей мере два олигонуклеотидных праймера, при этом каждый праймер гибридизуется с последовательностью ДНК, которая фланкирует представляющие интерес нуклеотидные последовательности. В предпочтительном варианте используют по меньшей мере два олигонуклеотидных праймера, при этом каждый праймер гибридизуется с последовательностью-мишенью, фланкирующей представляющие интерес нуклеотидные последовательности.
В одном варианте термин «фланкировать» в контексте гибридизации с праймерами означает, что по меньшей мере один праймер гибридизуется с последовательностью ДНК, граничащей с одним концом (например, 5'-концом) представляющей интерес нуклеотидной последовательности, и по меньшей мере один праймер гибридизуется с последовательностью ДНК на другом конце (например, 3'-конце) представляющей интерес нуклеотидной последовательности. Предпочтительно по меньшей мере один прямой праймер гибридизуется с последовательностью ДНК, граничащей с одним концом (например, с 5'-концом) представляющей интерес нуклеотидной последовательности, и по меньшей мере один обратный праймер гибридизуется с последовательностью ДНК на другом конце (например, на 3'-конце) представляющей интерес нуклеотидной последовательности.
В предпочтительном варианте термин «фланкировать» в контексте гибридизации с праймерами означает, что по меньшей мере один праймер гибридизуется с последовательностью-мишенью, граничащей с одним концом (например, с 5'-концом) представляющей интерес нуклеотидной последовательности, и по меньшей мере один праймер гибридизуется с последовательностью-мишенью на другом конце (например, на 3'-конце) представляющей интерес нуклеотидной последовательности. Предпочтительно по меньшей мере один прямой праймер гибридизуется с последовательностью-мишенью, граничащей с одним концом (например, 5'-концом) представляющей интерес нуклеотидной последовательности, и по меньшей мере один обратный праймер гибридизуется с последовательностью-мишенью на другом конце (например, на 3'-конце) представляющей интерес нуклеотидной последовательности.
В используемом в данном описании смысле термин «праймер» относится к олигонуклеотиду, либо встречающемуся в природе в виде очищенного продукта рестрикции, либо полученному синтетически, который способен действовать в качестве точки инициации синтеза при помещении в условия, в которых индуцируется синтез продукта удлинения праймера, который комплементарен цепи нуклеиновой кислоты (т.е. в присутствии нуклеотидов и индуцирующего агента, такого как ДНК-полимераза, и при подходящей температуре и pH). Праймер предпочтительно является однонитевым для максимальной эффективности амплификации, но может быть и двунитевым. Если праймер является двунитевым, то его сначала обрабатывают, чтобы разделить нити перед использованием для получения продуктов удлинения. Предпочтительно праймер является олигодезоксирибонуклеотидом. Праймер должен быть достаточно длинным, чтобы праймировать синтез продуктов удлинения в присутствии индуцирующего агента. Точные длины праймеров будут зависеть от многих факторов, включая температуру, источник праймера и применение способа.
Соответственно, праймеры будут иметь длину по меньшей мере 15, предпочтительно по меньшей мере 20, например, по меньшей мере 25, 30 или 40 нуклеотидов. Предпочтительно праймеры для амплификации имеют длину от 16 до 30 нуклеотидов.
Предпочтительно праймеры конструируют так, чтобы они были как можно ближе к сайтам узнавания первым и вторым ферментами рестрикции, которые разделяют первую нуклеотидную последовательность (мишень) и вторую нуклеотидную последовательность. Праймеры могут быть сконструированы так, чтобы они были на расстоянии примерно 100 нуклеотидов - например, 90, 80, 70, 60, 50, 40, 30, 20, 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1 нуклеотид, от сайтов узнавания первым и вторым ферментами рестрикции.
Соответственно, праймеры для амплификации конструируют так, чтобы их 3'-концы были обращены наружу в сторону сайтов узнавания первым и вторым ферментами рестрикции так, чтобы удлинение происходило непосредственно через сайты рестрикции ко второй нуклеотидной последовательности.
Если применяемый способ амплификации представляет собой инвертированную ПЦР, то предпочтительно осуществление реакций амплификации примерно на 100-400 нг ДНК 4C-матрицы (примерно на 50 мкл реакционной смеси для ПЦР) или другие количества ДНК, в случае которых репликативные реакции ПЦР дают воспроизводимые результаты (см. фиг.1) и включают максимальное количество событий лигирования на реакцию ПЦР.
Предпочтительно реакцию инвертированной ПЦР-амплификации осуществляют с применением системы ПЦР с элонгацией на длинных матрицах (Roche), используя буфер 1 согласно инструкциям производителя.
ОБРАЗЕЦ
Термин «образец» в используемом в данном описании смысле имеет свое естественное значение. Образец может представлять собой любой физический объект, содержащий ДНК, которая является поперечно сшитой или способна к поперечному сшиванию. Образец может представлять собой или может быть получен из биологического материала.
Образец может представлять собой или может быть получен из одного или нескольких объектов - таких как одна или несколько клеток, одно или несколько ядер или один или несколько образцов ткани. Объекты могут представлять собой или могут быть получены из объектов, в которых присутствует ДНК, например хроматин. Образец может представлять собой или может быть получен из одной или нескольких выделенных клеток или одного или нескольких выделенных образцов ткани, или одного или нескольких выделенных ядер.
Образец может представлять собой или может быть получен из живых клеток и/или мертвых клеток и/или ядерных лизатов и/или изолированного хроматина.
Образец может представлять собой или может быть получен от больного и/или здорового субъекта.
Образец может представлять собой или может быть получен от субъекта, у которого предполагается наличие заболевания.
Образец может представлять собой или может быть получен от субъекта, которого необходимо проверить в отношении вероятности того, что он будет поражен заболеванием в будущем.
Образец может представлять собой или может быть получен из материала живого или умершего пациента.
Фиксация клеток и тканей для применения в получении матрицы 3C подробно описана в Splinter et al., (2004) Methods Enzymol. 375, 493-507.
МЕТКА
Предпочтительно нуклеотидные последовательности (например, амплифицированные ДНК-матрицы 4C, праймеры или зонды и т.д.) метят, что способствует их дальнейшим применениям, таким как гибридизация с чипом. В качестве примера ДНК-матрицы 4C можно метить, используя мечение со случайными праймерами или ник-трансляцию.
Можно использовать широкое множество меток (например, репортеров), чтобы пометить нуклеотидные последовательности, описанные в данной публикации, в частности во время стадии амплификации. Подходящие метки включают радионуклиды, ферменты, флуоресцентные, хемилюминесцентные или хромогенные агенты, а также субстраты, кофакторы, ингибиторы, магнитные частицы и тому подобное. Патенты, в которых имеются инструкции по применению таких меток, включают US-A-3817837, US-A-3850752, US-A-3939350, US-A-3996345, US-A-4277437, US-A-4275149 и US-A-4366241.
Дополнительные метки включают, без ограничения, β-галактозидазу, инвертазу, белок с зеленой флуоресценцией, люциферазу, хлорамфеникол, ацетилтрансферазу, β-глюкуронидазу, экзоглюканазу и глюкоамилазу. Также можно использовать флуоресцентные метки, такие как флуоресцентные реагенты, специально синтезированные с конкретными химическим свойствами. Имеется широкое разнообразие способов для измерения флуоресценции. Например, некоторые флуоресцентные метки проявляют изменение в спектрах возбуждения или испускания, некоторые проявляют резонансный перенос энергии, когда флуоресценция одного флуоресцентного репортера уменьшается, в то время как флуоресценция второго увеличивается, некоторые проявляют потерю (тушение) или появление флуоресценции, в то время как некоторые дают вращательные движения.
Чтобы получить достаточное количество материала для мечения, можно объединить несколько амплификаций, вместо того, чтобы увеличивать количество циклов амплификации на реакцию. Альтернативно меченые нуклеотиды могут быть включены в последние циклы реакции амплификации (например, 30 циклов ПЦР (без метки) + 10 циклов ПЦР (с добавлением метки)).
ЧИП
В конкретном предпочтительном варианте ДНК-матрицы 4C, которые готовят согласно способам, описанным в данной публикации, можно гибридизовать с чипом. Соответственно, методику чипов (например, микроматриц) можно использовать для идентификации нуклеотидных последовательностей, таких как геномные фрагменты, которые часто имеют общее положение в ядре с первой нуклеотидной последовательностью (мишенью).
Существующие чипы, такие как экспрессирующие и геномные чипы, можно использовать согласно настоящему изобретению. Однако в настоящем изобретении предпринята попытка получить новые чипы (например, чипы ДНК), которые описаны в данной публикации.
«Чип» представляет собой намеренно созданную коллекцию нуклеиновых кислот, которая может быть получена либо синтетически, либо биосинтетически и подвергнута скринингу в отношении биологической активности в нескольких разных формах (например, библиотеки растворимых молекул и библиотеки олигомеров, связанных с шариками из смолы, чипы на диоксиде кремния или других твердых подложках). Кроме того, термин «чип» включает такие библиотеки нуклеиновых кислот, которые могут быть получены нанесением пятен нуклеиновых кислот, по существу, любой длины (например, от 1 до примерно 1000 нуклеотидных мономеров в длину) на подложку.
Основанная на чипах методика и различные связанные с ней способы и применения, в общем, описаны в многочисленных руководствах и документах. Публикации включают Lemieux et al., 1998, Molecular Breeding 4, 277-289, Schena and Davis. Parallel Analysis with Biological Chips, in PCR Methods Manual (eds. M. Innis, D. Gelfand, J. Sninsky), Schena and Davis, 1999, Genes, Genomes and Chips. In DNA Microarrays: A Practical Approach (ed. M. Schena), Oxford University Press, Oxford, UK, 1999), The Chipping Forecast (Nature Genetics special issue; January 1999 Supplement), Mark Schena (Ed.), Microarray Biochip Technology, (Eaton Publishing Company), Cortes, 2000, The Scientist 14[17]: 25, Gwynne and Page, Microarray analysis: the next revolution in molecular biology, Science, 1999 August 6; и Eakins and Chu, 1999, Trends in Biotechnology, 17, 217-218.
Основанная на чипах методика позволяет преодолеть недостатки традиционных способов молекулярной биологии, которые обычно работают на основе «один ген в одном эксперименте», приводя к низкой производительности и неспособности оценить «полную картину» функционирования генов. В настоящее время основные применения методики чипов включают идентификацию последовательности (ген/мутация гена) и определение уровня экспрессии (распространенности) генов. Методику чипов можно использовать при определении профиля экспрессии генов, необязательно в комбинации со способами протеомики (Celis et al., 2000, FEBS Lett., 480(1): 2-16; Lockhart and Winzeler, 2000, Nature 405(6788): 827-836; Khan et al., 1999, 20(2): 223-9). Другие применения основанной на чипах методики также известны в данной области; например, выявление генов, исследование злокачественной опухоли (Marx, 2000, Science 289: 1670-1672; Scherf, et al., 2000, Nat. Genet; 24(3): 236-44; Ross et al., 2000, Nat. Genet. 2000 Mar; 24(3): 227-35), анализ SNP (Wang et al., 1998, Science, 280(5366): 1077-82), выявление лекарственных средств, фармакогеномика, диагностика заболеваний (например, использование микрожидкостных устройств: Chemical and Engineering News, February 22, 1999, 77(8): 27-36), токсикология (Rockett and Dix (2000), Xenobiotica, 30(2): 155-77; Afshari et al., 1999, Cancer Resl; 59(19): 4759-60) и токсикогеномика (гибрид функциональной геномики и молекулярной токсикологии).
В общем, любая библиотека может быть упорядоченно распределена на чипе с пространственным разделением представителей библиотеки. Примеры подходящих библиотек для распределения на чипе включают библиотеки нуклеиновых кислот (включая библиотеки ДНК, кДНК, олигонуклеотидов и т.д.), библиотеки пептидов, полипептидов и белков, а также библиотеки, содержащие любые молекулы, например, такие как библиотеки лигандов.
Образцы (например, представители библиотеки) в общем фиксируют или иммобилизуют на твердой фазе, предпочтительно на твердой подложке, чтобы ограничить диффузию и смешивание образцов. В предпочтительном варианте могут быть получены библиотеки лигандов, связывающих ДНК. В частности, библиотеки могут быть иммобилизованы, по существу, на плоской твердой фазе, включая мембраны и непористые подложки, такие как пластик и стекло. Кроме того, образцы предпочтительно распределяют таким образом, чтобы облегчить индексацию (т.е. ссылки или доступ к конкретному образцу). Обычно образцы наносят пятнами в виде решетки. Для данной цели могут быть адаптированы общие системы анализа. Например, матрица может быть иммобилизована на поверхности микропланшета, либо с несколькими образцами в лунке, либо с одним образцом в каждой лунке. Кроме того, твердой подложкой может быть мембрана, такая как нитроцеллюлозная или нейлоновая мембрана (например, мембраны, используемые в экспериментах по блоттингу). Альтернативные подложки включают подложки на основе стекла или диоксида кремния. Таким образом, образцы иммобилизуют любым подходящим способом, известным в данной области, например, за счет взаимодействия зарядов или химическим связыванием со стенками или дном лунок или с поверхностью мембраны. Можно использовать другие средства распределения и фиксации, например нанесение пипеткой, каплями, пьезоэлектрические способы, струйную и пузырьково-струйную методику, электростатическое нанесение и т.д. В случае основанных на диоксиде кремния чипах можно использовать фотолитографию для распределения и фиксации образцов на чипе.
Образцы могут быть распределены посредством «нанесения пятнами» на твердую подложку; нанесение можно сделать вручную или с использованием робототехники для нанесения образца. В общем, чипы могут быть описаны как макроматрицы или микроматрицы, при этом различие заключается в размере пятен образца. Макроматрицы обычно содержат пятна образца размером примерно 300 микрон или больше, и их можно легко визуализировать с помощью существующих сканнеров гелей и блотов. Размеры пятен образца на микроматрице обычно составляют менее 200 микрон в диаметре, и такие чипы обычно содержат тысячи пятен. Таким образом, для микроматрицы может требоваться специальная робототехника и оборудование для визуализации, которые необходимо делать специально. Оборудование в общем описано в обзоре Cortese, 2000, The Scientist 14[11]: 26.
Способы получения иммобилизованных библиотек молекул ДНК описаны в данной области. В общем, в случае большинства способов известного уровня техники описано, как синтезировать библиотеки однонитевых молекул нуклеиновых кислот, используя, например, методику трафаретов для построения различных перестановок последовательностей в различных отдельных положениях на твердой подложке. В патенте США № 5837832 описан усовершенствованный способ получения матриц ДНК, иммобилизованных на кремниевых подложках, основанных на способе компоновки в очень крупном масштабе. В частности, в патенте США № 5837832 описана методика, называемая «мозаичным распределением», для синтеза специфичных наборов зондов в пространственно заданных положениях на подложке, которая может быть использована для получения иммобилизованных библиотек ДНК согласно настоящему изобретению. В патенте США № 5837832 также приведены ссылки на более ранние способы, которые также можно использовать.
Чипы также могут быть построены с использованием химии фотоосаждения.
Матрицы пептидов (или пептидомиметиков) также могут быть синтезированы на поверхности таким образом, чтобы разместить каждого отдельного представителя библиотеки (например, уникальную пептидную последовательность) в отдельном, заранее определенном положении на чипе. Опознавание каждого представителя библиотеки осуществляют по его пространственному положению на чипе. Определяют положение на чипе, в котором происходят взаимодействия связывания между предварительно определяемой молекулой (например, мишенью или зондом) и реакционно-способными представителями библиотеки, тем самым идентифицируя последовательности реакционно-способных представителей библиотеки на основе их пространственного положения. Такие способы описаны в патенте США № 5143854, WO 90/15070 и WO 92/10092, Fodor et al. (1991) Science, 251: 767; Dower and Fodor (1991) Ann. Rep. Med. Chem., 26: 271.
Для регистрации обычно используют метки (которые обсуждаются выше), такие как легко регистрируемый репортер, например, флуоресцирующий, биолюминесцентный, фосфоресцентный, радиоактивный и т.д. репортер. Такие репортеры, их регистрация, связывание с мишенями/зондами и т.д. обсуждаются в данном документе. Мечение зондов и мишеней также описано в Shalon et al., 1996, Genome Res. 6(7): 639-45.
Конкретными примерами ДНК-чипов являются следующие чипы:
Форма I: зонд кДНК (длиной 500-5000 оснований) иммобилизуют на твердой поверхности, такой как стекло, используя автоматическое нанесение пятен и подвергая воздействию набора мишеней либо по отдельности, либо в виде смеси. Указанный способ, который был разработан в Стэнфордском Университете (Ekins and Chu, 1999, Trends in Biotechnology, 1999, 17, 217-218), является широко признанным способом.
Форма II: матрицу олигонуклеотидов (20-25-мерных олигонуклеотидов, предпочтительно 40-60-мерных олигонуклеотидов) или зондов на основе пептидо-нуклеиновых кислот (PNA) синтезируют либо in situ (на чипе), либо обычным синтезом с последующей иммобилизацией на чипе. Чип подвергают воздействию меченого образца ДНК, гибридизуют и определяют идентичность/относительное содержание комплементарных последовательностей. Такой чип ДНК продается Affymetrix, Inc. под торговой маркой GeneChip®. Agilent и Nimblegen также предлагают подходящие чипы (например, геномные мозаичные чипы).
Примеры некоторых коммерчески доступных форм микроматрицы указаны в таблице 1 ниже (см. также Marshall and Hodgson, 1998, Nature Biotechnology, 16(1), 27-31).
Чтобы получить данные в основанных на чипах анализах, регистрируют сигнал, который свидетельствует о наличии или отсутствии гибридизации между зондом и нуклеотидной последовательностью. Настоящее изобретение, кроме того, включает в себя способы прямого и опосредованного мечения. Например, в случае прямого мечения флуоресцентные красители включают непосредственно в нуклеотидные последовательности, которые гибридизуются с зондами, связанными с чипом (например, красители включают в нуклеотидную последовательность посредством ферментативного синтеза в присутствии меченых нуклеотидов или ПЦР-праймеров). Схемы прямого мечения дают интенсивные сигналы гибридизации, обычно при использовании семейств флуоресцентных красителей со сходными химическими структурами и характеристиками, и являются простыми в осуществлении. В предпочтительных вариантах, включающих в себя прямое мечение нуклеиновых кислот, используют аналоги цианина или alexa в сравнительных анализах на чипах с применением нескольких флуоресцирующих агентов. В других вариантах можно использовать схемы опосредованного мечения, чтобы ввести эпитопы в нуклеиновые кислоты либо до, либо после гибридизации с зондами на микроматрице. Используют один или несколько способов окрашивания и реагентов, чтобы пометить гибридизованный комплекс (например, флуоресцентную молекулу, которая связывается с эпитопами, обеспечивая, таким образом, сигнал флуоресценции вследствие конъюгирования молекулы красителя с эпитопом гибридизованного вида).
Анализ данных также является важной частью эксперимента с применением чипов. Необработанные данные, полученные в основанном на использовании чипов эксперименте, обычно представляют собой изображения, которые необходимо преобразовать в матрицы - таблицы, в которых строки представляют, например, гены, столбцы представляют, например, различные образцы, такие как ткани, или условия эксперимента, и числа в каждой ячейке, например, характеризуют экспрессию конкретной последовательности (предпочтительно второй последовательности, которая была лигирована с первой нуклеотидной последовательностью (мишенью)), в конкретном образце. Указанные матрицы затем необходимо анализировать, если необходимо извлечь какие-либо данные о лежащих в основе биологических процессах. Способы анализа данных (включая контролируемый и неконтролируемый анализ данных, а также способы биоинформатики) описаны в Brazma and Vilo J. (2000) FEBS Lett. 480(1): 17-24.
Как описано в данной публикации, одна или несколько нуклеотидных последовательностей (например, ДНК-матрица), которые метят и затем гибридизуют на чипе, содержат нуклеотидную последовательность, которая обогащена небольшими участками последовательностей с отличительным характерным признаком, например, на протяжении нуклеотидной последовательности между сайтом узнавания первым ферментом рестрикции, который лигировали во время осуществления способа 3C с первой нуклеотидной последовательностью (мишенью), и соответствующими соседними сайтами узнавания вторым ферментом рестрикции.
Один чип может содержать несколько (например, две или более) последовательностей «приманок».
ЗОНДЫ
В используемом в данном описании смысле термин «зонд» относится к молекуле (например, олигонуклеотиду, либо встречающемуся в природе, например, в виде очищенного продукта рестрикции, либо полученному синтетически, рекомбинантно, либо при ПЦР-амплификации), которая способна гибридизоваться с другой представляющей интерес молекулой (например, другим олигонуклеотидом). В том случае, когда зонды представляют собой олигонуклеотиды, они могут быть однонитевыми или двунитевыми. Зонды применимы для регистрации, идентификации и выделения конкретных мишеней (например, генных последовательностей). Как описано в данной публикации, предполагается, что зонды, используемые в настоящем изобретении, можно метить меткой, которую можно регистрировать в любой системе регистрации, включая, без ограничения, фермент (например, ELISA, а также основанные на ферментах гистохимические анализы), флуоресцентные, радиоактивные и люминесцентные системы.
Что касается чипов и микроматриц, то термин «зонд» используют по отношению к любому способному к гибридизации материалу, который связан с чипом в целях выявления нуклеотидной последовательности, которая гибридизуется с указанным зондом. Предпочтительно такие зонды являются 25-60-мерами или имеют большую длину.
Методики конструирования зондов описаны в WO 95/11995, EP 717113 и WO 97/29212.
Так как 4C обеспечивает объективный поиск во всем геноме в отношении взаимодействий, то предпочтительно получение чипа с зондами, обеспечивающими запрос каждого возможного (например, уникального/неповторяющегося) сайта узнавания первым ферментом рестрикции в геноме. Таким образом, конструкция чипа зависит только от выбора первого фермента рестрикции, а не от реальной первой или второй нуклеотидных последовательностей.
Хотя согласно настоящему изобретению можно использовать существующие чипы, предпочтительно применение альтернативных конфигураций.
В одной конфигурации один или несколько зондов на чипе конструируют так, чтобы они могли гибридизоваться вблизи сайтов, которые расщепляются первым ферментом рестрикции. Более предпочтительно зонд (зонды) располагаются в пределах примерно 20 п.о. от сайта узнавания первым ферментом рестрикции. Более предпочтительно зонд (зонды) располагаются в пределах примерно 50 п.о. от сайта узнавания первым ферментом рестрикции.
Таким образом, зонд (зонды) располагаются в пределах примерно 100 п.о. (например, примерно 0-100 п.о., примерно 20-100 п.о.) от сайта узнавания первым ферментом рестрикции.
В предпочтительной конфигурации конструируют один уникальный зонд в пределах 100 п.о. с каждой стороны от сайтов, которые расщепляются первым ферментом рестрикции.
В другой предпочтительной конфигурации принимают во внимание положения сайтов, расщепляемых вторым ферментом рестрикции, относительно положения сайтов, расщепляемых первым ферментом рестрикции. В данной конфигурации конструируют один уникальный зонд только с каждой стороны от сайтов, расщепляемых первым ферментом рестрикции, которые имеют ближайший сайт узнавания вторым ферментом рестрикции на достаточно большом расстоянии, чтобы сконструировать зонд заданной длины между сайтами узнавания первым и вторым ферментами рестрикции. В такой конфигурации, например, не конструируют зонд со стороны конкретного сайта узнавания первым ферментом рестрикции, когда сайт узнавания вторым ферментом рестрикции находится в пределах 10 п.о. с той же самой стороны.
В другой конфигурации зонды на чипе конструируют так, чтобы они гибридизовались с любой стороны от сайтов, которые расщепляются первым ферментом рестрикции. Таким образом, можно использовать один зонд с каждой стороны от сайта узнавания первым ферментом рестрикции.
В еще одной конфигурации могут быть сконструированы два или несколько зондов (например, 3, 4, 5, 6, 7 или 8 или более) с каждой стороны сайта узнавания первым ферментом рестрикции, которые затем могут быть использованы для исследования такого же события лигирования. В отношении количества и положения зондов относительно каждого сайта узнавания первым ферментом рестрикции учитывают точное положение в геноме соседнего сайта узнавания вторым ферментом рестрикции.
В еще одной конфигурации могут быть сконструированы два или более зондов (например, 3, 4, 5, 6, 7 или 8 или более) вблизи каждого сайта узнавания первым ферментом рестрикции, независимо от ближайшего сайта узнавания вторым ферментом рестрикции. В данной конфигурации все зонды все же должны находиться близко к сайтам узнавания первым ферментом рестрикции (предпочтительно в пределах 300 п.о. от сайта рестрикции).
Преимущественно последняя конструкция, а также конструкция, в которой используют 1 зонд (сбоку от) сайта узнавания первым ферментом рестрикции, позволяют использовать разные вторые ферменты рестрикции в сочетании с данным первым ферментом рестрикции.
Преимущественно применение нескольких (например, 2, 3, 4, 5, 6, 7 или 8 или более) зондов на один сайт узнавания первым ферментом рестрикции может минимизировать проблему получения ложноотрицательных результатов вследствие слабой эффективности отдельных зондов. Кроме того, применение нескольких зондов также может повышать достоверность данных, полученных в эксперименте с использованием одного чипа, и уменьшать количество чипов, необходимых для того, чтобы сделать статистически значимые выводы.
Зонды для применения при получении чипа могут иметь длину более чем 40 нуклеотидов и могут быть изотермическими.
Предпочтительно зонды, содержащие повторяющиеся последовательности ДНК, исключают.
Зонды, являющиеся диагностическими в отношении сайтов рестрикции, которые непосредственно фланкируют или находятся вблизи первой нуклеотидной последовательности, предположительно дают очень интенсивные сигналы гибридизации и также могут быть исключены из конструкций зондов.
Чип может охватывать любой геном, включая геномы млекопитающих (например, человека, мыши (например, хромосому 7)), позвоночных (например, полосатый данио)) или беспозвоночных (например, бактерий, дрожжей, грибов или насекомых (например, Drosophila)).
В следующем предпочтительном варианте чип содержит 2-6 зондов вокруг каждого уникального первого сайта рестрикции и близко, насколько это возможно, к сайту расщепления ферментом рестрикции.
Предпочтительно максимальное расстояние от сайта расщепления ферментом рестрикции составляет примерно 300 п.о.
В следующем предпочтительном варианте настоящего изобретения предлагаются чипы для ферментов рестрикции, таких как HindIII, EcoRI, BglII и NotI, которые охватывают геномы млекопитающего или другого животного, отличного от млекопитающих. Преимущественно конструкция чипов, описанных в данной публикации, позволяет обойтись без необходимости повторно конструировать чипы для каждой последовательности-мишени, при условии, что анализ осуществляют у того же самого вида.
НАБОР ЗОНДОВ
В используемом в данном описании смысле термин «набор зондов» относится к набору или совокупности зондов, которые гибридизуются с каждым из первых сайтов узнавания ферментом рестрикции для первого фермента рестрикции в геноме.
Соответственно, в следующем аспекте предлагается набор зондов, имеющих последовательность, комплементарную последовательности нуклеиновой кислоты, граничащей с каждым из первых сайтов узнавания ферментом рестрикции для первого фермента рестрикции в геномной ДНК.
Таким образом, зонды в наборе комплементарны по последовательности первым 25-60 (например, 35-60, 45-60 или 50-60) или более нуклеотидам, которые граничат с каждым из сайтов узнавания первым ферментом рестрикции в геномной ДНК. Зонды в наборе могут быть комплементарны последовательности с одной (например, с любой) стороны или с обеих сторон от сайта узнавания первым ферментом рестрикции. Соответственно, последовательность зондов может быть комплементарна последовательности нуклеиновой кислоты, граничащей с каждой стороны с каждым из сайтов узнавания первым ферментом рестрикции в геномной ДНК.
Также можно определить окно (например, 300 п.о. или меньше - например, 250 п.о., 200 п.о., 150 п.о. или 100 п.о. - от сайта узнавания первым ферментом рестрикции), в котором может быть сконструирован один или несколько зондов для набора. Такими факторами, которые важны для определения окна, в пределах которого конструируют зонды, являются, например, содержание GC, отсутствие палиндромных последовательностей, которые могут образовывать структуры в виде шпилек, максимальный размер участков с нуклеотидами одного типа. Соответственно, по последовательности зонды в наборе могут быть комплементарны последовательности нуклеиновой кислоты в пределах менее 300 п.о. от каждого из сайтов узнавания первым ферментом рестрикции в геномной ДНК.
Также можно определить окно в пределах примерно 100 п.о. от сайта узнавания первым ферментом рестрикции, чтобы идентифицировать оптимальные зонды вблизи каждого сайта рестрикции.
В следующих вариантах осуществления настоящего изобретения зонды в наборе комплементарны последовательности, которая находится в пределах менее 300 п.о. от каждого из сайтов узнавания первым ферментом рестрикции в геномной ДНК, комплементарны последовательности, расположенной в пределах от 200 до 300 п.о. от каждого из сайтов узнавания первым ферментом рестрикции в геномной ДНК и/или комплементарны последовательности, расположенной в пределах от 100 до 200 п.о. от каждого из сайтов узнавания первым ферментом рестрикции в геномной ДНК.
В следующих вариантах осуществления настоящего изобретения зонды в наборе комплементарны последовательности, расположенной в пределах от 0 до 300 п.о. от каждого из сайтов узнавания первым ферментом рестрикции в геномной ДНК, комплементарны последовательности, расположенной в пределах от 0 до 200 п.о. от каждого из сайтов узнавания первым ферментом рестрикции в геномной ДНК и/или комплементарны последовательности, расположенной в пределах от 0 до 100 п.о. от каждого из сайтов узнавания первым ферментом рестрикции в геномной ДНК (например, примерно 10, 20, 30, 40, 50, 60, 70, 80 или 90 п.о. от каждого из сайтов узнавания первым ферментом рестрикции в геномной ДНК).
Также могут быть сконструированы два или более зондов, которые способны гибридизоваться с последовательностью, граничащей с каждым из сайтов узнавания первым ферментом рестрикции в геномной ДНК.
Зонды могут перекрываться или частично перекрываться. Если зонды перекрываются, то перекрывание предпочтительно составляет менее чем 10 нуклеотидов.
Также можно использовать ПЦР-фрагменты, представляющие собой первые 1-300 нуклеотидов (например, 1-20, 1-40, 1-60, 1-80, 1-100, 1-120, 1-140, 1-160, 1-180, 1-200, 1-220, 1-240, 1-260 или 1-280 нуклеотидов), которые фланкируют каждый сайт узнавания первым ферментом рестрикции.
ПЦР-фрагменты также можно использовать в качестве зондов, которые точно соответствуют каждому сайту в геноме, который фланкирован сайтом узнавания первым ферментом рестрикции и первым ближайшим сайтом узнавания вторым ферментом рестрикции. Соответственно, последовательность зонда может соответствовать всей или части последовательности между каждым из сайтов узнавания первым ферментом рестрикции и каждым первым ближайшим сайтом узнавания вторым ферментом рестрикции.
Как правило, зонды, матрица зондов или набор зондов будут иммобилизованы на подложке. Подложки (например, твердые подложки) могут быть изготовлены из различных материалов, таких как стекло, диоксид кремния, пластик, нейлон или нитроцеллюлоза. Подложки предпочтительно являются жесткими и имеют плоскую поверхность. Подложки обычно имеют примерно 1-10000000 отдельных имеющих определяемое пространственное положение областей или ячеек. Обычно подложки имеют примерно 10-1000000 или примерно 100-100000 или примерно 1000-100000 ячеек. Плотность ячеек обычно составляет по меньшей мере примерно 1000, 10000, 100000 или 1000000 ячеек на квадратный сантиметр. На некоторых подложках все ячейки заняты объединенными смесями зондов или набором зондов. На других подложках некоторые ячейки заняты объединенными смесями зондов или набором зондов, а другие ячейки заняты олигонуклеотидом одного типа, получаемым способами синтеза, по меньшей мере с определенной степенью чистоты.
Предпочтительно чип, описанный в данной публикации, содержит более одного зонда на сайт узнавания первым ферментом рестрикции, который в случае фермента рестрикции, разрезающего по 6 п.о., встречается, например, около 750000 раз в геноме человека или мыши.
В случае фермента рестрикции, узнающего последовательность узнавания длиной >6 п.о., можно использовать один чип, содержащий примерно 2×750000 зондов, чтобы охватить, например, полный геном человека или мыши, при этом 1 зонд используют для каждой стороны каждого сайта рестрикции.
В предпочтительной конструкции чипа общее количество молекул зондов с заданной нуклеотидной последовательностью, присутствующих на чипе, находится в большом избытке по отношению к гомологичным фрагментам, присутствующим в образце 4C, который гибридизуют с таким чипом. Учитывая природу способа 4C, фрагменты, представляющие области генома, расположенные вблизи анализируемой нуклеотидной последовательности на линейной хроматиновой матрице, будут в большом избытке в образце для гибридизации 4C (как описано на фиг.2). Чтобы получить количественную информацию об эффективности гибридизации таких часто встречающихся фрагментов, может быть необходимо уменьшение количества гибридизуемого образца и/или увеличение количества молекул зонда с заданной олигонуклеотидной последовательностью на чипе.
Таким образом, для определения регуляторных элементов ДНК, которые часто контактируют, например промоторного элемента генов, может быть необходимым применение чипа с зондами, которые представляют только выбранную область генома (например, примерно 0,5-10 млн п.о.), но при этом каждый уникальный зонд присутствует в разных (например, примерно в 100, 200, 1000) положениях на чипе. Такие конструкции также могут быть предпочтительными для диагностических целей, чтобы выявить локальные (например, в пределах около 10 млн п.о.) геномные перестройки, такие как делеции, инверсии, дупликации и т.д., вокруг сайта (например, представляющего интерес гена).
Чип может содержать примерно 3×750000 зондов, 4×750000 зондов, 5×750000 зондов или предпочтительно 6×750000 зондов. Более предпочтительно чип содержит 6×750000 зондов, по 2, 3, 4, 5, 6, 7 или 8 или более зондов с каждой стороны от каждого сайта рестрикции. Наиболее предпочтительно чип содержит 6×750000 зондов, по 3 зонда с каждой стороны от каждого сайта рестрикции.
Матрицы зондов или наборы зондов могут быть последовательно синтезированы на подложке или могут быть связаны в предварительно синтезируемой форме. Одним из способов синтеза является VLSIPSTM (который описан в US 5143854 и EP 476014), который подразумевает применение света для регуляции синтеза олигонуклеотидных зондов в виде микроматриц с высокой плотностью. Алгоритмы конструирования трафаретов для того, чтобы уменьшить количество циклов синтеза, описаны в US 5571639 и US 5593839. Матрицы также могут быть синтезированы комбинаторным способом посредством доставки мономеров в ячейки подложки механически ограниченными потоками, как описано в EP 624059. Матрицы также могут быть синтезированы посредством нанесения реагентов в виде пятен на подложку с использованием струйного принтера (см., например, EP 728520).
В контексте настоящего изобретения термины «по существу набор зондов», «по существу матрица зондов» означают, что набор или матрица зондов содержит по меньшей мере примерно 50, 60, 70, 80, 90, 95, 96, 97, 98 или 99% всего или полного набора или матрицы зондов. Предпочтительно набор или матрица зондов представляют собой весь или полный набор зондов (т.е. 100%).
В предпочтительном варианте чип содержит один уникальный зонд с каждой стороны каждого сайта узнавания первым ферментом рестрикции, который присутствует в данном геноме. Если такое количество зондов превышает количество зондов, которое может находиться на одном чипе, то чип предпочтительно еще может содержать полный геном данного вида, но с меньшим разрешением, например может содержать один из 2, 3, 4, 5, 6, 7, 8, 9, 10, 102, 103 или 104 и т.д. зондов, которые располагаются по порядку на линейной хромосомной матрице, присутствующей на чипе. Такие чипы, которые охватывают полный геном человека или другой геном при субоптимальном разрешении, могут быть предпочтительными по сравнению с чипами с высоким разрешением, которые охватывают часть того же самого генома, например, в случаях, когда необходимо найти партнеров при транслокации.
Предпочтительно представление полного генома данного вида при более низком разрешении получают благодаря зондам на чипе, каждый из которых представляет один фрагмент рестрикции, который получают после расщепления первым ферментом рестрикции. Предпочтительно такое представление получают, пропуская каждый второй, третий, четвертый, пятый, шестой, седьмой, восьмой, девятый, десятый, двадцатый, тридцатый, сороковой, пятидесятый, шестидесятый, семидесятый, восьмидесятый, девяностый или сотый и т.д. зонд, который гибридизуется с тем же самым фрагментом рестрикции.
Предпочтительно представление полного генома данного вида при более низком разрешении содержит зонды, которые равномерно распределены вдоль линейных хромосомных матриц. Предпочтительно такое представление получают, пропуская один или несколько зондов в тех областях генома, в которых наблюдается наиболее высокая плотность зондов.
ГИБРИДИЗАЦИЯ
Термин «гибридизация» в используемом в данном описании смысле будет включать «способ, с помощью которого нить нуклеиновой кислоты связывается с комплементарной нитью посредством спаривания оснований», а также способ амплификации, который осуществляют, например, в случае использования методики полимеразной цепной реакции (ПЦР).
Нуклеотидные последовательности, способные избирательно гибридизоваться, в общем будут по меньшей мере на 75%, предпочтительно по меньшей мере на 85% или на 90% и более предпочтительно по меньшей мере на 95% или на 98% гомологичны соответствующей комплементарной нуклеотидной последовательности на протяжении области, состоящей по меньшей мере из 20, предпочтительно по меньшей мере из 25 или 30, например, по меньшей мере из 40, 60 или 100 или более следующих друг за другом нуклеотидов.
«Специфичная гибридизация» относится к связыванию, образованию дуплекса или гибридизации молекулы только с конкретной нуклеотидной последовательностью в жестких условиях (например, при 65°C и 0,1×SSC (1×SSC=0,15М NaCl, 0,015М Na-цитрат, pH 7,0)). Жесткими условиями являются условия, в которых зонд будет гибридизоваться со своей последовательностью-мишенью, но не будет гибридизоваться с другими последовательностями. Жесткие условия зависят от последовательности и отличаются в разных случаях. Более длинные последовательности специфично гибридизуются при более высоких температурах. В общем, жесткие условия выбирают примерно на 5°C ниже, чем точка температуры плавления (Tm) для конкретной последовательности при определенной ионной силе и pH. Tm представляет собой температуру (при определенной ионной силе, pH и концентрации нуклеиновой кислоты), при которой 50% зондов, комплементарных последовательности-мишени, гибридизуются с последовательностью-мишенью в равновесии (Так как последовательности-мишени, как правило, присутствуют в избытке, то при Tm в равновесии 50% зондов заняты). Обычно жесткие условия включают концентрацию соли, составляющую по меньшей мере примерно от 0,01 до 1,0М концентрации ионов Na (или других солей) при pH от 7,0 до 8,3 и при температуре, составляющей по меньшей мере примерно 30°C, для коротких зондов. Жесткие условия также могут быть достигнуты при добавлении дестабилизирующих агентов, таких как формальдегид или соли тетраалкиламмония.
Как будет понятно специалистам в данной области, гибридизация в условиях максимальной жесткости может быть использована для идентификации или выявления идентичных нуклеотидных последовательностей, тогда как гибридизация в условиях умеренной (или низкой) жесткости может быть использована для идентификации или выявления сходных или родственных полинуклеотидных последовательностей.
Также описаны способы гибридизации матриц зондов с мечеными или немечеными нуклеотидными последовательностями. Конкретные условия реакции гибридизации можно регулировать, чтобы изменить гибридизацию (например, увеличить или уменьшить жесткость связывания зонд/мишень). Например, температура реакции, концентрации анионов и катионов, добавление детергентов и тому подобное могут изменять параметры гибридизации зондов на чипе и молекул-мишеней.
ЧАСТОТА ВЗАИМОДЕЙСТВИЯ
Количественное измерение частоты лигирования фрагментов рестрикции является мерой частоты их поперечного сшивания. Соответственно, количественное измерение можно осуществить, используя ПЦР, применяемую в обычном способе 3C, который описан Splinter et al. (2004) (выше). Коротко, образование продуктов ПЦР можно измерить сканированием интенсивностей сигнала после разделения в агарозных гелях, окрашенных бромидом этидия, с использованием устройства для визуализации Typhoon 9200 (Molecular Dynamics, Sunnyvale, CA). Соответственно, используют несколько контролей для правильной интерпретации данных, что также описано Splinter et al. (2004) (выше).
Так как способ 4C, описанный в данной публикации, обеспечивает высокопроизводительный анализ частоты взаимодействия двух или более нуклеотидных последовательностей в ядерном пространстве, предпочтительна количественная оценка частоты лигирования фрагментов рестрикции с использованием чипов, описанных в данной публикации.
В случае количественного анализа сигналы, получаемые для образца 4C, могут быть нормализованы по отношению к сигналам, получаемым для контрольного образца. Образец 4C и контрольный образец(цы) будут мечены разными и отличающимися метками (например, красителями) и будут одновременно гибридизованы с чипом. Контрольный образец(цы) обычно будет содержать все фрагменты ДНК (т.е. все возможные вторые нуклеотидные последовательности, которые лигированы с первой нуклеотидной последовательностью (мишенью)) в равных количествах, и, чтобы исключить ошибки в оценке эффективности гибридизации, он должен быть сходен по размеру со второй нуклеотидной последовательностью(ями). Таким образом, контрольная матрица обычно будет содержать геномную ДНК (того же генетического происхождения, что и геномная ДНК, используемая для получения матрицы 4C), расщепленную как первым, так и вторым ферментами рестрикции и меченую таким же способом (например, с использованием случайных праймеров), что и матрица 4C. Такая контрольная матрица делает возможным корректировку связанных с разными зондами различий в эффективности гибридизации. Нормализация сигналов матрицы 4C к сигналам контрольной матрицы обеспечивает возможность выражать результаты на основе сравнения по увеличению со случайными событиями.
Меченая 4C-матрица также может быть гибридизована с чипом в присутствии или в отсутствие иным образом меченого контрольного образца и в присутствии или в отсутствие одного или нескольких иначе меченых других 4C-матриц. Другие 4C-матрицы могут быть неродственными данной 4C-матрице, например, они могут быть получены из разных тканей и/или получены с использованием другого набора праймеров для инвертированной ПЦР. Например, первая 4C-матрица может быть получена от пациента, а вторая 4C-матрица может быть получена от здорового субъекта или контрольного образца.
Имея убедительные картины гибридизации, которые вероятны для генетических перестроек, не всегда будет необходимо сравнивать больных субъектов и здоровых. Соответственно, несколько (например, две или более) 4C-матрицы, каждая из которых позволяет исследовать разные локусы у одного и того же пациента или субъекта, можно гибридизовать с одним (например, одним или несколькими) чипом.
4C-матрицы можно метить по-разному (например, гибридизация с двумя или несколькими окрасками) и/или могут быть одинаково помечены в случае таких локусов, которые в норме находятся на разных хромосомах или на одной и той же хромосоме на достаточно далеком расстоянии, чтобы минимизировать перекрывание между сигналами ДНК-ДНК-взаимодействия. В качестве примера материал от субъекта с T-клеточным лейкозом можно обработать так, чтобы получить 4C-матрицы для TCRα/β (меченая одним цветом, чтобы выявить транслокации), и MLL, TAL1, HOX11 и LMO2 (каждая мечена одинаковым вторым цветом, чтобы выявить другие генетические перестройки). Указанные пять 4C-матриц можно гибридизовать с одним чипом, который позволит осуществить одновременный анализ в нескольких локусах в отношении генетической перестройки, связанной с заболеванием.
Для количественного анализа частоты взаимодействий также можно учитывать абсолютные интенсивности сигналов или отношения к контрольному образцу. Кроме того, сигналы зондов, расположенных рядом на линейной хромосомной матрице, можно использовать для идентификации взаимодействующих хромосомных областей. Такую информацию о положении предпочтительно анализируют, располагая зонды по порядку на линейной хромосомной матрице и анализируя абсолютные интенсивности сигналов или отношения к сигналам контрольных матриц способами скользящего окна, используя, например, способы скользящих средних или скользящих медианных значений.
СПОСОБ АНАЛИЗА
В следующем аспекте настоящего изобретения предлагается способ анализа для идентификации одного или нескольких агентов, которые модулируют ДНК-ДНК-взаимодействие.
В используемом в данном описании смысле термин «модулировать» относится к предотвращению, уменьшению, подавлению, восстановлению, повышению, увеличению или другому влиянию на ДНК-ДНК-взаимодействие.
В некоторых случаях может быть желательна оценка двух или более агентов вместе в отношении применения для модулирования ДНК-ДНК-взаимодействия. В таких случаях анализы можно легко модифицировать добавлением такого дополнительного агента(ов) либо одновременно, либо последовательно с первым агентом.
Способ согласно настоящему изобретению также может представлять собой скрининг, при этом ряд агентов тестируют в отношении модулирования активности ДНК-ДНК-взаимодействия.
Предполагается, что способы анализа согласно настоящему изобретению будут подходящими как в случае скрининга в небольшом масштабе, так и в случае крупномасштабного скрининга агентов, а также в количественных анализах.
Медицинские применения таких терапевтических агентов входят в объем настоящего изобретения, также как программы разработки лекарственных средств как таковых и фармацевтических композиций, содержащих такие агенты. Программа разработки лекарственного средства, например, может включать в себя выбор агента, который идентифицируют или который может быть идентифицирован способами, описанными в данной публикации, необязательно его модификацию (например, модификацию его структуры и/или создание новой композиции, содержащей указанный компонент) и осуществление других исследований (например, исследований токсичности и/или исследований активности, структуры или функции). Испытания могут быть осуществлены на животных, отличных от человека, и, в конце концов, могут быть осуществлены на человеке. Такие испытания в общем будут заключаться в определении эффекта (эффектов) разных уровней доз. В программах разработки лекарственных средств можно использовать компьютеры, чтобы проанализировать компоненты, идентифицированные скринингом (например, чтобы предсказать структуру и/или функцию, чтобы идентифицировать возможных агонистов или антагонистов, чтобы найти другие компоненты, которые могут иметь сходные структуры или функции, и т.д.).
ДИАГНОСТИЧЕСКОЕ ТЕСТИРОВАНИЕ
В настоящее время все еще сложно выявлять различные перестройки генома имеющимися молекулярно-цитогенетическими способами. Хотя методика сравнительной геномной гибридизации на чипе (CGH на чипе) является недавно разработанным способом выявления хромосомной амплификации и/или делеций в геноме с разрешением 35-300 т.п.о., указанная методика не подходит для выявления сбалансированных транслокаций и инверсий в хромосомах. С другой стороны, часто проводят спектральное кариотипирование (SKY) или обычное кариотипирование на материале, полученном от пациента, для выявления хромосомных транслокаций, а также многочисленных изменений, но разрешение для определения точек разрыва в случае транслокации является низким, обычно 10-50 млн.п.о. и 5-10 млн.п.о., соответственно. Поэтому результаты, получаемые обоими способами, и особенно SKY, будут приводить к требующим больших временных затрат и трудоемким подтверждающим экспериментам, подобным флуоресцентной гибридизации in situ (FISH) и методикам молекулярного клонирования точек разрыва.
Способ 4C заключается в осуществлении процедуры, в которой можно выявлять любые хромосомные перестройки на основе измененной частоты взаимодействия между физически связанными последовательностями ДНК. Поэтому способ 4C применим для идентификации (периодических) хромосомных перестроек в случае большинства злокачественных новообразований/множественных аномалий развития или задержки умственного развития у человека. Важное преимущество способа 4C заключается в том, что он позволяет осуществлять очень точное картирование точки разрыва в области, занимающей только несколько тысяч пар оснований. Другое преимущество способа 4C заключается в том, что не требуется предварительных сведений о точном положении точки разрыва, так как точки разрыва могут быть выявлены даже в том случае, когда последовательность - приманка 4C расположена на расстоянии 1-5 млн.п.о. от точки разрыва. Способ также имеет преимущество, состоящее в том, что одна и та же последовательность приманки может быть использована для выявления конкретных хромосомных перестроек, охватывающих большие области точек разрыва. Точное картирование геномных перестроек способом 4C будет в значительной степени облегчать идентификацию аномально экспрессированного гена (генов), лежащего в основе заболеваний или генетических расстройств, что внесет существенный вклад в понимание корреляций генотип-фенотип, поможет в принятии решения при лечении и даст важную дополнительную информацию для прогнозирования.
В одном варианте осуществления настоящего изобретения для обеспечения основы для диагностики или прогнозирования заболевания устанавливают нормальные или стандартные значения для субъекта. Это можно осуществить посредством тестирования образцов, взятых от здоровых субъектов, таких как животные или человек. Частоту ДНК-ДНК-взаимодействия можно количественно оценить посредством сравнения с серийными разведениями позитивных контролей. Затем стандартные значения, полученные для нормальных образцов, можно сравнить со значениями, полученными для образцов от субъектов, которые поражены или потенциально могут быть поражены заболеванием или расстройством. Отклонения значений для субъекта от стандартных значений позволяют установить наличие патологического состояния.
Такие диагностические анализы могут быть адаптированы для оценки эффективности конкретной схемы терапевтического лечения и могут быть использованы в исследованиях на животных, в клинических испытаниях или для того, чтобы контролировать лечение отдельного пациента. Чтобы обеспечить основу для диагностики заболевания, можно установить нормальный или стандартный профиль ДНК-ДНК-взаимодействия. Стандартные значения, полученные для нормальных образцов, можно сравнить со значениями, полученными для образцов от субъектов, которые потенциально могут быть поражены расстройством или заболеванием. Отклонение значений, полученных для субъекта, от стандартных значений позволяет установить наличие патологического состояния. Если устанавливают наличие заболевания, то можно ввести существующее терапевтическое средство и можно получить профиль или значения, характеризующие лечение. Наконец, способ можно повторять регулярно, чтобы оценить, изменяются ли значения или возвращаются ли значения к нормальной или стандартной картине. Последовательные профили лечения можно использовать для того, чтобы показать эффективность лечения в течение периода времени в несколько дней или несколько месяцев.
Способ 4C позволяет точно выявлять по меньшей мере 5 млн.п.о. геномной ДНК, связанной в цис-положении с нуклеотидной последовательностью, которую подвергают анализу (см. фиг. 2-3 и 5). Преимущественно способ 4C можно использовать для выявления любой геномной аберрации, которая сопровождается изменением расстояния между сайтами подвергнутых перестройке последовательностей и выбранной последовательности (приманки) 4C в геноме. Такое изменение, например, может представлять собой увеличение или уменьшение расстояния между сайтами в геноме или может представлять собой уменьшенное представительство (как в случае делеций) или чрезмерное представительство (как в случае дупликаций) последовательностей, расположенных проксимально (например, вплоть до 15 млн.п.о. или более) по отношению к последовательности 4C (приманка). Обычно такие геномные аберрации или перестройки являются причиной или связаны с заболеваниями, такими как злокачественная опухоль (например, лейкоз) и другие наследственные или врожденные заболевания, которые описаны в данной публикации.
Генетические аберрации (например, геномные или хромосомные аберрации, такие как сбалансированные и/или несбалансированные геномные или хромосомные аберрации) включают, без ограничения, перестройки, транслокации, инверсии, инсерции, делеции и другие мутации нуклеиновой кислоты (например, хромосом), а также утрату или добавление части или целых хромосом. Генетические аберрации являются главной причиной генетических расстройств или заболеваний, включая наследственные расстройства и приобретенные заболевания, такие как злокачественные заболевания. Во многие перестройки вовлечены две разных хромосомы. Таким образом, гены (или фрагменты генов) удаляются из нормального физиологического контекста конкретной хромосомы и помещаются в реципиентную хромосому, рядом с не связанными с ними генами или фрагментами генов (часто онкогенами или протоонкогенами).
Злокачественные заболевания могут включать острый лейкоз, злокачественные лимфомы и солидные опухоли. Неограничивающие примеры изменений включают t(14;18), которая часто происходит при NHL; t(12;21), которая часто встречается при ALL из предшественников B-клеток у детей; и наличие аберраций llq23 (ген MLL (миелоидный лимфоидный лейкоз или смешанный лейкоз)) при острых лейкозах.
Ген MLL в области хромосомы llq23 вовлечен в несколько транслокаций как при ALL, так и при острых миелоидных лейкозах (AML). До настоящего времени идентифицировано по меньшей мере десять генов-партнеров. Некоторые из указанных транслокаций, такие как t(4;11) (q21;q23), t(11;19) (q23;p13) и t(1;11) (p32;q23), преимущественно встречаются при ALL, тогда как другие, подобные t(1;11) (q21;q23), t(2;11) (p21;q23), t(6;11) (q27;q23) и t(9;11) (p22;q23) более часто наблюдаются при AML. Перестройки с участием области llq23 очень часто происходят при острых лейкозах у новорожденных (около 60-70%), и в намного меньшей степени при лейкозах у детей и взрослых (в каждом случае около 5%).
В перестройки при злокачественных лимфоидных заболеваниях часто вовлечены гены Ig или TCR. Примеры включают три типа транслокаций (t(8;14), t(2;8) и t(8;22)), которые встречаются при лимфомах Беркитта, при которых ген MYC связан с участками гена тяжелой цепи Ig (IGH), Ig-каппа (IGK) или Ig-лямбда (IGL), соответственно. Другим общим типом транслокации такой категории является t(14;18) (q32;q21), которая наблюдается примерно в 90% случаев фолликулярных лимфом, одним из основных типов NHL. В случае указанной транслокации ген BCL2 в результате перестройки оказывается в областях в пределах локуса IGH в участках гена JH или рядом с ними. Результатом такой хромосомной аберрации является сверхэкспрессия белка BCL2, который играет роль в качестве фактора жизнеспособности в регуляции роста посредством ингибирования запрограммированной гибели клеток.
Ген BCL2 состоит из трех экзонов, но они разбросаны в большой области. Из них последний экзон кодирует большую 3'-нетранслируемую область (3'-UTR). Указанная 3'-UTR представляет собой одну из двух областей, в которых сгруппировано несколько точек разрыва t(14;18), и названа «основной областью точек разрыва»; другая область точек разрыва, вовлеченная в транслокации t(14;18), расположена на 20-30 т.п.о. ниже локуса BCL2 и названа «областью второстепенного кластера». Третья область точек разрыва BCL2, VCR (вариант кластерной области), расположена с 5'-стороны локуса BCL2 и наряду с другими вовлечена в такие варианты транслокации, как t(2;18) и t(18;22), при которых участки генов IGK и IGL являются генами-партнерами.
Таким образом, в качестве примера способ 4C можно применять для скрининга материала, полученного от пациента, в отношении генетических аберраций вблизи локусов и в локусах, которые были выбраны на основе высокой частоты их связи с данным клиническим фенотипом. Дополнительными неограничивающими примерами таких локусов являются AML1, MLL, MYC, BCL, BCR, ABL1, локусы иммуноглобулинов, LYL1, TAL1, TAL2, LMO2, TCRα/δ, TCRβ, HOX и другие локусы при различных лимфобластных лейкозах.
Преимущество состоит в том, что если предполагается генетическая аберрация, то способ 4C можно применять в качестве первого и единственного скрининга для подтверждения и картирования наличия аберрации, как указано в данном описании.
Выявление геномных перестроек
В конкретном предпочтительном варианте осуществления настоящего изобретения способы, описанные в данной публикации, можно использовать для выявления геномных перестроек.
В настоящее время геномные перестройки, такие как точки разрыва при транслокациях, очень трудно выявлять. Например, с помощью микроматриц для сравнительной геномной гибридизации (CGH) можно выявить несколько типов перестроек, но невозможно выявить транслокации. Если у пациента предполагается наличие транслокации, но хромосомы-партнеры неизвестны, можно осуществить спектральное кариотипирование (SKY), чтобы найти партнеров в транслокациях и получить примерную оценку положения точек разрыва. Однако разрешение при этом очень плохое (обычно не лучше чем ~50 млн.п.о.), и обычно требуется дополнительное тонкое картирование (которое является и трудоемким, и дорогостоящим). Обычно это осуществляют с использованием флуоресцентной гибридизации in situ (FISH), которая также дает ограниченное разрешение. С использованием FISH точки разрыва могут быть локализованы при максимальном разрешении в области +/-50 т.п.о.
Частоты ДНК-ДНК-взаимодействий главным образом являются функцией разделения сайтов в геноме, т.е. частоты ДНК-ДНК-взаимодействий обратно пропорциональны линейному расстоянию (в тысячах оснований) между двумя локусами ДНК, присутствующими на одной и той же физической матрице ДНК (Dekker et al., 2002). Таким образом, транслокация, которая создает одну или несколько новых физических матриц ДНК, сопровождается измененными ДНК-ДНК-взаимодействиями вблизи точек разрыва, и указанные взаимодействия можно измерить способом 4C. Заболевания, основанные на транслокациях, обычно вызваны аномальными ДНК-ДНК-взаимодействиями, так как транслокация является результатом физического связывания (взаимодействия) плеч разорванных хромосом (ДНК).
Соответственно, в случае выявления транслокаций можно использовать способ 4C, чтобы идентифицировать такие ДНК-ДНК-взаимодействия, которые различаются у больных и здоровых субъектов.
В качестве примера способ 4C можно применять для скрининга материала, полученного от пациента, в отношении транслокаций вблизи локусов, которые были выбраны на основе их частой связи с данным клиническим фенотипом, которые описаны в данной публикации.
Если у пациента предполагается наличие транслокации, но хромосомные партнеры неизвестны, можно осуществить начальное картирование с использованием имеющихся в настоящее время способов, подобных спектральному кариотипированию (SKY). Таким способом можно идентифицировать партнеров в транслокации и получить очень грубую оценку положения точек разрыва (обычно с разрешением не лучше чем ~50 млн.п.о.). Затем можно применять способ 4C, используя последовательности-«приманки» в данной области, расположенные с интервалами, например, 2 млн.п.о., 5 млн.п.о., 10 млн.п.о., 20 млн.п.о. (или с другими интервалами, которые описаны в данной публикации), чтобы тонко картировать точку разрыва и идентифицировать, например, ген (гены), которые неправильно экспрессированы вследствие транслокации.
Обычно транслокацию идентифицируют на основе резкого перехода от низкой к высокой частоте взаимодействия на другой хромосоме, отличной от хромосомы, содержащей последовательность-приманку 4C, или в другом месте на той же самой хромосоме.
В предпочтительном варианте образец, полученный от субъекта, является образцом в предзлокачественном состоянии.
В предпочтительном варианте образец, полученный от субъекта, состоит из культивируемых или некультивируемых амниоцитов, полученных при амниоцентезе для пренатальной диагностики.
В предпочтительной конструкции чипа зонды, присутствующие на одном чипе, представляют полный геном данного вида с максимальным разрешением. Таким образом, чипы для выявления транслокаций и тому подобного способом 4C содержат зонды, которые описаны в данной публикации, комплементарные каждой стороне сайта узнавания первым ферментом рестрикции в геноме данного вида (например, человека).
В другой предпочтительной конструкции зонды, присутствующие на одном чипе, представляют полный геном данного вида, но не с максимальным разрешением. Таким образом, чипы для выявления транслокаций и тому подобного способом 4C содержат зонды, которые описаны в данной публикации, которые комплементарны только одной стороне каждого сайта узнавания первым ферментом рестрикции в геноме данного вида (например, человека).
В другой предпочтительной конструкции зонды, присутствующие на одном чипе, представляют полный геном данного вида, но не с максимальным разрешением. Таким образом, чипы для выявления транслокаций, делеций, инверсий, дупликаций и других геномных перестроек способом 4C содержат зонды, которые описаны в данной публикации, которые комплементарны одной стороне каждого следующего по порядку второго сайта узнавания первым ферментом рестрикции, который расположен вдоль линейной матрицы генома данного вида (например, человека).
Таким образом, чипы для выявления транслокаций, делеций, инверсий, дупликаций и других геномных перестроек способом 4C содержат зонды, которые описаны в данной публикации, каждый из которых представляет один фрагмент рестрикции, который получен после расщепления первым ферментом рестрикции. Предпочтительно, чип получают, пропуская каждый второй, третий, четвертый, пятый, шестой, седьмой, восьмой, девятый, десятый, двадцатый, тридцатый, сороковой, пятидесятый, шестидесятый, семидесятый, восьмидесятый, девяностый или сотый и т.д. зонд, который гибридизуется с одним и тем же фрагментом рестрикции. Чипы для выявления транслокаций, делеций, инверсий, дупликаций и других геномных перестроек способом 4C могут содержать зонды, которые описаны в данной публикации, которые равномерно распределены вдоль линейных хромосомных матриц. Предпочтительно этого достигают, пропуская один или несколько зондов в таких областях генома, в которых наблюдается наиболее высокая плотность зондов.
В другой предпочтительной конструкции зонды, присутствующие на одном чипе, представляют полный геном данного вида, но не при максимальном разрешении. Таким образом, чипы для выявления транслокаций, делеций, инверсий, дупликаций и других геномных перестроек способом 4C содержат зонды, которые описаны в данной публикации, комплементарные одной стороне каждого третьего, четвертого, пятого, шестого, седьмого, восьмого, девятого, десятого, двадцатого, тридцатого, сорокового, пятидесятого, шестидесятого, семидесятого, восьмидесятого, девяностого или сотого и т.д. сайта узнавания первым ферментом рестрикции, которые расположены по порядку вдоль линейной матрицы генома данного вида (например, человека). Чипы для выявления транслокаций, делеций, инверсий, дупликаций и других геномных перестроек способом 4C могут содержать зонды, которые описаны в данной публикации, которые представляют полный геном, но с использованием одного зонда на каждые 100 тысяч оснований. Чипы для выявления транслокаций, делеций, инверсий, дупликаций и других геномных перестроек способом 4C могут содержать зонды, которые описаны в данной публикации, которые представляют каждый отдельный сайт узнавания первым ферментом рестрикции в геноме, который может быть представлен уникальной последовательностью зонда.
В другой предпочтительной конструкции чипа зонды, которые описаны в данной публикации, на одном чипе представляют геномные области заданного размера, такие как примерно 50 т.п.о., 100 т.п.о., 200 т.п.о., 300 т.п.о., 400 т.п.о., 500 т.п.о., 1 млн.п.о., 2 млн.п.о., 3 млн.п.о., 4 млн.п.о., 5 млн.п.о., 6 млн.п.о., 7 млн.п.о., 8 млн.п.о., 9 млн.п.о. или 10 млн.п.о. (например, около 50 т.п.о. - 10 млн.п.о.), вокруг всех локусов, которые, как известно, вовлечены в транслокации, делеции, инверсии, дупликации и другие геномные перестройки.
В другой предпочтительной конструкции чипа зонды, которые описаны в данной публикации, на одном чипе представляют геномные области заданного размера, такие как примерно 50 т.п.о., 100 т.п.о., 200 т.п.о., 300 т.п.о., 400 т.п.о., 500 т.п.о., 1 млн.п.о., 2 млн.п.о., 3 млн.п.о., 4 млн.п.о., 5 млн.п.о., 6 млн.п.о., 7 млн.п.о., 8 млн.п.о., 9 млн.п.о. или 10 млн.п.о. (например, около 50 т.п.о. - 10 млн.п.о.), вокруг выбранных локусов, которые, как известно, вовлечены в транслокации, делеции, инверсии, дупликации и другие геномные перестройки. Выбор можно осуществить на основе известных критериев, например, они могут представлять только локусы, которые вовлечены в данный тип заболевания.
В другой предпочтительной конструкции чипа зонды, которые описаны в данной публикации, на одном чипе представляют геномную область, которая представляет интерес, например, 100 т.п.о., 200 т.п.о., 300 т.п.о., 400 т.п.о., 500 т.п.о., 600 т.п.о., 700 т.п.о., 800 т.п.о., 900 т.п.о., 1 млн.п.о., 2 млн.п.о., 3 млн.п.о., 4 млн.п.о., 5 млн.п.о., 6 млн.п.о., 7 млн.п.о., 8 млн.п.о., 9 млн.п.о., 10 млн.п.о., 20 млн.п.о., 30 млн.п.о., 40 млн.п.о., 50 млн.п.о., 60 млн.п.о., 70 млн.п.о., 80 млн.п.о., 90 млн.п.о. или 100 млн.п.о. (например, 100 т.п.о. - 10 млн.п.о.) (части) хромосомы или нескольких хромосом, при этом каждый зонд представлен много раз (например, 10, 100, 1000), обеспечивая возможность количественных измерений интенсивностей сигнала гибридизации для каждой последовательности зонда.
В предпочтительной экспериментальной конструкции последовательность 4C (приманка) находится в пределах примерно 0 т.п.о., 10 т.п.о., 20 т.п.о., 30 т.п.о., 40 т.п.о., 50 т.п.о., 100 т.п.о., 200 т.п.о., 300 т.п.о., 400 т.п.о., 500 т.п.о., 1 млн.п.о., 2 млн.п.о., 3 млн.п.о., 4 млн.п.о., 5 млн.п.о., 6 млн.п.о., 7 млн.п.о., 8 млн.п.о., 9 млн.п.о., 10 млн.п.о., 11 млн.п.о., 12 млн.п.о., 13 млн.п.о., 14 млн.п.о. или 15 млн.п.о. (например, примерно 0-15 млн.п.о.) или более от фактически перестроенной последовательности (т.е. точки разрыва в случае транслокации).
В предпочтительном случае гибридизации две по-разному меченые 4C-матрицы, полученные с использованием одной последовательности (4C-приманки) от больного и здорового субъектов, одновременно гибридизуют с одним и тем же чипом. Различия в ДНК-ДНК-взаимодействиях позволяют выявлять точку разрыва в цис-положении (на той же самой хромосоме, что и 4C-приманка) и в транс-положении (на партнере в транслокации).
В предпочтительном случае гибридизации множество дифференциально меченых 4C-матриц, полученных с использованием одной последовательности (4C-приманки) от больного и здорового субъектов, одновременно гибридизуют с одним и тем же чипом. Различия в ДНК-ДНК-взаимодействиях позволяют выявлять точку разрыва в цис-положении (на той же хромосоме, что и 4C-приманка) и в транс-положении (на партнере в транслокации).
Предпочтительно на микроматрицах можно использовать анализ с несколькими окрасками, вместо анализа с двумя окрасками, что обеспечивает возможность одновременной гибридизации более чем двух образцов с одним чипом. Соответственно, гибридизацию с несколькими окрасками можно использовать в способе 4C.
В предпочтительном случае гибридизации множество дифференциально меченых 4C-матриц, полученных с использованием одной последовательности (4C-приманки) от больных субъектов, и одну иным образом меченую 4C-матрицу от здорового субъекта одновременно гибридизуют с одним и тем же чипом. Различия в ДНК-ДНК-взаимодействиях позволяют выявлять точку разрыва в цис-положении (на той же хромосоме, что и 4C-приманка) и в транс-положении (на партнере в транслокации).
В другом предпочтительном случае гибридизации две по-разному меченые 4C-матрицы от одного и того же здорового субъекта, полученные с использованием разных последовательностей (4C-приманок), которые представляют разных возможных партнеров в транслокации, одновременно гибридизуют с одним и тем же чипом. Кластеры интенсивных сигналов гибридизации, наблюдаемые на линейной матрице хромосом, не родственных хромосоме, несущей представляющую интерес последовательность (4C-приманку), будут идентифицировать хромосому-партнера в транслокации и точку разрыва на партнере в транслокации.
В другом предпочтительном случае гибридизации несколько дифференциально меченых 4C-матриц от одного и того же здорового субъекта, полученные с использованием нескольких разных последовательностей (4C-приманок), которые представляют разных возможных партнеров в транслокации, одновременно гибридизуют с одним и тем же зондом. Кластеры интенсивных сигналов гибридизации, наблюдаемые на линейной матрице хромосом, не родственных хромосоме, несущей представляющую интерес последовательность (4C-приманку), будут идентифицировать хромосому-партнера в транслокации и точку разрыва в ней для представляющей интерес последовательности.
Материал, используемый для выявления транслокаций, делеций, инверсий, дупликаций и других геномных перестроек способом 4C, можно получить поперечным сшиванием (и дополнительной обработкой, как описано) живых клеток и/или мертвых клеток, и/или лизатов ядер и/или изолированного хроматина и т.д. (как описано в данной публикации) от больных и/или здоровых субъектов.
Выявление инверсий
Инверсии (например, сбалансированные инверсии) не могут быть выявлены такими способами, как способ сравнительной геномной гибридизации, но могут быть выявлены способом 4C, особенно в том случае, когда (сбалансированная) инверсия находится вблизи (например, примерно вплоть до 1-15 млн.п.о. или более) последовательности 4C (приманки).
Выявление (сбалансированных) инверсий основано на идентификации таких ДНК-ДНК-взаимодействий, которые отличаются у больных и здоровых субъектов. Инверсии будут изменять относительное положение (в тысячах оснований) на физической матрице ДНК всех (но главным образом центрально расположенных) последовательностей в перестроенной области, которое измеряют по отношению к последовательности, расположенной рядом на той же самой хромосоме, которую принимают в качестве последовательности 4C (приманки). Так как частоты ДНК-ДНК-взаимодействия обратно связаны с расстоянием между сайтами в геноме, то у больных субъектов будут получены обратные картины интенсивностей гибридизации для всех зондов, локализованных в подвергнутой перестройке области генома, по сравнению со здоровым субъектом. Таким образом, способ 4C позволяет идентифицировать положение и размер (сбалансированных) инверсий.
Согласно данному аспекту настоящего изобретения предпочтительная специальная конструкция чипа содержит зонды на одном чипе, представляющие области генома заданного размера, например, около 50 т.п.о., 100 т.п.о., 200 т.п.о., 300 т.п.о., 400 т.п.о., 500 т.п.о., 1 млн.п.о., 2 млн.п.о., 3 млн.п.о., 4 млн.п.о., 5 млн.п.о., 6 млн.п.о., 6 млн.п.о., 7 млн.п.о., 8 млн.п.о., 9 млн.п.о. или 10 млн.п.о.) (например, 50 т.п.о. - 10 млн.п.о.), вокруг локуса, в котором предполагается инверсия или другая перестройка.
В другой предпочтительной специальной конструкции чипа зонды на одном чипе представляют области генома заданного размера (50 т.п.о., 100 т.п.о., 200 т.п.о., 300 т.п.о., 400 т.п.о., 500 т.п.о., 1 млн.п.о., 2 млн.п.о. и т.д.) вокруг локуса, в котором предполагается инверсия или другая перестройка. Для достоверного количественного анализа интенсивностей сигнала количество зондов, присутствующих на чипе, обычно находится в большом избытке по сравнению с количеством родственных фрагментов, которые гибридизуют с чипом. Поэтому может требоваться присутствие каждого зонда на чипе несколько раз (например, 10, 20, 50, 100, 1000 раз и т.д.). Кроме того, может быть необходимо титровать количество матрицы, которое надо гибридизовать с чипом.
Выявление делеций
Выявление делеций основано на идентификации таких ДНК-ДНК-взаимодействий, которые отличаются у больных и здоровых субъектов. Делеции будут приводить к отсутствию ДНК-взаимодействий с последовательностью 4C (приманкой), расположенной вблизи (например, примерно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15 млн.п.о. или более) делетированной области. Это может приводить к полному отсутствию сигналов гибридизации всех зондов, локализованных в подвергнутой перестройке области, если делеция имеется в обоих аллелях (гомозиготная), или к уменьшению интенсивностей сигнала у больного субъекта по сравнению со здоровым, если делеция имеется только в одном аллеле (гетерозиготная). Делеция переводит более отдаленные последовательности ближе на физической матрице ДНК к анализируемой последовательности 4C (к приманке), что приведет к более интенсивным сигналам гибридизации для зондов, локализованных непосредственно после делетированной области.
Выявление дупликации (дупликаций)
Выявление дупликации обычно основано на идентификации таких ДНК-ДНК-взаимодействий, которые отличаются у больных и здоровых субъектов. Зонды в дуплицированной области будут давать увеличенные сигналы гибридизации с последовательностью 4C (приманкой), расположенной вблизи (например, примерно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15 млн.п.о. или больше) подвергнутой перестройке области, по сравнению с сигналами, полученными для контрольного здорового субъекта. Зонды за пределами дуплицированной области дополнительно отдаляются от последовательности 4C и, следовательно, будут давать пониженные сигналы гибридизации по сравнению с сигналами у контрольного здорового субъекта.
Предпочтительно, повышение или понижение частоты ДНК-ДНК-взаимодействия в образце от субъекта по сравнению с контролем является показателем дупликации или инсерции.
Предпочтительно, повышение частоты ДНК-ДНК-взаимодействия в образце от субъекта по сравнению с контролем и/или понижение частоты ДНК-ДНК-взаимодействия для более отдаленных областей является показателем дупликации или инсерции.
Пренатальная диагностика
Предпочтительно способ 4C также можно использовать в пренатальной диагностике.
Нуклеиновая кислота может быть получена из плода с использованием различных способов, которые известны в данной области. В качестве примера можно использовать амниоцентез, чтобы получить амниотическую жидкость, из которой выделяют клетки плода в суспензии и культивируют в течение нескольких дней (Mercier and Bresson (1995) Ann. Gnt., 38, 151-157). Затем из клеток можно выделить нуклеиновую кислоту. Сбор хорионических ворсинок может обеспечить возможность обойтись без стадии культивирования и избежать сбора амниотической жидкости. Указанные способы можно применять раньше (до 7 недель беременности в случае сбора хорионических ворсинок и 13-14 недель в случае амниоцентеза), но с немного повышенным риском выкидыша.
Прямой сбор крови плода на уровне пуповины также можно использовать для получения нуклеиновой кислоты, но обычно для этого требуется бригада врачей, специализирующихся в данной методике (Donner et al., (1996) Fetal Diagn. Ther., 10, 192-199).
На данной стадии преимущественно можно выявить генетические аберрации (например, геномные или хромосомные аберрации), такие как перестройки, транслокации, инверсии, инсерции, делеции и другие мутации в хромосомах и нуклеиновой кислоте.
Предпочтительно могут быть выявлены генетические аберрации (например, геномные или хромосомные аберрации), такие как перестройки, транслокации, инверсии, инсерции, делеции и другие мутации в хромосомах 21, 18, 13, X или Y, а также потери или добавления части или целых хромосом 21, 18, 13, X или Y, так как указанные хромосомы являются хромосомами, в которых происходит большинство аберраций у плода.
Определение геномных сайтов интеграции
Способ 4C также позволяет определять геномные сайты интеграции вирусов и трансгенов, и т.д., а также случая, когда множество копий встроены в разных положениях в геноме (как описано на фиг.4).
Определение предрасположенности к появлению некоторых транслокаций
Предпочтительно способ 4C также можно применять по отношению к здоровым субъектам для измерения геномного окружения локусов, которые часто вовлечены в генетические аберрации. Указанным способом можно определить предрасположенность субъекта к появлению у него некоторых генетических аберраций.
Таким образом, кроме медицинских применений, описанных в данной публикации, настоящее изобретение можно применять для диагностики.
СУБЪЕКТ
Термин «субъект» включает млекопитающих - таких как животные и человек.
АГЕНТ
Агентом может быть органическое соединение или другое химическое вещество. Агент может представлять собой соединение, которое может быть получено или которое получают из любого подходящего источника, либо природного, либо искусственного. Агентом может быть молекула аминокислоты, полипептид или их химическое производное или их комбинация. Агентом может быть также молекула полинуклеотида, которая может быть смысловой или антисмысловой молекулой, или антитело, например, поликлональное антитело, моноклональное антитело или моноклональное гуманизированное антитело.
Разработаны различные методики получения моноклональных антител, обладающих свойствами антител человека, которые не требуют использования линии клеток человека, продуцирующих антитела. Например, подходящие моноклональные антитела мыши были «гуманизированы» посредством связывания вариабельных областей грызунов и константных областей человека (Winter G. and Milstein C. (1991) Nature 349, 293-299). Это снижает иммунногеность антитела, связанную с ответом иммунной системы человека на антитело мыши, но остаточная иммуногенность сохраняется из-за чужеродного каркаса V-области. Кроме того, специфичность связывания антигена по существу такая, как у мышиного донора. Прививки CDR и обработка каркаса (EP 0239400) усовершенствовали и улучшили обработку антител по существу так, что стало возможным получение гуманизированных мышиных антител, которые приемлемы для терапевтического применения на человеке. Гуманизированные антитела могут быть получены с использованием других способов, хорошо известных в данной области (например, как описано в US-A-239400).
Агенты могут быть связаны со структурной единицей (например, органической молекулой) линкером, который может представлять собой гидролизируемый бифункциональный линкер.
Указанная структурная единица может быть сконструирована или получена из библиотеки соединений, которая может содержать пептиды, а также другие соединения, такие как малые органические молекулы.
В качестве примера структурная единица может быть природным веществом, биологической макромолекулой или экстрактом, полученным из биологических материалов, таких как бактерии, грибы или клетки или ткани животных (в частности, млекопитающих), органической или неорганической молекулой, синтетическим агентом, полусинтетическим агентом, структурным или функциональным миметиком, пептидом, пептидомиметиком, пептидом, полученным в результате расщепления целого белка, или пептидами, полученными синтетически (например, либо с использованием синтезатора пептидов, либо рекомбинантными способами, либо их комбинацией), рекомбинантным агентом, антителом, природным или неприродным агентом, слитым белком или их эквивалентами и мутантами, производными или комбинациями указанного.
Обычно структурная единица будет представлять собой органическое соединение. В некоторых случаях органические соединения будут содержать две или более углеводородных группы. В данном описании термин «углеводородная группа» означает группу, содержащую по меньшей мере C и H, и которая необязательно может содержать один или несколько других подходящих заместителей. Примеры таких заместителей могут включать галоген, алкоксигруппу, нитрогруппу, алкильную группу, циклическую группу и т.д. Кроме возможности того, что заместители представляют собой циклическую группу, комбинация заместителей может образовывать циклическую группу. Если углеводородная группа содержит более одного атома C, то такие атомы углерода не должны быть обязательно связаны друг с другом. Например, по меньшей мере два атома углерода могут быть связаны посредством подходящего элемента или группы. Таким образом, углеводородная группа может содержать гетероатомы. Подходящие гетероатомы будут очевидны для специалистов в данной области и включают, например, серу, азот и кислород. Для некоторых применений структурная единица предпочтительно содержит по меньшей мере одну циклическую группу. Циклическая группа может быть полициклической группой, такой как неконденсированная полициклическая группа. Для некоторых применений структурная единица содержит по меньшей мере одну из указанных циклических групп, связанную с другой углеводородной группой.
Структурная единица может содержать группы галогена, такие как фтор, хлор, бром или иод.
Структурная единица может содержать одну или несколько групп: алкил, алкоксигруппу, алкенил, алкинил и алкенилен, которые могут иметь неразветвленную или разветвленную цепь.
ПРОЛЕКАРСТВО
Специалистам в данной области будет понятно, что структурная единица может быть образована из пролекарства. Примеры пролекарств включают некоторые защищенные группы, которые могут не обладать фармакологической активностью как таковые, но в некоторых случаях могут быть введены (например, перорально или парентерально) и затем метаболизированы в организме до формы структурной единицы, которая является фармакологически активной.
Подходящие пролекарства могут включать, без ограничения, доксорубицин, митомицин, феноловый иприт, метотрексат, антифолаты, хлорамфеникол, камптотецин, 5-фторурацил, цианид, хинин, дипиридамол и паклитаксел.
Кроме того, будет понятно, что некоторые остатки, известные как «про-остатки», которые, например, описаны в «Design of Prodrugs» by H. Bundgaard, Elsevier, 1985, могут быть помещены на соответствующие функциональные группы агентов. Такие пролекарства также включены в объем изобретения.
Агент может быть в форме фармацевтически приемлемой соли, такой как кислотно-аддитивная соль или соль основания, или в форме сольватов, включая их гидраты. Обзор подходящих солей см. в Berge et al., J. Pharm. Sci., 1977, 66, 1-19.
Агент может быть способен проявлять другие терапевтические свойства.
Агент можно использовать в сочетании с одним или несколькими другими фармацевтически активными агентами.
Если вводят комбинации активных агентов, то комбинации активных агентов можно вводить одновременно, отдельно или последовательно.
СТЕРЕОИЗОМЕРЫ И ГЕОМЕТРИЧЕСКИЕ ИЗОМЕРЫ
Структурная единица может существовать в виде стереоизомеров и/или геометрических изомеров - например, структурная единица может иметь один или несколько асимметричных и/или геометрических центров и, таким образом, может существовать в двух или более стереоизомерных и/или геометрических формах. Настоящее изобретение охватывает применение всех отдельных стереоизомеров и геометрических изомеров указанных структурных единиц и их смесей.
ФАРМАЦЕВТИЧЕСКАЯ СОЛЬ
Агент можно вводить в форме фармацевтически приемлемой соли.
Фармацевтически приемлемые соли хорошо известны специалистам в данной области и, например, включают соли, указанные в Berge et al., J. Pharm. Sci., 66, 1-19 (1977). Подходящие кислотно-аддитивные соли образованы из кислот, которые образуют нетоксичные соли, и включают гидрохлорид, гидробромид, гидроиодид, нитрат, сульфат, бисульфат, фосфат, вторичный кислый фосфат, ацетат, трифторацетат, глюконат, лактат, салицилат, цитрат, тартрат, аскорбат, сукцинат, малеат, фумарат, глюконат, формиат, бензоат, метансульфонат, этансульфонат, бензолсульфонат и п-толуолсульфонат.
В том случае, когда присутствует один или несколько остатков кислоты, могут быть образованы подходящие фармацевтически приемлемые основно-аддитивные соли из оснований, которые образуют нетоксичные соли, и такие соли включают соли алюминия, кальция, лития, магния, калия, натрия, цинка и фармацевтически активных аминов, таких как диэтаноламин.
Фармацевтически приемлемую соль агента можно легко получить смешиванием вместе растворов агента и требуемой в соответствующем случае кислоты или основания. Соль может выпадать в осадок из раствора, и ее можно собирать фильтрованием или можно извлечь, выпаривая растворитель.
Агент может существовать в полиморфных формах.
Агент может содержать один или несколько асимметричных атомов углерода, и поэтому существует в двух или более стереоизомерных формах. В том случае, когда агент содержит группу алкенила или алкенилена, также может иметь место цис-(E) и транс-(Z) изомерия. Настоящее изобретение включает отдельные стереоизомеры агента, и в некоторых случаях отдельные таутомерные формы, а также их смеси.
Разделение диастереоизомеров или цис- и транс-изомеров можно осуществить обычными способами, например, фракционной кристаллизацией, хроматографией или ВЭЖХ смеси стереоизомеров агента или его подходящей соли или производного. Также можно получить отдельный энантиомер агента из соответствующего оптически чистого промежуточного продукта или разделением, таким как разделение с помощью ВЭЖХ, соответствующего рацемата с использованием подходящего хирального носителя, или фракционной кристаллизацией диастереоизомерных солей, образованных при взаимодействии соответствующего рацемата с подходящей оптически активной кислотой или основанием, в зависимости от ситуации.
Агент также может включать все подходящие изотопные варианты агента или его фармацевтически приемлемой соли. Изотопный вариант агента или его фармацевтически приемлемой соли определяют как вариант, в котором по меньшей мере один атом заменен атомом, имеющим такое же атомное число, но имеет атомную массу, отличающуюся от атомной массы, обычно встречающейся в природе. Примеры изотопов, которые могут быть введены в агент и его фармацевтически приемлемые соли, включают изотопы водорода, углерода, азота, кислорода, фосфора, серы, фтора и хлора, такие как2H,3H,13C,14C,15N,17O,18O,31P,32P,35S,18F и36Cl, соответственно. Некоторые изотопные варианты агента и его фармацевтически приемлемых солей, например, таких, в которые введен такой радиоактивный изотоп, как3H или14C, применимы в исследованиях тканевого распределения лекарственного средства и/или субстрата. Изотопы трития, т.е.3H, и углерода-14, т.е.14C, особенно предпочтительны благодаря тому, что их легко получать и регистрировать. Кроме того, замена изотопами, такими как дейтерий, т.е.2H, может давать некоторые терапевтические преимущества в результате более высокой метаболической стабильности, например, из-за увеличенного времени полужизни in vivo или уменьшенных потребностей в дозе, и поэтому может быть предпочтительной в некоторых случаях. Изотопные варианты агента и его фармацевтически приемлемых солей согласно настоящему изобретению в общем могут быть получены обычными способами с использованием подходящих изотопных вариантов соответствующих реагентов.
ФАРМАЦЕВТИЧЕСКИ АКТИВНАЯ СОЛЬ
Агент можно вводить в виде фармацевтически приемлемой соли. Обычно фармацевтически приемлемая соль может быть легко получена с использованием требуемой кислоты или основания, в зависимости от ситуации. Соль может выпадать в осадок из раствора и может быть собрана фильтрованием, или может быть извлечена выпариванием растворителя.
СПОСОБЫ ХИМИЧЕСКОГО СИНТЕЗА
Агент может быть получен способами химического синтеза.
Специалистам в данной области будет понятно, что может быть необходимо защищать чувствительные функциональные группы и удалять их защиту во время синтеза соединения согласно изобретению. Защиту и удаление защиты осуществляют обычными способами, например, как описано в «"Protective Groups in Organic Synthesis», T.W. Greene and P.G.M. Wuts, John Wiley and Sons Inc. (1991), и в P.J. Kocienski, «Protecting Groups», Georg Thieme Verlag (1994).
В ходе некоторых реакций возможно, что какие-либо присутствующие стереоцентры в определенных условиях могут быть рацемизированы, например, если используют основание в реакции с субстратом, имеющим оптический центр, содержащий чувствительную к основанию группу. Указанное возможно, например, на стадии гуанилирования. Как хорошо известно в данной области, потенциальные проблемы, такие как в указанном случае, можно обойти за счет выбора последовательности реакций, условий, реагентов, схем защиты/удаления защиты и т.д.
Соединения и соли могут быть разделены и очищены обычными способами.
Разделение диастереомеров можно осуществлять обычными способами, например, фракционной кристаллизацией, хроматографией или ВЭЖХ смеси стереоизомеров соединения формулы (I) или его подходящей соли или производного. Также можно получить отдельный энантиомер соединения формулы (I) из соответствующего оптически чистого промежуточного продукта или разделением, таким как разделение с помощью ВЭЖХ, соответствующего рацемата с использованием подходящего хирального носителя, или фракционной кристаллизацией диастереомерных солей, образованных при взаимодействии соответствующего рацемата с подходящей оптически активной кислотой или основанием.
Агент может быть получен с использованием химических способов синтеза агента целиком или частями. Например, если агент содержит пептид, то пептид может быть синтезирован способом твердофазного синтеза, отщеплен от смолы и очищен препаративной высокоэффективной жидкостной хроматографией (например, Creighton (1983) Proteins Structures and Molecular Principles, W.H. Freeman and Co, New York NY). Состав синтетических пептидов можно подтвердить анализом аминокислот или секвенированием (например, способом деградации по Эдману; Creighton, выше).
Синтез пептидных ингибирующих агентов (или их вариантов, гомологов, производных, фрагментов или миметиков) можно осуществить с использованием различных твердофазных способов (Roberge J.Y. et al. (1995) Science 269: 202-204), и можно проводить автоматизированный синтез, например, с использованием синтезатора пептидов ABI 431 A (Perkin Elmer) согласно инструкциям, предлагаемым производителем. Кроме того, аминокислотные последовательности, составляющие агент, могут быть изменены во время прямого синтеза и/или могут быть объединены с использованием химических способов с последовательностью других субъединиц или их любых частей, чтобы получить вариант агента.
ХИМИЧЕСКОЕ ПРОИЗВОДНОЕ
Термин «производное» или «дериватизированный» в используемом в данном описании смысле включает химическую модификацию агента. Иллюстрацией таких химических модификаций может быть замена водорода группой галогена, алкильной группой, ацильной группой или аминогруппой.
ХИМИЧЕСКАЯ МОДИФИКАЦИЯ
Агент может представлять собой модифицированный агент, такой как, без ограничения, химически модифицированный агент.
Химическая модификация агента может либо усиливать, либо уменьшать взаимодействие за свет водородных связей, взаимодействие зарядов, гидрофобное взаимодействие Ван-дер-Ваальса или взаимодействие диполей.
В одном аспекте агент может действовать в качестве модели (например, матрицы) для разработки других соединений.
ФАРМАЦЕВТИЧЕСКИЕ КОМПОЗИЦИИ
В следующем аспекте предлагается фармацевтическая композиция, содержащая агент, идентифицированный способом анализа, описанным в данной публикации, в смеси с фармацевтически приемлемым носителем, разбавителем, эксципиентом или адъювантом и/или их комбинации.
В следующем аспекте предлагается композиция вакцины, содержащая агент.
В следующем аспекте предлагается способ приготовления фармацевтической композиции, включающий в себя смешивание агента, идентифицированного в анализе, с фармацевтически приемлемым разбавителем, носителем, эксципиентом или адъювантом и/или их комбинациями.
В следующем аспекте предлагается способ профилактики и/или лечения заболевания, включающий в себя введение агента или фармацевтической композиции или вакцины субъекту.
Фармацевтические композиции могут быть предназначены для применения на человеке или животном в медицине и ветеринарии и, как правило, будут содержать любой один или несколько фармацевтически приемлемых разбавителей, носителей или эксципиентов. Приемлемые носители или разбавители для терапевтического применения хорошо известны в фармацевтической области и описаны, например, в Remington's Pharmaceutical Sciences, Mack Publishing Co. (A. R. Gennaro edit. 1985). Выбор фармацевтического носителя, эксципиента или разбавителя может быть осуществлен на основании планируемого пути введения и стандартной фармацевтической практики. Фармацевтические композиции могут содержать в качестве или в дополнение к носителю, эксципиенту или разбавителю любой подходящий связывающий агент (агенты), скользящее вещество (вещества), суспендирующий агент (агенты), покрывающий агент (агенты), солюбилизатор (солюбилизаторы).
В фармацевтической композиции могут присутствовать консерванты, стабилизаторы, красители и корригенты. Примеры консервантов включают бензоат натрия, сорбиновую кислоту и сложные эфиры п-гидроксибензойной кислоты. Также можно использовать антиоксиданты и суспендирующие агенты.
Могут предъявляться различные требования к композициям/препаратам, в зависимости от разных систем доставки. В качестве примера может быть приготовлена фармацевтическая композиция согласно настоящему изобретению для введения с использованием мининасоса или через слизистую оболочку, например, в виде назального спрея или аэрозоля для ингаляции, или принимаемого вовнутрь раствора, или для введения парентерально, при этом композицию готовят в инъекционной форме для доставки, например, внутривенным, внутримышечным или подкожным путем. Альтернативно может быть создан препарат для введения несколькими путями.
Если агент необходимо вводить через слизистую оболочку желудочно-кишечного тракта, то он должен обладать способностью оставаться стабильным во время прохождения желудочно-кишечного тракта; например, он должен быть резистентным к протеолитическому расщеплению, стабильным в кислых условиях pH и резистентным к влиянию детергентов желчи.
В соответствующих случаях фармацевтические композиции могут быть введены посредством ингаляции, в форме суппозитория или пессария, местно в форме примочки, раствора, крема, мази или опудривающего средства, с использованием кожного пластыря, перорально в форме таблеток, содержащих такие эксципиенты, как крахмал или лактоза, или в виде капсул или овальных таблеток, либо отдельно, либо в смеси с эксципиентами, или в форме эликсиров, растворов или суспензий, содержащих корригенты или красители, или фармацевтические композиции могут быть инъецированы парентерально, например, внутривенно, внутримышечно или подкожно. Для парентерального введения композиции лучше всего могут быть использованы в форме стерильного водного раствора, который может содержать другие вещества, например, достаточное количество солей или моносахаридов, чтобы сделать раствор изотоничным крови. В случае буккального или подъязычного введения композиции могут быть введены в форме таблеток или лепешек, которые могут быть приготовлены обычным способом.
Агенты можно использовать в сочетании с циклодекстрином. Известно, что циклодекстрины образуют комплексы включения и комплексы, не являющиеся комплексами включения, с молекулами лекарственных средств. Образование комплекса лекарственное средство-циклодекстрин может модифицировать растворимость, скорость растворения, биодоступность и/или свойство стабильности молекулы лекарственного средства. Комплексы лекарственное средство-циклодекстрин в общем применимы для большинства дозированных форм и путей введения. В качестве альтернативы прямому образованию комплекса с лекарственным средством циклодекстрин можно использовать в качестве вспомогательной добавки, например, в качестве носителя, разбавителя или солюбилизатора. Наиболее широко используют альфа-, бета- и гамма-циклодекстрины, и подходящие примеры описаны в WO-A-91/11172, WO-A-94/02518 и WO-A-98/55148.
Если агент является белком, то указанный белок может быть получен in situ у субъекта, подвергаемого лечению. В данном случае нуклеотидные последовательности, кодирующие указанный белок, могут быть доставлены с помощью способов без применения вирусов (например, с использованием липосом) и/или основанными на вирусах способами (например, с использованием ретровирусных векторов), для того чтобы указанный белок экспрессировался с указанной нуклеотидной последовательности.
Фармацевтические композиции согласно настоящему изобретению также можно использовать в сочетании с традиционными способами лечения.
ВВЕДЕНИЕ
Термин «введенный» включает доставку основанными на вирусах и невирусными способами. Механизмы вирусной доставки включают, без ограничения, доставку аденовирусными векторами, векторами на основе аденоассоциированных вирусов (AAV), векторами на основе вируса герпеса, ретровирусными векторами, лентивирусными векторами и бакуловирусными векторами. Механизмы невирусной доставки включают опосредованную липидами трансфекцию, липосомы, иммунолипосомы, липофектин, катионные поверхностные амфифилы (CFA) и их комбинации.
Компоненты можно вводить отдельно, но, как правило, их будут вводить в виде фармацевтической композиции, например, когда компоненты находятся в смеси с подходящим фармацевтическим эксципиентом, разбавителем или носителем, выбранным в соответствии с предполагаемым путем введения и стандартной фармацевтической практикой.
Например, компоненты могут быть введены в форме таблеток, капсул, овальных таблеток, эликсиров, растворов или суспензий, которые могут содержать корригенты или красители, для применений посредством немедленного, замедленного, модифицированного, длительного, импульсного или контролируемого высвобождения.
Если фармацевтический препарат представляет собой таблетку, то таблетка может содержать такие эксципиенты, как микрокристаллическая целлюлоза, лактоза, цитрат натрия, карбонат кальция, двухосновный фосфат кальция и глицин, дезинтегрирующие агенты, такие как крахмал (предпочтительно кукурузный, картофельный или крахмал, полученный из тапиоки), крахмалгликолят натрия, кроскармелоза натрия и некоторые комплексные силикаты, и связывающие агенты для гранулирования, такие как поливинилпирролидон, гидроксипропилметилцеллюлоза (HPMC), гидроксипропилцеллюлоза (HPC), сахароза, желатин и аравийская камедь. Кроме того, могут быть введены скользящие вещества, такие как стеарат магния, стеариновая кислота, глицерилбехенат и тальк.
Твердые композиции сходного типа также могут быть использованы в качестве наполнителей в желатиновых капсулах. Предпочтительные эксципиенты в данном случае включают лактозу, крахмал, целлюлозу, молочный сахар или высокомолекулярные полиэтиленгликоли. В случае водных суспензий и/или эликсиров, агент может быть в сочетании с разными подсластителями или корригентами, красящими веществами или красителями, эмульгаторами и/или суспендирующими агентами и разбавителями, такими как вода, этанол, пропиленгликоль и глицерин и их комбинации.
Пути введения (доставки) могут включать, без ограничения, один или несколько путей: пероральный (например, в виде таблетки, капсулы или в виде принимаемого вовнутрь раствора), местный, через слизистую оболочку (например, в виде назального спрея или аэрозоля для ингаляции), назальный, парентеральный (например, в виде инъекционной формы), желудочно-кишечный, интраспинальный, внутрибрюшинный, внутримышечный, внутривенный, внутриматочный, внутриглазной, интрадермальный, внутричерепной, внутритрахеальный, внутривагинальный, интрацеребровентрикулярный, интрацеребральный, подкожный, глазной (включая введение в стекловидное тело или в камеры), трансдермальный, ректальный, трансбуккальный, вагинальный, эпидуральный, подъязычный.
УРОВНИ ДОЗ
Обычно лечащий врач определяет реальную дозу, которая будет наиболее подходящей для отдельного субъекта. Конкретный уровень дозы и частота дозирования для конкретного пациента может варьировать и будет зависеть от множества факторов, включая активность конкретного используемого соединения, метаболическую стабильность и продолжительность действия данного соединения, возраст, массу тела, общее состояние здоровья, пол, режим питания, способ и время введения, скорость выведения, комбинацию лекарственных средств, тяжесть конкретного состояния и конкретного человека, подвергаемого лечению.
ПРИГОТОВЛЕНИЕ КОМПОЗИЦИИ
Компонент(ты) могут быть приготовлены в виде фармацевтической композиции, например, смешиванием с одним или несколькими подходящими носителями, разбавителями или эксципиентами способами, которые известны в данной области.
ЗАБОЛЕВАНИЕ
Аспекты настоящего изобретения могут быть использованы для лечения и/или профилактики и/или диагностики и/или прогнозирования заболеваний, например, таких, которые перечислены в WO-A-98/09985.
Для облегчения часть из указанного списка приведена ниже: ингибирующая макрофаги и/или ингибирующая T-клетки активность и таким образом, противовоспалительная активность; противоиммунная активность, т.е. ингибирующее влияние, направленное против клеточного и/или гуморального иммунного ответа, включая ответ, не связанный с воспалением; заболевания, связанные с вирусами и/или другими внутриклеточными патогенами; ингибирование способности макрофагов и T-клеток прилипать к внеклеточному матриксу и фибронектину, а также повышенной экспрессии fas-рецептора в T-клетках; ингибирование нежелательного иммунного ответа и воспаления, включая артрит, включая ревматоидный артрит, воспаление, связанное с гиперчувствительностью, аллергические реакции, астму, системную красную волчанку, коллагеновые заболевания и другие аутоиммунные заболевания, воспаление, связанное с атеросклерозом, артериосклероз, атеросклеротическую болезнь сердца, реперфузионное повреждение, остановку сердца, инфаркт миокарда, воспалительные заболевания сосудов, респираторный дистресс-синдром или другие сердечно-легочные заболевания, воспаление, связанное с пептической язвой, язвенный колит и другие заболевания желудочно-кишечного тракта, фиброз печени, цирроз печени или другие заболевания печени, тиреоидит или другие заболевания желез, гломерулонефрит или другие почечные и урологические болезни, отит или другие оториноларингологические болезни, дерматит или другие кожные заболевания, периодонтальные заболевания или другие болезни зубов, орхит или орхоэпидидимит, бесплодие, травма яичка или другие связанные с иммунной системой болезни семенников, дисфункцию плаценты, плацентарную недостаточность, привычный аборт, эклампсию, предэклампсию и другие иммунные и/или связанные с воспалением гинекологические заболевания, задний увеит, интермедиарный увеит, передний увеит, конъюнктивит, хориоретинит, увеоретинит, неврит зрительного нерва, внутриглазное воспаление, например, ретинит или кистоидный отек желтого пятна, симпатическую офтальмию, склерит, пигментную дегенерацию сетчатки, иммунные и воспалительные компоненты заболевания дегенеративного генеза, воспалительные компоненты при травме глаза, воспаление глаза, вызванное инфекцией, пролиферативные витреоретинопатии, острую ишемическую оптическую невропатию, избыточное рубцевание, например, после фильтрационной операции по поводу глаукомы, иммунную и/или воспалительную реакцию против глазных имплантатов и другие иммунные и связанные с воспалением глазные болезни, воспаление, связанное с аутоиммунными заболеваниями или состояниями или расстройствами, при которых может быть полезной иммуносупрессия и/или подавление воспаления как в центральной нервной системе (ЦНС), так и в любом другом органе, болезнь Паркинсона, осложнение и/или побочные эффекты лечения болезни Паркинсона, связанный со СПИДом комплекс деменции, ВИЧ-ассоциированную энцефалопатию, болезнь Девика, хорею Сиденгама, болезнь Альцгеймера и другие дегенеративные заболевания, состояния или расстройства ЦНС, воспалительные компоненты инсульта, постполиомиелитный синдром, иммунные и воспалительные компоненты психических расстройств, миелит, энцефалит, подострый склерозирующий пан-энцефалит, энцефаломиелит, острую невропатию, подострую невропатию, хроническую невропатию, синдром Гийена-Барре, хорею Сиденгама, бульбоспинальный паралич, ложную опухоль головного мозга, синдром Дауна, болезнь Гентингтона, боковой амиотрофический склероз, воспалительные компоненты компрессии ЦНС или травмы ЦНС или инфекции ЦНС, воспалительные компоненты мышечной атрофии и дистрофии, и связанные с иммунной системой и воспалением заболевания, состояния или расстройства центральной и периферической нервной систем, посттравматическое воспаление, септический шок, инфекционные болезни, воспалительные осложнения или побочные эффекты хирургии, трансплантацию костного мозга или другие трансплантационные осложнения и/или побочные эффекты, воспалительные и/или иммунные осложнения и побочные эффекты генной терапии, например, вследствие инфекции вирусным носителем, или воспаление, связанное со СПИдом, для подавления или ингибирования гуморального и/или клеточного иммунного ответа, для лечения или улучшения состояния в случае заболеваний, связанных с пролиферацией моноцитов или лейкоцитов, например, лейкоза, посредством снижения количества моноцитов или лимфоцитов, для профилактики и/или лечения отторжения трансплантата в случаях трансплантации природных или искусственных клеток, ткани и органов, таких как роговица, костный мозг, органы, линзы, электрокардиостимуляторы, природная или искусственная кожа. Специфичные связанные со злокачественной опухолью расстройства включают без ограничения: солидные опухоли; наследственные опухоли крови, такие как лейкозы; метастазы опухолей; доброкачественные опухоли, например, гемангиомы, невромы слухового нерва, нейрофибромы, трахомы и пиогенные гранулемы; ревматоидный артрит; псориаз; ангиогенные болезни глаз, например, диабетическую ретинопатию, ретинопатию недоношенных, дегенерацию желтого пятна, отторжение трансплантата роговицы, неоваскулярную глаукому, ретролентальную фиброплазию, покраснение радужки; синдром Ослера-Вебера; ангиогенез миокарда; неоваскуляризацию бляшек; телеангиэктазию; поражение суставов при гемофилии; ангиофиброму; гранулирование ран; коронарные коллатерали; коллатерали головного мозга; артериовенозные мальформации; ангиогенез в ишемизированной конечности; неоваскулярную глаукому; ретролентальную фиброплазию; диабетическую неоваскуляризацию; связанные с гелиобактериями заболевания, переломы, васкулогенез, гемопоэз, овуляцию, менструацию и плацентацию.
Предпочтительно заболевание представляет собой злокачественную опухоль - такую как острый лимфоцитарный лейкоз (ALL), острый миелоидный лейкоз (AML), злокачественную опухоль коры надпочечников, рак заднего прохода, рак мочевого пузыря, рак крови, рак костей, опухоль головного мозга, рак молочной железы, рак женской половой системы, рак мужской половой системы, лимфому центральной нервной системы, рак шейки матки, рабдомиосаркому у детей, саркому у детей, хронический лимфоцитарный лейкоз (CLL), хронический миелоидный лейкоз (CML), рак прямой и ободочной кишки, рак ободочной кишки, рак эндометрия, саркому эндометрия, рак пищевода, рак глаз, рак желчного пузыря, рак желудка, рак желудочно-кишечного тракта, лейкоз ворсистых клеток, рак головы и шеи, печеночно-клеточный рак, болезнь Ходжкина, гипофарингеальный рак, саркому Копоши, рак почки, рак гортани, лейкоз, рак печени, рак легкого, злокачественную фиброзную гистиоцитому, злокачественную тимому, меланому, мезотелиому, множественную миелому, миелому, рак носовой полости и околоносовой пазухи, рак носоглотки, рак нервной системы, нейробластому, неходжкинскую лимфому, рак ротовой полости, рак ротоглотки, остеосаркому, рак яичников, рак поджелудочной железы, рак околощитовидной железы, рак пениса, рак глотки, рак гипофиза, неоплазму плазматических клеток, первичную лимфому ЦНС, рак простаты, рак прямой кишки, рак дыхательной системы, ретинобластому, рак слюнных желез, рак кожи, рак тонкого кишечника, саркому мягких тканей, рак желудка, рак семенников, рак щитовидной железы, рак мочевой системы, саркому матки, рак влагалища, опухоли сосудистой системы, макроглобулинемию Вальденстрема и опухоль Вильмса.
НАБОРЫ
Материалы для применения в способах согласно настоящему изобретению, хорошо подходят для приготовления наборов.
Такой набор может содержать емкости, в каждой из которых находится один или несколько различных реагентов (обычно в концентрированной форме), используемых в способах, описанных в данной публикации, включая, например, первый фермент рестрикции, второй фермент рестрикции, поперечно сшивающий агент, фермент для лигирования (например, лигазу) и агент для удаления поперечных сшивок (например, протеиназу K).
В емкостях также могут быть представлены олигонуклеотиды, которые могут быть в любой форме, например, лиофилизованными или в растворе (например, в дистиллированной воде или забуференном растворе) и т.д.
В предпочтительном аспекте настоящего изобретения предлагается набор, содержащий набор зондов, который описан в данной публикации, чип и необязательно одну или несколько меток.
Также обычно вложен набор инструкций.
ПРИМЕНЕНИЯ
Предпочтительно настоящее изобретение можно применять для того, чтобы получить информацию о пространственной организации нуклеотидных последовательностей, таких как геномные локусы in vitro или in vivo.
В качестве примера способ 4C можно использовать для исследования трехмерной организации одного или нескольких генных локусов. В частности, такой способ можно применять для исследования роли одного или нескольких факторов транскрипции в трехмерной организации одного или нескольких генных локусов.
В качестве следующего примера способ 4C можно применять для исследования роли транс-действующих факторов и цис-регуляторных элементов ДНК.
В качестве следующего примера способ 4C можно применять для исследования регуляции генов дальнего действия in vitro или in vivo.
В качестве следующего примера способ 4C можно применять для исследования внутрихромосомной близости и взаимодействия.
В качестве следующего примера способ 4C можно применять для исследования межхромосомной близости и взаимодействия.
В качестве следующего примера способ 4C можно применять для идентификации нуклеотидных последовательностей, которые функционируют с промотором, энхансером, сайленсером, инсулятором, областью регуляции локуса, началом репликации, MAR, SAR, центромерой, теломерой или любой другой представляющей интерес последовательностью в регуляторной сети.
В качестве следующего примера способ 4C можно применять для идентификации генов, ответственных за фенотип (заболевание) в тех случаях, когда происходит мутация и/или делеция, влияющая на отдаленный регуляторный элемент, и поэтому их картирование не дает такой информации.
В качестве следующего примера способ 4C можно применять для того, чтобы в конечном итоге реконструировать пространственную конформацию генных локусов, больших геномных областей или даже полных хромосом.
В качестве следующего примера способ 4C можно применять для определения потенциальных якорных последовательностей, которые удерживают некоторые хромосомы вместе в ядерном пространстве.
В качестве следующего примера способ 4C можно применять для того, чтобы в конечном итоге реконструировать при высоком разрешении положение хромосом по отношению друг к другу.
В качестве следующего примера способ 4C можно использовать для диагностики (например, для пренатальной диагностики), чтобы определить или идентифицировать геномные перестройки и/или аберрации - такие как транслокации, делеции, инверсии, дупликации.
ОБЩИЕ СПОСОБЫ МЕТОДИКИ РЕКОМБИНАНТНОЙ ДНК
В настоящем изобретении, если не оговорено особо, используют обычные способы химии, молекулярной биологии, микробиологии, рекомбинантной ДНК и иммунологии, которые могут быть осуществлены специалистом в данной области. Такие способы описаны в литературе. См., например, J. Sambrook, E. F. Fritsch and T. Maniatis, 1989, Molecular Cloning: A Laboratory Manual, Second Edition, Books 1-3, Cold Spring Harbor Laboratory Press; Ausubel, F. M. et al. (1995 и периодические приложения; Current Protocols in Molecular Biology, ch. 9, 13 and 16, John Wiley and Sons, New York, N. Y.); B. Roe, J. Crabtree and A. Kahn, 1996, DNA Isolation and Sequencing: Essential Techniques, John Wiley and Sons; M. J. Gait (Editor), 1984, Oligonucleotide Synthesis: A Practical Approach, Irl Press; и D. M. J. Lilley and J. E. Dahlberg, 1992, Methods of Enzymology: DNA Structure Part A: Synthesis and Physical Analysis of DNA Methods in Enzymology, Academic Press. Каждая из указанных публикаций включена в виде ссылки.
Далее изобретение будет описано в виде примеров, при этом подразумевается, что примеры служат для того, чтобы помочь специалисту в осуществлении изобретения, и никоим образом не предназначены для ограничения объема изобретения.
ПРИМЕР 1
Материалы и способы
Способ 4C
Начальные стадии способа 3C осуществляли как описано ранее (Splinter et al. (2004). Methods Enzymol. 375, 493-507 (2004), получая продукты лигирования между HindIII-фрагментами. Полученную HindIII-лигированную матрицу 3C (~50 мкг) расщепляли в течение ночи при концентрации 100 нг/мкл с использованием 50 Ед. второго, часто разрезающего фермента рестрикции, либо DpnII (HS2, Rad23A), либо NlaIII (β-основной). Чтобы избежать ограничений при образовании колец ДНК (Rippe et al. (1995) Trends Biochem. Sci. 20, 500-6), предпринимали осторожность при выборе второго фермента рестрикции, чтобы он не делал разрезов в пределах примерно 350-400 п.о. от сайта рестрикции HindIII, который ограничивает представляющий интерес фрагмент рестрикции (т.е. «приманку»). После расщепления вторым ферментом рестрикции ДНК экстрагировали фенолом, преципитировали этанолом и затем лигировали при низкой концентрации (50 мкг образца в 14 мл, используя 200 Ед. лигазы (Roche), 4 часа при 16°C), чтобы стимулировать образование DpnII- или DpnII-колец. Продукты лигирования экстрагировали фенолом и преципитировали этанолом, используя гликоген (Roche) в качестве носителя (20 мкг/мл). Представляющие интерес кольца линеаризовали расщеплением в течение ночи, используя 50 Ед. третьего фермента рестрикции, который расщепляет приманку между сайтами узнавания первым и вторым ферментом рестрикции, при этом использовали следующие ферменты рестрикции: SpeI (HS2), PstI (Rad23A) и PflmI (β-основной). Указанную стадию линеаризации осуществляли для того, чтобы облегчить последующую гибридизацию с праймером во время первых раундов ПЦР-амплификации. Расщепленные продукты очищали, используя колонку для удаления нуклеотидов QIAquick (250) (Qiagen).
Реакции ПЦР осуществляли, используя систему ПЦР с элонгацией на длинных матрицах (Roche), с использованием тщательно оптимизированных условий, чтобы обеспечить линейную амплификацию фрагментов размером до 1,2 т.п.о. (80% 4C-ПЦР-фрагментов имеют размер менее 600 п.о.). Использовали следующие условия ПЦР: 94°C в течение 2 минут, 30 циклов 94°C в течение 15 секунд, 55°C в течение 1 минуты и 68°C в течение 3 минут, с последующей конечной стадией при 68°C в течение 7 минут. Определяли максимальное количество матрицы, при котором еще наблюдается линейный диапазон амплификации. С этой целью серийные разведения матрицы добавляли в реакционные смеси для ПЦР, амплифицированный материал ДНК разгоняли в агарозном геле и продукты ПЦР количественно определяли с использованием компьютерной программы ImageQuant. Обычно 100-200 нг матрицы на 50 мкл реакционной смеси для ПЦР давали продукты в линейном диапазоне амплификации. От 16 до 32 смесей после ПЦР-реакций объединяли и очищали 4C-матрицу, используя систему для удаления нуклеотидов QIAquick (250) (Qiagen). Очищенную 4C-матрицу метили и гибридизовали с чипами согласно стандартным протоколам для ChIP-чипов (Nimblegen Systems of Iceland, LLC). Геномная ДНК, меченная иным образом, которую расщепляли первым и вторым ферментами, используемыми в способе 4C, служила в качестве контрольной матрицы для корректировки различий в эффективности гибридизации. Для каждого эксперимента два независимо обработанных образца метили красителями с разной ориентацией.
Используемые последовательности праймеров 4C:
HS2: 5'-ACTTCCTACACATTAACGAGCC-3',
5'-GCTGTTATCCCTTTCTCTTCTAC-3'
Rad23A: 5'-TCACACGCGAAGTAGGCC-3',
5'-CCTTCCTCCACCATGATGA-3'
β-основной: 5'-AACGCATTTGCTCAATCAACTACTG-3',
5'-GTTGCTCCTCACATTTGCTTCTGAC-3'
Чипы 4C
Чипы и анализ были основаны на конструкции NCBI m34. Зонды (60-меры) отбирали из последовательностей длиной 100 п.о. выше и ниже сайтов HindIII. Содержание CG оптимизировали до 50% для получения единообразных сигналов гибридизации. Чтобы предотвратить перекрестную гибридизацию зонды, которые имели сходство с широко распространенными повторами (RepBase 10.09)3, удаляли из набора зондов. Кроме того, зонды, которые давали более двух совпадений в геноме при использовании BLAST, также удаляли из набора зондов. Выравнивание последовательностей осуществляли с использованием MegaBLAST (Zhang et al. (2000) J. Comput. Biol. 7, 203-14), используя стандартные установки параметров. Совпадение определяли в случае выравнивания на протяжении 30 нуклеотидов или более.
Анализ данных 4C
Отношение сигналов 4C-образец/геномная ДНК рассчитывали для каждого зонда и данные визуализировали с использованием компьютерной программы SignalMap, предоставляемой Nimblegen Systems. Данные анализировали, используя пакет R (http://www.r-project.org), Spotfire и Excel. Не подвергнутые обработке отношения гибридизации давали кластеры по 20-50 позитивных сигналов 4C вдоль хромосомной матрицы. Для определения указанных кластеров применяли способ скользящих средних. Использовали различные размеры окна в диапазоне 9-39 зондов, причем все они позволяли идентифицировать одни и те же кластеры. Показанные результаты были основаны на размере окна 29 зондов (в среднем 60 т.п.о.), и результаты сравнивали со скользящим средним, полученным для совокупности рандомизированных данных. Указанную обработку осуществляли для каждого чипа отдельно. Поэтому все измерения оценивали относительно амплитуды и шума данного конкретного чипа. Уровень ложных обнаружений (FDR), определяемый как (количество ложно-положительных результатов)/(количество ложно-положительных результатов + количество истинных положительных результатов), рассчитывали следующим образом: (количество положительных результатов в рандомизированном наборе)/(количество положительных результатов в полученных данных). Пороговый уровень определяли с использованием принципа нисходящего анализа, чтобы установить минимальное значение, для которого: FDR <0,05.
Затем сравнивали биологические эксперименты, проведенные в двух повторах. Окна, которые превышали порог в обоих повторах, считали позитивными. При сравнении рандомизированных данных не выявлено окон выше порога в обоих повторах. Позитивные окна, непосредственно расположенные рядом на хромосомной матрице, соединяли (пробелы недопустимы), создавая позитивные области.
Анализ экспрессии
Для каждой ткани использовали три независимых микроматрицы согласно протоколу Affymetrix (мышиные зонды 430_2). Данные нормализовали, используя программные средства RMA ca (www.bioconductor.org), и для каждого набора зондов усредняли измерения, полученные с использованием трех микроматриц. Кроме того, когда несколько наборов зондов представляли один и тот же ген, данные также усредняли. Mas5calls (библиотека Affy: www.bioconductor.org) использовали для установления сигналов «присутствует», «отсутствует» и «пограничный». Гены с сигналом «присутствует» на всех трех чипах и значением экспрессии выше 50 назвали экспрессируемыми. Гены, специфичные для печени плода, были классифицированы как гены, которые удовлетворяют критериям авторов, являясь экспрессируемыми в фетальной печени, и имели более чем в пять раз более высокие значения экспрессии по сравнению с фетальным мозгом. Чтобы получить меру общей транскрипционной активности вокруг каждого гена, применяли сумму скользящих средних. С этой целью авторы использовали log-преобразованные значения экспрессии. Для каждого гена авторы рассчитывали сумму экспрессии всех генов, обнаруженных в окне от 100 т.п.о. выше начала и до 100 т.п.о. ниже конца гена, включая сам ген. Полученные в результате значения для активных генов, обнаруженных внутри позитивных областей 4C (n=124, 123 и 208, соответственно, для HS2 в печени, Rad23A в головном мозге и Rad23A в печени), сравнивали со значениями, полученными для активных генов вне позитивных областей 4C (n=153, 301 и 186, соответственно, где n=153 соответствует количеству активных не взаимодействующих генов, присутствующих между наиболее близко расположенной к центромере взаимодействующей областью и теломерой хромосомы 7); две группы сравнивали, используя односторонний критерий суммы рангов Уилкоксона.
Зонды FISH
Использовали следующие BAC-клоны (BACPAC Resources Centre); RP23-370E12 для Hbb-1, RP23-317H16 для хромосомы 7 - 80,1 млн.п.о. (генный кластер OR), RP23-334E9 для Uros, RP23-32C19 для хромосомы 7 - 118,3 млн.п.о., RP23-143F10 для хромосомы 7 - 130,1 млн.п.о., RP23-470N5 для хромосомы 7 - 73,1 млн.п.о., RP23-247L11 для хромосомы 7 - 135,0 млн.п.о. (генный кластер OR), RP23-136A15 для Rad23A, RP23-307P24 для хромосомы 8 - 21,8 млн.п.о., и RP23-460F21 для хромосомы 8 - 122,4 млн.п.о. В качестве специфичного для центромеры хромосомы 7 зонда авторы использовали P1-клон 5279 (Genome Systems Inc.), который отжигается с участком ДНК D7Mit21. Зонды, меченые со случайными праймерами, получали, используя систему мечения генома BioPrime Array CGH (Invitrogen). Перед мечением ДНК расщепляли DpnII и очищали с помощью набора для очистки и концентрирования ДНК 5 (Zymo research). Расщепленную ДНК (300 нг) метили SpectrumGreen-dUTP (Vysis) или Alexa fluor 594-dUTP (Molecular probes) и очищали, используя набор для очистки, полученной в ПЦР ДНК, и очистки полос из геля GFX (Amersham Biosciences), чтобы удалить не включившиеся нуклеотиды. Специфичность меченых зондов тестировали на метафазных пластинках, приготовленных из ES-клеток мышей.
Крио-FISH
Крио-FISH осуществляли как описано выше5. Коротко, печень и головной мозг E14.5 фиксировали в течение 20 мин в смеси 4% параформальдегид/250 мМ HEPES, pH 7,5 и нарезали небольшие кусочки ткани с последующей новой стадией фиксации в течение 2 час в 8% параформальдегиде при 4°C. Фиксированные кусочки ткани погружали в 2,3М сахарозу на 20 мин при комнатной температуре, вставляли в держатель образца и быстро замораживали в жидком азоте. Кусочки ткани хранили в жидком азоте вплоть до получения срезов. Нарезали ультратонкие срезы замороженной ткани толщиной около 200 нм, используя ультрамикротом Reichert E, оборудованный криоприставкой (Leica). Используя петлю, заполненную сахарозой, срезы переносили на покровные стекла и хранили при -20°C. Для гибридизации срезы промывали PBS, чтобы удалить сахарозу, обрабатывали 250 нг/мл РНКазы в 2×SSC в течение 1 часа при 37°C, инкубировали в течение 10 мин в 0,1М HCl, дегидратировали серией проводок в этаноле и денатурировали в течение 8 мин при 80°C в 70% формамиде/2×SSC, pH 7,5. Срезы снова дегидратировали непосредственно перед гибридизацией с зондами. 500 нг меченого зонда преципитировали вместе с 5 мкг ДНК Cot1 мыши (Invitrogen) и растворяли в смеси hybmix (50% формальдегид, 10% сульфата декстрана, 2×SSC, 50 мМ фосфатный буфер, pH 7,5). Зонды денатурировали в течение 5 мин при 95°C, повторно отжигали в течение 30 мин при 37°C и гибридизовали по меньшей мере в течение 40 ч при 37°C. После постгибридизационных промывок ядра контрастно красили 20 нг/мл DAPI (Sigma) в PBS/0,05% твин-20 и помещали в реагент против обесцвечивания Prolong Gold (Molecular Probes).
Изображения получали с помощью эпифлуоресцентного микроскопа Zeiss Axio Imager Z1 (×100 апохроматический объектив с плоским полем, 1,4 масляно-иммерсионный объектив), оборудованного CCD-камерой, и используя компьютерную программу для системы визуализации FISH Isis (Metasystems). Анализировали минимум 250 аллелей β-глобина или Rad23A и оценивали как перекрывающиеся или не перерывающиеся с BAC, расположенными в других местах генома, при этом исследователь не знал комбинацию зондов, применяемых по отношению к срезам. Использовали повторяющиеся критерии согласия (G-статистика)6 для оценки значимости различий между значениями, измеренными для 4C-позитивных по сравнению с 4C-негативными областями. Общие результаты представлены в таблице 2.
Хотя авторы обнаружили статистически значимые различия между фоновыми (0,4-3,9%) и действительными частотами взаимодействия (5-20,4%), может быть понятно, что частоты, измеренные с помощью крио-FISH, ниже, чем частоты, измеряемые другими исследователями с использованием разных протоколов FISH. При получении срезов некоторые взаимодействующие локусы могут быть разделены, и поэтому измерения крио-FISH будут в некоторой степени занижать истинные частоты взаимодействия. С другой стороны, современные способы 2D- и 3D-FISH будут завышать указанные проценты вследствие ограниченного разрешения в z-направлении. В будущем улучшенные способы микроскопии в сочетании с более специфичными FISH-зондами будут лучше выявлять истинные частоты взаимодействий.
ПРИМЕР 2
Способ 3C (т.е. фиксацию формальдегидом, расщепление (первым) ферментом рестрикции, повторное лигирование поперечно сшитых фрагментов ДНК и очистку ДНК) осуществляют по существу как описано (Splinter et al., (2004) Methods Enzymol. 375: 493-507), получая смесь ДНК («3C-матрица»), содержащую фрагменты рестрикции, которые лигируют, так как исходно они находились близко друг к другу в ядерном пространстве.
Осуществляют инвертированную ПЦР, чтобы амплифицировать все фрагменты, лигированные с данным фрагментом рестрикции («приманкой»; выбранной потому, что она содержит промотор, энхансер, инсулятор, область связывания с матриксом, начало репликации или любую другую первую нуклеотидную последовательность (мишень)). С этой целью создают кольца ДНК расщеплением 3C-матрицы вторым ферментом рестрикции (предпочтительно часто разрезающим ферментом, узнающим тетра- или пента-нуклеотидные последовательности) с последующим лигированием в условиях разбавления, для того чтобы предпочтительно происходили внутримолекулярные взаимодействия. Чтобы минимизировать искажения при образовании колец вследствие топологических ограничений (Rippe et al., (2001) Trends in Biochem. Sciences 26, 733-40), второй фермент рестрикции необходимо выбрать так, чтобы он предпочтительно разрезал приманку на расстоянии >350-400 п.о. от первого сайта рестрикции. Чтобы увеличить эффективность и воспроизводимость инвертированной ПЦР-амплификации, кольца лучше всего линеаризуют перед ПЦР-амплификацией ферментом рестрикции (например, узнающим 6 или более п.о.), которые расщепляет приманку между диагностическим первым и вторым сайтами рестрикции.
Расщепление 3C-матрицы вторым ферментом рестрикции, образование колец посредством лигирования в условиях разбавления и линеаризация содержащих приманку колец осуществляют в условиях, стандартных для таких обработок ДНК, получая ДНК-матрицу для инвертированной ПЦР-амплификации («4C-матрицу»).
Соответственно, 10 мкг 3C-матрицы расщепляют в 100 мкл, используя 20 Ед. второго фермента рестрикции (в течение ночи), с последующей инактивацией фермента нагреванием и очисткой ДНК. Лигирование осуществляют в 10 мл (1 нг/мкл ДНК), используя 50 Ед. лигазы T4 (4 часа при 16°C, 30 мин при комнатной температуре) с последующей очисткой ДНК. Наконец, осуществляют линеаризацию представляющих интерес колец в 100 мкл, используя 20 Ед. фермента рестрикции (в течение ночи), после чего снова проводят очистку ДНК.
Для инвертированной ПЦР конструируют два специфичных для приманки праймера, каждый из которых расположен близко, насколько это возможно, к первому и непосредственно ближайшему к нему второму сайту узнавания ферментами рестрикции, соответственно, и каждый из них имеет 3'-конец, обращенный наружу так, чтобы удлинение происходило непосредственно через сайты рестрикции к фрагменту, лигированному с приманкой. Инвертированную ПЦР с указанными праймерами предпочтительно осуществляют на 100-400 нг ДНК 4C-матрицы (на 50 мкл реакционной смеси для ПЦР), чтобы включить максимальное количество событий лигирования на реакцию ПЦР. Авторы осуществляли инвертированную ПЦР с применением системы для ПЦР с удлинением на длинных матрицах (Roche), используя буфер 1, согласно способам, предложенным производителем.
Осуществляли следующие циклы ПЦР:
1) 2 минуты при 94°C;
2) 15 секунд при 94°C;
3) 1 минута при 55°C;
4) 3 минуты при 68°C;
5) повторяют стадии 2-4 29 раз (или от 25 до 40 раз);
6) 7 минут при 68°C;
7) остановка.
Проводят электрофорез в геле, чтобы проанализировать вопроизводимость отдельных реакций ПЦР. Как правило, должны быть получены идентичные картины продуктов.
Чтобы получить достаточное количество материала для мечения с использованием случайных праймеров и гибридизации с чипом, продукты нескольких ПЦР-реакций (в каждом случае после 30 циклов ПЦР) могут быть объединены (вместо того, чтобы увеличивать количество циклов ПЦР на реакцию). Альтернативно в случае мечения с использованием случайных праймеров меченые нуклеотиды могут быть включены в последние циклы ПЦР (например, 30 циклов ПЦР (без метки) + 10 циклов ПЦР (с добавлением метки)).
ПРИМЕР 3
Определение транслокации с использованием способа 4C
Способ 4C используют для измерения частоты взаимодействия данной последовательности X, присутствующей в данной хромосоме A в клетках здорового человека и в клетках пациента, несущего одну реципрокную транслокацию между хромосомами A и B с точкой разрыва, находящейся вблизи последовательности X (как показано на фиг.9).
В нормальных клетках с помощью такого анализа выявляют повышенные сигналы гибридизации (т.е. частые взаимодействия с X) (почти) для каждого зонда, расположенного в пределах 0,2-10 млн.п.о. последовательности X на хромосоме A (реальный размер области хромосомы, в которой наблюдаются интенсивные сигналы поперечного связывания, зависит главным образом от сложности образца, который гибридизовали с чипом). В других местах на той же хромосоме A, а также на других хромосомах не наблюдают такой большой области (на линейной матрице ДНК) зондов с повышенными сигналами гибридизации.
Однако в клетках пациента сигналы гибридизации со всеми зондами на хромосоме A, расположенными с другой стороны от точки разрыва, снижены на ~50% (одна копия хромосомы A еще остается интактной и будет давать нормальные сигналы), тогда как необычная (т.е. не встречающаяся в нормальных клетках) концентрация повышенных сигналов гибридизации наблюдается для зондов, граничащих с точкой разрыва на хромосоме B. Действительно, резкий переход от зондов, не дающих и дающих высокие сигналы гибридизации на хромосоме B, выявляет положение точки разрыва на хромосоме B.
ПРИМЕР 4
Анализ результатов, полученных способом 4C
Способ 4C использовали для характеристики геномного окружения регуляторной области локуса (LCR) β-глобина мыши, фокусируя внимание на фрагменте рестрикции, содержащем гиперчувствительный сайт 2 (HS2). LCR является сильным специфичным для эритроидных клеток элементом, регулирующим транскрипцию, необходимым для обеспечения высоких уровней экспрессии гена β-глобина. Локус β-глобина присутствует на хромосоме 7 в положении 97 млн.п.о., где он располагается в большом, занимающем 2,9 млн.п.о., кластере генов обонятельных рецепторов, которые транскрибируются только в обонятельных нейронах. Взаимодействия анализировали в двух тканях: фетальной печени E14.5, где LCR является активным и транскрипция генов β-глобина является высокой, и в головном мозге плода E14.5, где LCR является неактивным и гены глобина являются молчащими. В обеих тканях большую часть взаимодействий обнаружили с последовательностями в хромосоме 7 и очень немного взаимодействий LCR выявлено с шестью неродственными хромосомами (8, 10, 11, 12, 13, 14) (фиг.13a). Наиболее сильные сигналы на хромосоме 7 обнаружены в области 5-10 млн.п.о., сосредоточенных вокруг хромосомного положения β-глобина, что согласуется с представлением о том, что частоты взаимодействий обратно пропорциональны расстоянию (в парах оснований) между физически связанными последовательностями ДНК. Количественно интерпретировать взаимодействия в данной области было невозможно. Авторы рассуждали так, что указанные, близко расположенные последовательности настолько часто находились вместе с β-глобином, что их избыточное представление в образцах гибридизации насыщало соответствующие зонды. Предположение подтвердилось, когда авторы проводили гибридизации с использованием образцов, разбавленных 1:10 и 1:100, и обнаружили, что интенсивность сигнала была снижена для зондов за пределами данной области и по краю области, но не внутри данной области (данные не показаны).
Способ 4C давал высоко воспроизводимые данные. На фиг.2b-c показаны необработанные отношения 4C-сигналов по сравнению с контрольными сигналами гибридизации для двух областей длиной 1,5 млн.п.о. на хромосоме 7, приблизительно на расстоянии 25 млн.п.о. и 80 млн.п.о. от гена β-глобина. При таком уровне разрешения результаты для независимо обработанных образцов были почти идентичны. Как в фетальной печени, так и в головном мозге идентифицировали кластеры позитивных сигналов на хромосоме 7, часто в положениях хромосомы на расстоянии десятков миллионов оснований от β-глобина. Указанные кластеры обычно состояли минимально из 20-50 зондов с повышенными отношениями сигналов, расположенных рядом на хромосомной матрице (фиг.13b-c). Каждый зонд на чипе позволяет анализировать независимое событие лигирования. Кроме того, только две копии фрагмента рестрикции HS2 присутствуют в клетке, каждая из которых может лигировать только с одним другим фрагментом рестрикции. Таким образом, выявление независимых событий лигирования с 20 или более соседними фрагментами рестрикции строго показывает, что соответствующий локус контактирует с LCR β-глобина во множестве клеток.
Чтобы определить статистическую значимость выявления указанных кластеров, данные, полученные в отдельных экспериментах, располагали в определенном порядке на хромосомных картах и анализировали, используя алгоритм скользящих средних при размере окна примерно 60 т.п.о. Распределение скользящих средних случайным образом перемешанных данных использовали для установления порогового значения, допускающего уровень ложных обнаружений 5%. С помощью указанного анализа идентифицировали 66 кластеров в фетальной печени и 45 в головном мозге, которые воспроизводимо выявляли в экспериментах, проводимых в двух повторах (фиг.13 d-f). Действительно, FISH высокого разрешения подтвердила, что такие кластеры на самом деле представляют локусы, которые часто взаимодействуют (см. ниже).
Таким образом, способом 4C идентифицируют локусы, взаимодействующие на далеком расстоянии, посредством выявления независимых событий лигирования с несколькими фрагментами рестрикции, расположенными в виде кластера в определенном положении в хромосоме.
Проводили полностью независимую серию экспериментов 4C с другим набором праймеров для инвертированной ПЦР, в которой исследовали геномное окружение гена β-основного, локализованного на ~50 т.п.о. ниже HS2. В фетальной печени ген β-основного имеет высокий уровень транскрипции и часто контактирует с LCR. Обнаружены почти идентичные кластеры взаимодействий дальнего действия с β-основным, как и с HS2, и в фетальной печени и в головном мозге, что дополнительно подтверждает, что указанные локусы часто контактируют с локусом β-глобина (фиг.17).
ПРИМЕР 5
Активный и неактивный локус β-глобина имеют разную окружающую среду в геноме
Сравнение между двумя тканями выявило, что активно транскрибируемый локус β-глобина в фетальной печени взаимодействует с совершенно другим набором локусов, чем его транскрипционно молчащий аналог в головном мозге (τ=-0,03; ранговая корреляция по Спирману) (фиг.13f). Это исключало, что на результаты влиял состав последовательности зондов. В фетальной печени взаимодействующие участки ДНК располагались в пределах области 70 млн.п.о., сосредоточенной вокруг локуса β-глобина, при этом большая часть (40/66) были расположены по направлению к теломере хромосомы 7. В головном мозге плода взаимодействующие локусы были обнаружены на сходных или еще больших расстояниях от β-глобина по сравнению с фетальной печенью, и при этом большая часть взаимодействий (43/45) была локализована по направлению к центромере хромосомы 7. Полученные данные показали, что активный и неактивный локус β-глобина контактирует с разными частями хромосомы 7.
Шесть других хромосом (8, 10, 11, 12, 13 и 14) были представлены на микроматрице. Высокие сигналы гибридизации на указанных хромосомах были редкими, обычно выглядели изолированными на линейной матрице ДНК и часто отсутствовали в повторных экспериментах. Также уровни скользящих средних вдоль указанных хромосом никогда воспроизводимо не подходили к уровням, определяемым для хромосомы 7 (фиг.19). Таким образом, полученные авторами данные показали, что локус β-глобина главным образом контактирует с локусами в других местах на той же самой хромосоме, что согласуется с предпочтительной локализацией данного локуса внутри территории, занимаемой хромосомой, в которой он находится. Авторы отмечают, что локус α-глобина также присутствует на зонде (хромосома 11) и не оценивается позитивно в отношении взаимодействия с β-глобином, что согласуется с представленной недавно демонстрацией посредством FISH того, что мышиные локусы α- и β-глобина не часто встречаются в ядерном пространстве (Brown J.M. et al. (2006) J. Cell Biol. 172, 177-87).
Чтобы лучше понять значение наблюдаемых взаимодействий дальнего действия на хромосоме 7, авторы сравнили взаимодействующие локусы с положениями генов в хромосоме. Кроме того, осуществляли анализ чипа для исследования экспрессии Affymetrix, чтобы определить транскрипционную активность в указанных положениях в двух тканях. Хотя средний размер взаимодействующих областей в фетальной печени и головном мозге были сравнимы (183 т.п.о. и 159 т.п.о., соответственно), наблюдали существенные различия в генах, которые в них находятся, и в активности. В фетальной печени 80% взаимодействующих с β-глобином локусов содержали один или несколько активно транскрибируемых генов, тогда как в головном мозге плода большая часть (87%) не проявляла регистрируемой генной активности (фиг.15). Таким образом, локус β-глобина имеет очень разное геномное окружение в двух тканях. В головном мозге, где локус является неактивным, он главным образом контактирует с транскрипционно молчащими локусами, расположенными по направлению к центромере хромосомы 7. В фетальной печени, где локус имеет высокую активность, он предпочтительно взаимодействует с активно транскрибируемыми областями, расположенными ближе к теломерной стороне хромосомы 7. Важно, что способом 4C идентифицировали Uros и Eraf (~30 млн.п.о. от β-глобина) в качестве генов, взаимодействующих с активным локусом β-глобина в фетальной печени, что согласуется с предыдущими наблюдениями, сделанными с помощью FISH (Osborne C.S. et al. (2004) Nat. Genet. 36, 1065-71 (2004)). Интересно, что в головном мозге наблюдали контакты с двумя другими кластерами генов обонятельного рецептора, присутствующими на хромосоме 7, которые были локализованы с каждой стороны на расстоянии 17 и 37 млн.п.о. от β-глобина.
Не все транскрибируемые области в хромосоме 7 взаимодействуют с активным локусом β-глобина в фетальной печени. Поэтому авторы проводили поиск в отношении показателя, который является общим исключительно только для взаимодействующих локусов, но не для других активных областей в фетальной печени. Гены β-глобина, Uros и Eraf являются специфичными для эритроидных клеток генами, которые могут регулироваться одним и тем же набором факторов транскрипции, и привлекательна идея о том, что указанные факторы координируют экспрессию своих генов-мишеней в ядерном пространстве. Авторы сравнили данные, полученные с использованием чипа для исследования экспрессии Affymetrix, для фетальной печени E14.5 с данными для головного мозга плода, чтобы идентифицировать гены, экспрессируемые преимущественно (>5 раз больше) в фетальной печени. Как таковые 28% активных генов в хромосоме 7 были классифицированы как «специфичные для фетальной печени», из них 25% были локализованы в общей области. Таким образом, авторы не обнаружили обогащения «специфичными для фетальной печени» генами в областях совместной локализации. Более важно, что 49 из 66 (74%) взаимодействующих областей не содержали «специфичных для фетальной печени» генов, и поэтому сделан вывод, что полученные авторами данные не представили доказательства координированной экспрессии тканеспецифичных генов в ядерном пространстве. Гены β-глобина транскрибируются с необычайно высокими скоростями, и возникает следующий вопрос, взаимодействовал ли локус предпочтительно с другими областями с высокой транскрипционной активностью, либо областями, содержащими гены, имеющие высокий уровень экспрессии, либо областями с высокой плотностью активных генов. Используя подсчет на Affymetrix в качестве меры активности генов, авторы применили алгоритм скользящих сумм, чтобы измерить общую транскрипционную активность в областях размером 200 т.п.о. вокруг активно транскрибируемых генов. Указанный анализ выявил, что транскрипционная активность вокруг взаимодействующих генов не выше, чем вокруг не взаимодействующих активных генов на хромосоме 7 (p=0,9867; сумма рангов Уилкоксона).
ПРИМЕР 6
Геномное окружение гена домашнего хозяйства является высоко консервативным в разных тканях
Далее исследовали, изменяет ли свое геномное окружение ген, который сходным образом эксрессируется в обеих тканях. Rad23A является повсеместно экспрессируемым геном, который находится в имеющем высокую плотность генов кластере главным образом генов домашнего хозяйства, в хромосоме 8. И в фетальной печени E14.5, и в головном мозге плода указанный ген и многие его непосредственные соседи являются активными. Осуществляли анализ 4C и идентифицировали многие взаимодействия на больших расстояниях с локусами, находящимися до 70 млн.п.о. от Rad23A. Важно, что взаимодействия с Rad23A в высокой степени коррелировали в печени и головном мозге плода (τ=0,73; ранговая корреляция по Спирману) (фиг.15a). Общим признаком указанных локусов снова было то, что они содержали активно транскрибируемые гены. Таким образом, в обеих тканях около 70% содержали по меньшей мере один активный ген (фиг.15b-c). Области вокруг взаимодействующих генов имели статистически значимо более высокие уровни генной активности по сравнению с активными генами в других местах хромосомы, как определяли с помощью алгоритма сумм скользящих средних (p<0,001 для обеих тканей). Таким образом, в отличие от локуса β-глобина, ген Rad23A, который локализован в богатой генами области, предпочтительно взаимодействует на расстоянии с другими хромосомными областями с повышенной транскрипционной активностью. С помощью FISH обнаружено, что хромосомная область, содержащая Rad23A, находится главным образом на краю (90%) или вне (10%) территории, занимаемой хромосомой (неопубликованные данные, D. Noordermeer, M. Branco, A. Pombo and W. de Laat). Однако анализ 4C выявил только внутрихромосомные взаимодействия, и ни одна из областей на хромосоме 7, 10, 11, 12, 13 или 14 воспроизводимо не удовлетворяла строгим критериям авторов в отношении взаимодействия. Таким образом, Rad23A главным образом вовлечен в внутрихромосомные взаимодействия, которые сходны в двух очень разных тканях. Если Rad23A имеет предпочтительные прилегающие области на указанных неродственных хромосомах, то они не взаимодействуют достаточно часто, чтобы их можно было выявить в условиях, используемых в данном случае для способа 4C.
ПРИМЕР 7
Проверка способа 4C с помощью микроскопии с высоким разрешением
Чтобы подтвердить результаты, полученные способом 4C, осуществляли эксперименты крио-FISH. Крио-FISH является недавно разработанным способом микроскопии, который имеет преимущество по сравнению с современными протоколами 3D-FISH, так как лучше сохраняет ультраструктуру ядра, давая при этом улучшенное разрешение по z-оси за счет получения ультратонких срезов замороженной ткани (Branco M.R. and Pombo, A (2006). PLoS Biol. 4, e138). Данные 4C подтверждали, измеряя то, как часто аллели β-глобина или Rad23A (всегда n>250) локализованы совместно с более чем 15 выбранными хромосомными областями на ультратонких срезах толщиной 200 нм, полученных из печени и головного мозга E14.5. Важно, что все частоты взаимодействия, измеренные с помощью крио-FISH точно соответствовали результатам 4C (фиг.17). Например, отдаленные области, которые, как было идентифицировано способом 4C, взаимодействуют с β-глобином, были локализованы совместно более часто, чем промежуточные области, не выявленные способом 4C (7,4% и 9,7% по сравнению с 3,6% и 3,5%, соответственно). Также для двух отдаленных кластеров генов обонятельного рецептора, которые идентифицированы способом 4C как взаимодействующие с β-глобином в головном мозге плода, но не в печени, частоту совместной локализации оценивали соответственно 12,9% и 7% в головном мозге по сравнению с 3,6% и 1,9% на срезах печени. Итак, частоты совместной локализации, измеренной для локусов, позитивно идентифицированных способом 4C, были значимо выше, чем частоты, измеренные для фоновых локусов (p<0,05; G-критерий). Авторы пришли к выводу, что способ 4C точно идентифицировал взаимодействующие локусы ДНК. Наконец, авторы использовали крио-FISH, чтобы показать, что локусы, идентифицированные как взаимодействующие с β-глобином, также часто контактировали друг с другом. Указанное было справедливо для двух активных областей, разделенных большим расстоянием в хромосоме, в фетальной печени (фиг.19), а также для двух неактивных кластеров генов OR, находящихся на большом расстоянии на хромосоме в головном мозге (фиг.17). Интересно, что частые контакты между указанными двумя отдаленными кластерами генов OR также обнаружены в фетальной печени, где они не взаимодействовали с кластером генов OR, который содержал активно транскрибируемый локус β-глобина. Полученные данные показали, что взаимодействия в ядре между отдаленными кластерами генов OR не являются специфичными для анализируемой ткани головного мозга плода. Становится заманчивым предположение о том, что такие пространственные контакты способствуют взаимодействию между многими генами OR, необходимыми для того, чтобы обеспечить транскрипцию только одного аллеля в обонятельном нейроне (Shykind, B. (2005) Hum. Mol. Genet. 14 Spec. No 1, R33-9.
ПРИМЕР 8
Ядерная организация активных и неактивных доменов хроматина
Наблюдения, описанные в данной публикации, показывают, что не только активные, но также и неактивные геномные области образуют отдельные области в ядерном пространстве, в которые вовлечены контакты дальнего действия, что строго свидетельствует о том, что каждый участок ДНК имеет свой собственный предпочтительный набор взаимодействий. Полученные авторами данные свидетельствуют, что когда локус β-глобина включается, он покидает транскрипционно молчащее геномное окружение и входит в область, в которой поддерживаются взаимодействия с активными доменами. Предполагается, что такое существенное изменение положения при активации транскрипции вполне может быть признаком только тканеспецифичных генов, которые достигают определенного уровня экспрессии, и что еще более важно, располагаются изолированно от других активных генов на линейной хромосомной матрице, как в случае β-глобина. Предполагается, что разветвленная сеть взаимодействий дальнего действия, которые идентифицированы как между неактивными, так и между активными геномными локусами, отражает межклеточные различия конформации хромосом больше, чем различия, являющиеся следствием динамического движения во время интерфазы (Chakalova et al. (2005) Nat. Rev. Genet. 6, 669-77 (2005). По-видимому, деконденсация, в различной степени происходящая после клеточного деления, направляет активные области генома дальше от неактивного хроматина (Gilbert N. et al. (2004) Cell 118, 555-66 (2004)), а контакты между отдаленными локусами со сходным составом хроматина стабилизируются главным образом благодаря аффинности между связанными с хроматином белками. Пространственное сближение между отдаленными локусами может быть функциональным, но также может быть просто следствием развернутой конформации хромосом. Хотя отдельные локусы могут двигаться в ограниченном объеме ядра, общая конформация хромосом в значительной степени может сохраняться на протяжении клеточного цикла, и требуется деление клетки для перегруппировки. Указанная идея согласуется с исследованиями визуализации жизненного цикла клетки, показывающими ограниченное движение меченых локусов ДНК внутри ядра (Chubb et al. (2002) Curr. Biol. 12, 439-45 (2002)), и хорошо согласуется с исследованиями, показывающими, что информация о положении хроматина в ядре часто передается по наследству во время деления клеток, не будучи консервативной в популяции клеток (Essers J. et al. Mol. Biol. Cell 16, 769-75 (2005); Gerlich D. et al. Cell 112, 751-64 (2003)).
ДОПОЛНИТЕЛЬНЫЕ АСПЕКТЫ 1
Дополнительные аспекты настоящего изобретения указаны ниже в пронумерованных абзацах.
1. Набор зондов, комплементарных каждой стороне каждого сайта узнавания первым ферментом рестрикции в геноме данного вида (например, человека).
2. Набор зондов, комплементарных только одной стороне каждого сайта узнавания первым ферментом рестрикции в геноме данного вида (например, человека).
3. Набор зондов, комплементарных одной стороне каждого второго сайта узнавания первым ферментом рестрикции, который расположен по порядку вдоль линейной матрицы генома данного вида (например, человека).
4. Набор зондов, комплементарных одной стороне каждого третьего, четвертого, пятого, шестого, седьмого, восьмого, девятого, десятого, двадцатого, тридцатого, сорокового, пятидесятого, шестидесятого, семидесятого, восьмидесятого, девяностого или сотого сайта узнавания первым ферментом рестрикции, которые расположены по порядку вдоль линейной матрицы генома данного вида (например, человека).
5. Набор зондов, представляющих геномные области заданного размера (например, около 50 т.п.о., 100 т.п.о., 200 т.п.о., 300 т.п.о., 400 т.п.о., 500 т.п.о., 1 млн.п.о., 2 млн.п.о., 3 млн.п.о., 4 млн.п.о., 5 млн.п.о., 6 млн.п.о., 6 млн.п.о., 7 млн.п.о., 8 млн.п.о., 9 млн.п.о. или 10 млн.п.о.) (например, 50 т.п.о. - 10 млн.п.о.) вокруг всех локусов, которые, как известно, вовлечены в транслокации, делеции, инверсии, дупликации и другие геномные перестройки.
6. Набор зондов, представляющих геномные области заданного размера (например, около 50 т.п.о., 100 т.п.о., 200 т.п.о., 300 т.п.о., 400 т.п.о., 500 т.п.о., 1 млн.п.о., 2 млн.п.о., 3 млн.п.о., 4 млн.п.о., 5 млн.п.о., 6 млн.п.о., 6 млн.п.о., 7 млн.п.о., 8 млн.п.о., 9 млн.п.о. или 10 млн.п.о. (например, 50 т.п.о. - 10 млн.п.о.), вокруг выбранных локусов, которые, как известно, вовлечены в транслокации, делеции, инверсии, дупликации и другие геномные перестройки.
7. Предпочтительно, последовательность 4C (приманка) находится в пределах примерно 50 т.п.о., 100 т.п.о., 200 т.п.о., 300 т.п.о., 400 т.п.о., 500 т.п.о., 1 млн.п.о., 2 млн.п.о., 3 млн.п.о., 4 млн.п.о., 5 млн.п.о., 6 млн.п.о., 6 млн.п.о., 7 млн.п.о., 8 млн.п.о., 9 млн.п.о., 10 млн.п.о., 11 млн.п.о., 12 млн.п.о., 13 млн.п.о., 14 млн.п.о. или 15 млн.п.о. или более от фактически перестроенной последовательности (т.е. точки разрыва в случае транслокации).
8. Набор зондов, представляющих полный геном данного вида, при этом каждый зонд представляет один фрагмент рестрикции, который получен или может быть получен после расщепления первым ферментом рестрикции.
9. Набор зондов, представляющих полный геном данного вида, при этом зонды равномерно распределены вдоль линейных хромосомных матриц.
10. Чип, содержащий набор зондов по п.п.1-10.
11. Способ анализа частоты взаимодействий нуклеотидной последовательности-мишени с одной или несколькими нуклеотидными последовательностями (например, одним или несколькими геномными локусами), который включает в себя применение нуклеотидной последовательности или матрицы зондов, или набора зондов, или чипа, которые описаны в данной публикации.
12. Способ идентификации одного или нескольких ДНК-ДНК-взаимодействий, которые являются показателем конкретного патологического состояния, включающий в себя применение нуклеотидной последовательности или матрицы зондов, или набора зондов, или чипа, которые описаны в данной публикации.
13. Способ диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия, включающий в себя применение нуклеотидной последовательности или матрицы зондов или набора зондов или чипа, которые описаны в данной публикации.
14. Способ анализа для идентификации одного или нескольких агентов, которые модулируют ДНК-ДНК-взаимодействие, включающий в себя применение нуклеотидной последовательности или матрицы зондов, или набора зондов, или чипа, которые описаны в данной публикации.
15. Способ определения положения точки разрыва (например, транслокации), включающий в себя применение нуклеотидной последовательности или матрицы зондов, или набора зондов, или чипа, которые описаны в данной публикации.
16. Способ определения положения инверсии, включающий в себя применение нуклеотидной последовательности или матрицы зондов, или набора зондов, или чипа, которые описаны в данной публикации.
17. Способ определения положения делеции, включающий в себя применение нуклеотидной последовательности или матрицы зондов, или набора зондов, или чипа, которые описаны в данной публикации.
18. Способ определения положения дупликации, включающий в себя применение нуклеотидной последовательности или матрицы зондов, или набора зондов, или чипа, которые описаны в данной публикации.
19. Применение микроматриц в способе 4C для идентификации (всех) участков ДНК, которые находятся в пространственной близости с выбранным участком ДНК.
20. Микроматрица, содержащая зонды, гомологичные последовательностям ДНК, непосредственно граничащим с сайтами узнавания первым ферментом рестрикции, присутствующими в геномной области, которая включена в анализ (которая может представлять собой полный геном или часть генома): при этом каждый зонд локализован предпочтительно в пределах 100 п.о. или максимально в пределах 300 п.о. от уникального сайта узнавания первым ферментом рестрикции, или альтернативно зонд конструируют между каждым сайтом узнавания первым ферментом рестрикции и ближайшим к нему сайтом узнавания вторым ферментом рестрикции.
21. Чип, который описан в данной публикации, содержащий зонды, комплементарные последовательностям выбранных локусов, при этом указанный зонд представляет полный геном данного вида.
22. Чип по п.21, в котором локусы являются локусами, связанными с одним или несколькими заболеваниями.
23. Чип по п.21 или п.22, в котором последовательности выбранных локусов включают последовательности, которые расположены на расстоянии до 20 млн.п.о. от указанных локусов.
24. Способ анализа частоты взаимодействия нуклеотидной последовательности-мишени с одной или несколькими представляющими интерес нуклеотидными последовательностями (например, одним или несколькими геномными локусами), включающий в себя стадии
(a) получения образца поперечно сшитой ДНК;
(b) расщепления поперечно сшитой ДНК первым ферментом рестрикции;
(c) лигирования поперечно сшитых нуклеотидных последовательностей;
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым фермент рестрикции;
(f) лигирования нуклеотидных последовательностей;
(g) амплификации одной или нескольких представляющих интерес нуклеотидных последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью, с использованием по меньшей мере двух олигонуклеотидных праймеров, причем каждый праймер гибридизуется с известной последовательностью ДНК, которая фланкирует представляющие интерес нуклеотидные последовательности;
(h) гибридизации амплифицированной последовательности(ей) с чипом; и
(i) определения частоты взаимодействия между последовательностями ДНК.
ДОПОЛНИТЕЛЬНЫЕ АСПЕКТЫ 2
Следующие дополнительные аспекты настоящего изобретения указаны в пронумерованных пунктах.
1. Кольцевая нуклеотидная последовательность, содержащая первую и вторую нуклеотидные последовательности, разделенные сайтами узнавания первым и вторым ферментами рестрикции, при этом указанная первая нуклеотидная последовательность является нуклеотидной последовательностью-мишенью, а указанная вторая нуклеотидная последовательность может быть получена при поперечном сшивании геномной ДНК.
2. Кольцевая нуклеотидная последовательность по п.1, в которой нуклеотидная последовательность-мишень выбрана из группы, состоящей из промотора, энхансера, сайленсера, инсулятора, области связывания с матриксом, области регуляции локуса, транскрипционной единицы, начала репликации, горячей точки рекомбинации, точки разрыва при транслокации, центромеры, теломеры, области с высокой плотностью генов, области с низкой плотностью генов, повторяющегося элемента и сайта интеграции (вируса).
3. Кольцевая нуклеотидная последовательность по п.1, в которой нуклеотидная последовательность-мишень представляет собой нуклеотидную последовательность, которая связана или вызывает заболевание, или расположена на расстоянии менее чем 15 млн.п.о. на линейной матрице ДНК от локуса, который ассоциирован с заболеванием или вызывает заболевание.
4. Кольцевая нуклеотидная последовательность по любому из пп.1-3, в которой нуклеотидная последовательность-мишень выбрана из группы, состоящей из AML1, MLL, MYC, BCL, BCR, ABL1, IGH, LYL1, TAL1, TAL2, LM02, TCRα/δ, TCRβ и HOX или других локусов, связанных с заболеванием, которые описаны в «Catalogue of Unbalanced Chromosome Aberrations in Man» 2nd edition. Albert Schinzel. Berlin: Walter de Gruyter, 2001. ISBN 3-11-011607-3.
5. Кольцевая нуклеотидная последовательность по любому из пп.1-4, в которой сайт узнавания первым ферментом рестрикции является сайтом узнавания длиной 6-8 п.о., предпочтительно выбранным из группы, состоящей из сайтов узнавания BglII, HindIII, EcoRI, BamHI, SpeI, PstI и NdeI.
6. Кольцевая нуклеотидная последовательность по любому из предшествующих пунктов, в которой сайт узнавания вторым ферментом рестрикции является сайтом узнавания, состоящим из нуклеотидной последовательности длиной 4 или 5 п.о.
7. Кольцевая нуклеотидная последовательность по любому из предшествующих пунктов, в которой сайт узнавания вторым ферментом рестрикции расположен на расстоянии более чем около 350 п.о. от первого сайта рестрикции.
8. Кольцевая нуклеотидная последовательность по любому из предшествующих пунктов, в котором нуклеотидная последовательность является меченой.
9. Нуклеотидная последовательность, содержащая первую и вторую нуклеотидные последовательности, разделенные сайтами узнавания первым и вторым ферментами рестрикции, при этом указанная первая нуклеотидная последовательность является нуклеотидной последовательностью-мишенью, вторая нуклеотидная последовательность может быть получена при поперечном связывании геномной ДНК, и где указанная вторая нуклеотидная последовательность делит на части нуклеотидную последовательность-мишень.
10. Способ получения кольцевой нуклеотидной последовательности, включающий в себя стадии
(a) получения образца поперечно сшитой ДНК;
(b) расщепления поперечно сшитой ДНК первым ферментом рестрикции;
(c) лигирования поперечно сшитых нуклеотидных последовательностей;
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции; и
(f) образования кольцевых нуклеотидных последовательностей.
11. Способ получения нуклеотидной последовательности, включающий в себя стадии
(a) получения образца поперечно сшитой ДНК;
(b) расщепления поперечно сшитой ДНК первым ферментом рестрикции;
(c) лигирования поперечно сшитых нуклеотидных последовательностей;
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции;
(f) образования кольцевых нуклеотидных последовательностей; и
(g) амплификации одной или нескольких нуклеотидных последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью.
12. Способ по п.11, в котором кольцевую нуклеотидную последовательность-мишень линеаризуют перед амплификацией.
13. Способ по п.12, в котором кольцевую нуклеотидную последовательность-мишень линеаризуют, используя фермент рестрикции, который узнает сайт узнавания длиной 6 п.о. или более.
14. Способ по любому из п.п.10-13, в котором поперечно сшитую нуклеотидную последовательность амплифицируют с использованием ПЦР.
15. Способ по п.14, в котором поперечно сшитую нуклеотидную последовательность амплифицируют, используя инвертированную ПЦР.
16. Способ по п.14 или п.15, в котором используют систему ПЦР с элонгацией на длинных матрицах (Roche).
17. Способ анализа частоты взаимодействия нуклеотидной последовательности-мишени с одной или несколькими нуклеотидными последовательностями (например, одним или несколькими геномными локусами), включающий в себя применение нуклеотидной последовательности по любому из п.п.1-9.
18. Матрица зондов, иммобилизованных на подложке, содержащая один или несколько зондов, которые гибридизуются или способны гибридизоваться с нуклеотидной последовательностью по любому из п.п.1-9.
19. Набор зондов, последовательность которых комплементарна последовательности нуклеиновой кислоты, граничащей с каждым из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
20. Набор зондов по п.19, в котором зонды имеют последовательности, комплементарные последовательности нуклеиновой кислоты, граничащей с каждой стороны с каждым из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
21. Набор зондов по п.19 или п.20, в котором указанные зонды имеют последовательности, комплементарные последовательности нуклеиновой кислоты, которая находится на расстоянии менее чем 300 пар оснований от каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
22. Набор зондов по любому из п.п.19-21, в котором зонды комплементарны последовательности, которая расположена на расстоянии менее чем 300 п.о. от каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
23. Набор зондов по любому из п.п.19-22, в котором зонды комплементарны последовательности, расположенной в пределах от 200 до 300 п.о. от каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
24. Набор зондов по любому из п.п.19-23, в котором зонды комплементарны последовательности, расположенной в пределах от 100 до 200 п.о. от каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
25. Набор зондов по любому из п.п.19-24, в котором сконструированы два или более зондов, которые способны гибридизоваться с последовательностью, граничащей с каждым из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
26. Набор зондов по п.25, в котором зонды перекрываются или частично перекрываются.
27. Набор зондов по п.26, в котором перекрывание составляет менее 10 нуклеотидов.
28. Набор зондов по любому из п.п.19-27, в котором последовательность зондов соответствует всей или части последовательности между каждым из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции и каждым из первых соседних сайтов узнавания рестрикционными ферментами для второго фермента рестрикции.
29. Набор зондов по любому из п.п.19-28, в котором каждый зонд является по меньшей мере 25-мером.
30. Набор зондов по любому из п.п.19-29, в котором каждый зонд является 25-60-мером.
31. Способ получения набора зондов, включающий в себя стадии
(a) идентификации каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК;
(b) конструирования зондов, которые способны гибридизоваться с последовательностью, граничащей с каждым из сайтов узнавания первым ферментом рестрикции в геномной ДНК;
(c) синтеза зондов; и
(d) объединения зондов с образованием набора зондов или по существу набора зондов.
32. Способ по п.31, в котором зонды являются продуктами ПЦР-амплификации.
33. Набор зондов или по существу набор зондов, который получен или может быть получен способом по п.31 или п.32.
34. Чип, содержащий матрицу зондов по п.18 или по существу набор зондов по любому из п.п.19-30 или 33.
35. Чип, содержащий набор зондов по любому из п.п.19-30 или 33.
36. Чип по п.34 или п.35, в котором матрица содержит около 300000-400000 зондов.
37. Чип по любому из п.п.34-36, в котором матрица содержит примерно 385000 или более зондов, предпочтительно примерно 750000 зондов, более предпочтительно, 6×750000 зондов.
38. Чип по любому из п.п.34-37, в котором в том случае, если количество зондов превышает количество зондов, которое может находиться на одном чипе, чип содержит или состоит из представления полного генома данного вида при более низком разрешении.
39. Чип по п.38, в котором чип содержит один из каждых 2, 3, 4, 5, 6, 7, 8, 9 или 10 зондов, которые располагаются по порядку на линейной хромосомной матрице.
40. Способ получения чипа, включающий в себя стадию иммобилизации на твердой подложке по существу матрицы зондов по п.18 или по существу набора зондов по любому из п.п.19-30 или 33.
41. Способ получения чипа, включающий в себя стадию иммобилизации на твердой подложке матрицы зондов по п.18 или набора зондов по любому из п.п.19-30 или 33.
42. Чип, который получен или может быть получен способом по п.40 или п.41.
43. Способ анализа частоты взаимодействия нуклеотидной последовательности-мишени с одной или несколькими нуклеотидными последовательностями (например, одним или несколькими геномными локусами), включающий в себя стадии
(a) получения образца поперечно сшитой ДНК;
(b) расщепления поперечно сшитой ДНК первым ферментом рестрикции;
(c) лигирования поперечно сшитых нуклеотидных последовательностей;
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции;
(f) образования кольцевых нуклеотидных последовательностей;
(g) амплификации одной или нескольких нуклеотидных последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью;
(h) необязательно гибридизации амплифицированных последовательностей с чипом; и
(i) определения частоты взаимодействия между последовательностями ДНК.
44. Способ идентификации одного или нескольких ДНК-ДНК-взаимодействий, которые являются показателем конкретного патологического состояния, включающий в себя стадии
(a) получения образца поперечно сшитой ДНК из пораженной заболеванием и не пораженной заболеванием клетки;
(b) расщепления поперечно сшитой ДНК в каждом образце первым ферментом рестрикции;
(c) лигирования поперечно сшитых нуклеотидных последовательностей;
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции;
(f) образования кольцевых нуклеотидных последовательностей;
(g) амплификации одной или нескольких последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью;
(h) необязательно гибридизации амплифицированных последовательностей с чипом; и
(i) определения частоты взаимодействия между последовательностями ДНК,
при этом разница между частотой взаимодействия между последовательностями ДНК из пораженной заболеванием клетки и не пораженной заболеванием клетки свидетельствует о том, что ДНК-ДНК-взаимодействие является показателем конкретного патологического состояния.
45. Способ диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия, включающий в себя стадии
(a) получения образца поперечно сшитой ДНК от субъекта;
(b) расщепления поперечно сшитой ДНК первым ферментом рестрикции;
(c) лигирования поперечно сшитых нуклеотидных последовательностей;
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции;
(f) образования кольцевых нуклеотидных последовательностей;
(g) амплификации одной или нескольких последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью;
(h) необязательно гибридизации амплифицированных последовательностей с чипом; и
(i) определения частоты взаимодействия между последовательностями ДНК, и
(j) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в непораженном контроле;
при этом разница между значением, полученным в контроле, и значением, полученным для субъекта, является показателем того, что субъект имеет данное заболевание или синдромом, или является показателем того, что субъект будет иметь данное заболевание или синдром.
46. Способ по п.45, в котором переход от низкой к высокой частоте взаимодействия является показателем положения точки разрыва.
47. Способ по п.45, в котором обратная картина частоты ДНК-ДНК-взаимодействия для образца, полученного от субъекта, по сравнению с контролем является показателем инверсии.
48. Способ по п.45, в котором увеличение частоты ДНК-ДНК-взаимодействия для образца, полученного от субъекта, по сравнению с контролем, в комбинации с увеличением частоты ДНК-ДНК-взаимодействия для более отдаленных областей, является показателем делеции.
49. Способ по п.45, в котором увеличение или уменьшение частоты ДНК-ДНК-взаимодействия для образца, полученного от субъекта, по сравнению с контролем является показателем дупликации или инсерции.
50. Способ по любому из п.п.45-49, в котором используют спектральное кариотипирование и/или FISH перед осуществлением указанного способа.
51. Способ по любому из п.п.45-50, в котором заболевание является наследственным заболеванием.
52. Способ по любому из п.п.45-51, в котором заболевание является злокачественной опухолью.
53. Способ диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия, включающий в себя стадии
(a) получения образца поперечно сшитой ДНК от субъекта;
(b) расщепления поперечно сшитой ДНК первым ферментом рестрикции;
(c) лигирования поперечно сшитых нуклеотидных последовательностей;
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции;
(f) образования кольцевых нуклеотидных последовательностей;
(g) амплификации одной или нескольких последовательностей, которые лигированы с нуклеотидной последовательностью(ями)-мишенью(ями);
(h) мечения двух или более амплифицированных последовательностей;
(i) гибридизации нуклеотидных последовательностей с чипом;
(j) определения частоты взаимодействия между последовательностями ДНК; и
(j) идентификации одного или нескольких локусов, которые были подвергнуты геномной перестройке, которая связана с заболеванием.
54. Способ по п.53, в котором две или более амплифицированных последовательностей метят по-разному.
55. Способ по п.54, в котором две или более амплифицированных последовательностей метят идентично в том случае, когда последовательности находятся на разных хромосомах.
56. Способ по п.53, в котором две или более амплифицированных последовательностей метят идентично в том случае, когда последовательности находятся на одной и той же хромосоме на расстоянии, которое является достаточно большим, чтобы было минимальное перекрывание между сигналами ДНК-ДНК-взаимодействия.
57. Способ анализа для идентификации одного или нескольких агентов, которые модулируют ДНК-ДНК-взаимодействие, включающий в себя стадии
(a) контактирования образца с одним или несколькими агентами;
(b) получения поперечно сшитой ДНК из образца;
(c) расщепления поперечно сшитой ДНК первым ферментом рестрикции;
(d) лигирования поперечно сшитых нуклеотидных последовательностей;
(e) удаления поперечных сшивок;
(f) расщепления нуклеотидных последовательностей вторым ферментом рестрикции;
(g) образования кольцевых нуклеотидных последовательностей;
(h) амплификации одной или нескольких последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью;
(i) необязательно гибридизации амплифицированных последовательностей с чипом; и
(j) определения частоты взаимодействия между последовательностями ДНК,
при этом разница между (i) частотой взаимодействия между последовательностями ДНК в присутствии агента и (ii) частотой взаимодействия между последовательностями ДНК в отсутствие агента является показателем того, что агент модулирует ДНК-ДНК-взаимодействие.
58. Способ определения положения точки разрыва (например, при транслокации), включающий в себя стадии
(a) получения образца поперечно сшитой ДНК;
(b) расщепления поперечно сшитой ДНК первым ферментом рестрикции;
(c) лигирования поперечно сшитых нуклеотидных последовательностей;
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции;
(f) образования кольцевых нуклеотидных последовательностей;
(g) амплификации одной или нескольких последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью;
(h) необязательно гибридизации амплифицированных нуклеотидных последовательностей с чипом;
(j) определения частоты взаимодействия между последовательностями ДНК; и
(j) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле;
при этом переход от низкой частоты к высокой частоте ДНК-ДНК-взаимодействия в образце по сравнению с контролем является показателем положения точки разрыва.
59. Способ определения положения инверсии, включающий в себя стадии
(a) получения образца поперечно сшитой ДНК;
(b) расщепления поперечно сшитой ДНК первым ферментом рестрикции;
(c) лигирования поперечно сшитых нуклеотидных последовательностей;
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции;
(f) образования кольцевых нуклеотидных последовательностей;
(g) амплификации одной или нескольких последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью;
(h) необязательно гибридизации амплифицированных нуклеотидных последовательностей с чипом;
(j) определения частоты взаимодействия между последовательностями ДНК; и
(j) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле;
при этом обратная картина частоты ДНК-ДНК-взаимодействия в образце по сравнению с контролем является показателем инверсии.
60. Способ определения положения делеции, включающий в себя стадии
(a) получения образца поперечно сшитой ДНК;
(b) расщепления поперечно сшитой ДНК первым ферментом рестрикции;
(c) лигирования поперечно сшитых нуклеотидных последовательностей;
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции;
(f) образования кольцевых нуклеотидных последовательностей;
(g) амплификации одной или нескольких последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью;
(h) необязательно гибридизации амплифицированных нуклеотидных последовательностей с чипом;
(j) определения частоты взаимодействия между последовательностями ДНК; и
(j) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле;
при этом уменьшение частоты ДНК-ДНК-взаимодействия в образце по сравнению с контролем является показателем делеции.
61. Способ определения положения дупликации, включающий в себя стадии
(a) получения образца поперечно сшитой ДНК;
(b) расщепления поперечно сшитой ДНК первым ферментом рестрикции;
(c) лигирования поперечно сшитых нуклеотидных последовательностей;
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции;
(f) образования кольцевых нуклеотидных последовательностей;
(g) амплификации одной или нескольких последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью;
(h) необязательно гибридизации амплифицированных нуклеотидных последовательностей с чипом;
(j) определения частоты взаимодействия между последовательностями ДНК; и
(j) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле;
при этом увеличение или уменьшение частоты ДНК-ДНК-взаимодействия в образце, полученном от субъекта, по сравнению с контролем является показателем дупликации или инсерции.
62. Агент, который получен или может быть получен способом анализа по п.57.
63. Применение нуклеотидной последовательности по любому из п.п.1-9 для идентификации одного или нескольких ДНК-ДНК-взаимодействий в образце.
64. Применение нуклеотидной последовательности по любому из п.п.1-9 для диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия.
65. Применение матрицы зондов по п.18 или набора зондов по любому из п.п.19-30 или 33 для идентификации одного или нескольких ДНК-ДНК-взаимодействий в образце.
66. Применение матрицы зондов по п.18 или набора зондов по любому из п.п.19-30 или 33 для диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия.
67. Применение чипа по любому из п.п.34-39 или 42 для идентификации одного или нескольких ДНК-ДНК-взаимодействий в образце.
68. Применение чипа по любому из п.п.34-39 или 42 для диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия.
69. Применение по любому из п.п.64, 66 или 68, в котором диагностика или прогнозирование представляют собой пренатальную диагностику или прогнозирование.
70. Способ, по существу описанный в данной публикации со ссылками на любые примеры или фигуры.
71. Матрица зондов, по существу описанная в данной публикации со ссылками на любые примеры или фигуры.
72. Набор зондов, по существу описанный в данной публикации со ссылками на любые примеры или фигуры.
73. Способ, по существу описанный в данной публикации со ссылками на любые примеры или фигуры.
74. Чип, по существу описанный в данной публикации со ссылками на любые примеры или фигуры.
75. Способ анализа, по существу описанный в данной публикации со ссылками на любые примеры или фигуры.
76. Агент, по существу описанный в данной публикации со ссылками на любые примеры или фигуры.
77. Применение, по существу описанное в данной публикации со ссылками на любые примеры или фигуры.
ССЫЛКИ
Blanton J, Gaszner M, Schedl P. 2003. Protein:protein interactions and the pairing of boundary elements in vivo. Genes Dev 17:664-75.
Dekker, J., Rippe, K., Dekker, M., and Kleckner, N. 2002. Capturing chromosome conformation. Science 295: 1306-11.
Drissen R, Palstra RJ, Gillemans N, Splinter E, Grosveld F, Philipsen S, de Laat W. 2004. The active spatial organization of the beta-globin locus requires the transcription factor EKLF. Genes Dev 18:2485-90.
Horike S, Cai S, Miyano M, Cheng JF, Kohwi-Shigematsu T. 2005. Loss of silent-chromatin looping and impaired imprinting of DLX5 in Rett syndrome. Nat Genet 37:31-40.
Murrell A, Heeson S, Reik W. 2004. Interaction between differentially methylated regions partitions the imprinted genes Igf2 and H19 into parent-specific chromatin loops. Nat Genet 36:889-93.
Palstra, R.J., Tolhuis, B., Splinter, E., Nijmeijer, R., Grosveld, F., and de Laat, W. 2003. The beta-globin nuclear compartment in development and erythroid differentiation. Nat Genet 35: 190-4.
Patrinos, G.P., de Krom, M., de Boer, E., Langeveld, A., Imam, A.M.A, Strouboulis, J., de Laat, W., and Grosveld, F.G. (2004). Multiple interactions between regulatory regions are required to stabilize an active chromatin hub. Genes & Dev. 18: 1495-1509.
Spilianakis CG, Flavell RA. 2004. Long-range intrachromosomal interactions in the T helper type 2 cytokine locus. Nat Immunol 5:1017-27.
Tolhuis, B., Palstra, R.J., Splinter, E., Grosveld, F., and de Laat, W. 2002. Looping and interaction between hypersensitive sites in the active beta-globin locus. Molecular Cell 10: 1453-65.
Vakoc CR, Letting DL, Gheldof N, Sawado T, Bender MA, Groudine M, Weiss MJ, Dekker J, Blobel GA. 2005. Proximity among distant regulatory elements at the beta-globin locus requires GATA-1 and FOG-1. Mol Cell. 17:453-62.
Все публикации, указанные в приведенном выше описании, включены в данное описание в виде ссылки. Специалистам в данной области будут понятны различные модификации и варианты описанных способов и системы согласно изобретению, не выходящие за рамки объема и не отходящие от сути изобретения. Хотя изобретение описано в связи с конкретными предпочтительными вариантами его осуществления, следует понимать, что заявленное изобретение никоим образом не следует ограничивать такими конкретными вариантами. В действительности, различные модификации описанных способов осуществления изобретения, которые очевидны для специалистов в области молекулярной биологии или родственных областях, входят в объем приведенной ниже формулы изобретения.
Реферат
Изобретение относится к области биотехнологии, в частности к способу анализа частоты взаимодействия нуклеотидной последовательности-мишени с одной или несколькими представляющими интерес нуклеотидными последовательностями. Способ предусматривает (а) получение образца поперечно сшитой ДНК и (b) расщепление поперечно сшитой ДНК первым ферментом рестрикции. Далее проводят (с) лигирование поперечно сшитых нуклеотидных последовательностей, (d) удаление поперечных сшивок; (е) расщепление нуклеотидных последовательностей вторым ферментом рестрикции (f) лигирование одной или нескольких последовательностей ДНК, имеющих известный состав нуклеотидов, с доступным сайтом(ами) расщепления вторым ферментом рестрикции, который фланкирует одну или несколько представляющих интерес нуклеотидных последовательностей. Проводят (g) амплификацию одной или нескольких представляющих интерес нуклеотидных последовательностей с использованием по меньшей мере двух олигонуклеотидных праймеров, причем каждый праймер гибридизуется с последовательностями ДНК, которые фланкируют представляющие интерес нуклеотидные последовательности. Далее осуществляют (h) гибридизацию амплифицированной последовательности(ей) с чипом; и (i) определения частоты взаимодействия между последовательностями ДНК. 9 н. и 20 з.п. ф-лы, 19 ил., 2 табл., 8 пр.
Формула
(a) получения образца поперечно сшитой ДНК;
(b) расщепления поперечно сшитой ДНК первым ферментом рестрикции, который имеет, по меньшей мере, один сайт рестрикции, смежный с нуклеотидной последовательностью-мишенью, или находящийся внутри нее;
(c) лигирования поперечно сшитых нуклеотидных последовательностей при низкой концентрации ДНК, что благоприятствует лигированию поперечно сшитых фрагментов ДНК вместе (т.е. внутримолекулярному взаимодействию);
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции, который, предпочтительно, имеет, по меньшей мере, один сайт рестрикции, смежный с нуклеотидной последовательностью-мишенью или находящийся внутри нее;
(f) лигирования одной или более последовательностей ДНК с уникальным известным нуклеотидным составом с доступным сайтом (сайтами) расщепления вторым ферментом рестрикции, который фланкирует одну или более представляющих интерес нуклеотидных последовательностей, где последовательности ДНК с известным нуклеотидным составом, таким образом, лигируются с олигонуклеотидом, полученным на стадии (е);
(g) амплификации одной или более представляющих интерес нуклеотидных последовательностей с использованием, по меньшей мере, двух олигонуклеотидных праймеров, при этом каждый праймер гибридизуется с последовательностями ДНК, которые фланкируют представляющие интерес нуклеотидные последовательности;
(h) гибридизации амплифицированной последовательности(ей) с чипом; и
(i) определения частоты взаимодействия между последовательностями ДНК на основании относительного количества ДНК, гибридизованной с каждым зондом в чипе на стадии (h).
(a) получения образца поперечно сшитой ДНК;
(b) расщепления поперечно сшитой ДНК первым ферментом рестрикции, который имеет, по меньшей мере, один сайт рестрикции, смежный с нуклеотидной последовательностью-мишенью, или находящийся внутри нее;
(c) лигирования поперечно сшитых нуклеотидных последовательностей при низких концентрациях ДНК, что благоприятствует лигированию поперечно сшитых фрагментов ДНК вместе (т.е. внутримолекулярному взаимодействию);
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции, который, предпочтительно, имеет, по меньшей мере, один сайт рестрикции, смежный с нуклеотидной последовательностью-мишенью или находящийся внутри нее;
(f) образования кольцевых нуклеотидных последовательностей через внутримолекулярное лигирование между сайтами узнавания вторым ферментом рестрикции в условиях разбавления;
(g) амплификации одной или нескольких нуклеотидных последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью;
(h) гибридизации амплифицированных последовательностей с чипом; и
(j) определения частоты взаимодействия между последовательностями ДНК на основании относительного количества ДНК, гибридизованной с каждым зондом в чипе на стадии (h).
(a) получения образца поперечно сшитой ДНК;
(b) расщепления поперечно сшитой ДНК первым ферментом рестрикции, который имеет, по меньшей мере, один сайт рестрикции, смежный с нуклеотидной последовательностью-мишенью или находящийся внутри нее;
(c) лигирования поперечно сшитых нуклеотидных последовательностей при низких концентрация ДНК, что благоприятствует лигированию поперечно сшитых фрагментов ДНК вместе (т.е. внутримолекулярному взаимодействию);
(d) удаления поперечных сшивок;
(e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции, который имеет, по меньшей мере, один сайт рестрикции, смежный с нуклеотидной последовательностью-мишенью или находящийся внутри нее; и
(f) образования кольцевых нуклеотидных последовательностей через внутримолекулярное лигирование между сайтами узнавания вторым ферментом рестрикции в условиях разбавлени.
(a) осуществления стадий (a)-(i) по любому из пп.1-11; и
(b) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле;
где переход от низкой к высокой частоте ДНК-ДНК-взаимодействия в образце по сравнению с контролем является показателем положения точки разрыва.
(a) осуществления стадий (a)-(i) по любому из пп.1-11; и
(b) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле;
где обратная картина частот ДНК-ДНК-взаимодействий для образца по сравнению с контролем является показателем инверсии.
(a) осуществления стадий (a)-(i) по любому из пп.1-11; и
(b) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле;
где уменьшение частоты ДНК-ДНК-взаимодействия в образце по сравнению с контролем является показателем делеции.
(a) осуществления стадий (a)-(i) по любому из пп.1-11; и
(b) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле;
где увеличение или уменьшение частоты ДНК-ДНК-взаимодействия в случае образца от субъекта по сравнению с контролем является показателем дупликации или инсерции.

Комментарии