Фармацевтическая композиция для лечения и/или предотвращения злокачественной опухоли - RU2714205C2
Код документа: RU2714205C2
Чертежи




Описание
Область техники
[0001] Настоящее изобретение относится к новому медицинскому применению антитела к белку CSPG5 или его фрагмента, например, в качестве терапевтического и/или профилактического средства для злокачественных опухолей.
Предшествующий уровень техники
[0002] Злокачественная опухоль представляет собой заболевание занимающее лидирующее положение в причинах смертности. Используемые в настоящее время способы лечения преимущественно основаны на хирургическом лечении в комбинации с лучевой терапией и химиотерапией. Несмотря на недавние разработки новых хирургических способов и открытие новых средств против злокачественных опухолей, результаты лечения злокачественных опухолей, за исключением определенных злокачественных опухолей, в настоящее время пока еще не улучшены. В результате успехов молекулярной биологии и иммунологии злокачественных опухолей, идентифицированы антитела, которые специфически реагируют со злокачественными опухолями, антигены злокачественных опухолей, распознаваемые цитотоксическими T-клетками, гены, кодирующие антигены злокачественных опухолей и т.п. Таким образом, необходимо разработка конкретных средств лечения злокачественных опухолей, направленных на антигены злокачественных опухолей.
[0003] При терапии злокачественных опухолей для снижения побочных эффектов желательно, чтобы пептиды (включая полипептиды), распознаваемые в качестве антигенов, редко присутствовали в нормальных клетках и специфически присутствовали в злокачественных клетках. В 1991 году Boon et al. из Ludwig Laboratory в Бельгии способом экспрессионного клонирования кДНК с использованием аутологичной линии опухолевых клеток и реагирующих на опухоль T-клеток выделили антиген меланомы человека MAGE1, распознаваемый положительными по CD8 T-клетками (непатентная литература 1). Затем, был опубликован способ SEREX (серологической идентификации антигенов посредством рекомбинантного экспрессионного клонирования), который представляет собой способ идентификации антигена злокачественной опухоли, распознаваемого антителом, продуцируемым в ответ на аутологичную злокачественную опухоль в организме пациента, способом экспрессионного клонирования генов (патентная литература 1, непатентная литература 2). Этим способом выделено несколько антигенов злокачественных опухолей, которые редко экспрессируются в нормальных клетках и специфически экспрессируются в злокачественных клетках (непатентная литература 3). Кроме того, в клинических испытаниях проводят клеточную терапию, в которой применяют иммунные клетки, мишенью которых является часть аминокислотной последовательности такого антигена злокачественной опухоли и специфически реагирующие с антигеном злокачественной опухоли, и используют специфичные для злокачественных опухолей иммунотерапевтические средства, такие как вакцина, содержащая антиген злокачественной опухоли.
[0004] В то же время в мире известны различные типы лекарственных средств для лечения злокачественных опухолей на основе антител, направленных на конкретный антигенный белок на злокачественных клетках. Большинство из антигенных белков-мишеней обеспечивают определенные уровни лечебного действия в качестве специфического для злокачественной опухоли терапевтического средства и привлекают внимание; однако они также экспрессируются на множестве нормальных клеток. Вследствие этого серьезное беспокойство вызывают побочные эффекты, так как в результате введения антител повреждаются не только злокачественные клетки, но также и нормальные клетки. Таким образом, полагают, что можно осуществлять лечение лекарственным средством на основе антитела с меньшим количеством побочных эффектов, если можно идентифицировать антиген, который специфически экспрессирован только на поверхности злокачественных клеток и не экспрессирован на нормальных клетках, и в качестве лекарственного средства можно использовать антитело, направленное к этому антигену.
Список цитируемых документов
Патентная литература
[0005] Патентная литература 1: патент США № 5698396
Непатентная литература
[0006] Непатентная литература 1: Bruggen P. et al. Science, 254: 1643-1647 (1991)
Непатентная литература 2: Proc. Natl. Acad. Sci. USA, 92: 11810-11813 (1995)
Непатентная литература 3: Int. J. Cancer, 72: 965-971 (1997)
Сущность изобретения
Техническая задача
[0007] Целью настоящего изобретения является идентификация антигенного белка злокачественной опухоли, специфически экспрессируемого на поверхности злокачественных клеток, и обеспечение применения антитела, направленного к антигенному белку злокачественной опухоли, в качестве терапевтического и/или профилактического средства для злокачественной опухоли.
Решение задачи
[0008] Авторы настоящего изобретения выделили антиген, специфически экспрессируемый в злокачественных опухолях, способом SEREX с использованием библиотеки кДНК, полученной из ткани семенника собаки, и сыворотки несущей рак молочной железы собаки, а затем выделив кДНК, кодирующую белок CSPG5. Белок CSPG5 может связываться с антителами, присутствующими в сыворотке, полученной у различных несущих злокачественные опухоли живых организмов. Авторы настоящего изобретения также выявили, что белок CSPG5 специфически экспрессируется в клетках рака молочной железы, рака легких, опухоли головного мозга, лейкоза, злокачественной лимфомы, аденокарциномы, мастоцитомы, плоскоклеточной карциномы, меланомы или нейробластомы; и что часть белка CSPG5 специфически экспрессируется на поверхности этих злокачественных клеток. Белок CSPG5 (протеогликан с хондроитинсульфатами 5) представляет собой трансмембранный белок I типа и один из белков семейства нейрегулинов. Также опубликовано, что белок CSPG5 связывается с белком ErbB3, осуществляя функцию фактора роста; и что экспрессия белка CSPG5 возрастает при раке яичника с мутацией белка BRCA1 (Kinugasa, Y., et al., 2004, Biochem. Biophys. Res. Commun., 321: 1045; Press, J. Z., et al., 2010, Neoplasia., 12 (12): 993-1002). Также известно, что белок CSPG5 высоко экспрессирован в тканях нервной системы, таких как ганглиозные клетки сетчатки, клетки Пуркинье и гиппокамп, и осуществляет функцию фактора пролиферации/дифференцировки нервных клеток, участвующий в процессе удлинения аксонов нервных клеток (Yasuda, Y. et al., 1998, Neurosci. Res., 32: 313; Aono, S., et al., 2006, J. Neurosci. Res., 83: 110; Nakanishi, K., et al., 2006, J. Biol. Chem., 281: 24970). Однако публикаций о том, что белок CSPG5 обладает индуцирующей иммунитет против злокачественных клеток активностью и, таким образом, пригоден для лечения и предотвращения злокачественной опухоли не существует.
[0009] Также авторы изобретения получили молекулы белка CSPG5, состоящие из аминокислотных последовательностей, представленных SEQ ID NO: 2, 4, 6, 8, 10, 12, 14 и 16 на основе полученного гена CSPG5 собаки и гомологичных ему генов человека, кошки и мыши, и антитела к этим молекулам белка CSPG5. Затем они выявили, что антитела к частям каждой из этих молекул белка CSPG5, экспрессируемым на поверхностях отдельных злокачественных клеток, другими словами, их внеклеточным областям, повреждают злокачественные клетки, экспрессирующие белок CSPG5. На основе этого открытия осуществлено настоящее изобретение.
[0010] Таким образом, настоящее изобретение обладает указанными ниже признаками.
(1) Фармацевтическая композиция для лечения и/или предотвращения злокачественной опухоли, содержащая в качестве активного ингредиента антитело или его фрагмент, обладающие иммунологической реакционноспособностью в отношении белка CSPG5 или его фрагмента, состоящего по меньшей мере из 7 или более последовательных аминокислотных остатков.
(2) Фармацевтическая композиция по (1), где белок CSPG5 состоит из любой из аминокислотных последовательностей, представленных SEQ ID NO: 8, 4, 6, 10 и 12, или аминокислотных последовательностей с идентичностью аминокислот с этими аминокислотными последовательностями, составляющей 80% или более.
(3) Фармацевтическая композиция по (1) или (2), где злокачественная опухоль представляет собой лейкоз или злокачественную лимфому.
(4) Фармацевтическая композиция по любому из (1)-(3), где антитело представляет собой моноклональное антитело или поликлональное антитело.
(5) Фармацевтическая композиция по любому из (1)-(4), где антитело представляет собой антитело человека, гуманизированное антитело, химерное антитело, одноцепочечное антитело или биспецифическое антитело.
[0011] Описание включает описание патентной заявки Японии № 2015-093640, на основании которой по настоящей заявке испрашивается приоритет.
Полезные эффекты изобретения
[0012] Антитело к белку CSPG5, используемое по настоящему изобретению, повреждает злокачественные клетки. Таким образом, антитело к белку CSPG5 пригодно для лечения и/или профилактики злокачественных опухолей.
Краткое описание чертежей
[0013] [Фигура 1] на фигуре 1 представлены профили экспрессии идентифицированного гена CSPG5 в опухолевых тканях или линиях злокачественных клеток собаки. На фигуре под кодовым номером 1 продемонстрированы профили экспрессии гена CSPG5 собаки в отдельных тканях и линиях клеток собаки; а под кодовым номером 2 продемонстрированы профили экспрессии гена GAPDH собаки в отдельных тканях и линиях клеток собаки.
[Фигура 2] на фигуре 2 представлены профили экспрессии идентифицированного гена CSPG5 в опухолевых тканях или линиях злокачественных клеток человека.
[Фигура 3] на фигуре 3 представлены профили экспрессии идентифицированного гена CSPG5 в опухолевых тканях или линиях злокачественных клеток мыши. Под кодовым номером 3 продемонстрированы профили экспрессии гена CSPG5 мыши в отдельных тканях и линиях клеток мыши; под кодовым номером 4 продемонстрированы профили экспрессии гена GAPDH мыши в отдельных тканях и линиях клеток мыши.
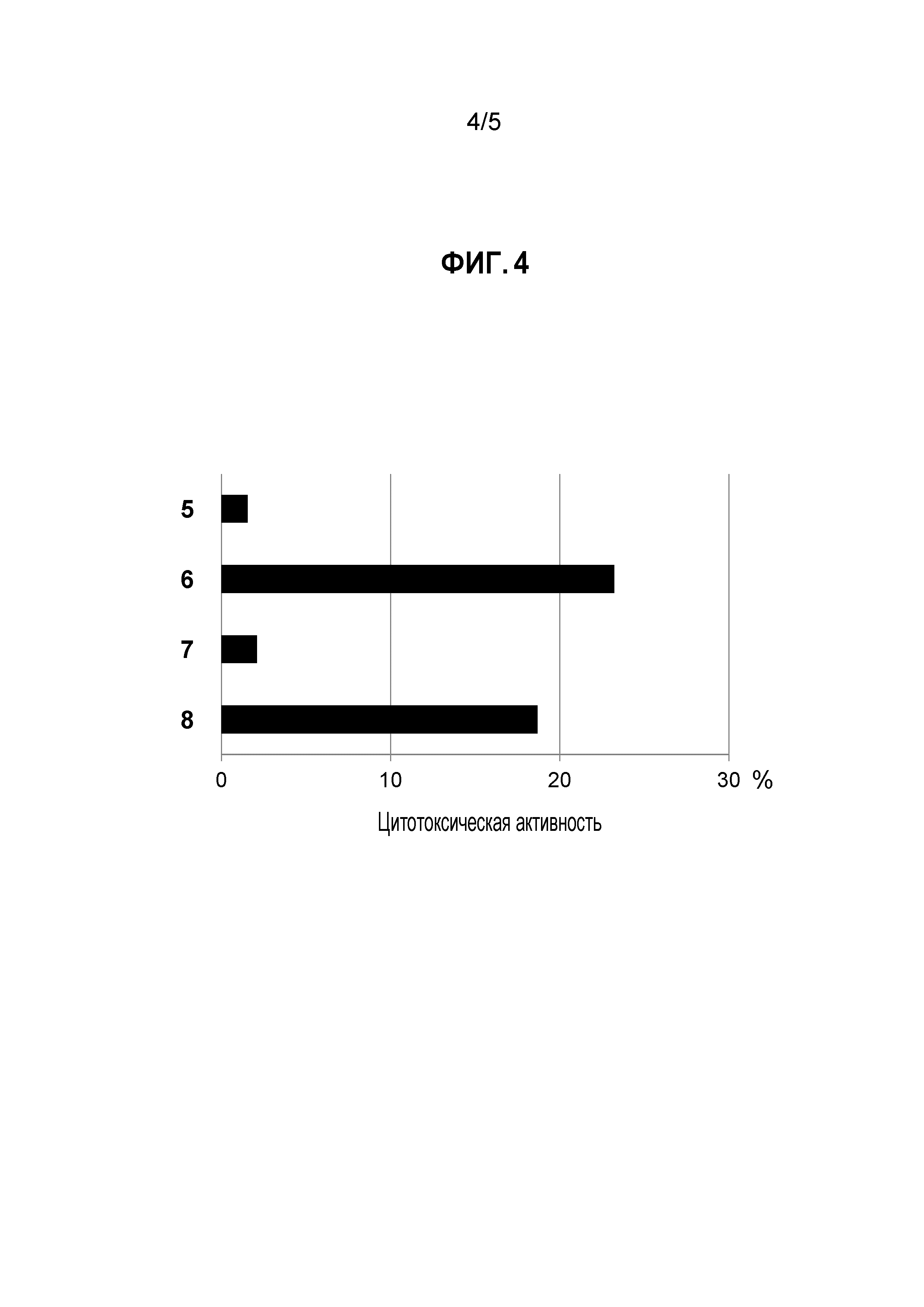
[Фигура 4] на фигуре 4 представлена цитотоксическая активность поликлонального антитела к белку CSPG5 (поликлональное антитело к CSPG5) в отношении линии клеток лейкоза (K562) и клеток злокачественной лимфомы (L-1236), экспрессирующих ген CSPG5. На фигуре под кодовым номером 5 продемонстрирована цитотоксическая активность против клеток K562 при добавлении контрольного поликлонального антитела; под кодовым номером 6 продемонстрирована цитотоксическая активность против клеток K562 при добавлении поликлонального антитела к CSPG5; под кодовым номером 7 продемонстрирована цитотоксическая активность против клеток L-1236 при добавлении контрольного поликлонального антитела; и под кодовым номером 8 продемонстрирована цитотоксическая активность против клеток L-1236 при добавлении поликлонального антитела к CSPG5.
[Фигура 5] на фигуре 5 представлена цитотоксическая активность моноклонального антитела к белку CSPG5 (моноклональное антитело к CSPG5) в отношении линии клеток лейкоза (K562) и клеток злокачественной лимфомы (L-1236), экспрессирующих ген CSPG5. На фигуре под кодовым номером 9 продемонстрирована цитотоксическая активность против клеток K562 при добавлении контрольного моноклонального антитела; под кодовым номером 10 продемонстрирована цитотоксическая активность против клеток K562 при добавлении моноклонального антитела к CSPG5; под кодовым номером 11 продемонстрирована цитотоксическая активность против клеток L-1236 при добавлении контрольного моноклонального антитела; и под кодовым номером 12 продемонстрирован цитотоксическая активность против клеток L-1236 при добавлении моноклонального антитела к CSPG5.
Описание вариантов осуществления
[0014] Противоопухолевая активность антитела, используемого по настоящему изобретению, против полипептида, состоящего из аминокислотной последовательности, представленной SEQ ID NO:2, 4, 6, 8, 10, 12, 14 или 16, можно оценивать, анализируя, подавление роста опухоли in vivo у несущего злокачественную опухоль животного или анализируя проявление антителом цитотоксической активности против опухолевых клеток, экспрессирующих полипептид, in vitro посредством иммунных клеток или комплемента, как описано далее.
[0015] Нуклеотидная последовательность полинуклеотида, кодирующего белок, состоящий из аминокислотной последовательности, представленной SEQ ID NO:2, 4, 6, 8, 10, 12, 14 или 16, представлена SEQ ID NO:1, 3, 5, 7, 9, 11, 13 или 15, соответственно.
[0016] Аминокислотная последовательность, представленная SEQ ID NO:2 в списке последовательностей, предоставляемом по настоящему изобретению, представляет собой аминокислотную последовательность белка CSPG5, выделенного как полипептид, связывающийся с антителом, специфически присутствующим в сыворотке, полученной у несущей злокачественную опухоль собаки способом SEREX с использованием библиотеки кДНК, полученной из ткани семенника собаки, и сыворотке несущей рак молочной железы собаки; аминокислотные последовательности, представленные SEQ ID NO: 4, 6, 8, 10 и 12, выделены как гомологи человека этого полипептида; аминокислотная последовательность, представленная SEQ ID NO:14, выделана как гомолог кошки этого полипептида; и аминокислотная последовательность, представленная SEQ ID NO:16, выделана как гомолог мыши этого полипептида (см., пример 1, описанный далее).
[0017] На основании аминокислотной последовательности известно, что белок CSPG5 представляет собой трансмембранный белок I типа, и что внеклеточная область, прогнозируемая на основе его последовательности, экспрессирована на поверхности нервных клеток. В настоящей заявке подтверждено, что внеклеточная область белка CSPG5 фактически экспрессирована (присутствует) на поверхностях различных типов злокачественных клеток. В настоящем изобретении предпочтительно используют антитело, связывающееся с внеклеточной областью белка CSPG5 на злокачественной клетке или связывающееся с полипептидом с идентичностью аминокислот с аминокислотной последовательностью этой внеклеточной области, составляющей 80% или более, предпочтительно 85% или более, более предпочтительно 90% или более, еще более предпочтительно 95% или более, 97% или более, 98% или более или 99% или более.
[0018] Антитело к белку CSPG5, используемое по настоящему изобретению, может представлять собой антитело любого типа при условии, что оно может проявлять противоопухолевую активность. Примеры антитела включают моноклональное антитело, поликлональное антитело, синтетическое антитело, полиспецифическое антитело, антитело человека, гуманизированное антитело, химерное антитело, одноцепочечное антитело (scFV) и фрагмент антитела (например, Fab, F(ab')2, Fv). Эти антитела и их фрагменты можно получать способами, известными специалистам в данной области. По настоящему изобретению необходимо антитело, способное к специфическому связыванию с белком CSPG5, и предпочтительным является моноклональное антитело; однако можно использовать поликлональные антитела при условии, что можно стабильно получать гомогенные антитела. Когда индивидуум представляет собой человека, во избежание или для подавления реакции отторжения необходимо антитело человека или гуманизированное антитело.
[0019] Фраза "специфическое связывание с белком CSPG5", используемая в настоящем документе, означает специфическое связывание с белком CSPG5 и по существу отсутствие связывания с белками, отличными от белка CSPG5.
[0020] Индивидуум, который является целью лечения и/или профилактика злокачественной опухоли по настоящему изобретению, представляет собой млекопитающее, такого как человек, комнатное животное, домашнее животное и животное для соревнований, предпочтительно человек.
[0021] Ниже описано получение антигена, получение антитела и фармацевтической композиции по настоящему изобретению.
[0022] <Получение антигена для получения антитела>
Белок или его фрагмент, используемый в качестве сенсибилизирующего антигена для получения антитела к белку CSPG5 (антитело к CSPG5) по настоящему изобретению, можно получать, например, у людей, собак, кошек, мышей, крыс, коров, лошадей и кур, и виды животных, у которых получают белок или его фрагмент, не ограничены ими. Белок или его фрагмент предпочтительно отбирают, учитывая совместимость с исходной клеткой, используемой в слиянии клеток. Как правило, белок, получаемый у млекопитающего, предпочтительно представляет собой белок, получаемый у человека, и конкретно предпочтителен белок, получаемый у человека. Например, если белок CSPG5 представляет собой белок CSPG5 человека, можно использовать белок CSPG5 человека, являющийся его частью полипептид, клетку, экспрессирующую белок CSPG5 человека, и т.п.
[0023] Аминокислотные последовательности белка CSPG5 и кодирующие их нуклеотидные последовательности и их гомологи можно получать с использованием, например, GenBank (NCBI of the United State) и с использованием такого алгоритма, как BLAST и FASTA (Karlin and Altschul, Proc. Natl. Acad. Sci. USA, 90: 5873-5877,1993; Altschul et al., Nucleic Acids Res. 25: 3389-3402, 1997).
[0024] Например, если в качестве основы используют белок CSPG5 человека, в качестве мишени используют нуклеотидную последовательность (SEQ ID NO:3, 5, 7, 9 или 11), кодирующую белок CSPG5 человека и нуклеиновую кислота с идентичностью нуклеотидов с этой нуклеотидной последовательностью, составляющей от 70% до 100%, предпочтительно от 80% до 100%, более предпочтительно от 90% до 100%, еще более предпочтительно от 95% до 100% (например, от 97% до 100%, от 98% до 100%, от 99% до 100% или от 99,5% до 100%). Также в качестве мишени используют аминокислотную последовательность (SEQ ID NO:4, 6, 8, 10 или 12) белка CSPG5 человека и полипептид с идентичностью аминокислот с этой аминокислотной последовательностью, составляющей от 70% до 100%, предпочтительно от 80% до 100%, более предпочтительно от 90% до 100% или еще более предпочтительно от 95% до 100% (например, от 97% до 100%, от 98% до 100%, от 99% до 100% или от 99,5% до 100%). Термин "идентичность нуклеотидов", используемый в настоящем документе, относится к проценту (%) количества идентичных нуклеотидов относительно общего количества нуклеотидов, когда две нуклеотидных последовательности выровнены так, что они обладают максимальной степенью сходства при соответствующем внесении пропуска(ов). Подобным образом, термин "идентичность аминокислот" относится к проценту (%) количества идентичных аминокислот относительно общего количества аминокислот, когда две аминокислотных последовательностей выровнены так, что они обладают максимальной степенью сходства при соответствующем внесении пропуска(ов).
[0025] Фрагмент белка CSPG5 указан так, чтобы его длина была равна или превышала длину эпитопа (антигенной детерминанты) в аминокислотах и была меньше, чем у полноразмерного белка. Эпитоп представляет собой полипептидный фрагмент, который представляет собой минимальный участок, распознаваемый антителом у млекопитающего, предпочтительно человека, и обладает антигенностью или иммуногенностью, и включает аминокислотные последовательности с длиной приблизительно от 7 до 12 аминокислот, например, от 8 до 11 аминокислот.
[0026] Белок CSPG5 человека и полипептид, содержащий являющийся его частью пептид, можно синтезировать, например, способом химического синтеза, таким как способ с Fmoc (способ с флуоренилметилоксикарбонилом) и способ с tBoc (способ с трет-бутилоксикарбонилом) (Biochemical Experiment Course 1, Chemistry of Protein IV, Chemical Modification and Synthesis of Peptide, edited by the Japan Biochemical Society, Tokyo Kagaku Dojin (Japan), 1981) или синтезировать стандартным способом с использованием коммерчески доступного синтезатора пептидов. Альтернативно, желаемый полипептид можно получать способом генетической инженерии, известным в данной области (например, Green, M. R. and Sambrook, J., 2012, Molecular Cloning: A Laboratory Manual 4th Ed., Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York, Ausubel et al., Short Protocols in Molecular Biology, third edition, A compendium of Methods from Current Protocols in Molecular Biology (1995), John Wiley & Sons); более конкретно, получая полинуклеотид, кодирующий полипептид, интегрируя полинуклеотид в экспрессирующий вектор, вводя вектор в клетку-хозяина и позволяя клетке-хозяину продуцировать полипептид.
[0027] Полинуклеотид, кодирующий полипептид, можно легко получать стандартным способом с использованием способа генетической инженерии, известного в данной области, или коммерчески доступного синтезатора нуклеиновых кислот. Например, ДНК с нуклеотидной последовательностью SEQ ID NO:3 можно получать, проводя ПЦР с использованием библиотеки хромосомной ДНК человека или библиотеки кДНК человека в качестве матрицы и пары праймеров, сконструированных так, чтобы амплифицировать нуклеотидную последовательность, представленную SEQ ID NO:3. Условия реакции ПЦР можно определить соответствующим образом; например, условия реакции включают условия, когда с использованием термостабильной ДНК-полимеразы (например, полимеразы Taq) и содержащего Mg2+ буфера для ПЦР цикл, состоящий из реакции денатурации при 94°C в течение 30 секунд, реакции отжига при 55°C в течение периода от 30 секунд до 1 минуты и реакции элонгации при 72°C в течение 2 минут, например, повторяют 30 раз, а затем проводят реакцию при 72°C в течение 7 минут; однако условия реакции этим конкретно не ограничены. Способ и условия ПЦР описаны, например, в Ausubel et al., Short Protocols in Molecular Biology, third edition, A compendium of Methods from Current Protocols in Molecular Biology (1995), John Wiley & Sons (particularly Chapter 15).
[0028] Необходимую ДНК можно выделять, получая соответствующий зонд и праймеры на основе информации о нуклеотидных последовательностях, представленных SEQ ID NO:1, 3, 5, 7, 9, 11, 13 или 15 в списке последовательностей настоящей заявки, и посредством зонда и праймеров подвергая скринингу библиотеку кДНК человека и т.д.. Библиотеку кДНК предпочтительно получать из клеток, органа или ткани, экспрессирующих белок, состоящий из аминокислотной последовательности, представленной SEQ ID NO:2, 4, 6, 8, 10, 12, 14 или 16. Примеры таких клеток или тканей в качестве неограничивающих примеров включают клетки или ткани, получаемые из злокачественных опухолей или незлокачественных опухолей, таких как опухоль семенников или лейкоз, рак молочной железы, лимфома, опухоль головного мозга, рак легких, рак толстого кишечника, мастоцитома, меланома и нейробластома. Указанные выше действия, такие как получение зонда или праймеров, конструирование библиотеки кДНК, скрининг библиотеки кДНК и клонирование требуемого гена, известны специалистам в данной области и их можно проводить способом, описанным, например, в Green, M. R. and Sambrook (описанной выше), Ausbel et al. (описанной выше). Из полученной таким образом ДНК можно получать ДНК, кодирующую белок CSPG5 и являющийся его частью пептид.
[0029] В качестве клеток-хозяев можно использовать клетки любого типа при условии, что они могут экспрессировать указанный выше полипептид. Примеры прокариотических клеток включают Escherichia coli, а примеры эукариотических клеток включают, но не ограничены ими, дрожжевые клетки, включая почкующиеся дрожжи и делящиеся дрожжи, клетки насекомых, такие как клетки тутового шелкопряда, яйцеклетки Xenopus и клетки млекопитающих, такие как клетки почки обезьяны COS1, клетки яичника китайского хомяка CHO, линия клеток эмбриональной почки человека HEK293 и линия клеток эмбриональной кожи мыши NIH3T3.
[0030] Когда в качестве клеток хозяев используют прокариотические клетки, в качестве экспрессирующего вектора используют экспрессирующий вектор, содержащий участок начала репликации в прокариотической клетке, промотор, участок связывания рибосомы, участок множественного клонирования, терминатор, ген устойчивости к лекарственному средству, ген комплемента ауксотрофии и т.п. Примеры экспрессирующего вектора для Escherichia coli могут включать систему pUC, pBluescriptII, экспрессирующую систему pET и экспрессирующую систему pGEX. Полипептид, кодируемый ДНК, можно экспрессировать в прокариотических клетках-хозяевах посредством интеграции ДНК, кодирующей полипептид, в такой экспрессирующий векторе; трансформации вектором прокариотических клеток-хозяев и культивирования полученных трансформантов. После этого полипептид можно экспрессировать в виде части слитого с другим белком белка.
[0031] Когда в качестве клеток хозяев используют эукариотические клетки, в качестве экспрессирующего вектора используют экспрессирующий вектор для эукариотических клеток, содержащий промотор, область сплайсинга, дополнительный участок поли (A) и т.п. Примеры таких экспрессирующих векторов могут включать pKA1, pCDM8, pSVK3, pMSG, pSVL, pBK-CMV, pBK-RSV, вектор EBV, pRS, pcDNA3.1 и pYES2. Полипептид, кодируемый ДНК, можно экспрессировать в эукариотических клетках-хозяевах, способом, подобным способу, указанному выше, интегрируя ДНК, кодирующую указанный выше полипептид, в такой экспрессирующий вектор, трансформируя вектором эукариотические клетки-хозяева и культивируя полученные в результате трансформанты., Когда в качестве экспрессирующего вектора используют pIND/V5-His, pFLAG-CMV-2, pEGFP-N1, pEGFP-C1 или т.п., цказанный выше полипептид можно экспрессировать в виде части слитого белка, связанного с меткой, такой как метка His (например, содержащая от (His)6 до (His)10), метка FLAG, метка myc, метка HA и GFP.
[0032] Введение экспрессирующего вектора в клетку-хозяина можно проводить способом, хорошо известным в данной области, таким как способ электропорации, способ с фосфатом кальция, способ с липосомами, способ с DEAE-декстраном, микроинъекция, инфекция вирусом, липофекция и связывание с проходящим через клеточную мембрану пептидом.
[0033] Для выделения/очистки требуемого полипептида из клеток-хозяев можно использовать комбинацию способов разделения, известных в данной области. Примеры способов разделения в качестве неограничивающих примеров включают обработку денатурирующим средством, таким как мочевина или поверхностно-активное вещество, обработку ультразвуком, ферментативное расщепление, высаливание и осаждение после разделения на фракции с селективным растворителем, диализ, центрифугирование, ультрафильтрация, гель-фильтрацию, SDS-PAGE, изоэлектрофокусирование, ионообменную хроматографию, гидрофобную хроматографию, аффинную хроматографию и хроматографию с обращенной фазой.
[0034] <Структура антитела>
Антитело представляет собой гетеромультимерный гликопротеин, как правило, содержащий по меньшей мере две тяжелых цепи и две легких цепи. Каждый из четырех типов иммуноглобулинов за исключением IgM представляет собой гетеротетрамерный гликопротеин массой приблизительно 150 кДа, преимущественно состоящий из двух идентичных легких (L) цепей и двух идентичных тяжелых (H) цепей. Как правило, каждая из легких цепей связана с тяжелой цепью одной дисульфидной ковалентной связью; в то время как количество дисульфидных связей между тяжелыми цепями варьирует в зависимости от изотипов иммуноглобулинов. Также каждая из тяжелых цепей и легких цепей содержит внутрицепочечную дисульфидную связь. Каждая из тяжелых цепей на одном из концов содержит вариабельный домен (область VH) с последующими несколькими константными областями. Каждая из легких цепей содержит вариабельный домен (область VL) на одном из концов, и одну константную область на другом конце. Вариабельный домен легкой цепи выровнен с вариабельным доменом тяжелой цепи. Константная область легкой цепи выровнена с первой константной областью, следующей за вариабельным доменом тяжелой цепи. В вариабельном домене антитела, находятся три особых области, называемых определяющие комплементарность области (CDR), которые представляют собой вариабельные части и на их основе антитело обладает специфичностью связывания. В вариабельной области относительно консервативную часть называют каркасной областью (FR). Каждый из полных вариабельных доменов тяжелых цепей и легких цепей содержит 4 FR (FR1, FR2, FR3 и FR4 последовательно от N-конца), связанные тремя CDR. Три CDR в тяжелой цепи последовательно от N-конца называют CDRH1, CDRH2 и CDRH3, а CDR в легкой цепи называют CDRL1, CDRL2 и CDRL3. Наиболее важным для специфичности связывания антитела с антигеном является CDRH3. CDR каждой цепи удерживаются вместе в непосредственной близости друг от друга областями FR и участвуют в формировании антигенсвязывающего участка антитела вместе с CDR другой цепи. Константная область непосредственно не участвует в связывании антитела с антигеном; однако константная область обладает различными эффекторными функциями, такими как участие в антителозависимой клеточной цитотоксичности (ADCC), фагоцитозе посредством связывания с рецептором Fcγ, скорости полувыведения/клиренса при посредстве неонатального рецептора Fc (FcRn) и обусловленной комплементом цитотоксичности (CDC) при посредстве компонента C1q каскада комплемента.
[0035] <Получение антител>
Антитела к CSPG5 по настоящему изобретению относятся к антителам с иммунологической реакционноспособностью в отношении полноразмерного белка CSPG5 или его фрагмента.
[0036] Термин "иммунологическая реакционноспособность", используемый в настоящем документе, относится к свойству связывания антитела с антигеном CSPG5 in vivo. При таком связывании проявляется повреждающее действие (например, уничтожение, подавление или индукция регрессии) в отношении опухоли. Более конкретно, тип антитела, используемого по настоящему изобретению, не ограничен при условии, что оно может связываться с белком CSPG5 с повреждением опухоли, такой как рак молочной железы, рак легких, опухоль головного мозга, лейкоз, злокачественная лимфома, аденокарцинома, мастоцитома, плоскоклеточная карцинома, меланома или нейробластома.
[0037] Примеры антитела включают моноклональное антитело, поликлональное антитело, генетически рекомбинантное антитело и фрагмент антитела (например, Fab и F(ab')2), как указано выше. Также антитело может представлять собой любой класс молекула иммуноглобулинов, такой как IgG, IgE, IgM, IgA, IgD и IgY или любой их подкласс, такой как IgG1, IgG2, IgG3, IgG4, IgA1 и IgA2.
[0038] Антитело можно дополнительно модифицировать посредством ацетилирования, формилирования, амидирования, фосфорилирования, пегилирования (PEG), или т.п., а также гликозилирования.
[0039] Ниже описаны примеры получения различных антител.
(1) Моноклональные антитела
Примеры моноклональных антител включают моноклональные антитела человека и моноклональные антитела животных (не являющихся человеком) (например, моноклональные антитела мыши, моноклональные антитела крысы, моноклональные антитела кролика и моноклональные антитела курицы).
[0040] В случае моноклональных антител антитела можно получать, проводя иммунизацию общепринятым способом иммунизации с использованием в качестве сенсибилизирующего антигена требуемого антигена (белок CSPG5 в настоящем документе) или клеток, экспрессирующих требуемый антиген, сливая полученные, таким образом, иммуноциты общепринятым способом слияния клеток с исходными клетками, известными в данной области, и подвергая продуцирующие моноклональные антитела клетки (гибридомы) скринингу общепринятым способом скрининга.
[0041] Сначала животное известным в данной области способом иммунизируют сенсибилизирующим антигеном. В качестве общепринятого способа сенсибилизирующий антиген интраперитонеально или подкожно инъецируют млекопитающему, например, мыши. Более конкретно, сенсибилизирующий антиген, т.е. белок CSPG5, разбавляют или суспендируют в подходящем количестве PBS (фосфатно-солевого буфера), физиологического раствора или т.п. и, при желании, в него в соответствующем количестве добавляют общепринятый адъювант, например, полный адъювант Фрейнда, и эмульгируют. Затем эмульсию несколько раз с интервалами от 4 до 21 суток вводят млекопитающему, например, мыши. Вместе с сенсибилизирующим антигеном во время иммунизации можно использовать подходящий носитель. Альтернативно, для иммунизации животного животному (которое необходимо иммунизировать) можно вводить линию клеток лейкоза K562, экспрессирующих ген CSPG5, или т.п.
[0042] После иммунизации млекопитающего как описано выше и подтверждения возрастания уровня требуемых антител в сыворотке, у животного проводят сбор иммуноцитов и подвергают их слиянию клеток для получения гибридом, продуцирующих моноклональные антитела. В качестве предпочтительных иммуноцитов для получения гибридом, конкретно предусмотрены спленоциты.
[0043] В качестве другого партнера для слияния с иммуноцитами используют миеломные клетки млекопитающих. Соответственно, в качестве миеломных клеток используют различные линии клеток, известные в данной области, такие как P3U1 (P3-X63Ag8U1), P3 (P3×63Ag8,653) (J. Immunol. (1979) 123, 1548-1550), P3×63Ag8U.1 (Current Topics in Microbiology and Immunology (1978) 81, 1-7), NS-1 (Kohler. G. и Milstein, C. Eur. J. Immunol. (1976) 6, 511-519), MPC-11 (Margulies. D. H. et al., Cell (1976) 8, 405-415), SP2/0 (Shulman, M. et al., Nature (1978) 276, 269-270), FO (deSt. Groth, S.F. et al., J. Immunol. Methods (1980) 35, 1-21), S194 (Trowbridge, I. S. J. Exp. Med. (1978) 148, 313-323), R210 (Galfre, G. et al., Nature (1979) 277, 131-133).
[0044] Слияние клеток у иммуноцитов и миеломных клеток можно проводить в основном известным в данной области способом, например, способом Kohler and Milstein et al., (Kohler, G. and Milstein, C. Methods Enzymol. (1981) 73, 3-46).
[0045] Более конкретно, слияние клеток проводят в присутствии, например, катализатора слияния клеток, в общепринятом питательном растворе для культивирования. В качестве катализатора слияния используют, например, полиэтиленгликоль (PEG) или вирус Сендай (HVJ) и, при желании, для увеличения эффективности слияния, можно добавлять вспомогательное средство, такое как диметилсульфоксид.
[0046] Отношение иммуноцитов и миеломных клеток для использования можно определять произвольно. Например, иммуноциты относительно миеломных клеток можно использовать в отношении от 1 до 10. В качестве раствора для культивирования, используемого при слиянии клеток, можно использовать, например, раствор для культивирования RPMI 1640 или раствор для культивирования MEM, подходящие для пролиферации линий миеломных клеток, и другие растворы для культивирования, как правило, используемые при культивировании этих клеток. Кроме того, в комбинации с раствором для культивирования можно использовать добавку сыворотки, такой как эмбриональная телячья сыворотка (ЭТС).
[0047] Слияние клеток проводят посредством достаточного смешивания предопределенных количеств иммуноцитов и миеломных клеток в растворе для культивирования и добавления раствора PEG (например, средней молекулярной массы приблизительно от 1000 до 6000), предварительно нагретого приблизительно до 37°C, как правило, в концентрации от 30 до 60% (масс./об.) с последующим перемешиванием с формированием требуемых гибридом. Затем, повторяют действие, состоящее из периодического добавления соответствующего раствора для культивирования, центрифугирования смеси и удаления супернатанта, для удаления средства для слияния клеток, отрицательно влияющего на рост гибридомы, и т.п.
[0048] Получаемые таким образом гибридомы подвергают селекции посредством культивирования в общепринятом растворе для селекции культур, таком как раствор для культивирования HAT (раствор для культивирования, содержащий гипоксантин, аминоптерин и тимидин). Культивирование в растворе для культивирования HAT проводят в течение определенного периода времени (как правило, от нескольких суток до нескольких недель), достаточного для гибели клеток, отличных от гибридомы (неслившихся клеток). Затем, проводят общепринятый способ лимитирующих разведений и скрининг гибридомы, продуцирующих требуемые антитела, и проводят клонирование одиночных клеток.
[0049] Кроме получения указанных выше гибридом посредством иммунизации животных, кроме человека, антигенами, также можно получать гибридомы, продуцирующие антитела человека с требуемой активностью (например, цитостатической активностью) посредством сенсибилизации лимфоцитов человека, таких как лимфоциты человека, инфицированные вирусом EB, белком, экспрессирующими белок клетками или их лизатами in vitro, и слияния сенсибилизированных лимфоцитов с полученными у человека миеломными клетками, например, U266 (регистрационный номер TIB196), с потенциалом длительного деления.
[0050] Полученные таким образом гибридомы, продуцирующие моноклональные антитела, можно субклонировать в общепринятом раствор для культивирования и хранить в жидком азоте в течение длительного времени.
[0051] (2) Поликлональные антитела
В случае поликлональных антител антитела можно получать посредством иммунизации небольшого животного, такого как мышь, продуцирующие антитела человека мышь или кролик, природным белком CSPG5, рекомбинантным белком CSPG5, экспрессированным в форме слитого с GST белка и т.п. в микроорганизме, таком как Escherichia coli, или являющимся его частью пептидом с получением сыворотки; и очистки антител, например, посредством осаждения сульфатом аммония, колонок с белком A, белком G, ионообменной хроматографии с DEAE, аффинной колонки, связанной с белком CSPG5, и синтетического пептида. В примерах, описанных далее, получают поликлональные антитела мыши к внеклеточной области (снаружи злокачественной клетки) аминокислотной последовательности белка CSPG5 и подтверждают наличие у них противоопухолевого действия.
[0052] В качестве продуцирующих антитела человека мышей, используемых по настоящему документу, известны, например, мышь KM (Kirin Pharma/Medarex) и мышь Xeno (Amgen) (например, международные публикации №№ WO 02/43478 и 02/092812). Когда таких мышей иммунизируют белком CSPG5 или его фрагментом, из крови можно получать полные поликлональные антитела человека.
[0053] Антиген можно получать, например, способом с использованием клеток животного (патентная публикация Японии (Kohyo) № 2007-530068A) или способом с использованием бакуловируса (например, международная публикация № WO 98/46777). Когда иммуногенность антигена является низкой, антиген можно связывать с макромолекулой, такой как альбумин, обладающей иммуногенностью, и подвергать иммунизации.
[0054] (3) Рекомбинантные антитела
В случае генетически рекомбинантных антител антитела можно получать в соответствии с технологией генетической рекомбинации, клонируя ген антитела из гибридомы, встраивая ген в соответствующий вектор и вводя вектор хозяину с получением рекомбинантного антитела (см., например, Carl, A. K. Borrebaeck, James, W. Larrick, THERAPEUTIC MONOCLONAL ANTIBODIES, Published in the United Kingdom by MACMILLAN PUBLISHERS LTD, 1990). Более конкретно, с использованием обратной транскриптазы из иРНК гибридомы синтезируют кДНК вариабельной области (V область) антитела. Когда получают ДНК, кодирующую V область требуемого антитела, ее лигируют с ДНК, кодирующей константную область (C область) требуемого антитела и полученную ДНК интегрируют в экспрессирующий вектор. Альтернативно, ДНК, кодирующую V область антитела можно интегрировать в экспрессирующий вектор, содержащий ДНК C области антитела. Указанную ДНК можно встраивать для экспрессии под контролем регулирующей экспрессию области, например, энхансера и промотора. Затем экспрессирующим вектором трансформируют клетки-хозяева и обеспечивают экспрессию генетически рекомбинантного антитела.
[0055] Примеры генетически рекомбинантных антител включают химерное антитело, гуманизированное антитело, одноцепочечное антитело и многоцепочечное антитело, такое как биспецифическое антитело.
[0056] "Химерное антитело" представляет собой антитело, сформированное из последовательностей, получаемых у разных животных, в комбинации, например, антитело, состоящее из вариабельной области тяжелой цепи и легкой цепи антитела мыши и константной область тяжелой цепи и легкой цепи антитела человека. Химерное антитело можно получать известным в данной области способом, например, лигируя ДНК, кодирующую V область антитела, и ДНК кодирующую C область антитела человека, встраивая лигат в экспрессирующий вектор и вводя экспрессирующий вектор хозяину, обеспечивая продукцию антитела хозяином. В качестве примера, ДНК, кодирующую химерное антитело человека/мыши можно получать, лигируя ДНК, кодирующую вариабельную область легкой цепи или тяжелой цепи антитела, получаемую у не являющегося человеком животного (например, мыши) с ДНК, кодирующей константную область легкой цепи или тяжелой цепи антитела, получаемую из антитела человека.
[0057] "Гуманизированное антитело" представляет собой модифицированное антитело, также называемое реконструированное антитело человека. Гуманизированное антитело конструируют, прививая CDR антитела, полученного у иммунизированного животного в определяющую комплементарность область антитела человека. Также в данной области известен способ общей генетической рекомбинации для получения гуманизированных антител. Конкретно, гуманизированное антитело получают, клонируя ДНК, кодирующую моноклональное антитело, с использованием результата в качестве матрицы для получения ДНК, кодирующей вариабельную область легкой цепи и вариабельную область тяжелой цепи способом ОТ-ПЦР или т.п.; определяя последовательности вариабельных областей легкой цепи и тяжелой цепи или последовательности CDR1, CDR2 и CDR3, на основе системы нумерации Kabat EU (Kabat et al., Sequences of proteins of Immunological Interest, 5th Ed. Public Health Service, National Institute of Health, Bethesda, Md. (1991)); затем, способом ПЦР с использованием нескольких олигонуклеотидов, сконструированных так, чтобы содержать на концах перекрывающиеся области, синтезируя последовательность ДНК, сконструированную так, чтобы было можно лигировать CDR антитела мыши и каркасные области (каркасная область; FR) антитела человека; лигируя полученную ДНК в ДНК, кодирующую константную область антитела человека, а затем встраивая ее в экспрессирующий вектор и вводя экспрессирующий вектор хозяину, а затем обеспечивая продукцию гуманизированного антитела хозяином (см., публикацию патента Европы № 239400, международную публикацию № WO 96/02576). FR антитела человека для лигирования CDR выбраны так, чтобы CDR (определяющие комплементарность области) формировали удовлетворительный антигенсвязывающий участок. Если необходимо, аминокислоты каркасной области в вариабельной области антитела можно замещать так, чтобы определяющие комплементарность области антитела человека реконструировали соответствующий антигенсвязывающий участок (Sato K., et al., Cancer Research, 1993, 53: 851-856). Кроме того, каркасные области можно замещать каркасными областями, получаемыми из различных антител человека (международная публикация № WO99/51743).
[0058] Каркасные области антитела человека для лигирования CDR выбраны так, что CDR (определяющие комплементарность области) формируют удовлетворительный антигенсвязывающий участок. Если необходимо, аминокислоты каркасных областей вариабельной области антитела можно замещать так, чтобы определяющие комплементарность области реконструированного антитела человека формировали соответствующий антигенсвязывающий участок (Sato K. et al., Cancer Research 1993, 53: 851-856).
[0059] Аминокислоту в вариабельной области (например, FR) и константной области можно замещать, например, другой аминокислотой после формирования химерного антитела и гуманизированного антитела.
[0060] Замены аминокислот включают замены аминокислот в количестве, например, менее 15, менее 10, 8 или менее, 7 или менее, 6 или менее, 5 или менее, 4 или менее, 3 или менее или 2 или менее, предпочтительно от 1 до 5 аминокислот, а более предпочтительно - 1 или 2 аминокислоты. Антитело с заменой должно быть функционально эквивалентно антителу без замены. Предпочтительно замена представляет собой консервативную замену аминокислоты, которая представляет собой замену аминокислот с аналогичными свойствами в отношении заряда, боковой цепи, полярности и ароматичности, и т.п. Аминокислоты с аналогичными свойствами можно классифицировать, например, на основные аминокислоты (аргинин, лизин, гистидин), кислые аминокислоты (аспарагиновая кислота, глутаминовая кислота), незаряженные полярные аминокислоты (глицин, аспарагин, глутамин, серин, треонин, цистеин, тирозин), неполярные аминокислоты (лейцин, изолейцин, аланин, валин, пролин, фенилаланин, триптофан, метионин), разветвленные аминокислоты (треонин, валин, изолейцин) и ароматические аминокислоты (фенилаланин, тирозин, триптофан, гистидин).
[0061] Модифицированные антитела включают, например, антитела, связанные с молекулами различного типа, такими как полиэтиленгликоль (PEG). В модифицированном антителе по настоящему изобретению вещество для связывания с антителом не ограничено. Такое модифицированное антитело можно получать посредством химической модификации получаемого антитела. Способы химической модификации уже общепризнаны в данной области.
[0062] Термин "функционально эквивалентный", используемый в настоящем документе, означает, что требуемое антитело обладает такой же биологической или биохимической активность, как антитело по настоящему изобретению, более конкретно, означает, что требуемое антитело обуславливает повреждающее опухоль действие, и при применении антитела у человека реакции отторжения по сути не происходит. Такая активность включает, например, цитостатическую активность или связывающую активность.
[0063] Способ внесения мутации в полипептид известен как хорошо известный специалистам в данной области способ получения полипептида, который функционально эквивалентен предопределенному полипептиду. Специалисты в данной области для надлежащего внесения мутации в антитело по настоящему изобретению могут использовать сайт-специфический мутагенез (Hashimoto-Gotoh, T.et al., (1995) Gene, 152,271-275: Zoller, MJ., and Smith, M. (1983) Methods Enzymol.100, 468-500; Kramer, W.et al., (1984) Nucleic Acids Res.12, 9441-9456; Kramer, W. and Fritz, HJ., (1987) Methods Enzymol.154, 350-367, Kunkel, TA., (1985) Proc. Natl. Acad. Sci. USA., 82, 488-492; Kunkel (1988) Methods Enzymol., 85, 2763-2766), таким образом, получая антитело, функционально эквивалентное указанному антителу.
[0064] Антитело, распознающее эпитоп белка CSPG5, который распознает антитело к CSPG5, можно получать способом, известным специалистам в данной области. Например, антитело получают, определяя общепринятым способом (например, картирования эпитопов) эпитоп белка CSPG5, распознаваемый антителом к CSPG5, и поучая антитело с использованием в качестве иммуногена полипептида с аминокислотной последовательностью, содержащейся в эпитопе. В дополнение к этому способу антитело можно получать способом, например, с определением эпитопов антител, получаемых общепринятым способом и отбирая антитела с тем же эпитопом, что и антитело к CSPG5.
[0065] Константа аффинности (константа связывания) Ka (kon/koff) антитела к CSPG5 по настоящему изобретению в отношении белка CSPG5 на поверхности клеток злокачественной опухоли составляет по меньшей мере 107 M-1, по меньшей мере 108 M-1, по меньшей мере 5 × 108 M-1, по меньшей мере 109 M-1, по меньшей мере 5 × 109 M-1, по меньшей мере 1010 M-1, по меньшей мере 5 × 1010 M-1, по меньшей мере 1011 M-1, по меньшей мере 5 × 1011 M-1, по меньшей мере 1012 M-1 или по меньшей мере 1013 M-1. При возрастании аффинности связывания можно получать более сильную противоопухолевую активность. Таким образом, если можно получить антитело к CSPG5 с высокой аффинностью связывания белка CSPG5, можно ожидать более сильного противоопухолевого действия, антитело можно применять для фармацевтической композиции для лечения и/или предотвращения злокачественной опухоли.
[0066] "Одноцепочечное антитело" представляет собой антитело, получаемое посредством последовательного связывания вариабельной области тяжелой цепи и вариабельной области легкой цепи посредством линкера. ДНК, кодирующую одноцепочечное антитело, можно получать посредством лигирования ДНК, кодирующей вариабельную область тяжелой цепи, ДНК, кодирующей линкер, и ДНК, кодирующей вариабельную область легкой цепи. Вариабельная область тяжелой цепи и вариабельная область легкой цепи, используемые в одноцепочечном антителе, представляют собой области, предпочтительно получаемые из антитела человека, или области, получаемые из антитела человека, в котором CDR заменены CDR антитела, получаемого у не являющегося человеком животного (например, мыши, крысы, курицы). Линкер состоит из 12-19 аминокислот и содержит, например, (G4S)3, состоящий из 15 аминокислот (G. B. Kim et al., Protein Engineering Design and Selection, 2007, 20 (9): 425-432).
[0067] В случае "биспецифического антитела (диатела)", которое представляет собой антитело, способное к специфическому связыванию с двумя различными эпитопами, ДНК, кодирующую биспецифическое антитело, можно получать, последовательно связывая, например, ДНК, кодирующую вариабельную область тяжелой цепи A, ДНК, кодирующую вариабельную область легкой цепи B, ДНК, кодирующую вариабельную область тяжелой цепи B, и ДНК, кодирующую вариабельную область легкой цепи A, в указанном порядке (при условии, что ДНК, кодирующая вариабельную область легкой цепи B, и ДНК, кодирующая вариабельную область тяжелой цепи B, связаны посредством ДНК, кодирующей линкер, как указано выше). Каждую из вариабельных областей тяжелых цепей и вариабельных областей легких цепей предпочтительно получают из антитела человека или получают из антитела человека, в котором CDR заменены CDR антитела, получаемого у не являющегося человеком животного (например, мыши, крысы, курицы).
[0068] Рекомбинантное антитело можно получать, интегрируя рекомбинантную ДНК, получаемую как описано выше, в один или несколько подходящих векторов, вводя вектор(ы) в клетку-хозяина (например, клетки млекопитающих, дрожжевые клетки, клетки насекомых) и обеспечивая (ко)экспрессию клеткой-хозяином рекомбинантной ДНК (P. J. Delves., ANTIBODY PRODUCTION ESSENTIAL TECHNIQUES., 1997 WILEY, P. Shepherd and C. Dean., Monoclonal Antibodies., 2000 OXFORD UNIVERSITY PRESS; J. W. Goding., Monoclonal Antibodies: principles and practice., 1993 ACADEMIC PRESS).
[0069] Антитело, как указано выше, предпочтительно обладает цитотоксической активностью и вследствие цитотоксической активности может обеспечивать противоопухолевое действие.
[0070] Антитело по настоящему изобретению можно конъюгировать с другим противоопухолевым средством. Антитело и противоопухолевое средство можно конъюгировать посредством спейсера, содержащего группу, способную к реакции с аминогруппой, карбоксильной группой, гидроксигруппой, тиольной группой и т.п. (примеры реакционноспособных групп включают сукцинимидильную группу, формильную группу, 2-пиридилдитиогруппу, малеимидильную группу, алкоксикарбонильную группу и гидроксигруппу).
[0071] Примеры противоопухолевого средства включают указанные ниже противоопухолевые средства, известные из литературы и т.д., такие как паклитаксел, доксорубицин, даунорубицин, циклофосфамид, метотрексат, 5-фторурацил, тиотепа, бусульфан, импросульфан, пипосульфан, бензодопа, карбоквон, метуредопа, уредопа, алтретамин, триэтиленмеламин, триэтиленфосфорамид, триэтилентиофосфорамид, триметилоломеламин, буллатацин, буллатацинон, камптотецин, бриостатин, каллистатин, криптофицин 1, криптофицин 8, доластатин, дуокармицин, элеутеробин, панкратистатин, саркодиктин, спонгистатин, хлорамбуцил, хлорнафазин, холофосфамид, эстрамустин, ифосфамид, мехлоретамин, мехлоретамин оксид гидрохлорид, мелфалан, новэмбихин, фенестерин, преднимустин, трофосфамид, урамустин, кармустин, хлорзотоцин, фотемустин, ломустин, нимустин, ранимустин, калихимицин, динемицин, клодронат, эсперамицин, аклациномицин, актиномицин, аутрамицин, азасерин, блеомицин, кактиномицин, карабицин, карминомицин, карцинофилин, хромомицин, дактиномицин, деторбицин, 6-диазо-5-оксо-L-норлейцин, адриамицин, эпирубицин, эзолубицин, идарубицин, марцелломицин, митомицин C, микофеноловая кислота, ногаламицин, оливомицин, пепромицин, потфиромицин, пуромицин, квеламицин, родорубицин, стрептонигрин, стрептозоцин, туберцидин, убенимекс, циностатин, зорубицин, деноптерин, птероптерин, триметрексат, флударабин, 6-меркаптопурин, тиамиприн, тиогуанин, анцитабин, азацитидин, 6-азауридин, кармофур, цитарабин, дидезоксиуридин, диоксифлуридин, эноцитабин, флоксуридин; андрогены, такие как калустерон, пропионат дромостанолона, эпитиостанол, мепитиостан, тестолактон, аминоглутетимид, митотан, трилостан, фолиновая кислота, ацеглатон, альдофосфамидгликозид, аминолевулиновая кислота, енилурацил, амсакрин, бестрабуцил, бизантрен, эдатраксат, дефофамин, демеколцин, диазиквон, эльфорнитин, ацетат эллиптиния, эпотилон, этоглюцид, лентинан, лонидамин, майтанзин, ансамитоцин, митогуазон, митоксантрон, мопиданмол, нитраэрин, пентостатин, фенамет, пирарубицин, лозоксантрон, подофиллиновая кислота, 2-этилгидразид, прокарбазин, разоксан, ризоксин, шизофиллан, спирогерманий, тенуазоновая кислота, триазиквон, роридин A, ангвидин, уретан, виндезин, дакарбазин, манномустин, митобронитол, митолактол, пипоброман, гацитозин, доцетаксел, хлорамбуцил, гемцитабин, 6-тиогуанин, меркаптопурин, цисплатин, оксалиплатин, карбоплатин, винбластин, этопозид, ифосфамид, митоксантрон, винкристин, винорелбин, новантрон, тенипозид, эдатрексат, дауномицин, аминоптерин, кселода, ибандронат, иринотекан, ингибитор топоизомеразы, дифторметилорнитин (DMFO), ретиноевая кислота, капецитабин и их фармацевтически приемлемые соли или производные.
[0072] С антителом по настоящему изобретению можно связывать радиоактивный изотоп, известный из литературы и т.д., такой как211At,131I,125I,90Y,186Re,188Re,153Sm,212Bi,32P,175Lu и176Lu. В качестве радиоактивного изотопа предпочтителен радиоактивный изотоп, эффективный для лечения и диагностики опухоли.
[0073] Антитело по настоящему изобретению представляет собой антитело, иммунологически реакционноспособное с белком CSPG5, или антитело, специфически распознающее белок CSPG5. Антитело должно представлять собой антитело с такой структурой, чтобы у подвергаемого обработке животного, которому вводят антиген, не происходила или происходила незначительная реакция отторжения. Примеры таких антител в случае, когда подвергаемое обработке животное представляет собой человек и т.д., включают антитело человека, гуманизированное антитело и химерное антитело (например, химерное антитело человека-мыши).
[0074] Получают гибридому, способную к продукции антитела человека или антитела не являющегося человеком животного (например, антитела мыши) к белку CSPG5 человека. Моноклональное антитело, получаемое посредством гибридомы, восстанавливают и определяют является ли оно требуемым антителом или нет на основе свойств иммунологического связывания с белком CSPG5 человека и цитотоксической активности в качестве индикатора. Таким образом, идентифицируют и отбирают продуцирующую требуемое моноклональное антитело гибридому. Затем, как описано выше, из гибридомы получают ДНК, кодирующую вариабельные области тяжелой цепи и легко цепи требуемого антитела и определяют ее нуклеотидную последовательность. Информацию о нуклеотидной последовательности ДНК используют для получения другого антитела.
[0075] Настоящее изобретение дополнительно относится к ДНК, кодирующей антитело по настоящему изобретению, описанное выше, ДНК, кодирующей тяжелую цепь или легкую цепь антитела, описанного выше, или к ДНК, кодирующей вариабельную область тяжелой цепи или легкой цепи антитела, описанного выше.
[0076] CDR, кодируемые этим молекулами ДНК, представляют собой области, определяющие специфичность антитела. Нуклеотидная последовательность, кодирующая область (более конкретно, константную область и каркасную область), отличающуюся от CDR антитела, может представлять собой нуклеотидную последовательность, происходящую из другого антитела. "Другое антитело", используемое в настоящем документе, может включать антитело, происходящее из организма, отличающегося от человека; однако для снижения побочных эффектов предпочтительно антитело, получаемое у человека. Более конкретно, в указанной выше ДНК, области, кодирующие отдельные каркасные области тяжелой цепи и легкой цепь, и области, кодирующие их отдельные константные области, предпочтительно содержат нуклеотидные последовательности, кодирующие соответствующие аминокислотные последовательности, происходящие из антитела человека.
[0077] ДНК антитела, служащего в качестве активного ингредиента по настоящему изобретению, можно получать, например, указанным выше способом или указанным ниже способом. Сначала, из гибридомы, относящейся к антителу по настоящему изобретению, посредством коммерчески доступного набора для выделения РНК получают тотальную РНК, а затем, синтезируют кДНК с использованием обратной транскриптазы с использованием случайных праймеров и т.д. Затем, кДНК, кодирующую антитело, амплифицируют способом ПЦР с использованием в качестве праймеров олигонуклеотидов из консервативных последовательностей в вариабельных областях гена тяжелой цепи и гена легкой цепь известного антитела мыши. Кодирующую последовательность константной области можно получать амплифицируя известную последовательность способом ПЦР. Нуклеотидную последовательность ДНК можно определять стандартным способом, например, интегрируя ДНК в секвенирующие плазмиду или фаг и т.д.
[0078] Полагают, что противоопухолевое действие антитела к CSPG5, используемого по настоящему изобретению для экспрессирующих белок CSPG5 злокачественных клеток, является результатом действия по механизму антителозависимой клеточной цитотоксичности (ADCC) посредством эффекторных клеток и обусловленной комплементом цитотоксичности (CDC).
[0079] Таким образом, активность антитела к CSPG5, используемого по настоящему изобретению, можно оценивать определяя активность ADCC или активность CDC в отношении злокачественных клеток, экспрессирующих белок CSPG5 in vitro, как конкретно описано в примерах.
[0080] Полагают, что антитело к CSPG5 пригодно для лечения или предотвращения злокачественной опухоли, так как антитело, используемое по настоящему изобретению, связывается с внеклеточной областью белка CSPG5, находящегося на поверхности клеток злокачественной опухоли, и проявляет противоопухолевое действие на основе указанных выше видов активности. Более конкретно, настоящее изобретение относится к фармацевтической композиции, содержащей в качестве активного ингредиента антитело к CSPG5 для лечения и/или предотвращения злокачественной опухоли. Когда антитело к CSPG5 используют для введении в организм человека (лечение антителом), антитело к CSPG5 предпочтительно получают в виде антитела человека или гуманизированного антитела для снижения иммуногенности.
[0081] <Связывание с экспрессирующими антиген клетками>
Способность антитела к связыванию с белком CSPG5 можно определять, например, посредством анализа связывания, такого как ELISA, вестерн-блоттинг, иммунофлуоресценый и проточный цитометрический анализ, как описано в примерах.
[0082] <Иммуногистохимическое окрашивание>
В отношении антитела, распознающего белок CSPG5, его реакционноспособность в отношении белка CSPG5 можно проверять с использованием тканей и их срезов в способе иммуногистохимического окрашивания, хорошо известного специалистам в данной области. Например, ткани, получаемой у пациента при хирургической операции; или ткани, получаемой у животного с гетерологичной тканью, прививаемой посредством введения клеток, экспрессирующих белок CSPG5 от природы или после трансфекции; среза замороженной ткани, фиксированной параформальдегидом или ацетоном; или среза погруженной в парафин ткани, фиксированной параформальдегидом.
[0083] Окрашивание на антитело с реакционноспособностью в отношении белка CSPG5 можно проводить различными способами иммуногистохимического окрашивания. Например, визуализацию можно проводить посредством реакции конъюгированных с пероксидазой хрена антител козы к антителам мыши и конъюгированных с пероксидазой хрена антител козы к антителам кролика.
[0084] <Фармацевтическая композиция>
Мишень фармацевтической композиции для лечения и/или предотвращения злокачественной опухоли по настоящему изобретению конкретно не ограничена при условии, что он представляет собой злокачественную опухоль (клетки) с экспрессией белка CSPG5 на поверхности клеток.
[0085] Термины "опухоль" и "злокачественная опухоль", используемые в настоящем документе, относятся к злокачественным неоплазиям и являются взаимозаменяемыми.
[0086] В настоящем изобретении, являющиеся мишенью злокачественные опухоли представляют собой злокачественные опухоли, экспрессирующие ген CSPG5, предпочтительно, конкретно, злокачественные опухоли, экспрессирующие гены, кодирующие аминокислотные последовательности, представленные SEQ ID NO: 2, 4, 6, 8, 10, 12, 14 и 16, или полипептиды, содержащие последовательности, являющиеся частями аминокислотных последовательностей, состоящих по меньшей мере из 7 последовательных аминокислоты, более предпочтительно, злокачественные опухоли, кроме рака яичника, дополнительно предпочтительно, рак молочной железы, рак легких, опухоль головного мозга, лейкоз, злокачественная лимфома, мастоцитома, меланома или нейробластома, более предпочтительно, лейкоз или злокачественная лимфома.
[0087] Примеры этих конкретных злокачественных опухолей в качестве неограничивающих примеров включают аденокарциному молочной железы, комплексную аденокарциному молочной железы, смешанную злокачественную опухоль молочной железы, протоковую папиллярную аденокарциному, аденокарциному легких, плоскоклеточную карциному, мелкоклеточную злокачественную опухоль, крупноклеточную злокачественную опухоль, глиому, которая представляет собой опухоль нейроэпителиальной ткани, эпендимому, нейроцитому, нейроэктодермальную опухоль плода, невриному, нейрофиброму, менингиому, хронический лимфоцитарный лейкоз, лимфому Ходжкина, лимфому желудочно-кишечного тракта, гастроинтестинальную лимфому, от мелко- до среднеклеточной лимфому, злокачественную опухоль слепой кишки, рак восходящей ободочной кишки, рак нисходящей ободочной кишки, рак поперечной ободочной кишки, рак сигмовидной кишки и рак прямой кишки.
[0088] Животные, лечение которых фармацевтической композицией по настоящему изобретению представляет интерес, являются млекопитающими; например, приматами, комнатными животными, домашними животными и животными для состязаний, особенно предпочтительно, людьми, собаками и кошками.
[0089] Когда антитело по настоящему изобретению используют в качестве фармацевтической композиции, антитело можно формулировать способом, известным специалистам в данной области. Например, его можно использовать парентерально в форме стерильного раствора или суспензии для инъекции, когда антитело смешивают с водой или фармацевтически приемлемой жидкостью. Также предусмотрено, что антитело соответствующим образом смешено с фармацевтически приемлемыми носителем или средой; например, стерильной водой, физиологическим раствором, растительным маслом, эмульгатором, суспендирующим средством, поверхностно-активным веществом, стабилизатором, ароматизатором, эксципиентом, носителем, антисептическим средством и/или связывающим средством, в стандартную дозу, требуемую для общепринятых фармацевтических средств для получения лекарственных средств. Количество активного ингредиента в этих лекарственных средствах определяют так, чтобы можно было соответствующим образом получить дозу в пределах предопределенного диапазона.
[0090] Стерильную композицию для инъекций можно формулировать с использованием такого носителя, как дистиллированная вода для инъекций, стандартным способом получения лекарственных средств.
[0091] Примеры водного раствора для инъекций включают физиологический раствор и изотонические растворы, содержащие глюкозу и другое вспомогательное средство(а); например, D-сорбит, D-маннозу, D-маннит и хлорид натрия, и его можно использовать в комбинации с соответствующим солюбилизатором, таким как спирт, например, этанол, полиспирт, такой как пропиленгликоль и полиэтиленгликоль, и неионное поверхностно-активное вещество, такое как полисорбат80(™) и HCO-60.
[0092] Примеры масляного раствора включают кунжутное масло и соевое масло. В комбинации можно использовать солюбилизатор, такой как бензилбензоат и бензиловый спирт. Кроме того, в комбинации можно смешивать буфер, такой как фосфатный буфер и ацетатно-натриевый буфер, болеутоляющее средство, такое как гидрохлорид прокаина, стабилизатор, такой как бензиловый спирт и фенол, и/или антиоксидант. Как правило, получаемый раствор для инъекций, хранят в подходящих ампулах.
[0093] Примеры способа введения включают пероральное введение или парентеральное введение, предпочтительно парентеральное введение, в частности, инъекцию, назальное введение, транспульмональное введение и трансдермальное введение. Примеры инъекции включают внутривенную инъекцию, внутримышечную инъекцию, интраперитонеальную инъекцию и подкожную инъекцию для системного введения или местного введения.
[0094] Способ введения можно соответствующим образом выбирать в зависимости от возраста, массы, пола и симптомов пациента. В качестве дозы фармацевтической композиции, содержащей антитело или полинуклеотид, кодирующий антитело, можно проводить выбор из диапазона например, от 0,0001 мг до 1000 мг на 1 кг массы тела, или из диапазона, например, от 0,001 до 100000 мг/массу тела пациента. Однако доза не ограничена этими числовыми значениями. Доза и способ введения могут варьировать в зависимости от массы тела, возраста, пола, симптомов пациента и т.п., и их соответствующим образом могут выбирать специалисты в данной области.
Примеры
[0095] Настоящее изобретение более конкретно описано ниже посредством примеров; однако область настоящего изобретения этими примерами не ограничена.
<Пример 1: Идентификация нового антигенного белка злокачественной опухоли способом SEREX>
(1) Получение библиотеки кДНК
Из тестикулярной ткани здоровой собаки способом с кислым гуанидином-фенол-хлороформом выделяли тотальную РНК, а затем выделяли поли-A РНК с использованием набора для очистки иРНК Oligotex-dT30 (производимого Takara Shuzo Co., Ltd.) в соответствии с протоколом, приложенным к набору.
[0096] С использованием иРНК (5 мкг), полученной выше, синтезировали фаговую библиотеку кДНК семенника собаки. Фаговую библиотеку кДНК получали с применением набора синтеза кДНК, набора синтеза ZAP-кДНК или набора клонирования ZAP-кДНК GigapackIII Gold (производимыми Agilent Technologies) в соответствии с протоколом, приложенным к набору. Размер полученной фаговой библиотеки кДНК составлял 7,73 × 105 БОЕ/мл.
[0097] (2) Скрининг библиотеки кДНК с использованием сыворотки
Иммуноскрининг проводили с использованием фаговой библиотеки кДНК семенников собаки, полученной выше. Более конкретно, хозяина Escherichia coli (XL1-Blue MRF') инфицировали фагами с получением 2210 клонов в планшете с NZY агарозой φ 90 × 15 мм и культивирование проводили при 42°C в течение периода от 3 до 4 часов с формированем бляшек. Планшет накрывали нитроцеллюлозной мембраной (Hybond C Extra, производимой GE Healthcare Bio-Scinece), импрегнированной IPTG (изопропил-β-D-тиогалактозид) при 37°C в течение 4 часов для индукции экспрессии белка и белок переносили на мембрану. Затем мембрана забирали, вымачивали в TBS (10 мМ Tris-HCl, 150 мМ NaCl pH7,5), содержащем 0,5% сухое обезжиренное порошковое молоко и перемешивали при 4°C в течение ночи для подавления неспецифических реакций. Этот фильтр подвергали воздействию разведенной в 500 раз сыворотки больной собаки при комнатной температуре в течение периода от 2 до 3 часов.
[0098] В качестве сыворотки больной собаки, указанной выше, использовали сыворотку, получаемую у собаки с раком молочной железы. Сыворотку хранили при -80°C и перед использованием подвергали предварительной обработке. Предварительную обработку проводят следующим способом. Сначала хозяина Escherichia coli (XL1-Blure MRF') инфицировали фагами λ ZAP Express без чужеродного гена и культивировали на среде для планшета NZY при 37°C в течение ночи. Затем в планшет добавляли буфер (0,2 M NaHCO3 pH8,3), содержащий 0,5 M NaCl и оставляли в покое при 4°C на 15 часов. Затем получали супернатант в виде экстракционной жидкости Escherichia coli/фагов. Затем, полученную экстракционную жидкость Escherichia coli/фагов пропускали через колонку с NHS (производимую GE Healthcare Bio-Science), обеспечивая иммобилизацию белков, происходящих из Escherichia coli/фагов на колонке. Сыворотку больной собаки пропускали через колонку с иммобилизованным белком для реакции с удалением из сыворотки антител, адсорбируемых на Escherichia coli и фаги (белок). Фракцию сыворотки, прошедшую через колонку, разбавляли в 500 раз TBS, содержащим 0,5% сухое обезжиренное порошковое молоко и использовали в качестве образца для иммуноскрининга.
[0099] Обработанную таким образом сыворотку и указанный выше слитый белок наносили на мембрану и мембрану четыре раза отмывали TBS-T (0,05% Tween (зарегистрированная торговая марка) 20/TBS), затем оставляли для реакции с IgG козы к IgG собаки (IgG козы к IgG-h собаки, конъюгированные с I HRP; производимые BETHYL Laboratories) в качестве вторичного антитела, которое разбавляли в 5000 раз TBS, содержащим 0,5% сухое обезжиренное порошковое молоко, при комнатной температуре в течение одного часа. Детекцию проводили посредством хромогенной ферментативной реакции с использованием реакционного раствора NBT/BCIP (производимого Roche). Колонии, соответствующие положительным участкам хромогенной реакции накалывали из планшета с агарозой NZY φ 90 × 15 мм и растворяли в 500 мкл буферного раствора SM (100 мМ NaCl, 10 мМ MgClSO4, 50 мМ Tris-HCl, 0,01% желатин, pH7,5). Вторичный и третичный скрининг проводили, повторяя указанный выше способ до унификации положительных в хромогенной реакции колоний. После скрининга 9110 фаговых клонов, реагирующих с IgG в сыворотке выделили один положительный клон.
[0100] (3) Поиск гомологии выделенного гена антигена
Для того, чтобы подвергнуть единственный выделенный положительный клон указанному выше способу анализа нуклеотидной последовательности, провели процесс переноса из фагового вектора в плазмидный вектор. Более конкретно, раствор (200 мкл), содержащий хозяина Escherichia coli (XL1-Blue MRF'), полученный так, чтобы его оптическая плотность составляла OD600 1,0, раствор очищенного фага (100 мкл) и дополнительно 1 мкл фага-помощника ExAssist (производимого Agilent Technologies) смешивали и оставляли для реакции при 37°C в течение 15 минут. Добавляли 3 мл среды LB и проводили культивирование при 37°C в течение периода от 2,5 до 3 часов. Непосредственно после культивирования среду поддерживали нагретой в водяной бане при 70°C в течение 20 минут и центрифугировали при 4°C и 1000×g в течение 15 минут. Супернатант отделяли в виде раствор фагмиды. Затем получали раствор (200 мкл), содержащий хозяина фагмиды Escherichia coli (SOLR), так, чтобы его оптическая плотность составляла OD600 1,0 и очищенный раствор фага (10 мкл) смешивали и проводили реакцию при 37°C в течение 15 минут. Полученный раствор (50 мкл) высевали на содержащий ампициллин (конечная концентрация: 50 мкг/мл) агарозную среду LB и культивировали при 37°C в течение ночи. Одну трансформированную колонию SOLR накалывали, культивировали в содержащей ампициллин (конечная концентрация: 50 мкг/мл) среде LB при 37°C, а затем очищали набором QIAGEN plasmid Miniprep Kit (производимым QIAGEN) с получением плазмидной ДНК, содержащей требуемую вставку.
[0101] Для анализа полноразмерной последовательности вставки очищенную плазмиду подвергали обработке способом "прогулки праймеров" с использованием праймера T3, представленного SEQ ID NO:17, и праймера T7, представленного SEQ ID NO:18. Посредством секвенирования получали последовательность гена, представленную SEQ ID NO:1. С использованием нуклеотидной последовательности и аминокислотной последовательности гена проводили поиск идентичных последовательностей (поиск идентичной последовательности у известных генов) посредством программы поиска идентичных последовательностей, поиска BLAST (http://www.ncbi.nlm.nih.gov/BLAST/). В результате выявлено, что полученный выше ген представляет собой ген CSPG5. У гена CSPG5 человека, который представляет собой фактор человека, гомологичный гену CSPG5 собаки, идентичность нуклеотидной последовательности составляла 87%, и у белка CSPG5 человека идентичность аминокислотных последовательностей составляла 87%. У гена CSPG5 кошки идентичность нуклеотидных последовательностей составляла 92%. у белка CSPG5 кошки идентичность аминокислотных последовательностей составляла 91%. В гомологичном факторе мыши, т.е., гене CSPG5 мыши, идентичность нуклеотидной последовательности составляла 84%. В белке CSPG5 мыши идентичность аминокислотных последовательностей составляла 85%. Нуклеотидные последовательности гена CSPG5 человека представлены SEQ ID NO: 3, 5, 7, 9 и 11. Аминокислотные последовательности белка CSPG5 человека представлены SEQ ID NO:4, 6, 8, 10 и 12. Нуклеотидная последовательность гена CSPG5 кошки представлена SEQ ID NO:13. Аминокислотная последовательность белка CSPG5 кошки представлена SEQ ID NO:14. Нуклеотидная последовательность гена CSPG5 мыши представлена SEQ ID NO:15. Аминокислотная последовательность белка CSPG5 мыши представлена SEQ ID NO:16.
[0102] (4) Анализ экспрессии гена в ткани
Экспрессия гена, полученного описанным выше способом в нормальных тканях и злокачественных тканях собаки, человека и мыши и в линиях злокачественных клеток анализировали способом ОТ-ПЦР (обратная транскрипция-ПЦР). Реакцию обратной транскрипции проводили следующим способом. Сначала выделяли тотальную РНК из конкретных тканей (от 50 до 100 мг) и конкретных линий клеток (от 5 до 10 × 106 клеток) с использованием реагента TRIZOL (производимого Thermo Fisher Scientific) в соответствии с приложенным протоколом. С использованием тотальной РНК синтезировали кДНК с использованием системы для ОТ-ПЦР Superscript First-Strand Synthesis System (производимой Thermo Fisher Scientific) в соответствии с приложенным протоколом. В качестве кДНК нормальных тканей человека (головного мозга, гиппокампа, семенников, кишечника, плаценты) использовали геномную кДНК (производимую Thermo Fisher Scientific), кДНК QUICK-Clone (производимую Clontech Laboratories, Inc.) и библиотеку кДНК Large-Insert cDNA Library (производимую Clontech Laboratories, Inc.). Реакцию ПЦР проводили с использованием полученных геноспецифических праймеров (праймеров собаки, представленных SEQ ID NO: 19 и 20, праймеров человека, представленных SEQ ID NO: 21 и 22, праймеров мыши, представленных SEQ ID NO: 23 и 24) как указано далее. Сначала реагенты и прилагаемый буфер добавляли в 0,25 мкл образца, полученного посредством реакции обратной транскрипции, с получением смеси, с общим количеством 25 мкл, содержащей указанные праймеры (2 мкМ каждого), dNTP (0,2 мМ каждого) и 0,65 Ед полимеразы ExTaq (производимой Takara Shuzo Co., Ltd.). Реакционную смесь подвергали обработке в термоциклере (производимом BIO RAD), в котором цикл состоял из реакции при 94°C в течение 30 секунд, реакции при 55°C в течение 30 секунд и реакции при 72°C в течение одной минута, повторенных 30 раз. В целях сравнения одновременно использовали специфичные к GAPDH праймеры (праймеры GAPDH собаки и человека представлены SEQ ID NO: 25 и 26, праймеры GAPDH мышь представлены SEQ ID NO: 27 и 28). В результате, как представлено на фигуре 1, выявлено, что ген CSPG5 собаки не экспрессирован почти ни в одной нормальной ткани собаки, но сильно экспрессирован в опухолевой ткани собаки. Подобно гену CSPG5 собаки, экспрессия генов CSPG5 человека и мыши в нормальных тканях человека и мыши подтверждалась редко; однако их экспрессию детектировали в злокачественных клетках, например, клетках рака молочной железы, рака легких, опухоли головного мозга, рака яичника, лейкоза, злокачественной лимфомы (фигуры 2 и 3).
[0103] <Пример 2: получение белка CSPG5 человека>
(1) Клонирование полноразмерной кДНК, кодирующей CSPG5 человека и кДНК, кодирующей внеклеточную область CSPG5 человека
Полноразмерную кДНК, кодирующую ген CSPG5 человека, получали клонированием приводимым ниже способом на основе гена, представленного SEQ ID NO:7, полученного в примере 1. ПЦР проводили следующим образом: реагенты и прилагаемый буфер смешивали с получением общего количества 50 мкл смеси, содержащей молекулу кДНК (1 мкл), которая представляла собой одну из молекул, полученных из различных тканей и клеток (полученных в примере 1) и экспрессию которой подтверждали способом ОТ-ПЦР, два типа праймеров (0,4 мкМ каждого) с последовательностями расщепления рестрикционных ферментов KpnI и EcoRI (представленные SEQ ID NO: 29 и 30), 0,2 мМ dNTP и 1,25 Ед полимеразы PrimeSTAR HS (производимой Takara Shuzo Co., Ltd.); и полученный раствор подвергали обработке в термоциклере (производимом BIO RAD), в котором цикл (ПЦР) состоял из реакции при 98°C в течение 10 секунд и реакции при 68°C в течение 2,5 минут, повторенных 30 раз. В данном случае указанные выше два типа праймеров использовали для амплификации области, кодирующей полноразмерную аминокислотную последовательность, представленную SEQ ID NO:7. После ПЦР амплифицированную ДНК подвергали электрофорезу на 1% агарозном геле и выделяли фрагмент ДНК приблизительно 1,7 т.п.н. с использованием набора для выделения из геля QIAquick Gel Extraction Kit (производимого QIAGEN). Продукт амплификации, полученный в указанной выше реакции ПЦР, вставляли в pcDNA3.1 (Thermo Fisher Scientific) (далее в настоящем документе, обозначаемый как CSPG5/pcDNA3.1), и проводили подтверждение наличия последовательности кДНК, кодирующей ген CSPG5 человека, посредством секвенирования с использованием ДНК-секвенатора. Последовательность, представленная SEQ ID NO:7, представляет нуклеотидную последовательность гена CSPG5 человека, а последовательность, представленная SEQ ID NO:8, представляет аминокислотную последовательность белка CSPG5 человека.
[0104] На основе SEQ ID NO:7 проводили реакцию ПЦР, как указано далее. Реагенты и прилагаемый буфер смешивали с получением общего количества 50 мкл смеси, содержащей два типа праймеров (0,4 мкМ каждого) (представленных SEQ ID NO: 29 и 30), содержащих последовательности расщепления рестрикционных ферментов KpnI и EcoRI, 0,2 мМ dNTP и 1,25 Ед полимеразы PrimeSTAR HS (производимой Takara Shuzo Co., Ltd.), и полученный раствор подвергали обработке в термоциклере (производимом BIO RAD), в котором цикл состоял из реакции при 98°C в течение 10 секунд и реакции при 68°C в течение 2,5 минут, повторенных 30 раз. В данном случае указанные два типа праймеров использовали для амплификации области, кодирующей аминокислотную последовательность внеклеточной области белка CSPG5, представленного SEQ ID NO:7. После ПЦР амплифицированную ДНК подвергали электрофорезу на 1% агарозном геле и выделяли фрагмент ДНК приблизительно 1,3 т.п.н. с использованием набора для выделения из геля QIAquick Gel Extraction Kit (производимого QIAGEN). Продукт амплификации, полученный в указанной выше реакции ПЦР, лигировали с pcDNA3.1, в которую вставлена кДНК, кодирующая белок Fc IgG2a мыши, с получением экспрессирующего вектора (далее в настоящем документе, обозначаемого как pcDNA-hCSPG5 ECD-IgG2aFc), кодирующего слитый белок внеклеточной области CSPG5 человека/Fc IgG2a мыши (далее в настоящем документе, обозначаемый как hCSPG5 ECD-mIgG2aFc), и проводили подтверждение наличия последовательности кДНК, кодирующей hCSPG5 ECD-mIgG2aFc, посредством секвенирования с использованием ДНК-секвенатора. Последовательность, представленная SEQ ID NO:32, представляет нуклеотидную последовательность, кодирующую hCSPG5 ECD-mIgG2aFc, а последовательность, представленная SEQ ID NO:33, представляет аминокислотную последовательность hCSPG5 ECD-mIgG2aFc.
[0105] (2) Получение hCSPG5 ECD-mIgG2aFc
Для получения антител к белку CSPG5 получали hCSPG5 ECD-mIgG2aFc в качестве иммунизирующего антигена.
[0106] Экспрессирующий вектор pcDNA-hCSPG5 ECD-mIgG2aFc способом липофекции вводили в линию клеток эмбриональной почки человека, клеток HEK293. Через 7 суток после введения из супернатанта культуры выделяли hCSPG5 ECD-mIgG2aFc. Супернатант культуры наносили на колонку Hi Trap proteinG HP (GE Healthcare Bioscience), промывали связывающим буфером (20 мМ фосфат натрия (pH 7,0)) и элюировали элюирующим буфером (0,1 M глицин-HCl (pH 2,7)). Элюированную жидкость помещали в пробирку, содержащую нейтрализующий буфер (1 M Tris-HCl (pH 9,0)) и сразу же нейтрализовали. Затем элюированную жидкость, полученную описанным выше способом, подвергали ультрафильтрации с использованием NANOSEP 10K OMEGA (производимым PALL) и замещению физиологическим раствором фосфатного буфера (производимым NISSUI PHARMACEUTICAL CO., LTD.), а затем асептически фильтровали посредством HT Tuffryn Acrodisc 0,22 мкм (производимой PALL) и использовали в описанных далее экспериментах.
[0107] <Пример 3: Получение поликлональных антител, связывающихся с внеклеточной областью CSPG5>
(1) Получение поликлональных антител к CSPG5
Для получения антител, связывающихся с внеклеточной областью CSPG5 смешивали 0,1 мг hCSPG5 ECD-mIgG2aFc, полученного, как описано выше, в качестве антигена с эквивалентным количеством раствора полного адъюванта Фрейнда (CFA) и полученную смесь подкожно вводили мыши 4 раза каждые две недели. Затем, получали кровь с получением антисыворотки, содержащей поликлональные антитела. Антисыворотку очищали посредством носителя белка G (производимого GE Healthcare Bioscience) с получением поликлональных антител к hCSPG5 ECD-mIgG2aFc. Сыворотку мыши, которой не вводили антиген, выделяли с использованием носителя белка G таким же образом, как описано выше, и использовали в качестве контрольных антител.
[0108] (2) Получение клеток, константно экспрессирующих полноразмерный CSPG5 человека
CSPG5/pcDNA3.1, полученный, как описано выше, вводили в клетки CHO-K1 (ATCC) способом липофекции. Проводили скрининг посредством 500 мкг/мл G418 (Nacalai) с получением линии клеток CHO, константно экспрессирующих полноразмерный CSPG5 человека (CHO-CSPG5). Экспрессирующий вектор без вставленной в него кДНК, кодирующей CSPG5 (далее в настоящем документе, обозначаемый как emp/pcDNA3.1), вводили и подвергали скринингу таким же способом, как указано выше, с получением клеток, используемых в качестве контрольных клеток (далее в настоящем документе, обозначаемых как CHO-emp).
[0109] Подобным образом способом липофекции CSPG5/pcDNA3.1 вводили в линию клеток лейкоза мыши EL4 (ATCC). Скрининг проводили посредством 500 мкг/мл G418 (Nacalai) с получением линии клеток EL4, константно экспрессирующих полноразмерный ген CSPG5 человека (EL4-CSPG5). Экспрессирующий вектор без вставленной в него кДНК, кодирующей CSPG5 (далее в настоящем документе, обозначаемый как emp/pcDNA3.1), вводили и подвергали скринингу таким же способом, как указано выше, с получением клеток, используемых в качестве контрольных клеток (далее в настоящем документе, обозначаемых как EL4-emp).
[0110] (3) Анализ экспрессии антигенного белка на клеточной поверхности
Проводили исследование специфичности реакции поликлонального антитела, полученного на этапе (1), с белком CSPG5, экспрессируемом на поверхности клеток, полученных на этапе (2). Клетки CHO-CSPG5 и клетки CHO-emp (106 клеток для каждого случая) раздельно вносили в микроцентрифужные пробирки объемом 1,5 мл и центрифугировали. В каждую из пробирок добавляли поликлональные антитела (2 мкг (5 мкл)) к белку CSPG5, полученные на описанном выше этапе (1). Смесь дополнительно суспендировали в PBS (95 мкл), содержащем 0,1% эмбриональную телячью сыворотку и оставляли на льду в течение одного часа. После промывки PBS смесь суспендировали с 5 мкл меченных FITC антител козы к IgG мыши (производимых Santacruz) и 95 мкл PBS, содержащего 0,1% эмбриональную телячью сыворотку (FBS) и оставляли на льду в течение одного часа. После промывки PBS измеряли интенсивность флуоресценции посредством FACS Calibur (производимого BD). С другой стороны вместо поликлональных антител к белку CSPG5 тем же способом, как описано выше, обрабатывали контрольные антитела, полученные на описанном выше этапе (1), и использовали в качестве контроля. В результате, клетки CHO-CSPG5, к которым добавляли антитело к CSPG5 человека, демонстрировали возрастание интенсивности флуоресценции приблизительно на 221% по сравнению с контролем. Тот же способ использовали для клеток CHO-emp. В результате, клетки CHO-emp, в которые добавляли антитела к CSPG5 человека, демонстрировали такую же интенсивность флуоресценции, что и контроль. На основе этих результатов продемонстрировано, что антитела к CSPG5 человека специфически связываются с белком CSPG5, экспрессируемым на поверхности клеточной мембраны.
[0111] Степень увеличения интенсивности флуоресценции выражали посредством степени увеличения средней интенсивности флуоресценции (значение MFI) каждой клетки и рассчитывали по следующей формуле:
Степень увеличения средней интенсивности флуоресценции (степень увеличения интенсивности флуоресценции) (%)=((значение MFI клеток, с которыми реагировали антитела к CSPG5 человека)-(контрольное значение MFI)) ÷(контрольное значение MFI) × 100
[0112] Затем проводили проверку экспрессии белка CSPG5 на клеточной поверхности в отношении двух типов линий клеток лейкоза (K562, THP-1) и двух типов линий клеток злокачественной лимфомы (L-1236, P3HR-1), в которых подтверждена высокая экспрессия гена CSPG5. Индивидуальные линии клеток человека (106 клетки), в которых ранее подтверждена экспрессия гена раздельно помещали в микроцентрифужные пробирки объемом 1,5 мл и центрифугировали. В каждую из пробирок добавляли поликлональные антитела (2 мкг (5 мкл)) к белку CSPG5, полученные на описанном выше этапе (1). Смесь дополнительно суспендировали с PBS (95 мкл), содержащим 0,1% эмбриональную телячью сыворотку и оставляли на льду в течение одного часа. После промывки PBS смесь суспендировали с 5 мкл меченных FITC антител козы к IgG мыши (производимых Santacruz) и 95 мкл PBS, содержащего 0,1% эмбриональную телячью сыворотку (FBS) и оставляли на льду в течение одного часа. После промывки PBS измеряли интенсивность флуоресценции посредством FACS Calibur (производимого BD). С другой стороны, вместо поликлональных антител к белку CSPG5 тем же способом, как описано выше, обрабатывали контрольные антитела, полученные на описанном выше этапе (1), и использовали в качестве контроля. В результате, клетки, к которым добавляли антитело к CSPG5 человека, демонстрировали усиление интенсивности флуоресценции на 30% или более по сравнению с контролем. Более конкретно, K562 демонстрировали возрастание интенсивности флуоресценции на 184%, THP-1 возрастание на 51%, L-1236 возрастание на 115% и P3HR-1 возрастание на 82%. На основе этих результатов подтверждено, что белок CSPG5 экспрессирован на поверхности мембраны клеток линий злокачественных клеток человека.
[0113] <Пример 4: Противоопухолевое действие (активность ADCC) поликлональных антител к белку CSPG5 на злокачественных клетках>
Затем проводили проверку способности поликлональных антител к белку CSPG5 к повреждению опухолевых клеток, экспрессирующих белок CSPG5. Оценку проводили с использованием поликлональных антител к CSPG5 человека, полученных в примере 3. Линию клеток лейкоза человека K562 и линию клеток злокачественной лимфомы L-1236 (106 клеток в каждом случае), на которых подтверждена экспрессия белка CSPG5, раздельно помещали в центрифужную пробирку объемом 50 мл. В пробирку добавляли 100 мкКи хрома 51 и пробирку инкубировали при 37°C в течение двух часов. Затем клетки три раза промывали средой RPMI 1640, содержащей 10% эмбриональную телячью сыворотку и добавляли в лунки 96-луночного (с V-образным дном) планшета в соотношении 103 клеток на лунку. К ним добавляли описанные выше поликлональные антитела к белку CSPG5 человека в отношении 1 мкг на лунку и дополнительно добавляли лимфоциты, выделенные из периферической крови кролика, в отношении 2×105 клеток на лунку. Планшет культивировали при 37°C в условиях 5% CO2 в течение 4 часов. После культивирования в супернатанте культуры определяли количество хрома (Cr) 51, высвободившегося из поврежденных опухолевых клеток, и для каждой из линий злокачественных клеток рассчитывали активность ADCC поликлональных антител к белку CSPG5 человека. В результате подтверждали, что активность ADCC в клетках K562 и клетках L-1236 составляла 23,2% и 18,7%, соответственно (см., фигура 4). С другой стороны, при проведении того же способа с использованием контрольных антител (пример 3), полученных из периферической крови мыши, не иммунизированной антигеном, и с использованием образца, к которому не добавляли антител, активность фактически не была подтверждена (см., фигуру 4). Таким образом, было наглядно продемонстрировано, что антитела к белку CSPG5 могут повреждать опухолевые клетки, экспрессирующие белок CSPG5, на основе активности ADCC.
[0114] Цитотоксическую активность определяли, смешивая антитела к белку CSPG5, используемые по настоящему изобретению, лимфоциты кролика и линии клеток (103 клеток), в которые вводили хром 51, культивируя смесь в течение 4 часов, определяя количество хрома 51, высвободившегося в среду после культивирования и рассчитывая цитотоксическую активность в отношении каждой из линий клеток лейкоза по следующей формуле*.
*Формула: Цитотоксическая активность (%) = (количество хрома 51, высвободившегося из K562 и L-1236, при добавлении антител к белку CSPG5 и лимфоцитов кролика) ÷ (количество хрома 51, высвободившегося из клеток-мишеней в которые добавляли 1Н соляную кислоту) × 100.
[0115] <Пример 5: Получение моноклональных антител к белку CSPG5>
Антигенный белок (hCSPG5 ECD-mIgG2aFc) (100 мкг), представленный SEQ ID NO:33 и полученный в примере 2, смешивали с эквивалентным количеством адъюванта MPL+TDM (производимого Sigma). Смесь использовали в качестве раствора антигена для мышей. Раствор антигена интраперитонеально вводили мышам Balb/c (выращиваемым Japan SLC, Inc.) в возрасте 6 недель и дополнительно вводили 4 раза каждую неделю для завершения иммунизации. Через трое суток после последней иммунизации вырезали селезенку и растирали, помещая каждую селезенку между двумя стерилизованными предметными стеклами, промывали PBS (-) (производимым Nissui) и центрифугировали при 1500 об./мин. в течение 10 минут, а затем удаляли супернатант. Эту операцию повторяли три раза с получением клеток селезенки. Полученные клетки селезенки и миеломные клетки мыши SP2/0 (приобретаемые в ATCC) смешивали в соотношении 5:1. К смеси добавляли раствор PEG, полученный посредством смешивания среды RPMI 1640 (200 мкл), содержащей 10% FBS и нагреваемой до 37°C, и 800 мкл PEG1500 (производимого Boehringer), нагретого до 37°C. Смесь оставляли в покое в течение 5 минут для прохождения слияния клеток. Смесь центрифугировали при 1700 об./мин. в течение 5 минут. После удаления супернатанта клетки суспендировали со 150 мл среды RPMI 1640 (селективная среда HAT), содержащей 15% FBS и раствор HAT, производимый Gibco в эквиваленте 2% и высевали в пятнадцать 96-луночных планшетов (производимых NUNC) в количестве 100 мкл на лунку. Клетки культивировали в течение 7 суток при 37°C в условиях 5% CO2 с получением гибридом, т.е., клеток, слитых из клеток селезенки и миеломных клеток.
[0116] Гибридомы подвергали скринингу на основе аффинности связывания антитела, продуцируемого полученной гибридомой, к hCSPG5 ECD-mIgG2aFc. 1 мкг/мл раствора белка hCSPG5 ECD-mIgG2aFc, полученного в примере 2, добавляли в 96-луночный планшет в количестве 100 мкл на лунку и оставляли в покое при 4°C в течение 18 часов. После трехкратного отмывания отдельных лунок PBS-T добавляли 0,5% раствор бычьего сывороточного альбумина (BSA) (производимого Sigma) в количестве 400 мкл на лунку и оставляли в покое при комнатной температуре в течение 3 часов. Раствор удаляли и лунки отмывали три раза 400 мкл PBS-T на лунку. Затем добавляли супернатант каждой культуры гибридомы, полученной выше, в количестве 100 мкл на лунку и оставляли в покое при комнатной температуре в течение 2 часов. После трехкратного отмывания отдельных лунок PBS-T добавляли меченное HRP антитело к IgG мыши (H+L) (производимое Invitrogen), разбавленное в 5000 раз PBS в количестве 100 мкл на лунку и оставляли в покое при комнатной температуре в течение одного часа. После трехкратного отмывания отдельных лунок PBS-T добавляли раствор субстрата TMB (производимого Thermo) в количестве 100 мкл на лунку и оставляли в покое в течение периода от 15 до 30 минут для прохождения хромогенной реакции. После развития окраски добавляли 1Н серную кислоту в количестве 100 мкл на лунку для завершения реакции. Значения оптической плотности при 450 нм и 595 нм определяли посредством абсорбционного спектрометра. В результате скрининг проходили гибридомы, продуцирующие антитела, демонстрирующие высокие значения оптической плотности.
[0117] Прошедшие скрининг гибридомы добавляли в 96-луночн планшет в отношении 0,5 клеток на лунку и культивировали. Через одну неделю наблюдали гибридомы, формирующие одиночные колонии в лунках. Клетки в этих лунках дополнительно культивировали. Гибридому подвергали скринингу на основе аффинности связывания антитела, продуцируемого клонированной гибридомой, к белку CSPG5. 1 мкг/мл раствора белка hCSPG5 ECD-mIgG2aFc, полученного в примере 2, добавляли в 96-луночный планшет в количестве 100 мкл на лунку и оставляли в покое при 4°C в течение 18 часов. После трехкратного отмывания отдельных лунок PBS-T добавляли 0,5% раствор BSA в количестве 400 мкл на лунку и оставляли в покое при комнатной температуре в течение 3 часов. Раствор удаляли и лунки три раза отмывали PBS-T в количестве 400 мкл на лунку. Каждый из супернатантов культур гибридом, полученных выше, добавляли в количестве 100 мкл на лунку и оставляли в покое при комнатной температуре в течение 2 часов. После трехкратного отмывания отдельных лунок PBS-T добавляли меченное HRP антитело к IgG мыши (H+L) (производимое Thermo Fisher Scientific), разбавленное в 5000 раз PBS в количестве 100 мкл на лунку и оставляли в покое при комнатной температуре в течение одного часа. После трехкратного отмывания отдельных лунок PBS-T добавляли раствор субстрата TMB (производимого Thermo) в количестве 100 мкл на лунку и оставляли в покое в течение периода от 15 до 30 минут для прохождения хромогенной реакции. После развития окраски добавляли 1Н серную кислоту в количестве 100 мкл на лунку для завершения реакции. Значения оптической плотности при 450 нм и 595 нм определяли посредством абсорбционного спектрометра. В результате получали 312 линий гибридомных клеток, продуцирующих моноклональные антитела, способные к реакции с белком CSPG5.
[0118] Затем в моноклональных антителах проводили скрининг моноклональных антител способных к реакции с поверхностью клеток, экспрессирующих белок CSPG5. Конкретно, 106 клеток (CHO-CSPG5) экспрессирующих белок CSPG5 и полученных в примере 2, помещали в микроцентрифужную пробирку объемом 1,5 мл и центрифугировали. К ним добавляли каждого из супернатантов культур гибридом, полученных выше (100 мкл) и оставляли на льду в течение одного часа. После промывки PBS добавляли меченные FITC антитела козы к IgG мыши (производимые Thermo Fisher Scientific), разбавленные в 500 раз PBS, содержащие 0,1% FBS, и смесь оставляли на льду в течение одного часа. После промывки PBS измеряли интенсивность флуоресценции посредством FACS Calibur (производимого BD). Тем же способом, что и описанный выше, обрабатывали клетки CHO (CHO-emp) не экспрессирующие белок CSPG5, и использовали в качестве контроля. В результате скрининг проходили моноклональные антитела, интенсивности флуоресценция которых превышали контроль, другими словами 18 моноклональных антител (№№ 1-18), реагирующих с поверхностью клеток, экспрессирующих белок CSPG5.
[0119] <Пример 6: Характеристики прошедших скрининг антител>
(1) Противоопухолевое действие (активность ADCC) моноклональных антитела к белку CSPG5 на злокачественных клетках
Оценивали цитотоксическую активность (активность ADCC) моноклонального антитела № 1 к белку CSPG5, прошедшего описанный выше скрининг, в отношении злокачественных клеток. Гибридому, продуцирующую моноклональное антитело, культивировали с использованием среды для гибридом SFM (производимой Thermo Fisher Scientific). Полученный супернатант очищали с использованием Hitrap proteinA SepharoseFF (производимой GE Healthcare), проводили замещение PBS (-) и фильтровали через 0,22 мкм фильтр (производимый Millipore), и полученный продукт использовали в качестве антитела для определения активности. Линию клеток лейкоза человека K562 и линию клеток злокачественной лимфомы L-1236 (106 клеток в каждом случае) собирали раздельно в центрифужные пробирки объемом 50 мл и добавляли 100 мкКи хрома 51 и инкубировали при 37°C в течение 2 часов. Затем клетки отмывали три раза средой RPMI 1640, содержащей 10% FBS и добавляли в 96-луночный (с V-образным дном) планшет в отношении 103 клеток на лунку и использовали в качестве клеток-мишеней. К ним добавляли выделенное выше антитело в количестве 1 мкг на клетку и добавляли лимфоциты мышь (2 × 105 клеток), выделенные из селезенки мыши, и культивировали при 37°C в условиях 5% CO2 в течение 4 часов. После культивирования определяли количество хрома 51, высвободившегося из поврежденных опухолевых клеток в супернатант культуры, и рассчитывали активность ADCC моноклонального антитело к CSPG5 в отношении злокачественных клеток.
[0120] (2) Противоопухолевое действие (активность CDC) моноклональных антител к белку CSPG5 на злокачественных клетках
Проводили оценку цитотоксической активности (активность CDC) моноклонального антитела № 1 к белку CSPG5, прошедшего скрининг, как описано выше, в отношении злокачественных клеток. Получали кровь кролика, помещали в пробирку Eppendorf, оставляли в покое при комнатной температуре в течение 60 минут и центрифугировали при 3000 об./мин. в течение 5 минут с получением сыворотки для определения активности CDC. Линию клеток лейкоза человека K562 и линию клеток злокачественной лимфомы L-1236 (105 клеток в каждом случае) собирали раздельно в центрифужные пробирки объемом 50 мл и добавляли 100 мкКи хрома 51 и инкубировали при 37°C в течение 2 часов, промывали три раза средой RPMI, содержащей 10% FBS, суспендировали средой RPMI, содержащей 50% сыворотку кролика, описанную, как описано выше, и добавляли в 96-луночный (с V-образным дном) планшет в отношении 103 клеток на лунку. К ним отдельно добавляли моноклональное антитело № 1, используемое на описанном выше этапе (1) в количестве 1 мкг и культивировали при 37°C в условиях 5% CO2 в течение 4 часов. После культивирования определяли количество хрома 51, высвободившегося из поврежденных опухолевых клеток в супернатант культуры, и рассчитывали активность CDC моноклонального антитела к CSPG5 в супернатанте гибридомы в отношении K562 и L-1236. В результате моноклональное антитело № 1 обладало активностью CDC 26%. Тем же способом обрабатывали моноклональное антитело, полученное в примере 5 и реагирующее с самим белком CSPG5, но не реагирующее с поверхностью злокачественных клеток. В результате цитотоксическую активность не наблюдали. Таким образом, продемонстрировано, что моноклональное антитело (№ 1) к белку CSPG5 также повреждает опухолевые клетки, экспрессирующие белок CSPG5, на основе активности CDC.
[0121] <Пример 7: противоопухолевое действие моноклональных антител к CSPG5 у мышей in vivo>
Проводили оценку противоопухолевого действие моноклонального антитела № 1 (полученного выше) к белку CSPG5 у несущих злокачественную опухоль мышей in vivo. Антитело, используемое в настоящем документе, получали, очищая супернатант каждой культуры гибридомы посредством колонки тем же способом, как описано выше.
[0122] Противоопухолевое действие моноклонального антитела № 1 к белку CSPG5 анализировали с использованием несущих злокачественную опухоль мышей, получаемых посредством трансплантации полученной у мышей линии клеток лейкоза EL4-CSPG5, которые экспрессируют белок CSPG5 и получены в примере 3-(2). В подкожную часть спины каждой из тридцати мышей C57BL/6 (выращиваемых Japan SLC, Inc.) трансплантировали клетки EL4-CSPG5 (106 клеток/мышь) и мышам позволяли расти до достижения опухолью размера приблизительно 7 мм в диаметре. Десяти из этих несущих злокачественную опухоль мышей интраперитонеально вводили моноклональное антитело № 1 к белку CSPG5 в дозе 100 мкг (100 мкл) на мышь. Другим десяти мышам интраперитонеально вводили моноклональное антитело, которое получали в примере 5 и которое реагирует с самим белком CSPG5, но не реагирует с поверхностью злокачественных клеток, в дозе 100 мкг (100 мкл) на мышь. Затем каждое антитело в той же дозе интраперитонеально вводили индивидуальным несущим злокачественную опухоль мышам один раз каждые трое суток всего три раза. Каждые сутки для наблюдения за противоопухолевым действием определяли размеры опухолей. Оставшимся десяти несущим злокачественную опухоль мышам вместо антитела вводили PBS (-), и их использовали в качестве контрольной группы. В результат наблюдения противоопухолевого действия в группе, которой вводили моноклональное антитело (№ 1) к белку CSPG5, объем опухоли снизился приблизительно на 90% на сутки 10, и приблизительно на 70% на сутки 20, и приблизительно на 60 и какое-то количество% на сутки 30 от объема опухоли на исходные сутки введения антитела, взятого за 100%. В отличие от этого в контрольной группе объем опухоли увеличился приблизительно на 260%, 350% и 550% на сутки 10, сутки 20 и сутки 30, соответственно. В группе, которой вводили моноклональное антитело, которое реагирует с самим белком CSPG5 и не реагирует с поверхностью злокачественных клеток, противоопухолевого действия не наблюдали и объем опухоли увеличивался таким же образом, как в контрольной группе. На основе результатов продемонстрировано, что моноклональное антитело (№ 1) к белку CSPG5 оказывает сильное противоопухолевое действие на злокачественные клетки лейкоза, экспрессирующие белок CSPG5 in vivo. Размер (объем) опухоли рассчитывали по формуле:
Большая ось × малая ось × малая ось × 0,5.
Применимость в промышленности
[0123] Антитело по настоящему изобретению пригодно для лечения и/или предотвращения злокачественной опухоли.
[0124] Все публикации, патенты и патентные заявки, цитируемые в описании, включены в описание полностью в качестве ссылки.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> TORAY INDUSTRIES, INC.
<120> ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ ДЛЯ ЛЕЧЕНИЯ И/ИЛИ ПРЕДОТВРАЩЕНИЯ ЗЛОКАЧЕСТВЕННОЙ ОПУХОЛИ
<130> PH-6535-PCT
<150> JP 2015-093640
<151> 2015-04-30
<160> 33
<170> PatentIn version 3.1
<210> 1
<211> 2270
<212> ДНК
<213> Canis familiaris
<220>
<221> Кодирующая последовательность
<222> (1)..(1434)
<223>
<400> 1
ggg gag gag gag acc tcg tgt act gca cct ggc ggc ctg ccg gcc gtg 48
Gly Glu Glu Glu Thr Ser Cys Thr Ala Pro Gly Gly Leu Pro Ala Val
1 5 10 15
gtg ggg cct ggg gtc ggg cca gag gag gcg ctg gag gcg tcc gcg gcc 96
Val Gly Pro Gly Val Gly Pro Glu Glu Ala Leu Glu Ala Ser Ala Ala
20 25 30
gtg acc ggc aca gcc tgg ctg gag gct gac agc ccg ggc ctg ggc gga 144
Val Thr Gly Thr Ala Trp Leu Glu Ala Asp Ser Pro Gly Leu Gly Gly
35 40 45
gcg acc gta gag gct ggc agc ggc gac acc cag gcc ctt ccg gcc acg 192
Ala Thr Val Glu Ala Gly Ser Gly Asp Thr Gln Ala Leu Pro Ala Thr
50 55 60
ctc ccg act ccg gag gag gcc ctc cga cgt gca tcg gtg gcc ccc gcc 240
Leu Pro Thr Pro Glu Glu Ala Leu Arg Arg Ala Ser Val Ala Pro Ala
65 70 75 80
acc ccc gag act aca gag gcc agc gga cca ccc tcc ccc act cct ggc 288
Thr Pro Glu Thr Thr Glu Ala Ser Gly Pro Pro Ser Pro Thr Pro Gly
85 90 95
gac cag cta cgc cca ggc ccc gaa ctc ccc aag gag agc ccc ttg gag 336
Asp Gln Leu Arg Pro Gly Pro Glu Leu Pro Lys Glu Ser Pro Leu Glu
100 105 110
gtt tgg ctg aac ctg gga ggc agc aca cat gac ccg cat ggg cca gag 384
Val Trp Leu Asn Leu Gly Gly Ser Thr His Asp Pro His Gly Pro Glu
115 120 125
ccc acg ttc ccc ttt cag ggc aca ctg gag ccc cgg ccg gcg tca gat 432
Pro Thr Phe Pro Phe Gln Gly Thr Leu Glu Pro Arg Pro Ala Ser Asp
130 135 140
atc att gac atc gac tac ttc gaa gga ttg gat ggt gag ggc cgt ggc 480
Ile Ile Asp Ile Asp Tyr Phe Glu Gly Leu Asp Gly Glu Gly Arg Gly
145 150 155 160
gcc gac ttg ggg agc ttc ccg gtg tcg cca gga acc tca gag cac cac 528
Ala Asp Leu Gly Ser Phe Pro Val Ser Pro Gly Thr Ser Glu His His
165 170 175
ccc gat act ggg gga gag acc cct tcc tgg agc ctg ctt gac tta tac 576
Pro Asp Thr Gly Gly Glu Thr Pro Ser Trp Ser Leu Leu Asp Leu Tyr
180 185 190
gat gac ttc acc ccc ttt gat gaa tct gac ttc tac ccc act aca tcc 624
Asp Asp Phe Thr Pro Phe Asp Glu Ser Asp Phe Tyr Pro Thr Thr Ser
195 200 205
ttc tat gat gac ttg gag gaa gag gag gag gaa gag gat gac gac aag 672
Phe Tyr Asp Asp Leu Glu Glu Glu Glu Glu Glu Glu Asp Asp Asp Lys
210 215 220
gat gca gcg gaa ggt gga gac ctg gaa gat gaa agt gac ctt ctg gtg 720
Asp Ala Ala Glu Gly Gly Asp Leu Glu Asp Glu Ser Asp Leu Leu Val
225 230 235 240
ccc act gag aag cct ggg ctg agg cca ggg cct ggc cag ccc acc agt 768
Pro Thr Glu Lys Pro Gly Leu Arg Pro Gly Pro Gly Gln Pro Thr Ser
245 250 255
cgg tgg cat gct gtc ccc cca cag cat act ctg ggg ttg gtc cct ggc 816
Arg Trp His Ala Val Pro Pro Gln His Thr Leu Gly Leu Val Pro Gly
260 265 270
agc agc atc gcc ctc aga ccc cgt ccg gga gag ccg ggc agg gac ctg 864
Ser Ser Ile Ala Leu Arg Pro Arg Pro Gly Glu Pro Gly Arg Asp Leu
275 280 285
gcc ccg agc gag aac ggc act gag tgc cgc agc ggc ttt gtg cgg cat 912
Ala Pro Ser Glu Asn Gly Thr Glu Cys Arg Ser Gly Phe Val Arg His
290 295 300
aac ggc tcc tgc cga tcc gtg tgc gac ctc ttc cca agt tac tgt cac 960
Asn Gly Ser Cys Arg Ser Val Cys Asp Leu Phe Pro Ser Tyr Cys His
305 310 315 320
aac ggc ggc cag tgc tac ctg gtg gac aac ata ggg gcc ttc tgc agg 1008
Asn Gly Gly Gln Cys Tyr Leu Val Asp Asn Ile Gly Ala Phe Cys Arg
325 330 335
tgt aac aca cag gac tac atc tgg cac aag ggg atg cgc tgc gag tcc 1056
Cys Asn Thr Gln Asp Tyr Ile Trp His Lys Gly Met Arg Cys Glu Ser
340 345 350
atc atc acc gac ttc cag gtg atg tgc gtg gcc gtc ggc tca gcc gcc 1104
Ile Ile Thr Asp Phe Gln Val Met Cys Val Ala Val Gly Ser Ala Ala
355 360 365
ctc gtg ctg ctc ctg ctc ttc atg atg aca gtg ttc ttc gcc aag aag 1152
Leu Val Leu Leu Leu Leu Phe Met Met Thr Val Phe Phe Ala Lys Lys
370 375 380
ctg tat ctg ctc aag aca gag aat acc aag ctg cgt agg acc aac aaa 1200
Leu Tyr Leu Leu Lys Thr Glu Asn Thr Lys Leu Arg Arg Thr Asn Lys
385 390 395 400
ttc cgg acc ccg tct gag ctc cac aac gat aac ttc tcc ctt tcc acc 1248
Phe Arg Thr Pro Ser Glu Leu His Asn Asp Asn Phe Ser Leu Ser Thr
405 410 415
att gcc gaa ggc tct cac cca aac gac gat ccc agt gct tcc cac aaa 1296
Ile Ala Glu Gly Ser His Pro Asn Asp Asp Pro Ser Ala Ser His Lys
420 425 430
atc cag gag gtt ctc aag tcc tgc ctg aaa gag gag gag tca ttt aac 1344
Ile Gln Glu Val Leu Lys Ser Cys Leu Lys Glu Glu Glu Ser Phe Asn
435 440 445
atc cag aac tcc atg tcc ccc aaa ctg gag ggt ggc aaa ggt gac cag 1392
Ile Gln Asn Ser Met Ser Pro Lys Leu Glu Gly Gly Lys Gly Asp Gln
450 455 460
gct gac ttg gag gtg aac tgt ctt cag aac aac ctc acc taa 1434
Ala Asp Leu Glu Val Asn Cys Leu Gln Asn Asn Leu Thr
465 470 475
agcagagcaa gaagagaggg aatgggggag ggcggggggt ggcaggggaa gaaacatgac 1494
ctcctcttgt acagagtcta tttcttgtaa ccatttgtta aactctcttc tttttctggt 1554
ctcatggcat gccttgatgt attttgtaca ggagggagaa aacacaaaat aagcaaagaa 1614
cctgaacaga atcgcataca ccgggttgtt tcgtctgtgc tgtctgtata ttgcttctgc 1674
tgctgtgatt tctaaaccta tgctgttatt caactgactt tttttttgta ctttgaccca 1734
cctttttttg aaataagagt taaaaaacaa agttcttgaa ataaaacttt ttaaaaagcc 1794
attttccatc agtgtgtcca cttcctaccc attcttgtca gcttgagttg aattcttacg 1854
ttccctgaag atgtatattt atatgtgttt gaaatcctgg aagtgctctc tgtattagcc 1914
taggttgccg taactaaaca tcatagactg gatcacttaa gcaatagaaa tttatttttg 1974
aacagtaata gaggctggat ccccaagatc aaggtgccaa cagagttggt ttctggcagg 2034
gcctctctgc ctggcttgca aagagccatc ttcttgctat gtcctcacaa ggccttttgt 2094
ctgtgcacat ctcttcctct tctgataagg acaccagtcc tattggccta ggatccattt 2154
aacctcaatt acctcctcat aggccctact ccagatacag tcacacttag gggttatggc 2214
ttcaacatga cttttggggt gacataattc agtccacaag tctgtagcac ctgatt 2270
<210> 2
<211> 477
<212> Белок
<213> Canis familiaris
<400> 2
Gly Glu Glu Glu Thr Ser Cys Thr Ala Pro Gly Gly Leu Pro Ala Val
1 5 10 15
Val Gly Pro Gly Val Gly Pro Glu Glu Ala Leu Glu Ala Ser Ala Ala
20 25 30
Val Thr Gly Thr Ala Trp Leu Glu Ala Asp Ser Pro Gly Leu Gly Gly
35 40 45
Ala Thr Val Glu Ala Gly Ser Gly Asp Thr Gln Ala Leu Pro Ala Thr
50 55 60
Leu Pro Thr Pro Glu Glu Ala Leu Arg Arg Ala Ser Val Ala Pro Ala
65 70 75 80
Thr Pro Glu Thr Thr Glu Ala Ser Gly Pro Pro Ser Pro Thr Pro Gly
85 90 95
Asp Gln Leu Arg Pro Gly Pro Glu Leu Pro Lys Glu Ser Pro Leu Glu
100 105 110
Val Trp Leu Asn Leu Gly Gly Ser Thr His Asp Pro His Gly Pro Glu
115 120 125
Pro Thr Phe Pro Phe Gln Gly Thr Leu Glu Pro Arg Pro Ala Ser Asp
130 135 140
Ile Ile Asp Ile Asp Tyr Phe Glu Gly Leu Asp Gly Glu Gly Arg Gly
145 150 155 160
Ala Asp Leu Gly Ser Phe Pro Val Ser Pro Gly Thr Ser Glu His His
165 170 175
Pro Asp Thr Gly Gly Glu Thr Pro Ser Trp Ser Leu Leu Asp Leu Tyr
180 185 190
Asp Asp Phe Thr Pro Phe Asp Glu Ser Asp Phe Tyr Pro Thr Thr Ser
195 200 205
Phe Tyr Asp Asp Leu Glu Glu Glu Glu Glu Glu Glu Asp Asp Asp Lys
210 215 220
Asp Ala Ala Glu Gly Gly Asp Leu Glu Asp Glu Ser Asp Leu Leu Val
225 230 235 240
Pro Thr Glu Lys Pro Gly Leu Arg Pro Gly Pro Gly Gln Pro Thr Ser
245 250 255
Arg Trp His Ala Val Pro Pro Gln His Thr Leu Gly Leu Val Pro Gly
260 265 270
Ser Ser Ile Ala Leu Arg Pro Arg Pro Gly Glu Pro Gly Arg Asp Leu
275 280 285
Ala Pro Ser Glu Asn Gly Thr Glu Cys Arg Ser Gly Phe Val Arg His
290 295 300
Asn Gly Ser Cys Arg Ser Val Cys Asp Leu Phe Pro Ser Tyr Cys His
305 310 315 320
Asn Gly Gly Gln Cys Tyr Leu Val Asp Asn Ile Gly Ala Phe Cys Arg
325 330 335
Cys Asn Thr Gln Asp Tyr Ile Trp His Lys Gly Met Arg Cys Glu Ser
340 345 350
Ile Ile Thr Asp Phe Gln Val Met Cys Val Ala Val Gly Ser Ala Ala
355 360 365
Leu Val Leu Leu Leu Leu Phe Met Met Thr Val Phe Phe Ala Lys Lys
370 375 380
Leu Tyr Leu Leu Lys Thr Glu Asn Thr Lys Leu Arg Arg Thr Asn Lys
385 390 395 400
Phe Arg Thr Pro Ser Glu Leu His Asn Asp Asn Phe Ser Leu Ser Thr
405 410 415
Ile Ala Glu Gly Ser His Pro Asn Asp Asp Pro Ser Ala Ser His Lys
420 425 430
Ile Gln Glu Val Leu Lys Ser Cys Leu Lys Glu Glu Glu Ser Phe Asn
435 440 445
Ile Gln Asn Ser Met Ser Pro Lys Leu Glu Gly Gly Lys Gly Asp Gln
450 455 460
Ala Asp Leu Glu Val Asn Cys Leu Gln Asn Asn Leu Thr
465 470 475
<210> 3
<211> 2175
<212> ДНК
<213> Homo sapiens
<220>
<221> Кодирующая последовательность
<222> (177)..(1796)
<223>
<400> 3
ggggaggcgc ggcgcgccgg ggacagcggc ggacggcggc ggcggcggca tgcggctcct 60
cgcgctgccc atcgtgggct gaggcggccg cagaaccggc gggaggcgcg gcggccgggc 120
gagccgaggg cgcagccagc cgggcggacc gcggacagcg gtcggggcgc cgcgcc atg 179
Met
1
ggg cga gcc ggg ggc ggg ggc ccg ggc cgg ggg ccg ccg cca ctg ctg 227
Gly Arg Ala Gly Gly Gly Gly Pro Gly Arg Gly Pro Pro Pro Leu Leu
5 10 15
ctg ttt ctg ggg gcc gcg ctg gtc ctg gcc tct ggg gcc gtg ccg gcg 275
Leu Phe Leu Gly Ala Ala Leu Val Leu Ala Ser Gly Ala Val Pro Ala
20 25 30
cgt gag gcg ggc agc gcg gtt gag gcc gaa gag ctg gtg aag ggc agc 323
Arg Glu Ala Gly Ser Ala Val Glu Ala Glu Glu Leu Val Lys Gly Ser
35 40 45
ccg gcg tgg gag ccg cct gcc aac gac acg cgg gaa gaa gcc ggc cca 371
Pro Ala Trp Glu Pro Pro Ala Asn Asp Thr Arg Glu Glu Ala Gly Pro
50 55 60 65
cca gcg gct ggg gaa gat gag gcg tcg tgg acg gcg ccc ggt ggc gag 419
Pro Ala Ala Gly Glu Asp Glu Ala Ser Trp Thr Ala Pro Gly Gly Glu
70 75 80
ctg gcc ggg cca gaa gag gtg ctg cag gag tcg gct gcg gtg acc ggc 467
Leu Ala Gly Pro Glu Glu Val Leu Gln Glu Ser Ala Ala Val Thr Gly
85 90 95
acc gcc tgg ctg gaa gct gac agc cca ggc ctg gga gga gtg acc gca 515
Thr Ala Trp Leu Glu Ala Asp Ser Pro Gly Leu Gly Gly Val Thr Ala
100 105 110
gag gcg ggc agc ggc gat gcc cag gcc ctt cca gct acg ctc cag gct 563
Glu Ala Gly Ser Gly Asp Ala Gln Ala Leu Pro Ala Thr Leu Gln Ala
115 120 125
ccc cac gag gtc ctc ggg cag tca atc atg ccc cct gcc att cct gag 611
Pro His Glu Val Leu Gly Gln Ser Ile Met Pro Pro Ala Ile Pro Glu
130 135 140 145
gct aca gag gcc agc ggg cca ccc tcc ccc acc ccc ggc gac aag ctg 659
Ala Thr Glu Ala Ser Gly Pro Pro Ser Pro Thr Pro Gly Asp Lys Leu
150 155 160
agc cca gct tct gaa ctc ccc aag gag agc ccc ttg gag gtt tgg ctg 707
Ser Pro Ala Ser Glu Leu Pro Lys Glu Ser Pro Leu Glu Val Trp Leu
165 170 175
aac ctg ggg ggc agc aca ccc gac cct caa ggg cca gag ctg act tac 755
Asn Leu Gly Gly Ser Thr Pro Asp Pro Gln Gly Pro Glu Leu Thr Tyr
180 185 190
cca ttt cag ggc acc ctg gag ccc caa ccg gca tca gat atc att gac 803
Pro Phe Gln Gly Thr Leu Glu Pro Gln Pro Ala Ser Asp Ile Ile Asp
195 200 205
atc gac tac ttc gaa gga ctg gat ggt gag ggt cgt ggc gca gat ctg 851
Ile Asp Tyr Phe Glu Gly Leu Asp Gly Glu Gly Arg Gly Ala Asp Leu
210 215 220 225
ggg agc ttc cca ggg tca cca gga acc tca gag aac cac cct gat act 899
Gly Ser Phe Pro Gly Ser Pro Gly Thr Ser Glu Asn His Pro Asp Thr
230 235 240
gag gga gag acc cct tcc tgg agc ctg ctt gac tta tac gat gat ttc 947
Glu Gly Glu Thr Pro Ser Trp Ser Leu Leu Asp Leu Tyr Asp Asp Phe
245 250 255
acc ccc ttc gat gaa tct gat ttc tac ccc acc aca tcc ttt tat gat 995
Thr Pro Phe Asp Glu Ser Asp Phe Tyr Pro Thr Thr Ser Phe Tyr Asp
260 265 270
gac ttg gat gaa gag gag gag gaa gag gag gat gac aaa gat gca gta 1043
Asp Leu Asp Glu Glu Glu Glu Glu Glu Glu Asp Asp Lys Asp Ala Val
275 280 285
gga ggt gga gac cta gaa gat gaa aat gag ctt cta gtg ccc act ggg 1091
Gly Gly Gly Asp Leu Glu Asp Glu Asn Glu Leu Leu Val Pro Thr Gly
290 295 300 305
aag cct ggt ctg ggg ccc ggg aca ggc cag ccc acc agt cgg tgg cat 1139
Lys Pro Gly Leu Gly Pro Gly Thr Gly Gln Pro Thr Ser Arg Trp His
310 315 320
gct gtc cct cca cag cac act ctg ggg tcg gtc ccc ggc agc agc atc 1187
Ala Val Pro Pro Gln His Thr Leu Gly Ser Val Pro Gly Ser Ser Ile
325 330 335
gcc ctc agg ccc cgc cca gga gag cca ggc agg gac ttg gcc tcc agt 1235
Ala Leu Arg Pro Arg Pro Gly Glu Pro Gly Arg Asp Leu Ala Ser Ser
340 345 350
gaa aat ggc act gag tgc cgc agt ggc ttt gtg cgg cat aac ggc tcc 1283
Glu Asn Gly Thr Glu Cys Arg Ser Gly Phe Val Arg His Asn Gly Ser
355 360 365
tgc cgg tca gtg tgc gac ctc ttc cca agt tac tgt cac aat ggc ggc 1331
Cys Arg Ser Val Cys Asp Leu Phe Pro Ser Tyr Cys His Asn Gly Gly
370 375 380 385
cag tgc tac ctg gtg gag aac ata ggg gcc ttc tgc agg tgc aac acg 1379
Gln Cys Tyr Leu Val Glu Asn Ile Gly Ala Phe Cys Arg Cys Asn Thr
390 395 400
cag gac tac atc tgg cac aag ggg atg cgc tgc gag tcc atc atc acc 1427
Gln Asp Tyr Ile Trp His Lys Gly Met Arg Cys Glu Ser Ile Ile Thr
405 410 415
gac ttc cag gtg atg tgc gtg gcc gtg ggc tcg gct gcc ctc gtc ctg 1475
Asp Phe Gln Val Met Cys Val Ala Val Gly Ser Ala Ala Leu Val Leu
420 425 430
ctc ctg ctc ttc atg atg acg gtg ttc ttt gcc aag aag ctc tac ctg 1523
Leu Leu Leu Phe Met Met Thr Val Phe Phe Ala Lys Lys Leu Tyr Leu
435 440 445
ctc aag acg gag aat acc aag ctg cgt agg acc aac aaa ttc cgg acc 1571
Leu Lys Thr Glu Asn Thr Lys Leu Arg Arg Thr Asn Lys Phe Arg Thr
450 455 460 465
cca tct gag ctc cac aat gat aac ttc tcc ctc tcc acc att gcc gag 1619
Pro Ser Glu Leu His Asn Asp Asn Phe Ser Leu Ser Thr Ile Ala Glu
470 475 480
ggc tct cac cca aat gat gat cct agt gct ccc cac aaa atc cag gag 1667
Gly Ser His Pro Asn Asp Asp Pro Ser Ala Pro His Lys Ile Gln Glu
485 490 495
gtt ctc aag tcc tgc ctg aaa gag gag gag tca ttt aac atc cag aac 1715
Val Leu Lys Ser Cys Leu Lys Glu Glu Glu Ser Phe Asn Ile Gln Asn
500 505 510
tcc atg tcg ccc aaa ctt gag ggt ggc aaa ggt gac cag gct gac ttg 1763
Ser Met Ser Pro Lys Leu Glu Gly Gly Lys Gly Asp Gln Ala Asp Leu
515 520 525
gat gtg aac tgt ctt cag aat aat tta acc taa agcagagcaa gaagagagga 1816
Asp Val Asn Cys Leu Gln Asn Asn Leu Thr
530 535
agcgggggta gtgggtgggg ggtaggggaa gaaacattat ctcctcttgt acagagtcta 1876
tttcttgtaa ccatttgtta aactcttttc tttttctgat ctcatggcat gcttttatgt 1936
attttgtaca ggaggcaaaa aaatacttaa aataagcaaa gaaactgaac agaattgcat 1996
acattgggtt gttttttctg tgctgtctgt acattgcttc tgctgctgtg atttctaaac 2056
ctgtgctgtt attcaactga cttttttttg tactttgacc cacgtttttt tgaaatacca 2116
gtaaaaaaca aagttcttga aataaaactt tttaaaaagt taaaaaaaaa aaaaaaaaa 2175
<210> 4
<211> 539
<212> Белок
<213> Homo sapiens
<400> 4
Met Gly Arg Ala Gly Gly Gly Gly Pro Gly Arg Gly Pro Pro Pro Leu
1 5 10 15
Leu Leu Phe Leu Gly Ala Ala Leu Val Leu Ala Ser Gly Ala Val Pro
20 25 30
Ala Arg Glu Ala Gly Ser Ala Val Glu Ala Glu Glu Leu Val Lys Gly
35 40 45
Ser Pro Ala Trp Glu Pro Pro Ala Asn Asp Thr Arg Glu Glu Ala Gly
50 55 60
Pro Pro Ala Ala Gly Glu Asp Glu Ala Ser Trp Thr Ala Pro Gly Gly
65 70 75 80
Glu Leu Ala Gly Pro Glu Glu Val Leu Gln Glu Ser Ala Ala Val Thr
85 90 95
Gly Thr Ala Trp Leu Glu Ala Asp Ser Pro Gly Leu Gly Gly Val Thr
100 105 110
Ala Glu Ala Gly Ser Gly Asp Ala Gln Ala Leu Pro Ala Thr Leu Gln
115 120 125
Ala Pro His Glu Val Leu Gly Gln Ser Ile Met Pro Pro Ala Ile Pro
130 135 140
Glu Ala Thr Glu Ala Ser Gly Pro Pro Ser Pro Thr Pro Gly Asp Lys
145 150 155 160
Leu Ser Pro Ala Ser Glu Leu Pro Lys Glu Ser Pro Leu Glu Val Trp
165 170 175
Leu Asn Leu Gly Gly Ser Thr Pro Asp Pro Gln Gly Pro Glu Leu Thr
180 185 190
Tyr Pro Phe Gln Gly Thr Leu Glu Pro Gln Pro Ala Ser Asp Ile Ile
195 200 205
Asp Ile Asp Tyr Phe Glu Gly Leu Asp Gly Glu Gly Arg Gly Ala Asp
210 215 220
Leu Gly Ser Phe Pro Gly Ser Pro Gly Thr Ser Glu Asn His Pro Asp
225 230 235 240
Thr Glu Gly Glu Thr Pro Ser Trp Ser Leu Leu Asp Leu Tyr Asp Asp
245 250 255
Phe Thr Pro Phe Asp Glu Ser Asp Phe Tyr Pro Thr Thr Ser Phe Tyr
260 265 270
Asp Asp Leu Asp Glu Glu Glu Glu Glu Glu Glu Asp Asp Lys Asp Ala
275 280 285
Val Gly Gly Gly Asp Leu Glu Asp Glu Asn Glu Leu Leu Val Pro Thr
290 295 300
Gly Lys Pro Gly Leu Gly Pro Gly Thr Gly Gln Pro Thr Ser Arg Trp
305 310 315 320
His Ala Val Pro Pro Gln His Thr Leu Gly Ser Val Pro Gly Ser Ser
325 330 335
Ile Ala Leu Arg Pro Arg Pro Gly Glu Pro Gly Arg Asp Leu Ala Ser
340 345 350
Ser Glu Asn Gly Thr Glu Cys Arg Ser Gly Phe Val Arg His Asn Gly
355 360 365
Ser Cys Arg Ser Val Cys Asp Leu Phe Pro Ser Tyr Cys His Asn Gly
370 375 380
Gly Gln Cys Tyr Leu Val Glu Asn Ile Gly Ala Phe Cys Arg Cys Asn
385 390 395 400
Thr Gln Asp Tyr Ile Trp His Lys Gly Met Arg Cys Glu Ser Ile Ile
405 410 415
Thr Asp Phe Gln Val Met Cys Val Ala Val Gly Ser Ala Ala Leu Val
420 425 430
Leu Leu Leu Leu Phe Met Met Thr Val Phe Phe Ala Lys Lys Leu Tyr
435 440 445
Leu Leu Lys Thr Glu Asn Thr Lys Leu Arg Arg Thr Asn Lys Phe Arg
450 455 460
Thr Pro Ser Glu Leu His Asn Asp Asn Phe Ser Leu Ser Thr Ile Ala
465 470 475 480
Glu Gly Ser His Pro Asn Asp Asp Pro Ser Ala Pro His Lys Ile Gln
485 490 495
Glu Val Leu Lys Ser Cys Leu Lys Glu Glu Glu Ser Phe Asn Ile Gln
500 505 510
Asn Ser Met Ser Pro Lys Leu Glu Gly Gly Lys Gly Asp Gln Ala Asp
515 520 525
Leu Asp Val Asn Cys Leu Gln Asn Asn Leu Thr
530 535
<210> 5
<211> 2253
<212> ДНК
<213> Homo sapiens
<220>
<221> Кодирующая последовательность
<222> (669)..(1874)
<223>
<400> 5
agtccagtga ggaataactg tagataagat gagatgatgg gcattaagtg taactgctga 60
gaagccttca tagagggaaa ggtgataaga actgagtttt tctggcatga ggagtttacc 120
aggcaggaga ggtggagacg ctaggctagg caaataggct gcagccatag ttctgctgag 180
aaacaggttt tgaaccaagg caactccatc tggagatatg tatcagaaag attaggagat 240
agaatctcca tcttggtcac ttatttggtc ttctaagaca gcaatgggac cgtctcttaa 300
atactggagt tgtcctatct ccttgggctg ataccctgaa ctgtcatttg gcgcgtgagg 360
cgggcagcgc ggttgaggcc gaagagctgg tgaagggcag cccggcgtgg gagccgcctg 420
ccaacgacac gcgggaagaa gccggcccac cagcggctgg ggaagatgag gcgtcgtgga 480
cggcgcccgg tggcgagctg gccgggccag aagaggtgct gcaggagtcg gctgcggtga 540
ccggcaccgc ctggctggaa gctgacagcc caggcctggg aggagtgacc gcagaggcgg 600
gcagcggcga tgcccaggcc cttccagcta cgctccaggc tccccacgag gtcctcgggc 660
agtcaatc atg ccc cct gcc att cct gag gct aca gag gcc agc ggg cca 710
Met Pro Pro Ala Ile Pro Glu Ala Thr Glu Ala Ser Gly Pro
1 5 10
ccc tcc ccc acc ccc ggc gac aag ctg agc cca gct tct gaa ctc ccc 758
Pro Ser Pro Thr Pro Gly Asp Lys Leu Ser Pro Ala Ser Glu Leu Pro
15 20 25 30
aag gag agc ccc ttg gag gtt tgg ctg aac ctg ggg ggc agc aca ccc 806
Lys Glu Ser Pro Leu Glu Val Trp Leu Asn Leu Gly Gly Ser Thr Pro
35 40 45
gac cct caa ggg cca gag ctg act tac cca ttt cag ggc acc ctg gag 854
Asp Pro Gln Gly Pro Glu Leu Thr Tyr Pro Phe Gln Gly Thr Leu Glu
50 55 60
ccc caa ccg gca tca gat atc att gac atc gac tac ttc gaa gga ctg 902
Pro Gln Pro Ala Ser Asp Ile Ile Asp Ile Asp Tyr Phe Glu Gly Leu
65 70 75
gat ggt gag ggt cgt ggc gca gat ctg ggg agc ttc cca ggg tca cca 950
Asp Gly Glu Gly Arg Gly Ala Asp Leu Gly Ser Phe Pro Gly Ser Pro
80 85 90
gga acc tca gag aac cac cct gat act gag gga gag acc cct tcc tgg 998
Gly Thr Ser Glu Asn His Pro Asp Thr Glu Gly Glu Thr Pro Ser Trp
95 100 105 110
agc ctg ctt gac tta tac gat gat ttc acc ccc ttc gat gaa tct gat 1046
Ser Leu Leu Asp Leu Tyr Asp Asp Phe Thr Pro Phe Asp Glu Ser Asp
115 120 125
ttc tac ccc acc aca tcc ttt tat gat gac ttg gat gaa gag gag gag 1094
Phe Tyr Pro Thr Thr Ser Phe Tyr Asp Asp Leu Asp Glu Glu Glu Glu
130 135 140
gaa gag gag gat gac aaa gat gca gta gga ggt gga gac cta gaa gat 1142
Glu Glu Glu Asp Asp Lys Asp Ala Val Gly Gly Gly Asp Leu Glu Asp
145 150 155
gaa aat gag ctt cta gtg ccc act ggg aag cct ggt ctg ggg ccc ggg 1190
Glu Asn Glu Leu Leu Val Pro Thr Gly Lys Pro Gly Leu Gly Pro Gly
160 165 170
aca ggc cag ccc acc agt cgg tgg cat gct gtc cct cca cag cac act 1238
Thr Gly Gln Pro Thr Ser Arg Trp His Ala Val Pro Pro Gln His Thr
175 180 185 190
ctg ggg tcg gtc ccc ggc agc agc atc gcc ctc agg ccc cgc cca gga 1286
Leu Gly Ser Val Pro Gly Ser Ser Ile Ala Leu Arg Pro Arg Pro Gly
195 200 205
gag cca ggc agg gac ttg gcc tcc agt gaa aat ggc act gag tgc cgc 1334
Glu Pro Gly Arg Asp Leu Ala Ser Ser Glu Asn Gly Thr Glu Cys Arg
210 215 220
agt ggc ttt gtg cgg cat aac ggc tcc tgc cgg tca gtg tgc gac ctc 1382
Ser Gly Phe Val Arg His Asn Gly Ser Cys Arg Ser Val Cys Asp Leu
225 230 235
ttc cca agt tac tgt cac aat ggc ggc cag tgc tac ctg gtg gag aac 1430
Phe Pro Ser Tyr Cys His Asn Gly Gly Gln Cys Tyr Leu Val Glu Asn
240 245 250
ata ggg gcc ttc tgc agg tgc aac acg cag gac tac atc tgg cac aag 1478
Ile Gly Ala Phe Cys Arg Cys Asn Thr Gln Asp Tyr Ile Trp His Lys
255 260 265 270
ggg atg cgc tgc gag tcc atc atc acc gac ttc cag gtg atg tgc gtg 1526
Gly Met Arg Cys Glu Ser Ile Ile Thr Asp Phe Gln Val Met Cys Val
275 280 285
gcc gtg ggc tcg gct gcc ctc gtc ctg ctc ctg ctc ttc atg atg acg 1574
Ala Val Gly Ser Ala Ala Leu Val Leu Leu Leu Leu Phe Met Met Thr
290 295 300
gtg ttc ttt gcc aag aag ctc tac ctg ctc aag acg gag aat acc aag 1622
Val Phe Phe Ala Lys Lys Leu Tyr Leu Leu Lys Thr Glu Asn Thr Lys
305 310 315
ctg cgt agg acc aac aaa ttc cgg acc cca tct gag ctc cac aat gat 1670
Leu Arg Arg Thr Asn Lys Phe Arg Thr Pro Ser Glu Leu His Asn Asp
320 325 330
aac ttc tcc ctc tcc acc att gcc gag ggc tct cac cca aat gat gat 1718
Asn Phe Ser Leu Ser Thr Ile Ala Glu Gly Ser His Pro Asn Asp Asp
335 340 345 350
cct agt gct ccc cac aaa atc cag gag gtt ctc aag tcc tgc ctg aaa 1766
Pro Ser Ala Pro His Lys Ile Gln Glu Val Leu Lys Ser Cys Leu Lys
355 360 365
gag gag gag tca ttt aac atc cag aac tcc atg tcg ccc aaa ctt gag 1814
Glu Glu Glu Ser Phe Asn Ile Gln Asn Ser Met Ser Pro Lys Leu Glu
370 375 380
ggt ggc aaa ggt gac cag gct gac ttg gat gtg aac tgt ctt cag aat 1862
Gly Gly Lys Gly Asp Gln Ala Asp Leu Asp Val Asn Cys Leu Gln Asn
385 390 395
aat tta acc taa agcagagcaa gaagagagga agcgggggta gtgggtgggg 1914
Asn Leu Thr
400
ggtaggggaa gaaacattat ctcctcttgt acagagtcta tttcttgtaa ccatttgtta 1974
aactcttttc tttttctgat ctcatggcat gcttttatgt attttgtaca ggaggcaaaa 2034
aaatacttaa aataagcaaa gaaactgaac agaattgcat acattgggtt gttttttctg 2094
tgctgtctgt acattgcttc tgctgctgtg atttctaaac ctgtgctgtt attcaactga 2154
cttttttttg tactttgacc cacgtttttt tgaaatacca gtaaaaaaca aagttcttga 2214
aataaaactt tttaaaaagt taaaaaaaaa aaaaaaaaa 2253
<210> 6
<211> 401
<212> Белок
<213> Homo sapiens
<400> 6
Met Pro Pro Ala Ile Pro Glu Ala Thr Glu Ala Ser Gly Pro Pro Ser
1 5 10 15
Pro Thr Pro Gly Asp Lys Leu Ser Pro Ala Ser Glu Leu Pro Lys Glu
20 25 30
Ser Pro Leu Glu Val Trp Leu Asn Leu Gly Gly Ser Thr Pro Asp Pro
35 40 45
Gln Gly Pro Glu Leu Thr Tyr Pro Phe Gln Gly Thr Leu Glu Pro Gln
50 55 60
Pro Ala Ser Asp Ile Ile Asp Ile Asp Tyr Phe Glu Gly Leu Asp Gly
65 70 75 80
Glu Gly Arg Gly Ala Asp Leu Gly Ser Phe Pro Gly Ser Pro Gly Thr
85 90 95
Ser Glu Asn His Pro Asp Thr Glu Gly Glu Thr Pro Ser Trp Ser Leu
100 105 110
Leu Asp Leu Tyr Asp Asp Phe Thr Pro Phe Asp Glu Ser Asp Phe Tyr
115 120 125
Pro Thr Thr Ser Phe Tyr Asp Asp Leu Asp Glu Glu Glu Glu Glu Glu
130 135 140
Glu Asp Asp Lys Asp Ala Val Gly Gly Gly Asp Leu Glu Asp Glu Asn
145 150 155 160
Glu Leu Leu Val Pro Thr Gly Lys Pro Gly Leu Gly Pro Gly Thr Gly
165 170 175
Gln Pro Thr Ser Arg Trp His Ala Val Pro Pro Gln His Thr Leu Gly
180 185 190
Ser Val Pro Gly Ser Ser Ile Ala Leu Arg Pro Arg Pro Gly Glu Pro
195 200 205
Gly Arg Asp Leu Ala Ser Ser Glu Asn Gly Thr Glu Cys Arg Ser Gly
210 215 220
Phe Val Arg His Asn Gly Ser Cys Arg Ser Val Cys Asp Leu Phe Pro
225 230 235 240
Ser Tyr Cys His Asn Gly Gly Gln Cys Tyr Leu Val Glu Asn Ile Gly
245 250 255
Ala Phe Cys Arg Cys Asn Thr Gln Asp Tyr Ile Trp His Lys Gly Met
260 265 270
Arg Cys Glu Ser Ile Ile Thr Asp Phe Gln Val Met Cys Val Ala Val
275 280 285
Gly Ser Ala Ala Leu Val Leu Leu Leu Leu Phe Met Met Thr Val Phe
290 295 300
Phe Ala Lys Lys Leu Tyr Leu Leu Lys Thr Glu Asn Thr Lys Leu Arg
305 310 315 320
Arg Thr Asn Lys Phe Arg Thr Pro Ser Glu Leu His Asn Asp Asn Phe
325 330 335
Ser Leu Ser Thr Ile Ala Glu Gly Ser His Pro Asn Asp Asp Pro Ser
340 345 350
Ala Pro His Lys Ile Gln Glu Val Leu Lys Ser Cys Leu Lys Glu Glu
355 360 365
Glu Ser Phe Asn Ile Gln Asn Ser Met Ser Pro Lys Leu Glu Gly Gly
370 375 380
Lys Gly Asp Gln Ala Asp Leu Asp Val Asn Cys Leu Gln Asn Asn Leu
385 390 395 400
Thr
<210> 7
<211> 2256
<212> ДНК
<213> Homo sapiens
<220>
<221> Кодирующая последовательность
<222> (177)..(1877)
<223>
<400> 7
ggggaggcgc ggcgcgccgg ggacagcggc ggacggcggc ggcggcggca tgcggctcct 60
cgcgctgccc atcgtgggct gaggcggccg cagaaccggc gggaggcgcg gcggccgggc 120
gagccgaggg cgcagccagc cgggcggacc gcggacagcg gtcggggcgc cgcgcc atg 179
Met
1
ggg cga gcc ggg ggc ggg ggc ccg ggc cgg ggg ccg ccg cca ctg ctg 227
Gly Arg Ala Gly Gly Gly Gly Pro Gly Arg Gly Pro Pro Pro Leu Leu
5 10 15
ctg ttt ctg ggg gcc gcg ctg gtc ctg gcc tct ggg gcc gtg ccg gcg 275
Leu Phe Leu Gly Ala Ala Leu Val Leu Ala Ser Gly Ala Val Pro Ala
20 25 30
cgt gag gcg ggc agc gcg gtt gag gcc gaa gag ctg gtg aag ggc agc 323
Arg Glu Ala Gly Ser Ala Val Glu Ala Glu Glu Leu Val Lys Gly Ser
35 40 45
ccg gcg tgg gag ccg cct gcc aac gac acg cgg gaa gaa gcc ggc cca 371
Pro Ala Trp Glu Pro Pro Ala Asn Asp Thr Arg Glu Glu Ala Gly Pro
50 55 60 65
cca gcg gct ggg gaa gat gag gcg tcg tgg acg gcg ccc ggt ggc gag 419
Pro Ala Ala Gly Glu Asp Glu Ala Ser Trp Thr Ala Pro Gly Gly Glu
70 75 80
ctg gcc ggg cca gaa gag gtg ctg cag gag tcg gct gcg gtg acc ggc 467
Leu Ala Gly Pro Glu Glu Val Leu Gln Glu Ser Ala Ala Val Thr Gly
85 90 95
acc gcc tgg ctg gaa gct gac agc cca ggc ctg gga gga gtg acc gca 515
Thr Ala Trp Leu Glu Ala Asp Ser Pro Gly Leu Gly Gly Val Thr Ala
100 105 110
gag gcg ggc agc ggc gat gcc cag gcc ctt cca gct acg ctc cag gct 563
Glu Ala Gly Ser Gly Asp Ala Gln Ala Leu Pro Ala Thr Leu Gln Ala
115 120 125
ccc cac gag gtc ctc ggg cag tca atc atg ccc cct gcc att cct gag 611
Pro His Glu Val Leu Gly Gln Ser Ile Met Pro Pro Ala Ile Pro Glu
130 135 140 145
gct aca gag gcc agc ggg cca ccc tcc ccc acc ccc ggc gac aag ctg 659
Ala Thr Glu Ala Ser Gly Pro Pro Ser Pro Thr Pro Gly Asp Lys Leu
150 155 160
agc cca gct tct gaa ctc ccc aag gag agc ccc ttg gag gtt tgg ctg 707
Ser Pro Ala Ser Glu Leu Pro Lys Glu Ser Pro Leu Glu Val Trp Leu
165 170 175
aac ctg ggg ggc agc aca ccc gac cct caa ggg cca gag ctg act tac 755
Asn Leu Gly Gly Ser Thr Pro Asp Pro Gln Gly Pro Glu Leu Thr Tyr
180 185 190
cca ttt cag ggc acc ctg gag ccc caa ccg gca tca gat atc att gac 803
Pro Phe Gln Gly Thr Leu Glu Pro Gln Pro Ala Ser Asp Ile Ile Asp
195 200 205
atc gac tac ttc gaa gga ctg gat ggt gag ggt cgt ggc gca gat ctg 851
Ile Asp Tyr Phe Glu Gly Leu Asp Gly Glu Gly Arg Gly Ala Asp Leu
210 215 220 225
ggg agc ttc cca ggg tca cca gga acc tca gag aac cac cct gat act 899
Gly Ser Phe Pro Gly Ser Pro Gly Thr Ser Glu Asn His Pro Asp Thr
230 235 240
gag gga gag acc cct tcc tgg agc ctg ctt gac tta tac gat gat ttc 947
Glu Gly Glu Thr Pro Ser Trp Ser Leu Leu Asp Leu Tyr Asp Asp Phe
245 250 255
acc ccc ttc gat gaa tct gat ttc tac ccc acc aca tcc ttt tat gat 995
Thr Pro Phe Asp Glu Ser Asp Phe Tyr Pro Thr Thr Ser Phe Tyr Asp
260 265 270
gac ttg gat gaa gag gag gag gaa gag gag gat gac aaa gat gca gta 1043
Asp Leu Asp Glu Glu Glu Glu Glu Glu Glu Asp Asp Lys Asp Ala Val
275 280 285
gga ggt gga gac cta gaa gat gaa aat gag ctt cta gtg ccc act ggg 1091
Gly Gly Gly Asp Leu Glu Asp Glu Asn Glu Leu Leu Val Pro Thr Gly
290 295 300 305
aag cct ggt ctg ggg ccc ggg aca ggc cag ccc acc agt cgg tgg cat 1139
Lys Pro Gly Leu Gly Pro Gly Thr Gly Gln Pro Thr Ser Arg Trp His
310 315 320
gct gtc cct cca cag cac act ctg ggg tcg gtc ccc ggc agc agc atc 1187
Ala Val Pro Pro Gln His Thr Leu Gly Ser Val Pro Gly Ser Ser Ile
325 330 335
gcc ctc agg ccc cgc cca gga gag cca ggc agg gac ttg gcc tcc agt 1235
Ala Leu Arg Pro Arg Pro Gly Glu Pro Gly Arg Asp Leu Ala Ser Ser
340 345 350
gaa aat ggc act gag tgc cgc agt ggc ttt gtg cgg cat aac ggc tcc 1283
Glu Asn Gly Thr Glu Cys Arg Ser Gly Phe Val Arg His Asn Gly Ser
355 360 365
tgc cgg tca gtg tgc gac ctc ttc cca agt tac tgt cac aat ggc ggc 1331
Cys Arg Ser Val Cys Asp Leu Phe Pro Ser Tyr Cys His Asn Gly Gly
370 375 380 385
cag tgc tac ctg gtg gag aac ata ggg gcc ttc tgc agg tgc aac acg 1379
Gln Cys Tyr Leu Val Glu Asn Ile Gly Ala Phe Cys Arg Cys Asn Thr
390 395 400
cag gac tac atc tgg cac aag ggg atg cgc tgc gag tcc atc atc acc 1427
Gln Asp Tyr Ile Trp His Lys Gly Met Arg Cys Glu Ser Ile Ile Thr
405 410 415
gac ttc cag gtg atg tgc gtg gcc gtg ggc tcg gct gcc ctc gtc ctg 1475
Asp Phe Gln Val Met Cys Val Ala Val Gly Ser Ala Ala Leu Val Leu
420 425 430
ctc ctg ctc ttc atg atg acg gtg ttc ttt gcc aag aag ctc tac ctg 1523
Leu Leu Leu Phe Met Met Thr Val Phe Phe Ala Lys Lys Leu Tyr Leu
435 440 445
ctc aag acg gag aat acc aag ctg cgt agg acc aac aaa ttc cgg acc 1571
Leu Lys Thr Glu Asn Thr Lys Leu Arg Arg Thr Asn Lys Phe Arg Thr
450 455 460 465
cca tct gag ctc cac aat gat aac ttc tcc ctc tcc acc att gcc gag 1619
Pro Ser Glu Leu His Asn Asp Asn Phe Ser Leu Ser Thr Ile Ala Glu
470 475 480
ggc tct cac cca aat gta agg aaa ctt tgc aac act ccc cgt acc tcc 1667
Gly Ser His Pro Asn Val Arg Lys Leu Cys Asn Thr Pro Arg Thr Ser
485 490 495
tcc ccc cat gcc cgt gcc ttg gct cac tat gat aac gtt atc tgt cag 1715
Ser Pro His Ala Arg Ala Leu Ala His Tyr Asp Asn Val Ile Cys Gln
500 505 510
gat gat cct agt gct ccc cac aaa atc cag gag gtt ctc aag tcc tgc 1763
Asp Asp Pro Ser Ala Pro His Lys Ile Gln Glu Val Leu Lys Ser Cys
515 520 525
ctg aaa gag gag gag tca ttt aac atc cag aac tcc atg tcg ccc aaa 1811
Leu Lys Glu Glu Glu Ser Phe Asn Ile Gln Asn Ser Met Ser Pro Lys
530 535 540 545
ctt gag ggt ggc aaa ggt gac cag gct gac ttg gat gtg aac tgt ctt 1859
Leu Glu Gly Gly Lys Gly Asp Gln Ala Asp Leu Asp Val Asn Cys Leu
550 555 560
cag aat aat tta acc taa agcagagcaa gaagagagga agcgggggta 1907
Gln Asn Asn Leu Thr
565
gtgggtgggg ggtaggggaa gaaacattat ctcctcttgt acagagtcta tttcttgtaa 1967
ccatttgtta aactcttttc tttttctgat ctcatggcat gcttttatgt attttgtaca 2027
ggaggcaaaa aaatacttaa aataagcaaa gaaactgaac agaattgcat acattgggtt 2087
gttttttctg tgctgtctgt acattgcttc tgctgctgtg atttctaaac ctgtgctgtt 2147
attcaactga cttttttttg tactttgacc cacgtttttt tgaaatacca gtaaaaaaca 2207
aagttcttga aataaaactt tttaaaaagt taaaaaaaaa aaaaaaaaa 2256
<210> 8
<211> 566
<212> Белок
<213> Homo sapiens
<400> 8
Met Gly Arg Ala Gly Gly Gly Gly Pro Gly Arg Gly Pro Pro Pro Leu
1 5 10 15
Leu Leu Phe Leu Gly Ala Ala Leu Val Leu Ala Ser Gly Ala Val Pro
20 25 30
Ala Arg Glu Ala Gly Ser Ala Val Glu Ala Glu Glu Leu Val Lys Gly
35 40 45
Ser Pro Ala Trp Glu Pro Pro Ala Asn Asp Thr Arg Glu Glu Ala Gly
50 55 60
Pro Pro Ala Ala Gly Glu Asp Glu Ala Ser Trp Thr Ala Pro Gly Gly
65 70 75 80
Glu Leu Ala Gly Pro Glu Glu Val Leu Gln Glu Ser Ala Ala Val Thr
85 90 95
Gly Thr Ala Trp Leu Glu Ala Asp Ser Pro Gly Leu Gly Gly Val Thr
100 105 110
Ala Glu Ala Gly Ser Gly Asp Ala Gln Ala Leu Pro Ala Thr Leu Gln
115 120 125
Ala Pro His Glu Val Leu Gly Gln Ser Ile Met Pro Pro Ala Ile Pro
130 135 140
Glu Ala Thr Glu Ala Ser Gly Pro Pro Ser Pro Thr Pro Gly Asp Lys
145 150 155 160
Leu Ser Pro Ala Ser Glu Leu Pro Lys Glu Ser Pro Leu Glu Val Trp
165 170 175
Leu Asn Leu Gly Gly Ser Thr Pro Asp Pro Gln Gly Pro Glu Leu Thr
180 185 190
Tyr Pro Phe Gln Gly Thr Leu Glu Pro Gln Pro Ala Ser Asp Ile Ile
195 200 205
Asp Ile Asp Tyr Phe Glu Gly Leu Asp Gly Glu Gly Arg Gly Ala Asp
210 215 220
Leu Gly Ser Phe Pro Gly Ser Pro Gly Thr Ser Glu Asn His Pro Asp
225 230 235 240
Thr Glu Gly Glu Thr Pro Ser Trp Ser Leu Leu Asp Leu Tyr Asp Asp
245 250 255
Phe Thr Pro Phe Asp Glu Ser Asp Phe Tyr Pro Thr Thr Ser Phe Tyr
260 265 270
Asp Asp Leu Asp Glu Glu Glu Glu Glu Glu Glu Asp Asp Lys Asp Ala
275 280 285
Val Gly Gly Gly Asp Leu Glu Asp Glu Asn Glu Leu Leu Val Pro Thr
290 295 300
Gly Lys Pro Gly Leu Gly Pro Gly Thr Gly Gln Pro Thr Ser Arg Trp
305 310 315 320
His Ala Val Pro Pro Gln His Thr Leu Gly Ser Val Pro Gly Ser Ser
325 330 335
Ile Ala Leu Arg Pro Arg Pro Gly Glu Pro Gly Arg Asp Leu Ala Ser
340 345 350
Ser Glu Asn Gly Thr Glu Cys Arg Ser Gly Phe Val Arg His Asn Gly
355 360 365
Ser Cys Arg Ser Val Cys Asp Leu Phe Pro Ser Tyr Cys His Asn Gly
370 375 380
Gly Gln Cys Tyr Leu Val Glu Asn Ile Gly Ala Phe Cys Arg Cys Asn
385 390 395 400
Thr Gln Asp Tyr Ile Trp His Lys Gly Met Arg Cys Glu Ser Ile Ile
405 410 415
Thr Asp Phe Gln Val Met Cys Val Ala Val Gly Ser Ala Ala Leu Val
420 425 430
Leu Leu Leu Leu Phe Met Met Thr Val Phe Phe Ala Lys Lys Leu Tyr
435 440 445
Leu Leu Lys Thr Glu Asn Thr Lys Leu Arg Arg Thr Asn Lys Phe Arg
450 455 460
Thr Pro Ser Glu Leu His Asn Asp Asn Phe Ser Leu Ser Thr Ile Ala
465 470 475 480
Glu Gly Ser His Pro Asn Val Arg Lys Leu Cys Asn Thr Pro Arg Thr
485 490 495
Ser Ser Pro His Ala Arg Ala Leu Ala His Tyr Asp Asn Val Ile Cys
500 505 510
Gln Asp Asp Pro Ser Ala Pro His Lys Ile Gln Glu Val Leu Lys Ser
515 520 525
Cys Leu Lys Glu Glu Glu Ser Phe Asn Ile Gln Asn Ser Met Ser Pro
530 535 540
Lys Leu Glu Gly Gly Lys Gly Asp Gln Ala Asp Leu Asp Val Asn Cys
545 550 555 560
Leu Gln Asn Asn Leu Thr
565
<210> 9
<211> 2099
<212> ДНК
<213> Homo sapiens
<220>
<221> Кодирующая последовательность
<222> (177)..(1610)
<223>
<400> 9
ggggaggcgc ggcgcgccgg ggacagcggc ggacggcggc ggcggcggca tgcggctcct 60
cgcgctgccc atcgtgggct gaggcggccg cagaaccggc gggaggcgcg gcggccgggc 120
gagccgaggg cgcagccagc cgggcggacc gcggacagcg gtcggggcgc cgcgcc atg 179
Met
1
ggg cga gcc ggg ggc ggg ggc ccg ggc cgg ggg ccg ccg cca ctg ctg 227
Gly Arg Ala Gly Gly Gly Gly Pro Gly Arg Gly Pro Pro Pro Leu Leu
5 10 15
ctg ttt ctg ggg gcc gcg ctg gtc ctg gcc tct ggg gcc gtg ccg gcg 275
Leu Phe Leu Gly Ala Ala Leu Val Leu Ala Ser Gly Ala Val Pro Ala
20 25 30
cgt gag gcg ggc agc gcg gtt gag gcc gaa gag ctg gtg aag ggc agc 323
Arg Glu Ala Gly Ser Ala Val Glu Ala Glu Glu Leu Val Lys Gly Ser
35 40 45
ccg gcg tgg gag ccg cct gcc aac gac acg cgg gaa gaa gcc ggc cca 371
Pro Ala Trp Glu Pro Pro Ala Asn Asp Thr Arg Glu Glu Ala Gly Pro
50 55 60 65
cca gcg gct ggg gaa gat gag gcg tcg tgg acg gcg ccc ggt ggc gag 419
Pro Ala Ala Gly Glu Asp Glu Ala Ser Trp Thr Ala Pro Gly Gly Glu
70 75 80
ctg gcc ggg cca gaa gag gtg ctg cag gag tcg gct gcg gtg acc ggc 467
Leu Ala Gly Pro Glu Glu Val Leu Gln Glu Ser Ala Ala Val Thr Gly
85 90 95
acc gcc tgg ctg gaa gct gac agc cca ggc ctg gga gga gtg acc gca 515
Thr Ala Trp Leu Glu Ala Asp Ser Pro Gly Leu Gly Gly Val Thr Ala
100 105 110
gag gcg ggc agc ggc gat gcc cag gcc ctt cca gct acg ctc cag gct 563
Glu Ala Gly Ser Gly Asp Ala Gln Ala Leu Pro Ala Thr Leu Gln Ala
115 120 125
ccc cac gag gtc ctc ggg cag tca atc atg ccc cct gcc att cct gag 611
Pro His Glu Val Leu Gly Gln Ser Ile Met Pro Pro Ala Ile Pro Glu
130 135 140 145
gct aca gag gcc agc ggg cca ccc tcc ccc acc ccc ggc gac aag ctg 659
Ala Thr Glu Ala Ser Gly Pro Pro Ser Pro Thr Pro Gly Asp Lys Leu
150 155 160
agc cca gct tct gaa ctc ccc aag gag agc ccc ttg gag gtt tgg ctg 707
Ser Pro Ala Ser Glu Leu Pro Lys Glu Ser Pro Leu Glu Val Trp Leu
165 170 175
aac ctg ggg ggc agc aca ccc gac cct caa ggg cca gag ctg act tac 755
Asn Leu Gly Gly Ser Thr Pro Asp Pro Gln Gly Pro Glu Leu Thr Tyr
180 185 190
cca ttt cag ggc acc ctg gag ccc caa ccg gca tca gat atc att gac 803
Pro Phe Gln Gly Thr Leu Glu Pro Gln Pro Ala Ser Asp Ile Ile Asp
195 200 205
atc gac tac ttc gaa gga ctg gat ggt gag ggt cgt ggc gca gat ctg 851
Ile Asp Tyr Phe Glu Gly Leu Asp Gly Glu Gly Arg Gly Ala Asp Leu
210 215 220 225
ggg agc ttc cca ggg tca cca gga acc tca gag aac cac cct gat act 899
Gly Ser Phe Pro Gly Ser Pro Gly Thr Ser Glu Asn His Pro Asp Thr
230 235 240
gag gga gag acc cct tcc tgg agc ctg ctt gac tta tac gat gat ttc 947
Glu Gly Glu Thr Pro Ser Trp Ser Leu Leu Asp Leu Tyr Asp Asp Phe
245 250 255
acc ccc ttc gat gaa tct gat ttc tac ccc acc aca tcc ttt tat gat 995
Thr Pro Phe Asp Glu Ser Asp Phe Tyr Pro Thr Thr Ser Phe Tyr Asp
260 265 270
gac ttg gat gaa gag gag gag gaa gag gag gat gac aaa gat gca gta 1043
Asp Leu Asp Glu Glu Glu Glu Glu Glu Glu Asp Asp Lys Asp Ala Val
275 280 285
gga ggt gga gac cta gaa gat gaa aat gag ctt cta gtg ccc act ggg 1091
Gly Gly Gly Asp Leu Glu Asp Glu Asn Glu Leu Leu Val Pro Thr Gly
290 295 300 305
aag cct ggt ctg ggg ccc ggg aca ggc cag ccc acc agt cgg tgg cat 1139
Lys Pro Gly Leu Gly Pro Gly Thr Gly Gln Pro Thr Ser Arg Trp His
310 315 320
gct gtc cct cca cag cac act ctg ggg tcg gtc ccc ggc agc agc atc 1187
Ala Val Pro Pro Gln His Thr Leu Gly Ser Val Pro Gly Ser Ser Ile
325 330 335
gcc ctc agg ccc cgc cca gga gag cca ggc agg gac ttg gcc tcc agt 1235
Ala Leu Arg Pro Arg Pro Gly Glu Pro Gly Arg Asp Leu Ala Ser Ser
340 345 350
gaa aat ggc act gag tgc cgc agt ggc ttt gtg cgg cat aac ggc tcc 1283
Glu Asn Gly Thr Glu Cys Arg Ser Gly Phe Val Arg His Asn Gly Ser
355 360 365
tgc cgg tca gtg tgc gac ctc ttc cca agt tac tgt cac aat ggc ggc 1331
Cys Arg Ser Val Cys Asp Leu Phe Pro Ser Tyr Cys His Asn Gly Gly
370 375 380 385
cag tgc tac ctg gtg gag aac ata ggg gcc ttc tgc agg tgc aac acg 1379
Gln Cys Tyr Leu Val Glu Asn Ile Gly Ala Phe Cys Arg Cys Asn Thr
390 395 400
cag gac tac atc tgg cac aag ggg atg cgc tgc gag tcc atc atc acc 1427
Gln Asp Tyr Ile Trp His Lys Gly Met Arg Cys Glu Ser Ile Ile Thr
405 410 415
gac ttc cag gtg atg tgc gtg gcc gtg ggc tcg gct gcc ctc gtc ctg 1475
Asp Phe Gln Val Met Cys Val Ala Val Gly Ser Ala Ala Leu Val Leu
420 425 430
ctc ctg ctc ttc atg atg acg gtg ttc ttt gcc aag aag ctc tac ctg 1523
Leu Leu Leu Phe Met Met Thr Val Phe Phe Ala Lys Lys Leu Tyr Leu
435 440 445
ctc aag acg gag aat acc aag ctg cgt agg acc aag atg atc cta gtg 1571
Leu Lys Thr Glu Asn Thr Lys Leu Arg Arg Thr Lys Met Ile Leu Val
450 455 460 465
ctc ccc aca aaa tcc agg agg ttc tca agt cct gcc tga aagaggagga 1620
Leu Pro Thr Lys Ser Arg Arg Phe Ser Ser Pro Ala
470 475
gtcatttaac atccagaact ccatgtcgcc caaacttgag ggtggcaaag gtgaccaggc 1680
tgacttggat gtgaactgtc ttcagaataa tttaacctaa agcagagcaa gaagagagga 1740
agcgggggta gtgggtgggg ggtaggggaa gaaacattat ctcctcttgt acagagtcta 1800
tttcttgtaa ccatttgtta aactcttttc tttttctgat ctcatggcat gcttttatgt 1860
attttgtaca ggaggcaaaa aaatacttaa aataagcaaa gaaactgaac agaattgcat 1920
acattgggtt gttttttctg tgctgtctgt acattgcttc tgctgctgtg atttctaaac 1980
ctgtgctgtt attcaactga cttttttttg tactttgacc cacgtttttt tgaaatacca 2040
gtaaaaaaca aagttcttga aataaaactt tttaaaaagt taaaaaaaaa aaaaaaaaa 2099
<210> 10
<211> 477
<212> Белок
<213> Homo sapiens
<400> 10
Met Gly Arg Ala Gly Gly Gly Gly Pro Gly Arg Gly Pro Pro Pro Leu
1 5 10 15
Leu Leu Phe Leu Gly Ala Ala Leu Val Leu Ala Ser Gly Ala Val Pro
20 25 30
Ala Arg Glu Ala Gly Ser Ala Val Glu Ala Glu Glu Leu Val Lys Gly
35 40 45
Ser Pro Ala Trp Glu Pro Pro Ala Asn Asp Thr Arg Glu Glu Ala Gly
50 55 60
Pro Pro Ala Ala Gly Glu Asp Glu Ala Ser Trp Thr Ala Pro Gly Gly
65 70 75 80
Glu Leu Ala Gly Pro Glu Glu Val Leu Gln Glu Ser Ala Ala Val Thr
85 90 95
Gly Thr Ala Trp Leu Glu Ala Asp Ser Pro Gly Leu Gly Gly Val Thr
100 105 110
Ala Glu Ala Gly Ser Gly Asp Ala Gln Ala Leu Pro Ala Thr Leu Gln
115 120 125
Ala Pro His Glu Val Leu Gly Gln Ser Ile Met Pro Pro Ala Ile Pro
130 135 140
Glu Ala Thr Glu Ala Ser Gly Pro Pro Ser Pro Thr Pro Gly Asp Lys
145 150 155 160
Leu Ser Pro Ala Ser Glu Leu Pro Lys Glu Ser Pro Leu Glu Val Trp
165 170 175
Leu Asn Leu Gly Gly Ser Thr Pro Asp Pro Gln Gly Pro Glu Leu Thr
180 185 190
Tyr Pro Phe Gln Gly Thr Leu Glu Pro Gln Pro Ala Ser Asp Ile Ile
195 200 205
Asp Ile Asp Tyr Phe Glu Gly Leu Asp Gly Glu Gly Arg Gly Ala Asp
210 215 220
Leu Gly Ser Phe Pro Gly Ser Pro Gly Thr Ser Glu Asn His Pro Asp
225 230 235 240
Thr Glu Gly Glu Thr Pro Ser Trp Ser Leu Leu Asp Leu Tyr Asp Asp
245 250 255
Phe Thr Pro Phe Asp Glu Ser Asp Phe Tyr Pro Thr Thr Ser Phe Tyr
260 265 270
Asp Asp Leu Asp Glu Glu Glu Glu Glu Glu Glu Asp Asp Lys Asp Ala
275 280 285
Val Gly Gly Gly Asp Leu Glu Asp Glu Asn Glu Leu Leu Val Pro Thr
290 295 300
Gly Lys Pro Gly Leu Gly Pro Gly Thr Gly Gln Pro Thr Ser Arg Trp
305 310 315 320
His Ala Val Pro Pro Gln His Thr Leu Gly Ser Val Pro Gly Ser Ser
325 330 335
Ile Ala Leu Arg Pro Arg Pro Gly Glu Pro Gly Arg Asp Leu Ala Ser
340 345 350
Ser Glu Asn Gly Thr Glu Cys Arg Ser Gly Phe Val Arg His Asn Gly
355 360 365
Ser Cys Arg Ser Val Cys Asp Leu Phe Pro Ser Tyr Cys His Asn Gly
370 375 380
Gly Gln Cys Tyr Leu Val Glu Asn Ile Gly Ala Phe Cys Arg Cys Asn
385 390 395 400
Thr Gln Asp Tyr Ile Trp His Lys Gly Met Arg Cys Glu Ser Ile Ile
405 410 415
Thr Asp Phe Gln Val Met Cys Val Ala Val Gly Ser Ala Ala Leu Val
420 425 430
Leu Leu Leu Leu Phe Met Met Thr Val Phe Phe Ala Lys Lys Leu Tyr
435 440 445
Leu Leu Lys Thr Glu Asn Thr Lys Leu Arg Arg Thr Lys Met Ile Leu
450 455 460
Val Leu Pro Thr Lys Ser Arg Arg Phe Ser Ser Pro Ala
465 470 475
<210> 11
<211> 2334
<212> ДНК
<213> Homo sapiens
<220>
<221> Кодирующая последовательность
<222> (669)..(1955)
<223>
<400> 11
agtccagtga ggaataactg tagataagat gagatgatgg gcattaagtg taactgctga 60
gaagccttca tagagggaaa ggtgataaga actgagtttt tctggcatga ggagtttacc 120
aggcaggaga ggtggagacg ctaggctagg caaataggct gcagccatag ttctgctgag 180
aaacaggttt tgaaccaagg caactccatc tggagatatg tatcagaaag attaggagat 240
agaatctcca tcttggtcac ttatttggtc ttctaagaca gcaatgggac cgtctcttaa 300
atactggagt tgtcctatct ccttgggctg ataccctgaa ctgtcatttg gcgcgtgagg 360
cgggcagcgc ggttgaggcc gaagagctgg tgaagggcag cccggcgtgg gagccgcctg 420
ccaacgacac gcgggaagaa gccggcccac cagcggctgg ggaagatgag gcgtcgtgga 480
cggcgcccgg tggcgagctg gccgggccag aagaggtgct gcaggagtcg gctgcggtga 540
ccggcaccgc ctggctggaa gctgacagcc caggcctggg aggagtgacc gcagaggcgg 600
gcagcggcga tgcccaggcc cttccagcta cgctccaggc tccccacgag gtcctcgggc 660
agtcaatc atg ccc cct gcc att cct gag gct aca gag gcc agc ggg cca 710
Met Pro Pro Ala Ile Pro Glu Ala Thr Glu Ala Ser Gly Pro
1 5 10
ccc tcc ccc acc ccc ggc gac aag ctg agc cca gct tct gaa ctc ccc 758
Pro Ser Pro Thr Pro Gly Asp Lys Leu Ser Pro Ala Ser Glu Leu Pro
15 20 25 30
aag gag agc ccc ttg gag gtt tgg ctg aac ctg ggg ggc agc aca ccc 806
Lys Glu Ser Pro Leu Glu Val Trp Leu Asn Leu Gly Gly Ser Thr Pro
35 40 45
gac cct caa ggg cca gag ctg act tac cca ttt cag ggc acc ctg gag 854
Asp Pro Gln Gly Pro Glu Leu Thr Tyr Pro Phe Gln Gly Thr Leu Glu
50 55 60
ccc caa ccg gca tca gat atc att gac atc gac tac ttc gaa gga ctg 902
Pro Gln Pro Ala Ser Asp Ile Ile Asp Ile Asp Tyr Phe Glu Gly Leu
65 70 75
gat ggt gag ggt cgt ggc gca gat ctg ggg agc ttc cca ggg tca cca 950
Asp Gly Glu Gly Arg Gly Ala Asp Leu Gly Ser Phe Pro Gly Ser Pro
80 85 90
gga acc tca gag aac cac cct gat act gag gga gag acc cct tcc tgg 998
Gly Thr Ser Glu Asn His Pro Asp Thr Glu Gly Glu Thr Pro Ser Trp
95 100 105 110
agc ctg ctt gac tta tac gat gat ttc acc ccc ttc gat gaa tct gat 1046
Ser Leu Leu Asp Leu Tyr Asp Asp Phe Thr Pro Phe Asp Glu Ser Asp
115 120 125
ttc tac ccc acc aca tcc ttt tat gat gac ttg gat gaa gag gag gag 1094
Phe Tyr Pro Thr Thr Ser Phe Tyr Asp Asp Leu Asp Glu Glu Glu Glu
130 135 140
gaa gag gag gat gac aaa gat gca gta gga ggt gga gac cta gaa gat 1142
Glu Glu Glu Asp Asp Lys Asp Ala Val Gly Gly Gly Asp Leu Glu Asp
145 150 155
gaa aat gag ctt cta gtg ccc act ggg aag cct ggt ctg ggg ccc ggg 1190
Glu Asn Glu Leu Leu Val Pro Thr Gly Lys Pro Gly Leu Gly Pro Gly
160 165 170
aca ggc cag ccc acc agt cgg tgg cat gct gtc cct cca cag cac act 1238
Thr Gly Gln Pro Thr Ser Arg Trp His Ala Val Pro Pro Gln His Thr
175 180 185 190
ctg ggg tcg gtc ccc ggc agc agc atc gcc ctc agg ccc cgc cca gga 1286
Leu Gly Ser Val Pro Gly Ser Ser Ile Ala Leu Arg Pro Arg Pro Gly
195 200 205
gag cca ggc agg gac ttg gcc tcc agt gaa aat ggc act gag tgc cgc 1334
Glu Pro Gly Arg Asp Leu Ala Ser Ser Glu Asn Gly Thr Glu Cys Arg
210 215 220
agt ggc ttt gtg cgg cat aac ggc tcc tgc cgg tca gtg tgc gac ctc 1382
Ser Gly Phe Val Arg His Asn Gly Ser Cys Arg Ser Val Cys Asp Leu
225 230 235
ttc cca agt tac tgt cac aat ggc ggc cag tgc tac ctg gtg gag aac 1430
Phe Pro Ser Tyr Cys His Asn Gly Gly Gln Cys Tyr Leu Val Glu Asn
240 245 250
ata ggg gcc ttc tgc agg tgc aac acg cag gac tac atc tgg cac aag 1478
Ile Gly Ala Phe Cys Arg Cys Asn Thr Gln Asp Tyr Ile Trp His Lys
255 260 265 270
ggg atg cgc tgc gag tcc atc atc acc gac ttc cag gtg atg tgc gtg 1526
Gly Met Arg Cys Glu Ser Ile Ile Thr Asp Phe Gln Val Met Cys Val
275 280 285
gcc gtg ggc tcg gct gcc ctc gtc ctg ctc ctg ctc ttc atg atg acg 1574
Ala Val Gly Ser Ala Ala Leu Val Leu Leu Leu Leu Phe Met Met Thr
290 295 300
gtg ttc ttt gcc aag aag ctc tac ctg ctc aag acg gag aat acc aag 1622
Val Phe Phe Ala Lys Lys Leu Tyr Leu Leu Lys Thr Glu Asn Thr Lys
305 310 315
ctg cgt agg acc aac aaa ttc cgg acc cca tct gag ctc cac aat gat 1670
Leu Arg Arg Thr Asn Lys Phe Arg Thr Pro Ser Glu Leu His Asn Asp
320 325 330
aac ttc tcc ctc tcc acc att gcc gag ggc tct cac cca aat gta agg 1718
Asn Phe Ser Leu Ser Thr Ile Ala Glu Gly Ser His Pro Asn Val Arg
335 340 345 350
aaa ctt tgc aac act ccc cgt acc tcc tcc ccc cat gcc cgt gcc ttg 1766
Lys Leu Cys Asn Thr Pro Arg Thr Ser Ser Pro His Ala Arg Ala Leu
355 360 365
gct cac tat gat aac gtt atc tgt cag gat gat cct agt gct ccc cac 1814
Ala His Tyr Asp Asn Val Ile Cys Gln Asp Asp Pro Ser Ala Pro His
370 375 380
aaa atc cag gag gtt ctc aag tcc tgc ctg aaa gag gag gag tca ttt 1862
Lys Ile Gln Glu Val Leu Lys Ser Cys Leu Lys Glu Glu Glu Ser Phe
385 390 395
aac atc cag aac tcc atg tcg ccc aaa ctt gag ggt ggc aaa ggt gac 1910
Asn Ile Gln Asn Ser Met Ser Pro Lys Leu Glu Gly Gly Lys Gly Asp
400 405 410
cag gct gac ttg gat gtg aac tgt ctt cag aat aat tta acc taa 1955
Gln Ala Asp Leu Asp Val Asn Cys Leu Gln Asn Asn Leu Thr
415 420 425
agcagagcaa gaagagagga agcgggggta gtgggtgggg ggtaggggaa gaaacattat 2015
ctcctcttgt acagagtcta tttcttgtaa ccatttgtta aactcttttc tttttctgat 2075
ctcatggcat gcttttatgt attttgtaca ggaggcaaaa aaatacttaa aataagcaaa 2135
gaaactgaac agaattgcat acattgggtt gttttttctg tgctgtctgt acattgcttc 2195
tgctgctgtg atttctaaac ctgtgctgtt attcaactga cttttttttg tactttgacc 2255
cacgtttttt tgaaatacca gtaaaaaaca aagttcttga aataaaactt tttaaaaagt 2315
taaaaaaaaa aaaaaaaaa 2334
<210> 12
<211> 428
<212> Белок
<213> Homo sapiens
<400> 12
Met Pro Pro Ala Ile Pro Glu Ala Thr Glu Ala Ser Gly Pro Pro Ser
1 5 10 15
Pro Thr Pro Gly Asp Lys Leu Ser Pro Ala Ser Glu Leu Pro Lys Glu
20 25 30
Ser Pro Leu Glu Val Trp Leu Asn Leu Gly Gly Ser Thr Pro Asp Pro
35 40 45
Gln Gly Pro Glu Leu Thr Tyr Pro Phe Gln Gly Thr Leu Glu Pro Gln
50 55 60
Pro Ala Ser Asp Ile Ile Asp Ile Asp Tyr Phe Glu Gly Leu Asp Gly
65 70 75 80
Glu Gly Arg Gly Ala Asp Leu Gly Ser Phe Pro Gly Ser Pro Gly Thr
85 90 95
Ser Glu Asn His Pro Asp Thr Glu Gly Glu Thr Pro Ser Trp Ser Leu
100 105 110
Leu Asp Leu Tyr Asp Asp Phe Thr Pro Phe Asp Glu Ser Asp Phe Tyr
115 120 125
Pro Thr Thr Ser Phe Tyr Asp Asp Leu Asp Glu Glu Glu Glu Glu Glu
130 135 140
Glu Asp Asp Lys Asp Ala Val Gly Gly Gly Asp Leu Glu Asp Glu Asn
145 150 155 160
Glu Leu Leu Val Pro Thr Gly Lys Pro Gly Leu Gly Pro Gly Thr Gly
165 170 175
Gln Pro Thr Ser Arg Trp His Ala Val Pro Pro Gln His Thr Leu Gly
180 185 190
Ser Val Pro Gly Ser Ser Ile Ala Leu Arg Pro Arg Pro Gly Glu Pro
195 200 205
Gly Arg Asp Leu Ala Ser Ser Glu Asn Gly Thr Glu Cys Arg Ser Gly
210 215 220
Phe Val Arg His Asn Gly Ser Cys Arg Ser Val Cys Asp Leu Phe Pro
225 230 235 240
Ser Tyr Cys His Asn Gly Gly Gln Cys Tyr Leu Val Glu Asn Ile Gly
245 250 255
Ala Phe Cys Arg Cys Asn Thr Gln Asp Tyr Ile Trp His Lys Gly Met
260 265 270
Arg Cys Glu Ser Ile Ile Thr Asp Phe Gln Val Met Cys Val Ala Val
275 280 285
Gly Ser Ala Ala Leu Val Leu Leu Leu Leu Phe Met Met Thr Val Phe
290 295 300
Phe Ala Lys Lys Leu Tyr Leu Leu Lys Thr Glu Asn Thr Lys Leu Arg
305 310 315 320
Arg Thr Asn Lys Phe Arg Thr Pro Ser Glu Leu His Asn Asp Asn Phe
325 330 335
Ser Leu Ser Thr Ile Ala Glu Gly Ser His Pro Asn Val Arg Lys Leu
340 345 350
Cys Asn Thr Pro Arg Thr Ser Ser Pro His Ala Arg Ala Leu Ala His
355 360 365
Tyr Asp Asn Val Ile Cys Gln Asp Asp Pro Ser Ala Pro His Lys Ile
370 375 380
Gln Glu Val Leu Lys Ser Cys Leu Lys Glu Glu Glu Ser Phe Asn Ile
385 390 395 400
Gln Asn Ser Met Ser Pro Lys Leu Glu Gly Gly Lys Gly Asp Gln Ala
405 410 415
Asp Leu Asp Val Asn Cys Leu Gln Asn Asn Leu Thr
420 425
<210> 13
<211> 2592
<212> ДНК
<213> Felis catus
<220>
<221> Кодирующая последовательность
<222> (1)..(2217)
<223>
<400> 13
atg ggc ttt ggc tat gga cgg att agg tct gat ccg att aga gcc cgg 48
Met Gly Phe Gly Tyr Gly Arg Ile Arg Ser Asp Pro Ile Arg Ala Arg
1 5 10 15
gcc gtc ggc ttc gcc ccc ttg cct ggc gct gcc agc gcc cgc ccc gcc 96
Ala Val Gly Phe Ala Pro Leu Pro Gly Ala Ala Ser Ala Arg Pro Ala
20 25 30
ctc gtg gac acc cgc aga cct ccc cga gac cct tcc cct ccc cga aca 144
Leu Val Asp Thr Arg Arg Pro Pro Arg Asp Pro Ser Pro Pro Arg Thr
35 40 45
cgg cat tgg cgc aga aat ctg aga ggt ccg tgc acc ccg ggc tcg gct 192
Arg His Trp Arg Arg Asn Leu Arg Gly Pro Cys Thr Pro Gly Ser Ala
50 55 60
cat ttc cac agc tcc agc tcg ggc ttc cgc ccc ctg cgg ccc ttc cga 240
His Phe His Ser Ser Ser Ser Gly Phe Arg Pro Leu Arg Pro Phe Arg
65 70 75 80
gcc ccg ccc caa gct tgc ggg tgt ctg ggc ccc cgc ctc cgt gct cgc 288
Ala Pro Pro Gln Ala Cys Gly Cys Leu Gly Pro Arg Leu Arg Ala Arg
85 90 95
cgc gtg gcg ggt ggg ttc ctc gca ggt ggg ggg ccc gtg cca gct ctc 336
Arg Val Ala Gly Gly Phe Leu Ala Gly Gly Gly Pro Val Pro Ala Leu
100 105 110
cac ggg gag ggc ggg ccc cgc ccc aca ggt ctc ccg ccc gtg cac ctg 384
His Gly Glu Gly Gly Pro Arg Pro Thr Gly Leu Pro Pro Val His Leu
115 120 125
tcg gct aac gcc acg cac ggc gct gtg ctc cgc acc cgc gct act cca 432
Ser Ala Asn Ala Thr His Gly Ala Val Leu Arg Thr Arg Ala Thr Pro
130 135 140
cgt ccg ttt gtc tcg gcg tcc cga gcc ggg ggt acc gac tgc gac cag 480
Arg Pro Phe Val Ser Ala Ser Arg Ala Gly Gly Thr Asp Cys Asp Gln
145 150 155 160
gac ccc cgc ggc cct cgc gcc cca ccc tgg gcc agg gtc ccg ctg gcc 528
Asp Pro Arg Gly Pro Arg Ala Pro Pro Trp Ala Arg Val Pro Leu Ala
165 170 175
tcg ggt aca ggc gga gtt agc gag ctg tgg caa ggg ggc ggg gca gct 576
Ser Gly Thr Gly Gly Val Ser Glu Leu Trp Gln Gly Gly Gly Ala Ala
180 185 190
cct tgc ccg cga ccg ggg cgg ggg aag ggg cgc gcg aag agg tgg gat 624
Pro Cys Pro Arg Pro Gly Arg Gly Lys Gly Arg Ala Lys Arg Trp Asp
195 200 205
act tgg ggg agg ccg agg ggt tgg ggg cgg ccc cgg ccc ggg tgt ccg 672
Thr Trp Gly Arg Pro Arg Gly Trp Gly Arg Pro Arg Pro Gly Cys Pro
210 215 220
gac aga gcc cgt gag gct ggc agc gcc gtc gag gcc cac gag cag gtg 720
Asp Arg Ala Arg Glu Ala Gly Ser Ala Val Glu Ala His Glu Gln Val
225 230 235 240
aag agc atc ctg gcg agg gag ccg act gcc aac gaa acg agg gag aag 768
Lys Ser Ile Leu Ala Arg Glu Pro Thr Ala Asn Glu Thr Arg Glu Lys
245 250 255
gcc ggc cca cca gca gct gag gaa gac gag acc tcg tgg acc gca cct 816
Ala Gly Pro Pro Ala Ala Glu Glu Asp Glu Thr Ser Trp Thr Ala Pro
260 265 270
ggc ggt gag cag gcc atg atg ggg cct agt gtc ggg cca gag gag gtg 864
Gly Gly Glu Gln Ala Met Met Gly Pro Ser Val Gly Pro Glu Glu Val
275 280 285
ctg gag gcg tcg gca gcg gtg acc ggc gca ccc tgg ctg gag gct gac 912
Leu Glu Ala Ser Ala Ala Val Thr Gly Ala Pro Trp Leu Glu Ala Asp
290 295 300
agc cct ggc ctg ggt gga gtg acc gca gag gcc ggc agc ggc gac acc 960
Ser Pro Gly Leu Gly Gly Val Thr Ala Glu Ala Gly Ser Gly Asp Thr
305 310 315 320
cag gcc ctt cca gct acg ctc ccg gct ccc aag gag gcc ctg gga cag 1008
Gln Ala Leu Pro Ala Thr Leu Pro Ala Pro Lys Glu Ala Leu Gly Gln
325 330 335
tca tcg atg gcc ccc tcc atc ccc aag gct aca gag gcc agc aga cca 1056
Ser Ser Met Ala Pro Ser Ile Pro Lys Ala Thr Glu Ala Ser Arg Pro
340 345 350
ccc tcc ccc aca cct ggc gac atg ctg agc ccc ggc cca gaa cac ccc 1104
Pro Ser Pro Thr Pro Gly Asp Met Leu Ser Pro Gly Pro Glu His Pro
355 360 365
aag gag agt ccc ttg gag gtt tgg ttg aac ctg gga ggc agc aca cat 1152
Lys Glu Ser Pro Leu Glu Val Trp Leu Asn Leu Gly Gly Ser Thr His
370 375 380
gac cct cat ggg cca gag ccc aca ttc ccc ttt cag ggc aca atg gag 1200
Asp Pro His Gly Pro Glu Pro Thr Phe Pro Phe Gln Gly Thr Met Glu
385 390 395 400
ccc cag cca gtg tca gat ata att gac atc gac tac ttc gaa gga ttg 1248
Pro Gln Pro Val Ser Asp Ile Ile Asp Ile Asp Tyr Phe Glu Gly Leu
405 410 415
gat ggt gag ggc cgt ggt gcc gac ctg gag agc ttc cca ggg tcg cca 1296
Asp Gly Glu Gly Arg Gly Ala Asp Leu Glu Ser Phe Pro Gly Ser Pro
420 425 430
gga acc tca gag cac cac cct gat act ggg gga gag acc cct tcc tgg 1344
Gly Thr Ser Glu His His Pro Asp Thr Gly Gly Glu Thr Pro Ser Trp
435 440 445
agc ctg ctt gac tta tac gat gac ttc acc ccc ttt gat gaa tct gac 1392
Ser Leu Leu Asp Leu Tyr Asp Asp Phe Thr Pro Phe Asp Glu Ser Asp
450 455 460
ttc tac ccc acc aca tcc ttc tat gat gac ctt gat gaa gag gag gag 1440
Phe Tyr Pro Thr Thr Ser Phe Tyr Asp Asp Leu Asp Glu Glu Glu Glu
465 470 475 480
gaa gag gat gac aag gat gca gcg gga ggt gaa gac ctg gaa gat gaa 1488
Glu Glu Asp Asp Lys Asp Ala Ala Gly Gly Glu Asp Leu Glu Asp Glu
485 490 495
agt gac ctt ctg gtg cct acc gag aag cct ggt ctg ggg ccc ggg act 1536
Ser Asp Leu Leu Val Pro Thr Glu Lys Pro Gly Leu Gly Pro Gly Thr
500 505 510
ggc cag cct acc agt cgg tgg cat gct gtg ccc cca cag cat act ctg 1584
Gly Gln Pro Thr Ser Arg Trp His Ala Val Pro Pro Gln His Thr Leu
515 520 525
ggg atg gtc cct ggc agc agc atc gcc ctc agg ccc cgc cct gga gag 1632
Gly Met Val Pro Gly Ser Ser Ile Ala Leu Arg Pro Arg Pro Gly Glu
530 535 540
cca ggc agg gac ctg acc cca agc gag aat ggc act gag tgc cgc agc 1680
Pro Gly Arg Asp Leu Thr Pro Ser Glu Asn Gly Thr Glu Cys Arg Ser
545 550 555 560
ggc ttt gtg cga cat aac ggc tcc tgc cgg tca gtg tgc gac ctc ttt 1728
Gly Phe Val Arg His Asn Gly Ser Cys Arg Ser Val Cys Asp Leu Phe
565 570 575
cca agt tac tgt cac aat ggc ggc cag tgc tac ctg gtg gag aac ata 1776
Pro Ser Tyr Cys His Asn Gly Gly Gln Cys Tyr Leu Val Glu Asn Ile
580 585 590
ggg gcc ttc tgc agg tgc aac aca cag gac tac atc tgg cac aag ggg 1824
Gly Ala Phe Cys Arg Cys Asn Thr Gln Asp Tyr Ile Trp His Lys Gly
595 600 605
atg cgc tgc gag tcc atc atc acc gac ttc cag gtg atg tgc gtg gcc 1872
Met Arg Cys Glu Ser Ile Ile Thr Asp Phe Gln Val Met Cys Val Ala
610 615 620
gtc ggc tcg gct gcc ctt gta ctg ctc ctg ctc ttc atg atg aca gtg 1920
Val Gly Ser Ala Ala Leu Val Leu Leu Leu Leu Phe Met Met Thr Val
625 630 635 640
ttc ttc gcc aag aag cta tat ctg ctc aag aca gag aat acc aag ctg 1968
Phe Phe Ala Lys Lys Leu Tyr Leu Leu Lys Thr Glu Asn Thr Lys Leu
645 650 655
cgt agg acc aac aaa ttc cgg acc ccg tct gaa ctc cac aac gat aac 2016
Arg Arg Thr Asn Lys Phe Arg Thr Pro Ser Glu Leu His Asn Asp Asn
660 665 670
ttc tcc ctc tcc acc att gcc gaa ggc tct cac cca aac gat gac cct 2064
Phe Ser Leu Ser Thr Ile Ala Glu Gly Ser His Pro Asn Asp Asp Pro
675 680 685
agt gct ccc cac aaa atc cag gaa gct ctc aag tcc tgc ctg aaa gag 2112
Ser Ala Pro His Lys Ile Gln Glu Ala Leu Lys Ser Cys Leu Lys Glu
690 695 700
gag gag tca ttt aac atc cag aac tcc atg tca ccc aaa ctt gag ggt 2160
Glu Glu Ser Phe Asn Ile Gln Asn Ser Met Ser Pro Lys Leu Glu Gly
705 710 715 720
ggc aaa ggt gac cag gct gac ttg gag gtg aac tgt ctt cag aat aac 2208
Gly Lys Gly Asp Gln Ala Asp Leu Glu Val Asn Cys Leu Gln Asn Asn
725 730 735
cta acc taa agcagaacaa gaagagagga aatgggggga gggggggtac 2257
Leu Thr
ggggagaaac atgacctcct cttgtacaga gtctatttct tgtaaccatt tgttaaactc 2317
ttttcttttt ctgatctcat ggcatgcttt gatgtatttt gtacaggagg ggaaacacac 2377
acacacacac acacacacac acacacacac acacacacta agcaaagaac ccagacaaaa 2437
ttgcatacgt tgggttgttt tgtctgtgct gtctgtacat tgcttctgct gctgtgattt 2497
ctaaacctac gctgttattc aactactttt tttttgtact ttgacccacc tttttttgaa 2557
ataagagtaa aaaacaaagt tcttgaaata aaact 2592
<210> 14
<211> 738
<212> Белок
<213> Felis catus
<400> 14
Met Gly Phe Gly Tyr Gly Arg Ile Arg Ser Asp Pro Ile Arg Ala Arg
1 5 10 15
Ala Val Gly Phe Ala Pro Leu Pro Gly Ala Ala Ser Ala Arg Pro Ala
20 25 30
Leu Val Asp Thr Arg Arg Pro Pro Arg Asp Pro Ser Pro Pro Arg Thr
35 40 45
Arg His Trp Arg Arg Asn Leu Arg Gly Pro Cys Thr Pro Gly Ser Ala
50 55 60
His Phe His Ser Ser Ser Ser Gly Phe Arg Pro Leu Arg Pro Phe Arg
65 70 75 80
Ala Pro Pro Gln Ala Cys Gly Cys Leu Gly Pro Arg Leu Arg Ala Arg
85 90 95
Arg Val Ala Gly Gly Phe Leu Ala Gly Gly Gly Pro Val Pro Ala Leu
100 105 110
His Gly Glu Gly Gly Pro Arg Pro Thr Gly Leu Pro Pro Val His Leu
115 120 125
Ser Ala Asn Ala Thr His Gly Ala Val Leu Arg Thr Arg Ala Thr Pro
130 135 140
Arg Pro Phe Val Ser Ala Ser Arg Ala Gly Gly Thr Asp Cys Asp Gln
145 150 155 160
Asp Pro Arg Gly Pro Arg Ala Pro Pro Trp Ala Arg Val Pro Leu Ala
165 170 175
Ser Gly Thr Gly Gly Val Ser Glu Leu Trp Gln Gly Gly Gly Ala Ala
180 185 190
Pro Cys Pro Arg Pro Gly Arg Gly Lys Gly Arg Ala Lys Arg Trp Asp
195 200 205
Thr Trp Gly Arg Pro Arg Gly Trp Gly Arg Pro Arg Pro Gly Cys Pro
210 215 220
Asp Arg Ala Arg Glu Ala Gly Ser Ala Val Glu Ala His Glu Gln Val
225 230 235 240
Lys Ser Ile Leu Ala Arg Glu Pro Thr Ala Asn Glu Thr Arg Glu Lys
245 250 255
Ala Gly Pro Pro Ala Ala Glu Glu Asp Glu Thr Ser Trp Thr Ala Pro
260 265 270
Gly Gly Glu Gln Ala Met Met Gly Pro Ser Val Gly Pro Glu Glu Val
275 280 285
Leu Glu Ala Ser Ala Ala Val Thr Gly Ala Pro Trp Leu Glu Ala Asp
290 295 300
Ser Pro Gly Leu Gly Gly Val Thr Ala Glu Ala Gly Ser Gly Asp Thr
305 310 315 320
Gln Ala Leu Pro Ala Thr Leu Pro Ala Pro Lys Glu Ala Leu Gly Gln
325 330 335
Ser Ser Met Ala Pro Ser Ile Pro Lys Ala Thr Glu Ala Ser Arg Pro
340 345 350
Pro Ser Pro Thr Pro Gly Asp Met Leu Ser Pro Gly Pro Glu His Pro
355 360 365
Lys Glu Ser Pro Leu Glu Val Trp Leu Asn Leu Gly Gly Ser Thr His
370 375 380
Asp Pro His Gly Pro Glu Pro Thr Phe Pro Phe Gln Gly Thr Met Glu
385 390 395 400
Pro Gln Pro Val Ser Asp Ile Ile Asp Ile Asp Tyr Phe Glu Gly Leu
405 410 415
Asp Gly Glu Gly Arg Gly Ala Asp Leu Glu Ser Phe Pro Gly Ser Pro
420 425 430
Gly Thr Ser Glu His His Pro Asp Thr Gly Gly Glu Thr Pro Ser Trp
435 440 445
Ser Leu Leu Asp Leu Tyr Asp Asp Phe Thr Pro Phe Asp Glu Ser Asp
450 455 460
Phe Tyr Pro Thr Thr Ser Phe Tyr Asp Asp Leu Asp Glu Glu Glu Glu
465 470 475 480
Glu Glu Asp Asp Lys Asp Ala Ala Gly Gly Glu Asp Leu Glu Asp Glu
485 490 495
Ser Asp Leu Leu Val Pro Thr Glu Lys Pro Gly Leu Gly Pro Gly Thr
500 505 510
Gly Gln Pro Thr Ser Arg Trp His Ala Val Pro Pro Gln His Thr Leu
515 520 525
Gly Met Val Pro Gly Ser Ser Ile Ala Leu Arg Pro Arg Pro Gly Glu
530 535 540
Pro Gly Arg Asp Leu Thr Pro Ser Glu Asn Gly Thr Glu Cys Arg Ser
545 550 555 560
Gly Phe Val Arg His Asn Gly Ser Cys Arg Ser Val Cys Asp Leu Phe
565 570 575
Pro Ser Tyr Cys His Asn Gly Gly Gln Cys Tyr Leu Val Glu Asn Ile
580 585 590
Gly Ala Phe Cys Arg Cys Asn Thr Gln Asp Tyr Ile Trp His Lys Gly
595 600 605
Met Arg Cys Glu Ser Ile Ile Thr Asp Phe Gln Val Met Cys Val Ala
610 615 620
Val Gly Ser Ala Ala Leu Val Leu Leu Leu Leu Phe Met Met Thr Val
625 630 635 640
Phe Phe Ala Lys Lys Leu Tyr Leu Leu Lys Thr Glu Asn Thr Lys Leu
645 650 655
Arg Arg Thr Asn Lys Phe Arg Thr Pro Ser Glu Leu His Asn Asp Asn
660 665 670
Phe Ser Leu Ser Thr Ile Ala Glu Gly Ser His Pro Asn Asp Asp Pro
675 680 685
Ser Ala Pro His Lys Ile Gln Glu Ala Leu Lys Ser Cys Leu Lys Glu
690 695 700
Glu Glu Ser Phe Asn Ile Gln Asn Ser Met Ser Pro Lys Leu Glu Gly
705 710 715 720
Gly Lys Gly Asp Gln Ala Asp Leu Glu Val Asn Cys Leu Gln Asn Asn
725 730 735
Leu Thr
<210> 15
<211> 3881
<212> ДНК
<213> Mus musculus
<220>
<221> Кодирующая последовательность
<222> (1816)..(3516)
<223>
<400> 15
atttctctgc agggagggga gaagatagtt gaggaaggaa atatttcaca ttcgcttgtt 60
tgttctactg atcaaaacag aatttaaaag gatagttacg ggtgggaaat agggtagata 120
gaaggatttg gttggttagt tggttctaga cagggattac agacataagc taccataacc 180
agcaaataca attattttgc agtactagga attgaacctg aagtctcact cattttaggc 240
aagtgctctc cctaactctt aagtgtttta ccataagtag gaaaagactt tattgtgacc 300
atgccctagc atgaaaggac ttacatagga tctcctgtgt ggtaggaact tccaccaaca 360
tccacttttg acaactacca cccaggaatt gaagagagaa ataggtatta tatctgggta 420
agcaagcccc ctcttgactg gaactccaaa ctaggacacg tatgtctggt tgccactctc 480
tagatgtcat acagacccta catagccacc tccgagccag gatataagag cctagctgct 540
ggaattttgt cagccttgag gggactctaa gctgcaacca tacttgtgag agaaaaggag 600
tgttgaacca aggcaactca ctagaagaca ggtatgagac agattaaatg atggtcctcc 660
atcttgttca tatttggtca ctgacagaag actatcaaca ccagcatcgc ccttggacta 720
acccttggac tgagctgaac atgatcattt aggtcgataa taaaacatca atatgtaggc 780
tatttggagg cggggaaagc caggtgacta agctgtagtt ctgcctccct tccaacaggt 840
ctcccagtaa gcagtgaacc tgggttagga acagagacct ctctctccac gttgcatgtg 900
cacctggcag tgatctagac ctcttggttt aaccaggaac ttgggtttgg gtagtactgg 960
tcacctgcgg aaggccgccc tggatttaga aaacgcaagg tggagtccaa atactgggga 1020
ccggataaca cgttgggtgg acctggctgt gcaagattta aactaatggc tttggctgtg 1080
gacagattag gtctgatcca attagagcct aggccgtagt attcgcctcc ttgccaggct 1140
ctgtcgatgt tctccctgct ctccggaaga agacttagag tcggactgga gaaccttctg 1200
gacaccgaat tctggattgg caccggacag tccgtgcccc gggctccgct ctctccacac 1260
tgggggcgcg ggcttccgcc ccctgtggcc cttccgagcc ccgccccgag cctgcgggtg 1320
tcggagcccg cgacgtcccc gcctcccctg cgcgcgcgtg gcgggtgggt tcctcgcagg 1380
tccgcggccc agcgcgctca gcactggaag cgcaagcggg ccccgtgggc tgccgccggt 1440
gcacctgtcg gcttccccca gtggctctgc tgccgcccct gcgcgccgcg cttgcatcgc 1500
tctccacgtc ccgtgccaga ggggtctgac tgtcccctgg cgaggaagac cgagaggggc 1560
cggcgtcaac gcgacgtgct gcggggcggg cggagtgggg gcggcgccga acgcggcagc 1620
ggcaagcggc agcggcggcg cgggaggcgg ggaggcgcgg cgctcggagg acagcggctg 1680
acggcggcat gcggcggctc atgctgccca ccgtgggctg aggcggccgc acacgggcgg 1740
caggcgcagc ggccgggcaa gccgagggcg cagccaagcc gcgcgcaccg cgcacagcgg 1800
caggggctcc gcgca atg ggc cga gct gga ggc ggg ggc ccg gac tgg ggg 1851
Met Gly Arg Ala Gly Gly Gly Gly Pro Asp Trp Gly
1 5 10
ccg ccg cca gtg ctg ctg ctt ctg ggg gtc acg ctg gtg ctc acc gct 1899
Pro Pro Pro Val Leu Leu Leu Leu Gly Val Thr Leu Val Leu Thr Ala
15 20 25
ggg gcc gta ccg gca cgg gaa aca ggc agt gcg atc gag gct gaa gag 1947
Gly Ala Val Pro Ala Arg Glu Thr Gly Ser Ala Ile Glu Ala Glu Glu
30 35 40
ctg gtg agg agc agc ctg gca tgg gag tcg cgt gcc aat gac acg cgg 1995
Leu Val Arg Ser Ser Leu Ala Trp Glu Ser Arg Ala Asn Asp Thr Arg
45 50 55 60
gag gaa gcc ggc ctg cca gca gct ggg gaa gat gag acc tcg tgg aca 2043
Glu Glu Ala Gly Leu Pro Ala Ala Gly Glu Asp Glu Thr Ser Trp Thr
65 70 75
gag cgg ggc agt gag atg gct gcg gtg ggc cct ggg gtc ggg cca gag 2091
Glu Arg Gly Ser Glu Met Ala Ala Val Gly Pro Gly Val Gly Pro Glu
80 85 90
gag gca cta gag gca tcg gct gca gtg act ggc act gcc tgg cta gag 2139
Glu Ala Leu Glu Ala Ser Ala Ala Val Thr Gly Thr Ala Trp Leu Glu
95 100 105
gca gat ggc cca ggc ctg ggt gga gtg act gca gag gct ggc agt ggc 2187
Ala Asp Gly Pro Gly Leu Gly Gly Val Thr Ala Glu Ala Gly Ser Gly
110 115 120
gac gcc cag acc ctt cca gct acg ctc cag gct cct gat gag gcc ctt 2235
Asp Ala Gln Thr Leu Pro Ala Thr Leu Gln Ala Pro Asp Glu Ala Leu
125 130 135 140
ggg tca tct aca atg ccc cct gcc atc cct gag gct act gaa acc agt 2283
Gly Ser Ser Thr Met Pro Pro Ala Ile Pro Glu Ala Thr Glu Thr Ser
145 150 155
gga cct ccc tcc cct gct gtc cat gat aag cct agt gta ggc cct gaa 2331
Gly Pro Pro Ser Pro Ala Val His Asp Lys Pro Ser Val Gly Pro Glu
160 165 170
ctc cct aaa gag atc ccc ttg gag gtt cgg ctg aac ctg gga ggc agc 2379
Leu Pro Lys Glu Ile Pro Leu Glu Val Arg Leu Asn Leu Gly Gly Ser
175 180 185
aca cca gag ccc act ttt ccc ctt cag ggc act ctc gag acc caa cca 2427
Thr Pro Glu Pro Thr Phe Pro Leu Gln Gly Thr Leu Glu Thr Gln Pro
190 195 200
gcc tca gat ata att gac att gat tac ttt gaa gga ttg gat agt gag 2475
Ala Ser Asp Ile Ile Asp Ile Asp Tyr Phe Glu Gly Leu Asp Ser Glu
205 210 215 220
ggt cgt ggt gca gac atg ggc agc ttc ccg ggg tca cca gga acc tca 2523
Gly Arg Gly Ala Asp Met Gly Ser Phe Pro Gly Ser Pro Gly Thr Ser
225 230 235
gaa aat cac cct gat acc gaa gga gag acc cct tcc tgg agc ctg ctt 2571
Glu Asn His Pro Asp Thr Glu Gly Glu Thr Pro Ser Trp Ser Leu Leu
240 245 250
gat ttg tat gat gac ttc acc cct ttt gat gag tct gat ttc tac ccc 2619
Asp Leu Tyr Asp Asp Phe Thr Pro Phe Asp Glu Ser Asp Phe Tyr Pro
255 260 265
acc aca tcc ttc tat gat gat ttg gaa gag gag gaa gaa gag gag gag 2667
Thr Thr Ser Phe Tyr Asp Asp Leu Glu Glu Glu Glu Glu Glu Glu Glu
270 275 280
gat aag gat aca gta gga ggt gga gac ctg gaa gat gaa aac gac ctt 2715
Asp Lys Asp Thr Val Gly Gly Gly Asp Leu Glu Asp Glu Asn Asp Leu
285 290 295 300
ctc ctg ccc tct caa aag cct ggt gtg ggg cct ggg aca gga cag ccc 2763
Leu Leu Pro Ser Gln Lys Pro Gly Val Gly Pro Gly Thr Gly Gln Pro
305 310 315
acc aac cgg tgg cat gct gtt ccc cca cag cat act ctg ggg atg gta 2811
Thr Asn Arg Trp His Ala Val Pro Pro Gln His Thr Leu Gly Met Val
320 325 330
cct ggc agc agc atc tct ctt agg ccc cgc ccc gga gat cca ggc aag 2859
Pro Gly Ser Ser Ile Ser Leu Arg Pro Arg Pro Gly Asp Pro Gly Lys
335 340 345
gac ctg gcc tca gga gaa aat ggc aca gag tgc cga gtt ggc ttc gtc 2907
Asp Leu Ala Ser Gly Glu Asn Gly Thr Glu Cys Arg Val Gly Phe Val
350 355 360
agg cac aat ggc tcc tgc cgg tca gtc tgt gac ctc ttt ccg agt tac 2955
Arg His Asn Gly Ser Cys Arg Ser Val Cys Asp Leu Phe Pro Ser Tyr
365 370 375 380
tgt cac aac ggc ggc cag tgc tac ctg gtg gag aac ata ggg gct ttc 3003
Cys His Asn Gly Gly Gln Cys Tyr Leu Val Glu Asn Ile Gly Ala Phe
385 390 395
tgc agg tgt aac acc cag gac tac atc tgg cac aag ggg atg cgc tgt 3051
Cys Arg Cys Asn Thr Gln Asp Tyr Ile Trp His Lys Gly Met Arg Cys
400 405 410
gag tcc atc atc acg gac ttc cag gtg atg tgc gtg gcc gtt ggc tcg 3099
Glu Ser Ile Ile Thr Asp Phe Gln Val Met Cys Val Ala Val Gly Ser
415 420 425
gct gct ctc gtg ctt ctc ctc ctg ttc atg atg act gtg ttc ttt gcc 3147
Ala Ala Leu Val Leu Leu Leu Leu Phe Met Met Thr Val Phe Phe Ala
430 435 440
aag aag ctc tat ctg ctc aag act gag aat acc aag ctg cgg agg acc 3195
Lys Lys Leu Tyr Leu Leu Lys Thr Glu Asn Thr Lys Leu Arg Arg Thr
445 450 455 460
aat aaa ttc cgg acc cca tct gag ctc cac aac gac aac ttc tcc ctc 3243
Asn Lys Phe Arg Thr Pro Ser Glu Leu His Asn Asp Asn Phe Ser Leu
465 470 475
tcc acc att gcc gag ggc tct cat cca aat gta agg aaa ttt tgc gac 3291
Ser Thr Ile Ala Glu Gly Ser His Pro Asn Val Arg Lys Phe Cys Asp
480 485 490
act ccc cgt gtc tcc tcc ccc cat gcc cgt gcc ttg gct cac tat gat 3339
Thr Pro Arg Val Ser Ser Pro His Ala Arg Ala Leu Ala His Tyr Asp
495 500 505
aac att gtc tgt cag gac gac ccc agc gct ccc cac aaa atc cag gac 3387
Asn Ile Val Cys Gln Asp Asp Pro Ser Ala Pro His Lys Ile Gln Asp
510 515 520
cct ctc aag tcc cgc ctg aag gag gaa gag tcc ttt aac atc cag aac 3435
Pro Leu Lys Ser Arg Leu Lys Glu Glu Glu Ser Phe Asn Ile Gln Asn
525 530 535 540
tcc atg tca ccc aaa ctt gag ggt ggc aaa ggt gac cag gat gac ttg 3483
Ser Met Ser Pro Lys Leu Glu Gly Gly Lys Gly Asp Gln Asp Asp Leu
545 550 555
ggg gtg aac tgt ctg cag aat aac cta acc tga gactgaggaa gaagagagga 3536
Gly Val Asn Cys Leu Gln Asn Asn Leu Thr
560 565
aaggggggtg ggggagggaa ggactgttgt ctcctctcgg gcagagtcgg cttcttgtaa 3596
ccatttgtta agcttttctt tttctgatct catggcatgc tctgatgtgt tttgtaggag 3656
gggaaacact taaaataagc aaagaaaccg agcaggattg catatatcgg atggttcttg 3716
tctgtgctct ctgtacgttg cttctgcagc tgtgatttct aaacctctgc tggcactcag 3776
ctgacttttt gttttgtact ttgacccgcc tttttttgga ataccaagtt aaaaaaaaaa 3836
agttcttgaa ataaaacttt ttaaaaagct gtccaaaaaa aaaaa 3881
<210> 16
<211> 566
<212> Белок
<213> Mus musculus
<400> 16
Met Gly Arg Ala Gly Gly Gly Gly Pro Asp Trp Gly Pro Pro Pro Val
1 5 10 15
Leu Leu Leu Leu Gly Val Thr Leu Val Leu Thr Ala Gly Ala Val Pro
20 25 30
Ala Arg Glu Thr Gly Ser Ala Ile Glu Ala Glu Glu Leu Val Arg Ser
35 40 45
Ser Leu Ala Trp Glu Ser Arg Ala Asn Asp Thr Arg Glu Glu Ala Gly
50 55 60
Leu Pro Ala Ala Gly Glu Asp Glu Thr Ser Trp Thr Glu Arg Gly Ser
65 70 75 80
Glu Met Ala Ala Val Gly Pro Gly Val Gly Pro Glu Glu Ala Leu Glu
85 90 95
Ala Ser Ala Ala Val Thr Gly Thr Ala Trp Leu Glu Ala Asp Gly Pro
100 105 110
Gly Leu Gly Gly Val Thr Ala Glu Ala Gly Ser Gly Asp Ala Gln Thr
115 120 125
Leu Pro Ala Thr Leu Gln Ala Pro Asp Glu Ala Leu Gly Ser Ser Thr
130 135 140
Met Pro Pro Ala Ile Pro Glu Ala Thr Glu Thr Ser Gly Pro Pro Ser
145 150 155 160
Pro Ala Val His Asp Lys Pro Ser Val Gly Pro Glu Leu Pro Lys Glu
165 170 175
Ile Pro Leu Glu Val Arg Leu Asn Leu Gly Gly Ser Thr Pro Glu Pro
180 185 190
Thr Phe Pro Leu Gln Gly Thr Leu Glu Thr Gln Pro Ala Ser Asp Ile
195 200 205
Ile Asp Ile Asp Tyr Phe Glu Gly Leu Asp Ser Glu Gly Arg Gly Ala
210 215 220
Asp Met Gly Ser Phe Pro Gly Ser Pro Gly Thr Ser Glu Asn His Pro
225 230 235 240
Asp Thr Glu Gly Glu Thr Pro Ser Trp Ser Leu Leu Asp Leu Tyr Asp
245 250 255
Asp Phe Thr Pro Phe Asp Glu Ser Asp Phe Tyr Pro Thr Thr Ser Phe
260 265 270
Tyr Asp Asp Leu Glu Glu Glu Glu Glu Glu Glu Glu Asp Lys Asp Thr
275 280 285
Val Gly Gly Gly Asp Leu Glu Asp Glu Asn Asp Leu Leu Leu Pro Ser
290 295 300
Gln Lys Pro Gly Val Gly Pro Gly Thr Gly Gln Pro Thr Asn Arg Trp
305 310 315 320
His Ala Val Pro Pro Gln His Thr Leu Gly Met Val Pro Gly Ser Ser
325 330 335
Ile Ser Leu Arg Pro Arg Pro Gly Asp Pro Gly Lys Asp Leu Ala Ser
340 345 350
Gly Glu Asn Gly Thr Glu Cys Arg Val Gly Phe Val Arg His Asn Gly
355 360 365
Ser Cys Arg Ser Val Cys Asp Leu Phe Pro Ser Tyr Cys His Asn Gly
370 375 380
Gly Gln Cys Tyr Leu Val Glu Asn Ile Gly Ala Phe Cys Arg Cys Asn
385 390 395 400
Thr Gln Asp Tyr Ile Trp His Lys Gly Met Arg Cys Glu Ser Ile Ile
405 410 415
Thr Asp Phe Gln Val Met Cys Val Ala Val Gly Ser Ala Ala Leu Val
420 425 430
Leu Leu Leu Leu Phe Met Met Thr Val Phe Phe Ala Lys Lys Leu Tyr
435 440 445
Leu Leu Lys Thr Glu Asn Thr Lys Leu Arg Arg Thr Asn Lys Phe Arg
450 455 460
Thr Pro Ser Glu Leu His Asn Asp Asn Phe Ser Leu Ser Thr Ile Ala
465 470 475 480
Glu Gly Ser His Pro Asn Val Arg Lys Phe Cys Asp Thr Pro Arg Val
485 490 495
Ser Ser Pro His Ala Arg Ala Leu Ala His Tyr Asp Asn Ile Val Cys
500 505 510
Gln Asp Asp Pro Ser Ala Pro His Lys Ile Gln Asp Pro Leu Lys Ser
515 520 525
Arg Leu Lys Glu Glu Glu Ser Phe Asn Ile Gln Asn Ser Met Ser Pro
530 535 540
Lys Leu Glu Gly Gly Lys Gly Asp Gln Asp Asp Leu Gly Val Asn Cys
545 550 555 560
Leu Gln Asn Asn Leu Thr
565
<210> 17
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер T3
<400> 17
aattaaccct cactaaaggg 20
<210> 18
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер T7
<400> 18
taatacgact cactatagg 19
<210> 19
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Смысловой праймер собаки RT
<400> 19
aagttactgt cacaacggcg 20
<210> 20
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Антисмысловой праймер собаки RT
<400> 20
tcatcatgaa gagcaggagc 20
<210> 21
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Смысловой праймер человека RT
<400> 21
aagttactgt cacaatggcg g 21
<210> 22
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Антисмысловой праймер человека RT
<400> 22
tcatcatgaa gagcaggagc 20
<210> 23
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Смысловой праймер мыши RT
<400> 23
gagttactgt cacaacggcg 20
<210> 24
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Антисмысловой праймер мыши RT
<400> 24
tcatcatgaa caggaggaga 20
<210> 25
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер GAPDH
<400> 25
gggctgcttt taactctg 18
<210> 26
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер GAPDH
<400> 26
ccaggaaatg agcttgac 18
<210> 27
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер GAPDH
<400> 27
cttcaccacc atggagaagg 20
<210> 28
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер GAPDH
<400> 28
tgaagtcgca ggagacaacc 20
<210> 29
<211> 51
<212> ДНК
<213> Искусственная последовательность
<220>
<223> homo CSPG5 kpn clo
<400> 29
ggtaccatgg ggcgagccgg gggcgggggc ccgggccggg ggccgccgcc a 51
<210> 30
<211> 49
<212> ДНК
<213> Искусственная последовательность
<220>
<223> homocspg ecoRI clo
<400> 30
gaattcttag gttaaattat tctgaagaca gttcacatcc aagtcagcc 49
<210> 31
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 31
gaattcgcac atcacctgga agtcggtgat gatgga 36
<210> 32
<211> 1998
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый с Fc белок
<220>
<221> Кодирующая последовательность
<222> (1)..(1998)
<223>
<400> 32
acc acg aag ctt gcc gcc acc atg ggg cga gcc ggg ggc ggg ggc ccg 48
Thr Thr Lys Leu Ala Ala Thr Met Gly Arg Ala Gly Gly Gly Gly Pro
1 5 10 15
ggc cgg ggg ccg ccg cca ctg ctg ctg ttt ctg ggg gcc gcg ctg gtc 96
Gly Arg Gly Pro Pro Pro Leu Leu Leu Phe Leu Gly Ala Ala Leu Val
20 25 30
ctg gcc tct ggg gcc gtg ccg gcg cgt gag gcg ggc agc gcg gtt gag 144
Leu Ala Ser Gly Ala Val Pro Ala Arg Glu Ala Gly Ser Ala Val Glu
35 40 45
gcc gaa gag ctg gtg aag ggc agc ccg gcg tgg gag ccg cct gcc aac 192
Ala Glu Glu Leu Val Lys Gly Ser Pro Ala Trp Glu Pro Pro Ala Asn
50 55 60
gac acg cgg gaa gaa gcc ggc cca cca gcg gct ggg gaa gat gag gcg 240
Asp Thr Arg Glu Glu Ala Gly Pro Pro Ala Ala Gly Glu Asp Glu Ala
65 70 75 80
tcg tgg acg gcg ccc ggt ggc gag ctg gcc ggg cca gaa gag gtg ctg 288
Ser Trp Thr Ala Pro Gly Gly Glu Leu Ala Gly Pro Glu Glu Val Leu
85 90 95
cag gag tcg gct gcg gtg acc ggc acc gcc tgg ctg gaa gct gac agc 336
Gln Glu Ser Ala Ala Val Thr Gly Thr Ala Trp Leu Glu Ala Asp Ser
100 105 110
cca ggc ctg gga gga gtg acc gca gag gcg ggc agc ggc gat gcc cag 384
Pro Gly Leu Gly Gly Val Thr Ala Glu Ala Gly Ser Gly Asp Ala Gln
115 120 125
gcc ctt cca gct acg ctc cag gct ccc cac gag gtc ctc ggg cag tca 432
Ala Leu Pro Ala Thr Leu Gln Ala Pro His Glu Val Leu Gly Gln Ser
130 135 140
atc atg ccc cct gcc att cct gag gct aca gag gcc agc ggg cca ccc 480
Ile Met Pro Pro Ala Ile Pro Glu Ala Thr Glu Ala Ser Gly Pro Pro
145 150 155 160
tcc ccc acc ccc ggc gac aag ctg agc cca gct tct gaa ctc ccc aag 528
Ser Pro Thr Pro Gly Asp Lys Leu Ser Pro Ala Ser Glu Leu Pro Lys
165 170 175
gag agc ccc ttg gag gtt tgg ctg aac ctg ggg ggc agc aca ccc gac 576
Glu Ser Pro Leu Glu Val Trp Leu Asn Leu Gly Gly Ser Thr Pro Asp
180 185 190
cct caa ggg cca gag ctg act tac cca ttt cag ggc acc ctg gag ccc 624
Pro Gln Gly Pro Glu Leu Thr Tyr Pro Phe Gln Gly Thr Leu Glu Pro
195 200 205
caa ccg gca tca gat atc att gac atc gac tac ttc gaa gga ctg gat 672
Gln Pro Ala Ser Asp Ile Ile Asp Ile Asp Tyr Phe Glu Gly Leu Asp
210 215 220
ggt gag ggt cgt ggc gca gat ctg ggg agc ttc cca ggg tca cca gga 720
Gly Glu Gly Arg Gly Ala Asp Leu Gly Ser Phe Pro Gly Ser Pro Gly
225 230 235 240
acc tca gag aac cac cct gat act gag gga gag acc cct tcc tgg agc 768
Thr Ser Glu Asn His Pro Asp Thr Glu Gly Glu Thr Pro Ser Trp Ser
245 250 255
ctg ctt gac tta tac gat gat ttc acc ccc ttc gat gaa tct gat ttc 816
Leu Leu Asp Leu Tyr Asp Asp Phe Thr Pro Phe Asp Glu Ser Asp Phe
260 265 270
tac ccc acc aca tcc ttt tat gat gac ttg gat gaa gag gag gag gaa 864
Tyr Pro Thr Thr Ser Phe Tyr Asp Asp Leu Asp Glu Glu Glu Glu Glu
275 280 285
gag gag gat gac aaa gat gca gta gga ggt gga gac cta gaa gat gaa 912
Glu Glu Asp Asp Lys Asp Ala Val Gly Gly Gly Asp Leu Glu Asp Glu
290 295 300
aat gag ctt cta gtg ccc act ggg aag cct ggt ctg ggg ccc ggg aca 960
Asn Glu Leu Leu Val Pro Thr Gly Lys Pro Gly Leu Gly Pro Gly Thr
305 310 315 320
ggc cag ccc acc agt cgg tgg cat gct gtc cct cca cag cac act ctg 1008
Gly Gln Pro Thr Ser Arg Trp His Ala Val Pro Pro Gln His Thr Leu
325 330 335
ggg tcg gtc ccc ggc agc agc atc gcc ctc agg ccc cgc cca gga gag 1056
Gly Ser Val Pro Gly Ser Ser Ile Ala Leu Arg Pro Arg Pro Gly Glu
340 345 350
cca ggc agg gac ttg gcc tcc agt gaa aat ggc act gag tgc cgc agt 1104
Pro Gly Arg Asp Leu Ala Ser Ser Glu Asn Gly Thr Glu Cys Arg Ser
355 360 365
ggc ttt gtg cgg cat aac ggc tcc tgc cgg tca gtg tgc gac ctc ttc 1152
Gly Phe Val Arg His Asn Gly Ser Cys Arg Ser Val Cys Asp Leu Phe
370 375 380
cca agt tac tgt cac aat ggc ggc cag tgc tac ctg gtg gag aac ata 1200
Pro Ser Tyr Cys His Asn Gly Gly Gln Cys Tyr Leu Val Glu Asn Ile
385 390 395 400
ggg gcc ttc tgc agg tgc aac acg cag gac tac atc tgg cac aag ggg 1248
Gly Ala Phe Cys Arg Cys Asn Thr Gln Asp Tyr Ile Trp His Lys Gly
405 410 415
atg cgc tgc gag tcc atc atc acc gac ttc cag ccg aat tct gca gat 1296
Met Arg Cys Glu Ser Ile Ile Thr Asp Phe Gln Pro Asn Ser Ala Asp
420 425 430
atc ccc aga ggg ccc aca atc aag ccc tgt cct cca tgc aaa tgc cca 1344
Ile Pro Arg Gly Pro Thr Ile Lys Pro Cys Pro Pro Cys Lys Cys Pro
435 440 445
gca cct aac ctc ttg ggt gga cca tcc gtc ttc atc ttc cct cca aag 1392
Ala Pro Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys
450 455 460
atc aag gat gta ctc atg atc tcc ctg agc ccc ata gtc aca tgt gtg 1440
Ile Lys Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val Thr Cys Val
465 470 475 480
gtg gtg gat gtg agc gag gat gac cca gat gtc cag atc agc tgg ttt 1488
Val Val Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile Ser Trp Phe
485 490 495
gtg aac aac gtg gaa gta cac aca gct cag aca caa acc cat aga gag 1536
Val Asn Asn Val Glu Val His Thr Ala Gln Thr Gln Thr His Arg Glu
500 505 510
gat tac aac agt act ctc cgg gtg gtc agt gcc ctc ccc atc cag cac 1584
Asp Tyr Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro Ile Gln His
515 520 525
cag gac tgg atg agt ggc aag gag ttc aaa tgc aag gtc aac aac aaa 1632
Gln Asp Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys
530 535 540
gac ctc cca gcg ccc atc gag aga acc atc tca aaa ccc aaa ggg tca 1680
Asp Leu Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro Lys Gly Ser
545 550 555 560
gta aga gct cca cag gta tat gtc ttg cct cca cca gaa gaa gag atg 1728
Val Arg Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu Glu Glu Met
565 570 575
act aag aaa cag gtc act ctg acc tgc atg gtc aca gac ttc atg cct 1776
Thr Lys Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp Phe Met Pro
580 585 590
gaa gac att tac gtg gag tgg acc aac aac ggg aaa aca gag cta aac 1824
Glu Asp Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr Glu Leu Asn
595 600 605
tac aag aac act gaa cca gtc ctg gac tct gat ggt tct tac ttc atg 1872
Tyr Lys Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser Tyr Phe Met
610 615 620
tac agc aag ctg aga gtg gaa aag aag aac tgg gtg gaa aga aat agc 1920
Tyr Ser Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu Arg Asn Ser
625 630 635 640
tac tcc tgt tca gtg gtc cac gag ggt ctg cac aat cac cac acg act 1968
Tyr Ser Cys Ser Val Val His Glu Gly Leu His Asn His His Thr Thr
645 650 655
aag agc ttc tcc cgg act ccg ggt aaa tga 1998
Lys Ser Phe Ser Arg Thr Pro Gly Lys
660 665
<210> 33
<211> 665
<212> Белок
<213> Искусственная последовательность
<220>
<223> Слитый с Fc белок
<400> 33
Thr Thr Lys Leu Ala Ala Thr Met Gly Arg Ala Gly Gly Gly Gly Pro
1 5 10 15
Gly Arg Gly Pro Pro Pro Leu Leu Leu Phe Leu Gly Ala Ala Leu Val
20 25 30
Leu Ala Ser Gly Ala Val Pro Ala Arg Glu Ala Gly Ser Ala Val Glu
35 40 45
Ala Glu Glu Leu Val Lys Gly Ser Pro Ala Trp Glu Pro Pro Ala Asn
50 55 60
Asp Thr Arg Glu Glu Ala Gly Pro Pro Ala Ala Gly Glu Asp Glu Ala
65 70 75 80
Ser Trp Thr Ala Pro Gly Gly Glu Leu Ala Gly Pro Glu Glu Val Leu
85 90 95
Gln Glu Ser Ala Ala Val Thr Gly Thr Ala Trp Leu Glu Ala Asp Ser
100 105 110
Pro Gly Leu Gly Gly Val Thr Ala Glu Ala Gly Ser Gly Asp Ala Gln
115 120 125
Ala Leu Pro Ala Thr Leu Gln Ala Pro His Glu Val Leu Gly Gln Ser
130 135 140
Ile Met Pro Pro Ala Ile Pro Glu Ala Thr Glu Ala Ser Gly Pro Pro
145 150 155 160
Ser Pro Thr Pro Gly Asp Lys Leu Ser Pro Ala Ser Glu Leu Pro Lys
165 170 175
Glu Ser Pro Leu Glu Val Trp Leu Asn Leu Gly Gly Ser Thr Pro Asp
180 185 190
Pro Gln Gly Pro Glu Leu Thr Tyr Pro Phe Gln Gly Thr Leu Glu Pro
195 200 205
Gln Pro Ala Ser Asp Ile Ile Asp Ile Asp Tyr Phe Glu Gly Leu Asp
210 215 220
Gly Glu Gly Arg Gly Ala Asp Leu Gly Ser Phe Pro Gly Ser Pro Gly
225 230 235 240
Thr Ser Glu Asn His Pro Asp Thr Glu Gly Glu Thr Pro Ser Trp Ser
245 250 255
Leu Leu Asp Leu Tyr Asp Asp Phe Thr Pro Phe Asp Glu Ser Asp Phe
260 265 270
Tyr Pro Thr Thr Ser Phe Tyr Asp Asp Leu Asp Glu Glu Glu Glu Glu
275 280 285
Glu Glu Asp Asp Lys Asp Ala Val Gly Gly Gly Asp Leu Glu Asp Glu
290 295 300
Asn Glu Leu Leu Val Pro Thr Gly Lys Pro Gly Leu Gly Pro Gly Thr
305 310 315 320
Gly Gln Pro Thr Ser Arg Trp His Ala Val Pro Pro Gln His Thr Leu
325 330 335
Gly Ser Val Pro Gly Ser Ser Ile Ala Leu Arg Pro Arg Pro Gly Glu
340 345 350
Pro Gly Arg Asp Leu Ala Ser Ser Glu Asn Gly Thr Glu Cys Arg Ser
355 360 365
Gly Phe Val Arg His Asn Gly Ser Cys Arg Ser Val Cys Asp Leu Phe
370 375 380
Pro Ser Tyr Cys His Asn Gly Gly Gln Cys Tyr Leu Val Glu Asn Ile
385 390 395 400
Gly Ala Phe Cys Arg Cys Asn Thr Gln Asp Tyr Ile Trp His Lys Gly
405 410 415
Met Arg Cys Glu Ser Ile Ile Thr Asp Phe Gln Pro Asn Ser Ala Asp
420 425 430
Ile Pro Arg Gly Pro Thr Ile Lys Pro Cys Pro Pro Cys Lys Cys Pro
435 440 445
Ala Pro Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys
450 455 460
Ile Lys Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val Thr Cys Val
465 470 475 480
Val Val Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile Ser Trp Phe
485 490 495
Val Asn Asn Val Glu Val His Thr Ala Gln Thr Gln Thr His Arg Glu
500 505 510
Asp Tyr Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro Ile Gln His
515 520 525
Gln Asp Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys
530 535 540
Asp Leu Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro Lys Gly Ser
545 550 555 560
Val Arg Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu Glu Glu Met
565 570 575
Thr Lys Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp Phe Met Pro
580 585 590
Glu Asp Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr Glu Leu Asn
595 600 605
Tyr Lys Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser Tyr Phe Met
610 615 620
Tyr Ser Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu Arg Asn Ser
625 630 635 640
Tyr Ser Cys Ser Val Val His Glu Gly Leu His Asn His His Thr Thr
645 650 655
Lys Ser Phe Ser Arg Thr Pro Gly Lys
660 665
<---
Реферат
Изобретение относится к области фармацевтики, а именно к фармацевтической композиции для лечения и/или предотвращения злокачественной опухоли. Композиция содержит в качестве активного ингредиента антитело или его фрагмент с иммунологической реакционноспособностью в отношении белка CSPG5 или его фрагмента, состоящего по меньшей мере из 7 или более последовательных аминокислотных остатков, где белок CSPG5 состоит из любой из аминокислотных последовательностей, представленных SEQ ID NO: 8, 4, 6, 10 и 12, или аминокислотной последовательности с идентичностью аминокислот с этими аминокислотными последовательностями 80% или более. Изобретение обеспечивает возможность создания композиции, эффективной для лечения и/или предотвращения злокачественной опухоли. 3 з.п. ф-лы, 5 ил., 7 пр.

Комментарии