Химерные белки на основе утрофина и дистрофина человека и их применение для лечения миодистрофии дюшенна - RU2767335C1
Код документа: RU2767335C1
Чертежи




Описание
Изобретение относится к области биотехнологии и медицины, в частности, к химерным белкам, содержащим последовательности дистрофина и утрофина человека, а также к последовательностям нуклеиновых кислот, кодирующим такие белки, и векторам, несущим такие последовательности. Генетические конструкции по настоящему изобретению могут использоваться для заместительной генной терапии миодистрофии Дюшенна при доставке в мышечные клетки пациентов с помощью известных экспрессионных векторов и дальнейшей экспрессии там химерного белка, способного функционально заменить отсутствующий дистрофии.
Уровень техники.
Миодистрофия Дюшенна занимает первое место среди мышечных дистрофий по распространенности. Одним из наиболее многообещающих способов ее лечения является заместительная терапия, а именно - доставка гена, кодирующего дистрофии либо его аналог, в мышцы. Среди самых популярных способов доставки генетического материалы следует выделить аденоассоциированные вирусные векторы (AAV) (Duan 2018). Преимущества AAV: вариабельность серотипа (что позволяет обеспечить таргетную доставку конструкции в целевую ткань), неспособность встраиваться в геном и низкая иммуногенность капсида. В то же время AAV имеют фундаментальное ограничение - с их помощью невозможна доставка генетической конструкции, длина которой превышает 4,7 тысяч пар оснований. Так как длина конструкции, кодирующий полноразмерный дистрофии, превышает 11 тысяч пар оснований, была предложена стратегия дизайна сокращенных вариантов дистрофина (т.н. микродистрофины), в которых отсутствует часть наименее критичных для функционирования исходного белка доменов. Этот подход имеет недостаток, связанный с иммуногенностью трансгена. Так как у большинства больных миодистрофией Дюшенна отсутствует полноразмерный дистрофии (хотя чаще всего присутствуют укороченные изоформы (Culligan et al. 1998), иммунная система может распознать трансген как чужеродный белок (Ferrer, Wells, and Wells 2000; Mendell et al. 2010). Для предотвращения иммунного ответа на трансген возможна разработка конструкций на основе ближайшего функционального и структурного гомолога дистрофина - утрофина (Tinsley and Davies 1993). Утрофин выполняет аналогичную дистрофину функцию, но в эмбриональном периоде развития, после чего заменяется дистрофином.
Все предложенные на данный момент варианты конструкций, способные быть доставленными с помощью AAV, и имеющие терапевтический потенциал, включают N-концевой домен (критически необходимый для связывания с актином) и цистеин-богатый домен (критически необходимый для связывания с белками сарколеммы). При этом возможно варьирование состава т.н. rod-домена, а именно - состава и количества спектрин-подобных повторов и неструктурированных пептидных доменов (т.н. доменов hinge - петля, стержень, далее - Н) (Duan 2018).
Из заявки WO2002029056A2 (дата публикации 11 апреля 2002) известны генетические конструкции, кодирующие «сокращенные» варианты дистрофина - микродистрофины. Сокращение происходит в основном за счет удаления из кодирующей последовательности фрагментов, кодирующих некоторые спектриновые повторы и неструктурированные линкеры. Авторы предлагают варианты ΔR4-R23 и ΔR2-R21 (указаны номера спектриновых повторов, отсутствующих в конечной конструкции). Конструкции, кодирующие такие белки, могут быть упакованы в аденоассоциированные вирусные векторы. Введение этих векторов мышам линии mdx (модельная линия, в которой не экспрессируется дистрофии, применяется для изучения и тестирования потенциальных препаратов для лечения миодистрофии Дюшенна) приводило к снижению негативных последствий делеции дистрофина.
В заявке WO2019078916A1 (дата публикации 16 мая 2018) раскрыт микродистрофии ΔR4-R23. Конструкцию, его кодирующую, доставляли с помощью аденоассоциированных векторов серотипа rAAVrh.74 под управлением промотора MHCK7. Авторы наблюдали восстановление мышечных функций и снижение тяжести симптомов дистрофии у мышей линии mdx. В Liu с соавторами (Liu, 2005) микродистрофин ΔR4-R23 доставляли в длинный разгибатель пальцев (extensior digitum longius) с помощью аденоассоциированного вектора серотипа 5. Работа проводилась на мышах линий В10 и mdx. Доставляемый микродистрофин локализуется в сарколеммах мышечных клеток аналогично полноразмерному нативному дистрофину. Отмечено уменьшение количества миофибрилл с центрально-локализованными ядрами, причем эффект был значительнее при доставке микродистрофина в мышцы молодых (пятинедельных) мышей по сравнению со взрослыми (девятимесячными). Частичная экспрессия микродистрофина ΔR4-R23 повышала специфическую сократительную силу мышц и предотвращала понижение сократительной способности мышцы под действием нескольких циклов электростимулированного сокращения.
В заявке US20180148488A1 (дата публикации 31 мая 2018) раскрыты 8 различных конструкций, кодирующих микродистрофины с 4-6 разными спектриновыми повторами и неструктурированными (hinge) доменами. Была продемонстрирована негативная роль неструктурированного домена 2 (hinge 2) в образовании кольцевых миотубул в мышцах (ringbinden). Были продемонстрированы преимущества микро дистрофинов, содержащих спектриновые повторы R15 и R16, обеспечивающие привлечение nNOS на мембрану. В целом, работа демонстрирует потенциал подхода по модификации микродистрофинов, заключающейся в замене/модификации отдельных спектриновых повторов.
Из заявки US20170368198A1 (дата публикации 28 декабря 2018) известны две конструкции минидистрофинов: первая включает N-концевой домен, «шарнирный» домен H1, спектриновые повторы 1, 2, «шарнирный» домен H3, спектриновые повторы 22, 23, 24, «шарнирный» домен Н4, цистеин-богатый домен и фрагмент С-концевого домена. Вторая конструкция аналогична первой, за исключением того, что в ней отсутствует «шарнирный» домен Н3. Применение AAV-векторов, кодирующих конструкцию с доменом Н3 ("miniDys3978") привело к предотвращению негативных последствий делеции дистрофина в мышиной (mdx), крысиной (Dmdmdx) и собачьей (DMD GRMD) модели миодистрофии Дюшенна. Авторы отдельно отмечают низкий иммунный ответ на трансген «miniDys3978». Однако эти данные были получены на крысах.
Все конструкции, основанные на модификации дистрофина, обладают общим недостатком: белок, кодируемый подобными конструкциями, отсутствует у значительной доли пациентов с миодистрофией Дюшенна, и его появление после введения аденоассоциированных вирусных векторов может привести к развитию иммунного ответа на трансген.
Этот недостаток был устранен в заявке WO2013009943A2 (дата публикации 17 января 2013), в которой была предложена конструкция, кодирующая микроутрофин. Этот белок представляет собой производное полноразмерного утрофина с делецией SR4-SR21. В этой работе белок доставлялся в виде микроутрофина, слитого с ТАТ-доменом, облегчающим транспорт белка в целевые ткани; либо с помощью нуклеотидной конструкции. Авторы отмечают положительные эффекты на мышах линии mdx. Однако выбранный способ доставки ограничивает возможности такого подхода.
В работе Song и соавторов (Song, 2019) был предложен ряд конструкций, основанных на утрофине человека (микроутрофин), содержащих N-терминальный, цистеин-богатый и некоторые спектриновые повторы исходного белка. Одним из требований к конструкциям была уменьшенная длина по сравнению с исходным утрофином, что должно было обеспечить сборку аденоассоциированного вектора, кодирующего такую конструкцию. Была предложена конструкция, включающая актин-связьгоающий домен 1, неструктурированный домен H1, спектриновые повторы 1-3, неструктурированный домен Н2, спектриновый домен 22, неструктурированный домен Н4 и цистеин-богатый домен. Данная конструкция продемонстрировала эффективность в предотвращении ряда проявлений миодистрофии Дюшенна в животной модели (дистрофин-дефицитная собака) и сниженную иммуногенность по сравнению с микродистрофином. Этот микроутрофин представляется наиболее близким аналогом заявленному изобретению.
По нашим данным, описанный выше микроутрофин уступает аналогичному по структурной композиции микродистрофину в эффективности, что может быть связано с различием в последовательностях и, соответственно, функциональности определенных доменов белков.
Задачей, на решение которой направлено настоящее изобретение, являлось расширение арсенала средств для заместительной генной терапии миодистрофии Дюшенна, а именно создание генетической конструкции, обеспечивающей равную или повышенную эффективность по сравнению с генетической конструкцией, кодирующей известный из уровня техники микроутрофин.
Задача решается тем, что созданы химерные белки на основе утрофина человека, в которых один или два функциональных домена утрофина заменены на соответствующие им домены дистрофина человека, а также генетические конструкции, кодирующие упомянутые химерные белки, и экспрессионные векторы для доставки упомянутых генетических конструкций в мышечные клетки пациентов. Для замены были выбраны наиболее значительно различающиеся между собой участки утрофина и дистрофина (короткий N-концевой пептид и так называемый «петлевой домен 1» (hinge 1).
Техническим результатом настоящего изобретения являются новые химерные белки для терапии миодистрофии Дюшенна с равной или повышенной эффективностью по сравнению с известным из уровня техники микроутрофином.
Сущность изобретения
Сущность созданного технического решения заключается в следующем:
Предлагаются химерные белки, имеющие в своем составе структурные домены дистрофина и утрофина человека; также предлагаются нуклеиновые кислоты, кодирующие упомянутые химерные белки. Упомянутые нуклеиновые кислоты могут быть доставлены в мышцы пациентов, страдающих от миодистрофии Дюшенна, с помощью известных из уровня техники экспрессионных векторов, например, аденоассоциированных вирусных векторов, плазмид, а также наночастиц и т.д.
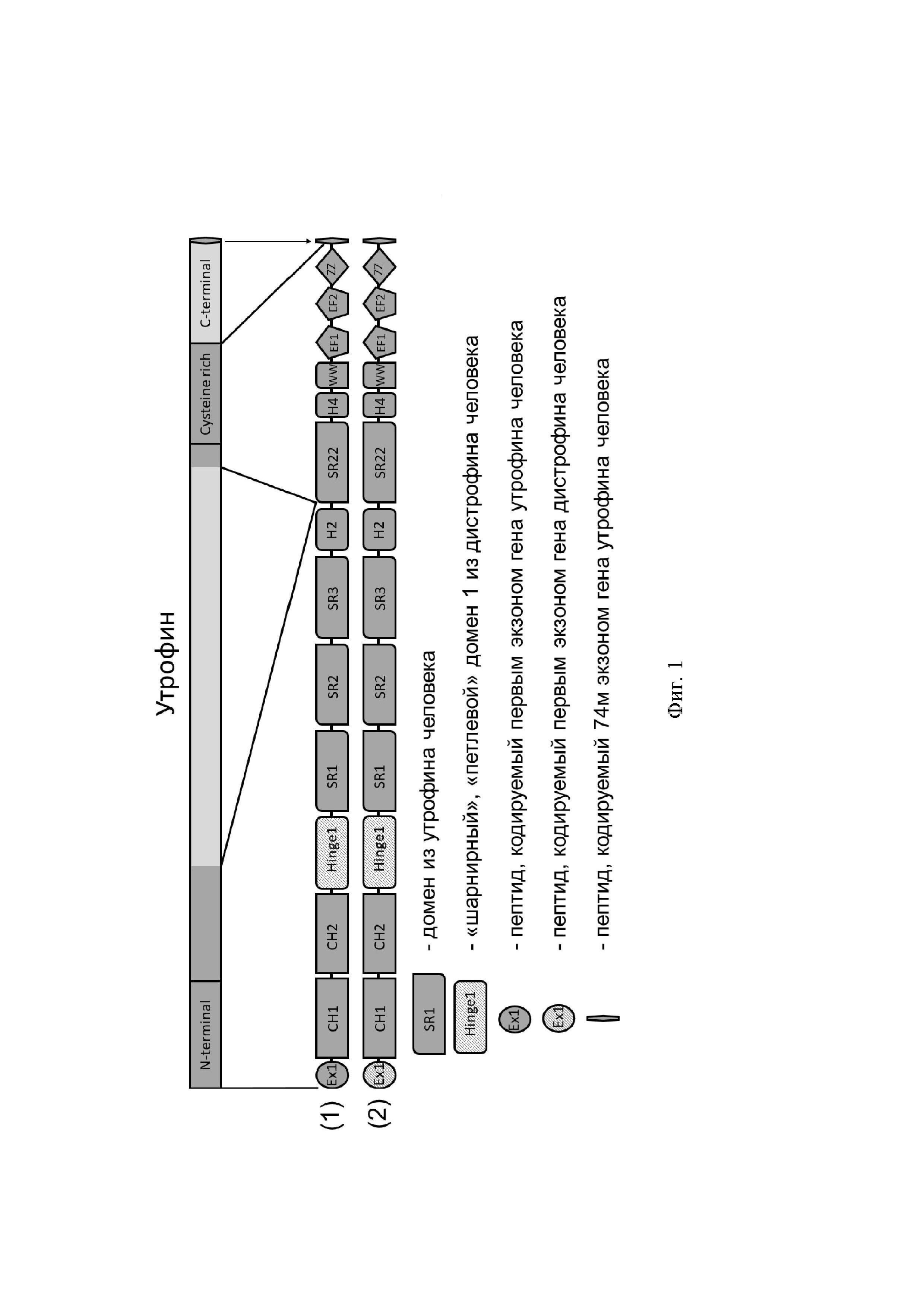
В состав первого химерного белка входят следующие структурные домены:
- N-терминальный домен утрофина (здесь и далее человека);
- первый неструктурированный домен дистрофина (hinge 1);
- спектриновые повторы 1, 2, 3 утрофина;
- второй неструктурированный домен утрофина (hinge 2);
- спектриновый повтор 22 утрофина;
- четвертый неструктурированный домен утрофина (hinge 4);
- цистеин-богатый (CR) домен утрофина;
- олигопептид QAM, кодируемый экзоном 74 утрофина человека (Фиг. 1).
Конструкция первого химерного белка по настоящему изобретению отличается от конструкции, описанной в статье Song с соавторами, заменой домена H1. Последовательность известного из статьи Song с соавторами белка содержит домен H1 утрофина, а в последовательности химерного белка по настоящему изобретению домен H1 дистрофина. Дополнительно в химерном белке по настоящему изобретению присутствует олигопептид, кодируемый концевым экзоном утрофина: три аминокислоты QAM, находящиеся на С-конце утрофина.
В состав второго химерного белка входят следующие структурные домены:
- пептид, кодируемый первым экзоном дистрофина;
- N-терминальный домен утрофина, не включающий пептид, кодируемый первым экзоном утрофина;
- первый неструктурированный домен дистрофина (hinge 1);
- спектриновые повторы 1,2,3 утрофина;
- второй неструктурированный домен утрофина (hinge 2);
- спектриновый повтор 22 утрофина;
- четвертый неструктурированный домен утрофина (hinge 4);
- цистеин-богатый (CR) домен утрофина;
- олигопептид QAM, кодируемый экзоном 74 утрофина человека (Фиг. 1).
В отличие от конструкции первого химерного белка по настоящему изобретению, конструкция второго химерного белка содержит пептид, кодируемый первым экзоном дистрофина вместо пептида, кодируемого первым экзоном утрофина.
Предлагаемые химерные белки содержат актин-связывающий домен, цистеин-богатый домен и 4 спектриновых повтора утрофина, что позволяет им связываться с актином и белками сарколеммы и эффективно встраиваться в дистрофин-ассоциированный белковый комплекс, что уже ранее было показано для аналогичных по структурной композиции микроутрофинов (Song, 2019). Кроме того, в химерных белках по настоящему изобретению укороченный первый неструктурированный домен утрофина (hinge 1) заменен на более длинный первый неструктурированный домен дистрофина. В одном из вариантов осуществления изобретения дополнительно N-терминальный пептид утрофина заменен на более короткий N-терминальный пептид дистрофина, что может оптимизировать длину химерного белка. Так как основная часть (более 90%) белковой последовательности предлагаемых конструкций гомологична утрофину человека, который всегда экспрессируется в эмбриональной стадии, ожидается сниженный иммунный ответ на химерные белки по настоящему изобретению.
Ранее нами были проведены эксперименты, в которых мы сравнивали эффективность микродистрофина и микроутрофина, сходных по доменной структуре (микродистрофин, аналогичный описанному у Sarepta - NCT02376816 и микроутрофин, аналогичный описанному в Song et al, 2019). Каждая из этих конструкций содержит N-концевой домен, первый неструктурированный домен (H1), три первых спектриновых повтора, второй неструктурированный домен (Н2), последний (24й - для дистрофина, и 22й - для утрофина) спектриновый повтор, четвертый неструктурированный домен (Н4), и цистеин-богатый домен. N-концевой домен и цистеин-богатый домен являются абсолютно необходимыми для функционирования белка, в то время как центральная часть может быть более вариабельна. В наших экспериментах микроутрофин демонстрировал сниженную эффективность по сравнению с микродистрофином. Это проявлялось в менее выраженных «защитных» эффектах микроутрофина на мышах линии mdx. Сравнение последовательностей микродистрофина и микроутрофина, использованных в экспериментах, показало наибольшие различия в двух доменах этих конструкций. Это N-концевой пептид, кодируемый первым экзоном каждого гена и предшествующий функциональной части актин-связывающего домена. Второй домен - это неструктурированный шарнирный повтор 1 (hinge 1), который у дистрофина почти в два раза длиннее, чем у утрофина (97 аминокислотных остатков против 51). Необходимо отметить, что микродистрофин и микроутрофин значительно короче полноразмерных форм соответствующих белков. Вероятно, именно дополнительное сокращение физической длины белка и снижение его гибкости за счет значительного сокращения самого гибкого из доменов белка приводит к снижению эффективности микроутрофина. Замена его на аналог из дистрофина может увеличить эффективность микроутрофина.
В состав предлагаемых конструкций также вводится С-концевой олигопептид QAM, который кодируется последним, 74-м экзоном полноразмерного утрофина человека. Данная модификация вводится для стабилизации белка, так как имеются данные по нестабильности изоформ дистрофина, в которых последний трехаминокислотный олигопептид заменен (Greener, 2002). Аналогичная модификация для микродистрофина применялась в конструкции, предлагаемой в заявке WO2001083695A2.
Также в настоящей заявке предлагаются рекомбинантные нуклеиновые кислоты, кодирующие химерные белки по заявленному изобретению. Рекомбинантная нуклеиновая кислота доставляется в составе доступного экспрессионного вектора в клетки организма, которому требуется лечение, с упомянутой нуклеиновой кислоты экспрессируется химерный белок, включающий в себя как структурные домены утрофина человека, так и структурные элементы дистрофина человека.
Также в настоящей заявке предлагаются экспрессионные векторы на основе AAV, несущие ген, кодирующий одну из заявленных химерных конструкций на основе микроутрофина.
Последовательность предлагаемых в данной заявке конструкций более чем на 90% совпадает с последовательностью доменов утрофина человека, что обеспечивает ее низкую иммуногенность в соответствии с данными, известными из уровня техники. В то же время, конструкции по настоящему изобретению демонстрируют лучшую эффективность по сравнению с микроутрофином при введении в мышиную модель миодистрофии Дюшенна.
Детальное описание изобретения
Определения
Если не указано иначе, предполагается, что все термины данной области, обозначения и другие научные термины или терминология, используемая в данной заявке, имеют значения, которые обычно понимают специалисты в области, к которой относится настоящее изобретение. В некоторых случаях, определения терминов с общепринятыми значениями приведены в данной заявке для ясности и/или для быстрой справки и понимания, и включение таких определений в настоящее описание не обязательно должно истолковываться как наличие существенного отличия значения термина от обычно подразумеваемого в данной области.
Термин «химерный белок» обозначает белок, в аминокислотной последовательности которого присутствуют фрагменты
последовательностей как минимум двух различных белков. Белки могут быть из одного организма или из различных организмов.
Термин «экспрессионный вектор» обозначает вектор, содержащий нуклеиновую кислоту, содержащую промоторную область, необходимые регуляторные элементы и открытую рамку считывания. С экспрессионного вектора должен экспрессироваться функциональный белок. Экспрессионный вектор может быть вирусным или невирусным.
«N-терминальный домен», или «N-концевой домен» - структурный домен белка, расположенный на N-конце белка. Применительно к дистрофину, под N-терминальным доменом понимается фрагмент белка, начинающийся с аминокислоты 1 и заканчивающийся аминокислотой 240 (в соответствии с последовательностью, приведенной в базе Uniprot, ID P11532, https://www.uniprot.org/). Применительно к утрофину, под N-терминальным доменом понимается фрагмент белка, начинающийся с аминокислоты 1 и заканчивающийся аминокислотой 255 (в соответствии с последовательностью, приведенной в базе Uniprot, ID Р46939).
«актин-связывающий домен» - структурный и функциональный домен, присутствующий в дистрофине, утрофине и еще ряде белков. Основным свойством актин-связывающего домена является способность эффективно связывать актин. Применительно к дистрофину, актин-связывающим доменом называют фрагмент белка, начинающийся с аминокислоты 15 и заканчивающийся аминокислотой 240 (в соответствии с последовательностью, приведенной в базе Uniprot, ID P11532). Применительно к утрофину, актин-связьшающим доменом называют фрагмент белка, начинающийся с аминокислоты 31 и заканчивающийся аминокислотой 255 (в соответствии с последовательностью, приведенной в базе Uniprot, ID Р46939). Актин-связывающий домен практически полностью перекрывается с N-терминальным доменом, однако не является полностью ему тождественным, так как короткий N-концевой пептид (14 ак для дистрофина, 30 ак для утрофина), по всей видимости, не несет известной структурной и функциональной нагрузки, и не включается в состав актин-связывающего домена.
«центральный домен», или «стержневой домен» - структурный и функциональный домен белков дистрофина и утрофина. Применительно к дистрофину, центральным доменом называют фрагмент белка, начинающийся с аминокислоты 241 и заканчивающийся аминокислотой 3054 (в соответствии с последовательностью, приведенной в базе Uniprot, ID P11532). Применительно к утрофину, центральным доменом называют фрагмент белка, начинающийся с аминокислоты 256 и заканчивающийся аминокислотой 2811 (в соответствии с последовательностью, приведенной в базе Uniprot, ID Р46939). Центральный домен состоит из 24х (для дистрофина) или 22х (для утрофина) спектриновых повторов и четырех неструктурированных доменов.
«спектриновые повторы» - структурные домены, свойственные дистрофину, утрофину и еще ряду белков. Применительно к дистрофину и утрофину, включаются в состав центрального домена. Представляют собой обособленные домены, каждый из которых образован тремя альфа-спиралями, организованными в т.н. coiled coil структуру.
«неструктурированные домены», так же «hinge domains» («петлевые домены», «hinge» (англ.) - «петля») - домены, свойственные дистрофину и утрофину. Входят в состав центрального домена. Для каждого белка выделяют четыре неструктурированных домена, называемые H1, Н2, Н3, Н4. Третичная структура этих доменов на данный момент неизвестна, предполагается, что в них отсутствуют какие-либо стандартные элементы вторичной структуры, такие, как альфа-спирали или бета-листы. Функции этих доменов также доподлинно неизвестны, предполагается, что за счет отсутствия элементов вторичной структуры эти домены обеспечивают дополнительную гибкость молекуле дистрофина/утрофина. Применительно к дистрофину, доменом H1 называют фрагмент белка, начинающийся с аминокислоты 241 и заканчивающийся аминокислотой 338; доменом Н2 называют фрагмент белка, начинающийся с аминокислоты 668 и заканчивающийся аминокислотой 718; доменом Н3 называют фрагмент белка, начинающийся с аминокислоты 2427 и заканчивающийся аминокислотой 2468; доменом Н4 называют фрагмент белка, начинающийся с аминокислоты 3041 и заканчивающийся аминокислотой 3054 (в соответствии с последовательностью, приведенной в базе Uniprot, ID P11532). Применительно к утрофину, доменом H1 называют фрагмент белка, начинающийся с аминокислоты 256 и заканчивающийся аминокислотой 308; доменом Н2 называют фрагмент белка, начинающийся с аминокислоты 638 и заканчивающийся аминокислотой 686; доменом Н3 называют фрагмент белка, начинающийся с аминокислоты 2187 и заканчивающийся аминокислотой 2228; доменом Н4 называют фрагмент белка, начинающийся с аминокислоты 2798 и заканчивающийся аминокислотой 2811 (в соответствии с последовательностью, приведенной в базе Uniprot, ID Р46939).
«цистеин-богатый (CR) домен» - структурный и функциональный домен дистрофина и утрофина, обеспечивающий связывание этих белков с белками дистрофин-ассоциированного белкового комплекса на сарколемме миотубул. Применительно к дистрофину, цистеин-богатым доменом называют фрагмент белка, начинающийся с аминокислоты 3055 и заканчивающийся аминокислотой 3345 (в соответствии с последовательностью, приведенной в базе Uniprot, ID P11532). Применительно к утрофину, цистеин-богатым доменом называют фрагмент белка, начинающийся с аминокислоты 2812 и заканчивающийся аминокислотой 3102 (в соответствии с последовательностью, приведенной в базе Uniprot, ID Р46939).
Предлагаются генетические конструкции, позволяющие с помощью экспрессионного вектора доставлять в клетки генетический материал, кодирующий химерные белки, предназначенные для функциональной замены отсутствующего дистрофина у пациентов с миодистрофией Дюшенна. Настоящее изобретение может применяться для лечения этого заболевания.
В состав первого химерного белка, именуемого в дальнейшем Н1-Н-Comi-UTRN, входят следующие структурные домены:
- N-терминальный домен утрофина (аминокислоты 1-255 утрофина человека Р46939 (здесь и далее - по базе данных Uniprot:);
- первый неструктурированный домен дистрофина (hinge 1) (аминокислоты 241-342 дистрофина человека P11532);
- спектриновые повторы 1, 2, 3 утрофина - второй неструктурированный домен утрофина (hinge 2) (координаты аминокислот 312-685 утрофина человека Р46939);
- спектриновый повтор 22 утрофина - четвертый неструктурированный домен утрофина (hinge 4) - цистеин-богатый (CR) домен и фрагмент С-концевого домена утрофина человека (координаты аминокислот 2690-3165 утрофина человека Р46939);
- олигопептид QAM, кодируемый экзоном 74 утрофина человека (координаты аминокислот 3431-3433 утрофина человека Р46939).
В состав химерного белка 2 (именуемого в дальнейшем Н1-Ех1-Н-Comi-UTRN входят следующие структурные домены:
- пептид, кодируемый первым экзоном дистрофина человека (координаты аминокислот 1-14 дистрофина человека Р11532);
- актин-связывающий домен утрофина, не включающий пептид, кодируемый первым экзоном утрофина человека (координаты аминокислот 31-255 утрофина человека Р46939);
- первый неструктурированный домен дистрофина (hinge 1) (аминокислоты 241-342 дистрофина человека P11532);
- спектриновые повторы 1, 2, 3 утрофина - второй неструктурированный домен утрофина (hinge 2) (координаты аминокислот 312-685 утрофина человека Р46939);
- спектриновый повтор 22 утрофина - четвертый неструктурированный домен утрофина (hinge 4) - цистеин-богатый (CR) домен и фрагмент С-концевого домена утрофина человека (координаты аминокислот 2690-3165 утрофина человека Р46939);
- олигопептид QAM, кодируемый экзоном 74 утрофина человека (координаты аминокислот 3431-3433 утрофина человека Р46939).
Принципиальные схемы предлагаемых конструкций представлены на Фиг. 1.
Изобретение иллюстрируется следующими графическими материалами.
На Фиг. 1 представлена принципиальная схема предложенных конструкций. Темным отмечены домены полноразмерного человеческого утрофина, включенные в состав предлагаемых конструкций. Светлым отмечены домены человеческого утрофина, не включенные в предлагаемые конструкции. Штриховкой отмечены домены предлагаемых конструкций, соответствующие доменам человеческого дистрофина. Белок 1 - Н1-Н-Comi-UTRN; белок 2 - Ex1-H1-H-Comi-UTRN.
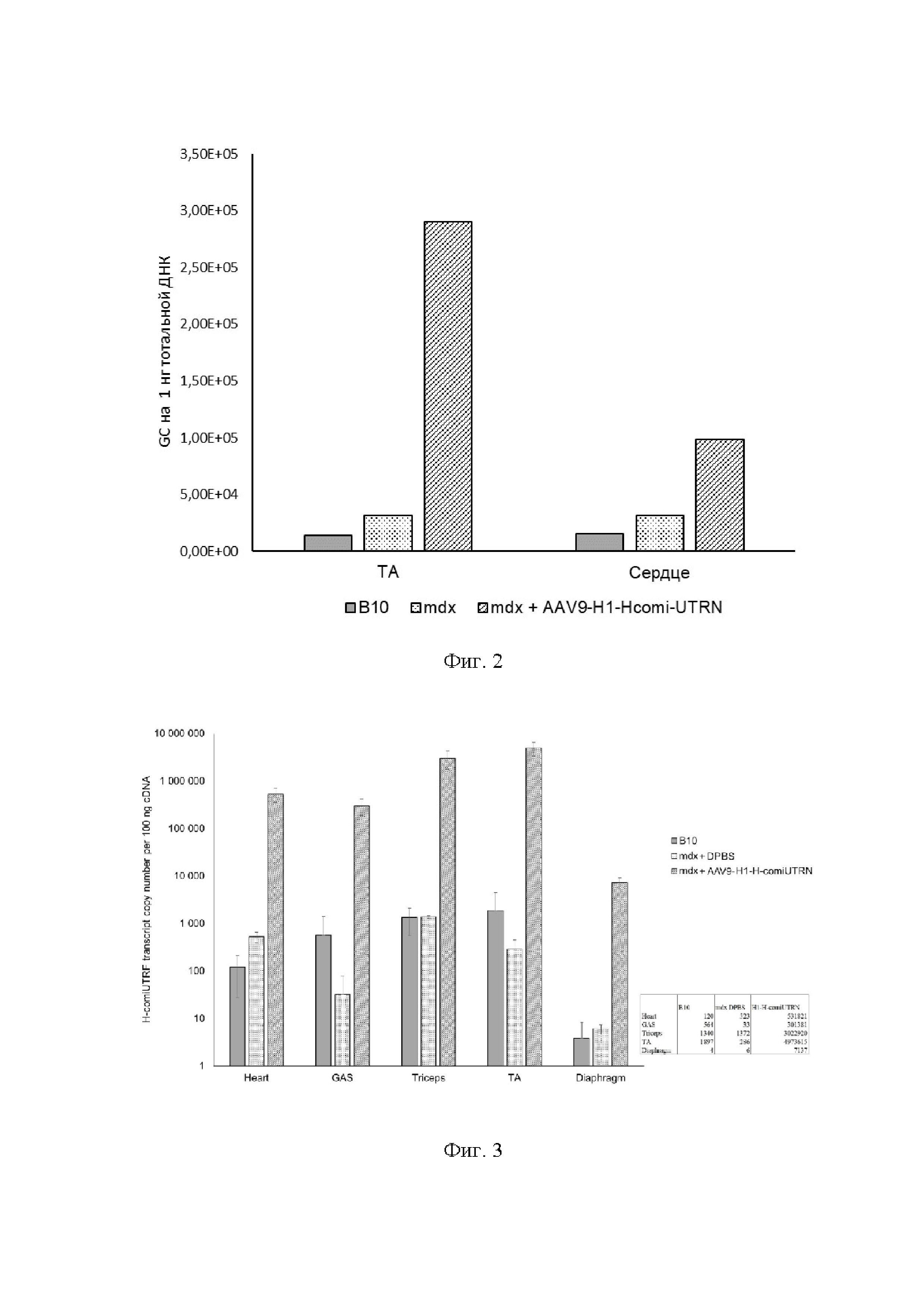
На Фиг. 2 представлено наличие геномных копий вектора AAV9 в передней большеберцовой мышце (tibialis anterior, ТА) и сердце мышей линии В10 (получали плацебо - DPBS), мышей линии mdx, получавших плацебо, и мышей линии mdx, получавших AAV9-H1-H-comi-UTRN.
На Фиг. 3 представлены уровни экспрессии мРНК H1-H-Comi-UTRN. Представлено количество копий транскрипта H1-H-Comi-UTRN на 100 нг кДНК в сердце, икроножной мышце (GAS), трицепсе, передней большеберцовой мышце (ТА) и диафрагме.
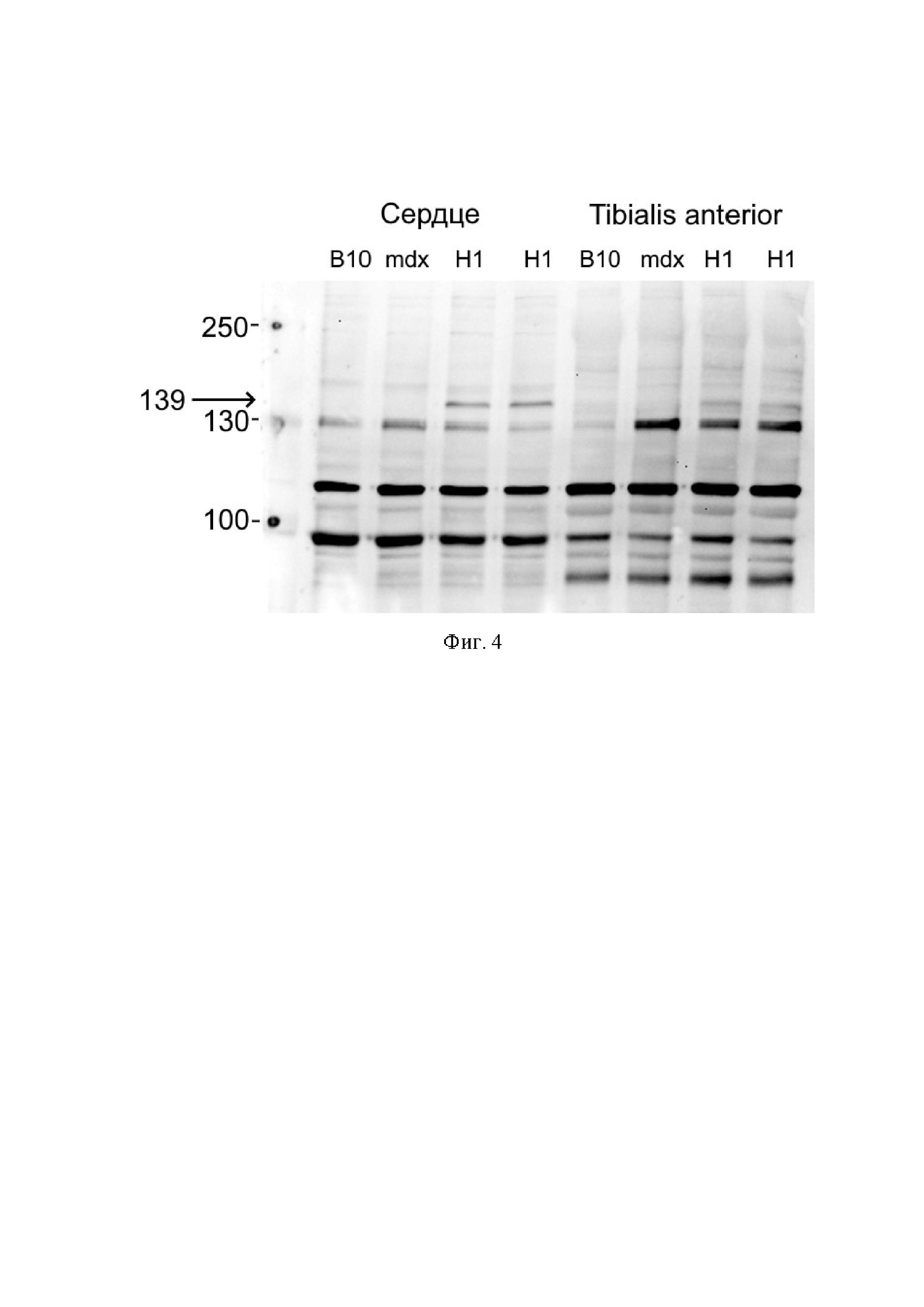
На Фиг. 4 представлена оценка содержания продукта трансляции H1-H-Comi-UTRN (белок H1-H-Comi-UTRN) с помощью вестерн блота в сердечной и передней большеберцовой мышцах мышей. Представлен анализ содержания тотального утрофина в сердце и передней большеберцовой мышце мыши дикого типа (В10), дистрофии-дефицитной мыши, получавшей плацебо (mdx), и двух дистрофии-дефицитных мышей, получавших AAV9-H1-H-Comi-UTRN (HI). Стрелкой указано расчетное положение полосы, соответствующей H1-H-Comi-UTRN (~139 кДа).
На Фиг. 5 представлено распределение альфа-саркогликана в большой передней большеберцовой мышце мыши линии В10 (А); мыши линии mdx, получавшей плацебо (Б) и мыши линии mdx, получавшей AAV9-H1-H-Comi-UTRN (В).
На Фиг. 6 представлены уровни креатинкиназы в сыворотке крови мышей. Измерение уровней креатинкиназы было проведено спустя 5 месяцев после введения тестируемых конструкций. Данные представлены в виде «ящика с усами». Представлены средние значения, квартили и предельные значения для каждой группы. Точками представлены единичные данные. В таблице представлены средние значения для каждой группы со стандартными отклонениями (U/L).
На Фиг. 7 представлена динамика изменения усредненного максимального времени висения на проволоке для мышей линии В10 (В10), получивших плацебо; мышей линии mdx, получивших плацебо (mdx); мышей линии mdx, получивших AAV9-miDMD (DMD); мышей линии mdx, получивших AAV9-H-Comi-UTRN (UTRN), мышей лиинии mdx, получивших AAV9-H1-H-Comi-UTRN (H1) и мышей линии mdx, получивших AAV9-H1-Exl-H-Comi-UTRN (Exl-H1). Представлены стандартные ошибки среднего для каждого измерения. * р-value<0.05 для групп "mdx+DPBS" и "mdx+AAV9-H1-H-Comi-UTRN"; | p-value<0.05 для групп "mdx+DPBS" и "mdx+AAV9-H-Comi-UTRN.
На Фиг. 8 представлены средние значения удельной силы передней большеберцовой мышцы для мышей линии В10; мышей линии mdx, получивших плацебо; мышей линии mdx, получивших AAV9-miDMD; мышей линии mdx, получивших AAV9-H-Comi-UTRN и мышей линии mdx, получивших AAV9-H1-H-Comi-UTRN. Указаны стандартные ошибки среднего для каждой группы.
На Фиг. 9 представлены средние значения площади поперечного сечения (CSA) передней большеберцовой мышцы для мышей линии В10; мышей линии mdx, получивших плацебо; мышей линии mdx, получивших AAV9-miDMD; мышей линии mdx, получивших AAV9-H-Comi-UTRN и мышей линии mdx, получивших AAV9-H1-H-Comi-UTRN. Указаны стандартные ошибки среднего для каждой группы.
На Фиг. 10 представлена динамика снижения силы передней большеберцовой мышцы при эксцентрических сокращениях передней большеберцовой мышцы мышей линии В10; мышей линии mdx, получивших плацебо; мышей линии mdx, получивших AAV9-miDMD (DMD); мышей линии mdx, получивших AAV9-H-Comi-UTRN (UTRN) и мышей линии mdx, получивших AAV9-H1-H-Comi-UTRN (H1). Указаны стандартные ошибки среднего для каждой группы.
Изобретение поясняется следующими примерами
Заявленные химерные белки синтезировались с генетических конструкций, доставленных с помощью аденоассоциированных вирусных экспрессионных векторов in vivo в животной модели миодистрофии Дюшенна: мышах линии mdx, которые не экспрессируют дистрофии и демонстрируют ряд фенотипических признаков, аналогичных симптомам у пациентов с миодистрофией Дюшенна (Bulfield et al. 1984).
Пример 1. Генетические конструкции.
Получение конструкций, использованных в данной работе, производилось с использованием стандартных методов клонирования.
Получение конструкций pAAV-miDMD и pAAV-H-Comi-UTRN. За основу были взяты последовательности, кодирующие полноразмерный дистрофии и полноразмерный утрофин, и представленные в базе данных NCBI: ID NM_004006.3 (Homo sapiens dystrophin (DMD), transcript variant Dp427m, mRNA) и ID: NM_007124.3 (Homo sapiens utrophin (UTRN), transcript variant 1, mRNA). На базе этих последовательностей были получены последовательности, кодирующие «укороченные» варианты дистрофина (ΔR4-R23/ΔCT) из статьи Liu et al., 2005, и утрофина (ΔR2-R21/ΔCT). Для этих последовательностей была проведена оптимизация кодонов (Foster at al, 2008). Синтез последовательностей, фланкированных сайтами рестрикции, заказывали в компании Shanghai ShineGene Molecular Bio-Technologies, Китай. В результате были получены конструкции pMV-miDMD и pMV-H-Comi-UTRN. Далее последовательности, кодирующие miDMD и H-Comi-UTRN, были лигированы в плазмиду pAAV-CMV-eGFP-sv40polyA вместо гена eGFP.
Получение pAAV-H1-H-Comi-UTRN и pAAV-H1-Exl-H-Comi-UTRN. Для получения последовательностей, кодирующих химерные белки Н-Comi-UTRN-H1 и Exl-H-Comi-UTRN-H1, плазмиду pAAV-CMV-H-Comi-UTRN разрезали по сайтам рестрикции BamHI/Eco72I. Фрагмент гена микродистрофина, кодирующий Hinge 1, аплифицировали с матрицы pAAV-CMV-miDMD. Оставшиеся фрагменты гена микроутрофина амплифицировали с матрицы pAAV-CMV-H-Comi-UTRN. Для создания конструкции pAAV-Exl-H1-H-Comi-UTRN амплифицировали фрагмент гена микродистрофина, кодирующий Exon 1. Цитирование всех фрагментов осуществляли по методу Гибсона с использованием набора NEBuilder® HiFi DNA Assembly Cloning Kit.
Аденоассоциированные вирусные экспрессионные векторы серотипа 9 были получены с помощью тройной трансфекции клеток HEK293T (метод широко применяется для получения AAV и многократно описан, например, в работе Xiao et al, 1998. Вкратце, готовится эквимолярная смесь трех плазмид: целевая плазмида, кодирующая одну из вышеуказанных генетических конструкций; плазмида, кодирующая белки аденоассоциированного вируса Rep и Сар; плазмида, кодирующая вспомогательные белки аденовируса (pHelper). Адгезионные клетки линии HEK293T трансфецируются указанной смесью в присутствии линейного полиэтиленимина). Аденоассоциированный вектор был очищен с помощью ультрацентрифугирования в градиенте иодиксанола с последующим диализом.
Для анализа результатов применялись стандартные методы. Для оценки содержания геномной ДНК и копий мРНК трансгена использовалась количественная ПЦР в реальном времени, для оценки содержания химерных белков - продуктов экспрессии, - использовался иммуноблотинг, для оценки содержания и локализации белка дистрофин-ассоциированного протеинового комплекса альфа-саркогликана использовалась иммуногистохимия. Для оценки функциональной эффективности тестируемых конструкций использовались стандартные методы физиологии - оценка уровня креатинфосфокиназы (СК), силы сокращения мышц, поперечного сечения мышц, среднего времени зависания на проволоке.
Пример 2. Использование экспрессионных векторов в мышиной модели миодистрофии Дюшенна.
Аденоассоциированный вирусный вектор, кодирующий химерный белок H1-H-Comi-UTRN (AAV9-H1-H-Comi-UTRN), был введен внутривенно 5-недельным мышам линии mdx в концентрации 6,414 гк/кг. Второй группе мышей линии mdx вводили аденоассоциированный вектор, кодирующий другой химерный белок: H1-Exl-H-Comi-UTRN (AAV9-H1-Exl-H-Comi-UTRN). Третьей группе мышей введен аденоассоциированный вектор, кодирующий H-Comi-UTRN (AAV9-HComi-UTRN). Четвертой группе был введен аденоассоциированный вектор, кодирующий микродистрофин (AAV9-miDMD), аналогичный микродистрофину компании Sarepta). Во всех случаях доза вектора составляла 6,414 гк/кг. Группа мышей линий В10 и еще одна группа мышей линии mdx использовались как контрольные, им было введено плацебо (DPBS). Эксперимент был терминирован через 19 недель после введения.
Пример 3. Эффективность химерных белков.
Эффективность доставки и наличие трансгена оценивалось для контрольных групп и группы мышей, получавших AAV9-H1-H-Comi-UTRN; функциональные параметры оценивались для контрольных групп, группы, получавшей AAV9-H-Comi-UTRN, AAV9-H1-H-Comi-UTRN, AAV9-Exl-H1-H-Comi-UTRN и AAV9-miDMD.
Пример 3.1 Оценка уровня экспрессии генетической конструкции.
В сердце и передней большеберцовой мышце (ТА) методом ПЦР в реальном времени было оценено количество копий вирусного генома. Оно превышало 105 на 1 мкг геномной ДНК для обеих мышц. Так же было оценено количество молекул мРНК трансгена на 100 нг кДНК в сердце, икроножной, передней большеберцовой мышцах, трицепсе и диафрагме. Наибольшее количество копий трансгена было обнаружено в трицепсе и передней большеберцовой мышцах (более 106 копий на 100 нг кДНК). Значительное количество копий целевого транскрипта было зафиксировано в икроножной мышце и сердце (более 105 копий на 100 нг кДНК). Более 103 копий на 100 нг кДНК трансгена было также зафиксировано в диафрагме (Фиг. 3). Таким образом, трансген был обнаружен в пяти различных мышцах, представляющих все три значимых типа мышц, испытывающих деградацию при миодистрофии Дюшенна (сердце, диафрагма, скелетные мышцы).
С помощью метода вестерн-блот было показано наличие белка весом 139 kDa в сердце и большой переднеберцовой мышцах (Фиг. 4). С помощью метода иммуногистохимии с применением антител против мышиного альфа-саркогликана у мышей линии mdx, получивших AAV9-H1-H-comi-UTRN детектировались волокна, в которых наблюдался мембранно-ассоциированный компонент дистрофин-ассоциированного протеинового комплекса альфа-саркогликан, в то время как у мышей линии mdx, получивших плацебо, альфа-саркогликан не детектировался (Фиг. 5). Это указывает на частичное восстановление дистрофин-ассоциированного протеинового комплекса в присутствии трансгена H1-H-comi-UTRN.
Пример 3.2. Оценка функционального состояния мышц в мышиной модели миодистрофии Дюшенна.
Введение конструкции, кодирующей химерный белок - аналог дистрофина, должно приводить к улучшению состояния модельных мышей. Анализ активности креатининфосфокиназы (маркера повреждения мышц при миодистрофии Дюшенна) в сыворотке крови мышей после терминации показал незначительное снижение активности CK у мышей, получивших AAV9-H1-H-comi-UTRN (Фиг. 6).
На всем протяжении эксперимента проводились еженедельные (кроме недели 2 и 9) оценки выносливости мышей - определение времени виса на проволоке (идентификатор СОП по TREAT-NMD: DMD_M.2.1.004). Мыши, получавшие AAV9-H1-H-comi-UTRN, уже с десятой недели эксперимента могли провисеть на проволоке в среднем дольше, чем мыши из группы mdx, получившие плацебо, что говорит о функциональном улучшении состояния мышц животных под действием AAV9-H1-H-comi-UTRN. Аналогичные результаты, однако с несколько меньшей эффективностью, были показаны и для группы мышей, получавшей AAV9-H-Comi-UTRN. Интересно, что мыши, получавшие AAV9-miDMD, демонстрировали лучшие результаты в течение первой половины эксперимента, однако несколько ухудшили результаты ближе к концу эксперимента (при сравнении с AAV9-H-Comi-UTRN). Мыши, получавшие AAV9-H1-Exl-H-comi-UTRN, также демонстрировали несколько лучшее среднее время висения по сравнению с мышами mdx, получавшими плацебо (наиболее заметна эта разница для 15 и 17 недели эксперимента), что позволяет нам уверенно говорить о тренде улучшения функциональности мышц после введения генетических конструкций по настоящему изобретению (Фиг. 7).
После окончания эксперимента была оценена удельная сила передней большеберцовой мышцы (ТА). Удельная сила ТА у мышей, получивших AAV9-H1-H-comi-UTRN (43 кН/мм2), превышала удельную силу ТА контрольных мышей линии mdx (33 кН/мм2), однако была значительно ниже удельной силы ТА мышей В10 (82 кН/мм2). Удельная сила ТА у мышей, получавших AAV9-H-Comi-UTRN (47 кН/мм2), так же превышала удельную силу у мышей линии mdx, получавших плацебо, и незначительно превышала удельную силу ТА у мышей, получивших AAV9-H1-H-comi-UTRN. Удельная сила ТА у мышей, получавших AAV9-miDMD (57 кН/мм2) значительно превышала удельную силу у мышей линии mdx, получавших плацебо, и превышала удельную силу ТА у мышей, получивших AAV9-H1-H-comi-UTRN и AAV9-H-Comi-UTRN (Фиг. 8). При этом в среднем площадь поперечного сечения мышцы ТА у мышей, получивших AAV9-H1-H-comi-UTRN (9.12 мм2) и AAV9-H-comi-UTRN (9.10 мм2) была меньше, чем у мышей линии mdx, получивших DPBS (10.73 мм2). Площадь поперечного сечения ТА у мышей линии mdx, получивших AAV9-miDMD, составляла 8,5 мм2 - несколько ниже, чем у групп, получавших AAV9-H1-H-comi-UTRN и AAV9-H-comi-UTRN. Площадь поперечного сечения ТА мышей линии В10 составила 6,23 мм2 (Фиг. 9).
Была также оценена относительная потеря силы мышцы ТА после серии из 7 эксцентрических сокращений. В то время, как относительная потеря в группе В10 составила примерно 1/3 (31%) от исходной, в группе мышей линии mdx потеря составила 2/3 (69%). Потеря силы мышц в группе мышей, получивших AAV9-H1-H-comi-UTRN (47%), была меньше, чем в группе контрольных линии mdx и незначительно меньше, чем в группе мышей, получавших AAV9-H-comi-UTRN (58%). Интересно, что в группе мышей, получавших AAV9-miDMD, потеря силы мышцы составила 80% (Фиг. 10).
Таким образом, экспериментально было показано, что с экспрессиоиного вектора AAV9-H1-H-Comi-UTRN экспрессируется РНК и белок, частично гомологичный утрофину. Было проведено сравнение функциональности векторов, кодирующих белки микродистрофин (miDMD) и микроутрофин (UTRN), обладающие сходной доменной организацией и в настоящее время рассматриваемые как кандидаты для заместительной терапии, и вектора, кодирующего химерный белок H1-H-Comi-UTRN. Все указанные белки частично снижали степень проявления дистрофии в мышах линии mdx. При оценке удельной силы сокращения мышцы ТА наилучший результат показала конструкция miDMD, в то время как H1 и UTRN были практически идентичны. При оценке снижения эффективности сокращения мышцы в результате эксцентрического сокращения наилучший результат продемонстрировала конструкция H1. Аналогично, H1 продемонстрировала лучший результат при длительной оценке общего физического состояния животных - теста зависания на проволоке. Это достаточно интересное наблюдение, которое может объясняться разной эффективностью встраивания и функционирования этих конструкций в клетках различных мышц и при различных типах нагрузки, так как распределение, экспрессия и стабильность, предположительно, у этих конструкций одинакова (выбраны одинаковый вектор и промотор, концентрации и способы введения идентичны). В тесте зависания на проволоке был также исследован экспрессионный вектор, кодирующий химерный белок Exl-H1-H-Comi-UTRN, который показал эффективность наравне с белком H1-H-Comi-UTRN.
Наиболее важным нам представляется тест зависания на проволоке, так как он является, во-первых, динамическим тестом, позволяющим оценить состояние тестируемых животных на протяжении длительного периода времени, и во-вторых, он является своеобразным отдаленным аналогом физических тестов, применяемых для оценки результатов терапии в клинических испытаниях. При прохождении этого теста задействуются разные группы мышц, к тому же, его результаты зависят и от общего состояния организма (хотя в некоторых случаях это может и нарушить результаты теста).
Таким образом, у мышей линии mdx, получивших AAV9-H1-H-comi-UTRN и AAV9-Exl-H1-H-Comi-UTRN, отмечалось снижение остроты проявления признаков, соответствующих миодистрофии Дюшенна. Белки H1-H-comi-UTRN и Exl-H1-H-Comi-UTRN превосходили по своей эффективности «стандартный» микроутрофин H-comi-UTRN и незначительно уступали, а в некоторых тестах и превосходили «стандартный» микродистрофин miDMD. Это подтверждает возможность применения конструкции H1-H-comi-UTRN или Exl-H1-H-Comi-UTRN для заместительной терапии у пациентов с миодистрофией Дюшенна. Полученные данные подтверждают решение настоящим изобретением поставленной технической задачи, поскольку полученные химерные белки более эффективны, чем микроутрофин, в комплексном тесте на выносливость в мышиной модели миодистрофии Дюшенна.
СПИСОК ЛИТЕРАТУРЫ
Bulfield, G., W.G. Siller, P.A. Wight, and K.J. Moore (1984) X Chromosome-Linked Muscular Dystrophy (mdx) in the Mouse // Proceedings of the National Academy of Sciences of the United States of America 81 (4): 1189-92.
Culligan, K.G., A.J. Mackey, D.M. Finn, P.B. Maguire, and K. Ohlendieck (1998) Role of Dystrophin Isoforms and Associated Proteins in Muscular Dystrophy (review) // International Journal of Molecular Medicine 2 (6): 639-48.
Duan, Dongsheng (2018) Systemic AAV Micro-Dystrophin Gene Therapy for Duchenne Muscular Dystrophy // Molecular Therapy: The Journal of the American Society of Gene Therapy 26 (10): 2337-56.
Ferrer, А., K.E. Wells, and D.J. Wells (2000) Immune Responses to Dystrophin: Implications for Gene Therapy of Duchenne Muscular Dystrophy // Gene Therapy 7 (17): 1439-46.
Foster H, Sharp PS, Athanasopoulos T, Trollet C, Graham IR, Foster K, Wells DJ, Dickson G. Codon and mRNA sequence optimization of microdystrophin transgenes improves expression and physiological outcome in dystrophic mdx mice following AAV2/8 gene transfer. Mol Ther. 2008 Nov; 16(11):1825-32. doi: 10.1038/mt.2008.186. Epub 2008 Sep 2. PMID: 18766174.
Greener MJ, Sewry CA, Muntoni F, Roberts RG. The 3'-untranslated region of the dystrophin gene - conservation and consequences of loss. Eur J Hum Genet. 2002 Jul; 10(7):413-20. doi: 10.1038/sj.ejhg.5200822. PMID: 12107815.
Liu M, Yue Y, Harper SQ, Grange RW, Chamberlain JS, Duan D. (2005) Adeno-associated virus-mediated microdystrophin expression protects young mdx muscle from contraction-induced injury // Molecular Therapy 11 (2): 245-56.
Mendell, Jerry R., Katherine Campbell, Louise Rodino-Klapac, Zarife Sahenk, Chris Shilling, Sarah Lewis, Dawn Bowles, et al. (2010) Dystrophin Immunity in Duchenne's Muscular Dystrophy // The New England Journal of Medicine 363 (15): 1429-37.
Song, Y., Morales, L., Malik, A.S. et al. Non-immunogenic utrophin gene therapy for the treatment of muscular dystrophy animal models // Nature Medicine 25: 1505-1511.
Tinsley, J.M., and K.E. Davies (1993) Utrophin: A Potential Replacement for Dystrophin? // Neuromuscular Disorders: NMD 3 (5-6): 537-39.
Xiao X, Li J, Samulski RJ. Production of high-titer recombinant adeno-associated virus vectors in the absence of helper adenovirus. J Virol. 1998 Mar; 72(3):2224-32. doi: 10.1128/JVI.72.3.2224-2232.1998. PMID: 9499080; PMCID: PMC109519.
--->
Перечень последовательностей
<110> Общество с ограниченной ответственностью «Марлин Биотех»
Marlin Biotech LLC
<120> Химерные белки на основе утрофина и дистрофина человека и
их применение для лечения миодистрофии Дюшенна
<130>
<160> 4
<210> 1
<211> 1209
<212> белок
<213> Искусственная последовательность
<220>
<222> (1)...(1209)
<223> H1-H-Comi-UTRN
<400> 1
Met Ala Lys Tyr Gly Glu His Glu Ala Ser Pro Asp Asn Gly Gln Asn
1 5 10 15
Glu Phe Ser Asp Ile Ile Lys Ser Arg Ser Asp Glu His Asn Asp Val
20 25 30
Gln Lys Lys Thr Phe Thr Lys Trp Ile Asn Ala Arg Phe Ser Lys Ser
35 40 45
Gly Lys Pro Pro Ile Asn Asp Met Phe Thr Asp Leu Lys Asp Gly Arg
50 55 60
Lys Leu Leu Asp Leu Leu Glu Gly Leu Thr Gly Thr Ser Leu Pro Lys
65 70 75 80
Glu Arg Gly Ser Thr Arg Val His Ala Leu Asn Asn Val Asn Arg Val
85 90 95
Leu Gln Val Leu His Gln Asn Asn Val Glu Leu Val Asn Ile Gly Gly
100 105 110
Thr Asp Ile Val Asp Gly Asn His Lys Leu Thr Leu Gly Leu Leu Trp
115 120 125
Ser Ile Ile Leu His Trp Gln Val Lys Asp Val Met Lys Asp Val Met
130 135 140
Ser Asp Leu Gln Gln Thr Asn Ser Glu Lys Ile Leu Leu Ser Trp Val
145 150 155 160
Arg Gln Thr Thr Arg Pro Tyr Ser Gln Val Asn Val Leu Asn Phe Thr
165 170 175
Thr Ser Trp Thr Asp Gly Leu Ala Phe Asn Ala Val Leu His Arg His
180 185 190
Lys Pro Asp Leu Phe Ser Trp Asp Lys Val Val Lys Met Ser Pro Ile
195 200 205
Glu Arg Leu Glu His Ala Phe Ser Lys Ala Gln Thr Tyr Leu Gly Ile
210 215 220
Glu Lys Leu Leu Asp Pro Glu Asp Val Ala Val Gln Leu Pro Asp Lys
225 230 235 240
Lys Ser Ile Ile Met Tyr Leu Thr Ser Leu Phe Glu Val Leu Pro Gln
245 250 255
Gln Val Ser Ile Glu Ala Ile Gln Glu Val Glu Met Leu Pro Arg Pro
260 265 270
Pro Lys Val Thr Lys Glu Glu His Phe Gln Leu His His Gln Met His
275 280 285
Tyr Ser Gln Gln Ile Thr Val Ser Leu Ala Gln Gly Tyr Glu Arg Thr
290 295 300
Ser Ser Pro Lys Pro Arg Phe Lys Ser Tyr Ala Tyr Thr Gln Ala Ala
305 310 315 320
Tyr Val Thr Thr Ser Asp Pro Thr Arg Ser Pro Phe Pro Ser Gln His
325 330 335
Leu Glu Ala Pro Glu Asp Lys Ser Phe Gly Ser Ser Leu Met Glu Ser
340 345 350
Glu Val Asn Leu Asp Ser Tyr Gln Ile Ala Leu Glu Glu Val Leu Thr
355 360 365
Trp Leu Leu Ser Ala Glu Asp Thr Phe Gln Glu Gln Asp Asp Ile Ser
370 375 380
Asp Asp Val Glu Glu Val Lys Asp Gln Phe Ala Thr His Glu Ala Phe
385 390 395 400
Met Met Glu Leu Thr Ala His Gln Ser Ser Val Gly Ser Val Leu Gln
405 410 415
Ala Gly Asn Gln Leu Ile Thr Gln Gly Thr Leu Ser Asp Glu Glu Glu
420 425 430
Phe Glu Ile Gln Glu Gln Met Thr Leu Leu Asn Ala Arg Trp Glu Ala
435 440 445
Leu Arg Val Glu Ser Met Asp Arg Gln Ser Arg Leu His Asp Val Leu
450 455 60
Met Glu Leu Gln Lys Lys Gln Leu Gln Gln Leu Ser Ala Trp Leu Thr
465 470 475 480
Leu Thr Glu Glu Arg Ile Gln Lys Met Glu Thr Cys Pro Leu Asp Asp
485 490 495
Asp Val Lys Ser Leu Gln Lys Leu Leu Glu Glu His Lys Ser Leu Gln
500 505 510
Ser Asp Leu Glu Ala Glu Gln Val Lys Val Asn Ser Leu Thr His Met
515 520 525
Val Val Ile Val Asp Glu Asn Ser Gly Glu Ser Ala Thr Ala Ile Leu
530 535 540
Glu Asp Gln Leu Gln Lys Leu Gly Glu Arg Trp Thr Ala Val Cys Arg
545 550 555 560
Trp Thr Glu Glu Arg Trp Asn Arg Leu Gln Glu Ile Asn Ile Leu Trp
565 570 575
Gln Glu Leu Leu Glu Glu Gln Cys Leu Leu Lys Ala Trp Leu Thr Glu
580 585 590
Lys Glu Glu Ala Leu Asn Lys Val Gln Thr Ser Asn Phe Lys Asp Gln
595 600 605
Lys Glu Leu Ser Val Ser Val Arg Arg Leu Ala Ile Leu Lys Glu Asp
610 615 620
Met Glu Met Lys Arg Gln Thr Leu Asp Gln Leu Ser Glu Ile Gly Gln
625 630 635 640
Asp Val Gly Gln Leu Leu Asp Asn Ser Lys Ala Ser Lys Lys Ile Asn
645 650 655
Ser Asp Ser Glu Glu Leu Thr Gln Arg Trp Asp Ser Leu Val Gln Arg
660 665 670
Leu Glu Asp Ser Ser Asn Gln Val Thr Gln Ala Val Ala Lys Leu Gly
675 680 685
Met Ser Gln Ile Pro Gln Lys Asp Leu Leu Glu Thr Val Arg Val Arg
690 695 700
Glu Gln Ala Ile Thr Lys Lys Ser Lys Gln Glu Leu Pro Pro Pro Pro
705 710 715 720
Pro Pro Lys Lys Arg Gln Ile His Val Asp Leu Glu Lys Leu Arg Asp
725 730 735
Leu Gln Gly Ala Met Asp Asp Leu Asp Ala Asp Met Lys Glu Ala Glu
740 745 750
Ser Val Arg Asn Gly Trp Lys Pro Val Gly Asp Leu Leu Ile Asp Ser
755 760 765
Leu Gln Asp His Ile Glu Lys Ile Met Ala Phe Arg Glu Glu Ile Ala
770 775 780
Pro Ile Asn Phe Lys Val Lys Thr Val Asn Asp Leu Ser Ser Gln Leu
785 790 795 800
Ser Pro Leu Asp Leu His Pro Ser Leu Lys Met Ser Arg Gln Leu Asp
805 810 815
Asp Leu Asn Met Arg Trp Lys Leu Leu Gln Val Ser Val Asp Asp Arg
820 825 830
Leu Lys Gln Leu Gln Glu Ala His Arg Asp Phe Gly Pro Ser Ser Gln
835 840 845
His Phe Leu Ser Thr Ser Val Gln Leu Pro Trp Gln Arg Ser Ile Ser
850 855 860
His Asn Lys Val Pro Tyr Tyr Ile Asn His Gln Thr Gln Thr Thr Cys
865 870 875 880
Trp Asp His Pro Lys Met Thr Glu Leu Phe Gln Ser Leu Ala Asp Leu
885 890 895
Asn Asn Val Arg Phe Ser Ala Tyr Arg Thr Ala Ile Lys Ile Arg Arg
900 905 910
Leu Gln Lys Ala Leu Cys Leu Asp Leu Leu Glu Leu Ser Thr Thr Asn
915 920 925
Glu Ile Phe Lys Gln His Lys Leu Asn Gln Asn Asp Gln Leu Leu Ser
930 935 940
Val Pro Asp Val Ile Asn Cys Leu Thr Thr Thr Tyr Asp Gly Leu Glu
945 950 955 960
Gln Met His Lys Asp Leu Val Asn Val Pro Leu Cys Val Asp Met Cys
965 970 975
Leu Asn Trp Leu Leu Asn Val Tyr Asp Thr Gly Arg Thr Gly Lys Ile
980 985 990
Arg Val Gln Ser Leu Lys Ile Gly Leu Met Ser Leu Ser Lys Gly Leu
995 1000 1005
Leu Glu Glu Lys Tyr Arg Tyr Leu Phe Lys Glu Val Ala Gly Pro Thr
1010 1015 1020
Glu Met Cys Asp Gln Arg Gln Leu Gly Leu Leu Leu His Asp Ala Ile
1025 1030 1035 1040
Gln Ile Pro Arg Gln Leu Gly Glu Val Ala Ala Phe Gly Gly Ser Asn
1045 1050 1055
Ile Glu Pro Ser Val Arg Ser Cys Phe Gln Gln Asn Asn Asn Lys Pro
1060 1065 1070
Glu Ile Ser Val Lys Glu Phe Ile Asp Trp Met His Leu Glu Pro Gln
1075 1080 1085
Ser Met Val Trp Leu Pro Val Leu His Arg Val Ala Ala Ala Glu Thr
1090 1095 1100
Ala Lys His Gln Ala Lys Cys Asn Ile Cys Lys Glu Cys Pro Ile Val
1105 1110 1115 1120
Gly Phe Arg Tyr Arg Ser Leu Lys His Phe Asn Tyr Asp Val Cys Gln
1125 1130 1135
Ser Cys Phe Phe Ser Gly Arg Thr Ala Lys Gly His Lys Leu His Tyr
1140 1145 1150
Pro Met Val Glu Tyr Cys Ile Pro Thr Thr Ser Gly Glu Asp Val Arg
1155 1160 1165
Asp Phe Thr Lys Val Leu Lys Asn Lys Phe Arg Ser Lys Lys Tyr Phe
1170 1175 1180
Ala Lys His Pro Arg Leu Gly Tyr Leu Pro Val Gln Thr Val Leu Glu
1185 1190 1195 1200
Gly Asp Asn Leu Glu Thr Gln Ala Met
1205 1209
<210> 2
<211> 3630
<212> ДНК
<213> Искусственная последовательность
<220>
<222> (1)...(3630)
<223> H1-H-Comi-UTRN gene.
<400> 2
ATG GCC AAG TAC GGC GAG CAC GAG GCC TCC CCC GAC AAC GGC CAG AAC 48
Met Ala Lys Tyr Gly Glu His Glu Ala Ser Pro Asp Asn Gly Gln Asn
1 5 10 15
GAG TTC TCC GAC ATC ATC AAG TCC CGC TCC GAC GAG CAC AAC GAC GTG 96
Glu Phe Ser Asp Ile Ile Lys Ser Arg Ser Asp Glu His Asn Asp Val
20 25 30
CAG AAG AAG ACC TTC ACC AAG TGG ATC AAC GCC CGC TTC TCC AAG TCC 144
Gln Lys Lys Thr Phe Thr Lys Trp Ile Asn Ala Arg Phe Ser Lys Ser
35 40 45
GGC AAG CCC CCC ATC AAC GAC ATG TTC ACC GAC CTG AAG GAC GGC CGC 192
Gly Lys Pro Pro Ile Asn Asp Met Phe Thr Asp Leu Lys Asp Gly Arg
50 55 60
AAG CTG CTG GAC CTG CTG GAG GGC CTG ACC GGC ACC TCC CTG CCC AAG 240
Lys Leu Leu Asp Leu Leu Glu Gly Leu Thr Gly Thr Ser Leu Pro Lys
65 70 75 80
GAG CGC GGC TCC ACC CGC GTG CAC GCC CTG AAC AAC GTG AAC CGC GTG 288
Glu Arg Gly Ser Thr Arg Val His Ala Leu Asn Asn Val Asn Arg Val
85 90 95
CTG CAG GTG CTG CAC CAG AAC AAC GTG GAG CTG GTG AAC ATC GGC GGC 336
Leu Gln Val Leu His Gln Asn Asn Val Glu Leu Val Asn Ile Gly Gly
100 105 110
ACC GAC ATC GTG GAC GGC AAC CAC AAG CTG ACC CTG GGC CTG CTG TGG 384
Thr Asp Ile Val Asp Gly Asn His Lys Leu Thr Leu Gly Leu Leu Trp
115 120 125
TCC ATC ATC CTG CAC TGG CAG GTG AAG GAC GTG ATG AAG GAC GTG ATG 432
Ser Ile Ile Leu His Trp Gln Val Lys Asp Val Met Lys Asp Val Met
130 135 140
TCC GAC CTG CAG CAG ACC AAC TCC GAG AAG ATC CTG CTG TCC TGG GTG 480
Ser Asp Leu Gln Gln Thr Asn Ser Glu Lys Ile Leu Leu Ser Trp Val
145 150 155 160
CGC CAG ACC ACC CGC CCC TAC TCC CAG GTG AAC GTG CTG AAC TTC ACC 528
Arg Gln Thr Thr Arg Pro Tyr Ser Gln Val Asn Val Leu Asn Phe Thr
165 170 175
ACC TCC TGG ACC GAC GGC CTG GCC TTC AAC GCC GTG CTG CAC CGC CAC 576
Thr Ser Trp Thr Asp Gly Leu Ala Phe Asn Ala Val Leu His Arg His
180 185 190
AAG CCC GAC CTG TTC TCC TGG GAC AAG GTG GTG AAG ATG TCC CCC ATC 624
Lys Pro Asp Leu Phe Ser Trp Asp Lys Val Val Lys Met Ser Pro Ile
195 200 205
GAG CGC CTG GAG CAC GCC TTC TCC AAG GCC CAG ACC TAC CTG GGC ATC 672
Glu Arg Leu Glu His Ala Phe Ser Lys Ala Gln Thr Tyr Leu Gly Ile
210 215 220
GAG AAG CTG CTG GAC CCC GAG GAC GTG GCC GTG CAG CTG CCC GAC AAG 720
Glu Lys Leu Leu Asp Pro Glu Asp Val Ala Val Gln Leu Pro Asp Lys
225 230 235 240
AAG TCC ATC ATC ATG TAC CTG ACC TCC CTG TTC GAG GTG CTG CCC CAG 768
Lys Ser Ile Ile Met Tyr Leu Thr Ser Leu Phe Glu Val Leu Pro Gln
245 250 255
CAG GTG TCC ATC GAG GCC ATC CAG GAA GTG GAA ATG CTG CCC AGG CCC 816
Gln Val Ser Ile Glu Ala Ile Gln Glu Val Glu Met Leu Pro Arg Pro
260 265 270
CCC AAA GTG ACC AAG GAG GAG CAC TTC CAG CTG CAC CAC CAG ATG CAC 864
Pro Lys Val Thr Lys Glu Glu His Phe Gln Leu His His Gln Met His
275 280 285
TAT AGC CAG CAG ATC ACC GTG TCC CTG GCC CAG GGC TAT GAG AGA ACC 912
Tyr Ser Gln Gln Ile Thr Val Ser Leu Ala Gln Gly Tyr Glu Arg Thr
290 295 300
AGC AGC CCC AAG CCC AGA TTC AAG AGC TAC GCC TAC ACC CAG GCC GCC 960
Ser Ser Pro Lys Pro Arg Phe Lys Ser Tyr Ala Tyr Thr Gln Ala Ala
305 310 315 320
TAC GTG ACC ACC TCC GAC CCC ACC AGA AGC CCC TTC CCC AGC CAG CAC 1008
Tyr Val Thr Thr Ser Asp Pro Thr Arg Ser Pro Phe Pro Ser Gln His
325 330 335
CTG GAG GCC CCC GAG GAC AAG AGC TTC GGC AGC AGC CTG ATG GAG AGC 1056
Leu Glu Ala Pro Glu Asp Lys Ser Phe Gly Ser Ser Leu Met Glu Ser
340 345 350
GAA GTG AAC CTG GAC TCC TAC CAG ATC GCC CTG GAG GAG GTG CTG ACC 1104
Glu Val Asn Leu Asp Ser Tyr Gln Ile Ala Leu Glu Glu Val Leu Thr
355 360 365
TGG CTG CTG TCC GCC GAG GAC ACC TTC CAG GAG CAG GAC GAC ATC TCC 1152
Trp Leu Leu Ser Ala Glu Asp Thr Phe Gln Glu Gln Asp Asp Ile Ser
370 375 380
GAC GAC GTG GAG GAG GTG AAG GAC CAG TTC GCC ACC CAC GAG GCC TTC 1200
Asp Asp Val Glu Glu Val Lys Asp Gln Phe Ala Thr His Glu Ala Phe
385 390 395 400
ATG ATG GAG CTG ACC GCC CAC CAG TCC TCC GTG GGC TCC GTG CTG CAG 1248
Met Met Glu Leu Thr Ala His Gln Ser Ser Val Gly Ser Val Leu Gln
405 410 415
GCC GGC AAC CAG CTG ATC ACC CAG GGC ACC CTG TCC GAC GAG GAG GAG 1296
Ala Gly Asn Gln Leu Ile Thr Gln Gly Thr Leu Ser Asp Glu Glu Glu
420 425 430
TTC GAG ATC CAG GAG CAG ATG ACC CTG CTG AAC GCC CGC TGG GAG GCC 1344
Phe Glu Ile Gln Glu Gln Met Thr Leu Leu Asn Ala Arg Trp Glu Ala
435 440 445
CTG CGC GTG GAG TCC ATG GAC CGC CAG TCC CGC CTG CAC GAC GTG CTG 1392
Leu Arg Val Glu Ser Met Asp Arg Gln Ser Arg Leu His Asp Val Leu
450 455 460
ATG GAG CTG CAG AAG AAG CAG CTG CAG CAG CTG TCC GCC TGG CTG ACC 1440
Met Glu Leu Gln Lys Lys Gln Leu Gln Gln Leu Ser Ala Trp Leu Thr
465 470 475 480
CTG ACC GAG GAG CGC ATC CAG AAG ATG GAG ACC TGC CCC CTG GAC GAC 1488
Leu Thr Glu Glu Arg Ile Gln Lys Met Glu Thr Cys Pro Leu Asp Asp
485 490 495
GAC GTG AAG TCC CTG CAG AAG CTG CTG GAG GAG CAC AAG TCC CTG CAG 1536
Asp Val Lys Ser Leu Gln Lys Leu Leu Glu Glu His Lys Ser Leu Gln
500 505 510
TCC GAC CTG GAG GCC GAG CAG GTG AAG GTG AAC TCC CTG ACC CAC ATG 1584
Ser Asp Leu Glu Ala Glu Gln Val Lys Val Asn Ser Leu Thr His Met
515 520 525
GTG GTG ATC GTG GAC GAG AAC TCC GGC GAG TCC GCC ACC GCC ATC CTG 1632
Val Val Ile Val Asp Glu Asn Ser Gly Glu Ser Ala Thr Ala Ile Leu
530 535 540
GAG GAC CAG CTG CAG AAG CTG GGC GAG CGC TGG ACC GCC GTG TGC CGC 1680
Glu Asp Gln Leu Gln Lys Leu Gly Glu Arg Trp Thr Ala Val Cys Arg
545 550 555 560
TGG ACC GAG GAG CGC TGG AAC CGC CTG CAG GAG ATC AAC ATC CTG TGG 1728
Trp Thr Glu Glu Arg Trp Asn Arg Leu Gln Glu Ile Asn Ile Leu Trp
565 570 575
CAG GAG CTG CTG GAG GAG CAG TGC CTG CTG AAG GCC TGG CTG ACC GAG 1776
Gln Glu Leu Leu Glu Glu Gln Cys Leu Leu Lys Ala Trp Leu Thr Glu
580 585 590
AAG GAG GAG GCC CTG AAC AAG GTG CAG ACC TCC AAC TTC AAG GAC CAG 1824
Lys Glu Glu Ala Leu Asn Lys Val Gln Thr Ser Asn Phe Lys Asp Gln
595 600 605
AAG GAG CTG TCC GTG TCC GTG CGC CGC CTG GCC ATC CTG AAG GAG GAC 1872
Lys Glu Leu Ser Val Ser Val Arg Arg Leu Ala Ile Leu Lys Glu Asp
610 615 620
ATG GAG ATG AAG CGC CAG ACC CTG GAC CAG CTG TCC GAG ATC GGC CAG 1920
Met Glu Met Lys Arg Gln Thr Leu Asp Gln Leu Ser Glu Ile Gly Gln
625 630 635 640
GAC GTG GGC CAG CTG CTG GAC AAC TCC AAG GCC TCC AAG AAG ATC AAC 1968
Asp Val Gly Gln Leu Leu Asp Asn Ser Lys Ala Ser Lys Lys Ile Asn
645 650 655
TCC GAC TCC GAG GAG CTG ACC CAG CGC TGG GAC TCC CTG GTG CAG CGC 2016
Ser Asp Ser Glu Glu Leu Thr Gln Arg Trp Asp Ser Leu Val Gln Arg
660 665 670
CTG GAG GAC TCC TCC AAC CAG GTG ACC CAG GCC GTG GCC AAG CTG GGC 2064
Leu Glu Asp Ser Ser Asn Gln Val Thr Gln Ala Val Ala Lys Leu Gly
675 680 685
ATG TCC CAG ATC CCC CAG AAG GAC CTG CTG GAG ACC GTG CGC GTG CGC 2112
Met Ser Gln Ile Pro Gln Lys Asp Leu Leu Glu Thr Val Arg Val Arg
690 695 700
GAG CAG GCC ATC ACC AAG AAG TCC AAG CAG GAG CTG CCC CCC CCC CCC 2160
Glu Gln Ala Ile Thr Lys Lys Ser Lys Gln Glu Leu Pro Pro Pro Pro
705 710 715 720
CCC CCC AAG AAG CGC CAG ATC CAC GTG GAC CTG GAG AAG CTG CGC GAC 2208
Pro Pro Lys Lys Arg Gln Ile His Val Asp Leu Glu Lys Leu Arg Asp
725 730 735
CTG CAG GGC GCC ATG GAC GAC CTG GAC GCC GAC ATG AAG GAG GCC GAG 2256
Leu Gln Gly Ala Met Asp Asp Leu Asp Ala Asp Met Lys Glu Ala Glu
740 745 750
TCC GTG CGC AAC GGC TGG AAG CCC GTG GGC GAC CTG CTG ATC GAC TCC 2304
Ser Val Arg Asn Gly Trp Lys Pro Val Gly Asp Leu Leu Ile Asp Ser
755 760 765
CTG CAG GAC CAC ATC GAG AAG ATC ATG GCC TTC CGC GAG GAG ATC GCC 2352
Leu Gln Asp His Ile Glu Lys Ile Met Ala Phe Arg Glu Glu Ile Ala
770 775 780
CCC ATC AAC TTC AAG GTG AAG ACC GTG AAC GAC CTG TCC TCC CAG CTG 2400
Pro Ile Asn Phe Lys Val Lys Thr Val Asn Asp Leu Ser Ser Gln Leu
785 790 795 800
TCC CCC CTG GAC CTG CAC CCC TCC CTG AAG ATG TCC CGC CAG CTG GAC 2448
Ser Pro Leu Asp Leu His Pro Ser Leu Lys Met Ser Arg Gln Leu Asp
805 810 815
GAC CTG AAC ATG CGC TGG AAG CTG CTG CAG GTG TCC GTG GAC GAC CGC 2496
Asp Leu Asn Met Arg Trp Lys Leu Leu Gln Val Ser Val Asp Asp Arg
820 825 830
CTG AAG CAG CTG CAG GAG GCC CAC CGC GAC TTC GGC CCC TCC TCC CAG 2544
Leu Lys Gln Leu Gln Glu Ala His Arg Asp Phe Gly Pro Ser Ser Gln
835 840 845
CAC TTC CTG TCC ACC TCC GTG CAG CTG CCC TGG CAG CGC TCC ATC TCC 2592
His Phe Leu Ser Thr Ser Val Gln Leu Pro Trp Gln Arg Ser Ile Ser
850 855 860
CAC AAC AAG GTG CCC TAC TAC ATC AAC CAC CAG ACC CAG ACC ACC TGC 2640
His Asn Lys Val Pro Tyr Tyr Ile Asn His Gln Thr Gln Thr Thr Cys
865 870 875 880
TGG GAC CAC CCC AAG ATG ACC GAG CTG TTC CAG TCC CTG GCC GAC CTG 2688
Trp Asp His Pro Lys Met Thr Glu Leu Phe Gln Ser Leu Ala Asp Leu
885 890 895
AAC AAC GTG CGC TTC TCC GCC TAC CGC ACC GCC ATC AAG ATC CGC CGC 2736
Asn Asn Val Arg Phe Ser Ala Tyr Arg Thr Ala Ile Lys Ile Arg Arg
900 905 910
CTG CAG AAG GCC CTG TGC CTG GAC CTG CTG GAG CTG TCC ACC ACC AAC 2784
Leu Gln Lys Ala Leu Cys Leu Asp Leu Leu Glu Leu Ser Thr Thr Asn
915 920 925
GAG ATC TTC AAG CAG CAC AAG CTG AAC CAG AAC GAC CAG CTG CTG TCC 2832
Glu Ile Phe Lys Gln His Lys Leu Asn Gln Asn Asp Gln Leu Leu Ser
930 935 940
GTG CCC GAC GTG ATC AAC TGC CTG ACC ACC ACC TAC GAC GGC CTG GAG 2880
Val Pro Asp Val Ile Asn Cys Leu Thr Thr Thr Tyr Asp Gly Leu Glu
945 950 955 960
CAG ATG CAC AAG GAC CTG GTG AAC GTG CCC CTG TGC GTG GAC ATG TGC 2928
Gln Met His Lys Asp Leu Val Asn Val Pro Leu Cys Val Asp Met Cys
965 970 975
CTG AAC TGG CTG CTG AAC GTG TAC GAC ACC GGC CGC ACC GGC AAG ATC 2976
Leu Asn Trp Leu Leu Asn Val Tyr Asp Thr Gly Arg Thr Gly Lys Ile
980 985 990
CGC GTG CAG TCC CTG AAG ATC GGC CTG ATG TCC CTG TCC AAG GGC CTG 3024
Arg Val Gln Ser Leu Lys Ile Gly Leu Met Ser Leu Ser Lys Gly Leu
995 1000 1005
CTG GAG GAG AAG TAC CGC TAC CTG TTC AAG GAG GTG GCC GGC CCC ACC 3072
Leu Glu Glu Lys Tyr Arg Tyr Leu Phe Lys Glu Val Ala Gly Pro Thr
1010 1015 1020
GAG ATG TGC GAC CAG CGC CAG CTG GGC CTG CTG CTG CAC GAC GCC ATC 3120
Glu Met Cys Asp Gln Arg Gln Leu Gly Leu Leu Leu His Asp Ala Ile
1025 1030 1035 1040
CAG ATC CCC CGC CAG CTG GGC GAG GTG GCC GCC TTC GGC GGC TCC AAC 3168
Gln Ile Pro Arg Gln Leu Gly Glu Val Ala Ala Phe Gly Gly Ser Asn
1045 1050 1055
ATC GAG CCC TCC GTG CGC TCC TGC TTC CAG CAG AAC AAC AAC AAG CCC 3216
Ile Glu Pro Ser Val Arg Ser Cys Phe Gln Gln Asn Asn Asn Lys Pro
1060 1065 1070
GAG ATC TCC GTG AAG GAG TTC ATC GAC TGG ATG CAC CTG GAG CCC CAG 3264
Glu Ile Ser Val Lys Glu Phe Ile Asp Trp Met His Leu Glu Pro Gln
1075 1080 1085
TCC ATG GTG TGG CTG CCC GTG CTG CAC CGC GTG GCC GCC GCC GAG ACC 3312
Ser Met Val Trp Leu Pro Val Leu His Arg Val Ala Ala Ala Glu Thr
1090 1095 1100
GCC AAG CAC CAG GCC AAG TGC AAC ATC TGC AAG GAG TGC CCC ATC GTG 3360
Ala Lys His Gln Ala Lys Cys Asn Ile Cys Lys Glu Cys Pro Ile Val
1105 1110 1115 1120
GGC TTC CGC TAC CGC TCC CTG AAG CAC TTC AAC TAC GAC GTG TGC CAG 3408
Gly Phe Arg Tyr Arg Ser Leu Lys His Phe Asn Tyr Asp Val Cys Gln
1125 1130 1135
TCC TGC TTC TTC TCC GGC CGC ACC GCC AAG GGC CAC AAG CTG CAC TAC 3456
Ser Cys Phe Phe Ser Gly Arg Thr Ala Lys Gly His Lys Leu His Tyr
1140 1145 1150
CCC ATG GTG GAG TAC TGC ATC CCC ACC ACC TCC GGC GAG GAC GTG CGC 3504
Pro Met Val Glu Tyr Cys Ile Pro Thr Thr Ser Gly Glu Asp Val Arg
1155 1160 1165
GAC TTC ACC AAG GTG CTG AAG AAC AAG TTC CGC TCC AAG AAG TAC TTC 3552
Asp Phe Thr Lys Val Leu Lys Asn Lys Phe Arg Ser Lys Lys Tyr Phe
1170 1175 1180
GCC AAG CAC CCC CGC CTG GGC TAC CTG CCC GTG CAG ACC GTG CTG GAG 3600
Ala Lys His Pro Arg Leu Gly Tyr Leu Pro Val Gln Thr Val Leu Glu
1185 1190 1195 1200
GGC GAC AAC CTG GAG ACC CAG GCC ATG TGA 3630
Gly Asp Asn Leu Glu Thr Gln Ala Met ***
1205 1209
<210> 3
<211> 1193
<212> белок
<213> Искусственная последовательность
<220>
<222> (1)...(1193)
<223> EX1-H1-H-Comi-UTRN
<400> 3
Met Leu Trp Trp Glu Glu Val Glu Asp Cys Tyr Glu Arg Glu Asp Val
1 5 10 15
Gln Lys Lys Thr Phe Thr Lys Trp Ile Asn Ala Arg Phe Ser Lys Ser
20 25 30
Gly Lys Pro Pro Ile Asn Asp Met Phe Thr Asp Leu Lys Asp Gly Arg
35 40 45
Lys Leu Leu Asp Leu Leu Glu Gly Leu Thr Gly Thr Ser Leu Pro Lys
50 55 60
Glu Arg Gly Ser Thr Arg Val His Ala Leu Asn Asn Val Asn Arg Val
65 70 75 80
Leu Gln Val Leu His Gln Asn Asn Val Glu Leu Val Asn Ile Gly Gly
85 90 95
Thr Asp Ile Val Asp Gly Asn His Lys Leu Thr Leu Gly Leu Leu Trp
100 105 110
Ser Ile Ile Leu His Trp Gln Val Lys Asp Val Met Lys Asp Val Met
115 120 125
Ser Asp Leu Gln Gln Thr Asn Ser Glu Lys Ile Leu Leu Ser Trp Val
130 135 140
Arg Gln Thr Thr Arg Pro Tyr Ser Gln Val Asn Val Leu Asn Phe Thr
145 150 155 160
Thr Ser Trp Thr Asp Gly Leu Ala Phe Asn Ala Val Leu His Arg His
165 170 175
Lys Pro Asp Leu Phe Ser Trp Asp Lys Val Val Lys Met Ser Pro Ile
180 185 190
Glu Arg Leu Glu His Ala Phe Ser Lys Ala Gln Thr Tyr Leu Gly Ile
195 200 205
Glu Lys Leu Leu Asp Pro Glu Asp Val Ala Val Gln Leu Pro Asp Lys
210 215 220
Lys Ser Ile Ile Met Tyr Leu Thr Ser Leu Phe Glu Val Leu Pro Gln
225 230 235 240
Gln Val Ser Ile Glu Ala Ile Gln Glu Val Glu Met Leu Pro Arg Pro
245 250 255
Pro Lys Val Thr Lys Glu Glu His Phe Gln Leu His His Gln Met His
260 265 270
Tyr Ser Gln Gln Ile Thr Val Ser Leu Ala Gln Gly Tyr Glu Arg Thr
275 280 285
Ser Ser Pro Lys Pro Arg Phe Lys Ser Tyr Ala Tyr Thr Gln Ala Ala
290 295 300
Tyr Val Thr Thr Ser Asp Pro Thr Arg Ser Pro Phe Pro Ser Gln His
305 310 315 320
Leu Glu Ala Pro Glu Asp Lys Ser Phe Gly Ser Ser Leu Met Glu Ser
325 330 335
Glu Val Asn Leu Asp Ser Tyr Gln Ile Ala Leu Glu Glu Val Leu Thr
340 345 350
Trp Leu Leu Ser Ala Glu Asp Thr Phe Gln Glu Gln Asp Asp Ile Ser
355 360 365
Asp Asp Val Glu Glu Val Lys Asp Gln Phe Ala Thr His Glu Ala Phe
370 375 380
Met Met Glu Leu Thr Ala His Gln Ser Ser Val Gly Ser Val Leu Gln
385 390 395 400
Ala Gly Asn Gln Leu Ile Thr Gln Gly Thr Leu Ser Asp Glu Glu Glu
405 410 415
Phe Glu Ile Gln Glu Gln Met Thr Leu Leu Asn Ala Arg Trp Glu Ala
420 425 430
Leu Arg Val Glu Ser Met Asp Arg Gln Ser Arg Leu His Asp Val Leu
435 440 445
Met Glu Leu Gln Lys Lys Gln Leu Gln Gln Leu Ser Ala Trp Leu Thr
450 455 460
Leu Thr Glu Glu Arg Ile Gln Lys Met Glu Thr Cys Pro Leu Asp Asp
465 470 475 480
Asp Val Lys Ser Leu Gln Lys Leu Leu Glu Glu His Lys Ser Leu Gln
485 490 495
Ser Asp Leu Glu Ala Glu Gln Val Lys Val Asn Ser Leu Thr His Met
500 505 510
Val Val Ile Val Asp Glu Asn Ser Gly Glu Ser Ala Thr Ala Ile Leu
515 520 525
Glu Asp Gln Leu Gln Lys Leu Gly Glu Arg Trp Thr Ala Val Cys Arg
530 535 540
Trp Thr Glu Glu Arg Trp Asn Arg Leu Gln Glu Ile Asn Ile Leu Trp
545 550 555 560
Gln Glu Leu Leu Glu Glu Gln Cys Leu Leu Lys Ala Trp Leu Thr Glu
565 570 575
Lys Glu Glu Ala Leu Asn Lys Val Gln Thr Ser Asn Phe Lys Asp Gln
580 585 590
Lys Glu Leu Ser Val Ser Val Arg Arg Leu Ala Ile Leu Lys Glu Asp
595 600 605
Met Glu Met Lys Arg Gln Thr Leu Asp Gln Leu Ser Glu Ile Gly Gln
610 615 620
Asp Val Gly Gln Leu Leu Asp Asn Ser Lys Ala Ser Lys Lys Ile Asn
625 630 635 640
Ser Asp Ser Glu Glu Leu Thr Gln Arg Trp Asp Ser Leu Val Gln Arg
645 650 655
Leu Glu Asp Ser Ser Asn Gln Val Thr Gln Ala Val Ala Lys Leu Gly
660 665 670
Met Ser Gln Ile Pro Gln Lys Asp Leu Leu Glu Thr Val Arg Val Arg
675 680 685
Glu Gln Ala Ile Thr Lys Lys Ser Lys Gln Glu Leu Pro Pro Pro Pro
690 695 700
Pro Pro Lys Lys Arg Gln Ile His Val Asp Leu Glu Lys Leu Arg Asp
705 710 715 720
Leu Gln Gly Ala Met Asp Asp Leu Asp Ala Asp Met Lys Glu Ala Glu
725 730 735
Ser Val Arg Asn Gly Trp Lys Pro Val Gly Asp Leu Leu Ile Asp Ser
740 745 750
Leu Gln Asp His Ile Glu Lys Ile Met Ala Phe Arg Glu Glu Ile Ala
755 760 765
Pro Ile Asn Phe Lys Val Lys Thr Val Asn Asp Leu Ser Ser Gln Leu
770 775 780
Ser Pro Leu Asp Leu His Pro Ser Leu Lys Met Ser Arg Gln Leu Asp
785 790 795 800
Asp Leu Asn Met Arg Trp Lys Leu Leu Gln Val Ser Val Asp Asp Arg
805 810 815
Leu Lys Gln Leu Gln Glu Ala His Arg Asp Phe Gly Pro Ser Ser Gln
820 825 830
His Phe Leu Ser Thr Ser Val Gln Leu Pro Trp Gln Arg Ser Ile Ser
835 840 845
His Asn Lys Val Pro Tyr Tyr Ile Asn His Gln Thr Gln Thr Thr Cys
850 855 860
Trp Asp His Pro Lys Met Thr Glu Leu Phe Gln Ser Leu Ala Asp Leu
865 870 875 880
Asn Asn Val Arg Phe Ser Ala Tyr Arg Thr Ala Ile Lys Ile Arg Arg
885 890 895
Leu Gln Lys Ala Leu Cys Leu Asp Leu Leu Glu Leu Ser Thr Thr Asn
900 905 910
Glu Ile Phe Lys Gln His Lys Leu Asn Gln Asn Asp Gln Leu Leu Ser
915 920 925
Val Pro Asp Val Ile Asn Cys Leu Thr Thr Thr Tyr Asp Gly Leu Glu
930 935 940
Gln Met His Lys Asp Leu Val Asn Val Pro Leu Cys Val Asp Met Cys
945 950 955 960
Leu Asn Trp Leu Leu Asn Val Tyr Asp Thr Gly Arg Thr Gly Lys Ile
965 970 975
Arg Val Gln Ser Leu Lys Ile Gly Leu Met Ser Leu Ser Lys Gly Leu
980 985 990
Leu Glu Glu Lys Tyr Arg Tyr Leu Phe Lys Glu Val Ala Gly Pro Thr
995 1000 1005
Glu Met Cys Asp Gln Arg Gln Leu Gly Leu Leu Leu His Asp Ala Ile
1010 1015 1020
Gln Ile Pro Arg Gln Leu Gly Glu Val Ala Ala Phe Gly Gly Ser Asn
1025 1030 1035 1040
Ile Glu Pro Ser Val Arg Ser Cys Phe Gln Gln Asn Asn Asn Lys Pro
1045 1050 1055
Glu Ile Ser Val Lys Glu Phe Ile Asp Trp Met His Leu Glu Pro Gln
1060 1065 1070
Ser Met Val Trp Leu Pro Val Leu His Arg Val Ala Ala Ala Glu Thr
1075 1080 1085
Ala Lys His Gln Ala Lys Cys Asn Ile Cys Lys Glu Cys Pro Ile Val
1090 1095 1100
Gly Phe Arg Tyr Arg Ser Leu Lys His Phe Asn Tyr Asp Val Cys Gln
1105 1110 1115 1120
Ser Cys Phe Phe Ser Gly Arg Thr Ala Lys Gly His Lys Leu His Tyr
1125 1130 1135
Pro Met Val Glu Tyr Cys Ile Pro Thr Thr Ser Gly Glu Asp Val Arg
1140 1145 1150
Asp Phe Thr Lys Val Leu Lys Asn Lys Phe Arg Ser Lys Lys Tyr Phe
1155 1160 1165
Ala Lys His Pro Arg Leu Gly Tyr Leu Pro Val Gln Thr Val Leu Glu
1170 1175 1180
Gly Asp Asn Leu Glu Thr Gln Ala Met ***
1185 1190 1193
<210> 4
<211> 3582
<212> ДНК
<213> Искусственная последовательность
<220>
<222> (1)...(3582)
<223> EX1-H1-H-Comi-UTRN gene
<400> 4
ATG CTG TGG TGG GAG GAA GTG GAG GAC TGC TAC GAG AGA GAG GAC GTG 48
Met Leu Trp Trp Glu Glu Val Glu Asp Cys Tyr Glu Arg Glu Asp Val
1 5 10 15
CAG AAG AAG ACC TTC ACC AAG TGG ATC AAC GCC CGC TTC TCC AAG TCC 96
Gln Lys Lys Thr Phe Thr Lys Trp Ile Asn Ala Arg Phe Ser Lys Ser
20 25 30
GGC AAG CCC CCC ATC AAC GAC ATG TTC ACC GAC CTG AAG GAC GGC CGC 144
Gly Lys Pro Pro Ile Asn Asp Met Phe Thr Asp Leu Lys Asp Gly Arg
35 40 45
AAG CTG CTG GAC CTG CTG GAG GGC CTG ACC GGC ACC TCC CTG CCC AAG 192
Lys Leu Leu Asp Leu Leu Glu Gly Leu Thr Gly Thr Ser Leu Pro Lys
50 55 60
GAG CGC GGC TCC ACC CGC GTG CAC GCC CTG AAC AAC GTG AAC CGC GTG 240
Glu Arg Gly Ser Thr Arg Val His Ala Leu Asn Asn Val Asn Arg Val
65 70 75 80
CTG CAG GTG CTG CAC CAG AAC AAC GTG GAG CTG GTG AAC ATC GGC GGC 288
Leu Gln Val Leu His Gln Asn Asn Val Glu Leu Val Asn Ile Gly Gly
85 90 95
ACC GAC ATC GTG GAC GGC AAC CAC AAG CTG ACC CTG GGC CTG CTG TGG 336
Thr Asp Ile Val Asp Gly Asn His Lys Leu Thr Leu Gly Leu Leu Trp
100 105 110
TCC ATC ATC CTG CAC TGG CAG GTG AAG GAC GTG ATG AAG GAC GTG ATG 384
Ser Ile Ile Leu His Trp Gln Val Lys Asp Val Met Lys Asp Val Met
115 120 125
TCC GAC CTG CAG CAG ACC AAC TCC GAG AAG ATC CTG CTG TCC TGG GTG 432
Ser Asp Leu Gln Gln Thr Asn Ser Glu Lys Ile Leu Leu Ser Trp Val
130 135 140
CGC CAG ACC ACC CGC CCC TAC TCC CAG GTG AAC GTG CTG AAC TTC ACC 480
Arg Gln Thr Thr Arg Pro Tyr Ser Gln Val Asn Val Leu Asn Phe Thr
145 150 155 160
ACC TCC TGG ACC GAC GGC CTG GCC TTC AAC GCC GTG CTG CAC CGC CAC 528
Thr Ser Trp Thr Asp Gly Leu Ala Phe Asn Ala Val Leu His Arg His
165 170 175
AAG CCC GAC CTG TTC TCC TGG GAC AAG GTG GTG AAG ATG TCC CCC ATC 576
Lys Pro Asp Leu Phe Ser Trp Asp Lys Val Val Lys Met Ser Pro Ile
180 185 190
GAG CGC CTG GAG CAC GCC TTC TCC AAG GCC CAG ACC TAC CTG GGC ATC 624
Glu Arg Leu Glu His Ala Phe Ser Lys Ala Gln Thr Tyr Leu Gly Ile
195 200 205
GAG AAG CTG CTG GAC CCC GAG GAC GTG GCC GTG CAG CTG CCC GAC AAG 672
Glu Lys Leu Leu Asp Pro Glu Asp Val Ala Val Gln Leu Pro Asp Lys
210 215 220
AAG TCC ATC ATC ATG TAC CTG ACC TCC CTG TTC GAG GTG CTG CCC CAG 720
Lys Ser Ile Ile Met Tyr Leu Thr Ser Leu Phe Glu Val Leu Pro Gln
225 230 235 240
CAG GTG TCC ATC GAG GCC ATC CAG GAA GTG GAA ATG CTG CCC AGG CCC 768
Gln Val Ser Ile Glu Ala Ile Gln Glu Val Glu Met Leu Pro Arg Pro
245 250 255
CCC AAA GTG ACC AAG GAG GAG CAC TTC CAG CTG CAC CAC CAG ATG CAC 816
Pro Lys Val Thr Lys Glu Glu His Phe Gln Leu His His Gln Met His
260 265 270
TAT AGC CAG CAG ATC ACC GTG TCC CTG GCC CAG GGC TAT GAG AGA ACC 864
Tyr Ser Gln Gln Ile Thr Val Ser Leu Ala Gln Gly Tyr Glu Arg Thr
275 280 285
AGC AGC CCC AAG CCC AGA TTC AAG AGC TAC GCC TAC ACC CAG GCC GCC 912
Ser Ser Pro Lys Pro Arg Phe Lys Ser Tyr Ala Tyr Thr Gln Ala Ala
290 295 300
TAC GTG ACC ACC TCC GAC CCC ACC AGA AGC CCC TTC CCC AGC CAG CAC 960
Tyr Val Thr Thr Ser Asp Pro Thr Arg Ser Pro Phe Pro Ser Gln His
305 310 315 320
CTG GAG GCC CCC GAG GAC AAG AGC TTC GGC AGC AGC CTG ATG GAG AGC 1008
Leu Glu Ala Pro Glu Asp Lys Ser Phe Gly Ser Ser Leu Met Glu Ser
325 330 335
GAA GTG AAC CTG GAC TCC TAC CAG ATC GCC CTG GAG GAG GTG CTG ACC 1056
Glu Val Asn Leu Asp Ser Tyr Gln Ile Ala Leu Glu Glu Val Leu Thr
340 345 350
TGG CTG CTG TCC GCC GAG GAC ACC TTC CAG GAG CAG GAC GAC ATC TCC 1104
Trp Leu Leu Ser Ala Glu Asp Thr Phe Gln Glu Gln Asp Asp Ile Ser
355 360 365
GAC GAC GTG GAG GAG GTG AAG GAC CAG TTC GCC ACC CAC GAG GCC TTC 1152
Asp Asp Val Glu Glu Val Lys Asp Gln Phe Ala Thr His Glu Ala Phe
370 375 380
ATG ATG GAG CTG ACC GCC CAC CAG TCC TCC GTG GGC TCC GTG CTG CAG 1200
Met Met Glu Leu Thr Ala His Gln Ser Ser Val Gly Ser Val Leu Gln
385 390 395 400
GCC GGC AAC CAG CTG ATC ACC CAG GGC ACC CTG TCC GAC GAG GAG GAG 1248
Ala Gly Asn Gln Leu Ile Thr Gln Gly Thr Leu Ser Asp Glu Glu Glu
405 410 415
TTC GAG ATC CAG GAG CAG ATG ACC CTG CTG AAC GCC CGC TGG GAG GCC 1296
Phe Glu Ile Gln Glu Gln Met Thr Leu Leu Asn Ala Arg Trp Glu Ala
420 425 430
CTG CGC GTG GAG TCC ATG GAC CGC CAG TCC CGC CTG CAC GAC GTG CTG 1344
Leu Arg Val Glu Ser Met Asp Arg Gln Ser Arg Leu His Asp Val Leu
435 440 445
ATG GAG CTG CAG AAG AAG CAG CTG CAG CAG CTG TCC GCC TGG CTG ACC 1392
Met Glu Leu Gln Lys Lys Gln Leu Gln Gln Leu Ser Ala Trp Leu Thr
450 455 460
CTG ACC GAG GAG CGC ATC CAG AAG ATG GAG ACC TGC CCC CTG GAC GAC 1440
Leu Thr Glu Glu Arg Ile Gln Lys Met Glu Thr Cys Pro Leu Asp Asp
465 470 475 480
GAC GTG AAG TCC CTG CAG AAG CTG CTG GAG GAG CAC AAG TCC CTG CAG 1488
Asp Val Lys Ser Leu Gln Lys Leu Leu Glu Glu His Lys Ser Leu Gln
485 490 495
TCC GAC CTG GAG GCC GAG CAG GTG AAG GTG AAC TCC CTG ACC CAC ATG 1536
Ser Asp Leu Glu Ala Glu Gln Val Lys Val Asn Ser Leu Thr His Met
500 505 510
GTG GTG ATC GTG GAC GAG AAC TCC GGC GAG TCC GCC ACC GCC ATC CTG 1584
Val Val Ile Val Asp Glu Asn Ser Gly Glu Ser Ala Thr Ala Ile Leu
515 520 525
GAG GAC CAG CTG CAG AAG CTG GGC GAG CGC TGG ACC GCC GTG TGC CGC 1632
Glu Asp Gln Leu Gln Lys Leu Gly Glu Arg Trp Thr Ala Val Cys Arg
530 535 540
TGG ACC GAG GAG CGC TGG AAC CGC CTG CAG GAG ATC AAC ATC CTG TGG 1680
Trp Thr Glu Glu Arg Trp Asn Arg Leu Gln Glu Ile Asn Ile Leu Trp
545 550 555 560
CAG GAG CTG CTG GAG GAG CAG TGC CTG CTG AAG GCC TGG CTG ACC GAG 1728
Gln Glu Leu Leu Glu Glu Gln Cys Leu Leu Lys Ala Trp Leu Thr Glu
565 570 575
AAG GAG GAG GCC CTG AAC AAG GTG CAG ACC TCC AAC TTC AAG GAC CAG 1776
Lys Glu Glu Ala Leu Asn Lys Val Gln Thr Ser Asn Phe Lys Asp Gln
580 585 590
AAG GAG CTG TCC GTG TCC GTG CGC CGC CTG GCC ATC CTG AAG GAG GAC 1824
Lys Glu Leu Ser Val Ser Val Arg Arg Leu Ala Ile Leu Lys Glu Asp
595 600 605
ATG GAG ATG AAG CGC CAG ACC CTG GAC CAG CTG TCC GAG ATC GGC CAG 1872
Met Glu Met Lys Arg Gln Thr Leu Asp Gln Leu Ser Glu Ile Gly Gln
610 615 620
GAC GTG GGC CAG CTG CTG GAC AAC TCC AAG GCC TCC AAG AAG ATC AAC 1920
Asp Val Gly Gln Leu Leu Asp Asn Ser Lys Ala Ser Lys Lys Ile Asn
625 630 635 640
TCC GAC TCC GAG GAG CTG ACC CAG CGC TGG GAC TCC CTG GTG CAG CGC 1968
Ser Asp Ser Glu Glu Leu Thr Gln Arg Trp Asp Ser Leu Val Gln Arg
645 650 655
CTG GAG GAC TCC TCC AAC CAG GTG ACC CAG GCC GTG GCC AAG CTG GGC 2016
Leu Glu Asp Ser Ser Asn Gln Val Thr Gln Ala Val Ala Lys Leu Gly
660 665 670
ATG TCC CAG ATC CCC CAG AAG GAC CTG CTG GAG ACC GTG CGC GTG CGC 2064
Met Ser Gln Ile Pro Gln Lys Asp Leu Leu Glu Thr Val Arg Val Arg
675 680 685
GAG CAG GCC ATC ACC AAG AAG TCC AAG CAG GAG CTG CCC CCC CCC CCC 2112
Glu Gln Ala Ile Thr Lys Lys Ser Lys Gln Glu Leu Pro Pro Pro Pro
690 695 700
CCC CCC AAG AAG CGC CAG ATC CAC GTG GAC CTG GAG AAG CTG CGC GAC 2160
Pro Pro Lys Lys Arg Gln Ile His Val Asp Leu Glu Lys Leu Arg Asp
705 710 715 720
CTG CAG GGC GCC ATG GAC GAC CTG GAC GCC GAC ATG AAG GAG GCC GAG 2208
Leu Gln Gly Ala Met Asp Asp Leu Asp Ala Asp Met Lys Glu Ala Glu
725 730 735
TCC GTG CGC AAC GGC TGG AAG CCC GTG GGC GAC CTG CTG ATC GAC TCC 2256
Ser Val Arg Asn Gly Trp Lys Pro Val Gly Asp Leu Leu Ile Asp Ser
740 745 750
CTG CAG GAC CAC ATC GAG AAG ATC ATG GCC TTC CGC GAG GAG ATC GCC 2304
Leu Gln Asp His Ile Glu Lys Ile Met Ala Phe Arg Glu Glu Ile Ala
755 760 765
CCC ATC AAC TTC AAG GTG AAG ACC GTG AAC GAC CTG TCC TCC CAG CTG 2352
Pro Ile Asn Phe Lys Val Lys Thr Val Asn Asp Leu Ser Ser Gln Leu
770 775 780
TCC CCC CTG GAC CTG CAC CCC TCC CTG AAG ATG TCC CGC CAG CTG GAC 2400
Ser Pro Leu Asp Leu His Pro Ser Leu Lys Met Ser Arg Gln Leu Asp
785 790 795 800
GAC CTG AAC ATG CGC TGG AAG CTG CTG CAG GTG TCC GTG GAC GAC CGC 2448
Asp Leu Asn Met Arg Trp Lys Leu Leu Gln Val Ser Val Asp Asp Arg
805 810 815
CTG AAG CAG CTG CAG GAG GCC CAC CGC GAC TTC GGC CCC TCC TCC CAG 2496
Leu Lys Gln Leu Gln Glu Ala His Arg Asp Phe Gly Pro Ser Ser Gln
820 825 830
CAC TTC CTG TCC ACC TCC GTG CAG CTG CCC TGG CAG CGC TCC ATC TCC 2544
His Phe Leu Ser Thr Ser Val Gln Leu Pro Trp Gln Arg Ser Ile Ser
835 840 845
CAC AAC AAG GTG CCC TAC TAC ATC AAC CAC CAG ACC CAG ACC ACC TGC 2592
His Asn Lys Val Pro Tyr Tyr Ile Asn His Gln Thr Gln Thr Thr Cys
850 855 860
TGG GAC CAC CCC AAG ATG ACC GAG CTG TTC CAG TCC CTG GCC GAC CTG 2640
Trp Asp His Pro Lys Met Thr Glu Leu Phe Gln Ser Leu Ala Asp Leu
865 870 875 880
AAC AAC GTG CGC TTC TCC GCC TAC CGC ACC GCC ATC AAG ATC CGC CGC 2688
Asn Asn Val Arg Phe Ser Ala Tyr Arg Thr Ala Ile Lys Ile Arg Arg
885 890 895
CTG CAG AAG GCC CTG TGC CTG GAC CTG CTG GAG CTG TCC ACC ACC AAC 2736
Leu Gln Lys Ala Leu Cys Leu Asp Leu Leu Glu Leu Ser Thr Thr Asn
900 905 910
GAG ATC TTC AAG CAG CAC AAG CTG AAC CAG AAC GAC CAG CTG CTG TCC 2784
Glu Ile Phe Lys Gln His Lys Leu Asn Gln Asn Asp Gln Leu Leu Ser
915 920 925
GTG CCC GAC GTG ATC AAC TGC CTG ACC ACC ACC TAC GAC GGC CTG GAG 2832
Val Pro Asp Val Ile Asn Cys Leu Thr Thr Thr Tyr Asp Gly Leu Glu
930 935 940
CAG ATG CAC AAG GAC CTG GTG AAC GTG CCC CTG TGC GTG GAC ATG TGC 2880
Gln Met His Lys Asp Leu Val Asn Val Pro Leu Cys Val Asp Met Cys
945 950 955 960
CTG AAC TGG CTG CTG AAC GTG TAC GAC ACC GGC CGC ACC GGC AAG ATC 2928
Leu Asn Trp Leu Leu Asn Val Tyr Asp Thr Gly Arg Thr Gly Lys Ile
965 970 975
CGC GTG CAG TCC CTG AAG ATC GGC CTG ATG TCC CTG TCC AAG GGC CTG 2976
Arg Val Gln Ser Leu Lys Ile Gly Leu Met Ser Leu Ser Lys Gly Leu
980 985 990
CTG GAG GAG AAG TAC CGC TAC CTG TTC AAG GAG GTG GCC GGC CCC ACC 3024
Leu Glu Glu Lys Tyr Arg Tyr Leu Phe Lys Glu Val Ala Gly Pro Thr
995 1000 1005
GAG ATG TGC GAC CAG CGC CAG CTG GGC CTG CTG CTG CAC GAC GCC ATC 3072
Glu Met Cys Asp Gln Arg Gln Leu Gly Leu Leu Leu His Asp Ala Ile
1010 1015 1020
CAG ATC CCC CGC CAG CTG GGC GAG GTG GCC GCC TTC GGC GGC TCC AAC 3120
Gln Ile Pro Arg Gln Leu Gly Glu Val Ala Ala Phe Gly Gly Ser Asn
1025 1030 1035 1040
ATC GAG CCC TCC GTG CGC TCC TGC TTC CAG CAG AAC AAC AAC AAG CCC 3168
Ile Glu Pro Ser Val Arg Ser Cys Phe Gln Gln Asn Asn Asn Lys Pro
1045 1050 1055
GAG ATC TCC GTG AAG GAG TTC ATC GAC TGG ATG CAC CTG GAG CCC CAG 3216
Glu Ile Ser Val Lys Glu Phe Ile Asp Trp Met His Leu Glu Pro Gln
1060 1065 1070
TCC ATG GTG TGG CTG CCC GTG CTG CAC CGC GTG GCC GCC GCC GAG ACC 3264
Ser Met Val Trp Leu Pro Val Leu His Arg Val Ala Ala Ala Glu Thr
1075 1080 1085
GCC AAG CAC CAG GCC AAG TGC AAC ATC TGC AAG GAG TGC CCC ATC GTG 3312
Ala Lys His Gln Ala Lys Cys Asn Ile Cys Lys Glu Cys Pro Ile Val
1090 1095 1100
GGC TTC CGC TAC CGC TCC CTG AAG CAC TTC AAC TAC GAC GTG TGC CAG 3360
Gly Phe Arg Tyr Arg Ser Leu Lys His Phe Asn Tyr Asp Val Cys Gln
1105 1110 1115 1120
TCC TGC TTC TTC TCC GGC CGC ACC GCC AAG GGC CAC AAG CTG CAC TAC 3408
Ser Cys Phe Phe Ser Gly Arg Thr Ala Lys Gly His Lys Leu His Tyr
1125 1130 1135
CCC ATG GTG GAG TAC TGC ATC CCC ACC ACC TCC GGC GAG GAC GTG CGC 3456
Pro Met Val Glu Tyr Cys Ile Pro Thr Thr Ser Gly Glu Asp Val Arg
1140 1145 1150
GAC TTC ACC AAG GTG CTG AAG AAC AAG TTC CGC TCC AAG AAG TAC TTC 3504
Asp Phe Thr Lys Val Leu Lys Asn Lys Phe Arg Ser Lys Lys Tyr Phe
1155 1160 1165
GCC AAG CAC CCC CGC CTG GGC TAC CTG CCC GTG CAG ACC GTG CTG GAG 3552
Ala Lys His Pro Arg Leu Gly Tyr Leu Pro Val Gln Thr Val Leu Glu
1170 1175 1180
GGC GAC AAC CTG GAG ACC CAG GCC ATG TGA 3582
Gly Asp Asn Leu Glu Thr Gln Ala Met ***
1185 1190 1193
<---
Реферат
Группа изобретений относится к области биотехнологии и медицины, в частности к химерным белкам, содержащим последовательности дистрофина и утрофина человека. Предложен химерный белок для лечения миодистрофии Дюшенна, содержащий N-терминальный домен утрофина человека, в котором пептид, кодируемый первым экзоном утрофина, необязательно заменен на пептид, кодируемый первым экзоном дистрофина человека, первый неструктурированный домен дистрофина человека (hinge 1), спектриновые повторы 1, 2, 3 утрофина человека, второй неструктурированный домен утрофина человека (hinge 2), спектриновый повтор 22 утрофина человека, четвёртый неструктурированный домен утрофина человека (hinge 4), цистеин-богатый (CR) домен утрофина человека, олигопептид QAM, кодируемый экзоном 74 утрофина человека. Также предложены нуклеиновые кислоты, кодирующие упомянутые белки и вирусные экспрессионные векторы для введения в организм млекопитающего, содержащие упомянутые нуклеиновые кислоты. Изобретения могут использоваться для заместительной генной терапии миодистрофии Дюшенна при доставке в мышечные клетки пациентов с помощью известных экспрессионных векторов и дальнейшей экспрессии там химерного белка, способного функционально заменить отсутствующий дистрофин. 4 н. и 4 з.п. ф-лы, 10 ил., 3 пр.

Комментарии