Последовательности 3'-utr для стабилизации рнк - RU2720934C2
Код документа: RU2720934C2
Чертежи







Описание
Последовательности 3'-UTR для стабилизации РНК
Применение РНК предоставляет перспективную альтернативу ДНК с возможностью избежать потенциальных рисков безопасности, связанных с терапевтическим применением ДНК. РНК, транскрибированная in vitro (IVT-РНК), представляет особый интерес для терапевтических подходов. Преимущества терапевтического применения РНК включают транзиентную экспрессию и нетрансформирующий характер. В случае РНК не требуется, чтобы она проникала в ядро для экспрессии и, кроме того, она не способна интегрироваться в геном хозяина, благодаря чему устраняется риск, связанный с онкогенезом. При применении для вакцинации инъекция РНК может обеспечивать индукцию как клеточного, так и гуморального иммунных ответов in vivo. Однако использование РНК для клинических вариантов применения в значительной степени ограничено, в первую очередь, коротким временем полужизни РНК.
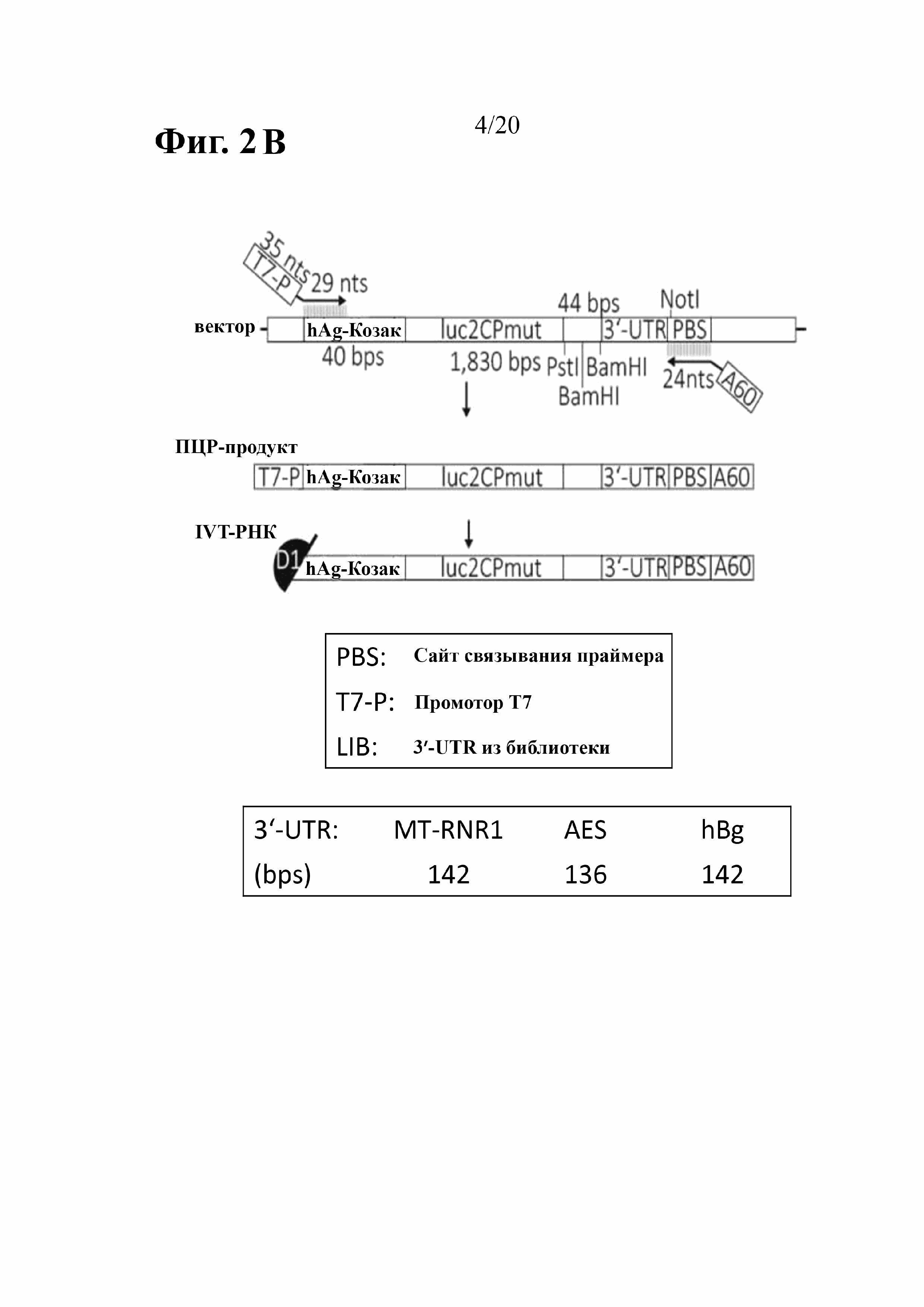
Векторы для использования IVT можно применять стандартным способом как матрицу для in vitro транскрипции. Такие векторы для использования IVT могут иметь следующую структуру: промотор для РНК-полимеразы, работающей в направлении от 5', обеспечивающий транскрипцию РНК, за которым следует представляющий интерес ген, фланкированный или 3'-, и/или 5'-нетранслируемыми областями (UTR), и кассета с 3'-полиадениловой последовательностью, содержащей нуклеотиды A. Перед осуществлением in vitro транскрипции кольцевую плазмиду подвергают линеаризации ниже кассеты с полиадениловой последовательностью с помощью ферментов рестрикции II типа (последовательность распознавания соответствует сайту расщепления). Кассета с полиадениловой последовательностью, таким образом, соответствует образующейся впоследствии поли(A)-последовательности в транскрипте.
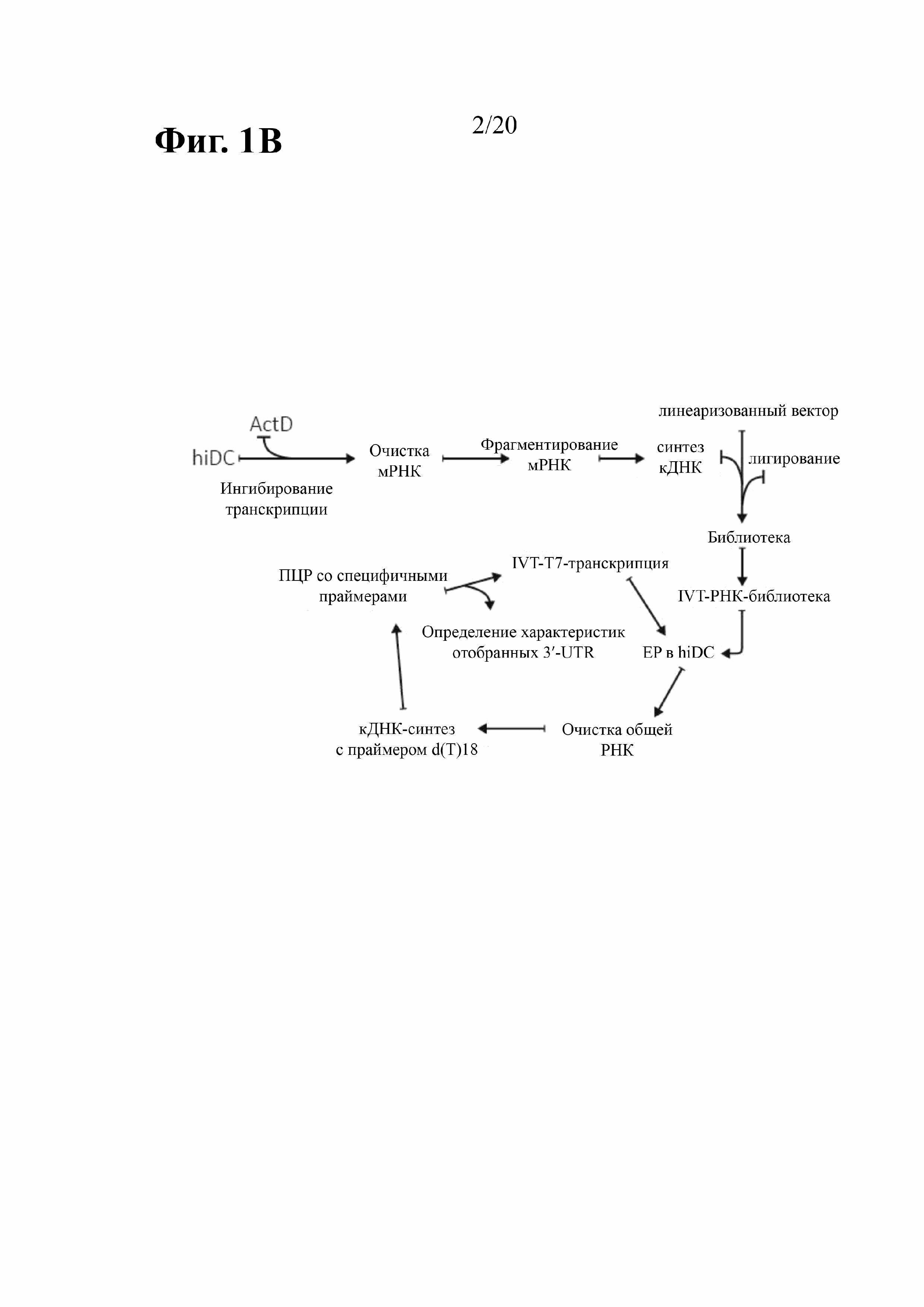
Незрелые дендритные клетки человека (hiDC) широко применяют для разработки и усовершенствования иммунотерапевтических препаратов для лечения злокачественной опухоли. Нагруженные транскрибированной in vitro (IVT)-мРНК, кодирующей специфичный опухолевый антиген (TA), hiDC способны индуцировать эффективный противоопухолевый ответ. Однако необходимым условием для получения эффективного иммунного ответа с применением противоопухолевых вакцин на основе РНК является высокая стабильность и эффективность трансляции РНК. Оба условия можно улучшить благодаря структурным модификациям, представляющим собой 5'-КЭП, 3'-поли(A)-хвост,а также 5'- и 3'-нетранслируемые области (UTR). Элементы последовательности в пределах UTR влияют на эффективность трансляции (главным образом 5'-UTR) и стабильность РНК (главным образом 3'-UTR).
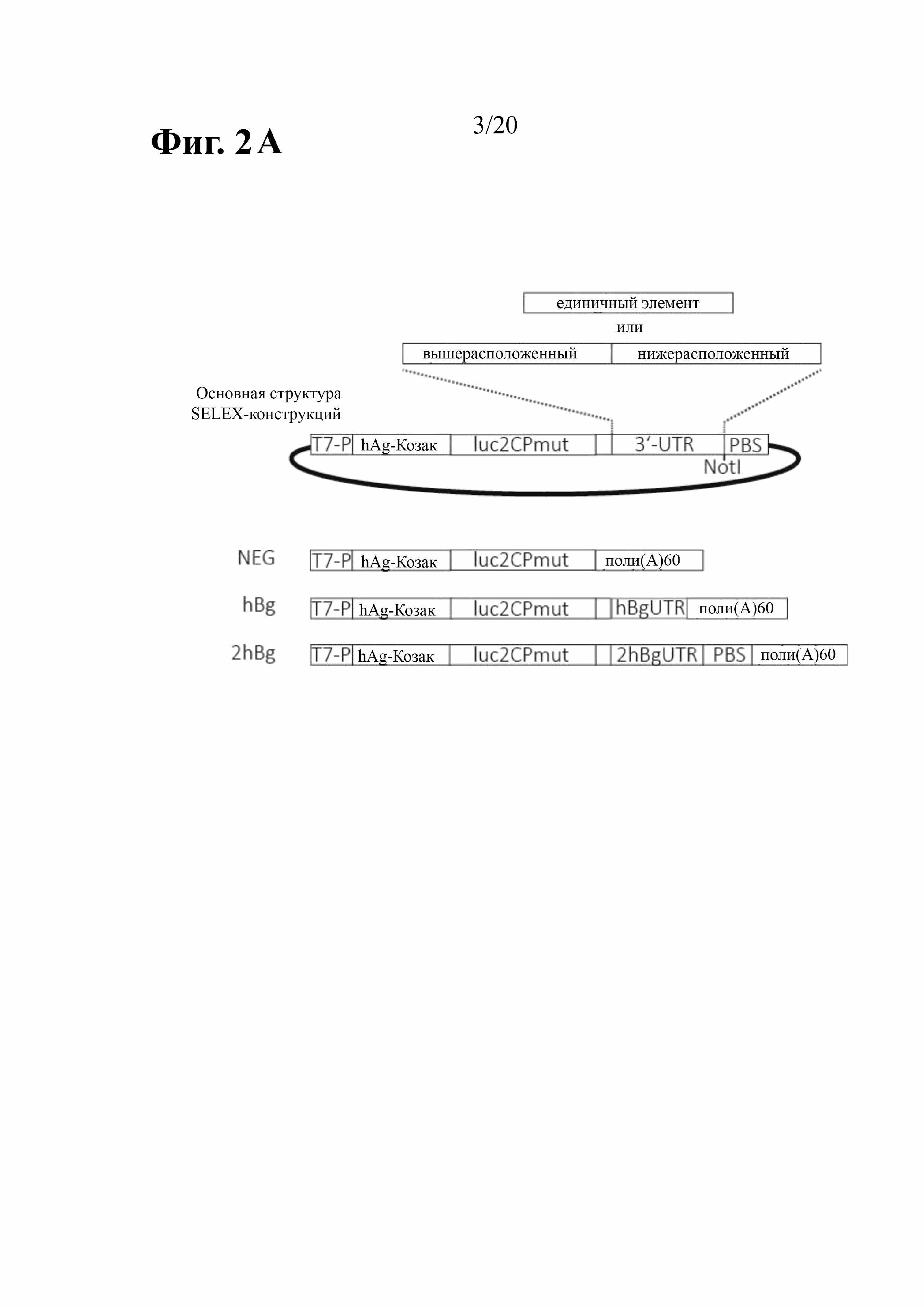
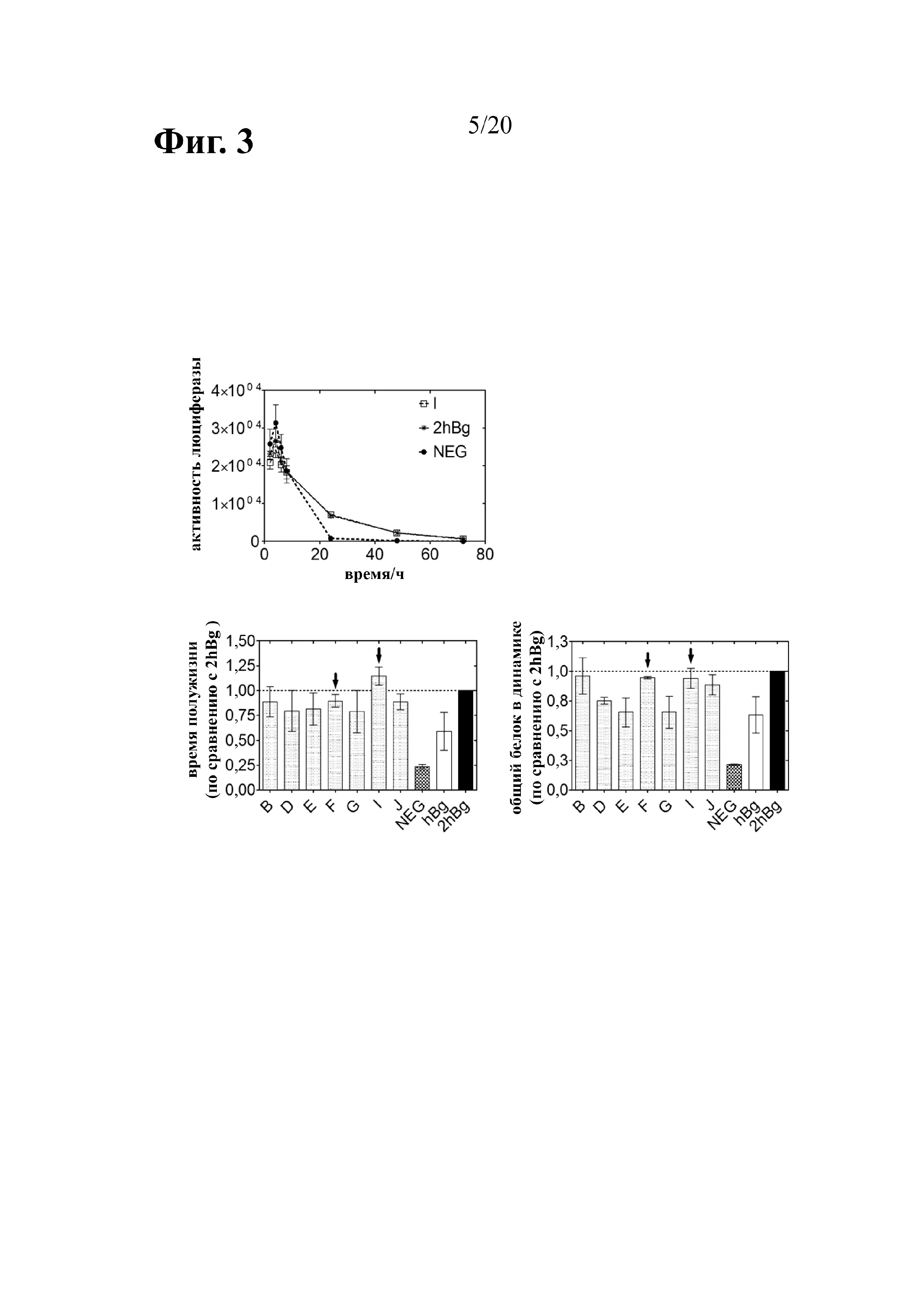
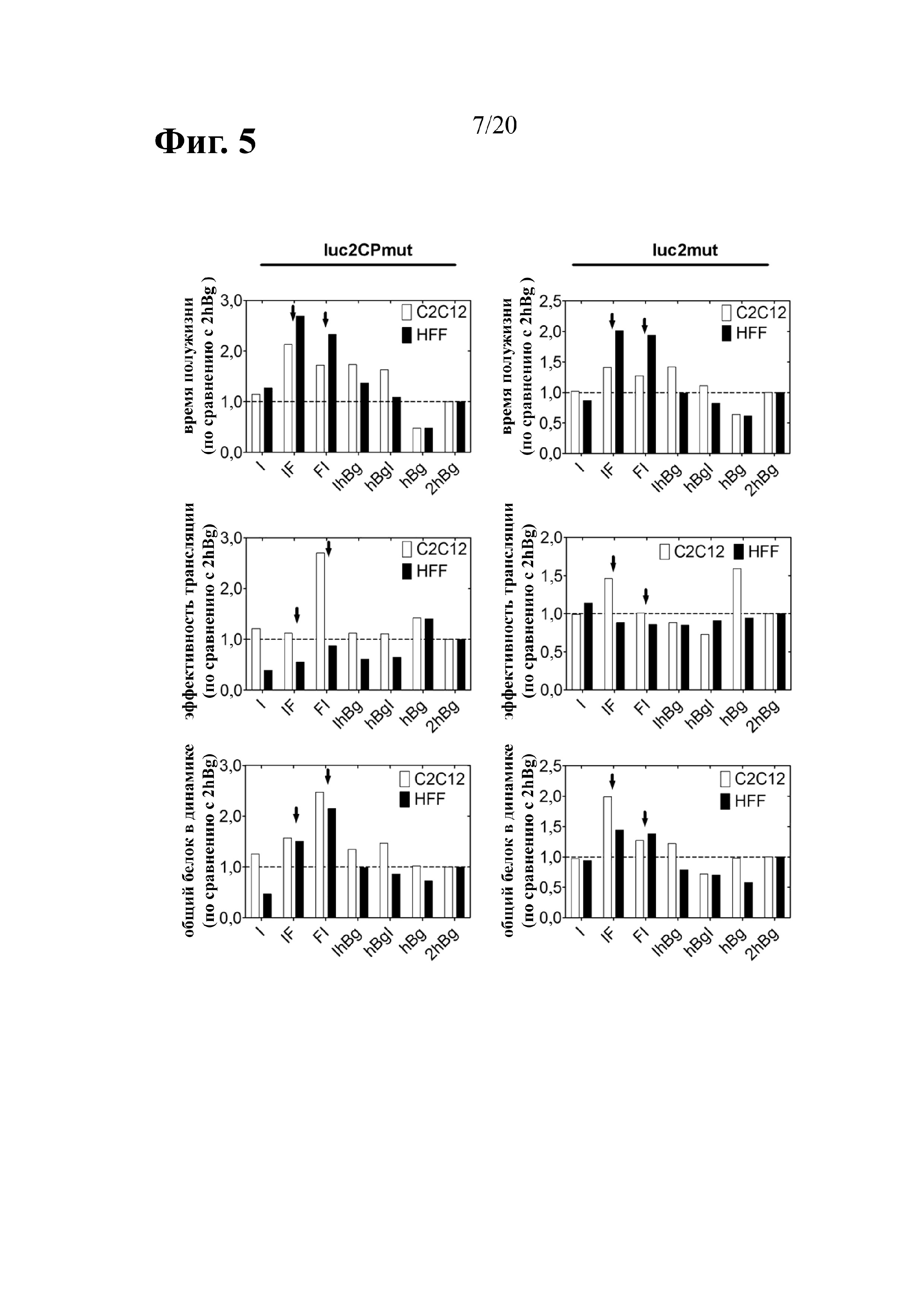
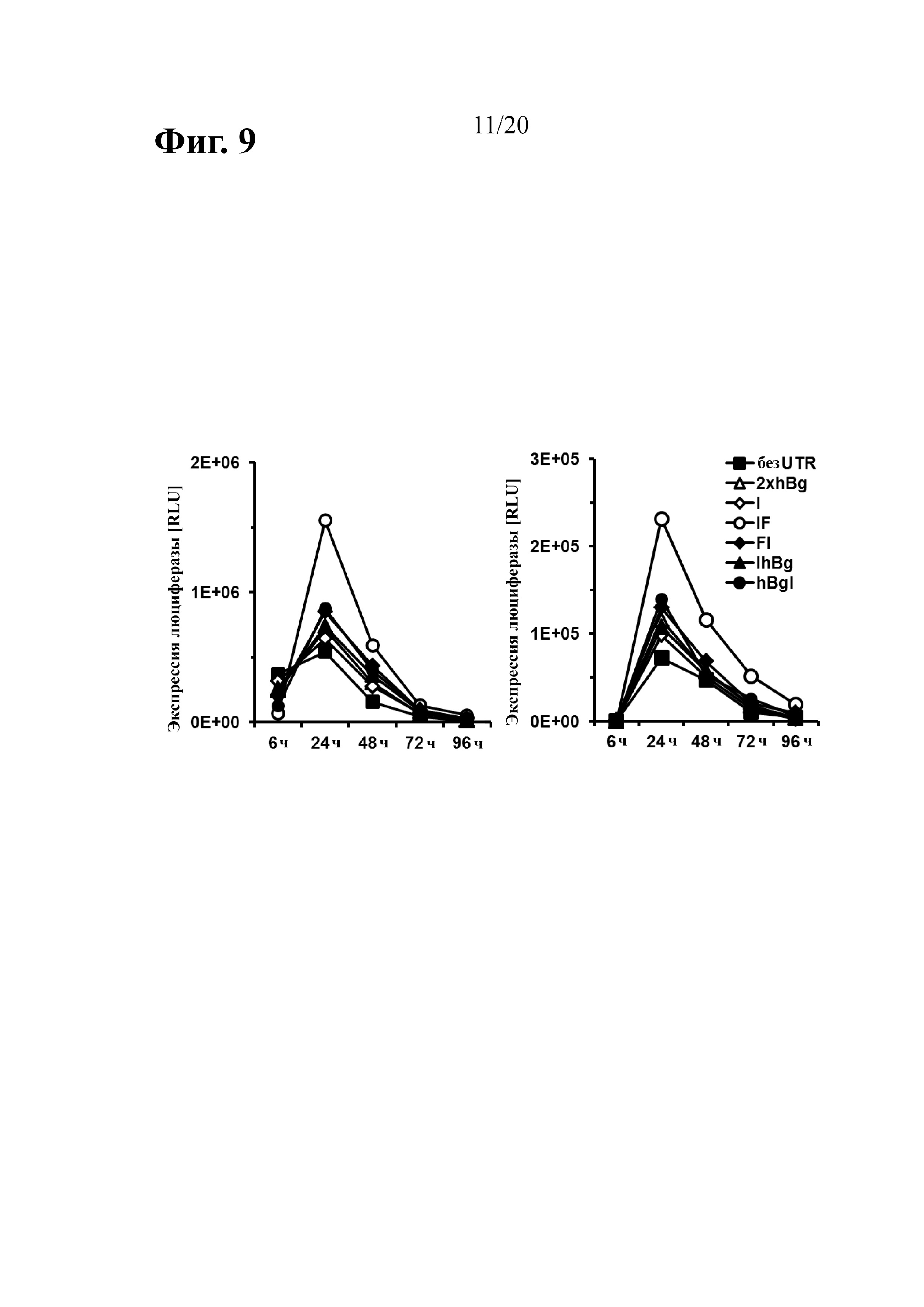
В предыдущей работе авторы настоящего изобретения продемонстрировали, что две последовательные копии 3'-UTR бета-глобина человека (на данный момент называемые 2hBg; ранее также 2βgUTR) способствуют повышению стабильности транскрипта и эффективности трансляции (Holtkamp (2006) Blood 108:4009-4017). Однако присутствие двух идентичных копий последовательности 3'-UTR бета-глобина человека в плазмидной ДНК, которую затем применяют в качестве матрицы для транскрипции РНК in vitro, создает риск рекомбинации в ходе ее размножения в E. coli. Аналогичным образом, любой подход с использованием клонирования, в частности, с применением ПЦР-амплификации, является очень сложным. То же самое касается ПЦР-амплификации РНК-кодирующей области с 2hBg на 3'-конце, которая будет использована в качестве матрицы для транскрипции in vitro, поскольку в этом случае наблюдали неспецифическое связывание праймера с матрицей, которое приводит к потере одной копии 3'-UTR бета-глобина человека. Во избежание таких проблем авторы настоящего изобретения сделали попытки идентифицировать новые последовательности, которые оказывают стабилизирующий эффект в отношении транскрибированной in vitro мРНК по меньшей мере аналогичным образом, в идеальном случае даже лучше, чем последовательность 2hBg.
Целью настоящего изобретения было получение РНК с повышенной стабильностью и/или эффективностью трансляции, а также средств для получения такой РНК. При применении указанной РНК в терапии должно быть возможным обеспечение высоких уровней экспрессии.
В соответствии с настоящим изобретением эта цель достигается с применением заявляемого объекта.
Настоящее изобретение относится к стабилизации РНК, в частности мРНК, и к повышению эффективности трансляции мРНК. Настоящее изобретение, в частности, относится к модификации РНК, в частности РНК, транскрибированной in vitro, которая обеспечивает повышенную стабильность транскрипта и/или эффективность трансляции.
В соответствии с настоящим изобретением было продемонстрировано, что определенные последовательности 3'-нетранслируемой области (UTR) молекулы РНК улучшают стабильность и эффективность трансляции.
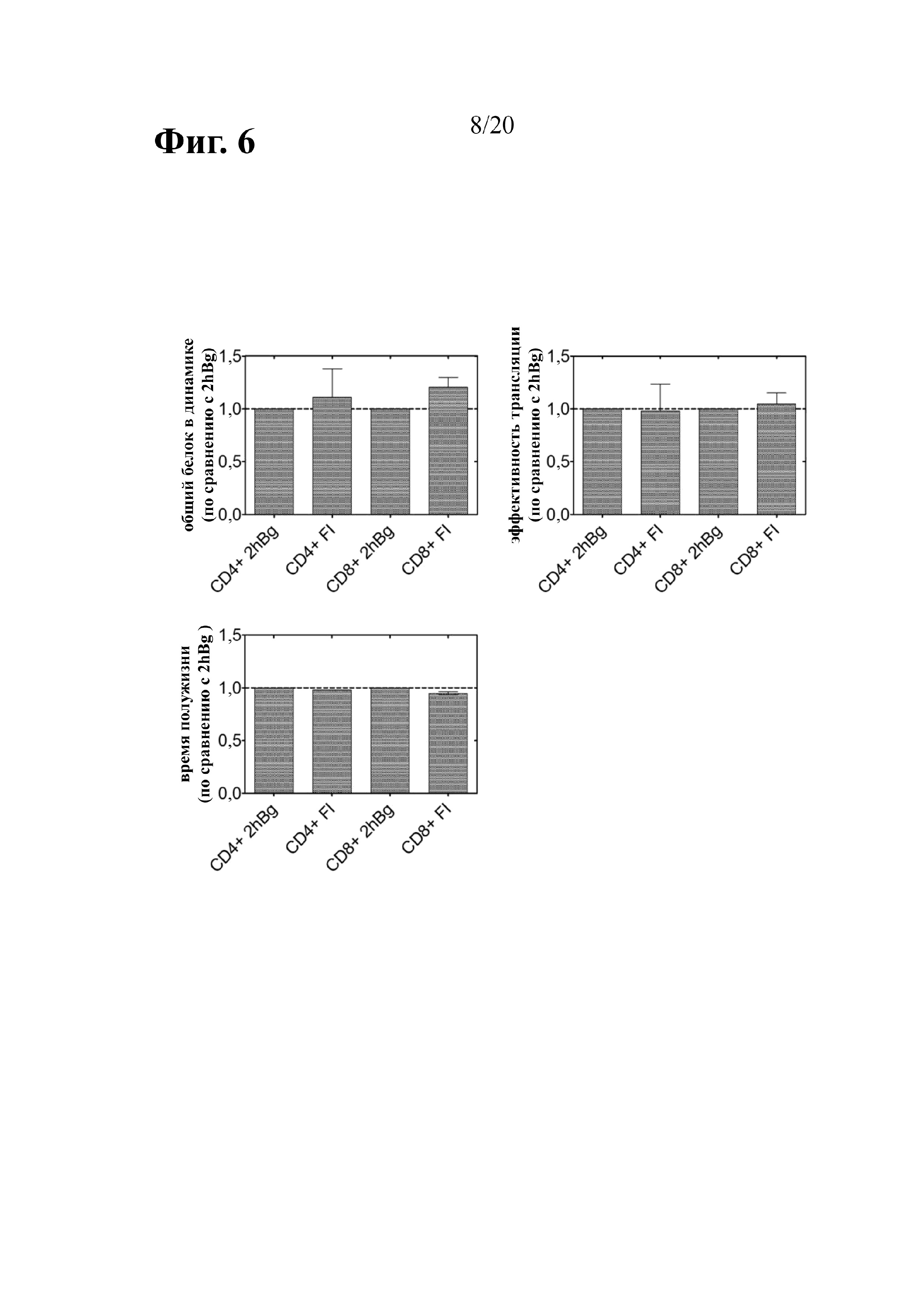
С применением модифицированной в соответствии с настоящим изобретением РНК для трансфекции дендритных клеток (DC) можно будет, например, повышать плотность комплексов антигенспецифичный пептид/MHC на поверхности трансфицированных клеток и их способность стимулировать и способствовать размножению антигенспецифичных CD4+ и CD8+ T-клеток. Таким образом, согласно одному варианту осуществления настоящее изобретение относится к стратегии оптимизации РНК-вакцин для трансфицирования DC или вакцин на основе трансфицированных РНК DC с применением РНК, которая была модифицирована с помощью модификаций РНК, описанных в соответствии с настоящим изобретением.
В соответствии с настоящим изобретением модификация и, следовательно, стабилизация и/или повышение эффективности трансляции РНК предпочтительно достигается с помощью применения генетически модифицированных векторов экспрессии, которые предпочтительно служат в качестве матрицы для транскрипции РНК in vitro. Такие векторы экспрессии обеспечивают транскрипцию РНК с 3'-нетранслируемой областью, описанной в соответствии с настоящим изобретением и предпочтительно расположенной между последовательностью, кодирующей пептид или белок (открытая рамка считывания), и поли(A)-последовательностью.
Такие векторы могут также обеспечивать транскрипцию РНК с поли(A)-последовательностью, которая в указанной РНК предпочтительно имеет открытый конец, т. е. нуклеотиды, отличные от нуклеотидов A, не фланкируют указанную поли(A)-последовательность на ее 3'-конце. Открытая поли(A)-последовательность в РНК может быть обеспечена путем введения сайта расщепления рестриктазами типа IIS в вектор экспрессии, который обеспечивает возможность транскрипции РНК под контролем промотора для РНК-полимеразы, работающей в направлении от 5', и который содержит кассету с полиадениловой последовательностью, причем последовательность распознавания расположена в направлении 3' от кассеты с полиадениловой последовательностью, тогда как сайт расщепления расположен выше и, следовательно, находится в пределах кассеты с полиадениловой последовательностью. Рестрикционное расщепление в сайте расщепления рестриктазами типа IIS позволяет осуществлять линеаризацию плазмиды в пределах кассеты с полиадениловой последовательностью. Линеаризованную плазмиду затем можно применять в качестве матрицы для in vitro транскрипции, причем полученный в результате транскрипт заканчивается немаскированной поли(A)-последовательностью. Кроме того, необязательное прерывание кассеты с 3'-полиадениловой последовательностью случайной нуклеотидной последовательностью с равным распределением 4 нуклеотидов (линкер) обеспечивает повышение стабильности кассеты с 3'-полиадениловой последовательностью в E.coli.
Краткое раскрытие настоящего изобретения
Согласно одному аспекту настоящее изобретение относится к молекуле нуклеиновой кислоты, содержащей, в направлении транскрипции 5' → 3':
(a) промотор;
(b) транскрибируемую последовательность нуклеиновой кислоты или последовательность нуклеиновой кислоты для введения транскрибируемой последовательности нуклеиновой кислоты; и
(c) последовательность нуклеиновой кислоты, которая, будучи транскрибированной под контролем промотора (a), кодирует 3'-нетранслируемую область транскрипта, причем указанная 3'-нетранслируемая область содержит последовательность нуклеиновой кислоты, которая выбрана из группы, состоящей из:
(c-1) последовательности нуклеиновой кислоты 3'-нетранслируемой области FCGRT, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(c-2) последовательности нуклеиновой кислоты 3'-нетранслируемой области LSP1, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(c-3) последовательности нуклеиновой кислоты 3'-нетранслируемой области CCL22, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(c-4) последовательности нуклеиновой кислоты 3'-нетранслируемой области AES, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(c-5) последовательности нуклеиновой кислоты 3'-нетранслируемой области PLD3, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(c-6) последовательности нуклеиновой кислоты некодирующей РНК MT-RNR1, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(c-7) последовательности нуклеиновой кислоты 3'-нетранслируемой области HLA-DRB4, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
и
(c-8) любой комбинации двух или более последовательностей нуклеиновой кислоты, фрагментов и/или вариантов, обозначенных (c-1), (c-2), (c-3), (c-4), (c-5), (c-6) и (c-7).
Согласно одному варианту осуществления последовательности нуклеиновой кислоты (b) и (c) под контролем промотора (a) могут быть транскрибированы с образованием общего транскрипта, в котором последовательность нуклеиновой кислоты, транскрибированная с последовательности нуклеиновой кислоты (c), является активной в отношении повышения эффективности трансляции и/или стабильности последовательности нуклеиновой кислоты, транскрибированной с транскрибируемой последовательности нуклеиновой кислоты (b).
Согласно одному варианту осуществления последовательности нуклеиновой кислоты (b) и (c) в естественных условиях не связаны.
Согласно одному варианту осуществления последовательность нуклеиновой кислоты (c-4) 3'-нетранслируемой области AES, ее фрагмент или вариант указанных последовательности нуклеиновой кислоты или фрагмента содержат последовательность нуклеиновой кислоты, выбранную из группы, состоящей из последовательности нуклеиновой кислоты, выбранной из SEQ ID NO: 86-89, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента.
Согласно одному варианту осуществления последовательность нуклеиновой кислоты (c-4) 3'-нетранслируемой области AES, ее фрагмент или вариант указанных последовательности нуклеиновой кислоты или фрагмента содержат последовательность нуклеиновой кислоты, выбранную из группы, состоящей из последовательности нуклеиновой кислоты под SEQ ID NO: 86, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента.
Согласно одному варианту осуществления последовательность нуклеиновой кислоты (c-6) некодирующей РНК MT-RNR1, ее фрагмент или вариант указанных последовательности нуклеиновой кислоты или фрагмента содержат последовательность нуклеиновой кислоты, выбранную из группы, состоящей из последовательности нуклеиновой кислоты, выбранной из SEQ ID NO: 105-121, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента.
Согласно одному варианту осуществления последовательность нуклеиновой кислоты (c-6) некодирующей РНК MT-RNR1, ее фрагмент или вариант указанных последовательности нуклеиновой кислоты или фрагмента содержат последовательность нуклеиновой кислоты, выбранную из группы, состоящей из последовательности нуклеиновой кислоты под SEQ ID NO: 115, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента.
Согласно одному варианту осуществления последовательность нуклеиновой кислоты (c8) содержит комбинацию двух или более идентичных или отличающихся последовательностей нуклеиновой кислоты, фрагментов и/или вариантов, обозначенных (c-1), (c-2), (c-3), (c-4), (c-5) (c-6) и (c-7). Согласно различным вариантам осуществления последовательность нуклеиновой кислоты (c8) содержит комбинацию (c-1) и (c-2), (c-1) и (c-3), (c-1) и (c-4), (c-1) и (c-5), (c-1) и (c-6), (c-1) и (c-7), (c-2) и (c-3), (c-2) и (c-4), (c-2) и (c-5), (c-2) и (c-6), (c-2) и (c-7), (c-3) и (c-4), (c-3) и (c-5), (c-3) и (c-6), (c-3) и (c-7), (c-4) и (c-5), (c-4) и (c-6), (c-4) и (c-7), (c-5) и (c-6), (c-5) и (c-7) или (c-6) и (c-7).
Согласно одному варианту осуществления последовательность нуклеиновой кислоты (c8) содержит комбинацию последовательности нуклеиновой кислоты (c-4) 3'-нетранслируемой области AES, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента и последовательности нуклеиновой кислоты (c-6) некодирующей РНК MT-RNR1, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента. Согласно одному варианту осуществления последовательность нуклеиновой кислоты (c-4) 3'-нетранслируемой области AES, ее фрагмент или вариант указанных последовательности нуклеиновой кислоты или фрагмента расположены в направлении 5' от последовательности нуклеиновой кислоты (c-6) некодирующей РНК MT-RNR1, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента. Согласно одному варианту осуществления комбинация последовательности нуклеиновой кислоты (c-4) 3'-нетранслируемой области AES, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента и последовательности нуклеиновой кислоты (c-6) некодирующей РНК MT-RNR1, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента содержит последовательность нуклеиновой кислоты, выбранную из группы, состоящей из последовательности нуклеиновой кислоты под SEQ ID NO: 174, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента.
Согласно одному варианту осуществления молекула нуклеиновой кислоты по настоящему изобретению дополнительно содержит (d) последовательность нуклеиновой кислоты, которая, будучи транскрибированной под контролем промотора (a), кодирует последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов А. Согласно одному варианту осуществления указанная полиадениловая последовательность содержит по меньшей мере 20 нуклеотидов A, предпочтительно по меньшей мере 40, по меньшей мере 80, по меньшей мере 100 или по меньшей мере 120 нуклеотидов A, предпочтительно последовательных нуклеотидов A. Согласно одному варианту осуществления указанная последовательность из одного или нескольких последовательных нуклеотидов, содержащая нуклеотиды, отличные от нуклеотидов A, представляет собой последовательность, предпочтительно произвольную последовательность, из 2 или более последовательных нуклеотидов, где первый и последний нуклеотиды указанной последовательности из 2 или более последовательных нуклеотидов представляют собой нуклеотиды, отличные от нуклеотида A. Согласно одному варианту осуществления указанная последовательность нуклеиновой кислоты (d) представляет собой последовательность нуклеиновой кислоты, которая, будучи транскрибированной под контролем промотора (a), кодирует последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A, и которая характеризуется более высокой стабильностью при размножении указанной молекулы нуклеиновой кислоты в Escherichia coli по сравнению с молекулой нуклеиновой кислоты, которая вместо указанной последовательности нуклеиновой кислоты (d)предусматривает последовательность нуклеиновой кислоты (d)', которая, будучи транскрибированной под контролем промотора (a), кодирует полиадениловую последовательность такой же длины, что и указанная последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, предусматривающих нуклеотиды, отличные от нуклеотидов A. Согласно одному варианту осуществления указанная последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A, содержит по меньшей мере 80 нуклеотидов, предпочтительно по меньшей мере 90 или 100 нуклеотидов. Согласно одному варианту осуществления указанная последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A, содержит по меньшей мере 90 нуклеотидов, предпочтительно по меньшей мере 100 нуклеотидов, предпочтительно по меньшей мере 110 нуклеотидов. Согласно одному варианту осуществления указанная последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A, содержит приблизительно 120 нуклеотидов. Согласно конкретным вариантам осуществления указанная последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A, содержит до 200, предпочтительно до 150, и, в частности, до 130 нуклеотидов. Согласно одному варианту осуществления по меньшей мере 90%, предпочтительно по меньшей мере 92%, предпочтительно по меньшей мере 95%, 97% или 98% нуклеотидов указанной последовательности нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A, представляют собой нуклеотиды A в указанной полиадениловой последовательности (не включая нуклеотиды A в указанной последовательности из одного или нескольких последовательных нуклеотидов, содержащей нуклеотиды, отличные от нуклеотидов A).
Согласно одному варианту осуществления указанная последовательность из одного или нескольких последовательных нуклеотидов, содержащая нуклеотиды, отличные от нуклеотидов A, расположена в пределах участка от положения 21 до положения 80, предпочтительно от положения 21 до положения 60, более предпочтительно от положения 31 до положения 50 указанной полиадениловой последовательности.
Согласно одному варианту осуществления указанной последовательности из одного или нескольких последовательных нуклеотидов, содержащей нуклеотиды, отличные от нуклеотидов A, предшествуют по меньшей мере 20 остатков A, предпочтительно по меньшей мере 30, 40 или 50 остатков A в указанной полиадениловой последовательности. Согласно конкретным вариантам осуществления указанной последовательности из одного или нескольких последовательных нуклеотидов, содержащей нуклеотиды, отличные от нуклеотидов A, предшествуют до 80 остатков A, предпочтительно до 70 или 60 остатков A в указанной полиадениловой последовательности.
Согласно одному варианту осуществления за указанной последовательностью из одного или нескольких последовательных нуклеотидов, содержащей нуклеотиды, отличные от нуклеотидов A, расположены по меньшей мере 20 остатков A, предпочтительно по меньшей мере 30, 40, 50, 60 или 70 остатков A в указанной полиадениловой последовательности. Согласно конкретным вариантам осуществления за указанной последовательностью из одного или нескольких последовательных нуклеотидов, содержащей нуклеотиды, отличные от нуклеотидов A, расположены до 100 остатков A, предпочтительно до 80 остатков A в указанной полиадениловой последовательности.
Согласно одному варианту осуществления указанной последовательности из одного или нескольких последовательных нуклеотидов, содержащей нуклеотиды, отличные от нуклеотидов A, предшествуют 20-50, предпочтительно 30-40 остатков A в указанной полиадениловой последовательности, и за ней расположены 30-80, предпочтительно 40-70 остатков A в указанной полиадениловой последовательности.
Согласно одному варианту осуществления длина указанной последовательности из одного или нескольких последовательных нуклеотидов, содержащей нуклеотиды, отличные от нуклеотидов A, составляет по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6, по меньшей мере 8, предпочтительно по меньшей мере 10, более предпочтительно по меньшей мере 15 нуклеотидов.
Согласно одному варианту осуществления длина указанной последовательности из одного или нескольких последовательных нуклеотидов, содержащей нуклеотиды, отличные от нуклеотидов A, составляет не более 50, предпочтительно не более 30, более предпочтительно не более 20 нуклеотидов.
Согласно одному варианту осуществления указанная последовательность из одного или нескольких последовательных нуклеотидов, содержащая нуклеотиды, отличные от нуклеотидов A, не содержит более 3, предпочтительно не более 2, предпочтительно не содержит последовательных остатков A.
Согласно одному варианту осуществления последовательности нуклеиновой кислоты (b), (c) и (d) под контролем промотора (a) могут быть транскрибированы с образованием общего транскрипта. Согласно одному варианту осуществления последовательности нуклеиновой кислоты, транскрибированные с последовательностей нуклеиновой кислоты (c) и необязательно (d), являются активными в отношении повышения эффективности трансляции и/или стабильности последовательности нуклеиновой кислоты, транскрибированной с транскрибируемой последовательности нуклеиновой кислоты (b).
Согласно одному варианту осуществления в транскрипте указанная последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A, расположена на 3'-конце.
Согласно одному варианту осуществления молекула нуклеиновой кислоты по настоящему изобретению представляет собой молекулу ДНК. Согласно одному варианту осуществления указанная молекула нуклеиновой кислоты представляет собой вектор или плазмиду экспрессии, как, например, вектор для использования IVT.
Согласно одному варианту осуществления молекула нуклеиновой кислоты по настоящему изобретению представляет собой замкнутую кольцевую молекулу или линейную молекулу.
Согласно одному варианту осуществления транскрибируемая последовательность нуклеиновой кислоты содержит последовательность нуклеиновой кислоты, кодирующую пептид или белок, а последовательность нуклеиновой кислоты для введения транскрибируемой последовательности нуклеиновой кислоты представляет собой сайт множественного клонирования.
Согласно одному варианту осуществления молекула нуклеиновой кислоты по настоящему изобретению дополнительно содержит один или несколько элементов, выбранных из группы, состоящей из: (i) репортерного гена; (ii) селектируемого маркера и (iii) точки начала репликации.
Согласно одному варианту осуществления молекула нуклеиновой кислоты по настоящему изобретению подходит, в частности после линеаризации, для in vitro транскрипции РНК, в частности мРНК.
Перед осуществлением in vitro транскрипции кольцевые векторы для использования IVT обычно подвергают линеаризации ниже кассеты с полиадениловой последовательностью с помощью ферментов рестрикции II типа (последовательность распознавания соответствует сайту расщепления). Кассета с полиадениловой последовательностью, таким образом, соответствует образующейся впоследствии поли(A)-последовательности в транскрипте. В результате этой процедуры некоторые нуклеотиды остаются частью сайта расщепления ферментами после линеаризация и продлевают или маскируют поли(A)-последовательность на 3'-конце. Однако, было обнаружено, что РНК с открытой поли(A)-последовательностью транслируется более эффективно,чем РНК с поли(A)-последовательностью с маскированным концом.
Таким образом, молекулы нуклеиновой кислоты по настоящему изобретению при применении в качестве векторов экспрессии предпочтительно обеспечивают транскрипцию РНК с поли(A)-последовательностью, которая в указанной РНК предпочтительно имеет открытый конец, т. е. нуклеотиды, отличные от нуклеотидов A, не фланкируют указанную поли(A)-последовательность на ее 3'-конце. Открытая поли(A)-последовательность в РНК может быть обеспечена путем введения сайта расщепления рестриктазами типа IIS в вектор экспрессии, который обеспечивает возможность транскрипции РНК под контролем промотора для РНК-полимеразы, работающей в направлении от 5', и который содержит кассету с полиадениловой последовательностью, причем последовательность распознавания расположена ниже кассеты с полиадениловой последовательностью, тогда как сайт расщепления расположен выше и, следовательно, находится в пределах кассеты с полиадениловой последовательностью. Рестрикционное расщепление в сайте расщепления рестриктазами типа IIS позволяет осуществлять линеаризацию плазмиды в пределах кассеты с полиадениловой последовательностью. Линеаризованную плазмиду затем можно применять в качестве матрицы для in vitro транскрипции, причем полученный в результате транскрипт заканчивается немаскированной поли(A)-последовательностью.
Соответственно, согласно одному варианту осуществления предпочтительным является, чтобы молекулу нуклеиновой кислоты по настоящему изобретению можно было расщеплять, предпочтительно ферментативно или другим биохимическим способом, в пределах последовательности нуклеиновой кислоты (d) таким образом, чтобы указанное расщепление приводило к образованию молекулы нуклеиновой кислоты, которая содержит, в направлении транскрипции 5' → 3', промотор (a), последовательности нуклеиновой кислоты (b) и (c) и по меньшей мере часть последовательности нуклеиновой кислоты (d), где по меньшей мере часть последовательности нуклеиновой кислоты (d), будучи транскрибированной под контролем промотора (a), кодирует указанную последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A, и где в транскрипте 3'-концевой нуклеотид представляет собой нуклеотид A указанной последовательности нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A.
Предпочтительно после расщепления молекула нуклеиновой кислоты на конце нити, которая служит в качестве матрицы для последовательности нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A, содержит нуклеотид T, который является частью последовательности нуклеиновой кислоты, которая служит в качестве матрицы для последовательности нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A.
Молекула нуклеиновой кислоты по настоящему изобретению предпочтительно до расщепления представляет собой замкнутую кольцевую молекулу, а после расщепления представляет собой линейную молекулу.
Предпочтительно расщепление осуществляют с использованием сайта расщепления рестриктазами, который предпочтительно представляет собой сайт расщепления рестриктазами для эндонуклеазы рестрикции типа IIS.
Согласно одному варианту осуществления последовательность распознавания для эндонуклеазы рестрикции типа IIS расположена в пределах 5-26 пар оснований, предпочтительно 24-26 пар оснований ниже 3'-конца последовательности нуклеиновой кислоты (d).
Согласно одному варианту осуществления молекула нуклеиновой кислоты по настоящему изобретению находится в конформации в виде замкнутого кольца и предпочтительно подходит для in vitro транскрипции РНК, в частности мРНК, в частности, после линеаризации.
Согласно дополнительным аспектам настоящее изобретение относится к молекуле нуклеиновой кислоты, получаемой путем линеаризации описанной выше молекулы нуклеиновой кислоты, предпочтительно путем расщепления в пределах последовательности нуклеиновой кислоты (d), и к РНК, получаемой в результате транскрипции, предпочтительно in vitro транскрипции, с использованием описанных выше молекул нуклеиновой кислоты под контролем промотора (a).
Таким образом, согласно одному аспекту настоящее изобретение относится к РНК, содержащей, в направлении 5' → 3':
(a) 5'-нетранслируемую область;
(b) последовательность нуклеиновой кислоты, кодирующую пептид или белок; и
(c) 3'-нетранслируемую область, причем указанная 3'-нетранслируемая область содержит последовательность нуклеиновой кислоты, которая выбрана из группы, состоящей из:
(c-1) последовательности нуклеиновой кислоты 3'-нетранслируемой области FCGRT, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(c-2) последовательности нуклеиновой кислоты 3'-нетранслируемой области LSP1, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(c-3) последовательности нуклеиновой кислоты 3'-нетранслируемой области CCL22, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(c-4) последовательности нуклеиновой кислоты 3'-нетранслируемой области AES, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(c-5) последовательности нуклеиновой кислоты 3'-нетранслируемой области PLD3, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(c-6) последовательности нуклеиновой кислоты некодирующей РНК MT-RNR1, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(c-7) последовательности нуклеиновой кислоты 3'-нетранслируемой области HLA-DRB4, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
и
(c-8) любой комбинации двух или более последовательностей нуклеиновой кислоты, фрагментов и/или вариантов, обозначенных (c-1), (c-2), (c-3), (c-4), (c-5), (c-6) и (c-7).
Согласно одному варианту осуществления последовательности нуклеиновой кислоты (b) и (c) в естественных условиях не связаны.
Согласно одному варианту осуществления РНК дополнительно содержит (d) последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A. Согласно одному варианту осуществления указанная последовательность нуклеиновой кислоты (d) расположена на 3'-конце указанной РНК.
Согласно одному варианту осуществления последовательности нуклеиновой кислоты (c) и необязательно (d) являются активными в отношении повышения эффективности трансляции и/или стабильности последовательности нуклеиновой кислоты, кодирующей пептид или белок.
Согласно одному варианту осуществления РНК дополнительно содержит (e) 5'-кэп.
Варианты осуществления 3'-нетранслируемой области и последовательности нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A, описаны выше для молекул нуклеиновой кислоты по настоящему изобретению.
Согласно дополнительному аспекту настоящее изобретение относится к способу получения РНК, предусматривающему:
(i) обеспечение молекулы нуклеиновой кислоты по настоящему изобретению и
(ii) обеспечение транскрибирования РНК с применением молекулы нуклеиновой кислоты в качестве матрицы.
Согласно дополнительному аспекту настоящее изобретение относится к способу получения пептида или белка, предусматривающему:
(i) получение РНК, кодирующей пептид или белок, в соответствии со способом получения РНК по настоящему изобретению и
(ii) обеспечение трансляции РНК.
Согласно одному варианту осуществления способ получения РНК или способ получения пептида или белка перед транскрипцией молекулы нуклеиновой кислоты дополнительно предусматривает расщепление молекулы нуклеиновой кислоты.
Согласно дополнительному аспекту настоящее изобретение относится к способу получения РНК, предусматривающему:
(i) присоединение последовательности нуклеиновой кислоты (b), которая, будучи транскрибированной, кодирует 3'-нетранслируемую область, к 3'-концу транскрибируемой последовательности нуклеиновой кислоты (a), содержащей последовательность нуклеиновой кислоты, кодирующую пептид или белок, и
(ii) обеспечение транскрибирования полученной нуклеиновой кислоты,
причем указанная 3'-нетранслируемая область содержит последовательность нуклеиновой кислоты, которая выбрана из группы, состоящей из:
(b-1) последовательности нуклеиновой кислоты 3'-нетранслируемой области FCGRT, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(b-2) последовательности нуклеиновой кислоты 3'-нетранслируемой области LSP1, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(b-3) последовательности нуклеиновой кислоты 3'-нетранслируемой области CCL22, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(b-4) последовательности нуклеиновой кислоты 3'-нетранслируемой области AES, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(b-5) последовательности нуклеиновой кислоты 3'-нетранслируемой области PLD3, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(b-6) последовательности нуклеиновой кислоты некодирующей РНК MT-RNR1, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
(b-7) последовательности нуклеиновой кислоты 3'-нетранслируемой области HLA-DRB4, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента,
и
(b-8) любой комбинации двух или более последовательностей нуклеиновой кислоты, фрагментов и/или вариантов, обозначенных (b-1), (b-2), (b-3), (b-4), (b-5), (b-6) и (b-7).
Согласно одному варианту осуществления последовательности нуклеиновой кислоты (a) и (b) могут быть транскрибированы с образованием общего транскрипта, в котором последовательность нуклеиновой кислоты, транскрибированная с последовательности нуклеиновой кислоты (b), является активной в отношении повышения эффективности трансляции и/или стабильности последовательности нуклеиновой кислоты, транскрибированной с транскрибируемой последовательности нуклеиновой кислоты (a).
Согласно одному варианту осуществления последовательности нуклеиновой кислоты (a) и (b) в естественных условиях не связаны.
Согласно одному варианту осуществления способ дополнительно предусматривает присоединение к 3'-концу последовательности нуклеиновой кислоты (b) последовательности нуклеиновой кислоты (c), которая, будучи транскрибированной, кодирует последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A.
Согласно одному варианту осуществления последовательности нуклеиновой кислоты (a), (b) и (c) могут быть транскрибированы с образованием общего транскрипта, в котором последовательности нуклеиновой кислоты, транскрибированные с последовательностей нуклеиновой кислоты (b) и необязательно (c), являются активными в отношении повышения эффективности трансляции и/или стабильности последовательности нуклеиновой кислоты, транскрибированной с транскрибируемой последовательности нуклеиновой кислоты (a).
Варианты осуществления 3'-нетранслируемой области и последовательности нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A, описаны выше для молекул нуклеиновой кислоты по настоящему изобретению.
Согласно дополнительному аспекту настоящее изобретение относится к способу получения пептида или белка, предусматривающему:
(i) получение РНК с помощью способа получения РНК по настоящему изобретению и
(ii) обеспечение трансляции РНК.
Способы по настоящему изобретению можно осуществлять in vitro или in vivo. Согласно одному варианту осуществления любого из способов по настоящему изобретению транскрипцию осуществляют in vitro.
Согласно одному варианту осуществления способ получения РНК или способ получения пептида или белка перед транскрипцией молекулы нуклеиновой кислоты дополнительно предусматривает расщепление молекулы нуклеиновой кислоты.
Согласно одному варианту осуществления расщепление в пределах последовательности нуклеиновой кислоты, которая, будучи транскрибированной, кодирует последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A, осуществляется таким образом, что в результате транскрипции полученной таким образом нуклеиновой кислоты образуется транскрипт, который на своем 3'-конце содержит указанную последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A, где 3'-концевой нуклеотид указанного транскрипта представляет собой нуклеотид A последовательности нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A.
Согласно всем аспектам способов в соответствии с настоящим изобретением расщепление предпочтительно осуществляют с использованием сайта расщепления рестриктазами, который предпочтительно представляет собой сайт расщепления рестриктазами для эндонуклеазы рестрикции типа IIS.
Согласно одному варианту осуществления последовательность распознавания для эндонуклеазы рестрикции типа IIS находится в пределах 5-26 пар оснований, предпочтительно 24-26 пар оснований ниже 3'-конца последовательности нуклеиновой кислоты, которая, будучи транскрибированной, кодирует последовательность нуклеиновой кислоты, которая представляет собой полиадениловую последовательность, необязательно содержащую, в пределах полиадениловой последовательности, последовательность из одного или нескольких последовательных нуклеотидов, содержащую нуклеотиды, отличные от нуклеотидов A.
Настоящее изобретение также относится к РНК, получаемой с помощью способов получения РНК в соответствии с настоящим изобретением.
Настоящее изобретение можно использовать, например, для повышения уровня экспрессии рекомбинантных белков при транскрипции и экспрессии в клетке. Более конкретно, в случае получении рекомбинантных белков для транскрипции рекомбинантных нуклеиновых кислот и экспрессии рекомбинантных белков в клеточных системах можно применять векторы экспрессии по настоящему изобретению. К такому случаю относится, например, получение рекомбинантных антител, гормонов, цитокинов, ферментов и т. д. Это позволяет, среди прочего, сократить производственные расходы.
Также молекулы нуклеиновой кислоты по настоящему изобретению можно использовать для применений в генной терапии. Соответственно, молекула нуклеиновой кислоты по настоящему изобретению может представлять собой вектор для генной терапии и ее применяют для экспрессии трансгена. С этой целью можно применять любые векторные системы на основе нуклеиновых кислот (ДНК/РНК)(например, плазмиды, аденовирусы, векторы на основе поксвируса, векторы на основе вируса гриппа, векторы на основе альфавируса и т. д.). Такими векторами можно трансфицировать клеткиin vitro, например, лимфоциты или дендритные клетки, или же in vivo посредством прямого введения.
РНК по настоящему изобретению (например, полученную с применением описанной в настоящем документе молекулы нуклеиновой кислоты в качестве матрицы для транскрипции) можно использовать, например, для транзиентной экспрессии генов, причем к возможным областям применения относятся вакцины на основе РНК, которые трансфицируют в клетки in vitro или непосредственно вводят in vivo, для транзиентной экспрессии функциональных рекомбинантных белков in vitro, например, с целью инициации процессов дифференцировки клеток или для изучения функций белков, и для транзиентной экспрессии функциональных рекомбинантных белков, таких как эритропоэтин, гормоны, ингибиторы коагуляции и т. п. in vivo, в частности, в качестве лекарственных препаратов.
РНК по настоящему изобретению можно применять, в частности, для трансфицирования антиген-представляющих клеток и, следовательно, в качестве инструмента для доставки представляемого антигена и для нагрузки антиген-представляющих клеток указанным представляемым антигеном, соответствующим пептиду или белку, экспрессированным с указанной РНК, или полученным из них, в частности, посредством внутриклеточного процессинга, такого как расщепление, т. е. представляемый антиген представляет собой, например, фрагмент пептида или белка, экспрессированных с РНК. Такие антиген-представляющие клетки можно применять для стимулирования T-клеток, в частности, CD4+ и/или CD8+ T-клеток.
Соответственно, согласно дополнительному аспекту настоящее изобретение относится к применению РНК по настоящему изобретению для трансфицирования клетки-хозяина. Согласно одному варианту осуществления клетка-хозяин представляет собой антиген-представляющую клетку, в частности дендритную клетку, моноцит или макрофаг.
Согласно дополнительному аспекту настоящее изобретение относится к применению РНК по настоящему изобретению для терапии, в частности для вакцинации.
Согласно дополнительному аспекту настоящее изобретение относится к фармацевтической композиции, такой как композиция, представляющая собой вакцину, содержащая РНК по настоящему изобретению.
Согласно дополнительному аспекту настоящее изобретение относится к РНК по настоящему изобретению для описанных в настоящем документе применений.
Подробное раскрытие настоящего изобретения
Хотя настоящее изобретение подробно описано ниже, следует понимать, что настоящее изобретение не ограничено описанными в настоящем документе конкретными методиками, протоколами и реагентами, поскольку они могут различаться. Также следует понимать, что применяемая в настоящем документе терминология предназначена только для описания конкретных вариантов осуществления, и не предназначена для ограничения объема настоящего изобретения, который будет ограничиваться только прилагаемой формулой изобретения. Если не указано иное, все применяемые в настоящем документе технические и научные термины имеют те же значения, которые обычно понятны рядовому специалисту в данной области.
Ниже будут описаны признаки настоящего изобретения. Такие признаки перечислены в конкретных вариантах осуществления, однако следует понимать, что их можно комбинировать любым способом и в любом количестве для создания дополнительных вариантов осуществления. Различные описанные примеры и предпочтительные варианты осуществления не должны толковаться как ограничивающие настоящее изобретение только до явно описанных вариантов осуществления. Следует понимать, что настоящее описание является основанием и охватывает варианты осуществления, которые объединяют явным образом описанные варианты осуществления с любым числом раскрытых и/или предпочтительных элементов. Кроме того, любые изменения и комбинации всех описанных признаков в настоящей заявке должны рассматриваться как раскрытые с помощью описания настоящей заявки, если контекстом не указано иное. Например, если согласно предпочтительному варианту осуществления последовательности из одного или нескольких последовательных нуклеотидов, содержащей нуклеотиды, отличные от нуклеотидов A, предшествуют по меньшей мере 20 остатков A в указанной полиадениловой последовательности, и если согласно другому предпочтительному варианту осуществления за последовательностью из одного или нескольких последовательных нуклеотидов, содержащей нуклеотиды, отличные от нуклеотидов A, расположены по меньшей мере 20 остатков A в указанной полиадениловой последовательности, имеется ввиду, что предпочтительным является вариант осуществления, в котором последовательности из одного или нескольких последовательных нуклеотидов, содержащей нуклеотиды, отличные от нуклеотидов A, предшествуют по меньшей мере 20 остатков A и за ней расположены по меньшей мере 20 остатков A в указанной полиадениловой последовательности.
Предпочтительно применяемые в настоящем документе термины определены, как описано в “A multilingual glossary of biotechnological terms: (IUPAC Recommendations)”, H.G.W. Leuenberger, B. Nagel, и H. Kölbl, Eds., Helvetica Chimica Acta, CH-4010 Basel, Switzerland, (1995).
При осуществлении настоящего изобретения на практике будут использовать, если не указано иное, традиционные в области химии, биохимии, клеточной биологии, иммунологии и методиках рекомбинантных ДНК способы, которые пояснены в литературе в данной области (ср., например, Molecular Cloning: A Laboratory Manual, 2nd Edition, J. Sambrook et al. eds., Cold Spring Harbor Laboratory Press, Cold Spring Harbor 1989).
В настоящем описании и нижеследующей формуле изобретения, если контекст не требует иного, слово «содержать» и варианты, такие как «содержит» и «содержащий», будут подразумевать включение заявленного элемента, целого числа или стадии или группы элементов, целых чисел или стадий, но не исключение любого другого элемента, целого числа или стадии или группы элементов, целых чисел или стадий. Формы единственного числа и аналогичные ссылки, применяемые в контексте описания настоящего изобретения (в особенности в контексте формулы изобретения), должны толковаться как охватывающие как единственное, так и множественное число, если в настоящем документе не указано иное или явно не противоречит контексту. Подразумевается, что описание диапазонов значений в настоящем документе служит лишь в качестве сокращенного способа индивидуального указания каждого отдельного значения, подпадающего под диапазон. Если не указано иное, в настоящем документе каждое отдельное значение включено в настоящее описание, как если бы оно было отдельно описано в настоящем документе. Все описанные в настоящем документе способы можно осуществлять в любом подходящем порядке, если в настоящем документе не указано иное или иным образом явно не противоречит контексту. Применение всех возможных примеров или уточняющих слов (например, «такой как»), предусмотренных в настоящем документе, предназначено просто для лучшей иллюстрации настоящего изобретения, и не является ограничением объема настоящего изобретения, заявленного иным образом. Никакая из формулировок в настоящем описании не должна истолковываться как указание какого-либо незаявленного элемента, необходимого для осуществления настоящего изобретения на практике.
В тексте настоящего описания приведены несколько документов. Каждый из документов, приведенных в настоящем документе (в том числе все патенты, патентные заявки, научные публикации, спецификации производителя, инструкции и т. д.), независимо от того, приведены они выше или ниже, включены в настоящий документ посредством ссылки во всей своей полноте. Ничто в настоящем документе не должно толковаться как признание того, что настоящее изобретение не имеет права претендовать на первенство в подаче настоящего раскрытия в силу предшествующего изобретения.
В настоящем изобретении описаны молекулы нуклеиновой кислоты, такие как ДНК-плазмиды, пригодные в качестве векторов для экспрессии РНК, содержащих последовательности нуклеиновой кислоты, кодирующие модифицированные 3'-нетранслируемые области (UTR) в РНК, оказывающие стабилизирующий эффект в отношении РНК и/или обеспечивающие повышение эффективности трансляции РНК.
Выражение «последовательность нуклеиновой кислоты, которая, будучи транскрибированной, кодирует 3'-нетранслируемую область транскрипта» относится к последовательности нуклеиновой кислоты,предусматривающей матричную нить, кодирующую указанную 3'-нетранслируемую область. Предпочтительно указанная последовательность нуклеиновой кислоты предусматривает кодирующую нить, содержащую ту же последовательность нуклеиновой кислоты, что и указанная 3'-нетранслируемая область образующегося РНК-транскрипта (за исключением замены тимина на урацил). Таким образом, в соответствии с настоящим изобретением «последовательность нуклеиновой кислоты, которая, будучи транскрибированной, кодирует 3'-нетранслируемую область транскрипта» согласно одному варианту осуществления предусматривает кодирующую нить, содержащую упоминаемую в настоящем документе 3'-нетранслируемую область (за исключением замены тимина на урацил).
Термин «FCGRT» относится к «Fc-фрагмент IgG, рецептор, транспортер, альфа» и относится к гену FCGRT. Этот ген кодирует рецептор, который связывает Fc-область мономерного иммуноглобулина G. Кодируемый белок переносит антитела, иммуноглобулины G, от матери к плоду через плаценту. Этот белок также связывает иммуноглобулин G для защиты антитела от разрушения.
Выражение «последовательность нуклеиновой кислоты 3'-нетранслируемой области FCGRT, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента» относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 1-50 из перечня последовательностей или ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента. Согласно одному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 1-50. Согласно одному особенно предпочтительному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты под SEQ ID NO: 27, или содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты под SEQ ID NO: 27.
Термин «LSP1» относится к специфичному для лимфоцитов белку 1 и относится к гену LSP1. Этот ген кодирует белок, связывающий внутриклеточный F-актин. Упоминаемый белок экспрессируется в лимфоцитах, нейтрофилах, макрофагах и эндотелии и может регулировать подвижность, адгезивную способность в отношении белков матрикса, представляющих собой фибриноген, и трансэндотелиальную миграцию нейтрофилов.
Выражение «последовательность нуклеиновой кислоты 3'-нетранслируемой области LSP1, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента» относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 51-72 из перечня последовательностей или ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента. Согласно одному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 51-72. Согласно одному особенно предпочтительному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты под SEQ ID NO: 52, или содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты под SEQ ID NO: 52.
Термин «CCL22» относится к хемокиновому лиганду (с мотивом C-C) 22 и относится к гену CCL22. Продукт этого гена связывается с рецептором хемокинов CCR4. Этот хемокин может играть роль в миграции активированных T-лимфоцитов к участкам воспаления и других аспектах физиологии активированных T-лимфоцитов.
Выражение «последовательность нуклеиновой кислоты 3'-нетранслируемой области CCL22, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента» относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 73-85 из перечня последовательностей или ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента. Согласно одному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 73-85. Согласно одному особенно предпочтительному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты под SEQ ID NO: 79, или содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты под SEQ ID NO: 79.
Термин «AES» относится к амино-концевому энхансеру Split и относится к гену AES. Белок, кодируемый этим геном, принадлежит к семейству белков groucho/TLE, может функционировать как гомоолигомер или как гетероолигомер вместе с другими представителями семейства преимущественно для репрессии экспрессии генов других представителей семейства.
Выражение «последовательность нуклеиновой кислоты 3'-нетранслируемой области AES, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента» относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 86-89 из перечня последовательностей или ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента. Согласно одному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 86-89. Согласно одному особенно предпочтительному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты под SEQ ID NO: 86, или содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты под SEQ ID NO: 86. Согласно одному особенно предпочтительному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, охватывающей положения 1-68, положения 1-102, положения 35-102, положения 35-136 или положения 68-136 SEQ ID NO: 86, или содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты, охватывающей положения 1-68, положения 1-102, положения 35-102, положения 35-136 или положения 68-136 SEQ ID NO: 86.
Термин «PLD3» относится к представителю 3 семейства фосфолипаз D и относится к гену PLD3. Этот ген кодирует представителя 3 семейства фосфолипаз D (PLD), ферментов, которые катализируют гидролиз мембранных фосфолипидов. Кодируемый белок представляет собой мембранный белок типа II, который пересекает мембрану один раз, и он содержит два фосфодиэстеразных домена PLD. Этот белок оказывает влияние на процессинг белка-предшественника бета-амилоида. Мутации в этом гене ассоциированы с риском развития болезни Альцгеймера.
Выражение «последовательность нуклеиновой кислоты 3'-нетранслируемой области PLD3, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента» относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 90-104 из перечня последовательностей или ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента. Согласно одному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 90-104. Согласно одному особенно предпочтительному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты под SEQ ID NO: 96, или содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты под SEQ ID NO: 96.
Термин «MT_RNR1» относится к кодируемой в митохондриях 12S РНК и относится к гену MT_RNR1. Этот ген РНК принадлежит к классу Mt_rRNA. К ассоциированным с MT-RNR1 заболеваниям относятся рестриктивная кардиомиопатия и слуховая нейропатия. Среди связанных с ним сигнальных путей - биогенез рибосом у эукариот и путь регуляции точности трансляции CFTR (мутации класса I).
Выражение «последовательность нуклеиновой кислоты 3'-нетранслируемой области MT_RNR1, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента» относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 105-121 из перечня последовательностей или ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента. Согласно одному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 105-121. Согласно одному особенно предпочтительному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты под SEQ ID NO: 115, или содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты под SEQ ID NO: 115. Согласно одному особенно предпочтительному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, охватывающей положения 1-71, положения 1-107, положения 37-107, положения 37-142 или положения 71-142 SEQ ID NO: 115, или содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты, охватывающей положения 1-71, положения 1-107, положения 37-107, положения 37-142 или положения 71-142 SEQ ID NO: 115.
Термин «HLA-DRB4» относится к DR-бета 4 главного комплекса гистосовместимости класса II и относится к гену HLA-DRB4. HLA-DRB4 относится к паралогам бета-цепи HLA класса II. Эта молекула класса II представляет собой гетеродимер, состоящий из альфа-цепи (DRA) и бета-цепи (DRB), при этом обе заякорены в мембране. Она играет центральную роль в иммунной системе, заключающуюся в представлении пептидов, происходящих из внеклеточных белков. Молекулы класса II экспрессируются в антиген-представляющих клетках (APC: B-лимфоциты, дендритные клетки, макрофаги).
Выражение «последовательность нуклеиновой кислоты 3'-нетранслируемой области HLA-DRB4, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента» относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 122-143 из перечня последовательностей или ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента. Согласно одному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 122-143. Согласно одному особенно предпочтительному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты под SEQ ID NO: 126, или содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты под SEQ ID NO: 126.
Выражение «любая комбинация двух или более последовательностей нуклеиновой кислоты, фрагментов и/или вариантов» в отношении последовательностей нуклеиновой кислоты 3'-нетранслируемых областей определенных генов, их фрагментов или вариантов указанных последовательностей нуклеиновой кислоты или фрагментов означает, что 2 или более, 3 или более или 4 или более и предпочтительно до 6 или до 5 указанных последовательностей нуклеиновой кислоты, фрагментов и/или вариантов выстроены линейно по принципу «голова к хвосту», при этом необязательно разнесены посредством линкеров. Согласно одному варианту осуществления комбинация двух или более последовательностей нуклеиновой кислоты, фрагментов и/или вариантов предусматривает две(два) или более отличающиеся(отличающихся) и/или две(два) или более идентичные(идентичных) последовательности нуклеиновой кислоты, фрагментов и/или вариантов. Согласно одному варианту осуществления комбинация двух или более последовательностей нуклеиновой кислоты, фрагментов и/или вариантов предусматривает две(два) или более отличающиеся(отличающихся) последовательности нуклеиновой кислоты, фрагментов и/или вариантов 3'-нетранслируемой области одного и тоже же и/или отличающихся генов.
Согласно одному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 144-220, предпочтительно SEQ ID NO: 174 и 208-220. Согласно одному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 144-220, предпочтительно SEQ ID NO: 174 и 208-220 или ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента. Согласно одному особенно предпочтительному варианту осуществления упоминаемое выражение относится к последовательности нуклеиновой кислоты, содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты под SEQ ID NO: 174, или содержащей, предпочтительно состоящей из последовательности нуклеиновой кислоты, которая по меньшей мере на 90%, предпочтительно по меньшей мере на 95%, более предпочтительно по меньшей мере на 98% идентична последовательности нуклеиновой кислоты под SEQ ID NO: 174.
Термин «линкер» в соответствии с настоящим изобретением относится к последовательности нуклеиновой кислоты, добавленной между двумя последовательностями нуклеиновой кислоты с соединением указанных двух последовательностей нуклеиновой кислоты. В отношении линкерной последовательности нет никаких особых ограничений.
В соответствии с настоящим изобретением молекула нуклеиновой кислоты или последовательность нуклеиновой кислоты относится к нуклеиновой кислоте, которая предпочтительно представляет собой дезоксирибонуклеиновую кислоту (ДНК) или рибонуклеиновую кислоту (РНК). В соответствии с настоящим изобретением нуклеиновые кислоты предусматривают геномную ДНК, кДНК, мРНК, полученные рекомбинантным путем и химически синтезированные молекулы. В соответствии с настоящим изобретением нуклеиновая кислота может находиться в форме однонитевой или двунитевой и линейной или ковалентно замкнутой кольцевой молекулы.
В контексте настоящего изобретения термин «РНК» относится к молекуле, которая содержит рибонуклеотидные остатки и предпочтительно полностью или преимущественно состоит из рибонуклеотидных остатков. Термин «рибонуклеотид» относится к нуклеотиду с гидроксильной группой в 2'-положении β-D-рибофуранозильной группы. Термин «РНК» предусматривает двунитевую РНК, однонитевую РНК, выделенную РНК, такую как частично или полностью очищенная РНК, в основном чистую РНК, синтетическую РНК и полученную рекомбинантным путем РНК, такую как модифицированная РНК, которая отличается от встречающейся в природе РНК добавлением, делецией, заменой и/или изменением одного или нескольких нуклеотидов. Такие изменения могут включать добавление вещества, отличного от нуклеотида, например, к концу(концам) РНК или внутрь нее, например, к одному или нескольким нуклеотидам РНК. Нуклеотиды в молекулах РНК могут также предусматривать нестандартные нуклеотиды, такие как не встречающиеся в природе нуклеотиды или химически синтезированные нуклеотиды или дезоксинуклеотиды. Такие измененные РНК могут называться аналогами, в частности, аналогами встречающихся в природе РНК. В соответствии с настоящим изобретением РНК включает мРНК.
Термин «мРНК» означает «матричную РНК» и относится к транскрипту, который получают с применением ДНК-матрицы и который кодирует пептид или белок. Как правило, мРНК содержит 5'-UTR, белок-кодирующую область, 3'-UTR и поли(A)-последовательность. мРНК может быть получена в результате транскрипции с ДНК-матрицы in vitro. Методика по in vitro транскрипции известна специалисту в данной области. Например, существует ряд коммерчески доступных наборов для in vitro транскрипции. В соответствии с настоящим изобретением в дополнение к модификациям в соответствии с настоящим изобретением мРНК можно модифицировать с помощью дополнительных стабилизирующих модификаций и с помощью присоединения кэпа.
Согласно одному варианту осуществления настоящего изобретения РНК представляет собой самореплицирующуюся РНК, такую как однонитевая самореплицирующаяся РНК. Согласно одному варианту осуществления самореплицирующаяся РНК представляет собой однонитевую плюс-смысловую РНК. Согласно одному варианту осуществления самореплицирующаяся РНК представляет собой вирусную РНК или РНК, происходящую из вирусной РНК. Согласно одному варианту осуществления самореплицирующаяся РНК представляет собой геномную РНК альфа-вируса или происходит из геномной РНК альфа-вируса. Согласно одному варианту осуществления самореплицирующаяся РНК представляет собой вектор экспрессии на основе вирусных генов. Согласно одному варианту осуществления вирус представляет собой вирус леса Семлики. Согласно одному варианту осуществления самореплицирующаяся РНК предусматривает один или несколько трансгенов. Согласно одному варианту осуществления, если РНК представляет собой вирусную РНК или происходит из вирусной РНК, трансгены могут частично или полностью заменять вирусные последовательности, такие как вирусные последовательности, кодирующие структурные белки. Согласно одному варианту осуществления самореплицирующаяся РНК представляет собой транскрибированную in vitro РНК.
Термин «5'-кэп» относится к кэп-структуре, которая находится на 5'-конце молекулы мРНК и обычно состоит из гуанозинового нуклеотида, присоединенного к мРНК посредством нетипичной 5'-5'-трифосфатной связи. Согласно одному варианту осуществления такой гуанозин метилирован по 7-положению. Термин «типичный 5'-кэп» относится к 5'-кэпу встречающейся в природе РНК, предпочтительно к кэпу, представляющему собой 7-метилгуанозин (m7G). В контексте настоящего изобретения термин «5'-кэп» включает аналог 5'-кэпа, который напоминает кэп-структуру РНК и модифицирован таким образом, что он обладает способностью стабилизировать РНК, когда присоединен к ней, предпочтительно in vivo и/или в клетке. Обеспечение РНК 5'-кэпом или аналогом 5'-кэпа может быть осуществлено посредством in vitro транскрипции ДНК-матрицы в присутствии указанного 5'-кэпа или аналога 5'-кэпа, где указанный 5'-кэп включается в образующуюся нить РНК в процессе транскрипции или РНК может быть образована, например, посредством in vitro транскрипции, а 5'-кэп может быть образован посттранскрипционно с помощью кэпирующих ферментов, например, кэпирующих ферментов вируса коровьей оспы.
Термин «нуклеиновая кислота» в соответствии с настоящим изобретением также предусматривает химическую дериватизацию нуклеиновой кислоты по нуклеотидному основанию, по сахару или по фосфату, а также нуклеиновые кислоты, содержащие синтетические нуклеотиды и аналоги нуклеотидов.
«Фрагмент» или «фрагмент последовательности нуклеиновой кислоты» относится к части последовательности нуклеиновой кислоты, т. е. последовательности, которая представляет собой последовательность нуклеиновой кислоты, укороченной с 5'- и/или 3'-конца(концов). Предпочтительно фрагмент, если он заменяет указанную последовательность нуклеиновой кислоты в молекуле РНК, сохраняет стабильность и/или эффективность трансляции РНК. Предпочтительно фрагмент последовательности нуклеиновой кислоты содержит по меньшей мере 80%, предпочтительно по меньшей мере 90%, 95%, 96%, 97%, 98% или 99% нуклеотидных остатков указанной последовательности нуклеиновой кислоты.
Термин «вариант» в отношении, например, последовательностей нуклеиновых кислот и аминокислотных последовательностей, в соответствии с настоящим изобретением включает любые варианты, в частности мутанты, сплайс-варианты, конформации, изоформы, аллельные варианты, видовые варианты и видовые гомологи, в частности таковые, существующие в природе. Аллельный вариант относится к изменению обычной последовательности гена, значение которого зачастую не установлено. С помощью полного секвенирования гена обычно идентифицируют множество аллельных вариантов для данного гена. Видовой гомолог представляет собой последовательность нуклеиновой кислоты или аминокислотную последовательность, происходящую от другого вида по сравнению с данной последовательностью нуклеиновой кислоты или аминокислотной последовательностью.
В соответствии с настоящим изобретением к вариантам нуклеиновой кислоты относятся делеции, добавления, мутации и/или вставки по одному или нескольким нуклеотидам по сравнению с эталонной нуклеиновой кислотой. Делеции предусматривают удаление одного или нескольких нуклеотидов из эталонной нуклеиновой кислоты. Варианты с добавлениями содержат 5'- и/или 3'-концевые слияния с одним или несколькими нуклеотидами, например, 1, 2, 3, 5, 10, 20, 30, 50 или более нуклеотидами. К мутациям могут относится без ограничения замены, где по меньшей мере один нуклеотид в последовательности удаляют, а другой нуклеотид вставляют на его место (как, например, трансверсии и транзиции), участки с удаленными азотистыми основаниями, участки с перекрестными сшивками и измененные химическим путем или модифицированные основания. К вставкам относится добавление по меньшей мере одного нуклеотида в эталонную нуклеиновую кислоту.
В отношении молекул нуклеиновой кислоты термин «вариант» включает вырожденные последовательности нуклеиновой кислоты, где вырожденная нуклеиновая кислота в соответствии с настоящим изобретением представляет собой нуклеиновую кислоту, которая отличается от эталонной нуклеиновой кислоты последовательностью кодонов ввиду вырожденности генетического кода.
Предпочтительно степень идентичности между данной последовательностью нуклеиновой кислоты и последовательностью нуклеиновой кислоты, которая представляет собой вариант указанной данной последовательности нуклеиновой кислоты, будет составлять по меньшей мере 70%, предпочтительно по меньшей мере 75%, предпочтительно по меньшей мере 80%, более предпочтительно по меньшей мере 85%, еще более предпочтительно по меньшей мере 90% или наиболее предпочтительно по меньшей мере 95%, 96%, 97%, 98% или 99%. Степень идентичности предпочтительно учитывается для области по меньшей мере из приблизительно 30, по меньшей мере приблизительно 50, по меньшей мере приблизительно 70, по меньшей мере приблизительно 90, по меньшей мере приблизительно 100, по меньшей мере приблизительно 150, по меньшей мере приблизительно 200, по меньшей мере приблизительно 250, по меньшей мере приблизительно 300 или по меньшей мере приблизительно 400 нуклеотидов. Согласно предпочтительным вариантам осуществления степень идентичности учитывается для полной длины эталонной последовательности нуклеиновой кислоты.
«Сходство последовательностей» означает процентную долю аминокислот, которые являются либо идентичными, либо представляют собой консервативные аминокислотные замены. «Идентичность последовательностей» между двумя полипептидными последовательностями или последовательностями нуклеиновой кислоты означает процентную долю аминокислот или нуклеотидов, которые являются идентичными среди последовательностей.
Подразумевается, что термин «% идентичность» относится, в частности, к процентной доле нуклеотидов, которые являются идентичными при оптимальном выравнивании двух сравниваемых последовательностей, причем указанная процентная доля является чисто статистической, и различия между двумя последовательностями могут случайным образом распределятся по всей длине последовательности, а сравниваемая последовательность может содержать добавления или делеции по сравнению с эталонной последовательностью, для получения оптимального выравнивания двух последовательностей. Для идентификации локальных областей соответствующих последовательностей, как правило, выполняют сравнения двух последовательностей путем сравнения указанных последовательностей, после оптимального выравнивания, относительно сегмента или «окна сравнения». Оптимальное выравнивание для сравнения можно выполнять вручную или с помощью алгоритма поиска локальной гомологии по Смиту-Уотерману, 1981, Ads App. Math. 2, 482, с помощью алгоритма поиска локальной гомологии по Нидлману-Вуншу, 1970, J. Mol. Biol. 48, 443 и с помощью алгоритма поиска сходства по Пирсону-Липману, 1988, Proc. Natl Acad. Sci. USA 85, 2444 или с помощью компьютерных программ с применением указанных алгоритмов (GAP, BESTFIT, FASTA, BLAST P, BLAST N и TFASTA в пакете программного обеспечения Wisconsin Genetics, Genetics Computer Group, 575 Science Drive, Мадисон, Висконсин).
Процентную идентичность получают путем определения числа идентичных положений, по которым сравниваемые последовательности совпадают, деления этого числа на число сравниваемых положений и умножения этого результата на 100.
Например, можно применять программу «BLAST 2 sequences» в рамках BLAST, которая доступна на веб-сайте http://www.ncbi.nlm.nih.gov/blast/bl2seq/wblast2.cgi.
Нуклеиновая кислота «способна гибридизироваться» или «гибридизируется» с другой нуклеиновой кислотой, если две последовательности являются комплементарными одна другой. Нуклеиновая кислота является «комплементарной» другой нуклеиновой кислоте, если две последовательности способны образовывать одна с другой стабильный дуплекс. В соответствии с настоящим изобретением гибридизацию предпочтительно осуществляют в условиях, при которых обеспечивается специфичная гибридизация полинуклеотидов (жесткие условия). Жесткие условия описаны, например, в Molecular Cloning: A Laboratory Manual, J. Sambrook et al., Editors, 2nd Edition, Cold Spring Harbor Laboratory press, Cold Spring Harbor, New York, 1989 или Current Protocols in Molecular Biology, F.M. Ausubel et al., Editors, John Wiley & Sons, Inc., New York и относятся, например, к гибридизации при 65°C в буфере для гибридизации (3,5 × SSC, 0,02% фиколл, 0,02% поливинилпирролидон, 0,02% бычий сывороточный альбумин, 2,5 мМ NaH2PO4 (pH 7), 0,5% SDS, 2 мМ EDTA). SSC представляет собой 0,15 M хлорид натрия/0,15 M цитрат натрия, pH 7. После гибридизации мембрану, на которую была перенесена ДНК, промывают, например, в 2 × SSC при комнатной температуре, а затем в 0,1-0,5 × SSC/0,1 × SDS при температурах, не превышающих 68°C.
Процентная комплементарность означает процентную долю смежных остатков в молекуле нуклеиновой кислоты, которая может образовывать водородные связи (например, спаривание оснований по Уотсону-Крику) со второй последовательностью нуклеиновой кислоты (например, 5, 6, 7, 8, 9, 10 из 10 соответствует 50%, 60%, 70%, 80%, 90% и 100% комплементарности). «Идеально комплементарный» или «полностью комплементарный» означает, что все смежные остатки последовательности нуклеиновой кислоты будут образовывать водородные связи с тем же числом смежных остатков во второй последовательности нуклеиновой кислоты. Предпочтительно степень комплементарности в соответствии с настоящим изобретением составляет по меньшей мере 70%, предпочтительно по меньшей мере 75%, предпочтительно по меньшей мере 80%, более предпочтительно по меньшей мере 85%, еще более предпочтительно по меньшей мере 90% или наиболее предпочтительно по меньшей мере 95%, 96%, 97%, 98% или 99%. Наиболее предпочтительно степень комплементарности в соответствии с настоящим изобретением составляет 100%.
Термин «производное» предусматривает любую химическую дериватизацию нуклеиновой кислоты по нуклеотидному основанию, по сахару или по фосфату. Термин «производное» также предусматривает нуклеиновые кислоты, которые содержат не встречающиеся в природе нуклеотиды и аналоги нуклеотидов. Предпочтительно дериватизация нуклеиновой кислоты повышает ее стабильность.
Фрагменты или варианты конкретных последовательностей нуклеиновой кислоты или последовательностей нуклеиновой кислоты, характеризующихся определенной степенью идентичности с конкретными последовательностями нуклеиновой кислоты, предпочтительно обладают по меньшей мере одним функциональным свойством указанных конкретных последовательностей и предпочтительно являются функционально эквивалентными указанным конкретным последовательностям, например, последовательностям нуклеиновой кислоты, характеризующимся свойствами, идентичными или подобными таковым у конкретных последовательностей нуклеиновой кислоты.
Одним важным свойством является сохранение или улучшение стабильности молекулы РНК и/или эффективности трансляции, и оно включает, в частности, способность повышать, будучи в функциональной связи с нуклеиновой кислотой, которая может быть транскрибирована в РНК (транскрибируемая последовательность нуклеиновой кислоты) или последовательностью нуклеиновой кислоты, кодирующей пептид или белок, стабильность и/или эффективность трансляции РНК, образующейся на основе этой нуклеиновой кислоты или последовательности нуклеиновой кислоты, кодирующей пептид или белок, в полной молекуле РНК.
Согласно одному варианту осуществления, если конкретная последовательность нуклеиновой кислоты является активной в отношении повышения эффективности трансляции и/или стабильности другой последовательности нуклеиновой кислоты, то фрагмент или вариант конкретной последовательности нуклеиновой кислоты или последовательности нуклеиновой кислоты, характеризующейся определенной степенью идентичности с конкретной последовательностью нуклеиновой кислоты, также является активным в отношении повышения эффективности трансляции и/или стабильности другой последовательности нуклеиновой кислоты (если он заменяет конкретную последовательность нуклеиновой кислоты). Фрагмент или вариант конкретной последовательности нуклеиновой кислоты или последовательности нуклеиновой кислоты, характеризующейся определенной степенью идентичности с конкретной последовательностью нуклеиновой кислоты, может быть активным в той же степени, или может быть более активным, чем конкретная последовательность нуклеиновой кислоты, или активность фрагмента или варианта конкретной последовательности нуклеиновой кислоты или последовательности нуклеиновой кислоты, характеризующейся определенной степенью идентичности с конкретной последовательностью нуклеиновой кислоты, может составлять по меньшей мере 50%, по меньшей мере 60%, по меньшей мере 70%, по меньшей мере 80% или по меньшей мере 90% активности конкретной последовательности нуклеиновой кислоты.
В соответствии с настоящим изобретением «функциональная связь» или «функционально связанный» относится к связи в пределах функционального взаимодействия. Нуклеиновая кислота является «функционально связанной», если она функционально зависит от другой последовательности нуклеиновой кислоты. Например, промотор является функционально связанным с кодирующей последовательностью, если он влияет на транскрипцию указанной кодирующей последовательности. Функционально связанные нуклеиновые кислоты, как правило, прилегают друг к другу, если необходимо, разделены дополнительными последовательностями нуклеиновой кислоты, и, согласно конкретным вариантам осуществления, транскрибируются с помощью РНК-полимеразы с образованием одной молекулы РНК (общий транскрипт). Предпочтительно последовательность, которая является вариантом по отношению к конкретной последовательности, если она заменяет конкретную последовательность в молекуле РНК, сохраняет стабильность и/или эффективность трансляции РНК.
В соответствии с настоящим изобретением «последовательность нуклеиновой кислоты, которая происходит из последовательности нуклеиновой кислоты» относится к нуклеиновой кислот, которая является вариантом нуклеиновой кислоты, из которой она происходит.
«3'-конец нуклеиновой кислоты» в соответствии с настоящим изобретением относится к тому концу, который имеет свободную гидроксильную группу. На схематическом изображении двунитевых нуклеиновых кислот, в частности ДНК, 3'-конец всегда находится в правой части. «5'-конец нуклеиновой кислоты» в соответствии с настоящим изобретением относится к тому концу, который имеет свободную фосфатную группу. На схематическом изображении двунитевых нуклеиновых кислот, в частности ДНК, 5'-конец всегда находится в левой части.
Согласно конкретным вариантам осуществления нуклеиновая кислота функционально связана, в соответствии с настоящим изобретением, с последовательностями контроля экспрессии, которые могут быть гомологичными или гетерологичными по отношению к нуклеиновой кислоте.
Транскрибируемая последовательность нуклеиновой кислоты, в частности последовательность нуклеиновой кислоты, кодирующая пептид или белок, и последовательность контроля экспрессии «функционально» связаны друг с другом, если они связаны ковалентно друг с другом таким образом, что транскрипция или экспрессия транскрибируемой и, в частности, кодирующей последовательности нуклеиновой кислоты находится под контролем или под влиянием последовательности контроля экспрессии. Если последовательность нуклеиновой кислоты подлежит трансляции в функциональный пептид или белок, индукция последовательности контроля экспрессии, функционально связанной с кодирующей последовательностью, приводит в результате к транскрипции указанной кодирующей последовательности, причем она не приводит к смещению рамки считывания в кодирующей последовательности или неспособности кодирующей последовательности транслироваться в пептид или белок.
Термин «последовательность контроля экспрессии» в соответствии с настоящим изобретением предусматривает промоторы, последовательности связывания рибосом и другие элементы контроля, которые осуществляют контроль транскрипции гена или трансляции результирующей РНК. Согласно конкретным вариантам осуществления настоящего изобретения можно осуществлять регуляцию последовательностей контроля экспрессии. Конкретная структура последовательностей контроля экспрессии может варьировать в зависимости от вида или типа клетки, но, как правило, включает 5'-нетранскрибируемые и 5'- и 3'-нетранслируемые последовательности, вовлеченные в инициацию транскрипции и трансляции соответственно, такие как TATA-бокс, кэпирующая последовательность, последовательность CAAT и т. д. Более конкретно, 5'-нетранскрибируемые последовательности контроля экспрессии включают промоторную область, которая охватывает промоторную последовательность для контроля транскрипции функционально связанного гена. Последовательности контроля экспрессии могут также включать энхансерные последовательности или вышележащие активаторные последовательности.
Определенные в настоящем документе последовательности нуклеиновой кислоты, в частности транскрибируемые и кодирующие последовательности нуклеиновой кислоты, можно объединять с любыми последовательностями контроля экспрессии, в частности промоторами, которые могут быть гомологичными или гетерологичными по отношению к указанным последовательностям нуклеиновой кислоты, причем термин «гомологичный» относится к тому факту, что последовательность нуклеиновой кислоты в естественных условиях также функционально связана с последовательностью контроля экспрессии, а термин «гетерологичный» относится к тому факту, что последовательность нуклеиновой кислоты в естественных условиях не связана функционально с последовательностью контроля экспрессии.
Термин «промотор» или «промоторная область» относится к последовательности ДНК, расположенной выше (5') от кодирующей последовательности гена, которая осуществляет контроль экспрессии указанной кодирующей последовательности путем обеспечения сайтов распознавания и связывания для РНК-полимеразы. Промоторная область может включать дополнительные сайты распознавания или связывания для дополнительных факторов, вовлеченных в регулирование транскрипции указанного гена. Промотор может осуществлять контроль транскрипции гена прокариотического или эукариотического организма. Промотор может быть «индуцируемым» и может инициировать транскрипцию в ответ на воздействие индуктора, или может быть «конститутивным», в случае если транскрипция не контролируется индуктором. Если индуктор отсутствует, индуцируемый промотор экспрессируется только в очень небольшой степени, или не экспрессируется вовсе. В присутствии индуктора ген «включается» или повышается уровень транскрипции. Этот процесс, как правило, опосредован связыванием специфичного фактора транскрипции.
Примерами промоторов, предпочтительных в соответствии с настоящим изобретением, являются промоторы для полимеразы SP6, T3 или T7.
В соответствии с настоящим изобретением термин «экспрессия» применяют в его наиболее общем значении и он предусматривает продукцию РНК или РНК и белка. Он также предусматривает частичную экспрессию нуклеиновых кислот. Кроме того, экспрессия может быть транзиентной или стабильной. В отношении РНК термин «экспрессия» или «трансляция» относится к процессу, осуществляемому на рибосомах клетки, посредством которого нить матричной РНК направляет сборку последовательности аминокислот с образованием пептида или белка.
Выражение «последовательности нуклеиновой кислоты, которые могут быть транскрибированы с образованием общего транскрипта» означает, что указанные последовательности нуклеиновой кислоты функционально связаны друг с другом таким образом, что, когда это уместно, после линеаризации, например, посредством расщепления ферментами рестрикции молекулы нуклеиновой кислоты, содержащей указанные последовательности нуклеиновой кислоты, в частности, замкнутой кольцевой молекулы нуклеиновой кислоты, транскрипция под контролем промотора приводит к образованию молекулы РНК, содержащей транскрипты указанных последовательностей нуклеиновой кислоты, ковалентно связанных друг с другом, когда это уместно, разделенных последовательностями, расположенными между ними.
В контексте настоящего изобретения термин «транскрипция» относится к процессу, в котором генетический код последовательности ДНК транскрибируется в РНК. Впоследствии РНК может быть транслирована в белок. В соответствии с настоящим изобретением термин «транскрипция» предусматривает «in vitro транскрипцию», причем термин «in vitro транскрипция» относится к процессу, в котором РНК, в частности мРНК, синтезируется in vitro в бесклеточной системе. Предпочтительно для получения транскриптов применяют клонирующие векторы. Такие клонирующие векторы обычно обозначаются как векторы транскрипции и в соответствии с настоящим изобретением охватываются термином «вектор». В соответствии с настоящим изобретением РНК предпочтительно представляет собой РНК, транскрибированную in vitro (IVT-РНК), и она может быть получена посредством in vitro транскрипции соответствующей ДНК-матрицы. Промотор для осуществления контроля транскрипции может представлять собой любой промотор для любой РНК-полимеразы. ДНК-матрица для in vitro транскрипции может быть получена путем клонирования нуклеиновой кислоты, в частности кДНК, и введения ее в соответствующий вектор для in vitro транскрипции. кДНК может быть получена посредством обратной транскрипции РНК.
Термин «последовательность нуклеиновой кислоты, транскрибированная с последовательности нуклеиновой кислоты» относится к РНК, когда это уместно, в виде части полной молекулы РНК, которая является продуктом транскрипции последней последовательности нуклеиновой кислоты.
Термин «последовательность нуклеиновой кислоты, которая является активной в отношении повышения эффективности трансляции и/или стабильности последовательности нуклеиновой кислоты» означает, что первая последовательность нуклеиновой кислоты способна модифицировать, в пределах общего транскрипта со второй последовательностью нуклеиновой кислоты, эффективность трансляции и/или стабильность указанной второй последовательности нуклеиновой кислоты таким образом, что указанные эффективность трансляции и/или стабильность повышаются в сравнении с эффективностью трансляции и/или стабильностью указанной второй последовательности нуклеиновой кислоты без указанной первой последовательности нуклеиновой кислоты. В этом контексте термин «эффективность трансляции» относится к количеству продукта трансляции, обеспечиваемому молекулой РНК за конкретный период времени, а термин «стабильность» относится к времени полужизни молекулы РНК.
Модификация и, следовательно, стабилизация и/или повышение эффективности трансляции РНК в соответствии с настоящим изобретением могут быть достигнуты с помощью генетически модифицированных молекул нуклеиновой кислоты по настоящему изобретению при применении их в качестве векторов экспрессии таким образом, что они обеспечивают транскрипцию РНК с описанными в настоящем документе 3'-нетранслируемыми областями на ее 3'-конце, и предпочтительно расположенными между последовательностью, кодирующей пептид или белок (открытая рамка считывания), и поли(A)-последовательностью.
Термин «3'-нетранслируемая область» относится к области, которая расположена на 3'-конце гена ниже кодона терминации белок-кодирующей области, и которая является транскрибированной, но не транслированной в аминокислотную последовательность, или к соответствующий области в молекуле РНК.
В соответствии с настоящим изобретением считается, что первая полинуклеотидная область расположена ниже второй полинуклеотидной области, если 5'-конец указанной первой полинуклеотидной области является частью указанной первой полинуклеотидной области, ближайшей к 3'-концу указанной второй полинуклеотидной области.
3'-нетранслируемая область, как правило, простирается от кодона терминации для продукта трансляции до поли(A)-последовательности, которая, как правило, присоединяется после процесса транскрипции. 3'-нетранслируемые области мРНК млекопитающих, как правило, содержат область гомологии, известную как гексануклеотидная последовательность AAUAAA. Эта последовательность, по-видимому, является сигналом присоединения поли(A) и часто расположена на 10-30 оснований выше сайта присоединения поли(A).
3'-Нетранслируемые области могут содержать один или несколько инвертированных повторов, которые могут складываться с образованием структур «петля на стебле», которые выполняют функцию барьеров для экзорибонуклеаз или взаимодействуют с белками, которые, как известно, повышают стабильность РНК (например, РНК-связывающие белки).
5'- И/или 3'-нетранслируемые области в соответствии с настоящим изобретением могут быть функционально связаны с транскрибируемой и, в частности, кодирующей нуклеиновой кислотой, следовательно, эти области должны быть ассоциированы с нуклеиновой кислотой таким образом, чтобы повышались стабильность и/или эффективность трансляции РНК, транскрибированной с указанной транскрибируемой нуклеиновой кислоты.
3'-Нетранслируемые области мРНК иммуноглобулинов являются относительно короткими (менее приблизительно 300 нуклеотидов), тогда как 3'-нетранслируемые области других генов являются относительно длинными. Например, длина 3'-нетранслируемой области tPA составляет приблизительно 800 нуклеотидов, длина таковой для фактора VIII составляет приблизительно 1800 нуклеотидов, а длина таковой для эритропоэтина составляет приблизительно 560 нуклеотидов.
В соответствии с настоящим изобретением можно определять, повышает ли 3'-нетранслируемая область или происходящая из нее последовательность нуклеиновой кислоты стабильность и/или эффективность трансляции РНК, путем включения 3'-нетранслируемой области или происходящей из нее последовательности нуклеиновой кислоты в 3'-нетранслируемую область гена и измерения того, обеспечивает ли указанное включение повышение количества синтезируемого белка.
Вышеприведенное применимо соответственно к случаю, когда в соответствии с настоящим изобретением нуклеиновая кислота содержит две или более 3'-нетранслируемые области, которые предпочтительно соединены последовательно, с линкером между ними или без него, предпочтительно с взаиморасположением по принципу «голова к хвосту» (т. е. 3'-нетранслируемые области имеют одинаковую ориентацию, предпочтительно ориентацию, встречающуюся в нуклеиновой кислоте в природе).
В соответствии с настоящим изобретением термин «ген» относится к определенной последовательности нуклеиновой кислоты, которая ответственна за продукцию одного или нескольких продуктов в клетке и/или за обеспечение одной или нескольких межклеточных или внутриклеточных функций. Более конкретно, указанный термин относится к отрезку ДНК, который предусматривает нуклеиновую кислоту, кодирующую конкретный белок или функциональную или структурную молекулу РНК.
Полиаденилирование представляет собой добавление поли(A)-последовательности или хвоста к первичному РНК-транскрипту. Поли(A)-последовательность состоит из множества аденозинмонофосфатов. Другими словами, это отрезок РНК, который содержит только адениновые основания. У эукариот полиаденилирование является частью процесса, в результате которого образуется зрелая матричная РНК (мРНК) для трансляция. Следовательно, оно образует часть более крупного процесса экспрессии генов. Процесс полиаденилирования начинается по завершению или терминации транскрипции гена. Наиболее близкий к 3'-концу отрезок новообразованной пре-мРНК сперва отщепляется под действием набора белков; эти белки затем синтезируют поли(A)-последовательность на 3'-конце РНК. Поли(A)-последовательность является важной для ядерного экспорта, трансляции и стабильности мРНК. Последовательность укорачивается с течением времени, и, когда она является достаточно короткой, мРНК разрушается под действием ферментов.
Термины «полиадениловая последовательность», «поли(A)-последовательность» или «поли(A)-хвост» относятся к последовательности из адениловых остатков, которая расположена, как правило, на 3'-конце молекулы РНК. Настоящее изобретение относится к такой последовательности, которая будет присоединена в ходе транскрипции РНК посредством ДНК-матрицы на основе повторяющихся тимидиловых остатков нити, комплементарной кодирующей нити, причем указанная последовательность в обычных условиях не закодирована в ДНК, а присоединяется к свободному 3'-концу РНК посредством независимой от матрицы РНК-полимеразы после транскрипции в ядре. В соответствии с настоящим изобретением согласно одному варианту осуществления поли(A)-последовательность содержит по меньшей мере 20, предпочтительно по меньшей мере 40, предпочтительно по меньшей мере 80, предпочтительно по меньшей мере 100 и предпочтительно до 500, предпочтительно до 400, предпочтительно до 300, предпочтительно до 200 и, в частности, до 150 нуклеотидов A, предпочтительно последовательных нуклеотидов A, и, в частности, приблизительно 120 нуклеотидов A. Термин «нуклеотиды A» или «A» относится к адениловым остаткам.
Согласно предпочтительному варианту осуществления молекула нуклеиновой кислоты в соответствии с настоящим изобретением представляет собой вектор. Термин «вектор» в настоящем документе применяют в его наиболее общем значении, и он предусматривает любые промежуточные средства для доставки нуклеиновой кислоты, которые, например, обеспечивают возможность введения указанной нуклеиновой кислоты в прокариотические и/или эукариотические клетки-хозяева и, при необходимости, интеграции в геном. Репликация и/или экспрессия таких векторов предпочтительно происходит в клетке. Векторы предусматривают плазмиды, фагмиды или вирусные геномы. Применяемый в настоящем документе термин «плазмида» обычно относится к конструкции из внехромосомного генетического материала, обычно кольцевому ДНК-дуплексу, который может реплицироваться независимо от хромосомной ДНК.
Описанные в настоящем документе нуклеиновые кислоты могут представлять собой рекомбинантные и/или выделенные молекулы.
Подразумевается, что в контексте настоящего документа «выделенная молекула» относится к молекуле, которая практически не содержит других молекул, таких как другой клеточный материал. Термин «выделенная нуклеиновая кислота» в соответствии с настоящим изобретением означает, что нуклеиновая кислота была (i) амплифицирована in vitro, например, с помощью полимеразной цепной реакции (ПЦР), (ii) получена рекомбинантным путем с помощью клонирования, (iii) очищена, например, с помощью расщепления и электрофоретического разделения в геле или (iv) синтезирована, например, с помощью химического синтеза. Выделенная нуклеиновая кислота представляет собой нуклеиновую кислоту, доступную для осуществления манипуляций с применением методик рекомбинантных ДНК.
Термин «рекомбинантный» в контексте настоящего изобретения означает «созданный с помощью генной инженерии». Предпочтительно «рекомбинантный объект», такой как рекомбинантная клетка, в контексте настоящего изобретения не встречается в природе.
Применяемый в настоящем документе термин «встречающийся в природе» относится к тому факту, что объект может быть найден в природе. Например, пептид или нуклеиновая кислота, которые находятся в организме (в том числе вирусах) и могут быть выделены из источника в природе, и которые не были намеренно модифицированы человеком в лаборатории, являются встречающимися в природе.
В соответствии с настоящим изобретением термин «клетка-хозяин» относится к любой клетке, которую можно трансформировать или трансфицировать экзогенной нуклеиновой кислотой. Термин «клетка-хозяин» в соответствии с настоящим изобретением предусматривает прокариотические (например, E.coli) или эукариотические клетки (например, клетки дрожжей и клетки насекомых). Особое предпочтение отдается клеткам млекопитающих, таким как клетки людей, мышей, хомяков, свиней, коз, приматов. Клетки могут быть получены из множества типов тканей и предусматривать первичные клетки и клеточные линии. К конкретным примерам относятся кератиноциты, лейкоциты периферической крови, стволовые клетки костного мозга и эмбриональные стволовые клетки. Согласно другим вариантам осуществления клетка-хозяин представляет собой антиген-представляющую клетку, в частности дендритную клетку, моноцит или макрофаг. Нуклеиновая кислота может находиться в клетке-хозяине в одной или в нескольких копиях и, согласно одному варианту осуществления, экспрессируется в клетке-хозяине.
E.coli представляет собой грамотрицательную факультативно анаэробную палочковидную бактерию рода Escherichia, которая обычно встречается в нижнем отделе кишечника теплокровных организмов. Бактерию можно легко и недорого выращивать в лабораторных условиях, и ее активно исследовали в течение более 60 лет. E.coli является наиболее широко изученным прокариотическим модельным организмом и важным видом в области биотехнологии и микробиологии, в которых он служит в качестве организма-хозяина для большей части работы с рекомбинантной ДНК. Штаммы E.coli в соответствии с настоящим изобретением включают: AG1, AB1157, B2155, BL21, BNN93, BNN97, BW26434, C600, CSH50, D1210, DB3.1, DH1, DH5α, DH10B, DH12S, DM1, E. cloni(r), E.coli K12 ER2738, ER2566, ER2267, HB101, IJ1126, IJ1127, JM83, JM101, JM103, JM105, JM106, JM107, JM108, JM109, JM110, JM2.300, LE392, Mach1, MC1061, MC4100, MFDpir, MG1655, OmniMAX2, RR1, RV308, SOLR, SS320, STBL2, STBL3, STBL4, SURE, SURE2, TG1, TOP10, Top10F', W3110, WM3064, XL1-Blue, XL2-Blue, XL1-Red и XL10-Gold.
В соответствии с настоящим изобретением термин «пептид» предусматривает олиго- и полипептиды и относится к веществам, которые содержат две или более, предпочтительно 3 или более, предпочтительно 4 или более, предпочтительно 6 или более, предпочтительно 8 или более, предпочтительно 10 или более, предпочтительно 13 или более, предпочтительно 16 или более, предпочтительно 20 или более и до предпочтительно 50, предпочтительно 100 или предпочтительно 150 последовательных аминокислот, связанных друг с другом с помощью пептидных связей. Термин «белок» относится к крупным пептидам, предпочтительно пептидам, содержащим по меньшей мере 151 аминокислоту, но при этом термины «пептид» и «белок» в настоящем документе обычно применяют как синонимы.
Термины «пептид» и «белок» в соответствии с настоящим изобретением предусматривают вещества, которые содержат не только аминокислотные компоненты, но также компоненты, отличные от аминокислот, такие как сахара и структуры на основе фосфата, а также предусматривают вещества, содержащие такие связи, как сложноэфирные, тиоэфирные или дисульфидные связи.
В соответствии с настоящим изобретением нуклеиновая кислота, такая как РНК, может кодировать пептид или белок. Соответственно, транскрибируемая последовательность нуклеиновой кислоты или ее транскрипт могут содержать открытую рамку считывания (ORF), кодирующую пептид или белок. С указанной нуклеиновой кислоты могут экспрессироваться кодируемые пептид или белок. Например, указанная нуклеиновая кислота может представлять собой нуклеиновую кислоту, с которой экспрессируются, и которая кодирует антиген или фармацевтически активные пептид или белок, как, например, иммунологически активное соединение (которое предпочтительно не является антигеном).
В соответствии с настоящим изобретением выражение «нуклеиновая кислота, кодирующая пептид или белок» означает, что нуклеиновая кислота, если она находится в соответствующем окружении, предпочтительно в клетке, в ходе процесса трансляции может направлять сборку аминокислот с образованием пептида или белка. Предпочтительно РНК в соответствии с настоящим изобретением способна взаимодействовать с трансляционным аппаратом клетки с обеспечением трансляции пептида или белка.
В соответствии с настоящим изобретением согласно одному варианту осуществления РНК содержит фармацевтически активную РНК, или состоит из нее. «Фармацевтически активная РНК» может представлять собой РНК, которая кодирует фармацевтически активные пептид или белок.
«Фармацевтически активные пептид или белок» при введении субъекту в терапевтически эффективном количестве оказывают положительный или благоприятный эффект в отношении состояния или течения заболевания у субъекта. Предпочтительно фармацевтически активные пептид или белок обладают терапевтическими или паллиативными свойствами, и их можно вводить для смягчения, облегчения, ослабления, содействия устранению, задержки проявления или уменьшения тяжести одного или нескольких симптомов заболевания или нарушения. Фармацевтически активные пептид или белок могут обладать профилактическими свойствами, и их можно применять для задержки проявления заболевания или для уменьшения тяжести такого заболевания или патологического состояния. Термин «фармацевтически активные пептид или белок» включает целые белки или полипептиды, и также может относиться к их фармацевтически активным фрагментам. Он также может включать фармацевтически активные аналоги пептида или белка. Термин «фармацевтически активные пептид или белок» включает пептиды и белки, которые представляют собой антигены, т. е. пептид или белок вызывают у субъекта иммунный ответ, который может быть терапевтическим или частично или полностью защитным.
К примерам фармацевтически активных белков относятся без ограничения цитокины и белки иммунной системы, как, например, иммунологически активные соединения (например, интерлейкины, колониестимулирующий фактор (CSF), колониестимулирующий фактор гранулоцитов (G-CSF), гранулоцитарно-макрофагальный колониестимулирующий фактор (GM-CSF), эритропоэтин, фактор некроза опухоли (TNF), интерфероны, интегрины, адрессины, селектины, хоминговые рецепторы, T-клеточные рецепторы, иммуноглобулины, растворимые антигены главного комплекса гистосовместимости, иммунологически активные антигены, такие как антигены бактериального, паразитарного или вирусного происхождения, аллергены, аутоантигены, антитела), гормоны (инсулин, тиреоидный гормон, катехоламины, гонадотропины, трофические гормоны, пролактин, окситоцин, дофамин, бычий соматотропин, лептины и т. д.), гормоны роста (например, гормон роста человека), факторы роста (например, эпидермальный фактор роста, фактор роста нервов, инсулиноподобный фактор роста и т. д.), рецепторы факторов роста, ферменты (тканевой активатор плазминогена, стрептокиназа, ферменты, участвующие в биосинтезе или распаде холестерина, стероидогенные ферменты, киназы, фосфодиэстеразы, метилазы, деметилазы, дегидрогеназы, целлюлазы, протеазы, липазы, фосфолипазы, ароматазы, цитохромы, аденилат- или гуанилатциклазы, нейрамидазы и т. д.), рецепторы (рецепторы стероидных гормонов, пептидные рецепторы), связывающие белки (белки, связывающие гормоны роста или факторы роста и т. д.), факторы транскрипции и трансляции, белки, подавляющие рост опухоли (например, белки, которые ингибируют ангиогенез), структурные белки (такие как коллаген, фиброин, фибриноген, эластин, тубулин, актин и миозин), белки крови (тромбин, сывороточный альбумин, фактор VII, фактор VIII, инсулин, фактор IX, фактор X, тканевой активатор плазминогена, белок C, фактор фон Виллебранда, антитромбин III, глюкоцереброзидаза, эритропоэтин, колониестимулирующий фактор гранулоцитов (GCSF) или модифицированный фактор VIII, антикоагулянты и т. д.
Согласно одному варианту осуществления фармацевтически активный белок в соответствии с настоящим изобретением представляет собой цитокин, который вовлечен в регулирование гомеостаза лимфоидной системы, предпочтительно цитокин, который вовлечен в развитие, примирование, размножение, дифференцировку и/или выживание T-клеток, и предпочтительно индуцирует или повышает указанное. Согласно одному варианту осуществления цитокин представляет собой интерлейкин. Согласно одному варианту осуществления фармацевтически активный белок в соответствии с настоящим изобретением представляет собой интерлейкин, выбранный из группы, состоящей из IL-2, IL-7, IL-12, IL-15 и IL-21.
Термин «иммунологически активное соединение» относится к любому соединению, изменяющему иммунный ответ, предпочтительно путем индуцирования и/или подавления созревания иммунных клеток, индуцирования и/или подавления биосинтеза цитокинов, и/или изменяющему гуморальный иммунный ответ путем стимулирования продукции антител B-клетками. Иммунологически активные соединения обладают сильной иммуностимулирующей активностью, в том числе без ограничения противовирусной и противоопухолевой активностью, и могут также осуществлять подавление в отношении других аспектов иммунного ответа, например, смещая иммунный ответ от TH2-иммунного ответа, что является применимым в лечении широкого диапазона TH2-опосредованных заболеваний. Иммунологически активные соединения могут быть пригодными в качестве адъювантов для вакцин.
Если в соответствии с настоящим изобретением требуется индукция или усиление иммунного ответа с применением описанной в настоящем документе РНК, иммунный ответ может быть вызван или усилен с помощью РНК. Например, белки или пептиды, кодируемые РНК или продуктами их процессирования, могут быть представлены белками главного комплекса гистосовместимости (MHC), экспрессируемыми на антиген-представляющих клетках. MHC-пептидный комплекс затем могут распознавать иммунные клетки, такие как T-клетки, что приводит к их активации.
Согласно одному варианту осуществления РНК, которая кодирует антиген, такой как ассоциированный с заболеванием антиген, вводят млекопитающему, в частности, если требуется лечение млекопитающего с заболеванием, предусматривающим антиген. РНК поглощается антиген-представляющими клетками млекопитающего (моноцитами, макрофагами, дендритными клетками или другими клетками). Образуется продукт трансляции антигена РНК, и продукт экспонируется на поверхности клеток для распознавания T-клетками. Согласно одному варианту осуществления антиген экспонируется на клеточной поверхности для распознавания сконструированными CAR-T-клетками, направленными на антиген. Согласно одному варианту осуществления антиген или образуемый в результате его необязательного процессирования продукт экспонируется на клеточной поверхности в контексте молекул MHC для распознавания T-клетками посредством их T-клеточного рецептора.
В качестве альтернативы, в настоящем изобретении предусмотрены варианты осуществления, где экспрессирующую антиген РНК вводят в антиген-представляющие клетки ex vivo, например, антиген-представляющие клетки, взятые от пациента, и антиген-представляющие клетки, необязательно клонально размноженные ex vivo, трансплантируют обратно тому же пациенту. Трансфицированные клетки можно повторно вводить пациенту с применением любых способов, известных в данной области, предпочтительно в стерильной форме путем внутривенного, внутриполостного, внутрибрюшинного или внутриопухолевого введения.
Способы по настоящему изобретению могут предусматривать антиген-представляющую клетку для экспрессирования РНК, кодирующей антиген. С этой целью способы по настоящему изобретению могут предусматривать введение РНК, кодирующей антигены, в антиген-представляющие клетки, такие как дендритные клетки. Для трансфекции антиген-представляющих клеток, таких как дендритные клетки, можно применять фармацевтическую композицию, содержащую РНК, кодирующую антиген. Средство для доставки, которое направляет РНК в дендритную или другую антиген-представляющую клетку, можно вводить пациенту, в результате чего происходит трансфекция in vivo.
В соответствии с настоящим изобретением предпочтительным является применение составов на основе РНК, кодирующей антиген, с помощью которых после системного введения РНК доставляется с более высокой избирательностью в антиген-представляющие клетки селезенки, такие как дендритные клетки (DC). Например, составы наночастиц на основе РНК с определенным размером частиц, где суммарный заряд частиц является отрицательным или приближается к нулю, например, электронейтральные или отрицательно заряженные липоплексы из РНК и липосом, например, липоплексы, содержащие DOTMA и DOPE или DOTMA и холестерин, после системного введения обеспечивают преимущественную экспрессию РНК в DC селезенки. Было определено, что в целевых клетках (селезенки) экспрессия была высокой, тогда как экспрессия в других органах была низкой.
В контексте настоящего документа термин «наночастица» относится к любой частице с диаметром, который делает частицу подходящей для системного, в частности, парентерального введения, в частности, нуклеиновых кислот, как правило, диаметром менее 1000 нанометров (нм). Согласно некоторым вариантам осуществления наночастица имеет диаметр менее 600 нм. Согласно некоторым вариантам осуществления наночастица имеет диаметр менее 400 нм.
В контексте настоящего документа термин «состав на основе наночастиц» или подобные термины относится к любому веществу, которое содержит по меньшей мере одну наночастицу. Согласно некоторым вариантам осуществления композиция на основе наночастиц представляет собой однородную совокупность наночастиц. Согласно некоторым вариантам осуществления композиции на основе наночастиц представляют собой дисперсии или эмульсии. Как правило, дисперсия или эмульсия образуются при объединении по меньшей мере двух несмешивающихся веществ.
Термин «липоплекс» или «липоплекс с нуклеиновой кислотой», в частности «липоплекс с РНК», относится к комплексу липидов и нуклеиновых кислот, в частности РНК. Липоплексы образуются спонтанно при смешивании катионных липосом, которые зачастую также содержат нейтральный «хелперный» липид, с нуклеиновыми кислотами.
Если в настоящем изобретении упоминается заряд, такой как положительный заряд, отрицательный заряд или нейтральный заряд или катионное соединение, отрицательно заряженное соединение или нейтрально заряженное соединение, то это обычно означает, что упоминаемый заряд имеет место при выбранном pH, таком как физиологический pH. Например, термин «катионный липид» означает липид с суммарным положительным зарядом при выбранном pH, таким как физиологический pH. Термин «нейтральный липид» означает липид, суммарный заряд которого не является положительным или отрицательным, и он может находиться в форме незаряженного или нейтрального амфотерного иона при выбранном pH, таком как физиологический pH. Под «физиологическим pH» в настоящем документе подразумевают pH, составляющий приблизительно 7,5.
Носители в виде наночастиц, такие как липидные носители, предназначенные для применения в настоящем изобретении, включают любые вещества или средства для доставки, с которыми может быть ассоциирована нуклеиновая кислота, такая как РНК, например, путем образования комплексов с нуклеиновой кислотой или образования везикул, в которые заключена или инкапсулирована нуклеиновая кислота. В результате этого может повышаться стабильность нуклеиновой кислоты по сравнению с «голой» нуклеиновой кислотой. В частности, может повышаться стабильность нуклеиновой кислоты в крови.
Катионные липиды, катионные полимеры и другие вещества с положительными зарядами могут образовывать комплексы с отрицательно заряженными нуклеиновыми кислотами. Такие катионные молекулы можно применять для связывания нуклеиновых кислот в комплекс, за счет чего образуются, например, так называемые липоплексы или полиплексы соответственно, и такие комплексы, как было показано, доставляют нуклеиновые кислоты в клетки.
Препараты наночастиц на основе нуклеиновых кислот для применение в настоящем изобретении можно получать с применением ряда протоколов и из любых соединений, образующих комплексы с нуклеиновыми кислотами. Комплексообразующими средствами, как правило, являются липиды, полимеры, олигомеры или амфифильные вещества. Согласно одному варианту осуществления комплексообразующее соединение предусматривает по меньшей мере одно средство, выбранное из группы, состоящей из протамина, полиэтиленимина, поли-L-лизина, поли-L-аргинина или гистона.
В соответствии с настоящим изобретением протамин является пригодным в качестве катионного средства-носителя. Термин «протамин» относится к любому из ряда богатых аргинином сильноосновных белков с относительно низкой молекулярной массой и встречающихся в ассоциированном, в частности, с ДНК состоянии в сперматозоидах ряда животных (таких как рыба), вместо соматических гистонов. В частности, термин «протамин» относится к встречающимся в молоках рыбы белкам, которые являются сильноосновными, растворяются в воде, не коагулируют при нагревании, и при их гидролизе главным образом образуется аргинин. В очищенной форме их применяют в составе на основе инсулина длительного действия и для нейтрализации антикоагулянтных эффектов гепарина.
В соответствии с настоящим изобретением подразумевается, что применяемый в настоящем документе термин «протамин» предусматривает любую аминокислотную последовательность протамина, полученную из нативных или биологических источников, или происходящую из них, в том числе ее фрагменты и мультимерные формы указанной аминокислотной последовательности или ее фрагмента. Кроме того, термин охватывает (синтезированные) полипептиды, которые являются искусственными и специально сконструированными для конкретных целей, и не могут быть выделены из нативных или биологических источников.
Применяемый в соответствии с настоящим изобретением протамин может представлять собой сульфатированный протамин или гидрохлорид протамина. Согласно предпочтительному варианту осуществления источник протамина, применяемого для получения описанных в настоящем документе наночастиц, представляет собой протамин 5000, который предусматривает протамин в концентрации более 10 мг/мл (5000 гепарин-нейтрализирующих единиц на мл) в изотоническом солевом растворе.
Липосомы представляют собой микроскопические липидные везикулы, часто имеющие один или несколько бислоев из образующего везикулу липида, такого как фосфолипид, и способны инкапсулировать лекарственное средство. В контексте настоящего изобретения можно использовать различные типы липосом, в том числе без ограничения многослойные везикулы (MLV), малые однослойные везикулы (SUV), крупные однослойные везикулы (LUV), стерически стабилизированные липосомы (SSL), мультивезикулярные тела (MV) и крупные мультивезикулярные тела (LMV), а также другие бислойные формы, известные в данной области. Размер и количество слоев липосомы будет зависеть от способа получения, а выбор типа подлежащих применению везикул будет зависеть от предпочтительного способа введения. Существует несколько других форм надмолекулярной организации, в виде которых липиды могут находиться в водной среде, предусматривающие ламеллярные фазы, гексагональные фазы и инверсные гексагональные фазы, кубические фазы, мицеллы, инвертированные мицеллярные фазы, состоящие из монослоев. Такие фазы также могут быть получены в комбинации с ДНК или РНК, и взаимодействие с РНК и ДНК может существенно влиять на фазовое состояние. Описанные фазы могут иметь место в составах наночастиц на основе нуклеиновых кислот по настоящему изобретению.
Для образования липоплексов с нуклеиновой кислотой из нуклеиновой кислоты и липосом можно применять любой подходящий способ образования липосом, если он обеспечивает получение ожидаемых липоплексов с нуклеиновой кислотой. Липосомы могут быть образованы с применением стандартных способов, таких как способ выпаривания в обращенной фазе (REV), способ инжекции этанола, способ дегидратации-регидратации (DRV), обработка ультразвуком или другие подходящие способы.
После образования липосом липосомы можно разделять по размеру с получением популяции липосом, характеризующейся в значительной степени гомогенным диапазоном частиц.
Образующие бислой липиды, как правило, содержат две углеводородные цепи, в частности, ацильные цепи, и концевую группу, либо полярную, либо неполярную. Образующие бислой липиды состоят либо из встречающихся в природе липидов, либо из липидов синтетического происхождения, в том числе фосфолипидов, таких как фосфатидилхолин, фосфатидилэтаноламин, фосфатидная кислота, фосфатидилинозитол и сфингомиелин, у которых длина углеводородных цепей, как правило, составляет приблизительно 14-22 атомов углерода, и они характеризуются отличающимися степенями ненасыщенности. Другие подходящие липиды для применения в композиции по настоящему изобретению включают гликолипиды и стерины, такие как холестерин, и их различные аналоги, которые также можно применять в липосомах.
Катионные липиды, как правило, содержат липофильный фрагмент, такой как стерин, ацильную или диацильную цепь, и имеют суммарный положительный заряд. Концевая группа липида, как правило, несет положительный заряд. Катионный липид предпочтительно имеет положительный заряд, соответствующий 1-10 валентностям, более предпочтительно положительный заряд, соответствующий 1-3 валентностям и более предпочтительно положительный заряд, соответствующий 1 валентности. К примерам катионных липидов относятся без ограничения 1,2-ди-O-октадеценил-3-триметиламмоний-пропан (DOTMA); диметилдиоктадециламмоний (DDAB); 1,2-диолеоил-3-триметиламмоний-пропан (DOTAP); 1,2-диолеоил-3-диметиламмоний-пропан (DODAP); 1,2-диацилокси-3-диметиламмоний-пропан; 1,2-диалкилокси-3-диметиламмоний-пропан; диоктадецилдиметиламмония хлорид (DODAC), 1,2-димиристоилоксипропил-1,3-диметилгидроксиэтиламмоний (DMRIE) и 2,3-диолеоилокси-N-[2(спермин-карбоксамид)этил]-N,N-диметил-1-пропанаминия трифторацетат (DOSPA). Предпочтительными являются DOTMA, DOTAP, DODAC и DOSPA. Наиболее предпочтительным является DOTMA.
Кроме того, описанные в настоящем документенаночастицы предпочтительно дополнительно содержат нейтральный липид с учетом стабильности структуры и т. д. Нейтральный липид может быть соответствующим образом выбран с учетом эффективности доставки комплекса нуклеиновая кислота-липид. К примерам нейтральных липидов относятся без ограничения 1,2-ди-(9Z-октадеценоил)-sn-глицеро-3-фосфоэтаноламин (DOPE), 1,2-диолеоил-sn-глицеро-3-фосфохолин (DOPC), диацилфосфатидилхолин, диацилфосфатидилэтаноламин, церамид, сфингомиелин, цефалин, стерин и цереброзид. Предпочтительными являются DOPE и/или DOPC. Наиболее предпочтительным является DOPE. В случае, когда катионная липосома содержит как катионный липид, так и нейтральный липид, молярное соотношение катионного липида и нейтрального липида может быть определено соответствующим образом с учетом стабильности липосомы и т. д.
В соответствии с одним вариантом осуществления описанные в настоящем документе наночастицы могут содержать фосфолипиды. Фосфолипиды могут представлять собой глицерофосфолипид. К примерам глицерофосфолипида относятся без ограничения три типа липидов: (i) цвиттерионные фосфолипиды, которые включают, например, фосфатидилхолин (PC), фосфатидилхолин яичного желтка, происходящий из соевых бобов PC в естественной, частично гидрогенизированной или полностью гидрогенизированной форме, димиристоилфосфатидилхолин (DMPC), сфингомиелин (SM); (ii) отрицательно заряженные фосфолипиды, которые включают, например, фосфатидилсерин (PS), фосфатидилинозитол (PI), фосфатидную кислоту (PA), фосфатидилглицерин (PG), дипальмитоил-PG, димиристоил-фосфатидилглицерин (DMPG); синтетические производные, в которых конъюгат содержит цвиттерионный фосфолипид с отрицательным зарядом, как в случае метоксиполиэтиленгликоль-дистеароилфосфатидилэтаноламина (mPEG-DSPE); и (iii) катионные фосфолипиды, которые включают, например, фосфатидилхолин или сфингомиелин, фосфомоноэфир которых был O-метилирован с образованием катионных липидов.
Ассоциация нуклеиновой кислоты с липидным носителем может происходить, например, путем заполнения нуклеиновой кислотой пустых пространств носителя так, что носитель физически захватывает нуклеиновую кислоту, или путем ковалентного, ионного или водородного связывания, или посредством адсорбции за счет неспецифических связей. Независимо от способа ассоциации, нуклеиновая кислота должна сохранять свои терапевтические, т. е. антиген-кодирующие свойства.
Термин «заболевание» относится к аномальному состоянию, которое негативно воздействует на организм индивидуума. Под заболеванием зачастую подразумевают медицинское состояние, ассоциированное со специфичными симптомами и признаками. Заболевание может быть вызвано факторами, происходящими от внешнего источника, как, например, инфекционное заболевание, или оно может быть вызвано внутренними функциональными нарушениями, как, например, аутоиммунные заболевания.
В соответствии с настоящим изобретением термин «заболевание» также относится к заболеваниям, связанным со злокачественной опухолью. Термины «заболевание, связанное со злокачественной опухолью» или «злокачественная опухоль» (медицинский термин: злокачественное новообразование) относятся к классу заболеваний, при которых группа клеток характеризуется неконтролируемым ростом (деление за пределами нормы), инвазией (проникновение в соседние ткани и их разрушение) и в некоторых случаях метастазированием (распространение в другие части организма через лимфу или кровь). Эти три свойства злокачественности, характерные для злокачественных опухолей, отличают их от доброкачественных опухолей, которые самоограничиваются и не проникают или не метастазируют. При большинстве онкологических заболеваний образуется опухоль, т. е. увеличение или поражение, образуемое при аномальном росте клеток (называемых неопластическими клетками или опухолевыми клетками), однако в некоторых случаях, как лейкоз, не образуется. К примерам злокачественных опухолей относятся без ограничения карцинома, лимфома, бластома, саркома, глиома и лейкоз. Более конкретно, к примерам таких злокачественных опухолей относятся злокачественная опухоль костей, гемобластоз, злокачественная опухоль легкого, злокачественная опухоль печени, злокачественная опухоль поджелудочной железы, злокачественная опухоль кожи, злокачественная опухоль головы или шеи, злокачественная интраокулярная меланома или злокачественная меланома кожи, злокачественная опухоль матки, злокачественная опухоль яичника, злокачественная опухоль прямой кишки, злокачественная опухоль анальной области, злокачественная опухоль желудка, злокачественная опухоль толстой кишки, злокачественная опухоль молочной железы, злокачественная опухоль предстательной железы, злокачественная опухоль матки, карцинома половых и репродуктивных органов, болезнь Ходжкина, злокачественная опухоль пищевода, злокачественная опухоль тонкого кишечника, злокачественная опухоль эндокринной системы, злокачественная опухоль щитовидной железы, злокачественная опухоль паращитовидной железы, злокачественная опухоль надпочечника, саркома мягкой ткани, злокачественная опухоль мочевого пузыря, злокачественная опухоль почки, почечно-клеточная карцинома, карцинома почечной лоханки, неоплазмы центральной нервной системы (ЦНС), нейроэктодермальная злокачественная опухоль, опухоли позвоночника, глиома, менингиома и аденома гипофиза. Термин «злокачественная опухоль» в соответствии с настоящим изобретением также предусматривает метастазы злокачественной опухоли.
Термин «инфекционное заболевание» относится к любому заболеванию, которое может передаваться от индивидуума к индивидууму или от организма к организму, и вызвано микробным агентом (например, вирусная инфекция верхних дыхательных путей). К примерам инфекционных заболеваний относятся вирусные инфекционные заболевания, такие как СПИД (ВИЧ), гепатит A, B или C, герпес, опоясывающий лишай (ветряная оспа), краснуха (rubella virus), желтая лихорадка, лихорадка денге и т. д, инфекции, вызванные флавивирусами, вирусами гриппа, геморрагические инфекционные заболевания (вирусы Марбург или Эбола) и тяжелый острый респираторный синдром (SARS), бактериальные инфекционные заболевания, такие как болезнь легионеров (Legionella), заболевания, передающиеся половым путем (например, chlamydia или gonorrhea), язва желудка (Helicobacter), холера (Vibrio), туберкулез, дифтерия, инфекции, вызванные E.coli, Staphylococci, Salmonella или Streptococci (столбняк); инфекции, вызванные патогенными простейшими, такие как малярия, сонная болезнь, лейшманиоз; токсоплазмоз, т. е. инфекции, вызванные представителями родов Plasmodium, Trypanosoma, Leishmania и Toxoplasma; или грибковые инфекции, вызванные, например, Cryptococcus neoformans, Histoplasma capsulatum, Coccidioides immitis, Blastomyces dermatitidis или Candida albicans.
Термин «аутоиммунное заболевание» относится к любому заболеванию, при котором в организме вырабатывается иммунный (т. е. в рамках иммунной системы) ответ на некоторые составляющие его же ткани. Другими словами, иммунная система теряет свою способность распознавать некоторые ткани или системы организма как свои, и нацеливается на них и атакует, как если бы они были чужеродными. Аутоиммунные заболевания можно классифицировать на такие, при которых поражается преимущественно один орган (например, гемолитическая анемия и аутоиммунный тиреоидит), и такие, при которых процесс аутоиммунного заболевания распространяется в другие многие ткани (например, системная красная волчанка). Например, полагают, что причина рассеянного склероза состоит в том, что T-клетки атакуют оболочки, которые окружают нервные волокна головного мозга и спинного мозга. Это приводит к потере координации, слабости и размытому зрению. Аутоиммунные заболевания известны в данной области и включают, к примеру, тиреоидит Хашимото, диффузный токсический зоб, волчанку, рассеянный склероз, ревматоидный артрит, гемолитическую анемию, аутоиммунный тиреоидит, системную красную волчанку, целиакию, болезнь Крона, колит, диабет, склеродермию, псориаз и т. д.
В соответствии с настоящим изобретением иммунный ответ можно стимулировать введением субъекту подходящей мРНК, которая кодирует антиген или его фрагмент, например, ассоциированный с заболеванием антиген.
Термин «антиген» относится к веществу, содержащему эпитоп, в отношении которого будет вырабатываться иммунный ответ. Термин «антиген» включает, в частности, белки, пептиды, полисахариды, нуклеиновые кислоты, в частности РНК и ДНК, и нуклеотиды. Термин «антиген» также включает вещества, которые становятся антигенными - и сенсибилизирующими - только посредством преобразования (например, внутримолекулярного преобразования или при объединении с белком организма). Антиген предпочтительно может быть представлен клетками иммунной системы, такими как антиген-представляющие клетки, например, дендритные клетки или макрофаги. Кроме того, антиген или продукт его процессирования предпочтительно может быть распознан T- или B-клеточным рецептором или молекулой иммуноглобулина, такой как антитело. Согласно предпочтительному варианту осуществления антиген представляет собой ассоциированный с заболеванием антиген, такой как опухоль-ассоциированный антиген, вирусный антиген или бактериальный антиген.
Термин «ассоциированный с заболеванием антиген», применяемый в его наиболее широком смысле, относится к антигену, ассоциированному с заболеванием. Ассоциированный с заболеванием антиген представляет собой молекулу, которая содержит эпитопы, которые будут стимулировать иммунную систему хозяина для выработки клеточного антигенспецифичного иммунного ответа и/или гуморального ответа с участием антител в отношении заболевания. Ассоциированный с заболеванием антиген, следовательно, можно применять в терапевтических целях. Ассоциированные с заболеванием антигены предпочтительно ассоциированы с инфекцией, вызванной микроорганизмами, как правило, антигены микроорганизмов, или ассоциированы со злокачественной опухолью, как правило, опухолевые антигены.
Термин «заболевание, предусматривающее антиген» относится к любому заболеванию, в которое вовлечен антиген, например, заболевание, которое характеризуется наличием и/или экспрессией антигена. Заболевание, предусматривающее антиген, может представлять собой инфекционное заболевание, аутоиммунное заболевание или заболевание, связанное со злокачественной опухолью, или просто злокачественную опухоль. Как уже упоминалось выше, антиген может представлять собой ассоциированный с заболеванием антиген, такой как опухоль-ассоциированный антиген, вирусный антиген или бактериальный антиген.
Согласно одному варианту осуществления ассоциированный с заболеванием антиген представляет собой опухоль-ассоциированный антиген. Согласно такому варианту осуществления настоящее изобретение может быть пригодным для лечение злокачественной опухоли или метастаза при злокачественной опухоли. Предпочтительно пораженный заболеванием орган или ткань характеризуются пораженными заболеваем клетками, такими как клетки злокачественной опухоли, экспрессирующие ассоциированный с заболеванием антиген, и/или характеризуются тем, что ассоциированный с заболеванием антиген ассоциирован с их поверхностью. Иммунизация интактными или практически интактными опухоль-ассоциированными антигенами или их фрагментами, такими как пептиды MHC класса I и класса II или нуклеиновые кислоты, в частности мРНК, кодирующие такой антиген или фрагмент, обеспечивает возможность вызова ответа II типа, опосредованного MHC класса I и/или класса II и, таким образом, стимуляции T-клеток, таких как CD8+ цитотоксические Т-лимфоциты, способные осуществлять лизис клеток злокачественной опухоли, и/или CD4+ T-клетки. С помощью такой иммунизации можно также вызвать гуморальный иммунный ответ (B-клеточный ответ), что приводит к выработке антител к опухоль-ассоциированному антигену. Кроме того, антиген-представляющие клетки (APC), такие как дендритные клетки (DC), можно нагружать пептидами, представляемыми MHC класса I, путем трансфекции кодирующими опухолевые антигены нуклеиновыми кислотами in vitro, и вводить пациенту. Согласно одному варианту осуществления термин «опухоль-ассоциированный антиген» относится к составляющей клетки злокачественной опухоли, которая может происходить из цитоплазмы, клеточной поверхности и клеточного ядра. В частности, она относится к таким антигенам, которые продуцируются, предпочтительно в большом количестве, внутриклеточно или в виде поверхностных антигенов на опухолевой клетке. К примерам опухолевых антигенов относятся без ограничения HER2, EGFR, VEGF, CAMPATH1-антиген, CD22, CA-125, HLA-DR, антигены, связанные с Лимфомой Ходжкина, или муцин-1.
В соответствии с настоящим изобретением опухоль-ассоциированный антиген предпочтительно предусматривает любой антиген, который является характерным для опухолей или злокачественных опухолей, а также для опухолевых клеток или клеток злокачественной опухоли, в отношении типа и/или уровня экспрессии. Согласно одному варианту осуществления термин «опухоль-ассоциированный антиген» относится к белкам, которые в нормальных условиях, т. е. у здорового субъекта, специфично экспрессируются в ограниченном числе органов и/или тканей или на определенных стадиях развития, например, опухоль-ассоциированный антиген в нормальных условиях может специфично экспрессироваться в ткани желудка, предпочтительно в слизистой оболочке желудка, в репродуктивных органах, например, в яичке, ткани трофобласта, например, в плаценте, или в клетках зародышевой линии, и которые экспрессируются или аномально экспрессируются в одной или нескольких тканях опухоли или злокачественной опухоли. В таком контексте «ограниченное число» предпочтительно означает не более 3, более предпочтительно не более 2 или 1. Опухоль-ассоциированные антигены в контексте настоящего изобретения включают, например, дифференцировочные антигены, предпочтительно дифференцировочные антигены, специфичные для типа клеток, т. е. белки, которые в нормальных условиях специфично экспрессируются в клетках определенного типа на определенной стадии дифференцировки, антигены злокачественной опухоли/яичка, т. е. белки, которые в нормальных условиях специфично экспрессируются в яичке и в некоторых случаях в плаценте, и специфичные для зародышевой линии антигены. В контексте настоящего изобретения опухоль-ассоциированный антиген предпочтительно не экспрессируется или экспрессируется только иногда в здоровых тканях, или является мутированным в опухолевых клетках. Предпочтительно опухоль-ассоциированный антиген или аномальная экспрессия опухоль-ассоциированного антигена определяют клетки злокачественной опухоли. В контексте настоящего изобретения опухоль-ассоциированный антиген, который экспрессируется клеткой злокачественной опухоли у субъекта, например, пациента, страдающего от заболевания, связанного со злокачественной опухолью, предпочтительно является собственным белком организма указанного субъекта. Согласно предпочтительным вариантам осуществления опухоль-ассоциированный антиген в контексте настоящего изобретения в нормальных условиях специфично экспрессируется в ткани или органе, которые не являются критически важными, т. е. тканях или органах, которые при повреждении с участием иммунной системы не приводят к смерти субъекта, или в органах или структурах организма, которые являются недоступными или труднодоступными для иммунной системы. Предпочтительно опухоль-ассоциированный антиген представляется в контексте молекул MHC клеткой злокачественной опухоли, в которой он экспрессируется.
К примерам дифференцировочных антигенов, которые идеально соответствуют критериям опухоль-ассоциированных антигенов, предполагаемых настоящим изобретением в качестве целевых структур для иммунотерапии опухолей, в частности, для вакцинации против опухоли, относятся белки клеточной поверхности семейства клаудинов, такие как CLDN6 и CLDN18.2. Такие дифференцировочные антигены экспрессируются в опухолях различного происхождения, и особенно подходят в качестве целевых структур применительно к опосредованной антителами иммунотерапии злокачественной опухоли ввиду их избирательной экспрессии (не экспрессируются в важной с точки зрения токсичности здоровой ткани) и локализации на плазматической мембране.
Дополнительными примерами антигенов, которые могут быть пригодны согласно настоящему изобретению, являются p53, ART-4, BAGE, бета-катенин/m, Bcr-abL CAMEL, CAP-1, CASP-8, CDC27/m, CDK4/m, CEA, КЛАУДИН-12, c-MYC, CT, Cyp-B, DAM, ELF2M, ETV6-AML1, G250, GAGE, GnT-V, Gap100, HAGE, HER-2/neu, HPV-E7, HPV-E6, HAST-2, hTERT (или hTRT), LAGE, LDLR/FUT, MAGE-A, предпочтительно MAGE-A1, MAGE-A2, MAGE-A3, MAGE-A4, MAGE-A5, MAGE-A6, MAGE-A7, MAGE-A8, MAGE-A9, MAGE-A10, MAGE-A11 или MAGE-A12, MAGE-B, MAGE-C, MART-1/Melan-A, MC1R, миозин/m, MUC1, MUM-1, -2, -3, NA88-A, NF1, NY-ESO-1, NY-BR-1, p190 минорный BCR-abL, Pm1/RARa, PRAME, протеиназа 3, PSA, PSM, RAGE, RU1 или RU2, SAGE, SART-1 или SART-3, SCGB3A2, SCP1, SCP2, SCP3, SSX, СУРВИВИН, TEL/AML1, TPI/m, TRP-1, TRP-2, TRP-2/INT2, TPTE и WT, предпочтительно WT-1.
Термин «вирусный антиген» относится к любому вирусному компоненту с антигенными свойствами, т. е. способному вызывать иммунный ответ у индивидуума. Вирусный антиген может представлять собой вирусный рибонуклеопротеин или белок оболочки.
Термин «бактериальный антиген» относится к любому бактериальному компоненту с антигенными свойствами, т. е. способному вызывать иммунный ответ у индивидуума. Бактериальный антиген может происходить из клеточной стенки или цитоплазматической мембраны бактерии.
«Процессирование антигена» относится к разрушению антигена с образованием продуктов процессирования, которые представляют собой фрагменты указанного антигена (например, разрушению белка с образованием пептидов) и ассоциации одного или нескольких из таких фрагментов (например, посредством связывания) с молекулами MHC для представления клетками, предпочтительно антиген-представляющими клетками, специфичным T-клеткам.
Применяемый в настоящем документе термин «иммунный ответ» относится к реакции иммунной системы, например, на иммуногенные организмы, такие как бактерии или вирусы, клетки или вещества. Термин «иммунный ответ» включает врожденный иммунный ответ и адаптивный иммунный ответ. Предпочтительно иммунный ответ относится к активации иммунных клеток, индукции биосинтеза цитокинов и/или выработке антител. Предпочтительно, чтобы иммунный ответ предусматривал стадии активации антиген-представляющих клеток, таких как дендритные клетки и/или макрофаги, представления антигена или его фрагмента указанными антиген-представляющими клетками и активации цитотоксических T-клеток вследствие такого представления.
Термин «лечить» или «лечение» относится к любому лечению, которое обеспечивает улучшение состояния здоровья и/или продление (увеличение) продолжительности жизни индивидуума. Указанное лечение может обеспечивать устранение заболевания у индивидуума, приостановку или замедление развития заболевания у индивидуума, ингибирование или замедление развития заболевания у индивидуума, снижение частоты проявления или тяжести симптомов у индивидуума и/или снижение вероятности повторного проявления у индивидуума, у которого на данный момент наблюдают заболевание или у которого ранее наблюдали заболевание.
В частности, термин «лечение заболевания» включает излечивание, сокращение продолжительности, смягчение, замедление или ингибирование прогрессирования или обострения заболевания или его симптомов.
Термин «иммунотерапия» относится к лечению, предпочтительно с вовлечением специфичной иммунной реакции и/или иммунной(иммунных) эффекторной(эффекторных) функции(функций).
С помощью термина «иммунизация» или «вакцинация» описывают процесс лечения субъекта по терапевтическим или профилактическим причинам.
Применяемый в настоящем документе термин «субъект» или «индивидуум» предпочтительно относится к млекопитающим. Например, млекопитающие в контексте настоящего изобретения представляют собой людей, приматов, отличных от людей, домашних животных, таких как собаки, кошки, овца, крупный рогатый скот, козы, свиньи, лошади и т. д., лабораторных животных, таких как мыши, крысы, кролики, морские свинки и т. д., а также животных в неволе, таких как животные зоопарков. Согласно предпочтительному варианту осуществления субъектом является человек.
Термин «антиген-представляющая клетка» (APC) относится к клетке из числа клеток, способных экспонировать, захватывать и/или представлять по меньшей мере один антиген или антигенный фрагмент на (или в пределах) их клеточной поверхности. Антиген-представляющие клетки можно дифференцировать на профессиональные антиген-представляющие клетки и непрофессиональные антиген-представляющие клетки.
Термин «профессиональные антиген-представляющие клетки» относится к антиген-представляющим клеткам, которые конститутивно экспрессируют молекулы главного комплекса гистосовместимости класса II (MHC класса II), необходимые для взаимодействия с наивными T-клетками. Если T-клетка взаимодействует с молекулярным комплексом MHC класса II на мембране антиген-представляющей клетке, антиген-представляющая клетка продуцирует костимулирующую молекулу, индуцирующую активацию T-клетки. Профессиональные антиген-представляющие клетки предусматривают дендритные клетки и макрофаги.
Термин «непрофессиональные антиген-представляющие клетки» относится к антиген-представляющим клеткам, которые экспрессируют молекулы MHC класса II не конститутивно, а при стимуляции определенными цитокинами, такими как интерферон гамма. В качестве примера, непрофессиональные антиген-представляющие клетки включают фибробласты, тимусные эпителиальные клетки, эпителиальные клетки щитовидной железы, глиальные клетки, бета-клетки поджелудочной железы или васкулярные эндотелиальные клетки.
Термин «главный комплекс гистосовместимости» и сокращение «MHC» включают молекулы MHC класса I и MHC класса II, и относятся к комплексу генов, которые встречаются у всех позвоночных. Белки или молекулы MHC являются важными для передачи сигналов между лимфоцитами и антиген-представляющими клетками или пораженными заболеванием клетками в рамках иммунных реакций, где белки или молекулы MHC связывают пептиды и представляют их для распознавания T-клеточными рецепторами. Белки, кодируемые MHC, экспрессируются на поверхности клеток, и они экспонируют как свои собственные антигены (пептидные фрагменты из самой клетки), так и чужие антигены (например, фрагменты инвазивных микроорганизмов), для T-клетки.
В соответствии с настоящим изобретением термин «химерный антигенный рецептор (CAR)» является синонимичным с терминами «химерный T-клеточный рецептор» и «искусственный T-клеточный рецептор».
Такие термины относятся к сконструированным рецепторам, которые придают иммунной эффекторной клетке, такой как T-клетка, определенную специфичность, такую как специфичность моноклонального антитела. Таким образом, для адоптивного переноса клеток можно получать большое число специфичных в отношении злокачественной опухоли T-клеток. Таким образом, CAR могут присутствовать на T-клетках, например, в дополнение к собственному T-клеточному рецептору T-клеток, или вместо него. Для таких T-клеток не является обязательным процессинг и представление антигена для распознавания целевой клетки, а скорее они могут распознавать предпочтительно с определенной специфичностью любой антиген, присутствующий на целевой клетке. Предпочтительно указанный CAR экспрессируется на поверхности клеток. Для целей настоящего изобретения T-клетки, содержащие CAR, охвачены применяемым в настоящем документе термином «T-клетка».
В соответствии с настоящим изобретением термин «CAR» (или «химерный антигенный рецептор») относится к искусственному рецептору, содержащему одну молекулу или комплекс молекул, которые распознают, т. е. связываются с целевой структурой (например, антигеном) на целевой клетке, такой как клетка злокачественной опухоли (например, путем связывания антигенсвязывающего домена с антигеном, экспрессируемым на поверхности целевой клетки), и который может придавать специфичность иммунной эффекторной клетке, такой как T-клетка, экспрессирующая на клеточной поверхности указанный CAR. Предпочтительно распознавание целевой структуры CAR приводит к активации иммунной эффекторной клетки, экспрессирующей указанный CAR. CAR может содержать один или несколько белковых компонентов, причем указанные белковые компоненты предусматривают один или несколько описанных в настоящем документе доменов. Термин «CAR» не включает T-клеточные рецепторы.
Согласно одному варианту осуществления одноцепочечный вариабельный фрагмент (scFv), происходящий из моноклонального антитела, сливают с трансмембранным и эндодоменом CD3-зета. Такие молекулы обеспечивают передачу сигнала через компонент зета в ответ на распознавание scFv его антигенной мишени на целевой клетке и уничтожение целевой клетки, которая экспрессирует целевой антиген. Домены распознавания антигена, которые также можно применять, включают, среди прочих, одиночные альфа- и бета-цепи T-клеточного рецептора (TCR). Фактически, в качестве домена распознавания антигена можно применять практически все объекты, которые с высокой аффинностью связывают данную мишень.
После распознавания антигена происходит кластеризация рецепторов и передача сигнала в клетку. В этом отношении «сигнальный домен T-клетки» представляет собой домен, предпочтительно эндодомен, который после связывания антигена передает сигнал активации T-клеткам. Наиболее широко применяемым компонентом эндодомена является CD3-зета.
Терапия с использованием адоптивного переноса клеток с применением сконструированных CAR-T-клеток, экспрессирующих химерные антигенные рецепторы, является перспективным видом терапии злокачественной опухоли, поскольку CAR-модифицированные T-клетки можно сконструировать для целенаправленного воздействия практически на любой опухолевый антиген. Например, T-клетки пациента можно сконструировать с помощью методов генной инженерии (генетически модифицированные) с возможностью экспрессии CAR, специфично направленных на антигены, присутствующие на опухолевых клетках пациента, а затем их вводят обратно пациенту.
В соответствии с настоящим изобретением CAR могут заменять функцию T-клеточного рецептора и, в частности, могут придавать клетке, такой как T-клетка, реактивность, такую как цитотоксическая активность. Однако в отличие от связывания T-клеточного рецептора с комплексом антигенный пептид-MHC, CAR может связываться с антигеном, в частности, когда он экспрессирован на поверхности клетки.
В соответствии с настоящим изобретением CAR, как правило, могут содержать три домена.
Первый домен представляет собой связывающий домен, который распознает и связывает антиген.
Второй домен представляет собой домен, вовлеченный в костимуляцию. Домен, вовлеченный в костимуляцию, служит для повышения степени пролиферации и выживания цитотоксических лимфоцитов после связывания CAR с целевым фрагментом. Особенность домена, вовлеченного в костимуляцию, ограничена только тем, что он имеет способность повышать степень пролиферации и выживания клеток после связывания CAR целевого фрагмента. Подходящие домены, вовлеченные в костимуляцию, включают CD28, CD137 (4-1BB), представителя семейства рецепторов фактора некроза опухоли (TNF), CD134 (OX40), представителя суперсемейства рецепторов TNFR и CD278 (ICOS), экспрессируемую на активированных T-клетках костимулирующую молекулу суперсемейства CD28. Специалисту в данной области будет понятно, что варианты последовательностей этих указанных доменов, вовлеченных в костимуляцию, можно применять без негативного влияния на настоящее изобретение, причем варианты обладают активностью, аналогичной или подобной таковой у домена, на основе которого они смоделированы. Такие варианты будут характеризоваться по меньшей мере приблизительно 80% идентичностью последовательностей с аминокислотной последовательностью домена, из которого они происходят. Согласно некоторым вариантам осуществления настоящего изобретения конструкции CAR содержат два домена, вовлеченные в костимуляцию. Хотя определенные комбинации включают все возможные варианты четырех указанных доменов, к конкретным примерам относятся CD28+CD137 (4-1BB) и CD28+CD134 (OX40).
Третий домен представляет собой домен передачи сигнала активации (или сигнальный домен T-клетки). Домен передачи сигнала активации служит для активации цитотоксических лимфоцитов после связывания CAR с антигеном. Особенность домена передачи сигнала активации ограничена только тем, что он имеет способность индуцировать активацию выбранного цитотоксического лимфоцита после связывания антигена CAR. Подходящие домены передачи сигнала активации включают CD3[зета]-цепь T-клетки и Fc-рецептор [гамма]. Специалисту в данной области будет понятно, что варианты последовательностей этих указанных доменов передачи сигнала активации можно применять без негативного влияния на настоящее изобретение, причем варианты обладают активностью, аналогичной или подобной таковой у домена, на основе которого они смоделированы. Такие варианты будут характеризоваться по меньшей мере приблизительно 80% идентичностью последовательностей с аминокислотной последовательностью домена, из которого они происходят.
CAR могут содержать три домена, вместе находящиеся в форме слитого белка. Такие слитые белки обычно будут содержать связывающий домен, один или несколько доменов, вовлеченных в костимуляцию, и домен передачи сигнала активации, связанные в направлении от N-конца к C-концу. Однако CAR не ограничиваются таким расположением, и другие варианты расположения являются допустимыми и предусматривают связывающий домен, домен передачи сигнала активации и один или несколько доменов, вовлеченных в костимуляцию. Будет понятно, что, поскольку связывающий домен должен быть свободным для возможности связывания антигена, размещение связывающего домена в слитом белке обычно будет таким, чтобы обеспечивалось экспонирование области на внешней поверхности клетки. Аналогичным образом, поскольку домен, вовлеченный в костимуляцию, и домен передачи сигнала активации служат для индукции активности и пролиферации цитотоксических лимфоцитов, в слитом белке эти два домена обычно будут экспонированы на внешней поверхности клетки. CAR могут включать дополнительные элементы, такие как сигнальный пептид для обеспечения надлежащего экспорта слитого белка на поверхность клеток, трансмембранный домен для обеспечения поддержания слитого белка как интегрального мембранного белка и шарнирный домен (или спейсерная область), который придает гибкость связывающему домену и обеспечивает сильное связывание с антигеном.
Клетки, применяемые с системой CAR по настоящему изобретению, предпочтительно представляют собой T-клетки, в частности цитотоксические лимфоциты, предпочтительно выбранные из цитотоксических T-клеток, естественных клеток-киллеров (NK) и лимфокин-активированных клеток-киллеров (LAK). После активации каждый из этих типов цитотоксических лимфоцитов запускает разрушение целевых клеток. Например, цитотоксические T-клетки запускают разрушение целевых клеток с помощью одного или обоих из следующих способов. Первый заключается в том, что после активации из T-клеток высвобождаются цитотоксины, такие как перфорин, гранзимы и гранулизин. Перфорин и гранулизин создают поры в целевой клетке, а гранзимы проникают в клетку и запускают каспазный каскад в цитоплазме, что индуцирует апоптоз (запрограммированную гибель клеток) клетки. Второй заключается в том, что апоптоз может быть индуцирован посредством взаимодействия Fas-лиганд Fas между T-клетками и целевыми клетками. Цитотоксические лимфоциты предпочтительно будут представлять собой аутологичные клетки, хотя можно применять и гетерологичные клетки или аллогенные клетки.
Для включения CAR-конструкций в T-клетки можно применять ряд способов, в том числе трансфекцию на основе ДНК, отличной от вирусной, системы на основе транспозонов и системы на основе вирусов. Трансфекция на основе ДНК, отличной от вирусной, характеризуется незначительным риском инсерционого мутагенеза. С помощью систем на основе транспозонов трансгены можно интегрировать более эффективно, чем с помощью плазмид, которые не содержат интегрирующий элемент. Системы на основе вирусов включают применение γ-ретровирусов и лентивирусных векторов. γ-Ретровирусы относительно просто получать, они эффективно и стабильно трансдуцируют T-клетки, и для них была предварительно доказана безопасность с точки зрения интеграции в первичные T-клетки человека. Лентивирусные векторы также эффективно и стабильно трансдуцируют T-клетки, но являются более дорогостоящими в производстве. Они также потенциально более безопасны, чем системы на основе ретровирусов.
Описанная в настоящем документе РНК (например, полученная с применением описанной в настоящем документе молекулы нуклеиновой кислоты в качестве матрицы для транскрипции) также является пригодной для осуществления перепрограммирования или дедифференцировки соматических клеток в клетки, подобные стволовым, т. е. клетки, обладающие характеристиками стволовых клеток, in vitro или in vivo. Такой случай может предусматривать транзиентную экспрессию факторов перепрограммирования in vitro или in vivo с целью инициации процессов перепрограммирования или дедифференцировки клеток. Таким образом, согласно одному варианту осуществления пептид или белок, кодируемые нуклеиновой кислотой, такой как описанная в настоящем документе РНК, представляет собой фактор, обеспечивающий перепрограммирование соматических клеток в клетки, обладающие характеристиками стволовых клеток. Клетки, подобные стволовым, можно получать в соответствии с настоящим изобретением без получения эмбрионов или зародышей. Дедифференцировку соматических клеток в клетки, обладающие характеристиками стволовых клеток, в частности плюрипотентностью, можно осуществлять путем введения РНК, кодирующей факторы, индуцирующие дедифференцировку соматических клеток, в соматические клетки (также называемые транскрипционными факторами перепрограммирования (rTF)), и культивирования соматических клеток, позволяя клеткам дедифференцироваться. После дедифференцировки клетки можно индуцировать в отношении повторной дифференцировки в тот же или в другой тип соматических клеток, таких как нервные, гемопоэтические, мышечные, эпителиальные и другие типы клеток. Таким образом, такие клетки, подобные стволовым, находят медицинское применение для лечения дегенеративных заболеваний с помощью «клеточной терапии», и могут быть использованы для новых терапевтических стратегий лечения нарушений со стороны сердечно-сосудистой, нервной, эндокринной, сосудистой систем, поражений сетчатки, дерматологических и скелетно-мышечных нарушений и других заболеваний.
Соответственно, настоящее изобретение также относится к способу получения клеток, обладающих характеристиками стволовых клеток, предусматривающему стадии (i) получения популяции клеток, содержащей соматические клетки, (ii) введения в соматические клетки РНК по настоящему изобретению, способной экспрессировать один или несколько факторов, обеспечивающих перепрограммирование соматических клеток в клетки, обладающие характеристиками стволовых клеток, и (iii) обеспечения развития клеток, обладающих характеристиками стволовых клеток. Согласно одному варианту осуществления способ дополнительно предусматривает введение в соматические клетки миРНК, усиливающей перепрограммирование соматических клеток в клетки, обладающие характеристиками стволовых клеток.
Согласно одному варианту осуществления один или несколько факторов предусматривают OCT4 и SOX2. Один или несколько факторов могут дополнительно предусматривать KLF4 и/или c-MYC и/или NANOG и/или LIN28. Согласно одному варианту осуществления один или несколько факторов предусматривают OCT4, SOX2, KLF4 и c-MYC, и могут дополнительно предусматривать LIN28 и необязательно NANOG. Согласно одному варианту осуществления один или несколько факторов предусматривают OCT4, SOX2, NANOG и LIN28.
Согласно одному варианту осуществления способ дополнительно предусматривает стадию культивирования соматических клеток в присутствии по меньшей мере одного ингибитора деацетилазы гистонов, где по меньшей мере один ингибитор деацетилазы гистонов предпочтительно предусматривает вальпроевую кислоту, маслянокислый натрий, трихостатин A и/или скриптаид.
Согласно одному варианту осуществления стадия (iii) предусматривает культивирование соматических клеток в условиях культивирования эмбриональных стволовых клеток.
Согласно одному варианту осуществления характеристики стволовых клеток предусматривают морфологию эмбриональных стволовых клеток.
Согласно одному варианту осуществления клетки, обладающие характеристиками стволовых клеток, имеют нормальный кариотип, характеризуются теломеразной активностью, экспрессируют маркеры клеточной поверхности, характерные для эмбриональных стволовых клеток, и/или экспрессируют гены, характерные для эмбриональных стволовых клеток.
Согласно одному варианту осуществления клетки, обладающие характеристиками стволовых клеток, характеризуются плюрипотентным статусом.
Согласно одному варианту осуществления клетки, обладающие характеристиками стволовых клеток, обладают дифференцировочным потенциалом к дифференцировке в развившиеся производные всех трех первичных зародышевых листков.
Согласно одному варианту осуществления соматические клетки представляют собой фибробласты, такие как фибробласты легких, фибробласты крайней плоти или фибробласты кожи. Предпочтительно соматические клетки представляют собой клетки человека.
Согласно одному варианту осуществления РНК вводят в соматические клетки путем электропорации или липофекции. Согласно одному варианту осуществления РНК вводят в соматические клетки повторно.
Согласно одному варианту осуществления введение в соматические клетки РНК, способной экспрессировать определенные раскрытые в настоящем документе факторы, приводит в результате к экспрессии указанных факторов в течение продолжительного периода времени, предпочтительно в течение по меньшей мере 10 дней, предпочтительно в течение по меньшей мере 11 дней и более предпочтительно в течение по меньшей мере 12 дней. Для обеспечения такой продолжительной экспрессии РНК предпочтительно периодически (т. е. повторно) вводят в клетки более одного раза, предпочтительно с применением электропорации. Предпочтительно для обеспечения экспрессии одного или несколько факторов в течение продолжительного периода времени РНК вводят в клетки по меньшей мере дважды, более предпочтительно по меньшей мере 3 раза, более предпочтительно по меньшей мере 4 раза, еще более предпочтительно по меньшей мере 5 раз и до предпочтительно 6 раз, более предпочтительно до 7 раз или даже до 8, 9 или 10 раз, предпочтительно в течение периода времени, составляющего по меньшей мере 10 дней, предпочтительно в течение по меньшей мере 11 дней и более предпочтительно в течение по меньшей мере 12 дней. Предпочтительно периоды времени, прошедшие между повторными введениями РНК, составляют от 24 часов до 120 часов, предпочтительно от 48 часов до 96 часов. Согласно одному варианту осуществления периоды времени, прошедшие между повторными введениями РНК, составляют не более 72 часов, предпочтительно не более 48 часов или 36 часов. Согласно одному варианту осуществления перед очередной электропорацией клеткам дают восстановиться после предыдущей электропорации. В любом случае, условия должны быть выбраны так, чтобы факторы были экспрессированы в клетках в количествах и в течение периодов времени, которые содействуют процессу перепрограммирования.
«Стволовая клетка» представляет собой клетку со способностью к самообновлению, оставаться в недифференцированном состоянии и проходить дифференцировку. Стволовая клетка может делиться без ограничения в течение по меньшей мере всей жизни животного, в организме которого она находится в естественных условиях. Стволовая клетка не является окончательно дифференцированной; она не пребывает на конечной стадии пути дифференцировки. Когда стволовая клетка делиться, каждая дочерняя клетка может либо остаться стволовой клеткой, либо пойти по пути, который ведет к терминальной дифференцировке.
Тотипотентные стволовые клетки представляют собой клетки с тотипотентными свойствами дифференцировки и способны развиться в целый организм. После оплодотворения яйцеклетки сперматозоидом таким свойством клетки обладают до достижения стадии 8 клеток. Если такие клетки выделить и пересадить в матку, они могут развиться в целый организм.
Плюрипотентные стволовые клетки представляют собой клетки, способные развиться в различные клетки и ткани, происходящие из эктодермального, мезодермального и эндодермального листков. Плюрипотентные стволовые клетки, которые происходят из внутренней клеточной массы, расположенные внутри бластоцист, образующихся на 4-5 дни после оплодотворения, называют «эмбриональными стволовыми клетками» и они могут дифференцироваться в клетки различных других тканей, но из них не могут образоваться новые живые организмы.
Мультипотентные стволовые клетки представляют собой стволовые клетки, дифференциирующиеся обычно только в типы клеток, специфичные для ткани и органа, из которого они происходят. Мультипотентные стволовые клетки вовлечены не только в рост и развитие различных тканей и органов во время фетального, неонатального периодов развития и взрослого периода жизни, но также в поддержание гомеостаза тканей взрослого организма и функцию индуцирования регенерации после повреждения тканей. Специфичные для ткани мультипотентные клетки вместе называют «взрослыми стволовыми клетками».
«Эмбриональная стволовая клетка» или «ESC» представляет собой стволовую клетку, которая присутствует у эмбриона или выделена из него. Она может быть плюрипотентной, обладающей способностью дифференцироваться в каждую без исключения клетку, присутствующую в организме, или мультипотентной, со способностью дифференцироваться в более чем один тип клеток.
В контексте настоящего документа «эмбрион» относится к животному на ранних стадиях его развития. Эти стадии характеризуются имплантацией и гаструляцией, при которых определяются и закладываются три зародышевых листка, и дифференцировкой зародышевых листков в соответствующие органы и системы органов. Тремя зародышевыми листками являются эндодерма, эктодерма и мезодерма.
«Бластоциста» представляет собой эмбрион на ранней стадии развития, на которой оплодотворенная яйцеклетка подвергается дроблению и образуется, или образован, сферический слой клеток, окружающий заполненную жидкостью полость. Этот сферический слой клеток представляет собой трофэктодерму. Внутри трофэктодермы находится кластер клеток, называемый внутренней клеточной массой (ICM). Трофэктодерм является предшественником плаценты, а ICM является предшественником эмбриона.
Взрослая стволовая клетка, также называемая соматической стволовой клеткой, представляет собой стволовую клетку, присутствующую во взрослом организме. Взрослая стволовая клетка присутствует в дифференцированной ткани, может самообновляться и может дифференцироваться, с некоторыми ограничениями, с образованием специализированных типов клеток ткани, из которой они происходят. К примерам относятся мезенхимальные стволовые клетки, гемопоэтические стволовые клетки и нейрональные стволовые клетки.
«Дифференцированная клетка» представляет собой зрелую клетку, которая подверглась прогрессирующим изменениям в рамках развития в более специализированную форму или функцию. Дифференцировка клеток представляет собой процесс, при котором клетка подвергается созреванию до явно специализированного типа клеток. Дифференцированные клетки обладают отличительными характеристиками, характеризуются специфичными функциями и делятся с меньшей вероятностью, чем их менее дифференцированные эквиваленты.
«Недифференцированная» клетка, например, незрелая, эмбриональная или зародышевая клетка, как правило, не имеет специфичного внешнего вида, может характеризоваться несколькими неспецифичными видами активности, и может обладать слабо выраженными, если такие вообще имеются, функциями, обычно характерными для дифференцированных клеток.
«Соматическая клетка» относится ко всем возможным дифференцированным клеткам, и не включает стволовые клетки, зародышевые клетки или гаметы. Предпочтительно «соматическая клетка» в контексте настоящего документа относится к окончательно дифференцированной клетке.
В контексте настоящего документа «коммитированный» относится к клеткам, которые считаются необратимо коммитированными в отношении определенной функции. Коммитированные клетки также называют «окончательно дифференцированными клетками».
В контексте настоящего документа «дифференцировка» относится к преобразованию клетки в отношении определенной формы или функции. В случае клеток дифференцировка приводит к образованию более коммитированных клеток.
В контексте настоящего документа «дедифференцировка» относится к потере специализации в отношении формы или функции. В случае клеток дедифференцировка приводит к образованию менее коммитированных клеток.
В контексте настоящего документа «перепрограммирование» относится к обращению генетической программы клетки. Перепрограммированная клетка предпочтительно характеризуется плюрипотентностью.
Термины «дедифференцированный» и «перепрограммированный» или подобные термины применяют в настоящем документе взаимозаменяемо для обозначения клеток, происходящих из соматических клеток, обладающих характеристиками стволовых клеток. Однако указанные термины не предназначены для ограничения раскрытого в настоящем документе заявляемого объекта теоретическими или функциональными соображениями.
Термин «РНК, индуцирующая проявление характеристик стволовых клеток» или «РНК, способная экспрессировать один или несколько факторов, обеспечивающих перепрограммирование соматических клеток в клетки, обладающие характеристиками стволовых клеток», относится к РНК, которая при введении в соматическую клетку индуцирует дедифференцировку клетки.
В контексте настоящего документа «зародышевая клетка» относится к репродуктивной клетке, такой как сперматоцит или ооцит, или клетке, которая будет развиваться в репродуктивную клетку.
В контексте настоящего документа «плюрипотентный» относится к клеткам, которые могут давать начало любому типу клеток, за исключением клеток плаценты или других сопутствующих клеток матки.
Такие термины как «клетка, обладающая характеристиками стволовых клеток», «клетка, обладающая свойствами стволовых клеток» или «клетка, подобная стволовой» применяют в настоящем документе для обозначения клеток, которые, несмотря на то, что они происходят из дифференцированных соматических клеток, не являющихся стволовыми, проявляют один или несколько признаков, типичных для стволовых клеток, в частности, эмбриональных стволовых клеток. Такие признаки включают морфологию эмбриональной стволовой клетки, как например, образование плотных колоний, высокое ядерно-цитоплазматическое отношение и четко выраженные ядрышки, нормальный кариотип, экспрессия теломеразной активности, экспрессия маркеров клеточной поверхности, характерных для эмбриональных стволовых клеток, и/или экспрессия генов, характерных для эмбриональных стволовых клеток. Маркеры клеточной поверхности, характерные для эмбриональных стволовых клеток, например, выбраны из группы, состоящей из специфичного для стадии развития клетки эмбрионального антигена 3 (SSEA-3), SSEA-4, связанного с опухолью антигена 1-60 (TRA-1-60), TRA-1-81 и TRA-2-49/6E. Гены, характерные для эмбриональных стволовых клеток выбраны, например, из группы, состоящей из эндогенного OCT4, эндогенного NANOG, гена фактора роста и дифференцировки 3 (GDF3), гена reduced expression 1 (REX1), гена фактора роста фибробластов 4 (FGF4), специфичного для эмбриональных клеток гена 1 (ESG1), гена белка 2, ассоциированного с плюрипотентностью при развитии (DPPA2), DPPA4 и гена обратной транскриптазы теломеразы (TERT). Согласно одному варианту осуществления один или несколько признаков, типичных для стволовых клеток, включают плюрипотентность.
Согласно одному варианту осуществления настоящего изобретения характеристики стволовых клеток предусматривают морфологию эмбриональной стволовой клетки, где указанная морфология эмбриональной стволовой клетки предпочтительно предусматривает морфологические критерии, выбранные из группы, состоящей из образования плотных колоний, высокого ядерно-цитоплазматического отношения и четко выраженных ядрышек. Согласно определенным вариантам осуществления клетки, обладающие характеристиками стволовых клеток, имеют нормальный кариотип, характеризуются теломеразной активностью, экспрессируют маркеры клеточной поверхности, характерные для эмбриональных стволовых клеток, и/или экспрессируют гены, характерные для эмбриональных стволовых клеток. Маркеры клеточной поверхности, характерные для эмбриональных стволовых клеток, могут быть выбраны из группы, состоящей из специфичного для стадии развития клетки эмбрионального антигена 3 (SSEA-3), SSEA-4, связанного с опухолью антигена 1-60 (TRA-1-60), TRA-1-81 и TRA-2-49/6E, а гены, характерные для эмбриональных стволовых клеток, могут быть выбраны из группы, состоящей из эндогенного OCT4, эндогенного NANOG, гена фактора роста и дифференцировки 3 (GDF3), гена reduced expression 1 (REX1), гена фактора роста фибробластов 4 (FGF4), специфичного для эмбриональных клеток гена 1 (ESG1), гена белка 2, ассоциированного с плюрипотентностью при развитии (DPPA2), DPPA4 и гена обратной транскриптазы теломеразы (TERT).
Предпочтительно клетки, обладающие характеристиками стволовых, представляют собой дедифференцированные и/или перепрограммированные соматические клетки. Предпочтительно клетки, обладающие характеристиками стволовых клеток, обладают ключевыми характеристиками эмбриональных стволовых клеток, такими как плюрипотентный статус. Предпочтительно клетки, обладающие характеристиками стволовых клеток, обладают дифференцировочным потенциалом к дифференцировке в развившиеся производные всех трех первичных зародышевых листков. Согласно одному варианту осуществления первичный зародышевый листок представляет собой эндодерму, а развившимся производным является эпителиальная ткань, подобная кишечному эпителию. Согласно дополнительному варианту осуществления первичный зародышевый листок представляет собой мезодерму, а развившимся производным является поперечнополосатая мышца и/или хрящ. Согласно еще одному варианту осуществления первичный зародышевый листок представляет собой эктодерму, а развившимся производным является нейрональная ткань и/или эпидермальная ткань. Согласно одному предпочтительному варианту осуществления клетки, обладающие характеристиками стволовых клеток, обладают дифференцировочным потенциалом к дифференцировке в нервные клетки и/или кардиомиоциты.
Согласно одному варианту осуществления соматические клетки представляют собой происходящие из эмбриональных стволовых клеток соматические клетки с мезенхимальным фенотипом. Согласно предпочтительному варианту осуществления соматические клетки представляют собой фибробласты, такие как фетальные фибробласты или постнатальные фибробласты, или кератиноциты, предпочтительно кератиноциты, происходящие из волосяных фолликулов. Согласно дополнительным вариантам осуществления фибробласты представляют собой фибробласты легких, фибробласты крайней плоти или фибробласты кожи. Согласно конкретным вариантам осуществления фибробласты представляют собой фибробласты, депонированные в Американской коллекции типовых культур (ATCC) с номером по каталогу CCL-186, депонированные в Американской коллекции типовых культур (ATCC) с номером по каталогу CRL-2097, или депонированные в Американской коллекции типовых культур (ATCC) с номером по каталогу CRL-2522, или предоставленные компанией System Biosciences с номером по каталогу PC501A-HFF. Согласно одному варианту осуществления фибробласты представляют собой фибробласты кожи взрослого человека. Предпочтительно соматические клетки представляют собой клетки человека. В соответствии с настоящим изобретением соматические клетки могут быть генетически модифицированными.
Термин «фактор» в соответствии с настоящим изобретением при применении в отношении его экспрессии с РНК включает белки и пептиды, а также их производные и варианты. Например, термин «фактор» предусматривает OCT4, SOX2, NANOG, LIN28, KLF4 и c-MYC.
Факторы могут происходить из любого вида животного; например, млекопитающих и грызунов. К примерам млекопитающих относятся без ограничения человек и приматы, отличные от людей. Приматы включают без ограничения людей, шимпанзе, павианов, яванских макаков и любых других обезьян Нового или Старого Света. Грызуны включают без ограничения мышь, крысу, морскую свинку, хомяка и песчанку.
В соответствии с настоящим изобретением один или несколько факторов, способных обеспечивать перепрограммирование соматических клеток в клетки, обладающие характеристиками стволовых клеток, предусматривают совокупность факторов, выбранных из группы, состоящей из (i) OCT4 и SOX2, (ii) OCT4, SOX2 и одного или обоих из NANOG и LIN28, (iii) OCT4, SOX2 и одного или обоих из KLF4 и c-MYC. Согласно одному варианту осуществления указанные один или несколько факторов, способных экспрессироваться с РНК, предусматривают OCT4, SOX2, NANOG и LIN28 или OCT4, SOX2, KLF4 и c-MYC. Предпочтительно РНК вводят в указанные соматические клетки путем электропорации или микроинъекции. Предпочтительно в настоящем изобретении предусмотрено обеспечение развития клеток, обладающих характеристиками стволовых клеток, например, путем культивирования соматической клетки в условиях культивирования эмбриональных стволовых клеток, предпочтительно в условиях, подходящих для поддержания плюрипотентных стволовых клеток в недифференцированном состоянии.
OCT4 представляет собой фактор транскрипции из семейства эукариотических факторов транскрипции POU, и является индикатором плюрипотентности эмбриональных стволовых клеток. Он представляет собой экспрессируемый в материнском организме октамер-связывающий белок. Было обнаружено,что он присутствует в ооцитах, внутренней клеточной массе бластоцитов, а также в примордиальных клетках. Белок OCT4 кодирует ген POU5F1. Синонимы названия гена включают OCT3, OCT4, OTF3 и MGC22487. Присутствие OCT4 в определенных концентрациях необходимо для того, чтобы эмбриональные стволовые клетки оставались недифференцированными. Предпочтительно «белок OCT4» или просто «OCT4» относится к OCT4 человека.
Sox2 является представителем семейства генов Sox (SRY-связанный HMG-бокс), которые кодируют факторы транскрипции с одним ДНК-связывающим доменом HMG. Было обнаружено, что SOX2 регулирует клетки-предшественницы нейронов путем ингибирования их способности дифференцироваться. Репрессия фактора приводит к деламинации из желудочковой зоны, после чего происходит выход из клеточного цикла. Такие клетки также начинают терять свои прогениторные свойства в результате потери маркеров прогениторных клеток и ранних маркеров дифференцировки нейронов. Предпочтительно «белок SOX2» или просто «SOX2» относится к SOX2 человека.
NANOG представляет собой ген гомеодомена типа NK-2, и, как было предположено, играет ключевую роль в сохранении плюрипотентности стволовых клеток, вероятно, путем регулирования экспрессии генов, крайне важных для самообновления и дифференцировки эмбриональных стволовых клеток. NANOG функционирует как активатор транскрипции с двумя доменами особенно сильной активации, расположенными на его C-конце. Снижение уровня экспрессии NANOG индуцирует дифференцировку эмбриональных стволовых клеток. Предпочтительно «белок NANOG» или просто «NANOG» относится к NANOG человека.
LIN28 представляет собой консервативный цитоплазматический белок с нетипичным спариванием РНК-связывающих мотивов: домен холодового шока и пара цинковых пальцев CCHC ретровирусного типа. У млекопитающих он в избытке присутствует в различных типах недифференцированных клеток. В плюрипотентных клетках млекопитающих LIN28 обнаруживают в чувствительных к РНКазе комплексах с поли(A)-связывающим белком и в полисомальных фракциях при разделении в градиенте плотности сахарозы, что указывает на то, что он ассоциирован с транслирующимися мРНК. Предпочтительно «белок LIN28» или просто «LIN28» относится к LIN28 человека.
Krueppel-подобный фактор (KLF4) представляет собой фактор транскрипции, содержащий цинковые пальцы, который на высоком уровне экспрессируется в постмитотических эпителиальных клетках различных тканей, например, толстой кишки, желудка и кожи. KLF4 является крайне важным для терминальной дифференцировки таких клеток и вовлечен в регуляцию клеточного цикла. Предпочтительно «белок KLF4» или просто «KLF4» относится к KLF4 человека.
MYC (cMYC) представляет собой протоонкоген,который сверхэкспрессируется в целом ряде злокачественных опухолей человека. Если он специфически мутирован или сверхэкспрессирован, он повышает степень пролиферации клеток и функционирует как онкоген. Ген MYC кодирует фактор транскрипции, который регулирует экспрессию 15% всех генов посредством связывания с последовательностями энхансер-бокса (E-боксы) и рекрутирования гистонацетилтрансфераз (HAT).
. MYC принадлежит к семейству MYC факторов транскрипции, которое также включает гены N-MYC и L-MYC. Факторы транскрипции семейства MYC содержат домен bHLH/LZ (содержащий основную структуру спираль-петля-спираль и лейциновую застежку). Предпочтительно «белок cMYC» или просто «cMYC» относится к cMYC человека.
Ссылку в настоящем документе на определенные факторы, такие как OCT4, SOX2, NANOG, LIN28, KLF4 или c-MYC, следует понимать как включающую также все варианты таких факторов. В частности, ее следует понимать также как включающую все сплайс-варианты, посттрансляционно модифицированные варианты, конформации, изоформы и видовые гомологи таких факторов, которые в естественных условиях экспрессируются клетками.
Термин «миРНК» (микроРНК) относится к некодирующей РНК длиной 21-23 нуклеотида, обнаруживаемой в эукариотических клетках, которая, путем препятствования трансляции целевых мРНК и/или индуцирования их разрушения, модулирует множество клеточных функций, в том числе таковых, которые связаны с самообновлением/дифференцировкой ESC и прохождением клеточного цикла. миРНК являются посттранскрипционными регуляторами, которые связываются с комплементарными последовательностями на транскриптах целевых матричных РНК (мРНК), что обычно приводит к репрессии трансляции или разрушению мишени и сайленсингу гена. Было обнаружено, что миРНК в правильной комбинации способны индуцировать прямое клеточное перепрограммирование соматических клеток в клетки, обладающие характеристиками стволовых клеток in vitro. Например, было обнаружено, что кластер миРНК 302-367 усиливает перепрограммирование соматических клеток.
Предпочтительно стадия обеспечения развития клеток, обладающих характеристиками стволовых клеток, предусматривает культивирование соматических клетках в условиях культивирования эмбриональных стволовых клеток, предпочтительно в условиях, подходящих для поддержания плюрипотентных стволовых клеток в недифференцированном состоянии.
Предпочтительно для обеспечения развития клеток, обладающих характеристиками стволовых клеток, клетки культивируют в присутствии одного или нескольких ингибиторов ДНК-метилтрансферазы и/или одного или нескольких ингибиторов деацетилазы гистонов. Предпочтительные соединения выбраны из группы, состоящей из 5'-азацитидина (5'-azaC), субероиланилидгидроксамовой кислоты (SAHA), дексаметезона, трихостатина A (TSA), маслянокислого натрия (NaBu), скриптаида и вальпроевой кислоты (VPA). Предпочтительно клетки культивируют в присутствии вальпроевой кислоты (VPA), предпочтительно в концентрации 0,5-10 мМ, более предпочтительно 1-5 мМ, наиболее предпочтительно в концентрации приблизительно 2 мМ.
Способы по настоящему изобретению можно применять для осуществления дедифференцировки соматической клетки любого типа. Клетки, которые можно применять, включают клетки, которые могут быть дедифференцированы или перепрограммированы с помощью способов по настоящему изобретению, в частности клетки, которые полностью или частично дифференцированы, более предпочтительно окончательно дифференцированы. Предпочтительно соматическая клетка представляет собой диплоидную клетку, происходящую из организма на пре-эмбриональной, эмбриональной, фетальной стадиях развития и многоклеточных организмов на постнатальной стадии развития. К примерам клеток, которые можно применять, относятся без ограничения фибробласты, такие как фетальные и неонатальные фибробласты или фибробласты взрослого организма, кератиноциты, в частности первичные кератиноциты, более предпочтительно кератиноциты, происходящие из волос, жировые клетки, эпителиальные клетки, эпидермальные клетки, хондроциты, кумулюсные клетки, нервные клетки, глиальные клетки, астроциты, кардиомиоциты, клетки пищевода, мышечные клетки, меланоциты, гемопоэтические клетки, остеоциты, макрофаги, моноциты, и мононуклеарные клетки.
Клетки, в отношении которых можно применять способы по настоящему изобретению, могут принадлежать любому виду животного; например, млекопитающим и грызунам. К примерам клеток млекопитающих, которые могут быть дедифференцированы и повторно дифференцированы в соответствии с настоящим изобретением, включают без ограничения клетки людей и приматов, отличных от человека. Клетки приматов, в отношении которых можно осуществлять настоящее изобретение, включают без ограничения клетки людей, шимпанзе, павианов, яванских макаков и любых других обезьян Нового или Старого Света. Клетки грызунов, в отношении которых можно осуществлять настоящее изобретение, включают без ограничения клетки мышей, крыс, морских свинок, хомяков и песчанок.
Ожидается, что дедифференцированные клетки, полученные в соответствии с настоящим изобретением, будут соответствовать многим из тех же требований, что и плюрипотентные стволовые клетки, и их можно размножать и поддерживать в условиях, применяемых для эмбриональных стволовых клеток, например, в среде для ES-клеток или любой другой среде, которая поддерживает рост эмбриональных клеток. Эмбриональные стволовые клетки сохраняют свою плюрипотентность in vitro при поддержании их в культуре на слое инактивированных фетальных фибробластов, таких как облученные эмбриональные фибробласты мыши или фибробласты человека (например, фибробласты крайней плоти человека, фибробласты кожи человека, фибробласты эндометрия человека, фибробласты яйцевода человека). Согласно одному варианту осуществления питающие клетки человека могут быть аутологичными питающими клетками, полученными из той же культуры перепрограммированных клеток путем прямой дифференцировки.
Кроме того, эмбриональные стволовые клетки человека можно успешно размножать на матригеле в среде, кондиционированной фетальными фибробластами мыши. Стволовые клетки человека можно выращивать в культуре в течение продолжительного периода времени и сохранять в недифференцированном состоянии при определенных условиях культивирования.
Согласно определенным вариантам осуществления условия культивирования клеток могут включать приведение клеток в контакт с факторами, которые могут ингибировать дифференцировку или, в противоположном случае, стимулировать дедифференцировку клеток, например, препятствовать дифференцировке клеток в клетки, отличные от ES, клетки трофэктодермы или другие типы клеток.
Дедифференцированные клетки, полученные в соответствии с настоящим изобретением, можно оценивать с помощью способов, включающих отслеживание изменений фенотипа клеток и определение характеристик экспрессии их генов и белков. Экспрессию генов можно определять с помощью RT-PCR, а продукты трансляции можно определять с помощью иммуноцитохимии и вестерн-блоттинга. В частности, дедифференцированные клетки можно охарактеризовать для определения профиля экспрессии генов и того, характеризуются ли перепрограммированные клетки профилем экспрессии генов, подобным профилю экспрессии, предполагаемому для недифференцированных, плюрипотентных контрольных клеток, таких как эмбриональные стволовые клетки, с применением методик, хорошо известных в данной области, в том числе методик транскриптомики.
В этом отношении можно оценивать экспрессию следующих генов дедифференцированных клеток: OCT4, NANOG, ген фактора роста и дифференцировки 3 (GDF3), ген reduced expression 1 (REX1), ген фактора роста фибробластов 4 (FGF4), специфичный для эмбриональных клеток ген 1 (ESG1), ген белка 2, ассоциированного с плюрипотентностью при развитии (DPPA2), DPPA4, ген обратной транскриптазы теломеразы (TERT), ген эмбрионального антигена 3 (SSEA-3), SSEA-4, ген связанного с опухолью антигена 1-60 (TRA-1-60), TRA-1-81 и TRA-2-49/6E.
Недифференцированные или эмбриональные стволовые клетки, с которыми можно сравнивать перепрограммированные клетки, могут происходить из того же вида, что и дифференцированные соматические клетки. В качестве альтернативы, недифференцированные или эмбриональные стволовые клетки, с которыми можно сравнивать перепрограммированные клетки, могут происходить из вида, отличного от такового для дифференцированных соматических клеток.
Согласно некоторым вариантам осуществления, если определенные гены, которые специфично экспрессируются в недифференцированной клетке, также экспрессируются и в перепрограммированной клетке, имеет место сходство по профилю экспрессии генов между перепрограммированной клеткой и недифференцированной клеткой, например, эмбриональной стволовой клеткой. Например, определенные гены, например, теломеразы, обычно не выявляемые в дифференцированных соматических клетках, можно применять для отслеживания степени перепрограммирования. Подобным образом, для определенных генов, для оценки степени перепрограммирования можно применять факт отсутствия экспрессии.
Способность к самообновлению, признаком которой является индукция теломеразной активности, представляет собой другую характеристику стволовых клеток, которую можно отслеживать у дедифференцированных клеток.
Кариотипический анализ можно осуществлять с применением хромосомных препаратов из митотических клеток, спектрального кариотипирования, анализов длины теломераз, полногеномной гибридизации или других методик, хорошо известных в данной области.
При применении настоящего изобретения РНК, кодирующую соответствующие факторы, вводят в одну или несколько соматических клеток, например, путем электропорации. После введения клетки предпочтительно культивируют с применением условий, которые способствуют поддержанию дедифференцированных клеток (т. е. условия культивирования стволовых клеток). Дедифференцированные клетки затем можно размножать и индуцировать в отношении повторной дифференцировки в другой тип соматических клеток, необходимых для клеточной терапии. Дедифференцированные клетки, полученные в соответствии с настоящим изобретением, можно индуцировать в отношении дифференцировки в один или несколько требуемых типов соматических клеток in vitro или in vivo.
Предпочтительно дедифференцированные клетки, полученные в соответствии с настоящим изобретением, могут давать начало клеткам из любого из трех эмбриональных зародышевых листков, т. е. эндодермы, мезодермы и эктодермы. Например, дедифференцированные клетки могут дифференцироваться в клетки скелетной мышцы, скелета, дермы кожи, соединительной ткани, мочеполовой системы, сердца, крови (лимфатические клетки) и селезенки (мезодерма); желудка, прямой кишки, печени, поджелудочной железы, мочевого пузыря; выстилки уретры, эпителиальных частей трахеи, легких, глотки, щитовидной железы, паращитовидной железы, кишечника (эндодерма); или центральной нервной системы, сетчатки и хрусталика глаза, краниальные и сенсорные клетки, ганглия и нервов, пигментные клетки, соединительной ткани головы, эпидермиса, волос, молочных желез (эктодерма). Дедифференцированные клетки, полученные в соответствии с настоящим изобретением, могут быть повторно дифференцированы in vitro или in vivo с применением методик, известных в данной области.
Согласно одному варианту осуществления настоящего изобретения перепрограммированные клетки, полученные с помощью способов по настоящему изобретению, применяют для получения дифференцированного потомства. Таким образом, согласно одному аспекту настоящее изобретение относится к способу получения дифференцированных клеток, предусматривающему: (i) получение перепрограммированных клеток с применением способов по настоящему изобретению; и (ii) индуцирование дифференцировки перепрограммированных клеток с получением дифференцированных клеток. Стадию (ii) можно осуществлять in vivo или in vitro. Кроме того, дифференцировка может быть индуцирована за счет присутствия соответствующих факторов дифференцировки, которые могут быть либо добавлены, либо присутствовать in situ, например, в организме, органе или ткани, в которые были введены перепрограммированные клетки. Дифференцированные клетки можно применять для получения клеток, тканей и/или органов, которые преимущественно применяют в области трансплантации клеток, тканей и/или органов. При необходимости можно вводить генетические модификации, например, в соматические клетки перед перепрограммированием. Дифференцированные клетки по настоящему изобретению предпочтительно не обладают плюрипотентностью эмбриональной стволовой клетки или эмбриональной зародышевой клетки, и, в сущности, являются специфичными для ткани частично или полностью дифференцированными клетками.
Одним из преимуществ способов по настоящему изобретению является то, что перепрограммированные клетки, полученные в соответствии с настоящим изобретением, могут быть дифференцированы без предварительного отбора или очистки или создания клеточной линии. Соответственно, согласно определенным вариантам осуществления гетерогенная популяция клеток содержит перепрограммированные клетки, дифференцированные в требуемый тип клеток. Согласно одному варианту осуществления смесь клеток, полученных с помощью способов по настоящему изобретению, подвергают воздействию одного или нескольких факторов дифференцировки и культивируют in vitro.
Способы обеспечения дифференцировки перепрограммированных клеток, полученных с помощью раскрытых в настоящем документе способов, могут предусматривать стадию пермеабилизации перепрограммированной клетки. Например, клетки, полученные с помощью описанных в настоящем документе методик перепрограммирования, или, в качестве альтернативы, гетерогенную смесь клеток, содержащую перепрограммированные клетки, можно пермеабилизовать перед подверганием воздействию одного или нескольких факторов дифференцировки или клеточного экстракта или другого препарата, содержащего факторы дифференцировки.
Например, дифференцированные клетки можно получать путем культивирования недифференцированных перепрограммированных клеток в присутствии по меньшей мере одного фактора дифференцировки и отбора дифференцированных клеток из культуры. Отбор дифференцированных клеток может быть основан на фенотипе, таком как экспрессия определенных клеточных маркеров, присутствующих на дифференцированных клетках, или результатах функциональных анализов (например, способность проявлять одну или несколько функций дифференцированных клеток конкретного типа).
Согласно другому варианту осуществления клетки, перепрограммированные в соответствии с настоящим изобретением, генетически модифицируют посредством добавления, делеции или модификации их последовательности(последовательностей) ДНК.
Перепрограммированные или дедифференцированные клетки, полученные в соответствии с настоящим изобретением, или клетки, происходящие из перепрограммированных или дедифференцированных клеток, являются пригодными для исследований и терапии. Перепрограммированные плюрипотентные клетки могут быть дифференцированы в любые клетки организма, в том числе без ограничения клетки кожи, хряща, кости, скелетной мышцы, сердечной мышцы, почки, печени, крови и кроветворения, предшественники сосудистых клеток и клетки эндотелия сосудов, бета-клетки поджелудочной железы, нейроны, клетки глии, клетки сетчатки, нейрональные клетки, клетки кишечника, клетки легкого и гепатоциты.
Перепрограммированные клетки являются пригодными для регенеративной/репаративной терапии и могут быть трансплантированы нуждающемуся в этом пациенту. Согласно одному варианту осуществления клетки являются аутологичными по отношению к пациенту.
Перепрограммированные клетки, полученные в соответствии с настоящим изобретением, можно применять, например, для терапевтических стратегий лечения нарушений со стороны сердечно-сосудистой, нервной, эндокринной, сосудистой систем, поражений сетчатки, дерматологических и скелетно-мышечных нарушений и других заболеваний.
Например, и без ограничения, перепрограммированные клетки по настоящему изобретению можно применять для восполнения клеток у животных, естественные клетки которых были сокращены в количестве вследствие старения или абляционной терапии, например, лучевой терапии и химиотерапии злокачественной опухоли. Согласно другому неограничивающему примеру перепрограммированные клетки по настоящему изобретению являются пригодными для регенерации органов и восстановления тканей. Согласно одному варианту осуществления настоящего изобретения перепрограммированные клетки можно применять для возобновления поврежденной мышечной ткани, в том числе мышц, пораженных дистрофическим заболеванием, и мышц, поврежденных вследствие ишемического события, такого как инфаркт миокарда. Согласно другому варианту осуществления настоящего изобретения раскрытые в настоящем документе перепрограммированные клетки можно применять для сглаживания рубцевания у животных, в том числе людей, после травматического повреждения или хирургического вмешательства. Согласно такому варианту осуществления перепрограммированные клетки по настоящему изобретению вводят системно, как, например, внутривенно, и при этом они мигрируют в участок недавно травмированной ткани, рекрутированные циркулирующими цитокинами, секретируемыми поврежденными клетками. Согласно другому варианту осуществления настоящего изобретения перепрограммированные клетки можно вводить локально в участок, нуждающийся в лечении, или восстановлении, или регенерации.
Согласно одному варианту осуществления настоящего изобретения нуклеиновые кислоты, такие как РНК, вводят пациенту ex vivo способами, т. е. путем взятия клеток у пациента, генетического модифицирования указанных клеток и повторного введения модифицированных клеток пациенту. Способы трансфекции и трансдукции известны специалисту в данной области.
Термин «трансфекция» относится к введению нуклеиновых кислот, в частности РНК, в клетку. Для целей настоящего изобретения термин «трансфекция» также включает введение нуклеиновой кислоты в клетку или поглощение нуклеиновой кислоты такой клеткой, где клетка может находиться в организме субъекта, например, пациента. Таким образом, в соответствии с настоящим изобретением клетка, подлежащая трансфекции описанной в настоящем документе нуклеиновой кислотой, может находиться in vitro или in vivo, например, клетка может образовывать часть органа, ткани и/или организма пациента. В соответствии с настоящим изобретением трансфекция может быть транзиентной или стабильной. Для некоторых вариантов применения трансфекции достаточно, чтобы трансфицированный генетический материал был экспрессирован только транзиентно. Поскольку нуклеиновая кислота, включенная в процесс трансфекции, обычно не интегрируется в ядерный геном, чужеродная нуклеиновая кислота будет утрачена после митоза или будет разрушена. В клетках, допускающих эписомную амплификацию нуклеиновых кислот, скорость утрачивания снижена. Если требуется, чтобы трансфицированная нуклеиновая кислота постоянно сохранялась в геноме клетки и ее дочерних клетках, должна иметь место стабильная трансфекция. РНК можно трансфицировать в клетки для транзиентной экспрессии кодируемого ею белка.
В соответствии с настоящим изобретением для введения нуклеиновых кислот в клетки, т. е. переноса или трансфицирования, можно применять любой пригодный метод. Предпочтительно РНК трансфицируют в клетки с помощью стандартных методик. Такие методики включают электропорацию, липофекцию и микроинъекцию. Согласно одному особенно предпочтительному варианту осуществления настоящего изобретения РНК вводят в клетки путем электропорации. Электропорация или электропермеабилизация относится к значительному повышению электропроводимости и проницаемости плазматической мембраны клетки, обусловленному прилагаемым извне электрическим полем. Указанную методику обычно применяют в молекулярной биологии в качестве способа введения в клетки некоторых веществ. В соответствии с настоящим изобретением предпочтительно, чтобы введение в клетки нуклеиновой кислоты, кодирующей белок или пептид, приводило в результате к экспрессии указанных белка или пептида.
В соответствии с настоящим изобретением нуклеиновые кислоты могут быть направлены в определенные клетки. Согласно таким вариантам осуществления носитель, применяемый для введения нуклеиновой кислоты в клетку (например, ретровирус или липосома), может содержать связанную нацеливающую молекулу. Например, молекула, такая как антитело, специфичное к поверхностному мембранному белку на целевой клетке или лиганду рецептора на целевой клетке, может быть включена в носитель нуклеиновой кислоты, или связана с ним. Если требуется введение нуклеиновой кислоты с помощью липосом, белки, связывающиеся с поверхностным мембранным белком, ассоциированным с эндоцитозом, могут быть включены в состав на основе липосом для обеспечения нацеливания и/или поглощения. Такие белки включают капсидные белки или их фрагменты, которые являются специфичными для определенного типа клеток, антитела к интернализированным белкам, белки, нацеливающиеся на внутриклеточный участок, и т. д.
«Репортер» относится к молекуле, как правило, пептиду или белку, кодируемому репортерным геном, и измеряемому в анализе по гену-репортеру. В традиционных системах обычно используют фермент в качестве репортера и измеряют активность указанного репортера.
Термин «сайт множественного клонирования» относится к области нуклеиновой кислоты, содержащей сайты для ферментов рестрикции, любой из которых можно применять для расщепления, например, вектора и вставки нуклеиновой кислоты.
В соответствии с настоящим изобретением два элемента, такие как нуклеотиды или аминокислоты, являются последовательными, если они непосредственно прилегают друг к другу, без какого-либо прерывания. Например, последовательность из x последовательных нуклеотидов N относится к последовательности (N)x.
«Эндонуклеаза рестрикции» или «фермент рестрикции» относится к классу ферментов, которые расщепляют фосфодиэфирные связи в обоих нитях молекулы ДНК в пределах специфичных последовательностей оснований. Они распознают специфичные сайты связывания, называемые последовательностями распознавания, в двунитевой молекуле ДНК. Сайты, по которым указанные фосфодиэфирные связи в ДНК расщепляются указанными ферментами, называют сайтами расщепления. В случае ферментов типа IIS, сайт расщепления расположен на определенном расстоянии от сайта связывания ДНК. В соответствии с настоящим изобретением термин «эндонуклеаза рестрикции» предусматривает, например, ферменты SapI, EciI, BpiI, AarI, AloI, BaeI, BbvCI, PpiI и PsrI, BsrD1, BtsI, EarI, BmrI, BsaI, BsmBI, FauI, BbsI, BciVI, BfuAI, BspMI, BseRI, EciI, BtgZI, BpuEI, BsgI, MmeI, CspCI, BaeI, BsaMI, Mva1269I, PctI, Bse3DI, BseMI, Bst6I, Eam1104I, Ksp632I, BfiI, Bso31I, BspTNI, Eco31I, Esp3I, BfuI, Acc36I, AarI, Eco57I, Eco57MI, GsuI, AloI, Hin4I, PpiI и PsrI.
Термин «стабильность» РНК относится к «времени полужизни» РНК. «Время полужизни» относится к периоду времени, который требуется для устранения половины активности, количества или числа молекул. В контексте настоящего изобретения время полужизни РНК является показателем стабильности указанной РНК.
Нуклеиновые кислоты, такие как описанная в настоящем документе РНК, в частности, при применении для описанных в настоящем документе вариантов лечения, могут находиться в форме фармацевтической композиции или набора, содержащих нуклеиновую кислоту и необязательно один или несколько фармацевтически приемлемых носителей, разбавителей и/или вспомогательных веществ.
Фармацевтические композиции предпочтительно являются стерильными и содержат эффективное количество нуклеиновой кислоты.
Фармацевтической композиции обычно предусмотрены в единообразной лекарственной форме и могут быть получены способом, известным в данной области. Фармацевтическая композиция, например, может находиться в форме раствора или суспензии.
Фармацевтическая композиция может содержать соли, буферные вещества, консерванты, носители, разбавители и/или вспомогательные вещества, все из которых предпочтительно являются фармацевтически приемлемыми. Термин «фармацевтически приемлемый» относится к нетоксичному веществу, которое не нарушает действие активного(активных) компонента(компонентов) фармацевтической композиции.
Соли, которые не являются фармацевтически приемлемыми, можно применять для получения фармацевтически приемлемых солей, и они включены в настоящее изобретение. Фармацевтически приемлемые соли такого типа предусматривают без ограничения таковые, полученные из следующих кислот: соляная, бромистоводородная, серная, азотная, фосфорная, малеиновая, уксусная, салициловая, лимонная, муравьиная, малоновая, янтарная кислоты и т. д. Фармацевтически приемлемые соли также могут быть получены в виде солей щелочных металлов или солей щелочноземельных металлов, таких как соли натрия, соли калия или соли кальция.
Подходящие буферные вещества для применение в фармацевтической композиции включают уксусную кислоту в виде соли, лимонную кислоту в виде соли, борную кислоту в виде соли и фосфорную кислоту в виде соли.
Подходящие консерванты для применения в фармацевтической композиции включают хлорид бензалкония, хлорбутанол, парабен и тимеросал.
Термин «носитель» относится к органическому или неорганическому компоненту естественного или неестественного (синтетического) происхождения, с которым активный компонент объединяют с целью содействия, усиления его действия или обеспечения возможности применения. В соответствии с настоящим изобретением термин «носитель» также включает один или несколько совместимых твердых или жидких наполнителей, разбавителей или инкапсулирующих веществ, которые подходят для введения пациенту.
Возможные вещества-носители для парентерального введения представляют собой, например, стерильную воду, растворы на основе глюкозы, раствор Рингера, раствор Рингера с лактатом, стерильный раствор хлорида натрия, полиалкиленгликоли, гидрированные нафталины и, в частности, биологически совместимые полимеры молочной кислоты, сополимеры молочной и гликолевой кислот или сополимеры полиоксиэтилена и полиоксипропилена.
Подразумевается, что термин «вспомогательное вещество» при применении в настоящем документе означает все вещества, которые могут присутствовать в фармацевтической композиции, и которые не являются активными ингредиентами, как, например, носители, связующие вещества, скользящие вещества, загустители, поверхностно-активные средства, консерванты, эмульгаторы, буферы, ароматизаторы или красители.
Описанные в настоящем документе фармацевтические композиции можно вводить любым традиционным путем, таким как парентеральное введение, в том числе путем инъекции или инфузии. Введение предпочтительно осуществляют парентерально, например, внутривенно, внутриартериально, подкожно, в лимфатический узел, внутрикожно или внутримышечно.
Композиции, подходящие для парентерального введения, обычно предусматривают стерильный водный или безводный препарат активного соединения, который предпочтительно изотоничен крови реципиента. Примерами совместимых носителей и растворителей являются раствор Рингера и изотоничный раствор хлорида натрия. Кроме того, обычно в качестве раствора или суспензионной среды применяют стерильные нелетучие масла.
Описанные в настоящем документе средства и композиции предпочтительно вводят в эффективных количествах. «Эффективное количество» относится к количеству, которое позволяет достичь требуемой реакции или требуемого эффекта в отдельности или вместе с дополнительными дозами. В случае лечения определенного заболевания или определенного состояния требуемая реакция предпочтительно относится к подавлению течения заболевания. Такой случай предусматривает замедление прогрессирования заболевания и, в частности, прерывание или обращение прогрессирования заболевания. Требуемая реакция при лечении заболевания или состояния также может обеспечивать задержку проявления или предотвращение проявления указанного заболевания или указанного состояния.
Эффективное количество описанных в настоящем документе средства или композиции будет зависеть от состояния, подлежащего лечению, тяжести заболевания, индивидуальных параметров пациента, в том числе возраста, физиологического состояния, размера и веса, длительности лечения, типа сопутствующей терапии (если имеется), конкретного пути введения и подобных факторов. Соответственно, дозы вводимых описанных в настоящем документе средств могут зависеть от нескольких из таких параметров. В случае, когда реакция у пациента является недостаточной при начальной дозе, можно применять более высокие дозы (или более высокие дозы, эффективность которых достигается другим, более локализованным путем введения).
Настоящее изобретение более детально описано с помощью следующих фигур и примеров, которые должны толковаться только как иллюстративные, а не ограничивающие. Основываясь на описании и примерах, дополнительные варианты осуществления доступны для специалиста в данной области и также находятся в пределах объема настоящего изобретения.
Чертежи
Фигура 1. Краткое представление процесса отбора in vivo.
Для получения исходной библиотеки незрелые дендритные клетки человека в течение пяти часов выращивали в присутствии актиномицина D, ингибитора транскрипции, для предварительного отбора стабильных РНК. Оставшуюся клеточную мРНК экстрагировали и очищали с применением набора Poly(A)Purist (Ambion), а затем подвергали фрагментации с помощью нуклеазы P1 (Roche). С этой целью 10 мкг РНК инкубировали в течение 45 мин. с 0,3 Ед. NP-1 в 8 мкл 50 мМ NaAc буфера (pH 5,5) в общем объеме реакции 24 мкл. После очистки на колонках RNeasy (Qiagen) фрагменты были готовы для проведения обратной транскрипции в кДНК. Синтез первой и второй нитей выполняли с применением, и следуя его протоколу, набора для синтеза 1-ой нити кДНК RevertAid Premium (Fermentas) и гексамерного праймера с определенной праймерной последовательностью и сайтом для рестриктазы сNotI. Для достройки 5'-выступающих концов и удаления 3'-выступающих концов кДНК затем инкубировали с 12,5 Ед. ДНК-полимеразы T4 в течение 5 мин. при 15°C. Реакцию останавливали добавлением 5 мкл 0,5 мМ EDTA, pH 8,0, и кДНК очищали с применением колонок NucleoBond (Macherey-Nagel). Расщепление кДНК-библиотеки с помощью NotI (NEB) приводило к образованию фрагментов с тупым и липким концом. Фрагменты дополнительно отбирали по размеру путем получения в геле с целью обеспечения удаления всех фрагментов размером менее 150 п.о. Для клонирования библиотеки вектор, как показано на панели A, расщепляли с помощью EcoRV и NotI, оставляя соответственно тупой и липкий концы. На следующей стадии библиотеку лигировали в вектор с применением ДНК-лигазы T4 (Fermentas). Смесь после лигирования непосредственно применяли в качестве матрицы для ПЦР, описанной в табл. 4, с применением высокоточной ДНК-полимеразы для ПЦР с горячим стартом Phusion™ (Finnzymes). После очистки ПЦР-продукт применяли в качестве матрицы для T7-транскрипции, как показано в табл. 5. Инкубацию проводили при 37°C. Через каждые 30 мин. в реакционную смесь добавляли 0,75 мкл 100 мМ GTP. Через 2,5 ч реакцию останавливали добавлением ДНКазы TURBO (2 Ед./мкл, Ambion), и инкубировали еще 15 мин. при 37°C. Наконец, продукт реакции очищали на колонках RNeasy (Qiagen). РНК-библиотеку затем можно было применять для процедуры отбора, начиная с введения РНК путем электропорации в hiDC, как описано ранее (Kuhn et al, 2010). После культивирования с целью отбора проводили экстракцию и очистку РНК с применением колонок RNeasy (Qiagen), и следуя инструкциям производителя. РНК затем применяли в качестве матрицы для синтеза кДНК с применением обратной транскриптазы Superscript II (Invitrogen), и следуя инструкциям производителя, и dT18-праймера. кДНК затем применяли в качестве матрицы для ПЦР, как описано выше. Наконец, ПЦР-продукты можно было применять в качестве матрицы для T7-транскрипции (см. выше) для начала следующего раунда отбора (панель B). Контроль качества образцов ДНК/кДНК и РНК осуществляли в агарозном геле и на биоанализаторе AGILENT 2100 соответственно.
Фиг. 2. Схематическое представление образца в векторе luc2CPmut.
(A) Одиночный элемент или два (вышерасположенный и нижерасположенный) элемента клонировали в качестве 3'-UTR в вектор, как описано. Также показаны контрольные образцы NEG (отрицательный контроль без вставки 3'-UTR), hBg и 2hBg. Получение РНК для раундов отбора. (B) Для введения в hiDC путем электропорации вектор применяли в качестве матрицы для ПЦР с применением удлиненных праймеров, содержащих последовательности промотора T7 и поли(A)-хвоста. ПЦР-продукт затем применяли в качестве матрицы для T7-синтеза in vitro с получением соответствующей IVT-РНК.
Фиг. 3. Эффект отобранных последовательностей в отношении стабильности РНК, кодирующей luc2CPmut.
В результатах продемонстрирована активность люциферазы, время полужизни и общий белок в динамике для РНК, содержащих отобранные последовательности в качестве 3'-UTR, по сравнению с полученной авторами настоящего изобретения эталонной 2hBg, после введения путем электропорации в незрелые дендритные клетки человека (NEG определен на фиг. 2). В верхней панели приведен график зависимости от времени для трех иллюстративных РНК с 3'-UTR, как указано. В нижней левой панели показано время полужизни РНК с соответствующей 3'-UTR, как указано, по сравнению с РНК с 2hBg. Аналогичным образом, в нижней правой панели представлена относительная экспрессия общего белка по сравнению с РНК с 2hBG.
Фиг. 4. Репрезентативная активность люциферазы при применении luc2mut в качестве репортерного гена и новых отобранных 3'-UTR.
После введения путем электропорации РНК с 3'-UTR, как указано, в незрелые дендритные клетки человека через 72 ч измеряли активность люциферазы.
Фиг. 5. Репрезентативные результаты по введению IVT-РНК в фибробласты путем электропорации.
Левая панель: вектор на основе luc2CPmut. Правая панель: вектор на основе luc2mut.
Фиг. 6. Репрезентативные результаты по введению IVT-РНК в Т-клетки путем электропорации.
На самой левой панели представлена относительная экспрессия общего белка для РНК с 3'-UTR FI по сравнению с РНК с 2hBG в CD4+ и CD8+ T-клетках. Аналогичным образом, на средней и самой правой панелях соответственно представлены относительная эффективность трансляции и время полужизни мРНК для РНК с 3'-UTR FI по сравнению с РНК с 2hBG в CD4+ и CD8+ T-клетках.
Фиг. 7. Структура и целостность РНК для тестируемых РНК с модифицированными нуклеотидами.
A: РНК, применяемые в анализах с люциферазой, были сконструированы, как показано здесь. В качестве 5'-кэпа применяли β-S-ARCA(D2). В качестве 5'-UTR применяли 5'-UTR альфа-глобина человека, в том числе последовательность Козак. За геном люциферазы светлячка клонировали две сравниваемые 3'-UTR. В качестве поли-A-хвоста применяли последовательность A30L70.
B: Перед трансфекцией РНК проверяли на предмет их целостности на приборе 2100 Bioanalyzer (Agilent). Все РНК характеризовались достаточно высокой и также сопоставимой целостностью, и, следовательно, могли быть использованы в экспериментах.
Фиг. 8. Эффект 3'-UTR FI в отношении стабильности и функциональности РНК in vivo.
мРНК люциферазы и gp70, содержащую 3'-UTR FI или 3'-UTR 2hBg, составляли вместе с F12 и вводили i.v. мышам BALB/c. После введения мРНК люциферазы спустя 6 ч и 24 ч отслеживали экспрессию; мРНК gp70 вводили в день 0 и день 6, и в день 10 анализировали иммунную активацию посредством окрашивания по CD8 и gp70 tet+.
A) Показаны уровни экспрессии люциферазы через 6 ч и 24 ч после инъекции немодифицированной и m1Y-модифицированной мРНК, содержащей 3'-UTR FI или 3'-UTR 2hBg. Как для немодифицированной, так и для m1Y-модифицированной мРНК люциферазы, содержащей 3'-UTR FI, были показаны сопоставимые уровни экспрессии, как и для соответствующей мРНК, содержащей 3'-UTR 2hBg.
B) Показана процентная доля gp70-специфичных CD8 T-клеток, образующихся в ответ на мРНК gp70, содержащую 3'-UTR FI или 3'-UTR 2hBg. Для двух 3'-UTR были продемонстрированы одинаково удовлетворительные результаты в отношении индуцирования антигенспецифичного иммунного ответа после двух иммунизаций, со значительным повышением числа антигенспецифичных CD8 T-клеток в селезенке тех мышей, которые получали мРНК gp70, содержащую 3'-UTR FI.
Статистика: однофакторный дисперсионный анализ и апостериорный критерий Тьюки, *p<0,05.
Фиг. 9. Эффект стабилизирующих UTR в отношении стабильности самореплицирующейся РНК.
Дестабилизированную люциферазу (Luc2CP) клонировали непосредственно выше 3'-консервативного элемента последовательности нецитотоксической самореплицирующейся (репликон) РНК, происходящей из вируса леса Семлики. РНК-репликон был получен посредством in vitro транскрипции с соответствующей линеаризованной плазмиды и введения путем электропорации в клетки. Экспрессию люциферазы измеряли за период времени от 96 ч до 120 ч путем добавления люминисцентного субстрата. (A) Динамика экспрессии люциферазы в репрезентативном эксперименте с клетками BHK21. (B) Динамика экспрессии люциферазы в репрезентативном эксперименте с фибробластами крайней плоти человека (HFF. Для снижения цитотоксичности высвобождаемых интерферонов типа I для каждого образца осуществляли котрансфекцию с мРНК B18R вируса коровьей оспы. Для ингибирования активации протеинкиназы R и повышения общего уровня трансляция для каждого образца осуществляли котрансфекцию с мРНК E3 вируса коровьей оспы.
Фиг. 10. Отрезки гомологии в FI-элементе.
Подчеркнутые отрезки последовательности согласно предсказанию образуют друг с другом пары оснований. В случае конструкции с «8-нуклеотидной мутацией» первый элемент превращали путем мутирования в aaagggcu с целью нарушения взаимодействий со вторым элементом.
Фиг. 11. Артефакты в IVT на основе ПЦР-матрицы при применении 2hBgUTR.
A: Схематическое представление получения матрицы для IVT с помощью ПЦР. 5'-Праймер гибридизируется выше промотора T7, 3'-праймер содержит последовательность поли-A-хвоста из 120 нуклеотидов и гибридизируется с кодируемой плазмидой поли-A и частью 3'-UTR. В случае 2hBgUTR может иметь место неспецифическое связывание праймера с матрицей вследствие гибридизации с первым повтором. B: ПЦР-продукты из плазмиды, содержащей 2hBgUTR. Красной стрелкой показан побочный продукт, что указывает на усечение 1hBg. C: РНК, транскрибированная с такого ПЦР-продукта, следовательно, также представлена укороченным побочным продуктом (стрелка). D: ПЦР-продукты из плазмиды, содержащей FI-элемент в качестве 3'-UTR. Не наблюдаются какие-либо побочные продукты. E: Полученная мРНК характеризуется ожидаемой высокой степенью целостности без каких-либо дополнительных побочных пиков.
Фиг. 12. Схематическое представление усеченных UTR-элементов и время полужизни соответствующих мРНК-конструкций.
На верхней панели фиг. A показано схематическое представление усеченных UTR-элементов с привязкой к положениям нуклеиновой кислоты в полноразмерной последовательности F-элемента под SEQ ID NO: 86, перекрываемой такими усеченными вариантами.
На нижней панели фиг. A показано относительное время полужизни мРНК, содержащей усеченную UTR, с привязкой к мРНК, содержащей полноразмерную последовательность F-элемента под SEQ ID NO: 86. мРНК, кодирующие репортерную люциферазу, вводили путем электропорации в hiDC, и их экспрессию отслеживали в динамике путем измерений люциферазы с целью определения относительного времени полужизни РНК.
На верхней панели фиг. B показано схематическое представление усеченных UTR-элементов с привязкой к положениям нуклеиновой кислоты в полноразмерной последовательности I-элемента под SEQ ID NO: 115, перекрываемой такими усеченными вариантами.
На нижней панели фиг. B показано относительное время полужизни мРНК, содержащей усеченную UTR, с привязкой к мРНК, содержащей полноразмерную последовательность I-элемента под SEQ ID NO: 115. мРНК, кодирующие репортерную люциферазу, вводили путем электропорации в hiDC, и их экспрессию отслеживали в динамике путем измерений люциферазы с целью определения относительного времени полужизни РНК.
Фиг. 13. Относительное время полужизни и уровень экспрессии белка с мРНК-конструкций, содержащих F-, I- или FI-элементы, по отношению к случайным UTR.
На фиг. 13 показаны относительное время полужизни и экспрессия белка с мРНК-конструкций, содержащих F-, I- или FI-элементы, по отношению к случайным UTR. С этой целью полноразмерные отдельные F- и I-элементы, а также комбинацию FI сравнивали со случайными 3'-UTR (длиной 257 нуклеотидов). Все элементы клонировали в кодирующие люциферазу конструкции, подвергали транскрибированию in vitro в мРНК, вводили путем электропорации в hiDC, измеряли уровень экспрессии люциферазы в динамике и рассчитывали относительные показатели времени полужизни и экспрессии общего белка.
Фиг. 14. UTR-элементы для перепрограммирования клеток.
На фиг. 14A показана хронология перепрограммирования первичных фибробластов крайней плоти человека. 40000 клеток высевали в 12-луночный планшет и в течение трех (3x) или четырех (4x) дней подряд подвергали липофекции смесями мРНК, которые состояли из 0,33 мкг немодифицированной in vitro транскрибированной (IVT)-РНК, кодирующей TF перепрограммирования OCT4, SOX2, KLF4, cMYC, NANOG и LIN28 (OSKMNL) (1:1:1:1:1:1), с 0,08 мкг каждого из B18R, E3 и K3 (EKB), и 0,17 мкг смеси миРНК, состоящей из миРНК 302a-d и 367 (1:1:1:1:1:1). РНК-конструкции, таким образом, отличались только по их 3'-UTR, состоящей из тандемного повтора 3'-UTR β-глобина человека (2hBg), F-I-элемента (FI) или I-F-элемента (IF). Начиная с дня 9 наблюдали образование колоний, а в день 11 проводили анализ колоний.
На фиг. 14B показано окрашивание по щелочной фосфатазе (AP) сформировавшихся колоний, а на фиг. 14C показана соответствующая гистограмма, на которой представлено подсчитанное число AP-положительных колоний.
На фиг. 14D показана морфология полученных колоний клеток iPS при применении РНК, содержащих FI-UTR. Она была подобна таковой у клеток hES, с плотно группирующимися небольшими клетками в отдельных колониях и четко выраженными краями.
На фиг. 14E показаны колонии, полученные как на D, положительно окрашенные по AP, в четырех- и десятикратном увеличении.
На фиг. 14F показаны колонии, полученные как на D, при окрашивании живых клеток по маркеру клеточной поверхности TRA-1-60 hES.
На фиг. 14G показаны результаты экспрессии мРНК маркеров hES OCT4 (эндогенный), NANOG (эндогенный), LIN28 (эндогенный), TERT и REX1 оценивали путем осаждения колоний, выделения общей РНК и количественного определения с помощью qRT-PCR.
Примеры
Пример 1. Идентификация элементов последовательности, которые стабилизируют мРНК.
Для идентификации новых элементов последовательности, которые стабилизируют мРНК, авторы настоящего изобретения разработали процесс in vivo отбора с применением hiDC в качестве селективного окружения для in vitro транскрибированной РНК. Исходную РНК-библиотеку создавали с применением встречающихся в природе последовательностей мРНК, полученных из hiDC. Перед выделением РНК клетки выращивали в течение 5 часов в присутствии ингибитора транскрипции актиномицина D (ActD) для предварительного отбора стабильных РНК. Оставшуюся мРНК затем экстрагировали и укорачивали с получением фрагментов длиной 200-800 нуклеотидов, осуществляли обратную транскрипцию и клонировали как 3'-UTR в вектор, несущий последовательность 5'UTR hAg и репортерный ген, который был выбран в качестве основы для процесса отбора. ДНК-матрицу, применяемую для последующей транскрипции мРНК из библиотеки, амплифицировали с помощью ПЦР, в ходе которой промотор T7 был включен с помощью 5'-, а поли-A-хвост A60 был включен с помощью 3'-праймера. Транскрибированную мРНК затем включали в процесс in vivo отбора, который состоял из нескольких раундов in vitro транскрипции библиотеки, введения соответствующей РНК в hiDC путем электропорации и экстракции и амплификации стабильных последовательностей через определенные моменты времени. После синтеза кДНК осуществляли амплификацию выбранных последовательностей с помощью ПЦР со специфичными праймерами. Полученные ПЦР-продукты затем применяли в качестве матриц для новой мРНК-библиотеки. Это осуществляли в шесть раундов, с экстракцией оставшихся РНК через 24 часа в раунде 1, 48 часов в раундах 2 и 3, 72 часа в раундах 4 и 5 и, в конечном счете, 96 часов, а также через одну и две недели в раунде 6 (после электропорации клетки разделяли на три части, а затем собирали по отдельности в заданные моменты времени).
В результате наблюдений за процессом отбора после проведения раундов 1-5 было продемонстрировано значительное повышение среднего времени полужизни соответствующего пула РНК, что было характерно в случае обогащения стабилизирующими 3'-UTR-элементами (табл. 1). Однако повышение стабильности было менее выраженным в раундах более высокого порядка. Таким образом, процесс отбора прекращали по окончанию последнего шестого раунда, в котором РНК экстрагировали из клеток спустя 96 часов, одну неделю и две недели. Для определения характеристик отобранных последовательностей осуществляли секвенирование более 350 отдельных клонов, 108 из раунда 5, 88 из раунда 6/96 часов, 110 из раунда 6/1 неделя и 96 из раунда 6/2 недели. Все последовательности сравнивали друг с другом, а также проводили поиск BLAST для идентификации их геномного происхождения. В данном случае особенно обращали внимание на то, происходят последовательности из эндогенных 5'- или 3'-UTR, или из кодирующей области. Наконец, данные по уровню их экспрессии в hiDC загружали из NextBio (Illumina). Всего можно было идентифицировать семь групп, (i) для которых было обнаружено множество последовательностей, (ii) которые происходили из 3'-UTR эндогенной РНК или из эндогенной некодирующей РНК и (iii) которые были явно экспрессированы в hiDC (табл. 2). Они происходили из следующих генов: Fc-фрагмент IgG, рецептор, транспортер, альфа (B, FCGRT, NM_001136019), специфичный для лимфоцитов белок 1 (D, LSP1, NM_002339), хемокиновый лиганд 22 (E, CCL22, NM_002990), амино-концевой энхансер split (F, AES, NM_198969), представитель 3 семейства фосфолипаз D (G, PLD3, NM_001031696), кодируемая в митохондриях 12S РНК (I, MT_RNR1, NC_012920), DR-бета 4 главного комплекса гистосовместимости класса II (J, HLA-DRB4, NM_021983). Следует отметить, что для простоты заглавные буквы от B до I, указанные в круглых скобках, применяют в качестве сокращений для этих элементов. Важно отметить, что во всех случаях клоны одной последовательности отличаются по их точным 5'- и 3'-концам, что демонстрирует, что это является результатом различий исходных клонов, и не является просто искусственным обогащением в ходе процесса (см. дополнения к документу для полного перечня всех последовательностей, идентифицированных в ходе скрининга).
Пример 2. Определение характеристик отдельных идентифицированных элементов последовательности.
Для определения характеристик идентифицированных элементов последовательности из каждой группы выбирали репрезентативного кандидата (конкретные последовательности отмечены в дополнении к документу). Эту последовательность затем клонировали как 3'-UTR в вектор с репортерным геном люциферазы, уровень экспрессии которого можно анализировать в динамике после переноса в клетки. Ранее было продемонстрировано, что из профиля экспрессии, наблюдаемого для белка, можно точно определить относительную стабильность и эффективность трансляции РНК (Kuhn 2010 Gene Ther.). Специфичный репортер, применяемый в этом эксперименте, luc2CPmut, представляет собой дестабилизированную форму люциферазы (Promega). Это позволяет выявить даже малые изменения в стабильности РНК. In vitro транскрибированную РНК, полученную из таких векторов, затем сравнивают в отношении стабильности и эффективности трансляции РНК с полученной авторами настоящего изобретения эталонной мРНК, т. е. содержащей 3'-UTR 2hBg. В качестве контрольных образцов применяли in vitro транскрибированную РНК без 3'-UTR (т. е. содержащую только последовательности, применяемые для клонирования вставок) и одну только с одним бета-глобиновым элементом (1hB).
Начиная с UTR-содержащих векторов, подлежащую транскрибированию область амплифицировали с помощью ПЦР с применением 5'-праймера, содержащего последовательность промотора T7, и 3'-праймера с последовательностью поли(A)-хвоста из 60 нуклеотидов. Очистку ПЦР-фрагментов выполняли с применением AGENCOURT AMPURE XP (Beckman Coulter). К каждой реакционной смеси после ПЦР добавляли 0,6 объема гранул и перемешивали. Спустя 15 мин. инкубации при RT ПЦР, ПЦР-продукты, связанные с гранулами, разделяли на магнитной подставке от избытка праймеров, нуклеотидов, солей и ферментов. Гранулы дважды промывали в течение 30 с 80% этанолом для дополнительного удаления контаминирующих веществ. Требуемые ПЦР-продукты в конечном счете дважды элюировали 30 мкл ddH2O и применяли в качестве матрицы для in vitro транскрипции соответствующих РНК. Для in vitro транскрипции применяли РНК-полимеразу T7 (Fermentas), соответствующий буфер для реакции и 6 мМ NTP. Для эффективного кэппирования РНК концентрацию GTP снижали до 1,5 мМ, и к реакционной смеси добавляли 6 мМ β-S-ARCA(D2) и инкубировали в течение 2,5 ч. при 37°C. РНК очищали на магнитных гранулах с карбоксильными группами (Invitrogen) и оценивали концентрацию и качество РНК с помощью спектрофотометрии и анализа на 2100 Bionanalyzer (Agilent).
В соответствии с их идентификацией в скрининговом подходе, для всех новых последовательностей наблюдали очень схожие характеристики в отношении стабильности РНК, сравниваемые с 2hBg, причем наилучшие показаны для группы I (mtRNR1) (фиг. 3; табл. 3). Важно отметить, что каждый отдельный элемент обеспечивает стабилизацию РНК по сравнению с РНК без 3'-UTR, и даже по сравнению с РНК только с одной копией бета-глобинового элемента. Эффективность трансляции не была существенно затронута, как было видно из прямой корреляции между стабильностью РНК и общим белком, экспрессированным за определенный промежуток времени.
Пример 3. Комбинация отдельных элементов последовательности.
В дополнительном эксперименте попарно комбинировали отдельные последовательности из каждой группы (фиг. 2). Мотивацией для этого было предыдущее наблюдение авторов настоящего изобретения, что комбинация двух 3'-UTR оказывала дополнительное эффект в отношении стабильности и эффективности трансляции РНК (Holtkamp et al. 2006). Стабильность и эффективность трансляции РНК рассчитывали в R путем интерполяции измеренных значений для люциферазы кубическими сплайнами, из которых самый крутой нарастающий наклон был определен как эффективность трансляции и время полужизни, как показатель стабильности. Интеграл интерполированных кубических сплайнов интерпретировали как экспрессию общего белка. В общей сложности клонировали 64 комбинаций, т. е. все возможные комбинации семи новых идентифицированных последовательностей и 3'-UTR бета-глобина человека (табл. 6). Как описано выше, РНК получали на основе этих ДНК-матриц, а затем вводили в hiDC путем электропорации. В качестве контролей также были включены РНК, содержащие отдельные элементы. Для большинства из семи новых элементов наблюдали, что по меньшей мере одна комбинация с другим элементом дает РНК с более высокой стабильностью, чем в случае только одного элемента (от табл. 7 до табл. 13). Интересно, что в большинстве случаев комбинация с элементом I (mtRNR1) повышала время полужизни РНК. В данном случае стабильность РНК была в целом даже выше по сравнению с РНК с 3'-UTR 2hBg (от табл. 7 до табл. 13). Почти все комбинации оказывали положительный эффект в отношении эффективности трансляции РНК. В конечном итоге, комбинированные эффекты в отношении стабильности и эффективности трансляции РНК приводили к повышению уровня экспрессии общего белка до 1,74 раза. Таким образом, авторы настоящего изобретения смогли идентифицировать отдельные элементы (со значениями длины менее 233 нуклеотидов), а также комбинации двух разных элементов, которые приводят к образованию РНК с повышенной стабильностью и/или эффективностью трансляции, но в тоже время позволяют избежать проблем, связанных с наличием идентичных копий одного элемента, как описано выше для 2hBg.
Для подтверждения результатов, полученных с применением дестабилизированной формы люциферазы, предыдущие эксперименты повторяли с РНК, несущими последовательность стандартной люциферазы (Promega), и следующие отобранные 3'-UTR: mtRNR1 (I), mtRNR1-AES (IF), AES-mtRNR1 (FI), mtRNR1-hBg (IhBg) и hBg-mtRNR1 (IhBg). Как видно из фиг. 4 и табл. 14, можно было получить результаты, эквивалентные рассматриваемым выше, подтверждающие, что новые элементы, отдельно или в комбинации, повышают стабильность и/или эффективность трансляции мРНК, как в случае 2hBg-элемента.
Пример 4. Анализ мРНК, несущих отобранные элементы последовательности, в других типах клеток.
Новые отобранные 3'-UTR mtRNR1 и AES также тестировали в различных типах клеток и клеточных линиях, чтобы определить, имеет ли место специфичность для hiDC. Последовательности тестировали на фибробластах человека (HFF), миобластах мыши (C2C12) (фиг. 5) и T-клетках (фиг. 6) для оценки того, являются ли они стабилизирующими также и в этих клетках.
Клетки HFF и C2C12 собирали и готовили для электропорации. 2,0 мкг IVT-РНК затем вводили путем электропорации вместе с 1,0 мкг кодирующей GFP РНК, содержащей указанные 3'-UTR. После электропорации клетки разделяли. Для измерения активности люциферазы 5000 клеток на лунку распределяли в трех повторностях в 96-луночном планшете в соответствии с всего 7 моментами времени (2, 4, 8, 24, 48 и 72 ч.). 2E+05 клеток на лунку высевали в 6-луночные планшеты для сбора через 24 ч. для FACS (GFP-сигнал). Это позволяло отслеживать показатели эффективности трансфекции. Они различались в пределах от 72 до 90% и могли быть включены в расчет времени полужизни. Результаты, полученные для HFF и C2C12, а также T-клеток, подтверждали результаты, полученные ранее для hiDC. Комбинация I с F, в частности, в 2-3 раза превосходила 2hBg по времени полужизни. Кроме того, FI, как было показано, превосходит полученный авторами настоящего изобретения эталон в 3 раза по эффективности трансляции в клетках C2C12 и в 2 раза по продукции белка за определенный промежуток времени. Их этих результатов видно, что I и F не являются специфичными для hiDC, но также повышают стабильность и эффективность трансляции мРНК в других клетках.
Пример 5. 3'-UTR FI повышает уровень экспрессии с модифицированной мРНК.
Для некоторых вариантов применения, в том числе заместительной терапии белком, мРНК с модифицированными нуклеотидами предпочтительнее немодифицированных мРНК ввиду их сниженной иммуногенности (Kariko et al., 2008). Однако модификации оснований могут оказывать эффект в отношении стабильности мРНК либо прямо влияя на взаимодействие с соответствующим РНК-связывающим белком, либо изменяя формирование вторичной структуры РНК. Соответственно, отобранные 3'-UTR могут по-разному себя вести в контексте модифицированных мРНК. Таким образом, авторы настоящего изобретения сравнили комбинацию F и I с 2hBgUTR в контексте m1Y-модифицированной мРНК в hiDC, HFF, CD8+ и CD4+ T-клетках и MEF мыши, C2C12 и bmDC. В качестве репортера применяли люциферазу (см. Фиг. 7A касательно схемы конструкции). Для получения модифицированных мРНК в IVT-реакции U полностью заменяли m1Y. Во всех экспериментах немодифицированные РНК были включены в качестве контроля. Целостность полученных мРНК не была затронута заменой UTP на m1YTP (фиг. 7B). Клетки подвергали электропорации с применением условий, описанных в табл. 15, и измеряли уровни люциферазы спустя 3, 6, 12, 24, 48, 72 и 96 ч.
С помощью электропорации с использованием немодифицированной мРНК люциферазы, можно было воспроизвести наблюдаемые ранее эффекты: Во всех типах клеток FI-элемент превосходил контроль 2hBg в отношении обеспечения стабильности РНК, или был эквивалентным ему (табл. 16A). В то время как в DC мыши и T-клетках человека показатели времени полужизни мРНК были сопоставимы между двумя 3'-UTR, в клетках HFF FI-элемент обеспечивал увеличение показателей времени полужизни мРНК в 1,69 раза. Количество общего белка было повышено во всех клеточных линиях, наиболее заметно в клетках HFF (в 2,45 раза).
В случае модифицированной мРНК FI-элемент также обеспечивал увеличение времени полужизни мРНК по сравнению с 2hBg в hiDC, причем количество общего белка повышалось более чем в два раза (табл. 16B). Результаты для других типов клеток также схожи с результатами, полученными с немодифицированной мРНК: FI-элемент превосходил 2hBg во всех экспериментах, включающих клетки HFF, MEF и C2C12, и был сопоставим в T-клетках и DC мыши (табл. 16B). Таким образом, U-модификация не изменяла способность FI-элемента стабилизировать мРНК.
Пример 6. 3'-UTR FI повышает уровень экспрессия с мРНК независимо от способа трансфекции.
До сих пор эксперименты выполняли с применением электропорации в качестве способа трансфекции. При электропорации доставляемая мРНК попадает непосредственно в цитоплазму, в обход пути эндосомального поглощения, который имеет место при трансфекции посредством липофекции. Чтобы определить, функционирует ли FI-элемент также и в этих условиях, клетки подвергали липофекции этими же содержащими FI и 2hBg мРНК люциферазы, которые применяли в предыдущих экспериментах, с применением RNAiMAX в качестве реагента для трансфекции. Аналогично, при липофекции FI-элемент обеспечивал повышение уровня экспрессии люциферазы, хотя повышении было менее выраженным, по сравнению с экспериментами, в которых РНК доставляли посредством электропорации (табл. 16C). Таким образом, способ трансфекции не влияет на стабилизирующий эффект FI-элемента.
Пример 7. Содержащая 3'-UTR FI и 2hBgUTR мРНК обеспечивает сопоставимую экспрессию белка и иммунную активацию in vivo.
Для оценки in vivo экспрессии белка с мРНК, содержащей 3'-UTR FI, те же содержащие FI и 2hBg мРНК люциферазы, что применяли в предыдущих экспериментах, составляли с F12 и вводили i.v. мыши BALB/c. Как показано на фиг. 8, экспрессия люциферазы была сопоставима для обоих 3'-UTR. Антигенспецифичный иммунный ответ также был индуцирован в сопоставимой степени, причем эффект содержащей 3'-UTR FI мРНК был немного сильнее в селезенке.
Пример 8. UTR IF обеспечивает повышение стабильности самореплицирующейся РНК in vitro.
In vitro транскрибированные самореплицирующиеся РНК (РНК-репликон), происходящие из геномов альфавирусов, являются перспективными вакцинными векторами. РНК-репликон кодирует на первых двух третях РНК-репликона ферментный комплекс, необходимый для репликации в цитоплазме (репликаза). Эта репликаза распознает внутреннюю структуру РНК, которая выполняет функцию субгеномного промотора для репликаза-зависимого синтеза субгеномной РНК. Трансгены или антигены для вакцинации кодируются этой субгеномной РНК, которая значительно короче полного репликона. В целом как геномная (т. е. полноразмерный РНК-репликон), так и субгеномная РНК напоминают клеточную мРНК. Обе фланкированы UTR, обе кэпированы и полиаденилированы. Ферменты, ответственные за кэпирование и полиаденилирование, входят в состав репликазного ферментного комплекса. Консервативные элементы последовательности (CSE) в пределах UTR - перекрывающиеся с ORF репликазы в случае 5'-CSE - требуются для связывания репликазы и функционируют как промоторы для синтеза минус-нити (3'-CSE) или синтеза плюс-нити (5'-CSE).
Для оценки того, обеспечивают ли новые стабилизирующие UTR, идентифицированные и утвержденные для использования в нереплицирующейся in vitro транскрибированной мРНК, более высокую стабильность, и, следовательно, более высокий уровень экспрессии трансгена, РНК-репликона, авторы настоящего изобретения клонировали соответствующие последовательности в векторы в качестве матрицы с РНК-репликоном. Поскольку необходимо, чтобы 3'-CSE были расположены непосредственно возле поли-A-хвоста, авторы настоящего изобретения вставили новые UTR непосредственно выше 3'-CSE репликона, кодирующего дестабилизированную люциферазу (Luc2CP). РНК-репликон был синтезирован путем in vitro транскрипции линеаризованных плазмид в качестве матрицы, аналогично IVT-мРНК. РНК-репликон путем электропорации вводили в клетки (BHK21 и HFF) и оценивали экспрессию люциферазы. Как показано на фиг. 9, все вставленные UTR повышали уровень трансляции Luc2CP в обоих применяемых клеточных линиях. Интересно, что комбинации UTR «IF» обеспечивала исключительное повышение уровня трансляции.
Пример 9. Нуклеотидные замены до достижения 90% гомологии не влияли на стабилизирующие свойства FI-элемента.
С учетом процедуры отбора, которую применяли для идентификации новых стабилизирующих UTR-элементов, получали последовательности в определенном диапазоне размеров. Идентификация таких же последовательностей с удлиненными 5'- и 3'-концами дала первоначальное представление о требуемой минимальной длине. Однако, минимальная область, требуемая для каждого элемента для проявления его стабилизирующего эффекта, может быть еще короче. Кроме того, небольшие вариации последовательностей могут все еще быть функциональными, т. е. идентичность любого отдельного нуклеотида может не иметь первостепенной важности для стабилизирующих свойств FI-элемента. Чтобы определить, до какой степени элементы устойчивы к нуклеотидным заменам, последовательности 3'-UTR с 97,5%, 95,0%, 92,5% и 90,0% гомологией с исходным FI-элементом тестировали на предмет экспрессии общего белка и времени полужизни мРНК в hiDC. Нуклеотиды, которые подлежали изменению, были выбраны случайным образом по всей длине последовательности (последовательности 208-211, случайные модификации). мРНК люциферазы с такими модифицированными элементами в качестве 3'-UTR были транскрибированы in vitro, введены путем электропорации в hiDC, и их экспрессию отслеживали в динамике путем измерений люциферазы через 3, 6, 24, 48 и 72 ч. мРНК люциферазы с модифицированным FI-элементом давали то же количество общего белка и характеризовались примерно таким же временем полужизни (табл. 17).
В дополнение к случайным заменам в возрастающем количестве, как описано выше, другой набор модифицированных FI-элементов получали путем рационального введения нуклеотидных замен, которые, вероятно, нарушают вторичную структуру FI-элемента. Известно, что для множества естественных последовательностей 3'-UTR их вторичная структура является важной, поскольку она обеспечивает сайты связывания для регуляторных белков, которые влияют на стабильность мРНК (Addess et al., 1997; Putland et al., 2002; Crucs et al., 2000; Adams et al., 2003). В FI-элементе присутствуют две последовательности длиной 8 нуклеотидов, идеально комплементарные одна другой, одна находится в F-, а другая в I-элементе (фиг. 10). Спаривание оснований таких двух областей также можно наблюдать в большинстве предсказаний mfold. mFold (Zuker, 2003) представляет собой компьютерную программу, позволяющую получать предсказания о вторичной структуре. Для проверки важности такого элемента со специфичной вторичной структурой последовательность изменяли таким образом, чтобы спаривание оснований не происходило (последовательность 212, 8-нуклеотидная мутация). Помимо этих довольно длинных комплементарных последовательностей предсказания mfold для 3'-UTR FI подвергали скринингу в отношении структурных элементов, присутствующих в большинстве исходных укладок, которые, следовательно, должны с высокой вероятностью образовываться in vivo. Нуклеотиды, вовлеченные в спаривание оснований таких укладок, изменяли до достижения 97,5%, 95,0%, 92,5% и 90,0% гомологии относительно исходной FI-последовательности путем обмена их на их партнеры по спариванию оснований, сохраняя таким образом вторичную структуру последовательности (последовательности 217-220, модификации, сохраняющие структуру). Кроме того, эти же последовательности изменяли только в одной нити двунитевой части, тем самым преднамеренно разрушая вторичную структуру. В таких случаях идентичность с исходной последовательностью составляла 98,75%, 97,50%, 96,25% и 95,00% соответственно (последовательности 213-216, модификации, дестабилизирующие структуру).
РНК люциферазы с описанными модифицированными 3'-UTR элементами были транскрибированы in vitro, введены путем электропорации в hiDC, и их экспрессию отслеживали в динамике путем измерений люциферазы через 3, 6, 24, 48 и 72 ч.
Ни в одной из стратегий модификации не наблюдали значительного влияния на время полужизни мРНК. Таким образом, стабилизирующие свойства FI-элемента, по-видимому, являются устойчивыми к изменениям его нуклеотидной последовательности или вторичной структуры по меньшей мере на не более чем 10,0% измененных нуклеотидов. Кроме того, при модификации последовательности FI не наблюдалось снижения количества общего белка (табл. 18 A и B).
Пример 10. Применение FI-элемента вместо 2hBg позволяет избежать неспецифического связывания праймера с матрицей при ПЦР-амплификация РНК-кодирующей область.
Как было показано, FI-элемент является аналогичным или превосходит 3'-UTR 2hBg в отношении стабильности и эффективности трансляции мРНК. Другим преимуществом FI-элемента является его неповторяющаяся последовательность, тогда как две копии 3'-UTR hBg в некоторых случаях могут быть причиной определенных проблем.
Это наиболее наглядно, когда ДНК-матрицу для транскрипции РНК амплифицируют с помощью ПЦР. В таких случаях полноразмерный поли-A-хвост добавляют с помощью олигонуклеотидного 3'-праймера, который связывается с самого края 3'-конца (фиг. 11 A). В случае 2hBgUTR в ходе ПЦР появляются усеченные побочные продукты, которые после секвенирования, как оказывается, состоят из мРНК только с повтором 1hBg в UTR (фиг. 11 B). После транскрипции усечение также видно в случае мРНК (фиг. 11C). Такое явление имеет место в большинстве ПЦР-реакций с конструкциями, содержащими 2hBgUTR-элемент, и не может быть полностью устранено с помощью мер оптимизации, в том числе температуры отжига праймера, состава буфера, последовательности праймера или альтернативных полимераз. Проблема остается даже после вставки уникальной линкерной последовательности между 3'-UTR и поли-A-хвостом. Важно отметить, что интенсивность побочного пика коррелировала с выходом ПЦР-реакции, что указывает на неспецифическое связывание праймера с короткими усеченными ПЦР-фрагментами, которые увеличиваются в количестве с каждым циклом ПЦР, как вероятную причину проблемы. Таким образом, для ДНК-матриц, кодирующих РНК с 2hBg 3'-UTR, не может быть выявлено удовлетворительных условий.
В отличие от этого, ПЦР ДНК-матриц с FI-элементом не приводила к образованию каких-либо усеченных побочных продуктов (фиг. 11 D), а также для полученной мРНК не было показано наличие дополнительного пика в профиле Bioanalyzer (фиг. 11 E). Таким образом, FI-элемент представляет собой значительное улучшение как 3'-UTR по сравнению с 2hBgUTR в отношении целостности матрицы для ПЦР и соответствующего качества РНК.
Пример 11. РНК-стабилизирующие свойства субфрагментов F- и I-элементов.
С учетом процедуры отбора, которую применяли для идентификации новых стабилизирующих UTR-элементов, получали последовательности в определенном диапазоне размеров. Идентификация таких же последовательностей с удлиненными 5'- и 3'-концами дала первоначальное представление о требуемой минимальной длине. Однако, минимальная область, требуемая для каждого элемента для проявления его стабилизирующего эффекта, может быть еще короче.
С этой целью, как для F-, так и для I-элемента разработали пять конструкций репортерной люциферазы, причем каждая содержала укороченную UTR, перекрывающую отличающийся фрагмент исходного элемента, укороченного с 5'- и/или 3'-конца (см. фиг. 12, верхние панели A и B соответственно). Такие репортерные конструкции были транскрибированы in vitro, введены путем электропорации в hiDC, и их экспрессию отслеживали в динамике путем измерений люциферазы через 3, 6, 24, 48 и 72 ч. Полученные кривые экспрессии анализировали на предмет относительного времени полужизни РНК, причем РНК, содержащая соответствующую полноразмерную последовательность, принята за 1 (см. фиг. 12, нижние панели A и B соответственно).
В случае F-элемента, значимого уменьшения времени полужизни мРНК не наблюдали ни для одной из тестируемых подпоследовательностей, что указывает на избыточное, некооперативное вовлечение различных подпоследовательностей вдоль F-элемента в его стабилизирующую функцию. Аналогичный результат можно было получить для I-элемента, хотя в данном случае при применении в качестве 3'-UTR только центральной области (нуклеотиды 37-107) можно было наблюдать незначительное снижение показателей.
Чтобы перенести эти результаты на концепцию, отдельные полноразмерные F- и I-элементы, а также комбинацию FI, сравнивали со случайно выбранным 3'-UTR из исходной библиотеки (длиной 257 нуклеотидов). Это осуществляли путем клонирования исходного пула ДНК и выбора отдельного случайного клона. Как описано выше, кодирующие люциферазу РНК с соответствующими последовательностями UTR вводили путем электропорации в hiDC, измеряли уровень экспрессии люциферазы в динамике и рассчитывали относительные показатели времени полужизни и экспрессии общего белка. По сравнению с F-, I- и FI-элементами РНК со случайно выбранными 3'-UTR были значительно менее стабильными (фиг. 13, верхняя панель). Эффект выбранных UTR в отношении экспрессии общего белка был еще более выраженным (фиг. 13, нижняя панель). Это явно указывает на то, что эффект фрагментов F- и I-элементов, описанных выше, является специфичным в отношении выбранных последовательностей, а не просто обусловлен присутствием последовательности 3'-UTR. Это согласовывается с наблюдаемым повышением стабильности РНК из пула в ходе отбора (см. выше).
Пример 12. Применение стабилизирующих UTR-элементов для перепрограммирования стволовых клеток.
40000 клеток высевали в 12-луночный планшет и в течение трех (3x) или четырех (4x) дней подряд подвергали липофекции смесями мРНК, которые состояли из 0,33 мкг немодифицированной in vitro транскрибированной (IVT)-РНК, кодирующей TF перепрограммирования OCT4, SOX2, KLF4, cMYC, NANOG и LIN28 (OSKMNL) (1:1:1:1:1:1), с 0,08 мкг каждого из B18R, E3 и K3 (EKB), и 0,17 мкг смеси миРНК, состоящей из миРНК 302a-d и 367 (1:1:1:1:1:1). РНК-конструкции, таким образом, отличались только по их 3'-UTR, состоящей из тандемного повтора 3'-UTR β-глобина человека (2hBg), F-I-элемента (FI) или I-F-элемента (IF). Клетки культивировали в среде для культивирования эмбриональных стволовых (hES) клеток, и осуществляли процедуры липофекции с применением RNAiMAX в соответствии с инструкциями производителя. Начиная с дня 9 наблюдали образование колоний, а в день 11 проводили анализ колоний (см. фиг. 14A касательно информации по хронологии). Сформировавшиеся колонии в день 11 окрашивали по щелочной фосфатазе (AP) с применением набора AP Staining Kit. Обзор репрезентативных результатов окрашивания представлен на фиг. 14B. Становится очевидно, что включение FI-элемента приводит к образованию больших количеств AP-положительных колоний (темные). Колонии, окрашенные по AP, считали, и результаты из обзора были подтверждены. По сравнению с ранее применяемой 2hBg-UTR, в случае, когда клетки подвергали липофекции 3 раза, замена на FI-UTR приводит к 3-4-кратному увеличению числа колоний. Замена на IF-UTR приводит к 2-кратному увеличению. В случае четырех трансфекций такие эффекты выражены меньше. В данном случае для IF-UTR никакого улучшения не наблюдали. С одной стороны, по-видимому, при четырех трансфекциях происходит насыщение процесса, а с другой стороны, подсчет колоний в данном случае в какой-то степени является необъективным ввиду чрезмерного разрастания колоний (см. фиг. 14C). Морфология колоний полученных колоний клеток iPS при применении РНК, содержащей FI-UTR, была подобна таковой у клеток hES, с плотно группирующимися небольшими клетками в отдельных колониях и четко выраженными краями (фиг. 14D). Такие колонии могли быть положительно окрашены по AP (фиг. 14E) и маркера клеточной поверхности TRA-1-60 hES (фиг. 14F). Окрашивание живых клеток по TRA-1-60 осуществляли с применением антитела к TRA-1-60 Stain-Alive (Stemgent) в соответствии с инструкциями производителя. Репрезентативные изображения колоний приведены. Для дополнительной оценки плюрипотентности колоний клетки осаждали, выделяли общую РНК и с помощью qRT-PCR количественно определяли экспрессию мРНК маркеров hES OCT4 (эндогенный), NANOG (эндогенный), LIN28 (эндогенный), TERT и REX1. Экспрессию мРНК нормализовали к таковой для HPRT, и она представлена как кратность повышения в сравнении с уровнями транскриптов исходных клеток. Результаты анализа колоний после 3 липофекций представлены на фиг. 14G. Все проанализированные маркеры были экспрессированы на высоком уровне по сравнению с исходными клетками, что указывало на плюрипотентность перепрограммированных клеток. Превосходство содержащей FI синтетической мРНК было подтверждено более высоким уровнем экспрессии эндогенных маркеров по сравнению с перепрограммированием с помощью содержащих 2hBg и IF мРНК.
Из этих результатов видно, что замена 2hBg-UTR на FI-UTR обеспечивает более быструю и эффективную методику перепрограммирования на основе использования РНК. Возможно, это обусловлено более продолжительной и высокой экспрессией транскрипционных факторов перепрограммирования вследствие осуществления замены на FI-элемент. Таким образом, ориентация FI-элемента, по-видимому, имеет важное значение, поскольку в случае IF-конструкций положительный эффект не наблюдали. Успешное перепрограммирование клеток с помощью содержащей FI мРНК подтверждали по морфологии, подобной таковой у hES, AP-активности и экспрессии поверхностных и эндогенных маркеров клеток hES, полученных колоний клеток iPS.
Таблицы
Табл. 1. Время полужизни мРНК в часах (ч), рассчитанное исходя из данных экспериментов по отслеживанию прогресса отбора с использованием ПЦР с обратной транскрипцией в режиме реального времени (RT-PCR). мРНК количественно определяли через 8, 24 и 48 часов после электропорации. В эксперименте I (слева) каждый образец анализировали только один раз. Соответственно, стандартное отклонение не приведено.
Табл. 2. Обзор 7 основных групп со связывающими областями (BR) в пределах 3'-UTR последовательности, в отношении которой проводили поиск BLAST. Приведены сокращение группы, число клонов, идентифицированных для группы (номер), геномное происхождение с соответствующим сокращением (сокр.), код NCBI и положение в последовательности по отношению к кодирующей области. В соответствии с NextBio экспрессия всех последовательностей в hiDC была повышена.
Табл. 3. Значения, рассчитанные относительно полученной авторами настоящего изобретения эталонной 2hBg для времени полужизни и экспрессии общего белка в динамике. Приведены название группы и соответствующий ген.
Табл. 4. Условия ПЦР для амплификации библиотеки и последующих раундов отбора.
Табл. 5. Реакция IVT-транскрипции с T7
Табл. 6. Комбинации, клонированные и подлежащие сравнению с полученной авторами настоящего изобретения эталонной 2hBg (нижний правый угол). Одиночные элементы, клонированные дважды, выделены.
Табл. 7. Результат по FCGRT (группа B), клонированному в качестве одиночного или вышерасположенного элемента в комбинации с одной из последовательностей из другой группы, в качестве нижерасположенного элемента. Жирным выделены значения > 1,0. Значения приведены относительно 2hBg.
Табл. 8. Результат по LSP1 (группа D), клонированному в качестве одиночного или вышерасположенного элемента в комбинации с одной из последовательностей из другой группы, в качестве нижерасположенного элемента. Жирным выделены значения > 1,0. Значения приведены относительно 2hBg.
Табл. 9. Результат по CCL22 (группа E), клонированному в качестве одиночного или вышерасположенного элемента в комбинации с одной из последовательностей из другой группы, в качестве нижерасположенного элемента. Жирным выделены значения > 1,0. Значения приведены относительно 2hBg.
Табл. 10. Результат по AES (группа F), клонированному в качестве одиночного или вышерасположенного элемента в комбинации с одной из последовательностей из другой группы, в качестве нижерасположенного элемента. Жирным выделены значения > 1,0. Значения приведены относительно 2hBg.
Табл. 11. Результат по PLD3 (группа G), клонированному в качестве одиночного или вышерасположенного элемента в комбинации с одной из последовательностей из другой группы, в качестве нижерасположенного элемента. Жирным выделены значения > 1,0. Значения приведены относительно 2hBg.
Табл. 12. Результат по mtRNR1 (группа I), клонированному в качестве одиночного или вышерасположенного элемента в комбинации с одной из последовательностей из другой группы, в качестве нижерасположенного элемента. Жирным выделены значения. Значения приведены относительно 2hBg.
Табл. 13. Результат по HLA-DRB4 (группа J), клонированному в качестве одиночного или вышерасположенного элемента в комбинации с одной из последовательностей из другой группы, в качестве нижерасположенного элемента. Жирным выделены значения > 1,0. Значения приведены относительно 2hBg.
Табл. 14. Репрезентативные результаты, полученные при применении luc2mut в качестве репортерного гена и новых отобранных 3'-UTR после введения в hiDC путем электропорации. Активность люциферазы измеряли за период времени 96 ч. Значения приведены относительно 2hBg.
Табл. 15. Условия электропорации
В таблице приведено подробное описание протокола электропорации для всех применяемых типов клеток. Количество клеток, указанное в ячейке «подсчет клеток», смешивали с количеством РНК, указанным либо в мкг, либо в пмоль, либо в кюветах для электропорации, либо в 96-луночных планшетах для электропорации (как указано в ячейке «формат») в среде X-VIVO15 (Lonza). Электропорацию осуществляли путем прикладывания импульса с указанной частотой и напряжением, указанным в ячейке «V». Затем суспензию клеток разбавляли в среде для выращивания и распределяли в 96-луночные планшеты при плотности, указанной в ячейке «клетки/момент времени».
Табл. 16
Показатели времени полужизни и общий белок для содержащей FI-элемент немодифицированной и модифицированной мРНК после электропорации и немодифицированной РНК после липофекции по сравнению с таковыми, содержащими 2hBgUTR.
Плазмиды, кодирующие ген люциферазы светлячка, содержащий либо FI, либо 2hBg в качестве 3'-UTR, подвергали линеаризации ниже поли(dA:dT) с помощью фермента рестрикции класса IIS, за счет чего получали матрицу без дополнительного нуклеотида после поли(dA:dT). Линеаризованную плазмидную ДНК очищали с применением гранул с карбоксильными группами (Invitrogen), количественно определяли спектрофотометрическим путем и подвергали in vitro транскрипции. Для in vitro транскрипции полученную в лаборатории РНК-полимеразу T7, дополнительно с ингибиторами РНКазы, и пирофосфатазу применяли с 7,5 мМ NTP в буфере с 125 мМ Hepes, pH 8,35, 34 мМ MgOAc2, 10 мМ DTT и 2 мМ спермидином. Для эффективного кэпирования РНК к реакционной смеси добавляли 6 мМ β-S-ARCA(D2) и исходную концентрацию GTP снижали до 1,5 мМ, которую доводили до 7,5 мМ в периодическом процессе с подпиткой в течение 2,5 ч при 37°C. РНК очищали на гранулах с карбоксильными группами (Invitrogen) и оценивали концентрацию и качество РНК с помощью спектрофотометрии и анализа на 2100 Bionanalyzer (Agilent).
В A) показано, что в нескольких клеточных линиях человека и мыши показатели времени полужизни немодифицированных мРНК, содержащих FI-элемент, были более высокими или сопоставимыми с таковыми, содержащими 3'-UTR 2hBg. Количество фибробластов человека (HFF), CD8+ и CD4+ T-клеток, эмбриональных фибробластов мыши (MEF), клеток миобластомы (C2C12) и DC мыши, приведенное в табл. 15, смешивали с соответствующим количеством РНК (табл. 15) в среде X-VIVO15 (Lonza) и подвергали электропорации. Указанное число клеток высевали в 96-луночные планшеты в 100 мкл соответствующей среды для выращивания с добавками. Через 2, 6, 24, 48, 72 и 96 часов после посева путем добавления люциферина (Promega) на флуоресцентном ридере (TECAN) определяли показатели активности люциферазы светлячка.
В B) показано, что в различных клеточных линиях человека и мыши показатели времени полужизни m1Y-модифицированных мРНК, содержащих FI-элемент, были более высокими или сопоставимыми с таковыми, содержащими 3'-UTR 2hBg. Количество незрелых дендритных клеток человека (iDC), фибробластов человека (HFF), CD8+ и CD4+ T-клеток, эмбриональных фибробластов мыши (MEF), клеток миобластомы (C2C12) и DC мыши, приведенное в табл. 15, смешивали с соответствующим количеством m1Y-модифицированной РНК (табл. 15) в среде X-VIVO15 (Lonza) и подвергали электропорации. Указанное число клеток высевали в 96-луночные планшеты в 100 мкл соответствующей среды для выращивания с добавками. Через 2, 6, 24, 48, 72 и 96 часов после посева путем добавления люциферина (Promega) на флуоресцентном ридере (TECAN) определяли показатели активности люциферазы светлячка.
В C) показано, что в различных клеточных линиях показатели времени полужизни немодифицированных мРНК, содержащих FI-элемент, были более высокими или сопоставимыми с таковыми, содержащими 3'-UTR 2hBg, также и в том случае, когда РНК трансфицировали посредством липофекции. 50 нг РНК инкубировали в течение 15-30 мин. с 0,2 мкл RNAiMAX и вносили в 96-луночные планшеты с клетками 1E04 HFF, MEF или C2C12. Через 3, 6, 12, 24, 48, 72 и 96 часов путем добавления люциферина (Promega) на флуоресцентном ридере (TECAN) измеряли уровни люциферазы.
Табл. 17. 10 мкг кодирующей люциферазу светлячка РНК, содержащей в качестве 3'-UTR либо FI-элемент, либо вариации FI-элемента с определенной гомологией с исходной FI-последовательностью, вводили путем электропорации в hiDC в формате с 96-луночным планшетом. Экспрессию люциферазы отслеживали в динамике через 3, 6, 24, 48 и 72 ч, и из полученной кривой экспрессии рассчитывали время полужизни мРНК и количество общего белка, транслированного с РНК.
Табл. 18. 10 мкг кодирующей люциферазу светлячка РНК, содержащей в качестве 3'-UTR либо FI-элемент, либо вариации FI-элемента, предусматривающие мутации сохранения и разрушения структуры, и вариации с определенной гомологией с исходной FI-последовательностью, вводили путем электропорации в hiDC в формате с 96-луночным планшетом. Экспрессию люциферазы отслеживали в динамике через 3, 6, 24, 48 и 72 ч, и из полученной кривой экспрессии рассчитывали время полужизни мРНК и количество общего белка.
Описанные в настоящем документе последовательности являются следующими:
Группа B
>Rn5-2pl-A4_For2
CAUCCUGCUGCUGCUGCUGCUGCUGCUGCGGGUCUUCCUGGAAUCUGACCAUUCGUUGUC
UGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUG
AGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUG
CCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn5-2pl-A3_For2
GCUGCUGCUGCUGCUGCUGCUGCGGGUCUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAU
GCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUG
ACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUG
CUGAUCCAUUGCCGGUGUGACC
>Rn5C5_For2
UUCCUGCUGCUGCUGCUGCUGCUGCUGCUGCGGGUCUUCCUAGAAUCUGACCAUUCGUUG
UCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCA
UGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCU
UGCCGCUGCUGAUCCAUUGCCGGUGAGACC
>Rn5E6_For2
UGCUGCUGCUGCUGCGGGUCUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCC
UCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCA
CUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCA
UUGCCGGCGGACA
>Rn6-1WoC3_For2
GCUGCUGCGGGUCUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAA
GACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCU
GCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGG
CGUACC
>Rn6-1WoB12_For2
CUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUG
CUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCAC
UGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGCGGACC
>Rn6-1WoB1_For2
UCCUGCUGCUGCUGCUGCUGCUGCUGCGGGUCUUCCUGGAAUCUGACCAUUCGUUGUCUG
CUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAG
ACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCC
GCUGCUGAUCCAUUGCCGGUGGGACC
>Rn6-1WoF3_For2
CUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGC
CUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUAGAAC
C
>Rn6-1Wo_H11_b
UCCUGCUGCUGCUGCUGCUGCUGCUGCGGGUCUUCCUGGAAUCUGACCAUUUGUUGUCUG
CUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAG
ACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCC
GCUGCUGAUCCAUUGCCGGUGGGACC
>Rn6-2WoG8_b
GCUGCUGCUGCUGCUGCGGGUCUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGU
CCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCC
CACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUC
CAUUGCCGGUGUGACC
>Rn5-2pl-B3_For2
UCUGGCCUCACUGAGUCUGAAGAGCUGUUAACUACCAUGGCCAGUCCUCCCUGAGUCUGA
CCAUCUUCCAUCCUGCUGCUGCUGCUGCUGCUGCUGCGGGUCUUCCUGGAAUCUGACCAU
UCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCG
GGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCC
CCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn5_F5_b
UCUGGCCUCACUGAGUCUGAAGAGCUGUUAACUACCAUGGCCAGUCCUCCCUGAGUCUGA
CCAUCUUCCAUCCUGCUGCUGCUGCUGCUGCUGCUGCGGGUCUUCCUGGAAUCUGACCAU
UCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCG
GGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCC
CCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn5B8_For2
CUACCAUGGCCAGUCCUCCCUGAGUCUGACCAUCUUCCAUCCUGCUGCUGCUGCUGCUGC
UGCUGCGGGUCUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGA
CUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGC
CUGCCUUUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUG
UGACC
>Rn6-1WoH9_For2
GUCCUCCCUGAGUCUGACCAUCUUCCAUCCUGCUGCUGCUGCUGCUGCUGCUGCGGGUCU
UCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGC
UGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCC
ACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUCUGACA
>Rn6-2WoC11_For2
GUCCUCCCUGAGUCUGACCAUCUUCCAUCCUGCUGCUGCUGCUGCUGCUGCUGCGGGUCU
UCCUGGAAUCUGACCAUUUGUUGUCUGCUAUGCCCCUCCUCACCAAGACUGACUGCCUGC
UGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUUCCCCA
CUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn5_C3_b
CCAUCCUGCUGCUGCUGCUGCUGCUGCUGCGGGUCUUCCUGGAAUCUGACCAUUCGUUGU
CUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUGCUACUGCCCGGGCCCAUG
AGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUG
CCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-2WoH5_For2
GCCAGUCCUCCCUGAGUCUGACCAUCUUCCAUCCUGCUGCUGCUGCUGCUGCUGCUGCGG
GUCUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGC
CUGCUGAUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCU
CCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-96hE12_For2
UGCCUUCCGUCUCCUGCUGCUUCUGGCCUCACUGAGUCUGAAGAGCUGUUAACUACCAUG
GCCAGUCCUCCCUGAGUCUGACCAUCUUCCAUCCUGCUGCUGCUGCUGCUGCUGCUGCGG
GUCUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGC
CUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCU
CCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-96h-2pl-E9_F
GGCCAGUCCUCCCUGAGUCUGACCAUCUUCCAUCCUGCUGCUGCUGCUGCUGCUGCUGCG
GGUCUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUG
CCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUC
UCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-96h-2pl-H10_
GGCCAGUCCUCCCUGAGUCUGACCAUCUUCCAUCCUGCUGCUGCUGCUGCUGCUGCUGCG
GGUCUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUG
CCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUC
UCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-1WoB11_For2
UGACCAUCUUCCAUCCUGCUGCUGCUGCUGCUGCUGCUGCGGGUCUUCCUGGAAUCUGAC
CAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGC
CCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACA
GCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-1WoF7_For2
CCAGUCCUCCCUGAGUCUGACCAUCUUCCAUCCUGCUGCUGCUGCUGCUGCUGCUGCGGG
UCUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCC
UGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCUCUGCUCUGCCUGCCUCUC
CCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCUGUGUGACCA
>Rn6-1WoA7_For2
UGACCAUCUUCCAUCCUGCUGCUGCUGCUGCUGCGGGUCUUCCUGGAAUCUGACCAUUCG
UUGUCUGCUAUGCCCGUCCUUACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGC
CCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCG
CCUUGUCGCUGCUGAUCCAUUGCCGGUGUGACAC
>Rn6-2WoD11_b
GACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCU
GCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGG
UGUGACCC
>Rn6-2WoG3_For2
CUCCCUGAGUCUGACCAUCUUCCAUCCUGCUGCUGCUGCUGCUGCUGCUGCGGGUCUUCC
UGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGC
UUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACU
GCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-2WoC2_For2
UUCCAUCCUGCUGCUGCUGCUGCUGCUGCUGCGGGUCUUCCUGGAAUCUGACCAUUCGUU
GUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCC
AUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCC
UUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-1WoD6_For2
UGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACU
GACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCU
GCUGAUCCAUUGCCGGUGUGACC
>Rn6-1WoD10_For2
UGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACU
GACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCU
GCUGAUCCAUUGCCGGUGUGACC
>Rn6-2WoG5_For2
GCGGGUCUUCCUGGAAUCUGAACAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGA
CUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGC
CUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGAC
C
>Rn6-96h-2pl-G8_F
GUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGG
CCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCC
GCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-1WoE7_For2
CAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGC
UCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGC
CGGUGUGACC
>Rn6-1Wo_A12_b
CCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGACCCAUGAGACUGACUUCC
CACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUC
CAUUGCCGGUGUGACUGC
>Rn6-1WoG11_For2
CUUCCAUCCUGCUGCUGCUGCUGCUGCUGCUGCGGGUCUUCCUGGAAUCUGACCAUUCGU
UGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCC
CAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGC
CUUGCCGCUGCUGAUCCAUUGCCGGUGUGACCCC
>Rn6-1WoH5_For2
CUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCC
ACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCC
AUUGCCGGUGUGACC
>Rn6-1WoH4_For2
AAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCU
CUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCC
GGUGUGACC
>Rn6-2WoB4_For2
CUCCAGCUCGCUUCCAUUUGCUUGCAGAAGUUCUCGCUGUGCUCACGAAGCUUGCGCUCC
UUGGAGGCCUCAGCAACAGCAUCAUCAAGCUGAGCUUCCAGCUCUUUCCUGAGCUUCUCA
GCUCUCCGCAUUUCCUGCCGCAUGGCGUCCACCUUCUGCGUGGCCACCUCCAUCUCCUCC
UCCUUGUCUCGCAGCUGCCGGGACACCUUCUGCGCUAAGAUGGGAUACGGCAUUGAGGGA
UCAAUGUGUAAGGAUCCGAUCUGCUUCUGGCCUCACUGAGUCUGAAGAGCUGUUAACUAC
CAUGGCCAGUCCUCCCUGAGUCUGACCAUCUUCCAUCCUGCUGCUGCUGCUGCUGCUGCU
GCGGGUCUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGUCCGUCCUCACCAAGACUGA
CUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGC
CUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGAC
C
>Rn6-96h-2pl-A5_F
CUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGC
CUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUG
UGACC
>Rn6-1WoC8_For2
CCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUG
CUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUG
ACGGUGUGACC
>Rn5D1_For2
UAACUACCAUGGCCAGUCCUCCCUGAGUCUGACCAUCUUCCAUCCUGCUGCUGCUGCUGC
UGCUGCGGGUCUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGA
CUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGC
CUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGUUGAUCCAUUGCCGGUG
UGACC
>Rn6-2WoG10_For2
CCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUG
CUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGUUGAUCCAUUG
UCGGUGUGACC
>Rn6-1Wo_E4_b
CCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUG
CUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUG
CCGGUGUGACC
>Rn6-2WoF3_For2
CUGCUAUGCCUGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAU
GAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCAAUGCACUGGCACAGCCCCGCCUU
GCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-96h-2pl-B10
CUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAU
GAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUU
GCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-96h-2pl-C10
GCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGA
GACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGC
CGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-1WoB6_For2
UCUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCC
UGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUC
CCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-96h-2pl-D6_F
CUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUG
CUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCAC
UGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-96h-2pl-E6_F
CUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUG
CUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCAC
UGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-2WoF10_For2
GGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCU
UUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUG
CACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>Rn6-1WoG9_For2
CCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUG
CUCUGCCUGCCUCUCCCCACUGCACUGGCAUAGCCCCGCCUUGCCGCUGCUGAUCCAUUG
CCGGUGUGACC
>Rn6-96hC12_For2
CUUCCUGGAAUCUGACCAUUCGUUGUCUGCUAUGCCCGUCCUCACCAAGACUGACUGCCU
GCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCC
CCACUGCACUGGCAUAGCCCCGCCUUGCCGCUGCUGAUCCAUUUCCGGUGUGACC
Группа D
>Rn6-1WoF2_For2
CAGACACCCGCCCCCCGGCCCUGGCUAAGAAUUUGCUUCCUGUUGCCAGCAUGACCUACC
CUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUC
UGCCCUUCCACUCUCUGACC
>Rn6-2WoD8_For2
UUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUG
ACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUA
GCCUCUCUGCCCUUCCACUCUCUGACCCC
>Rn6-1WoD5_For2
CUCGCUUCCUGGGUCUGCAGGUCCAGCCGGCUGGCACCCUCCAUGUACCCAGGGGAGAUU
CCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGAC
CUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGC
CUCUCUGCCCUUCCACUCUCUGACCACCGCCC
>Rn5-2pl-D3_For2
UCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGA
CCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAG
CUUCUCUGCCCUUCCACUCUCUGG
>Rn6-2WoA8_For2
CGCUUCCUGGGUCUGCAGGUCCAGCCGGCUGGCACCCUCCACGUACCCAGGGGAGAUUCC
AGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCU
ACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCU
CUCUGCCCUUCCACUCUCUGACCACCG
>Rn6-2WoD7_For2
CAUGUACCCAGGGGAGAUUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGC
UUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUU
UGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCG
>Rn6-2WoB8_For2
CUCGCUUCCUGGGUCUGCAGGUCCAGCCGGCUGGCACCCUCCAUGUACCCAGGGGAGAUU
CCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGAC
CUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGC
CUCUCUGCCCCCCCGAU
>Rn6-96h-2pl-H6_F
CCCAGCUCCCUAGGCGUCCCAUCUCGCUUCCUGGGUCUGCAGGUCCAGCCGGCUGGCACC
CUCCAUGUACCCAGGGGAGAUUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGU
UGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCC
UUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUUUGACCCCCAUCUUA
>Rn6-96h-2pl-F10
GGCCACCGGGCAUGGGAAGUAUGAGAAGGUGCUUGUGGAAGGGGGCCCGGCUCCCUAGGC
GUCCCAUCUCGCUUCCUGGGUCUGCAGGUCCAGCCGGCUGGCACCCUCCAUGUACCCAGG
GAGAUUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAG
CAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCC
UUUAGCCUCUCUGCCCUUCCACUCUCUGACCCC
>Rn5H3_For2
UGUACCCAGGGGAGAUUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUU
CCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUG
CUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCACCACCCCC
>Rn5G7_For2
CCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAU
GCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCU
CUGACCACAGCCCC
>Rn6-1WoG5_For2
CCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUG
AUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACU
CUCUGACCACCGCCCCCGCC
>Rn6-1WoA8_For2
CCGGCUGGCACCCUCCAUGUACCCAGGGGAGAUUCCAGCCAGACACCCGCCCCCCGGCCC
UGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCC
GCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCA
CCGCCCCC
>Rn6-96h_D3_b
GCCGGCUGGCACCCUCCAUGUACCCAGGGGAGAUUCCAGCCAGACACCCGCCCCCCGGCC
CUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUAGCCUCUUUGAUGCCAUC
CGCUGCCACCUCCUUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGAC
CACCGCCCCC
>Rn6-96hC11_For2
UCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGA
CCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAG
CCUCUCUGCCCUUCCACUCUCUGACCACCACCCC
>Rn5H1_For2
GCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUA
CCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUC
UCUGCCCUUCCACUCUCUGACCCCCC
>Rn6-1WoG2_For2
UCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGA
CCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAG
CCUCUCUGCCCUUCCACUCUCUGACCCCCC
>Rn6-1WoG7_For2
CGGCUCCCUAGGCGUCCCAUCUCGCUUCCUGGGUCUGCAGGUCCAGCCGGCUGGCACCCU
CCAUGUACCCAGGGGAGAUUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUG
CUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUU
UUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCACUGCCCC
>Rn6-96hB11_For2
UGCAGGUCCAGCCGGCUGGCACCCUCCAUGUACCCAGGGGAGAUUCCAGCCAGACACCCA
CCCCCCGGCCCUGGCUAAGAAGUUGCUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUU
GAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCAC
UCUCUGACCACUACCCC
>Rn6-2WoF8_For2
UUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUG
ACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUA
GCCUCUCUGCCCUUCCACUCUCUGACCACUGCCCC
>Rn6-96h_A9_b
CCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCC
UCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCU
UCCACUCUCUGACC
>Rn6-1WoH3_For2
CAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACC
UACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCC
UCUCUGCCCUUCCACUCUCUGAACACC
Группа E
>Rn6-2WoE2_For2
GAGCCUACUCUGAUGACCGUGGCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCC
CUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUC
CUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCUGACCCCUC
GAACCCAUCCUA
>Rn6-1WoD3_For2
GAGCCUACUCUGAUGACCGUGGCCUUGGCUCCUCCAGGAAGGCUCAGGCGCCCUACCUCC
CUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUC
CAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCUGAC
CCCUCUAACCC
>Rn6-2WoG7_For2
GCCUACUCUGAUGACCGUGGCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCU
GCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCA
UCCCUGUGGCUGUCACCCUUGGUCCACUGCCAUCUCCCCCCC
>Rn6-2WoH2_For2
GAGCCUACUCUGAUGACCGUGGCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCC
CUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUC
CAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUUACUGCCAUCUCCCCCCUGAC
CCC
>Rn6-2WoC1_For2
GAAGAGCCUACUCUGAUGACCGUGGCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACC
UCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAU
CUCCAUCCCUGUGGCUGUCAUCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCU
GACCCCUCUAACCCAUCCUCUGCCUCCCUCCCUGCAGUCAGAGGGUCCUGUUCCCAACCA
>Rn6-1Wo_C12_b
UGUGGCCUUGGCUCCUCCAGGAAGGCUAAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCU
CCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCA
CCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCUGACCCC
>Rn6-1WoE12_For2
GCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCC
GCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCU
UGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCC
>Rn6-2WoF5_For2
AGAGCCUACUCUGAUGACCGUGGCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUC
CCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCU
CCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCUGA
CCCCUCUAACCCAUCCUCUGCCUCCCUCCCUGCAGUCAGAGGGUCCUGUUCCCAUCAGCG
AUUCCCCUGCUUAAACCCUUCCAUGACUCCCCACUGCCCUAAGCUGAGGUCAGUCUCCCA
AGCCUGACAU
>Rn5-2pl-H3_For2
UAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCU
GUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCUGACCCCUCUA
ACCCAUCCUCUGCCUCCCUCCCUGCAGUCAGAGGGUCCUGUUCCCAUCAGCGAUUCCCCU
GCUUAAACCCUUCCAUGACAGCCC
>Rn6-2WoA3_For2
UCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCAC
UGCCAUCUCCCCCCUGACCCCUCUAACCCAUCCUCUGCCUCCCUCCCUGCAGUCAGAGGG
UCCUGUUCCCAUCAGCGAUUCCCCUGCUUAAGCCCUUCCAUGACUCCCC
>Rn6-96hF12_For2
CUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGA
UCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCC
UGACCCCUCUAACCCAUCCUCUGCCUCCCUCCCUGCAGUCAGAGGGUCCUGUUCCCAUCA
GCGAUUCCCCUGCUUAAACCCUUCCAUGACUCCCCAA
>Rn6-96hE11_For2
GCCUACUCUGAUGACCGUGGCCUUGGGUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCU
GCCAUUAUAGCUGCUCCCCGCCAGAAGUCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCA
UCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCUGACCC
CUCUAACCCAUCCUCUGCCUCCCUCCCUGCAGUCAGAGGGUCCUGUUCCCAUCAGCGAUU
CCCCUGCUUAAACCCUUCCAUGACUCCCCUCU
>Rn6-96h-2pl-A11
CUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCC
CUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCC
CCCCUGACCCC
Группа F
>Rn6-1WoB5_For2
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUC
CCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCU
AGUUCCAGACACCUCC
>Rn6-2WoE11_a
CCGGCCCUUCCCCCGUUUUGAACAUGUGUAACCGACAGUCUGCCUGGGCCACAGCCCUCU
CACCCUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGCACCCCGAG
UCUCCCCCGACCCCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUC
UGCUAGUUCCAGACACCCCCGCG
>Rn6-96h_E3_b
CCUUCCCCCGUUUUGAACAUGUGUAACCGACAGUCUGCCUGGGCCACAGCCCUCUCACCC
UGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGCCCUGGGCACCCCGAGUCUCC
CCCGACCCCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUA
GUUCCAGACACCUCCAC
>Rn6-96h-2pl-B6_F
UCUGCCUGGGCCACAGCCCUCUCACCCUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCU
UUCCCGUCCUGGGCACCCCGAGUCUCCCCCGACCCCGGGUCCCAGGUAUGCUCCCACCUC
CACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCACGCCCACCUGGUCCUCU
CCCAUCGCCCACAAAAGGGGGGGCACGAGGGACGAGCUUAGCUGAGCUGGGAGGAGCAGG
GUGAGGGUGGGCGACCCAGGAUUCCCCCACCCC
Группа G
>Rn5_D5_b
UGACACCUCAGCUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUG
GGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUC
CCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCC
CACCUCUACCUCCACCCCA
>Rn5B2_For2
CUCAGCUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGG
CCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCA
CCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUC
UACCUCCACUCCC
>Rn5G3_For2
UCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCU
CUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAG
GCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCC
>Rn6-96hF11_For2
GGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGG
UCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCU
CUCCCCUGCUCUCCCACCUCUACCUCCACCCCC
>Rn6-96h-2pl-D8_F
GCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCC
GCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCU
CCGCCCCC
>Rn5C4_For2
CGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCC
CAUUGUGGCUCCUUAGGCUCUCUCCCCUGCUCUCCCACCUUUACCUCCACCCCUAC
>Rn6-2WoD3_For2
CUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAG
GCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCC
GCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCU
CCACCCCCAC
>Rn6-96h-2pl-C6_F
CUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAG
GCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCC
GCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCU
CCACCCCCAAC
>Rn6-96h-2pl-C7_F
CUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAG
GCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCC
GCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCU
CCACCCCCAAC
>Rn6-96h-2pl-F8_F
CGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCU
GGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUG
UCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCA
CACC
>Rn6-96hH9_For2
UCCUGAGGGACUGGGACUCCCCUUACAGCCAUGACCUUGACACCUCAGCUGACAGCGUGG
GCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCC
CCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGC
CCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACGCCCAC
>Rn5_F10_b
CUGAGGGACUGGGACUCCCCUUACAGCCAUGACCUUGACACCUCAGCUGACAGCGUGGGC
AACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCC
CGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCC
CAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACACCU
>Rn6-2WoF11_For2
UGCGGAGCCAGCUGGAGGCCAUUUUCCUGAGGGACUGGGACUCCCCUUACAGCCAUGACC
UUGACACCUCAGCUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGU
GGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGU
CCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUC
CCACCUCUACCUCCACCC
>Rn6-1WoA9_For2
CUGGAGGCCAUUUUCCUGAGGGACUGGGACUCCCCUUACAGCCAUGACCUUGACACCUCA
GCUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAA
GGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCC
CGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACC
UCCCCCCAC
>Rn6-1WoF9_For2
CUGGGACUCCCCUUACAGCCAUGACCUUGACACCUCAGCUGACAGCGUGGGCAACGCCUG
CCGCCUGCUCUGAGGCCCAAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACC
CAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGC
CUCCUUAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCC
Группа I
>Rn5_A7_b
GCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGUAAACAGCAGUGAUU
AACUUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUU
CGUGCCAGCCACC
>Rn5_B6_b
CUUUCUAUUAGCUCUUAGUAAGAUUACACAUGCAAGCAUCCCCGUUCCAGUGAGUUCACC
CUCUAAAUCACCACGAUAAAAAGGGACAAGCAUCAAGCACGCAGCAAUGCAGCUCAAAAC
GCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAA
AGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCUACC
>Rn5D4_For2
GUUCCAGUGAGUUCACCCUCUAAAUCACCACGAUCAAAAGGGACAAGCAUCAAGCACGCA
GCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAAC
CUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGU
GCCAGCCACC
>Rn5D2_For2
AAAGGGACAAGCAUCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCC
CCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACU
AACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACC
>Rn6-1Wo_D7_b
UCAAAAGGGACAAGCAUCAAGCACGCAACAAUGCAGCUCAAAAACGCUUAGCCUAGCCAC
ACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUA
CACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACC
>Rn6-96h-2pl-A9_F
UACACAUGCAAGCAUCCCCGUUCCAGUGAGUUCACCCUCUAAAUCACCACGAUCAAAAGG
GACAAGCAUCAAGCACGCAGCAAUGCAGCUCAAAAACGCUUAGCCUAGCCACACCCCCAC
GGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACC
CCAGGGUUGGUCAAUUUCGUGCCAGCCACC
>Rn6-2WoH3_For2
CAUCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAAC
AGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGU
UGGUCAAUUUCGUGCCAACCACC
>Rn6-96hG11_For2
AAAGGGACAAGCAUCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCC
CCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACU
AACCCCAGGGUUGGUCAAUUUCGUGCCAACCACC
>Rn5E1_For2
CAAGCACGCAACAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGC
AGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGG
UCAAUUUCGUGCCAACCACC
>Rn6-1WoA11_For2
CAUCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCAUGGGAAAC
AGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGU
UGGUCAAUUUCGUGCCAGCUCACC
>Rn6-2WoE7_For2
CAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGC
AGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGG
UCAAUUUCGUGCCAGCCACACC
>Rn6-96h-2pl-B5_F
CAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGC
AGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGG
UCAAUUUCGUGCCAGCCACC
>Rn5H2_For2
CACGAUCAAAAGGGACAAGCAUCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAG
CCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAA
GCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACC
>Rn6-1WoF11_For2
UAAAUCACCACGAUCAAAAGGGACAAGCAUCAAGCACGCAGCAAUGCAGCUCAAAACGCU
UAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGU
UUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACC
>Rn6-2WoB11_For2
AGCCUUUCUAUUAGCUCUUAGUAAGAUUACACAUGCAAGCAUCCCCGUUCCAGUGAGUUC
ACCCUCUAAAUCACCACGAUCAAAAGGGACAAGCAUCAAGCACGCAGCAAUGCAGCUCAA
AACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAAC
GAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACC
>Rn6-1WoA3_For2
GGGACAAGCAUCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCA
CGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAAC
CCCAGGGUUGGUCAAUUUCGUGCCAGCCACC
>Rn6-1Wo_D2_b
GGGACAAGCAUCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCA
CGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAAC
CCCAGGGUUGGUCAAUUUCGUGCCAGCCACC
Группа J
>Rn5A1_For2
UUCUGCCCCAGCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGA
GAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCC
CUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCA
UCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUC
CUUCUGUGCCACU
>Rn5B1_For2
UUCUGCCCCAGCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGA
GAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCC
CUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCA
UCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUC
CUUCUGUGCCACU
>Rn5_A10_b
CCCCAGCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGAC
CUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUG
GCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUC
CAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCCU
GUGCCACAAA
>Rn5_G1_b
CCCCAGCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGAC
CUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUG
GCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUC
CAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCCU
GUGCCACAAA
>Rn6-1WoF5_For2
CUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCU
CCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCC
UCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUU
UUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCC
>Rn6-2WoA5_For2
UGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAGGAGAGACCUUUCUCCG
GACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCA
GCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUG
UGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCA
>Rn6-2WoA7_For2
GUGAAGAUGACCACAUUCAAGGAAGAACCUUCUGCCCCAGCUUUGCAGGAUGAAACACUU
CCCCGCUUGGCUCUCCUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGG
UUCAGCAGCUCUGCAGAAAAUGUCCUCCCUUGUGGCUGCCUCAGCUCGUACCUUUGGCCU
GAAGUCCCAGCAUUAAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAA
UGCUUCCUGCCUCCCAUGCAUCUGUACUCCUGCUGUGCCA
>Rn6-2WoG2_For2
UCCACAAGAGAGACCUUUCUCCGGACCUGGCUGCUACUGGUUCAGCAGCUCUGCAGAAAA
UGUCCUCCCUUGUGGCUGCCUCAGCUCGUACCUUUGGCCUGAAGUCCCAGCAUUAAUGGC
AGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAAUGCUUCCUGCCUCCCAUGCA
UCUGUACUCCUGCUGUGCCACAAACAC
>Rn6-2WoH10_For2
UCCACAAGAGAGACCUUUCUCCGGACCUGGCUGCUACUGGUUCAGCAGCUCUGCAGAAAA
UGUCCUCCCUUGUGGCUGCCUCAGCUCGUACCUUUGGCCUGAAGUCCCAGCAUUAAUGGC
AGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAAUGCUUCCUGCCUCCCAUGCA
UCUGUACUCCUGCUGUGCCACAAACAC
>Rn6-96h-2pl-G7_F
GCUACUGGUUCAGCAGCUCUGCAGAAAAUGUCCUCCCUUGUGGCUGCCUCAGCUCGUACC
UUUGGCCUGAAGUCCCAGCAUUAAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCU
UUACCUAAUGCUUCCUGCCUCCCAUGCAUCUGUACUCCUGCGU
>Rn5-2pl-B2_For2
AGAACCUUCUGCCCCAGCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCC
ACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAGCUCUGCAGAAAAUGU
CCUCCCUUGUGGCUGCCUCAGCUCGUACCUUUGGCCUGAAGUCCCAGCAUUAAUGGCAGC
CCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAAUGCUUCCUGCCUCCCAUGCAUCU
GUACUCCUG
>Rn5-2pl-D1_For2
AGAACCUUCUGCCCCAGCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCC
ACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAGCUCUGCAGAAAAUGU
CCUCCCUUGUGGCUGCCUCAGCUCGUACCUUUGGCCUGAAGUCCCAGCAUUAAUGGCAGC
CCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAAUGCUUCCUGCCUCCCAUGCAUCU
GUACUCCUG
>Rn6-1WoA5_For2
UGAAGAUGACCACAUUCAAGGAAGAACCUUCUGCCCCAGCUUUGCAGGAUGAAACACUUC
CCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGUUCAGCAG
CUCUGCAGAAAAUGUCCUCCCUUGUGGCUGCCUCAGCUCGUACCUUUGGCCUGAAGUCCC
AGCAUUAAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAAUGCUUCCU
GCCUCCCAUGCAUCUGUACUCCUG
>Rn6-1Wo_G10_b
UGAAGAUGACCACAUUCAAGGAAGAACCUUCUGCCCCAGCUUUGCAGGAUGAAACACUUC
CCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGU
UCAGCAGCUCUGCAGAAAAUGUCCUCCCUUGUGGCUGCCUCAGCUCGUACCUUUGGCCUG
AAGUCCCAGCAUUAAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAAU
GCUUCCUGCCUCCCAUGCAUCUGUACUCCC
>Rn6-2WoE4_For2
CCACAUUCAAGGAAGAACCUUCUGCCCCAGCUUUGCAGGAUGAAACACUUCCCCGCUUGG
CUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAGCU
CUGCAGAAAAUGUCCUCCCUUGUGGCUGCCUCAGCUCGUACCUUUGGCCUGAAGUCCCAG
CAUUAAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAAUGCUUCCUGC
CCCCC
>Rn6-96hG12_For2
GUGAAGAUGACCACAUUCAAGGAAGAACCUUCUGCCCCAGCUUUGCAGGAUGAAACACUU
CCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGG
UUCAGCAGCUCUGCAGAAAAUGUCCUCCCUUGUGGCUGCCUCAGCUCGUACCUUUGGCCU
GAAGUCCCAGCAUUAAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAA
UGCUUCCUGCCCCCCAU
>Rn6-96h-2pl-C12
AGAACCUUCUGCCCCAGCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCC
ACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAGCUCUGCAGAAAAUGU
CCUCCCUUGUGGCUGCCUCAGCUCGUACCUUUGGCCUGAAGUCCCAGCAUUAAUGGCAGC
CCCUCAUCUUCCAAGUUUUGUGCUCCCCC
>Rn6-96h-2pl-A6_F
CUGAAGUGAAGAUGACCACAUUCAAGGAAGAACCUUCUGCCCCAGCUUUGCAGGAUGAAA
CACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCU
ACUGGUUCAGCAGCUCUGCAGAAAAUGUCCUCCCUUGUGGCUGCCUCAGCUCGUACCUUU
GGCCUGAAGUCCCAGCAUUAAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUA
CCUAAUGCUUCCUGCCUCCCAUGCAUCUGUACUCCU
>Rn6-96h-2pl-H5_F
CUGAAGUGAAGAUGACCACAUUCAAGGAAGAACCUUCUGCCCCAGCUUUGCAGGAUGAAA
CACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCU
ACUGGUUCAGCAGCUCUGCAGAAAAUGUCCUCCCUUGUGGCUGCCUCAGCUCGUACCUUU
GGCCUGAAGUCCCAGCAUUAAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUA
CCUAAUGCUUCCUGCCUCCCAUGCAUCUGUACUCCU
>Rn6-2WoG1_For2
AAGAUGACCACAUUCAAGGAAGAACCUUCUGCCCCAGCUUUGCAGGAUGAAACACUUCCC
CGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUC
AGCAGCUCUGCAGAAAAUGUCCUCCCUUGUGGCUGCCUCAGCUCGUACCUUUGGCCUGAA
GUCCCAGCAUUAAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAAUGC
UUCCUGCCUCCCAUGCAUCUGUACUCCUGC
>Rn6-96h-2pl-D11
CCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGG
UUCAGCAGCUCUGCAGAAAAUGUCCUCCCUUGUGGCUGCCUCAGCUCGUACCUUUGGCCU
GAAGUCCCAGCAUUAAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAA
UGCUUCCUGCCUCCCAUGCAUCUGUACUCCU
>Rn6-96h-2pl-F9_F
CCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGG
UUCAGCAGCUCUGCAGAAAAUGUCCUCCCUUGUGGCUGCCUCAGCUCGUACCUUUGGCCU
GAAGUCCCAGCAUUAAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAA
UGCUUCCUGCCUCCCAUGCAUCUGUACUCCU
>hBg:
GAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUC
Без UTR:
>
>BB
UGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACCUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>BD
UGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACCUUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCC
>BE
UGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACCGCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCC
>BF
UGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACCCUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCC
>BG
UGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACCCUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCAC
>BhBg
UGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACCGAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUC
>BI
UGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>BJ
UGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACCCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCC
>DB
UUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCCUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>DD
UUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCCUUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCC
>DE
UUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCCGCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCC
>DF
UUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCCCUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCC
>DG
UUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCCCUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCAC
>DhBg
UUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCCGAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUC
>DI
UUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>DJ
UUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCCCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCC
>EB
GCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>ED
GCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCUUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCC
>EE
GCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCGCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCC
>EF
GCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCCUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCC
>EG
>GCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCCUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCAC
>EhBg
GCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCGAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUC
>EI
GCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>EJ
GCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCCCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCC
>FB
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>FD
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCUUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCC
>FE
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCGCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCC
>FF
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCC
>FG
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCAC
>FhBg
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCGAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUC
>FI
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>FJ
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCC
>GB
CUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCACUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>GD
CUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCACUUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCC
>GE
CUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCACGCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCC
>GF
CUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCACCUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCC
>GG
CUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCACCUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCAC
>GhBg
CUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCACGAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUC
>GI
CUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCACCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>GJ
CUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCACCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCC
>hBgB
GAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUCUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>hBgD
GAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUCUUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCC
>hBgE
GAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUCGCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCC
>hBgF
GAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUCCUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCC
>hBgG
GAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUCCUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCAC
>hBghBg
GAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUCGAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUC
>hBgI
GAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>hBgJ
GAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUCCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCC
>IB
CAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACCUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>ID
CAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACCUUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCC
>IE
CAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACCGCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCC
>IF
CAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACCCUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCC
>IG
CAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACCCUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCAC
>IhBg
CAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACCGAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUC
>II
CAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>IJ
CAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACCCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCC
>JB
CUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCCUGCCCGUCCUCACCAAGACUGACUGCCUGCUGCUUUGCUACUGCCCGGGCCCAUGAGACUGACUUCCCACUGCUCUGCCUGCCUCUCCCCACUGCACUGGCACAGCCCCGCCUUGCCGCUGCUGAUCCAUUGCCGGUGUGACC
>JD
CUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCCUUCCAGCCAGACACCCGCCCCCCGGCCCUGGCUAAGAAGUUGCUUCCUGUUGCCAGCAUGACCUACCCUCGCCUCUUUGAUGCCAUCCGCUGCCACCUCCUUUUGCUCCUGGACCCUUUAGCCUCUCUGCCCUUCCACUCUCUGACCCC
>JE
CUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCCGCCUUGGCUCCUCCAGGAAGGCUCAGGAGCCCUACCUCCCUGCCAUUAUAGCUGCUCCCCGCCAGAAGCCUGUGCCAACUCUCUGCAUUCCCUGAUCUCCAUCCCUGUGGCUGUCACCCUUGGUCACCUCCGUGCUGUCACUGCCAUCUCCCCCC
>JF
CUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCCCUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCC
>JG
CUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCCCUGACAGCGUGGGCAACGCCUGCCGCCUGCUCUGAGGCCCGAUCCAGUGGGCAGGCCAAGGCCUGCUGGGCCCCCGCGGACCCAGGUGCUCUGGGUCACGGUCCCUGUCCCCGCACCCCCGCUUCUGUCUGCCCCAUUGUGGCUCCUCAGGCUCUCUCCCCUGCUCUCCCACCUCUACCUCCACCCCCAC
>JhBg
CUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCCGAGAGCUCGCUUUCUUGCUGUCCAAUUUCUAUUAAAGGUUCCUUUGUUCCCUAAGUCCAACUACUAAACUGGGGGAUAUUAUGAAGGGCCUUGAGCAUCUGGAUUCUGCCUAAUAAAAAACAUUUAUUUUCAUUGCUGCGUC
>JI
CUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>JJ
CUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCCCUUUGCAGGAUGAAACACUUCCCCGCUUGGCUCUCAUUCUUCCACAAGAGAGACCUUUCUCCGGACCUGGUUGCUACUGGUUCAGCAACUCUGCAGAAAAUGUCCUCCCCUGUGGCUGCCUCAGCUCAUGCCUUUGGCCUGAAGUCCCAGCAUUGAUGGCAGCCCCUCAUCUUCCAAGUUUUGUGCUCCCCUUUACCUAACGCUUCCUGCCUCCCAUGCAUCUGUACUCCUCC
>FI-UTR с 97,5% гомологией (случайные модификации)
CUGGUACUGCAUGGACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCACCCCCGACCUCGGGUCCCAGGUAUCGUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCAUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAUCGAAUGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>FI-UTR с 95% гомологией (случайные модификации)
CUCGUACUGCAUGGACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCACCACCGACCUCGGGUCCCAGGUAUCGUCCCACCUCCACGUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCAUAGCCACACCCCCACGGGAAACAGUAGUGAUUAACCUUUAGCAAUAAUCGAAUGUCUAACUAAGCUAUACUAACCCCAGGGUUGAUCAAUUACGUGCCAGCCACACC
>FI-UTR с 92,5% гомологией (случайные модификации)
CUCGUACUGCAUGGACGCAAUGCUAGCUGCCCCUUUCCCGUCCUGGGUACCCCGAGUCACCACCGACCUCGGGUCCCAGGUAUCGUCCCACCUCCACGUGCCCCACUCACCACCUUUGCUAGUUCCAGACACCUCCCAUGCACGCAGCAAUGCAGAUCAAAACGCUUAGCAUAGCCACACCCCCACGGGAAACAGUAGUGAUUAACCUUUAGCAAUAAUCGAAUGUCUAACUAAGCUAUACUAACCCCAGGGUUGAUCAAUUACGUGCCAGCCACACC
>FI-UTR с 90% гомологией (случайные модификации)
GUCGUACUGCAUGGACGCAAUGCUAGCAGCACCUUUCCCGUCCUGGGUACCCCGAGUCACCACCGACCUCGGGUCCCAGGUAUCGUCCCACCUCCACGUGCCCCACCCACCACCUUUGCUAGUUCCAGAGACCUCCCAUGCACGCAGCAAUGCAGAUCAAAACGCUUAGCAUAGCCACACCGCCACGGGAAACAGUAGUGAUCAACCUUUAGCUAUAAUCGAAUGUCUAACUAAGCUAUUCUAACCACAGGGUUGAUCAAUUACGUGCCAGCCAGACC
>8-нуклеотидная мутация в FI
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCAAAGGGCUCCUGGGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>FI-UTR с 98,75% гомологией (модификации, дестабилизирующие структуру)
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUGGUCCGUACCCCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>FI-UTR с 97,5% гомологией (модификации, дестабилизирующие структуру)
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUGGACCGUACGGCGAGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>FI-UTR с 96,25% гомологией (модификации, дестабилизирующие структуру)
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUGCCGUGGACCGUACGGGCUGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>FI-UTR с 95% гомологией (модификации, дестабилизирующие структуру)
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUGGGCUGGACCGUACGGGCUGUCUCCCCCGACCUCGGGUCCCAGGUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>FI-UTR с 97,5% гомологией (модификации, сохраняющие структуру)
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUGGUCCGUACCCCGAGUCUCCCCCGACCUCGGGUCGGACCUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>FI-UTR 95% (модификации, сохраняющие структуру)
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUCCCGUGGACCGUACGGCGAGUCUCCCCCGACCUCGCCUCGGUCCUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGGAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>FI-UTR 92,5% (модификации, сохраняющие структуру)
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUGCCGUGGACCGUACGGGCUGUCUCCCCCGACCAGCCCUCGGUCCUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCACGGCAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
>FI-UTR 90% (модификации, сохраняющие структуру)
CUGGUACUGCAUGCACGCAAUGCUAGCUGCCCCUUUGGGCUGGACCGUACGGGCUGUCUCCCCCGACCAGCCCUCGGUCCUAUGCUCCCACCUCCACCUGCCCCACUCACCACCUCUGCUAGUUCCAGACACCUCCCAAGCACGCAGCAAUGCAGCUCAAAACGCUUAGCCUAGCCACACCCCCAGCCCAAACAGCAGUGAUUAACCUUUAGCAAUAAACGAAAGUUUAACUAAGCUAUACUAACCCCAGGGUUGGUCAAUUUCGUGCCAGCCACACC
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> BioNTech RNA Pharmaceuticals GmbH et al.
<120> ПОСЛЕДОВАТЕЛЬНОСТИ 3'-UTR ДЛЯ СТАБИЛИЗАЦИИ РНК
<130> 674-146 PCT
<150> PCT/EP2015/073180
<151> 2015-10-07
<160> 221
<170> PatentIn version 3.5
<210> 1
<211> 208
<212> RNA
<213> Homo sapiens
<400> 1
cauccugcug cugcugcugc ugcugcugcg ggucuuccug gaaucugacc auucguuguc 60
ugcuaugccc guccucacca agacugacug ccugcugcuu ugcuacugcc cgggcccaug 120
agacugacuu cccacugcuc ugccugccuc uccccacugc acuggcacag ccccgccuug 180
ccgcugcuga uccauugccg gugugacc 208
<210> 2
<211> 202
<212> RNA
<213> Homo sapiens
<400> 2
gcugcugcug cugcugcugc ugcgggucuu ccuggaaucu gaccauucgu ugucugcuau 60
gcccguccuc accaagacug acugccugcu gcuuugcuac ugcccgggcc caugagacug 120
acuucccacu gcucugccug ccucucccca cugcacuggc acagccccgc cuugccgcug 180
cugauccauu gccgguguga cc 202
<210> 3
<211> 210
<212> RNA
<213> Homo sapiens
<400> 3
uuccugcugc ugcugcugcu gcugcugcug cgggucuucc uagaaucuga ccauucguug 60
ucugcuaugc ccguccucac caagacugac ugccugcugc uuugcuacug cccgggccca 120
ugagacugac uucccacugc ucugccugcc ucuccccacu gcacuggcac agccccgccu 180
ugccgcugcu gauccauugc cggugagacc 210
<210> 4
<211> 193
<212> RNA
<213> Homo sapiens
<400> 4
ugcugcugcu gcugcggguc uuccuggaau cugaccauuc guugucugcu augcccgucc 60
ucaccaagac ugacugccug cugcuuugcu acugcccggg cccaugagac ugacuuccca 120
cugcucugcc ugccucuccc cacugcacug gcacagcccc gccuugccgc ugcugaucca 180
uugccggcgg aca 193
<210> 5
<211> 186
<212> RNA
<213> Homo sapiens
<400> 5
gcugcugcgg gucuuccugg aaucugacca uucguugucu gcuaugcccg uccucaccaa 60
gacugacugc cugcugcuuu gcuacugccc gggcccauga gacugacuuc ccacugcucu 120
gccugccucu ccccacugca cuggcacagc cccgccuugc cgcugcugau ccauugccgg 180
cguacc 186
<210> 6
<211> 170
<212> RNA
<213> Homo sapiens
<400> 6
cuggaaucug accauucguu gucugcuaug cccguccuca ccaagacuga cugccugcug 60
cuuugcuacu gcccgggccc augagacuga cuucccacug cucugccugc cucuccccac 120
ugcacuggca cagccccgcc uugccgcugc ugauccauug ccggcggacc 170
<210> 7
<211> 206
<212> RNA
<213> Homo sapiens
<400> 7
uccugcugcu gcugcugcug cugcugcggg ucuuccugga aucugaccau ucguugucug 60
cuaugcccgu ccucaccaag acugacugcc ugcugcuuug cuacugcccg ggcccaugag 120
acugacuucc cacugcucug ccugccucuc cccacugcac uggcacagcc ccgccuugcc 180
gcugcugauc cauugccggu gggacc 206
<210> 8
<211> 121
<212> RNA
<213> Homo sapiens
<400> 8
cugccugcug cuuugcuacu gcccgggccc augagacuga cuucccacug cucugccugc 60
cucuccccac ugcacuggca cagccccgcc uugccgcugc ugauccauug ccgguagaac 120
c 121
<210> 9
<211> 206
<212> RNA
<213> Homo sapiens
<400> 9
uccugcugcu gcugcugcug cugcugcggg ucuuccugga aucugaccau uuguugucug 60
cuaugcccgu ccucaccaag acugacugcc ugcugcuuug cuacugcccg ggcccaugag 120
acugacuucc cacugcucug ccugccucuc cccacugcac uggcacagcc ccgccuugcc 180
gcugcugauc cauugccggu gggacc 206
<210> 10
<211> 196
<212> RNA
<213> Homo sapiens
<400> 10
gcugcugcug cugcugcggg ucuuccugga aucugaccau ucguugucug cuaugcccgu 60
ccucaccaag acugacugcc ugcugcuuug cuacugcccg ggcccaugag acugacuucc 120
cacugcucug ccugccucuc cccacugcac uggcacagcc ccgccuugcc gcugcugauc 180
cauugccggu gugacc 196
<210> 11
<211> 276
<212> RNA
<213> Homo sapiens
<400> 11
ucuggccuca cugagucuga agagcuguua acuaccaugg ccaguccucc cugagucuga 60
ccaucuucca uccugcugcu gcugcugcug cugcugcggg ucuuccugga aucugaccau 120
ucguugucug cuaugcccgu ccucaccaag acugacugcc ugcugcuuug cuacugcccg 180
ggcccaugag acugacuucc cacugcucug ccugccucuc cccacugcac uggcacagcc 240
ccgccuugcc gcugcugauc cauugccggu gugacc 276
<210> 12
<211> 276
<212> RNA
<213> Homo sapiens
<400> 12
ucuggccuca cugagucuga agagcuguua acuaccaugg ccaguccucc cugagucuga 60
ccaucuucca uccugcugcu gcugcugcug cugcugcggg ucuuccugga aucugaccau 120
ucguugucug cuaugcccgu ccucaccaag acugacugcc ugcugcuuug cuacugcccg 180
ggcccaugag acugacuucc cacugcucug ccugccucuc cccacugcac uggcacagcc 240
ccgccuugcc gcugcugauc cauugccggu gugacc 276
<210> 13
<211> 245
<212> RNA
<213> Homo sapiens
<400> 13
cuaccauggc caguccuccc ugagucugac caucuuccau ccugcugcug cugcugcugc 60
ugcugcgggu cuuccuggaa ucugaccauu cguugucugc uaugcccguc cucaccaaga 120
cugacugccu gcugcuuugc uacugcccgg gcccaugaga cugacuuccc acugcucugc 180
cugccuuucc ccacugcacu ggcacagccc cgccuugccg cugcugaucc auugccggug 240
ugacc 245
<210> 14
<211> 233
<212> RNA
<213> Homo sapiens
<400> 14
guccucccug agucugacca ucuuccaucc ugcugcugcu gcugcugcug cugcgggucu 60
uccuggaauc ugaccauucg uugucugcua ugcccguccu caccaagacu gacugccugc 120
ugcuuugcua cugcccgggc ccaugagacu gacuucccac ugcucugccu gccucucccc 180
acugcacugg cacagccccg ccuugccgcu gcugauccau ugccggucug aca 233
<210> 15
<211> 232
<212> RNA
<213> Homo sapiens
<400> 15
guccucccug agucugacca ucuuccaucc ugcugcugcu gcugcugcug cugcgggucu 60
uccuggaauc ugaccauuug uugucugcua ugccccuccu caccaagacu gacugccugc 120
ugcuuugcua cugcccgggc ccaugagacu gacuucccac ugcucugccu gccuucccca 180
cugcacuggc acagccccgc cuugccgcug cugauccauu gccgguguga cc 232
<210> 16
<211> 208
<212> RNA
<213> Homo sapiens
<400> 16
ccauccugcu gcugcugcug cugcugcugc gggucuuccu ggaaucugac cauucguugu 60
cugcuaugcc cguccucacc aagacugacu gccugcugcu ugcuacugcc cgggcccaug 120
agacugacuu cccacugcuc ugccugccuc uccccacugc acuggcacag ccccgccuug 180
ccgcugcuga uccauugccg gugugacc 208
<210> 17
<211> 237
<212> RNA
<213> Homo sapiens
<400> 17
gccaguccuc ccugagucug accaucuucc auccugcugc ugcugcugcu gcugcugcgg 60
gucuuccugg aaucugacca uucguugucu gcuaugcccg uccucaccaa gacugacugc 120
cugcugauuu gcuacugccc gggcccauga gacugacuuc ccacugcucu gccugccucu 180
ccccacugca cuggcacagc cccgccuugc cgcugcugau ccauugccgg ugugacc 237
<210> 18
<211> 297
<212> RNA
<213> Homo sapiens
<400> 18
ugccuuccgu cuccugcugc uucuggccuc acugagucug aagagcuguu aacuaccaug 60
gccaguccuc ccugagucug accaucuucc auccugcugc ugcugcugcu gcugcugcgg 120
gucuuccugg aaucugacca uucguugucu gcuaugcccg uccucaccaa gacugacugc 180
cugcugcuuu gcuacugccc gggcccauga gacugacuuc ccacugcucu gccugccucu 240
ccccacugca cuggcacagc cccgccuugc cgcugcugau ccauugccgg ugugacc 297
<210> 19
<211> 238
<212> RNA
<213> Homo sapiens
<400> 19
ggccaguccu cccugagucu gaccaucuuc cauccugcug cugcugcugc ugcugcugcg 60
ggucuuccug gaaucugacc auucguuguc ugcuaugccc guccucacca agacugacug 120
ccugcugcuu ugcuacugcc cgggcccaug agacugacuu cccacugcuc ugccugccuc 180
uccccacugc acuggcacag ccccgccuug ccgcugcuga uccauugccg gugugacc 238
<210> 20
<211> 238
<212> RNA
<213> Homo sapiens
<400> 20
ggccaguccu cccugagucu gaccaucuuc cauccugcug cugcugcugc ugcugcugcg 60
ggucuuccug gaaucugacc auucguuguc ugcuaugccc guccucacca agacugacug 120
ccugcugcuu ugcuacugcc cgggcccaug agacugacuu cccacugcuc ugccugccuc 180
uccccacugc acuggcacag ccccgccuug ccgcugcuga uccauugccg gugugacc 238
<210> 21
<211> 219
<212> RNA
<213> Homo sapiens
<400> 21
ugaccaucuu ccauccugcu gcugcugcug cugcugcugc gggucuuccu ggaaucugac 60
cauucguugu cugcuaugcc cguccucacc aagacugacu gccugcugcu uugcuacugc 120
ccgggcccau gagacugacu ucccacugcu cugccugccu cuccccacug cacuggcaca 180
gccccgccuu gccgcugcug auccauugcc ggugugacc 219
<210> 22
<211> 237
<212> RNA
<213> Homo sapiens
<400> 22
ccaguccucc cugagucuga ccaucuucca uccugcugcu gcugcugcug cugcugcggg 60
ucuuccugga aucugaccau ucguugucug cuaugcccgu ccucaccaag acugacugcc 120
ugcugcuuug cuacugcccg ggcccaugag acugacuucc cucugcucug ccugccucuc 180
cccacugcac uggcacagcc ccgccuugcc gcugcugauc cauugccugu gugacca 237
<210> 23
<211> 214
<212> RNA
<213> Homo sapiens
<400> 23
ugaccaucuu ccauccugcu gcugcugcug cugcgggucu uccuggaauc ugaccauucg 60
uugucugcua ugcccguccu uaccaagacu gacugccugc ugcuuugcua cugcccgggc 120
ccaugagacu gacuucccac ugcucugccu gccucucccc acugcacugg cacagccccg 180
ccuugucgcu gcugauccau ugccggugug acac 214
<210> 24
<211> 128
<212> RNA
<213> Homo sapiens
<400> 24
gacugacugc cugcugcuuu gcuacugccc gggcccauga gacugacuuc ccacugcucu 60
gccugccucu ccccacugca cuggcacagc cccgccuugc cgcugcugau ccauugccgg 120
ugugaccc 128
<210> 25
<211> 230
<212> RNA
<213> Homo sapiens
<400> 25
cucccugagu cugaccaucu uccauccugc ugcugcugcu gcugcugcug cgggucuucc 60
uggaaucuga ccauucguug ucugcuaugc ccguccucac caagacugac ugccugcugc 120
uuugcuacug cccgggccca ugagacugac uucccacugc ucugccugcc ucuccccacu 180
gcacuggcac agccccgccu ugccgcugcu gauccauugc cggugugacc 230
<210> 26
<211> 211
<212> RNA
<213> Homo sapiens
<400> 26
uuccauccug cugcugcugc ugcugcugcu gcgggucuuc cuggaaucug accauucguu 60
gucugcuaug cccguccuca ccaagacuga cugccugcug cuuugcuacu gcccgggccc 120
augagacuga cuucccacug cucugccugc cucuccccac ugcacuggca cagccccgcc 180
uugccgcugc ugauccauug ccggugugac c 211
<210> 27
<211> 143
<212> RNA
<213> Homo sapiens
<400> 27
ugcccguccu caccaagacu gacugccugc ugcuuugcua cugcccgggc ccaugagacu 60
gacuucccac ugcucugccu gccucucccc acugcacugg cacagccccg ccuugccgcu 120
gcugauccau ugccggugug acc 143
<210> 28
<211> 143
<212> RNA
<213> Homo sapiens
<400> 28
ugcccguccu caccaagacu gacugccugc ugcuuugcua cugcccgggc ccaugagacu 60
gacuucccac ugcucugccu gccucucccc acugcacugg cacagccccg ccuugccgcu 120
gcugauccau ugccggugug acc 143
<210> 29
<211> 181
<212> RNA
<213> Homo sapiens
<400> 29
gcgggucuuc cuggaaucug aacauucguu gucugcuaug cccguccuca ccaagacuga 60
cugccugcug cuuugcuacu gcccgggccc augagacuga cuucccacug cucugccugc 120
cucuccccac ugcacuggca cagccccgcc uugccgcugc ugauccauug ccggugugac 180
c 181
<210> 30
<211> 154
<212> RNA
<213> Homo sapiens
<400> 30
guugucugcu augcccgucc ucaccaagac ugacugccug cugcuuugcu acugcccggg 60
cccaugagac ugacuuccca cugcucugcc ugccucuccc cacugcacug gcacagcccc 120
gccuugccgc ugcugaucca uugccggugu gacc 154
<210> 31
<211> 130
<212> RNA
<213> Homo sapiens
<400> 31
caagacugac ugccugcugc uuugcuacug cccgggccca ugagacugac uucccacugc 60
ucugccugcc ucuccccacu gcacuggcac agccccgccu ugccgcugcu gauccauugc 120
cggugugacc 130
<210> 32
<211> 138
<212> RNA
<213> Homo sapiens
<400> 32
ccucaccaag acugacugcc ugcugcuuug cuacugcccg gacccaugag acugacuucc 60
cacugcucug ccugccucuc cccacugcac uggcacagcc ccgccuugcc gcugcugauc 120
cauugccggu gugacugc 138
<210> 33
<211> 214
<212> RNA
<213> Homo sapiens
<400> 33
cuuccauccu gcugcugcug cugcugcugc ugcgggucuu ccuggaaucu gaccauucgu 60
ugucugcuau gcccguccuc accaagacug acugccugcu gcuuugcuac ugcccgggcc 120
caugagacug acuucccacu gcucugccug ccucucccca cugcacuggc acagccccgc 180
cuugccgcug cugauccauu gccgguguga cccc 214
<210> 34
<211> 135
<212> RNA
<213> Homo sapiens
<400> 34
cucaccaaga cugacugccu gcugcuuugc uacugcccgg gcccaugaga cugacuuccc 60
acugcucugc cugccucucc ccacugcacu ggcacagccc cgccuugccg cugcugaucc 120
auugccggug ugacc 135
<210> 35
<211> 129
<212> RNA
<213> Homo sapiens
<400> 35
aagacugacu gccugcugcu uugcuacugc ccgggcccau gagacugacu ucccacugcu 60
cugccugccu cuccccacug cacuggcaca gccccgccuu gccgcugcug auccauugcc 120
ggugugacc 129
<210> 36
<211> 541
<212> RNA
<213> Homo sapiens
<400> 36
cuccagcucg cuuccauuug cuugcagaag uucucgcugu gcucacgaag cuugcgcucc 60
uuggaggccu cagcaacagc aucaucaagc ugagcuucca gcucuuuccu gagcuucuca 120
gcucuccgca uuuccugccg cauggcgucc accuucugcg uggccaccuc caucuccucc 180
uccuugucuc gcagcugccg ggacaccuuc ugcgcuaaga ugggauacgg cauugaggga 240
ucaaugugua aggauccgau cugcuucugg ccucacugag ucugaagagc uguuaacuac 300
cauggccagu ccucccugag ucugaccauc uuccauccug cugcugcugc ugcugcugcu 360
gcgggucuuc cuggaaucug accauucguu gucugcuaug uccguccuca ccaagacuga 420
cugccugcug cuuugcuacu gcccgggccc augagacuga cuucccacug cucugccugc 480
cucuccccac ugcacuggca cagccccgcc uugccgcugc ugauccauug ccggugugac 540
c 541
<210> 37
<211> 125
<212> RNA
<213> Homo sapiens
<400> 37
cugacugccu gcugcuuugc uacugcccgg gcccaugaga cugacuuccc acugcucugc 60
cugccucucc ccacugcacu ggcacagccc cgccuugccg cugcugaucc auugccggug 120
ugacc 125
<210> 38
<211> 131
<212> RNA
<213> Homo sapiens
<400> 38
ccaagacuga cugccugcug cuuugcuacu gcccgggccc augagacuga cuucccacug 60
cucugccugc cucuccccac ugcacuggca cagccccgcc uugccgcugc ugauccauug 120
acggugugac c 131
<210> 39
<211> 245
<212> RNA
<213> Homo sapiens
<400> 39
uaacuaccau ggccaguccu cccugagucu gaccaucuuc cauccugcug cugcugcugc 60
ugcugcgggu cuuccuggaa ucugaccauu cguugucugc uaugcccguc cucaccaaga 120
cugacugccu gcugcuuugc uacugcccgg gcccaugaga cugacuuccc acugcucugc 180
cugccucucc ccacugcacu ggcacagccc cgccuugccg cuguugaucc auugccggug 240
ugacc 245
<210> 40
<211> 131
<212> RNA
<213> Homo sapiens
<400> 40
ccaagacuga cugccugcug cuuugcuacu gcccgggccc augagacuga cuucccacug 60
cucugccugc cucuccccac ugcacuggca cagccccgcc uugccgcugu ugauccauug 120
ucggugugac c 131
<210> 41
<211> 131
<212> RNA
<213> Homo sapiens
<400> 41
ccaagacuga cugccugcug cuuugcuacu gcccgggccc augagacuga cuucccacug 60
cucugccugc cucuccccac ugcacuggca cagccccgcc uugccgcugc ugauccauug 120
ccggugugac c 131
<210> 42
<211> 149
<212> RNA
<213> Homo sapiens
<400> 42
cugcuaugcc uguccucacc aagacugacu gccugcugcu uugcuacugc ccgggcccau 60
gagacugacu ucccacugcu cugccugccu cuccccaaug cacuggcaca gccccgccuu 120
gccgcugcug auccauugcc ggugugacc 149
<210> 43
<211> 149
<212> RNA
<213> Homo sapiens
<400> 43
cugcuaugcc cguccucacc aagacugacu gccugcugcu uugcuacugc ccgggcccau 60
gagacugacu ucccacugcu cugccugccu cuccccacug cacuggcaca gccccgccuu 120
gccgcugcug auccauugcc ggugugacc 149
<210> 44
<211> 147
<212> RNA
<213> Homo sapiens
<400> 44
gcuaugcccg uccucaccaa gacugacugc cugcugcuuu gcuacugccc gggcccauga 60
gacugacuuc ccacugcucu gccugccucu ccccacugca cuggcacagc cccgccuugc 120
cgcugcugau ccauugccgg ugugacc 147
<210> 45
<211> 176
<212> RNA
<213> Homo sapiens
<400> 45
ucuuccugga aucugaccau ucguugucug cuaugcccgu ccucaccaag acugacugcc 60
ugcugcuuug cuacugcccg ggcccaugag acugacuucc cacugcucug ccugccucuc 120
cccacugcac uggcacagcc ccgccuugcc gcugcugauc cauugccggu gugacc 176
<210> 46
<211> 171
<212> RNA
<213> Homo sapiens
<400> 46
cuggaaucug accauucguu gucugcuaug cccguccuca ccaagacuga cugccugcug 60
cuuugcuacu gcccgggccc augagacuga cuucccacug cucugccugc cucuccccac 120
ugcacuggca cagccccgcc uugccgcugc ugauccauug ccggugugac c 171
<210> 47
<211> 171
<212> RNA
<213> Homo sapiens
<400> 47
cuggaaucug accauucguu gucugcuaug cccguccuca ccaagacuga cugccugcug 60
cuuugcuacu gcccgggccc augagacuga cuucccacug cucugccugc cucuccccac 120
ugcacuggca cagccccgcc uugccgcugc ugauccauug ccggugugac c 171
<210> 48
<211> 169
<212> RNA
<213> Homo sapiens
<400> 48
ggaaucugac cauucguugu cugcuaugcc cguccucacc aagacugacu gccugcugcu 60
uugcuacugc ccgggcccau gagacugacu ucccacugcu cugccugccu cuccccacug 120
cacuggcaca gccccgccuu gccgcugcug auccauugcc ggugugacc 169
<210> 49
<211> 131
<212> RNA
<213> Homo sapiens
<400> 49
ccaagacuga cugccugcug cuuugcuacu gcccgggccc augagacuga cuucccacug 60
cucugccugc cucuccccac ugcacuggca uagccccgcc uugccgcugc ugauccauug 120
ccggugugac c 131
<210> 50
<211> 175
<212> RNA
<213> Homo sapiens
<400> 50
cuuccuggaa ucugaccauu cguugucugc uaugcccguc cucaccaaga cugacugccu 60
gcugcuuugc uacugcccgg gcccaugaga cugacuuccc acugcucugc cugccucucc 120
ccacugcacu ggcauagccc cgccuugccg cugcugaucc auuuccggug ugacc 175
<210> 51
<211> 140
<212> RNA
<213> Homo sapiens
<400> 51
cagacacccg ccccccggcc cuggcuaaga auuugcuucc uguugccagc augaccuacc 60
cucgccucuu ugaugccauc cgcugccacc uccuuuugcu ccuggacccu uuagccucuc 120
ugcccuucca cucucugacc 140
<210> 52
<211> 149
<212> RNA
<213> Homo sapiens
<400> 52
uuccagccag acacccgccc cccggcccug gcuaagaagu ugcuuccugu ugccagcaug 60
accuacccuc gccucuuuga ugccauccgc ugccaccucc uuuugcuccu ggacccuuua 120
gccucucugc ccuuccacuc ucugacccc 149
<210> 53
<211> 212
<212> RNA
<213> Homo sapiens
<400> 53
cucgcuuccu gggucugcag guccagccgg cuggcacccu ccauguaccc aggggagauu 60
ccagccagac acccgccccc cggcccuggc uaagaaguug cuuccuguug ccagcaugac 120
cuacccucgc cucuuugaug ccauccgcug ccaccuccuu uugcuccugg acccuuuagc 180
cucucugccc uuccacucuc ugaccaccgc cc 212
<210> 54
<211> 144
<212> RNA
<213> Homo sapiens
<400> 54
uccagccaga cacccgcccc ccggcccugg cuaagaaguu gcuuccuguu gccagcauga 60
ccuacccucg ccucuuugau gccauccgcu gccaccuccu uuugcuccug gacccuuuag 120
cuucucugcc cuuccacucu cugg 144
<210> 55
<211> 207
<212> RNA
<213> Homo sapiens
<400> 55
cgcuuccugg gucugcaggu ccagccggcu ggcacccucc acguacccag gggagauucc 60
agccagacac ccgccccccg gcccuggcua agaaguugcu uccuguugcc agcaugaccu 120
acccucgccu cuuugaugcc auccgcugcc accuccuuuu gcuccuggac ccuuuagccu 180
cucugcccuu ccacucucug accaccg 207
<210> 56
<211> 158
<212> RNA
<213> Homo sapiens
<400> 56
cauguaccca ggggagauuc cagccagaca cccgcccccc ggcccuggcu aagaaguugc 60
uuccuguugc cagcaugacc uacccucgcc ucuuugaugc cauccgcugc caccuccuuu 120
ugcuccugga cccuuuagcc ucucugcccu uccacucg 158
<210> 57
<211> 197
<212> RNA
<213> Homo sapiens
<400> 57
cucgcuuccu gggucugcag guccagccgg cuggcacccu ccauguaccc aggggagauu 60
ccagccagac acccgccccc cggcccuggc uaagaaguug cuuccuguug ccagcaugac 120
cuacccucgc cucuuugaug ccauccgcug ccaccuccuu uugcuccugg acccuuuagc 180
cucucugccc ccccgau 197
<210> 58
<211> 236
<212> RNA
<213> Homo sapiens
<400> 58
cccagcuccc uaggcguccc aucucgcuuc cugggucugc agguccagcc ggcuggcacc 60
cuccauguac ccaggggaga uuccagccag acacccgccc cccggcccug gcuaagaagu 120
ugcuuccugu ugccagcaug accuacccuc gccucuuuga ugccauccgc ugccaccucc 180
uuuugcuccu ggacccuuua gccucucugc ccuuccacuc uuugaccccc aucuua 236
<210> 59
<211> 273
<212> RNA
<213> Homo sapiens
<400> 59
ggccaccggg caugggaagu augagaaggu gcuuguggaa gggggcccgg cucccuaggc 60
gucccaucuc gcuuccuggg ucugcagguc cagccggcug gcacccucca uguacccagg 120
gagauuccag ccagacaccc gccccccggc ccuggcuaag aaguugcuuc cuguugccag 180
caugaccuac ccucgccucu uugaugccau ccgcugccac cuccuuuugc uccuggaccc 240
uuuagccucu cugcccuucc acucucugac ccc 273
<210> 60
<211> 171
<212> RNA
<213> Homo sapiens
<400> 60
uguacccagg ggagauucca gccagacacc cgccccccgg cccuggcuaa gaaguugcuu 60
ccuguugcca gcaugaccua cccucgccuc uuugaugcca uccgcugcca ccuccuuuug 120
cuccuggacc cuuuagccuc ucugcccuuc cacucucuga ccaccacccc c 171
<210> 61
<211> 134
<212> RNA
<213> Homo sapiens
<400> 61
ccggcccugg cuaagaaguu gcuuccuguu gccagcauga ccuacccucg ccucuuugau 60
gccauccgcu gccaccuccu uuugcuccug gacccuuuag ccucucugcc cuuccacucu 120
cugaccacag cccc 134
<210> 62
<211> 140
<212> RNA
<213> Homo sapiens
<400> 62
ccccggcccu ggcuaagaag uugcuuccug uugccagcau gaccuacccu cgccucuuug 60
augccauccg cugccaccuc cuuuugcucc uggacccuuu agccucucug cccuuccacu 120
cucugaccac cgcccccgcc 140
<210> 63
<211> 188
<212> RNA
<213> Homo sapiens
<400> 63
ccggcuggca cccuccaugu acccagggga gauuccagcc agacacccgc cccccggccc 60
uggcuaagaa guugcuuccu guugccagca ugaccuaccc ucgccucuuu gaugccaucc 120
gcugccaccu ccuuuugcuc cuggacccuu uagccucucu gcccuuccac ucucugacca 180
ccgccccc 188
<210> 64
<211> 190
<212> RNA
<213> Homo sapiens
<400> 64
gccggcuggc acccuccaug uacccagggg agauuccagc cagacacccg ccccccggcc 60
cuggcuaaga aguugcuucc uguugccagc augaccuacc cuagccucuu ugaugccauc 120
cgcugccacc uccuuuuugc uccuggaccc uuuagccucu cugcccuucc acucucugac 180
caccgccccc 190
<210> 65
<211> 154
<212> RNA
<213> Homo sapiens
<400> 65
uccagccaga cacccgcccc ccggcccugg cuaagaaguu gcuuccuguu gccagcauga 60
ccuacccucg ccucuuugau gccauccgcu gccaccuccu uuugcuccug gacccuuuag 120
ccucucugcc cuuccacucu cugaccacca cccc 154
<210> 66
<211> 146
<212> RNA
<213> Homo sapiens
<400> 66
gccagacacc cgccccccgg cccuggcuaa gaaguugcuu ccuguugcca gcaugaccua 60
cccucgccuc uuugaugcca uccgcugcca ccuccuuuug cuccuggacc cuuuagccuc 120
ucugcccuuc cacucucuga cccccc 146
<210> 67
<211> 150
<212> RNA
<213> Homo sapiens
<400> 67
uccagccaga cacccgcccc ccggcccugg cuaagaaguu gcuuccuguu gccagcauga 60
ccuacccucg ccucuuugau gccauccgcu gccaccuccu uuugcuccug gacccuuuag 120
ccucucugcc cuuccacucu cugacccccc 150
<210> 68
<211> 233
<212> RNA
<213> Homo sapiens
<400> 68
cggcucccua ggcgucccau cucgcuuccu gggucugcag guccagccgg cuggcacccu 60
ccauguaccc aggggagauu ccagccagac acccgccccc cggcccuggc uaagaaguug 120
cuuccuguug ccagcaugac cuacccucgc cucuuugaug ccauccgcug ccaccuccuu 180
uugcuccugg acccuuuagc cucucugccc uuccacucuc ugaccacugc ccc 233
<210> 69
<211> 197
<212> RNA
<213> Homo sapiens
<400> 69
ugcaggucca gccggcuggc acccuccaug uacccagggg agauuccagc cagacaccca 60
ccccccggcc cuggcuaaga aguugcuccu guugccagca ugaccuaccc ucgccucuuu 120
gaugccaucc gcugccaccu ccuuuugcuc cuggacccuu uagccucucu gcccuuccac 180
ucucugacca cuacccc 197
<210> 70
<211> 155
<212> RNA
<213> Homo sapiens
<400> 70
uuccagccag acacccgccc cccggcccug gcuaagaagu ugcuuccugu ugccagcaug 60
accuacccuc gccucuuuga ugccauccgc ugccaccucc uuuugcuccu ggacccuuua 120
gccucucugc ccuuccacuc ucugaccacu gcccc 155
<210> 71
<211> 134
<212> RNA
<213> Homo sapiens
<400> 71
cccgcccccc ggcccuggcu aagaaguugc uuccuguugc cagcaugacc uacccucgcc 60
ucuuugaugc cauccgcugc caccuccuuu ugcuccugga cccuuuagcc ucucugcccu 120
uccacucucu gacc 134
<210> 72
<211> 147
<212> RNA
<213> Homo sapiens
<400> 72
cagccagaca cccgcccccc ggcccuggcu aagaaguugc uuccuguugc cagcaugacc 60
uacccucgcc ucuuugaugc cauccgcugc caccuccuuu ugcuccugga cccuuuagcc 120
ucucugcccu uccacucucu gaacacc 147
<210> 73
<211> 192
<212> RNA
<213> Homo sapiens
<400> 73
gagccuacuc ugaugaccgu ggccuuggcu ccuccaggaa ggcucaggag cccuaccucc 60
cugccauuau agcugcuccc cgccagaagc cugugccaac ucucugcauu cccugaucuc 120
cuguggcugu cacccuuggu caccuccgug cugucacugc caucuccccc cugaccccuc 180
gaacccaucc ua 192
<210> 74
<211> 191
<212> RNA
<213> Homo sapiens
<400> 74
gagccuacuc ugaugaccgu ggccuuggcu ccuccaggaa ggcucaggcg cccuaccucc 60
cugccauuau agcugcuccc cgccagaagc cugugccaac ucucugcauu cccugaucuc 120
caucccugug gcugucaccc uuggucaccu ccgugcuguc acugccaucu ccccccugac 180
cccucuaacc c 191
<210> 75
<211> 162
<212> RNA
<213> Homo sapiens
<400> 75
gccuacucug augaccgugg ccuuggcucc uccaggaagg cucaggagcc cuaccucccu 60
gccauuauag cugcuccccg ccagaagccu gugccaacuc ucugcauucc cugaucucca 120
ucccuguggc ugucacccuu gguccacugc caucuccccc cc 162
<210> 76
<211> 183
<212> RNA
<213> Homo sapiens
<400> 76
gagccuacuc ugaugaccgu ggccuuggcu ccuccaggaa ggcucaggag cccuaccucc 60
cugccauuau agcugcuccc cgccagaagc cugugccaac ucucugcauu cccugaucuc 120
caucccugug gcugucaccc uuggucaccu ccgugcuguu acugccaucu ccccccugac 180
ccc 183
<210> 77
<211> 240
<212> RNA
<213> Homo sapiens
<400> 77
gaagagccua cucugaugac cguggccuug gcuccuccag gaaggcucag gagcccuacc 60
ucccugccau uauagcugcu ccccgccaga agccugugcc aacucucugc auucccugau 120
cuccaucccu guggcuguca uccuugguca ccuccgugcu gucacugcca ucuccccccu 180
gaccccucua acccauccuc ugccucccuc ccugcaguca gaggguccug uucccaacca 240
<210> 78
<211> 166
<212> RNA
<213> Homo sapiens
<400> 78
uguggccuug gcuccuccag gaaggcuaag gagcccuacc ucccugccau uauagcugcu 60
ccccgccaga agccugugcc aacucucugc auucccugau cuccaucccu guggcuguca 120
cccuugguca ccuccgugcu gucacugcca ucuccccccu gacccc 166
<210> 79
<211> 155
<212> RNA
<213> Homo sapiens
<400> 79
gccuuggcuc cuccaggaag gcucaggagc ccuaccuccc ugccauuaua gcugcucccc 60
gccagaagcc ugugccaacu cucugcauuc ccugaucucc aucccugugg cugucacccu 120
uggucaccuc cgugcuguca cugccaucuc ccccc 155
<210> 80
<211> 310
<212> RNA
<213> Homo sapiens
<400> 80
agagccuacu cugaugaccg uggccuuggc uccuccagga aggcucagga gcccuaccuc 60
ccugccauua uagcugcucc ccgccagaag ccugugccaa cucucugcau ucccugaucu 120
ccaucccugu ggcugucacc cuuggucacc uccgugcugu cacugccauc uccccccuga 180
ccccucuaac ccauccucug ccucccuccc ugcagucaga ggguccuguu cccaucagcg 240
auuccccugc uuaaacccuu ccaugacucc ccacugcccu aagcugaggu cagucuccca 300
agccugacau 310
<210> 81
<211> 204
<212> RNA
<213> Homo sapiens
<400> 81
uauagcugcu ccccgccaga agccugugcc aacucucugc auucccugau cuccaucccu 60
guggcuguca cccuugguca ccuccgugcu gucacugcca ucuccccccu gaccccucua 120
acccauccuc ugccucccuc ccugcaguca gaggguccug uucccaucag cgauuccccu 180
gcuuaaaccc uuccaugaca gccc 204
<210> 82
<211> 169
<212> RNA
<213> Homo sapiens
<400> 82
ucugcauucc cugaucucca ucccuguggc ugucacccuu ggucaccucc gugcugucac 60
ugccaucucc ccccugaccc cucuaaccca uccucugccu cccucccugc agucagaggg 120
uccuguuccc aucagcgauu ccccugcuua agcccuucca ugacucccc 169
<210> 83
<211> 217
<212> RNA
<213> Homo sapiens
<400> 83
cucccugcca uuauagcugc uccccgccag aagccugugc caacucucug cauucccuga 60
ucuccauccc uguggcuguc acccuugguc accuccgugc ugucacugcc aucucccccc 120
ugaccccucu aacccauccu cugccucccu cccugcaguc agaggguccu guucccauca 180
gcgauucccc ugcuuaaacc cuuccaugac uccccaa 217
<210> 84
<211> 272
<212> RNA
<213> Homo sapiens
<400> 84
gccuacucug augaccgugg ccuugggucc uccaggaagg cucaggagcc cuaccucccu 60
gccauuauag cugcuccccg ccagaagucu gugccaacuc ucugcauucc cugaucucca 120
ucccuguggc ugucacccuu ggucaccucc gugcugucac ugccaucucc ccccugaccc 180
cucuaaccca uccucugccu cccucccugc agucagaggg uccuguuccc aucagcgauu 240
ccccugcuua aacccuucca ugacuccccu cu 272
<210> 85
<211> 131
<212> RNA
<213> Homo sapiens
<400> 85
cuaccucccu gccauuauag cugcuccccg ccagaagccu gugccaacuc ucugcauucc 60
cugaucucca ucccuguggc ugucacccuu ggucaccucc gugcugucac ugccaucucc 120
ccccugaccc c 131
<210> 86
<211> 136
<212> RNA
<213> Homo sapiens
<400> 86
cugguacugc augcacgcaa ugcuagcugc cccuuucccg uccuggguac cccgagucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucc 136
<210> 87
<211> 203
<212> RNA
<213> Homo sapiens
<400> 87
ccggcccuuc ccccguuuug aacaugugua accgacaguc ugccugggcc acagcccucu 60
cacccuggua cugcaugcac gcaaugcuag cugccccuuu cccguccugg gcaccccgag 120
ucucccccga ccccgggucc cagguaugcu cccaccucca ccugccccac ucaccaccuc 180
ugcuaguucc agacaccccc gcg 203
<210> 88
<211> 197
<212> RNA
<213> Homo sapiens
<400> 88
ccuucccccg uuuugaacau guguaaccga cagucugccu gggccacagc ccucucaccc 60
ugguacugca ugcacgcaau gcuagcugcc ccuuucccgc ccugggcacc ccgagucucc 120
cccgaccccg ggucccaggu augcucccac cuccaccugc cccacucacc accucugcua 180
guuccagaca ccuccac 197
<210> 89
<211> 273
<212> RNA
<213> Homo sapiens
<400> 89
ucugccuggg ccacagcccu cucacccugg uacugcaugc acgcaaugcu agcugccccu 60
uucccguccu gggcaccccg agucuccccc gaccccgggu cccagguaug cucccaccuc 120
caccugcccc acucaccacc ucugcuaguu ccagacaccu ccacgcccac cugguccucu 180
cccaucgccc acaaaagggg gggcacgagg gacgagcuua gcugagcugg gaggagcagg 240
gugagggugg gcgacccagg auucccccac ccc 273
<210> 90
<211> 199
<212> RNA
<213> Homo sapiens
<400> 90
ugacaccuca gcugacagcg ugggcaacgc cugccgccug cucugaggcc cgauccagug 60
ggcaggccaa ggccugcugg gcccccgcgg acccaggugc ucugggucac ggucccuguc 120
cccgcacccc cgcuucuguc ugccccauug uggcuccuca ggcucucucc ccugcucucc 180
caccucuacc uccacccca 199
<210> 91
<211> 193
<212> RNA
<213> Homo sapiens
<400> 91
cucagcugac agcgugggca acgccugccg ccugcucuga ggcccgaucc agugggcagg 60
ccaaggccug cugggccccc gcggacccag gugcucuggg ucacgguccc uguccccgca 120
cccccgcuuc ugucugcccc auuguggcuc cucaggcucu cuccccugcu cucccaccuc 180
uaccuccacu ccc 193
<210> 92
<211> 158
<212> RNA
<213> Homo sapiens
<400> 92
ucugaggccc gauccagugg gcaggccaag gccugcuggg cccccgcgga cccaggugcu 60
cugggucacg gucccugucc ccgcaccccc gcuucugucu gccccauugu ggcuccucag 120
gcucucuccc cugcucuccc accucuaccu ccaccccc 158
<210> 93
<211> 153
<212> RNA
<213> Homo sapiens
<400> 93
ggcccgaucc agugggcagg ccaaggccug cugggccccc gcggacccag gugcucuggg 60
ucacgguccc uguccccgca cccccgcuuc ugucugcccc auuguggcuc cucaggcucu 120
cuccccugcu cucccaccuc uaccuccacc ccc 153
<210> 94
<211> 128
<212> RNA
<213> Homo sapiens
<400> 94
gccugcuggg cccccgcgga cccaggugcu cugggucacg gucccugucc ccgcaccccc 60
gcuucugucu gccccauugu ggcuccucag gcucucuccc cugcucuccc accucuaccu 120
ccgccccc 128
<210> 95
<211> 116
<212> RNA
<213> Homo sapiens
<400> 95
cgcggaccca ggugcucugg gucacggucc cuguccccgc acccccgcuu cugucugccc 60
cauuguggcu ccuuaggcuc ucuccccugc ucucccaccu uuaccuccac cccuac 116
<210> 96
<211> 190
<212> RNA
<213> Homo sapiens
<400> 96
cugacagcgu gggcaacgcc ugccgccugc ucugaggccc gauccagugg gcaggccaag 60
gccugcuggg cccccgcgga cccaggugcu cugggucacg gucccugucc ccgcaccccc 120
gcuucugucu gccccauugu ggcuccucag gcucucuccc cugcucuccc accucuaccu 180
ccacccccac 190
<210> 97
<211> 191
<212> RNA
<213> Homo sapiens
<400> 97
cugacagcgu gggcaacgcc ugccgccugc ucugaggccc gauccagugg gcaggccaag 60
gccugcuggg cccccgcgga cccaggugcu cugggucacg gucccugucc ccgcaccccc 120
gcuucugucu gccccauugu ggcuccucag gcucucuccc cugcucuccc accucuaccu 180
ccacccccaa c 191
<210> 98
<211> 191
<212> RNA
<213> Homo sapiens
<400> 98
cugacagcgu gggcaacgcc ugccgccugc ucugaggccc gauccagugg gcaggccaag 60
gccugcuggg cccccgcgga cccaggugcu cugggucacg gucccugucc ccgcaccccc 120
gcuucugucu gccccauugu ggcuccucag gcucucuccc cugcucuccc accucuaccu 180
ccacccccaa c 191
<210> 99
<211> 184
<212> RNA
<213> Homo sapiens
<400> 99
cgugggcaac gccugccgcc ugcucugagg cccgauccag ugggcaggcc aaggccugcu 60
gggcccccgc ggacccaggu gcucuggguc acggucccug uccccgcacc cccgcuucug 120
ucugccccau uguggcuccu caggcucucu ccccugcucu cccaccucua ccuccaccca 180
cacc 184
<210> 100
<211> 238
<212> RNA
<213> Homo sapiens
<400> 100
uccugaggga cugggacucc ccuuacagcc augaccuuga caccucagcu gacagcgugg 60
gcaacgccug ccgccugcuc ugaggcccga uccagugggc aggccaaggc cugcugggcc 120
cccgcggacc caggugcucu gggucacggu cccugucccc gcacccccgc uucugucugc 180
cccauugugg cuccucaggc ucucuccccu gcucucccac cucuaccucc acgcccac 238
<210> 101
<211> 234
<212> RNA
<213> Homo sapiens
<400> 101
cugagggacu gggacucccc uuacagccau gaccuugaca ccucagcuga cagcgugggc 60
aacgccugcc gccugcucug aggcccgauc cagugggcag gccaaggccu gcugggcccc 120
cgcggaccca ggugcucugg gucacggucc cuguccccgc acccccgcuu cugucugccc 180
cauuguggcu ccucaggcuc ucuccccugc ucucccaccu cuaccuccac accu 234
<210> 102
<211> 258
<212> RNA
<213> Homo sapiens
<400> 102
ugcggagcca gcuggaggcc auuuuccuga gggacuggga cuccccuuac agccaugacc 60
uugacaccuc agcugacagc gugggcaacg ccugccgccu gcucugaggc ccgauccagu 120
gggcaggcca aggccugcug ggcccccgcg gacccaggug cucuggguca cggucccugu 180
ccccgcaccc ccgcuucugu cugccccauu guggcuccuc aggcucucuc cccugcucuc 240
ccaccucuac cuccaccc 258
<210> 103
<211> 249
<212> RNA
<213> Homo sapiens
<400> 103
cuggaggcca uuuuccugag ggacugggac uccccuuaca gccaugaccu ugacaccuca 60
gcugacagcg ugggcaacgc cugccgccug cucugaggcc cgauccagug ggcaggccaa 120
ggccugcugg gcccccgcgg acccaggugc ucugggucac ggucccuguc cccgcacccc 180
cgcuucuguc ugccccauug uggcuccuca ggcucucucc ccugcucucc caccucuacc 240
uccccccac 249
<210> 104
<211> 226
<212> RNA
<213> Homo sapiens
<400> 104
cugggacucc ccuuacagcc augaccuuga caccucagcu gacagcgugg gcaacgccug 60
ccgccugcuc ugaggcccaa uccagugggc aggccaaggc cugcugggcc cccgcggacc 120
caggugcucu gggucacggu cccugucccc gcacccccgc uucugucugc cccauugugc 180
cuccuuaggc ucucuccccu gcucucccac cucuaccucc accccc 226
<210> 105
<211> 133
<212> RNA
<213> Homo sapiens
<400> 105
gcagcaaugc agcucaaaac gcuuagccua gccacacccc cacgguaaac agcagugauu 60
aacuuuuagc aauaaacgaa aguuuaacua agcuauacua accccagggu uggucaauuu 120
cgugccagcc acc 133
<210> 106
<211> 233
<212> RNA
<213> Homo sapiens
<400> 106
cuuucuauua gcucuuagua agauuacaca ugcaagcauc cccguuccag ugaguucacc 60
cucuaaauca ccacgauaaa aagggacaag caucaagcac gcagcaaugc agcucaaaac 120
gcuuagccua gccacacccc cacgggaaac agcagugauu aaccuuuagc aauaaacgaa 180
aguuuaacua agcuauacua accccagggu uggucaauuu cgugccagcu acc 233
<210> 107
<211> 190
<212> RNA
<213> Homo sapiens
<400> 107
guuccaguga guucacccuc uaaaucacca cgaucaaaag ggacaagcau caagcacgca 60
gcaaugcagc ucaaaacgcu uagccuagcc acacccccac gggaaacagc agugauuaac 120
cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc ccaggguugg ucaauuucgu 180
gccagccacc 190
<210> 108
<211> 154
<212> RNA
<213> Homo sapiens
<400> 108
aaagggacaa gcaucaagca cgcagcaaug cagcucaaaa cgcuuagccu agccacaccc 60
ccacgggaaa cagcagugau uaaccuuuag caauaaacga aaguuuaacu aagcuauacu 120
aaccccaggg uuggucaauu ucgugccagc cacc 154
<210> 109
<211> 158
<212> RNA
<213> Homo sapiens
<400> 109
ucaaaaggga caagcaucaa gcacgcaaca augcagcuca aaaacgcuua gccuagccac 60
acccccacgg gaaacagcag ugauuaaccu uuagcaauaa acgaaaguuu aacuaagcua 120
cacuaacccc aggguugguc aauuucgugc cagccacc 158
<210> 110
<211> 210
<212> RNA
<213> Homo sapiens
<400> 110
uacacaugca agcauccccg uuccagugag uucacccucu aaaucaccac gaucaaaagg 60
gacaagcauc aagcacgcag caaugcagcu caaaaacgcu uagccuagcc acacccccac 120
gggaaacagc agugauuaac cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc 180
ccaggguugg ucaauuucgu gccagccacc 210
<210> 111
<211> 143
<212> RNA
<213> Homo sapiens
<400> 111
caucaagcac gcagcaaugc agcucaaaac gcuuagccua gccacacccc cacgggaaac 60
agcagugauu aaccuuuagc aauaaacgaa aguuuaacua agcuauacua accccagggu 120
uggucaauuu cgugccaacc acc 143
<210> 112
<211> 154
<212> RNA
<213> Homo sapiens
<400> 112
aaagggacaa gcaucaagca cgcagcaaug cagcucaaaa cgcuuagccu agccacaccc 60
ccacgggaaa cagcagugau uaaccuuuag caauaaacga aaguuuaacu aagcuauacu 120
aaccccaggg uuggucaauu ucgugccaac cacc 154
<210> 113
<211> 140
<212> RNA
<213> Homo sapiens
<400> 113
caagcacgca acaaugcagc ucaaaacgcu uagccuagcc acacccccac gggaaacagc 60
agugauuaac cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc ccaggguugg 120
ucaauuucgu gccaaccacc 140
<210> 114
<211> 144
<212> RNA
<213> Homo sapiens
<400> 114
caucaagcac gcagcaaugc agcucaaaac gcuuagccua gccacacccc caugggaaac 60
agcagugauu aaccuuuagc aauaaacgaa aguuuaacua agcuauacua accccagggu 120
uggucaauuu cgugccagcu cacc 144
<210> 115
<211> 142
<212> RNA
<213> Homo sapiens
<400> 115
caagcacgca gcaaugcagc ucaaaacgcu uagccuagcc acacccccac gggaaacagc 60
agugauuaac cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc ccaggguugg 120
ucaauuucgu gccagccaca cc 142
<210> 116
<211> 140
<212> RNA
<213> Homo sapiens
<400> 116
caagcacgca gcaaugcagc ucaaaacgcu uagccuagcc acacccccac gggaaacagc 60
agugauuaac cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc ccaggguugg 120
ucaauuucgu gccagccacc 140
<210> 117
<211> 162
<212> RNA
<213> Homo sapiens
<400> 117
cacgaucaaa agggacaagc aucaagcacg cagcaaugca gcucaaaacg cuuagccuag 60
ccacaccccc acgggaaaca gcagugauua accuuuagca auaaacgaaa guuuaacuaa 120
gcuauacuaa ccccaggguu ggucaauuuc gugccagcca cc 162
<210> 118
<211> 170
<212> RNA
<213> Homo sapiens
<400> 118
uaaaucacca cgaucaaaag ggacaagcau caagcacgca gcaaugcagc ucaaaacgcu 60
uagccuagcc acacccccac gggaaacagc agugauuaac cuuuagcaau aaacgaaagu 120
uuaacuaagc uauacuaacc ccaggguugg ucaauuucgu gccagccacc 170
<210> 119
<211> 236
<212> RNA
<213> Homo sapiens
<400> 119
agccuuucua uuagcucuua guaagauuac acaugcaagc auccccguuc cagugaguuc 60
acccucuaaa ucaccacgau caaaagggac aagcaucaag cacgcagcaa ugcagcucaa 120
aacgcuuagc cuagccacac ccccacggga aacagcagug auuaaccuuu agcaauaaac 180
gaaaguuuaa cuaagcuaua cuaaccccag gguuggucaa uuucgugcca gccacc 236
<210> 120
<211> 151
<212> RNA
<213> Homo sapiens
<400> 120
gggacaagca ucaagcacgc agcaaugcag cucaaaacgc uuagccuagc cacaccccca 60
cgggaaacag cagugauuaa ccuuuagcaa uaaacgaaag uuuaacuaag cuauacuaac 120
cccaggguug gucaauuucg ugccagccac c 151
<210> 121
<211> 151
<212> RNA
<213> Homo sapiens
<400> 121
gggacaagca ucaagcacgc agcaaugcag cucaaaacgc uuagccuagc cacaccccca 60
cgggaaacag cagugauuaa ccuuuagcaa uaaacgaaag uuuaacuaag cuauacuaac 120
cccaggguug gucaauuucg ugccagccac c 151
<210> 122
<211> 253
<212> RNA
<213> Homo sapiens
<400> 122
uucugcccca gcuuugcagg augaaacacu uccccgcuug gcucucauuc uuccacaaga 60
gagaccuuuc uccggaccug guugcuacug guucagcaac ucugcagaaa auguccuccc 120
cuguggcugc cucagcucau gccuuuggcc ugaaguccca gcauugaugg cagccccuca 180
ucuuccaagu uuugugcucc ccuuuaccua acgcuuccug ccucccaugc aucuguacuc 240
cuucugugcc acu 253
<210> 123
<211> 253
<212> RNA
<213> Homo sapiens
<400> 123
uucugcccca gcuuugcagg augaaacacu uccccgcuug gcucucauuc uuccacaaga 60
gagaccuuuc uccggaccug guugcuacug guucagcaac ucugcagaaa auguccuccc 120
cuguggcugc cucagcucau gccuuuggcc ugaaguccca gcauugaugg cagccccuca 180
ucuuccaagu uuugugcucc ccuuuaccua acgcuuccug ccucccaugc aucuguacuc 240
cuucugugcc acu 253
<210> 124
<211> 250
<212> RNA
<213> Homo sapiens
<400> 124
ccccagcuuu gcaggaugaa acacuucccc gcuuggcucu cauucuucca caagagagac 60
cuuucuccgg accugguugc uacugguuca gcaacucugc agaaaauguc cuccccugug 120
gcugccucag cucaugccuu uggccugaag ucccagcauu gauggcagcc ccucaucuuc 180
caaguuuugu gcuccccuuu accuaacgcu uccugccucc caugcaucug uacuccuccu 240
gugccacaaa 250
<210> 125
<211> 250
<212> RNA
<213> Homo sapiens
<400> 125
ccccagcuuu gcaggaugaa acacuucccc gcuuggcucu cauucuucca caagagagac 60
cuuucuccgg accugguugc uacugguuca gcaacucugc agaaaauguc cuccccugug 120
gcugccucag cucaugccuu uggccugaag ucccagcauu gauggcagcc ccucaucuuc 180
caaguuuugu gcuccccuuu accuaacgcu uccugccucc caugcaucug uacuccuccu 240
gugccacaaa 250
<210> 126
<211> 233
<212> RNA
<213> Homo sapiens
<400> 126
cuuugcagga ugaaacacuu ccccgcuugg cucucauucu uccacaagag agaccuuucu 60
ccggaccugg uugcuacugg uucagcaacu cugcagaaaa uguccucccc uguggcugcc 120
ucagcucaug ccuuuggccu gaagucccag cauugauggc agccccucau cuuccaaguu 180
uugugcuccc cuuuaccuaa cgcuuccugc cucccaugca ucuguacucc ucc 233
<210> 127
<211> 228
<212> RNA
<213> Homo sapiens
<400> 127
ugcaggauga aacacuuccc cgcuuggcuc ucauucuucc acaggagaga ccuuucuccg 60
gaccugguug cuacugguuc agcaacucug cagaaaaugu ccuccccugu ggcugccuca 120
gcucaugccu uuggccugaa gucccagcau ugauggcagc cccucaucuu ccaaguuuug 180
ugcuccccuu uaccuaacgc uuccugccuc ccaugcaucu guacucca 228
<210> 128
<211> 280
<212> RNA
<213> Homo sapiens
<400> 128
gugaagauga ccacauucaa ggaagaaccu ucugccccag cuuugcagga ugaaacacuu 60
ccccgcuugg cucuccuucu uccacaagag agaccuuucu ccggaccugg uugcuacugg 120
uucagcagcu cugcagaaaa uguccucccu uguggcugcc ucagcucgua ccuuuggccu 180
gaagucccag cauuaauggc agccccucau cuuccaaguu uugugcuccc cuuuaccuaa 240
ugcuuccugc cucccaugca ucuguacucc ugcugugcca 280
<210> 129
<211> 207
<212> RNA
<213> Homo sapiens
<400> 129
uccacaagag agaccuuucu ccggaccugg cugcuacugg uucagcagcu cugcagaaaa 60
uguccucccu uguggcugcc ucagcucgua ccuuuggccu gaagucccag cauuaauggc 120
agccccucau cuuccaaguu uugugcuccc cuuuaccuaa ugcuuccugc cucccaugca 180
ucuguacucc ugcugugcca caaacac 207
<210> 130
<211> 207
<212> RNA
<213> Homo sapiens
<400> 130
uccacaagag agaccuuucu ccggaccugg cugcuacugg uucagcagcu cugcagaaaa 60
uguccucccu uguggcugcc ucagcucgua ccuuuggccu gaagucccag cauuaauggc 120
agccccucau cuuccaaguu uugugcuccc cuuuaccuaa ugcuuccugc cucccaugca 180
ucuguacucc ugcugugcca caaacac 207
<210> 131
<211> 163
<212> RNA
<213> Homo sapiens
<400> 131
gcuacugguu cagcagcucu gcagaaaaug uccucccuug uggcugccuc agcucguacc 60
uuuggccuga agucccagca uuaauggcag ccccucaucu uccaaguuuu gugcuccccu 120
uuaccuaaug cuuccugccu cccaugcauc uguacuccug cgu 163
<210> 132
<211> 249
<212> RNA
<213> Homo sapiens
<400> 132
agaaccuucu gccccagcuu ugcaggauga aacacuuccc cgcuuggcuc ucauucuucc 60
acaagagaga ccuuucuccg gaccugguug cuacugguuc agcagcucug cagaaaaugu 120
ccucccuugu ggcugccuca gcucguaccu uuggccugaa gucccagcau uaauggcagc 180
cccucaucuu ccaaguuuug ugcuccccuu uaccuaaugc uuccugccuc ccaugcaucu 240
guacuccug 249
<210> 133
<211> 249
<212> RNA
<213> Homo sapiens
<400> 133
agaaccuucu gccccagcuu ugcaggauga aacacuuccc cgcuuggcuc ucauucuucc 60
acaagagaga ccuuucuccg gaccugguug cuacugguuc agcagcucug cagaaaaugu 120
ccucccuugu ggcugccuca gcucguaccu uuggccugaa gucccagcau uaauggcagc 180
cccucaucuu ccaaguuuug ugcuccccuu uaccuaaugc uuccugccuc ccaugcaucu 240
guacuccug 249
<210> 134
<211> 264
<212> RNA
<213> Homo sapiens
<400> 134
ugaagaugac cacauucaag gaagaaccuu cugccccagc uuugcaggau gaaacacuuc 60
cccgcuuggc ucucauucuu ccacaagaga gaccuuucuc cggaccuggu uguucagcag 120
cucugcagaa aauguccucc cuuguggcug ccucagcucg uaccuuuggc cugaaguccc 180
agcauuaaug gcagccccuc aucuuccaag uuuugugcuc cccuuuaccu aaugcuuccu 240
gccucccaug caucuguacu ccug 264
<210> 135
<211> 270
<212> RNA
<213> Homo sapiens
<400> 135
ugaagaugac cacauucaag gaagaaccuu cugccccagc uuugcaggau gaaacacuuc 60
cccgcuuggc ucucauucuu ccacaagaga gaccuuucuc cggaccuggu ugcuacuggu 120
ucagcagcuc ugcagaaaau guccucccuu guggcugccu cagcucguac cuuuggccug 180
aagucccagc auuaauggca gccccucauc uuccaaguuu ugugcucccc uuuaccuaau 240
gcuuccugcc ucccaugcau cuguacuccc 270
<210> 136
<211> 245
<212> RNA
<213> Homo sapiens
<400> 136
ccacauucaa ggaagaaccu ucugccccag cuuugcagga ugaaacacuu ccccgcuugg 60
cucucauucu uccacaagag agaccuuucu ccggaccugg uugcuacugg uucagcagcu 120
cugcagaaaa uguccucccu uguggcugcc ucagcucgua ccuuuggccu gaagucccag 180
cauuaauggc agccccucau cuuccaaguu uugugcuccc cuuuaccuaa ugcuuccugc 240
ccccc 245
<210> 137
<211> 257
<212> RNA
<213> Homo sapiens
<400> 137
gugaagauga ccacauucaa ggaagaaccu ucugccccag cuuugcagga ugaaacacuu 60
ccccgcuugg cucucauucu uccacaagag agaccuuucu ccggaccugg uugcuacugg 120
uucagcagcu cugcagaaaa uguccucccu uguggcugcc ucagcucgua ccuuuggccu 180
gaagucccag cauuaauggc agccccucau cuuccaaguu uugugcuccc cuuuaccuaa 240
ugcuuccugc cccccau 257
<210> 138
<211> 209
<212> RNA
<213> Homo sapiens
<400> 138
agaaccuucu gccccagcuu ugcaggauga aacacuuccc cgcuuggcuc ucauucuucc 60
acaagagaga ccuuucuccg gaccugguug cuacugguuc agcagcucug cagaaaaugu 120
ccucccuugu ggcugccuca gcucguaccu uuggccugaa gucccagcau uaauggcagc 180
cccucaucuu ccaaguuuug ugcuccccc 209
<210> 139
<211> 276
<212> RNA
<213> Homo sapiens
<400> 139
cugaagugaa gaugaccaca uucaaggaag aaccuucugc cccagcuuug caggaugaaa 60
cacuuccccg cuuggcucuc auucuuccac aagagagacc uuucuccgga ccugguugcu 120
acugguucag cagcucugca gaaaaugucc ucccuugugg cugccucagc ucguaccuuu 180
ggccugaagu cccagcauua auggcagccc cucaucuucc aaguuuugug cuccccuuua 240
ccuaaugcuu ccugccuccc augcaucugu acuccu 276
<210> 140
<211> 276
<212> RNA
<213> Homo sapiens
<400> 140
cugaagugaa gaugaccaca uucaaggaag aaccuucugc cccagcuuug caggaugaaa 60
cacuuccccg cuuggcucuc auucuuccac aagagagacc uuucuccgga ccugguugcu 120
acugguucag cagcucugca gaaaaugucc ucccuugugg cugccucagc ucguaccuuu 180
ggccugaagu cccagcauua auggcagccc cucaucuucc aaguuuugug cuccccuuua 240
ccuaaugcuu ccugccuccc augcaucugu acuccu 276
<210> 141
<211> 270
<212> RNA
<213> Homo sapiens
<400> 141
aagaugacca cauucaagga agaaccuucu gccccagcuu ugcaggauga aacacuuccc 60
cgcuuggcuc ucauucuucc acaagagaga ccuuucuccg gaccugguug cuacugguuc 120
agcagcucug cagaaaaugu ccucccuugu ggcugccuca gcucguaccu uuggccugaa 180
gucccagcau uaauggcagc cccucaucuu ccaaguuuug ugcuccccuu uaccuaaugc 240
uuccugccuc ccaugcaucu guacuccugc 270
<210> 142
<211> 211
<212> RNA
<213> Homo sapiens
<400> 142
ccccgcuugg cucucauucu uccacaagag agaccuuucu ccggaccugg uugcuacugg 60
uucagcagcu cugcagaaaa uguccucccu uguggcugcc ucagcucgua ccuuuggccu 120
gaagucccag cauuaauggc agccccucau cuuccaaguu uugugcuccc cuuuaccuaa 180
ugcuuccugc cucccaugca ucuguacucc u 211
<210> 143
<211> 211
<212> RNA
<213> Homo sapiens
<400> 143
ccccgcuugg cucucauucu uccacaagag agaccuuucu ccggaccugg uugcuacugg 60
uucagcagcu cugcagaaaa uguccucccu uguggcugcc ucagcucgua ccuuuggccu 120
gaagucccag cauuaauggc agccccucau cuuccaaguu uugugcuccc cuuuaccuaa 180
ugcuuccugc cucccaugca ucuguacucc u 211
<210> 144
<211> 286
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 144
ugcccguccu caccaagacu gacugccugc ugcuuugcua cugcccgggc ccaugagacu 60
gacuucccac ugcucugccu gccucucccc acugcacugg cacagccccg ccuugccgcu 120
gcugauccau ugccggugug accugcccgu ccucaccaag acugacugcc ugcugcuuug 180
cuacugcccg ggcccaugag acugacuucc cacugcucug ccugccucuc cccacugcac 240
uggcacagcc ccgccuugcc gcugcugauc cauugccggu gugacc 286
<210> 145
<211> 292
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 145
ugcccguccu caccaagacu gacugccugc ugcuuugcua cugcccgggc ccaugagacu 60
gacuucccac ugcucugccu gccucucccc acugcacugg cacagccccg ccuugccgcu 120
gcugauccau ugccggugug accuuccagc cagacacccg ccccccggcc cuggcuaaga 180
aguugcuucc uguugccagc augaccuacc cucgccucuu ugaugccauc cgcugccacc 240
uccuuuugcu ccuggacccu uuagccucuc ugcccuucca cucucugacc cc 292
<210> 146
<211> 298
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 146
ugcccguccu caccaagacu gacugccugc ugcuuugcua cugcccgggc ccaugagacu 60
gacuucccac ugcucugccu gccucucccc acugcacugg cacagccccg ccuugccgcu 120
gcugauccau ugccggugug accgccuugg cuccuccagg aaggcucagg agcccuaccu 180
cccugccauu auagcugcuc cccgccagaa gccugugcca acucucugca uucccugauc 240
uccaucccug uggcugucac ccuuggucac cuccgugcug ucacugccau cucccccc 298
<210> 147
<211> 279
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 147
ugcccguccu caccaagacu gacugccugc ugcuuugcua cugcccgggc ccaugagacu 60
gacuucccac ugcucugccu gccucucccc acugcacugg cacagccccg ccuugccgcu 120
gcugauccau ugccggugug acccugguac ugcaugcacg caaugcuagc ugccccuuuc 180
ccguccuggg uaccccgagu cucccccgac cucggguccc agguaugcuc ccaccuccac 240
cugccccacu caccaccucu gcuaguucca gacaccucc 279
<210> 148
<211> 333
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 148
ugcccguccu caccaagacu gacugccugc ugcuuugcua cugcccgggc ccaugagacu 60
gacuucccac ugcucugccu gccucucccc acugcacugg cacagccccg ccuugccgcu 120
gcugauccau ugccggugug acccugacag cgugggcaac gccugccgcc ugcucugagg 180
cccgauccag ugggcaggcc aaggccugcu gggcccccgc ggacccaggu gcucuggguc 240
acggucccug uccccgcacc cccgcuucug ucugccccau uguggcuccu caggcucucu 300
ccccugcucu cccaccucua ccuccacccc cac 333
<210> 149
<211> 285
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 149
ugcccguccu caccaagacu gacugccugc ugcuuugcua cugcccgggc ccaugagacu 60
gacuucccac ugcucugccu gccucucccc acugcacugg cacagccccg ccuugccgcu 120
gcugauccau ugccggugug accgagagcu cgcuuucuug cuguccaauu ucuauuaaag 180
guuccuuugu ucccuaaguc caacuacuaa acugggggau auuaugaagg gccuugagca 240
ucuggauucu gccuaauaaa aaacauuuau uuucauugcu gcguc 285
<210> 150
<211> 285
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 150
ugcccguccu caccaagacu gacugccugc ugcuuugcua cugcccgggc ccaugagacu 60
gacuucccac ugcucugccu gccucucccc acugcacugg cacagccccg ccuugccgcu 120
gcugauccau ugccggugug acccaagcac gcagcaaugc agcucaaaac gcuuagccua 180
gccacacccc cacgggaaac agcagugauu aaccuuuagc aauaaacgaa aguuuaacua 240
agcuauacua accccagggu uggucaauuu cgugccagcc acacc 285
<210> 151
<211> 376
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 151
ugcccguccu caccaagacu gacugccugc ugcuuugcua cugcccgggc ccaugagacu 60
gacuucccac ugcucugccu gccucucccc acugcacugg cacagccccg ccuugccgcu 120
gcugauccau ugccggugug acccuuugca ggaugaaaca cuuccccgcu uggcucucau 180
ucuuccacaa gagagaccuu ucuccggacc ugguugcuac ugguucagca acucugcaga 240
aaauguccuc cccuguggcu gccucagcuc augccuuugg ccugaagucc cagcauugau 300
ggcagccccu caucuuccaa guuuugugcu ccccuuuacc uaacgcuucc ugccucccau 360
gcaucuguac uccucc 376
<210> 152
<211> 292
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 152
uuccagccag acacccgccc cccggcccug gcuaagaagu ugcuuccugu ugccagcaug 60
accuacccuc gccucuuuga ugccauccgc ugccaccucc uuuugcuccu ggacccuuua 120
gccucucugc ccuuccacuc ucugaccccu gcccguccuc accaagacug acugccugcu 180
gcuuugcuac ugcccgggcc caugagacug acuucccacu gcucugccug ccucucccca 240
cugcacuggc acagccccgc cuugccgcug cugauccauu gccgguguga cc 292
<210> 153
<211> 298
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 153
uuccagccag acacccgccc cccggcccug gcuaagaagu ugcuuccugu ugccagcaug 60
accuacccuc gccucuuuga ugccauccgc ugccaccucc uuuugcuccu ggacccuuua 120
gccucucugc ccuuccacuc ucugaccccu uccagccaga cacccgcccc ccggcccugg 180
cuaagaaguu gcuuccuguu gccagcauga ccuacccucg ccucuuugau gccauccgcu 240
gccaccuccu uuugcuccug gacccuuuag ccucucugcc cuuccacucu cugacccc 298
<210> 154
<211> 304
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 154
uuccagccag acacccgccc cccggcccug gcuaagaagu ugcuuccugu ugccagcaug 60
accuacccuc gccucuuuga ugccauccgc ugccaccucc uuuugcuccu ggacccuuua 120
gccucucugc ccuuccacuc ucugaccccg ccuuggcucc uccaggaagg cucaggagcc 180
cuaccucccu gccauuauag cugcuccccg ccagaagccu gugccaacuc ucugcauucc 240
cugaucucca ucccuguggc ugucacccuu ggucaccucc gugcugucac ugccaucucc 300
cccc 304
<210> 155
<211> 285
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 155
uuccagccag acacccgccc cccggcccug gcuaagaagu ugcuuccugu ugccagcaug 60
accuacccuc gccucuuuga ugccauccgc ugccaccucc uuuugcuccu ggacccuuua 120
gccucucugc ccuuccacuc ucugaccccc ugguacugca ugcacgcaau gcuagcugcc 180
ccuuucccgu ccuggguacc ccgagucucc cccgaccucg ggucccaggu augcucccac 240
cuccaccugc cccacucacc accucugcua guuccagaca ccucc 285
<210> 156
<211> 339
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 156
uuccagccag acacccgccc cccggcccug gcuaagaagu ugcuuccugu ugccagcaug 60
accuacccuc gccucuuuga ugccauccgc ugccaccucc uuuugcuccu ggacccuuua 120
gccucucugc ccuuccacuc ucugaccccc ugacagcgug ggcaacgccu gccgccugcu 180
cugaggcccg auccaguggg caggccaagg ccugcugggc ccccgcggac ccaggugcuc 240
ugggucacgg ucccuguccc cgcacccccg cuucugucug ccccauugug gcuccucagg 300
cucucucccc ugcucuccca ccucuaccuc cacccccac 339
<210> 157
<211> 291
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 157
uuccagccag acacccgccc cccggcccug gcuaagaagu ugcuuccugu ugccagcaug 60
accuacccuc gccucuuuga ugccauccgc ugccaccucc uuuugcuccu ggacccuuua 120
gccucucugc ccuuccacuc ucugaccccg agagcucgcu uucuugcugu ccaauuucua 180
uuaaagguuc cuuuguuccc uaaguccaac uacuaaacug ggggauauua ugaagggccu 240
ugagcaucug gauucugccu aauaaaaaac auuuauuuuc auugcugcgu c 291
<210> 158
<211> 291
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 158
uuccagccag acacccgccc cccggcccug gcuaagaagu ugcuuccugu ugccagcaug 60
accuacccuc gccucuuuga ugccauccgc ugccaccucc uuuugcuccu ggacccuuua 120
gccucucugc ccuuccacuc ucugaccccc aagcacgcag caaugcagcu caaaacgcuu 180
agccuagcca cacccccacg ggaaacagca gugauuaacc uuuagcaaua aacgaaaguu 240
uaacuaagcu auacuaaccc caggguuggu caauuucgug ccagccacac c 291
<210> 159
<211> 382
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 159
uuccagccag acacccgccc cccggcccug gcuaagaagu ugcuuccugu ugccagcaug 60
accuacccuc gccucuuuga ugccauccgc ugccaccucc uuuugcuccu ggacccuuua 120
gccucucugc ccuuccacuc ucugaccccc uuugcaggau gaaacacuuc cccgcuuggc 180
ucucauucuu ccacaagaga gaccuuucuc cggaccuggu ugcuacuggu ucagcaacuc 240
ugcagaaaau guccuccccu guggcugccu cagcucaugc cuuuggccug aagucccagc 300
auugauggca gccccucauc uuccaaguuu ugugcucccc uuuaccuaac gcuuccugcc 360
ucccaugcau cuguacuccu cc 382
<210> 160
<211> 298
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 160
gccuuggcuc cuccaggaag gcucaggagc ccuaccuccc ugccauuaua gcugcucccc 60
gccagaagcc ugugccaacu cucugcauuc ccugaucucc aucccugugg cugucacccu 120
uggucaccuc cgugcuguca cugccaucuc cccccugccc guccucacca agacugacug 180
ccugcugcuu ugcuacugcc cgggcccaug agacugacuu cccacugcuc ugccugccuc 240
uccccacugc acuggcacag ccccgccuug ccgcugcuga uccauugccg gugugacc 298
<210> 161
<211> 304
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 161
gccuuggcuc cuccaggaag gcucaggagc ccuaccuccc ugccauuaua gcugcucccc 60
gccagaagcc ugugccaacu cucugcauuc ccugaucucc aucccugugg cugucacccu 120
uggucaccuc cgugcuguca cugccaucuc cccccuucca gccagacacc cgccccccgg 180
cccuggcuaa gaaguugcuu ccuguugcca gcaugaccua cccucgccuc uuugaugcca 240
uccgcugcca ccuccuuuug cuccuggacc cuuuagccuc ucugcccuuc cacucucuga 300
cccc 304
<210> 162
<211> 310
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 162
gccuuggcuc cuccaggaag gcucaggagc ccuaccuccc ugccauuaua gcugcucccc 60
gccagaagcc ugugccaacu cucugcauuc ccugaucucc aucccugugg cugucacccu 120
uggucaccuc cgugcuguca cugccaucuc cccccgccuu ggcuccucca ggaaggcuca 180
ggagcccuac cucccugcca uuauagcugc uccccgccag aagccugugc caacucucug 240
cauucccuga ucuccauccc uguggcuguc acccuugguc accuccgugc ugucacugcc 300
aucucccccc 310
<210> 163
<211> 291
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 163
gccuuggcuc cuccaggaag gcucaggagc ccuaccuccc ugccauuaua gcugcucccc 60
gccagaagcc ugugccaacu cucugcauuc ccugaucucc aucccugugg cugucacccu 120
uggucaccuc cgugcuguca cugccaucuc ccccccuggu acugcaugca cgcaaugcua 180
gcugccccuu ucccguccug gguaccccga gucucccccg accucggguc ccagguaugc 240
ucccaccucc accugcccca cucaccaccu cugcuaguuc cagacaccuc c 291
<210> 164
<211> 345
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 164
gccuuggcuc cuccaggaag gcucaggagc ccuaccuccc ugccauuaua gcugcucccc 60
gccagaagcc ugugccaacu cucugcauuc ccugaucucc aucccugugg cugucacccu 120
uggucaccuc cgugcuguca cugccaucuc ccccccugac agcgugggca acgccugccg 180
ccugcucuga ggcccgaucc agugggcagg ccaaggccug cugggccccc gcggacccag 240
gugcucuggg ucacgguccc uguccccgca cccccgcuuc ugucugcccc auuguggcuc 300
cucaggcucu cuccccugcu cucccaccuc uaccuccacc cccac 345
<210> 165
<211> 297
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 165
gccuuggcuc cuccaggaag gcucaggagc ccuaccuccc ugccauuaua gcugcucccc 60
gccagaagcc ugugccaacu cucugcauuc ccugaucucc aucccugugg cugucacccu 120
uggucaccuc cgugcuguca cugccaucuc cccccgagag cucgcuuucu ugcuguccaa 180
uuucuauuaa agguuccuuu guucccuaag uccaacuacu aaacuggggg auauuaugaa 240
gggccuugag caucuggauu cugccuaaua aaaaacauuu auuuucauug cugcguc 297
<210> 166
<211> 297
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 166
gccuuggcuc cuccaggaag gcucaggagc ccuaccuccc ugccauuaua gcugcucccc 60
gccagaagcc ugugccaacu cucugcauuc ccugaucucc aucccugugg cugucacccu 120
uggucaccuc cgugcuguca cugccaucuc ccccccaagc acgcagcaau gcagcucaaa 180
acgcuuagcc uagccacacc cccacgggaa acagcaguga uuaaccuuua gcaauaaacg 240
aaaguuuaac uaagcuauac uaaccccagg guuggucaau uucgugccag ccacacc 297
<210> 167
<211> 388
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 167
gccuuggcuc cuccaggaag gcucaggagc ccuaccuccc ugccauuaua gcugcucccc 60
gccagaagcc ugugccaacu cucugcauuc ccugaucucc aucccugugg cugucacccu 120
uggucaccuc cgugcuguca cugccaucuc ccccccuuug caggaugaaa cacuuccccg 180
cuuggcucuc auucuuccac aagagagacc uuucuccgga ccugguugcu acugguucag 240
caacucugca gaaaaugucc uccccugugg cugccucagc ucaugccuuu ggccugaagu 300
cccagcauug auggcagccc cucaucuucc aaguuuugug cuccccuuua ccuaacgcuu 360
ccugccuccc augcaucugu acuccucc 388
<210> 168
<211> 279
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 168
cugguacugc augcacgcaa ugcuagcugc cccuuucccg uccuggguac cccgagucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accuccugcc cguccucacc aagacugacu gccugcugcu uugcuacugc 180
ccgggcccau gagacugacu ucccacugcu cugccugccu cuccccacug cacuggcaca 240
gccccgccuu gccgcugcug auccauugcc ggugugacc 279
<210> 169
<211> 285
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 169
cugguacugc augcacgcaa ugcuagcugc cccuuucccg uccuggguac cccgagucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accuccuucc agccagacac ccgccccccg gcccuggcua agaaguugcu 180
uccuguugcc agcaugaccu acccucgccu cuuugaugcc auccgcugcc accuccuuuu 240
gcuccuggac ccuuuagccu cucugcccuu ccacucucug acccc 285
<210> 170
<211> 291
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 170
cugguacugc augcacgcaa ugcuagcugc cccuuucccg uccuggguac cccgagucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accuccgccu uggcuccucc aggaaggcuc aggagcccua ccucccugcc 180
auuauagcug cuccccgcca gaagccugug ccaacucucu gcauucccug aucuccaucc 240
cuguggcugu cacccuuggu caccuccgug cugucacugc caucuccccc c 291
<210> 171
<211> 272
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 171
cugguacugc augcacgcaa ugcuagcugc cccuuucccg uccuggguac cccgagucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccugg uacugcaugc acgcaaugcu agcugccccu uucccguccu 180
ggguaccccg agucuccccc gaccucgggu cccagguaug cucccaccuc caccugcccc 240
acucaccacc ucugcuaguu ccagacaccu cc 272
<210> 172
<211> 326
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 172
cugguacugc augcacgcaa ugcuagcugc cccuuucccg uccuggguac cccgagucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccuga cagcgugggc aacgccugcc gccugcucug aggcccgauc 180
cagugggcag gccaaggccu gcugggcccc cgcggaccca ggugcucugg gucacggucc 240
cuguccccgc acccccgcuu cugucugccc cauuguggcu ccucaggcuc ucuccccugc 300
ucucccaccu cuaccuccac ccccac 326
<210> 173
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 173
cugguacugc augcacgcaa ugcuagcugc cccuuucccg uccuggguac cccgagucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accuccgaga gcucgcuuuc uugcugucca auuucuauua aagguuccuu 180
uguucccuaa guccaacuac uaaacugggg gauauuauga agggccuuga gcaucuggau 240
ucugccuaau aaaaaacauu uauuuucauu gcugcguc 278
<210> 174
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 174
cugguacugc augcacgcaa ugcuagcugc cccuuucccg uccuggguac cccgagucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccaag cacgcagcaa ugcagcucaa aacgcuuagc cuagccacac 180
ccccacggga aacagcagug auuaaccuuu agcaauaaac gaaaguuuaa cuaagcuaua 240
cuaaccccag gguuggucaa uuucgugcca gccacacc 278
<210> 175
<211> 369
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 175
cugguacugc augcacgcaa ugcuagcugc cccuuucccg uccuggguac cccgagucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccuuu gcaggaugaa acacuucccc gcuuggcucu cauucuucca 180
caagagagac cuuucuccgg accugguugc uacugguuca gcaacucugc agaaaauguc 240
cuccccugug gcugccucag cucaugccuu uggccugaag ucccagcauu gauggcagcc 300
ccucaucuuc caaguuuugu gcuccccuuu accuaacgcu uccugccucc caugcaucug 360
uacuccucc 369
<210> 176
<211> 333
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 176
cugacagcgu gggcaacgcc ugccgccugc ucugaggccc gauccagugg gcaggccaag 60
gccugcuggg cccccgcgga cccaggugcu cugggucacg gucccugucc ccgcaccccc 120
gcuucugucu gccccauugu ggcuccucag gcucucuccc cugcucuccc accucuaccu 180
ccacccccac ugcccguccu caccaagacu gacugccugc ugcuuugcua cugcccgggc 240
ccaugagacu gacuucccac ugcucugccu gccucucccc acugcacugg cacagccccg 300
ccuugccgcu gcugauccau ugccggugug acc 333
<210> 177
<211> 339
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 177
cugacagcgu gggcaacgcc ugccgccugc ucugaggccc gauccagugg gcaggccaag 60
gccugcuggg cccccgcgga cccaggugcu cugggucacg gucccugucc ccgcaccccc 120
gcuucugucu gccccauugu ggcuccucag gcucucuccc cugcucuccc accucuaccu 180
ccacccccac uuccagccag acacccgccc cccggcccug gcuaagaagu ugcuuccugu 240
ugccagcaug accuacccuc gccucuuuga ugccauccgc ugccaccucc uuuugcuccu 300
ggacccuuua gccucucugc ccuuccacuc ucugacccc 339
<210> 178
<211> 345
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 178
cugacagcgu gggcaacgcc ugccgccugc ucugaggccc gauccagugg gcaggccaag 60
gccugcuggg cccccgcgga cccaggugcu cugggucacg gucccugucc ccgcaccccc 120
gcuucugucu gccccauugu ggcuccucag gcucucuccc cugcucuccc accucuaccu 180
ccacccccac gccuuggcuc cuccaggaag gcucaggagc ccuaccuccc ugccauuaua 240
gcugcucccc gccagaagcc ugugccaacu cucugcauuc ccugaucucc aucccugugg 300
cugucacccu uggucaccuc cgugcuguca cugccaucuc ccccc 345
<210> 179
<211> 326
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 179
cugacagcgu gggcaacgcc ugccgccugc ucugaggccc gauccagugg gcaggccaag 60
gccugcuggg cccccgcgga cccaggugcu cugggucacg gucccugucc ccgcaccccc 120
gcuucugucu gccccauugu ggcuccucag gcucucuccc cugcucuccc accucuaccu 180
ccacccccac cugguacugc augcacgcaa ugcuagcugc cccuuucccg uccuggguac 240
cccgagucuc ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac 300
caccucugcu aguuccagac accucc 326
<210> 180
<211> 380
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 180
cugacagcgu gggcaacgcc ugccgccugc ucugaggccc gauccagugg gcaggccaag 60
gccugcuggg cccccgcgga cccaggugcu cugggucacg gucccugucc ccgcaccccc 120
gcuucugucu gccccauugu ggcuccucag gcucucuccc cugcucuccc accucuaccu 180
ccacccccac cugacagcgu gggcaacgcc ugccgccugc ucugaggccc gauccagugg 240
gcaggccaag gccugcuggg cccccgcgga cccaggugcu cugggucacg gucccugucc 300
ccgcaccccc gcuucugucu gccccauugu ggcuccucag gcucucuccc cugcucuccc 360
accucuaccu ccacccccac 380
<210> 181
<211> 332
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 181
cugacagcgu gggcaacgcc ugccgccugc ucugaggccc gauccagugg gcaggccaag 60
gccugcuggg cccccgcgga cccaggugcu cugggucacg gucccugucc ccgcaccccc 120
gcuucugucu gccccauugu ggcuccucag gcucucuccc cugcucuccc accucuaccu 180
ccacccccac gagagcucgc uuucuugcug uccaauuucu auuaaagguu ccuuuguucc 240
cuaaguccaa cuacuaaacu gggggauauu augaagggcc uugagcaucu ggauucugcc 300
uaauaaaaaa cauuuauuuu cauugcugcg uc 332
<210> 182
<211> 332
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 182
cugacagcgu gggcaacgcc ugccgccugc ucugaggccc gauccagugg gcaggccaag 60
gccugcuggg cccccgcgga cccaggugcu cugggucacg gucccugucc ccgcaccccc 120
gcuucugucu gccccauugu ggcuccucag gcucucuccc cugcucuccc accucuaccu 180
ccacccccac caagcacgca gcaaugcagc ucaaaacgcu uagccuagcc acacccccac 240
gggaaacagc agugauuaac cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc 300
ccaggguugg ucaauuucgu gccagccaca cc 332
<210> 183
<211> 423
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 183
cugacagcgu gggcaacgcc ugccgccugc ucugaggccc gauccagugg gcaggccaag 60
gccugcuggg cccccgcgga cccaggugcu cugggucacg gucccugucc ccgcaccccc 120
gcuucugucu gccccauugu ggcuccucag gcucucuccc cugcucuccc accucuaccu 180
ccacccccac cuuugcagga ugaaacacuu ccccgcuugg cucucauucu uccacaagag 240
agaccuuucu ccggaccugg uugcuacugg uucagcaacu cugcagaaaa uguccucccc 300
uguggcugcc ucagcucaug ccuuuggccu gaagucccag cauugauggc agccccucau 360
cuuccaaguu uugugcuccc cuuuaccuaa cgcuuccugc cucccaugca ucuguacucc 420
ucc 423
<210> 184
<211> 285
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 184
gagagcucgc uuucuugcug uccaauuucu auuaaagguu ccuuuguucc cuaaguccaa 60
cuacuaaacu gggggauauu augaagggcc uugagcaucu ggauucugcc uaauaaaaaa 120
cauuuauuuu cauugcugcg ucugcccguc cucaccaaga cugacugccu gcugcuuugc 180
uacugcccgg gcccaugaga cugacuuccc acugcucugc cugccucucc ccacugcacu 240
ggcacagccc cgccuugccg cugcugaucc auugccggug ugacc 285
<210> 185
<211> 291
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 185
gagagcucgc uuucuugcug uccaauuucu auuaaagguu ccuuuguucc cuaaguccaa 60
cuacuaaacu gggggauauu augaagggcc uugagcaucu ggauucugcc uaauaaaaaa 120
cauuuauuuu cauugcugcg ucuuccagcc agacacccgc cccccggccc uggcuaagaa 180
guugcuuccu guugccagca ugaccuaccc ucgccucuuu gaugccaucc gcugccaccu 240
ccuuuugcuc cuggacccuu uagccucucu gcccuuccac ucucugaccc c 291
<210> 186
<211> 297
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 186
gagagcucgc uuucuugcug uccaauuucu auuaaagguu ccuuuguucc cuaaguccaa 60
cuacuaaacu gggggauauu augaagggcc uugagcaucu ggauucugcc uaauaaaaaa 120
cauuuauuuu cauugcugcg ucgccuuggc uccuccagga aggcucagga gcccuaccuc 180
ccugccauua uagcugcucc ccgccagaag ccugugccaa cucucugcau ucccugaucu 240
ccaucccugu ggcugucacc cuuggucacc uccgugcugu cacugccauc ucccccc 297
<210> 187
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 187
gagagcucgc uuucuugcug uccaauuucu auuaaagguu ccuuuguucc cuaaguccaa 60
cuacuaaacu gggggauauu augaagggcc uugagcaucu ggauucugcc uaauaaaaaa 120
cauuuauuuu cauugcugcg uccugguacu gcaugcacgc aaugcuagcu gccccuuucc 180
cguccugggu accccgaguc ucccccgacc ucggguccca gguaugcucc caccuccacc 240
ugccccacuc accaccucug cuaguuccag acaccucc 278
<210> 188
<211> 332
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 188
gagagcucgc uuucuugcug uccaauuucu auuaaagguu ccuuuguucc cuaaguccaa 60
cuacuaaacu gggggauauu augaagggcc uugagcaucu ggauucugcc uaauaaaaaa 120
cauuuauuuu cauugcugcg uccugacagc gugggcaacg ccugccgccu gcucugaggc 180
ccgauccagu gggcaggcca aggccugcug ggcccccgcg gacccaggug cucuggguca 240
cggucccugu ccccgcaccc ccgcuucugu cugccccauu guggcuccuc aggcucucuc 300
cccugcucuc ccaccucuac cuccaccccc ac 332
<210> 189
<211> 284
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 189
gagagcucgc uuucuugcug uccaauuucu auuaaagguu ccuuuguucc cuaaguccaa 60
cuacuaaacu gggggauauu augaagggcc uugagcaucu ggauucugcc uaauaaaaaa 120
cauuuauuuu cauugcugcg ucgagagcuc gcuuucuugc uguccaauuu cuauuaaagg 180
uuccuuuguu cccuaagucc aacuacuaaa cugggggaua uuaugaaggg ccuugagcau 240
cuggauucug ccuaauaaaa aacauuuauu uucauugcug cguc 284
<210> 190
<211> 284
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 190
gagagcucgc uuucuugcug uccaauuucu auuaaagguu ccuuuguucc cuaaguccaa 60
cuacuaaacu gggggauauu augaagggcc uugagcaucu ggauucugcc uaauaaaaaa 120
cauuuauuuu cauugcugcg uccaagcacg cagcaaugca gcucaaaacg cuuagccuag 180
ccacaccccc acgggaaaca gcagugauua accuuuagca auaaacgaaa guuuaacuaa 240
gcuauacuaa ccccaggguu ggucaauuuc gugccagcca cacc 284
<210> 191
<211> 375
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 191
gagagcucgc uuucuugcug uccaauuucu auuaaagguu ccuuuguucc cuaaguccaa 60
cuacuaaacu gggggauauu augaagggcc uugagcaucu ggauucugcc uaauaaaaaa 120
cauuuauuuu cauugcugcg uccuuugcag gaugaaacac uuccccgcuu ggcucucauu 180
cuuccacaag agagaccuuu cuccggaccu gguugcuacu gguucagcaa cucugcagaa 240
aauguccucc ccuguggcug ccucagcuca ugccuuuggc cugaaguccc agcauugaug 300
gcagccccuc aucuuccaag uuuugugcuc cccuuuaccu aacgcuuccu gccucccaug 360
caucuguacu ccucc 375
<210> 192
<211> 285
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 192
caagcacgca gcaaugcagc ucaaaacgcu uagccuagcc acacccccac gggaaacagc 60
agugauuaac cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc ccaggguugg 120
ucaauuucgu gccagccaca ccugcccguc cucaccaaga cugacugccu gcugcuuugc 180
uacugcccgg gcccaugaga cugacuuccc acugcucugc cugccucucc ccacugcacu 240
ggcacagccc cgccuugccg cugcugaucc auugccggug ugacc 285
<210> 193
<211> 291
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 193
caagcacgca gcaaugcagc ucaaaacgcu uagccuagcc acacccccac gggaaacagc 60
agugauuaac cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc ccaggguugg 120
ucaauuucgu gccagccaca ccuuccagcc agacacccgc cccccggccc uggcuaagaa 180
guugcuuccu guugccagca ugaccuaccc ucgccucuuu gaugccaucc gcugccaccu 240
ccuuuugcuc cuggacccuu uagccucucu gcccuuccac ucucugaccc c 291
<210> 194
<211> 297
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 194
caagcacgca gcaaugcagc ucaaaacgcu uagccuagcc acacccccac gggaaacagc 60
agugauuaac cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc ccaggguugg 120
ucaauuucgu gccagccaca ccgccuuggc uccuccagga aggcucagga gcccuaccuc 180
ccugccauua uagcugcucc ccgccagaag ccugugccaa cucucugcau ucccugaucu 240
ccaucccugu ggcugucacc cuuggucacc uccgugcugu cacugccauc ucccccc 297
<210> 195
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 195
caagcacgca gcaaugcagc ucaaaacgcu uagccuagcc acacccccac gggaaacagc 60
agugauuaac cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc ccaggguugg 120
ucaauuucgu gccagccaca cccugguacu gcaugcacgc aaugcuagcu gccccuuucc 180
cguccugggu accccgaguc ucccccgacc ucggguccca gguaugcucc caccuccacc 240
ugccccacuc accaccucug cuaguuccag acaccucc 278
<210> 196
<211> 332
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 196
caagcacgca gcaaugcagc ucaaaacgcu uagccuagcc acacccccac gggaaacagc 60
agugauuaac cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc ccaggguugg 120
ucaauuucgu gccagccaca cccugacagc gugggcaacg ccugccgccu gcucugaggc 180
ccgauccagu gggcaggcca aggccugcug ggcccccgcg gacccaggug cucuggguca 240
cggucccugu ccccgcaccc ccgcuucugu cugccccauu guggcuccuc aggcucucuc 300
cccugcucuc ccaccucuac cuccaccccc ac 332
<210> 197
<211> 284
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 197
caagcacgca gcaaugcagc ucaaaacgcu uagccuagcc acacccccac gggaaacagc 60
agugauuaac cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc ccaggguugg 120
ucaauuucgu gccagccaca ccgagagcuc gcuuucuugc uguccaauuu cuauuaaagg 180
uuccuuuguu cccuaagucc aacuacuaaa cugggggaua uuaugaaggg ccuugagcau 240
cuggauucug ccuaauaaaa aacauuuauu uucauugcug cguc 284
<210> 198
<211> 284
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 198
caagcacgca gcaaugcagc ucaaaacgcu uagccuagcc acacccccac gggaaacagc 60
agugauuaac cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc ccaggguugg 120
ucaauuucgu gccagccaca cccaagcacg cagcaaugca gcucaaaacg cuuagccuag 180
ccacaccccc acgggaaaca gcagugauua accuuuagca auaaacgaaa guuuaacuaa 240
gcuauacuaa ccccaggguu ggucaauuuc gugccagcca cacc 284
<210> 199
<211> 375
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 199
caagcacgca gcaaugcagc ucaaaacgcu uagccuagcc acacccccac gggaaacagc 60
agugauuaac cuuuagcaau aaacgaaagu uuaacuaagc uauacuaacc ccaggguugg 120
ucaauuucgu gccagccaca cccuuugcag gaugaaacac uuccccgcuu ggcucucauu 180
cuuccacaag agagaccuuu cuccggaccu gguugcuacu gguucagcaa cucugcagaa 240
aauguccucc ccuguggcug ccucagcuca ugccuuuggc cugaaguccc agcauugaug 300
gcagccccuc aucuuccaag uuuugugcuc cccuuuaccu aacgcuuccu gccucccaug 360
caucuguacu ccucc 375
<210> 200
<211> 376
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 200
cuuugcagga ugaaacacuu ccccgcuugg cucucauucu uccacaagag agaccuuucu 60
ccggaccugg uugcuacugg uucagcaacu cugcagaaaa uguccucccc uguggcugcc 120
ucagcucaug ccuuuggccu gaagucccag cauugauggc agccccucau cuuccaaguu 180
uugugcuccc cuuuaccuaa cgcuuccugc cucccaugca ucuguacucc uccugcccgu 240
ccucaccaag acugacugcc ugcugcuuug cuacugcccg ggcccaugag acugacuucc 300
cacugcucug ccugccucuc cccacugcac uggcacagcc ccgccuugcc gcugcugauc 360
cauugccggu gugacc 376
<210> 201
<211> 382
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 201
cuuugcagga ugaaacacuu ccccgcuugg cucucauucu uccacaagag agaccuuucu 60
ccggaccugg uugcuacugg uucagcaacu cugcagaaaa uguccucccc uguggcugcc 120
ucagcucaug ccuuuggccu gaagucccag cauugauggc agccccucau cuuccaaguu 180
uugugcuccc cuuuaccuaa cgcuuccugc cucccaugca ucuguacucc uccuuccagc 240
cagacacccg ccccccggcc cuggcuaaga aguugcuucc uguugccagc augaccuacc 300
cucgccucuu ugaugccauc cgcugccacc uccuuuugcu ccuggacccu uuagccucuc 360
ugcccuucca cucucugacc cc 382
<210> 202
<211> 388
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 202
cuuugcagga ugaaacacuu ccccgcuugg cucucauucu uccacaagag agaccuuucu 60
ccggaccugg uugcuacugg uucagcaacu cugcagaaaa uguccucccc uguggcugcc 120
ucagcucaug ccuuuggccu gaagucccag cauugauggc agccccucau cuuccaaguu 180
uugugcuccc cuuuaccuaa cgcuuccugc cucccaugca ucuguacucc uccgccuugg 240
cuccuccagg aaggcucagg agcccuaccu cccugccauu auagcugcuc cccgccagaa 300
gccugugcca acucucugca uucccugauc uccaucccug uggcugucac ccuuggucac 360
cuccgugcug ucacugccau cucccccc 388
<210> 203
<211> 369
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 203
cuuugcagga ugaaacacuu ccccgcuugg cucucauucu uccacaagag agaccuuucu 60
ccggaccugg uugcuacugg uucagcaacu cugcagaaaa uguccucccc uguggcugcc 120
ucagcucaug ccuuuggccu gaagucccag cauugauggc agccccucau cuuccaaguu 180
uugugcuccc cuuuaccuaa cgcuuccugc cucccaugca ucuguacucc ucccugguac 240
ugcaugcacg caaugcuagc ugccccuuuc ccguccuggg uaccccgagu cucccccgac 300
cucggguccc agguaugcuc ccaccuccac cugccccacu caccaccucu gcuaguucca 360
gacaccucc 369
<210> 204
<211> 423
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 204
cuuugcagga ugaaacacuu ccccgcuugg cucucauucu uccacaagag agaccuuucu 60
ccggaccugg uugcuacugg uucagcaacu cugcagaaaa uguccucccc uguggcugcc 120
ucagcucaug ccuuuggccu gaagucccag cauugauggc agccccucau cuuccaaguu 180
uugugcuccc cuuuaccuaa cgcuuccugc cucccaugca ucuguacucc ucccugacag 240
cgugggcaac gccugccgcc ugcucugagg cccgauccag ugggcaggcc aaggccugcu 300
gggcccccgc ggacccaggu gcucuggguc acggucccug uccccgcacc cccgcuucug 360
ucugccccau uguggcuccu caggcucucu ccccugcucu cccaccucua ccuccacccc 420
cac 423
<210> 205
<211> 375
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 205
cuuugcagga ugaaacacuu ccccgcuugg cucucauucu uccacaagag agaccuuucu 60
ccggaccugg uugcuacugg uucagcaacu cugcagaaaa uguccucccc uguggcugcc 120
ucagcucaug ccuuuggccu gaagucccag cauugauggc agccccucau cuuccaaguu 180
uugugcuccc cuuuaccuaa cgcuuccugc cucccaugca ucuguacucc uccgagagcu 240
cgcuuucuug cuguccaauu ucuauuaaag guuccuuugu ucccuaaguc caacuacuaa 300
acugggggau auuaugaagg gccuugagca ucuggauucu gccuaauaaa aaacauuuau 360
uuucauugcu gcguc 375
<210> 206
<211> 375
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 206
cuuugcagga ugaaacacuu ccccgcuugg cucucauucu uccacaagag agaccuuucu 60
ccggaccugg uugcuacugg uucagcaacu cugcagaaaa uguccucccc uguggcugcc 120
ucagcucaug ccuuuggccu gaagucccag cauugauggc agccccucau cuuccaaguu 180
uugugcuccc cuuuaccuaa cgcuuccugc cucccaugca ucuguacucc ucccaagcac 240
gcagcaaugc agcucaaaac gcuuagccua gccacacccc cacgggaaac agcagugauu 300
aaccuuuagc aauaaacgaa aguuuaacua agcuauacua accccagggu uggucaauuu 360
cgugccagcc acacc 375
<210> 207
<211> 466
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 207
cuuugcagga ugaaacacuu ccccgcuugg cucucauucu uccacaagag agaccuuucu 60
ccggaccugg uugcuacugg uucagcaacu cugcagaaaa uguccucccc uguggcugcc 120
ucagcucaug ccuuuggccu gaagucccag cauugauggc agccccucau cuuccaaguu 180
uugugcuccc cuuuaccuaa cgcuuccugc cucccaugca ucuguacucc ucccuuugca 240
ggaugaaaca cuuccccgcu uggcucucau ucuuccacaa gagagaccuu ucuccggacc 300
ugguugcuac ugguucagca acucugcaga aaauguccuc cccuguggcu gccucagcuc 360
augccuuugg ccugaagucc cagcauugau ggcagccccu caucuuccaa guuuugugcu 420
ccccuuuacc uaacgcuucc ugccucccau gcaucuguac uccucc 466
<210> 208
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 208
cugguacugc auggacgcaa ugcuagcugc cccuuucccg uccuggguac cccgagucac 60
ccccgaccuc gggucccagg uaucguccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccaag cacgcagcaa ugcagcucaa aacgcuuagc auagccacac 180
ccccacggga aacagcagug auuaaccuuu agcaauaauc gaauguuuaa cuaagcuaua 240
cuaaccccag gguuggucaa uuucgugcca gccacacc 278
<210> 209
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 209
cucguacugc auggacgcaa ugcuagcugc cccuuucccg uccuggguac cccgagucac 60
caccgaccuc gggucccagg uaucguccca ccuccacgug ccccacucac caccucugcu 120
aguuccagac accucccaag cacgcagcaa ugcagcucaa aacgcuuagc auagccacac 180
ccccacggga aacaguagug auuaaccuuu agcaauaauc gaaugucuaa cuaagcuaua 240
cuaaccccag gguugaucaa uuacgugcca gccacacc 278
<210> 210
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 210
cucguacugc auggacgcaa ugcuagcugc cccuuucccg uccuggguac cccgagucac 60
caccgaccuc gggucccagg uaucguccca ccuccacgug ccccacucac caccuuugcu 120
aguuccagac accucccaug cacgcagcaa ugcagaucaa aacgcuuagc auagccacac 180
ccccacggga aacaguagug auuaaccuuu agcaauaauc gaaugucuaa cuaagcuaua 240
cuaaccccag gguugaucaa uuacgugcca gccacacc 278
<210> 211
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 211
gucguacugc auggacgcaa ugcuagcagc accuuucccg uccuggguac cccgagucac 60
caccgaccuc gggucccagg uaucguccca ccuccacgug ccccacccac caccuuugcu 120
aguuccagag accucccaug cacgcagcaa ugcagaucaa aacgcuuagc auagccacac 180
cgccacggga aacaguagug aucaaccuuu agcuauaauc gaaugucuaa cuaagcuauu 240
cuaaccacag gguugaucaa uuacgugcca gccagacc 278
<210> 212
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 212
cugguacugc augcacgcaa ugcuagcugc cccaaagggc uccuggguac cccgagucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccaag cacgcagcaa ugcagcucaa aacgcuuagc cuagccacac 180
ccccacggga aacagcagug auuaaccuuu agcaauaaac gaaaguuuaa cuaagcuaua 240
cuaaccccag gguuggucaa uuucgugcca gccacacc 278
<210> 213
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 213
cugguacugc augcacgcaa ugcuagcugc cccuuucccg ugguccguac cccgagucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccaag cacgcagcaa ugcagcucaa aacgcuuagc cuagccacac 180
ccccacggga aacagcagug auuaaccuuu agcaauaaac gaaaguuuaa cuaagcuaua 240
cuaaccccag gguuggucaa uuucgugcca gccacacc 278
<210> 214
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 214
cugguacugc augcacgcaa ugcuagcugc cccuuucccg uggaccguac ggcgagucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccaag cacgcagcaa ugcagcucaa aacgcuuagc cuagccacac 180
ccccacggga aacagcagug auuaaccuuu agcaauaaac gaaaguuuaa cuaagcuaua 240
cuaaccccag gguuggucaa uuucgugcca gccacacc 278
<210> 215
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 215
cugguacugc augcacgcaa ugcuagcugc cccuuugccg uggaccguac gggcugucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccaag cacgcagcaa ugcagcucaa aacgcuuagc cuagccacac 180
ccccacggga aacagcagug auuaaccuuu agcaauaaac gaaaguuuaa cuaagcuaua 240
cuaaccccag gguuggucaa uuucgugcca gccacacc 278
<210> 216
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 216
cugguacugc augcacgcaa ugcuagcugc cccuuugggc uggaccguac gggcugucuc 60
ccccgaccuc gggucccagg uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccaag cacgcagcaa ugcagcucaa aacgcuuagc cuagccacac 180
ccccacggga aacagcagug auuaaccuuu agcaauaaac gaaaguuuaa cuaagcuaua 240
cuaaccccag gguuggucaa uuucgugcca gccacacc 278
<210> 217
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 217
cugguacugc augcacgcaa ugcuagcugc cccuuucccg ugguccguac cccgagucuc 60
ccccgaccuc gggucggacc uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccaag cacgcagcaa ugcagcucaa aacgcuuagc cuagccacac 180
ccccacggga aacagcagug auuaaccuuu agcaauaaac gaaaguuuaa cuaagcuaua 240
cuaaccccag gguuggucaa uuucgugcca gccacacc 278
<210> 218
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 218
cugguacugc augcacgcaa ugcuagcugc cccuuucccg uggaccguac ggcgagucuc 60
ccccgaccuc gccucggucc uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccaag cacgcagcaa ugcagcucaa aacgcuuagc cuagccacac 180
ccccacggga aacagcagug auuaaccuuu agcaauaaac gaaaguuuaa cuaagcuaua 240
cuaaccccag gguuggucaa uuucgugcca gccacacc 278
<210> 219
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 219
cugguacugc augcacgcaa ugcuagcugc cccuuugccg uggaccguac gggcugucuc 60
ccccgaccag cccucggucc uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccaag cacgcagcaa ugcagcucaa aacgcuuagc cuagccacac 180
ccccacggca aacagcagug auuaaccuuu agcaauaaac gaaaguuuaa cuaagcuaua 240
cuaaccccag gguuggucaa uuucgugcca gccacacc 278
<210> 220
<211> 278
<212> RNA
<213> Artificial Sequence
<220>
<223> Combination of 3'-UTR sequence elements
<400> 220
cugguacugc augcacgcaa ugcuagcugc cccuuugggc uggaccguac gggcugucuc 60
ccccgaccag cccucggucc uaugcuccca ccuccaccug ccccacucac caccucugcu 120
aguuccagac accucccaag cacgcagcaa ugcagcucaa aacgcuuagc cuagccacac 180
ccccagccca aacagcagug auuaaccuuu agcaauaaac gaaaguuuaa cuaagcuaua 240
cuaaccccag gguuggucaa uuucgugcca gccacacc 278
<210> 221
<211> 142
<212> RNA
<213> Artificial Sequence
<220>
<223> 3'-UTR sequence element
<400> 221
gagagcucgc uuucuugcug uccaauuucu auuaaagguu ccuuuguucc cuaaguccaa 60
cuacuaaacu gggggauauu augaagggcc uugagcaucu ggauucugcc uaauaaaaaa 120
cauuuauuuu cauugcugcg uc 142
<---
Реферат
Изобретение относится к биотехнологии. Описана молекула нуклеиновой кислоты, содержащая, в направлении транскрипции 5' → 3': (a) промотор, (b) транскрибируемую последовательность нуклеиновой кислоты или последовательность нуклеиновой кислоты для введения транскрибируемой последовательности нуклеиновой кислоты и (c) последовательность нуклеиновой кислоты, которая, будучи транскрибированной под контролем промотора (a), кодирует 3'-нетранслируемую область транскрипта, которая в естественных условиях не связана с нуклеиновой кислотой (b), причем указанная 3'-нетранслируемая область содержит последовательность нуклеиновой кислоты, которая выбрана из группы, состоящей из: (c-1) последовательности нуклеиновой кислоты 3'-нетранслируемой области FCGRT, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента, (c-2) последовательности нуклеиновой кислоты 3'-нетранслируемой области LSP1, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента, (c-3) последовательности нуклеиновой кислоты 3'-нетранслируемой области CCL22, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента, (c-4) последовательности нуклеиновой кислоты 3'-нетранслируемой области AES, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента, (c-5) последовательности нуклеиновой кислоты 3'-нетранслируемой области PLD3, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента, (c-6) последовательности нуклеиновой кислоты некодирующей РНК MTRNR1, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента, (c-7) последовательности нуклеиновой кислоты 3'-нетранслируемой области HLA-DRB4, ее фрагмента или варианта указанных последовательности нуклеиновой кислоты или фрагмента и (c-8) любой комбинации двух или более последовательностей нуклеиновой кислоты, фрагментов и/или вариантов, обозначенных под номерами (c-1), (c-2), (c-3), (c-4), (c-5), (c-6) и (c-7), где последовательности нуклеиновой кислоты (b) и (c) под контролем промотора (a) могут быть транскрибированы с образованием общего транскрипта, в котором последовательность нуклеиновой кислоты, транскрибированная с последовательности нуклеиновой кислоты (c), является активной в отношении повышения эффективности трансляции и/или стабильности последовательности нуклеиновой кислоты, транскрибированной с транскрибируемой последовательности нуклеиновой кислоты (b). Изобретение обеспечивает повышенную стабильность транскрипта и/или эффективность трансляции. 12 н. и 41 з.п. ф-лы, 14 ил., 18 табл., 12 пр.
Формула
Документы, цитированные в отчёте о поиске
Днк-конструкция (варианты), способ получения трансгенных растений и фруктаны
Новая полноразмерная геномная phk вируса японского энцефалита, полученная из нее инфекционная кднк jev и их применение
Способ получения генетически трансформированных растений с повышенным содержанием крахмала и рекомбинантная двухцепочечная днк-молекула

Комментарии