Способ множественного параллельного скрининга специфичности связывания биологически активных соединений с нуклеиновыми кислотами с использованием биочипа (варианты) - RU2182708C2
Код документа: RU2182708C2
Чертежи






Описание
Изобретение относится к молекулярной биологии, фармакологии, медицине и охране окружающей среды и рассматривает способ определения относительного сродства к связыванию природных и химических биологически активных соединений с нуклеиновыми кислотами со всеми возможными последовательностями нуклеотидов в пределах участка заданного размера нуклеиновой кислоты. Специфичность связывания химических соединений с различными последовательностями нуклеиновых кислот анализируют с помощью универсального олигонуклеотидного биочипа, в ячейках которого иммобилизованы все возможные последовательности синтетических олигонуклеотидов определенной длины.
Изобретение также включает оригинальную методику подготовки универсального олигонуклеотидного биочипа к процедуре анализа специфичности связывания химических соединений с различными последовательностями, методику регистрации, анализа и интерпретации полученных результатов.
Предшествующий уровень техники
Множество
соединений тестируют и анализируют на
способность специфически связываться с определенной последовательностью пар оснований ДНК, среди этих соединений присутствуют регуляторы репликации,
транскрипции и трансляции - потенциальные
лекарства
для лечения определенных заболеваний. Представляет также интерес выявление всех последовательностей нуклеиновых кислот, с которыми способно
специфически взаимодействовать то или иное
соединение,
попадающее в человеческий организм, поскольку необходимо предвидеть возможные негативные последствия таких взаимодействий, если
соответствующие
последовательности входят в состав генов
или
регуляторных участков генома человека.
Такой скрининг включает тестирование соединений на специфичность связывания и стабильность их комплексов с нуклеиновыми кислотами. Наиболее исчерпывающие результаты были достигнуты в ходе экспериментов по связыванию и плавлению с последующим сравнительным термодинамическим анализом комплексов лиганда с нуклеиновыми кислотами различной последовательности. Однако такой анализ комплексов в растворе требует больших усилий и времени.
Альтернативные, более
простые и
удобные в практическом отношении процедуры анализа
избирательности связывания различных соединений с определенными последовательностями пар оснований ДНК включают два основных подхода:
I.
Защита нуклеиновой кислоты в составе комплекса с
химическим или природным соединением от химической модификации, действия нуклеаз, футпринтинг;
Maxam A. , Mirzabekov A. and
Gilbert W.
(1976). In: Control of Ribosome Synthesis (Alfred
Benzon
Symp. IX), p. 139.
Fox K.R. (1997) Methods Mol. Biol., 90, 1-22.
Petri V. and Brenowitz M. (1997) Curr. Opin. Biotechnol., 8, 36-44.
II. Разделение
комплексов с помощью электрофореза;
Hamdan I.I., Skellern G.G. and Waigh R.D. (1998) Nucleic Acids Res., 26, 3053-3058.
Принципиальным недостатком перечисленных выше подходов является существенное ограничение по количеству анализируемых последовательностей. Эти методы не дают возможности осуществить сравнительный экспериментальный анализ степени сродства химического соединения ко всем возможным последовательностям нуклеиновой кислоты в пределах участка связывания.
Данные недостатки устраняются в настоящем изобретении.
Сущность
изобретения
Сущность изобретения заключается в разработке способа множественного параллельного скрининга
специфичности
связывания биологически активных соединений с двухцепочечными
нуклеиновыми кислотами с использованием биочипа, включающего изготовление биочипа с иммобилизованными на нем
олигонуклеотидами,
гибридизацию иммобилизованных на биочипе олигонуклеотидов со смесью
флуоресцентно меченных олигонуклеотидов для формирования на биочипе дуплексов, плавление дуплексов с
одновременной регистрацией
данных и после отмывки биочипа повторную гибридизацию с той же смесью
олигонуклеотидов с последующей инкубацией с изучаемым соединением. После повторного плавления на
биочипе определяют разности
температур плавления дуплексов в комплексе с изучаемым соединением и в
свободном состоянии, на основе чего определяется специфичность взаимодействия изучаемого
соединения с двухцепочечными
нуклеиновыми кислотами.
Способ предусматривает использование универсального биочипа, в ячейках которого иммобилизованы все возможные гексануклеотиды в количестве 46=4096 и смеси флуоресцентно меченных несамокомплементарных олигонуклеотидов, каждый из которых комплементарен олигонуклеотиду, иммобилизованному на универсальном биочипе. В качестве флуоресцентного красителя можно использовать, например, Техасский красный (Texas Red). Плавление олигонуклеотидных дуплексов, иммобилизованых на биочипе, проводят с помощью специального термостолика, а регистрацию плавления проводят с помощью флуоресцентного микроскопа, оборудованного высокочувствительной телекамерой, подключенной к компьютеру. Массив экспериментальных данных, полученных в ходе плавления, обрабатывают с помощью специальной компьютерной программы, которая вычисляет температуры плавления Тпл двухцепочечных олигонуклеотидов в присутствии и в отсутствии изучаемого соединения и вычисляет величины сдвига Тпл для всех двухцепочечных олигонуклеотидов, иммобилизованных на биочипе, на основе которых определяется специфичность взаимодействия. С помощью этого способа можно анализировать различные соединения, в частности низкомолекулярные или высокомолекулярные химические, природные или рекомбинантные.
Способ множественного параллельного скрининга специфичности связывания биологически активных соединений с одноцепочечными нуклеиновыми кислотами с использованием биочипа включает изготовление биочипа с иммобилизованными на нем олигонуклеотидами; инкубацию биочипа с флуоресцирующим изучаемым соединением; плавление на биочипе комплексов олигонуклеотидов с флуоресцирующим изучаемым соединением с определением температуры плавления и определение специфичности связывания изучаемого соединения с нуклеиновыми кислотами.
Способ также предусматривает использование универсального биочипа. Изучаемое соединение может быть природно флуоресцирующим или нести на себе флуоресцентную метку. В качестве флуоресцентной метки можно использовать краситель FITC. Плавление комплексов олигонуклеотидов с изучаемым веществом проводят с помощью специального термостолика, а регистрацию плавления проводят с помощью флуоресцентного микроскопа, оборудованного высокочувствительной телекамерой, подключенной к компьютеру. Массив экспериментальных данных, полученных в ходе плавления, обрабатывают с помощью специальной компьютерной программы, которая в дальнейшем определяет температуры плавления комплексов изучаемого соединения со всеми олигонуклеотидами, иммобилизованными на биочипе, на основе которых определяется специфичность взаимодействия. С помощью этого способа можно анализировать различные соединения, в частности низкомолекулярные или высокомолекулярные химические, природные или рекомбинантные.
Перечень фигур
Фигура 1. Неравновесные кривые плавления дуплексов ДНК на
биочипах, снятые в присутствии (А) и в отсутствии (В) Hoechst.
Двухцепочечный олигонуклеотид гель-МТТTТСGМ-3'/5'-МСGАAААМ-ТR-3'
формировался при гибридизации смеси олигонуклеотидов
5'-MM(A/G)MM(A/G)MM-TexasRed на универсальном биочипе. Неравновесную температуру
павления Тпл определяли как температуру, при которой
гибридизационный сигнал уменьшается до 1/10 исходной величины,
измеренной при 0oС. Степень сродства Hoechst к дуплексу определяли как
ΔТпл= TплВ-TплА.
Фигура 2. Увеличение температуры плавления ΔТпл, вызванное связыванием Hoechst с дуплексами, сформированными на мирочипе. Универсальный 6-мерный биочип гибридизовали последовательно с двумя смесями флуоресцентно меченных олигонуклеотидов 5'-MM(A/G)MM(A/G)MM-TR и 5'-MM(A/C)MM(A/C)MM-TR в отсутствие и в присутствии Hoechst, после чего снимали кривые плавления дуплексов.
Дуплексы на биочипе расположены как двумерная матрица таким образом, что 5'-половины 6-меров, составляющих середину олигонуклеотида, располагаются построчно, а 3'-половины - по колонкам. Величина ΔТпл для дуплексов показана величиной черного сектора внутри круга для соответствующего элемента матрицы.
Фигура 3. Гистограмма, показывающая число дуплексов N с указанным значением Δ Тпл. Около 300 дуплексов имеют сильное сродство к Hoechst, соответствующие им величины ΔТпл>1.5oС (указаны стрелкой).
Фигура 4. Неравновесные кривые плавления двухцепочечного олигонуклеотида на универсальном биочипе в отсутствии и присутствии белка HU.
Последовательность олигонуклеотида, иммобилизованного на биочипе: 5'-MAGTCTGM-3'.
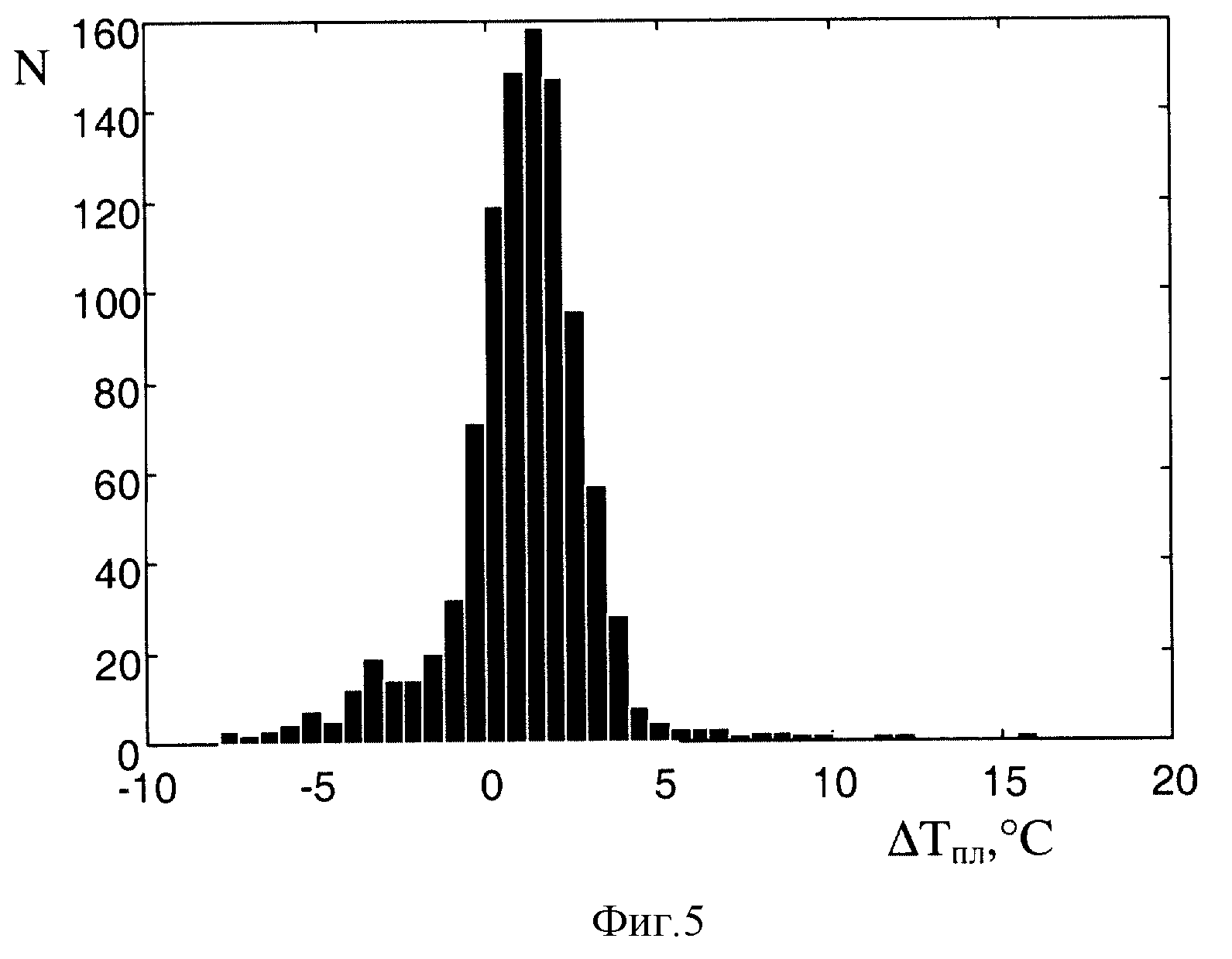
Фигура 5. Гистограмма, показывающая число дуплексов, демонстрирующих определенную ΔТпл.
Фигура 6. Средние величины Δ Тпл для дуплексов, содержащих различные последовательности из трех оснований ДНК.
Фигура 7. Неравновесные кривые плавления комплексов белка HU, меченого FITС, с иммобилизованными олигонуклеотидами на универсальном биочипе. Последовательности олигонуклеотидов указаны на фигуре.
Фигура 8. Зависимость температуры плавления комплексов белок HU - ДНК от А/Т состава иммобилизованного одноцепочечного олигонуклеотида.
Раскрытие изобретения
Изобретения заключается в
проведении массированного скрининга специфичности
связывания с ДНК природных и химических соединений с помощью анализа температур
плавления или сдвигов температур
плавления комплексов этих
соединений со всеми возможными последовательностями
олигонуклеотидных дуплексов или одноцепочечных олигонуклеотидов, иммобилизованных в
гелевых ячейках универсального
биочипа.
В
различных ячейках биочипа иммобилизованы олигонуклеотиды,
имеющие одинаковую длину, но различающиеся последовательностями оснований. При этом
в каждой ячейке биочипа
иммобилизован олигонуклеотид с
определенной последовательностью оснований, а число ячеек
биочипа равно числу всех возможных последовательностей оснований для нуклеотида данной
длины. С помощью такого
биочипа можно количественно
анализировать специфичность связывания природных или
химических соединений со всеми представленными в его ячейках последовательностями нуклеотидов
как однонитчатых, так и
входящих в состав двойной
спирали ДНК:
1. Определение специфичности
связывания природных или химических соединений со всеми последовательностями олигонуклеотидных
дуплексов, сформированных в
ячейках биочипа.
При анализе специфичности связывания с олигонуклеотидными дуплексами в гибридизационную камеру универсального биочипа сначала добавляют раствор, содержащий смесь не комплементарных друг другу флуоресцентно меченных олигонуклеотидов, каждый из которых комплементарен одному из нуклеотидов, иммобилизованных в ячейках биочипа, и в процессе гибридизации образует с ним двухцепочечный комплекс. Образование комплексов или их плавление регистрируется по нарастанию или убыванию интенсивности флуоресцентного сигнала одновременно во всех ячейках биочипа.
Гибридизацию проводят при низкой температуре, после чего биочип постепенно нагревают, регистрируя по убыванию флуоресцентных сигналов кривые плавления, и определяют температуры плавления всех дуплексов, образованных данной гибридизационной смесью олигонуклеотидов.
После этого проводят повторную гибридизацию при низкой температуре и добавляют в гибридизационную камеру раствор, содержащий химическое соединение, исследуемое на специфичность связывания с нуклеиновыми кислотами. Тестируемое соединение проникает в ячейки биочипа и образует с олигонуклеотидными дуплексами комплексы, характеризуемые в общем случае различной энергией связывания. Далее биочип нагревается с той же скоростью, как и в случае плавления свободных дуплексов, и повторно снимают кривые плавления всех дуплексов.
Если химическое соединение взаимодействует с какими-то последовательностями двухцепочечной ДНК, то кривые плавления, регистрируемые по убыванию флуоресцентного сигнала в ячейках биочипа, содержащих олигонуклеотиды с соответствующими последовательностями, окажутся сдвинутыми в область более высоких или более низких температур. Такие сдвиги кривых плавления указывают на наличие взаимодействия тестируемого соединения с соответствующими последовательностями оснований ДНК: положительный сдвиг указывает на взаимодействие, стабилизирующее структуру двойной спирали, а отрицательный сдвиг - на взаимодействие, дестабилизирующее структуру двойной спирали.
По сдвигам кривых плавления определяют сдвиги температур плавления ΔТпл, которые являются термодинамическими параметрами, характеризующими специфичность связывания химического соединения с соответствующими последовательностями пар оснований в составе двойной спирали ДНК. Наборы значений ΔТпл, определенные для различных смесей не комплементарных друг другу флуоресцентно меченных олигонуклеотидов, объединяют в массив термодинамических параметров, характеризующих сродство тестируемого соединения к различным последовательностям пар оснований в составе двойной спирали ДНК.
2. Определение специфичности связывания природных или химических соединений со всеми последовательностями одноцепочечных олигодезоксирибонуклеотидов, иммобилизованных в ячейках биочипа.
При анализе специфичности связывания с одноцепочечными олигонуклеотидами в гибридизационную камеру описанного выше универсального биочипа добавляют раствор, содержащий молекулы тестируемого соединения, предварительно меченные флуоресцентной меткой. При достаточно низкой температуре гибридизации молекулы тестируемого соединения проникают во все ячейки биочипа и образуют комплексы с теми олигонуклеотидами, к которым обнаруживают сродство к связыванию. В результате, в соответствующих ячейках появляются флуоресцентные сигналы, распределение интенсивностей которых может служить комплексной характеристикой сродства тестируемого соединения к представленным последовательностям олигонуклеотидов.
Далее проводят постепенное повышение температуры биочипа, регистрируя одновременно диссоциацию комплексов тестируемого соединения со всеми олигонуклеотидами по падению интенсивности флуоресценции в каждой ячейке. По полученным кривым плавления определяют температуры плавления Тпл образовавшихся комплексов тестируемого соединения с олигонуклеотидами.
Задача настоящего изобретения состоит в создании ускоренного метода, позволяющего проводить массированный скрининг специфичности связывания природных и химических соединений с ДНК. Скрининг специфичности связывания предлагается проводить с помощью универсального биочипа. В различных ячейках биочипа иммобилизованы олигонуклеотиды, имеющие одинаковую длину, но различающиеся последовательностями оснований. При этом в каждой ячейке биочипа иммобилизован олигонуклеотид с определенной последовательностью оснований, а число ячеек биочипа равно числу всех возможных последовательностей оснований для нуклеотида данной длины.
С помощью такого биочипа предлагается количественно анализировать специфичность связывания природных или химических соединений со всеми представленными в его ячейках последовательностями нуклеотидов как одноцепочечных, так и входящих в состав двойной спирали ДНК. Стереоспецифичность или сродство тестируемых соединений к определенным последовательностям оснований определяется по температурам плавления комплексов этих соединений со всеми возможными последовательностями олигонуклеотидных дуплексов или одноцепочечных олигонуклеотидов, иммобилизованных в гелевых ячейках универсального биочипа. Дополнительный способ оценки стереоспецифичности связывания с одноцепочечными олигонуклеотидами основан на измерении относительных количеств связанных молекул тестируемых соединений во всех ячейках биочипа.
Отличительной особенностью предлагаемого метода является большое количество термодинамических параметров (сдвигов температур плавления ΔТпл или температур плавления Тпл), характеризующих взаимодействие тестируемого соединения с полным перебором всех возможных последовательностей универсального биочипа.
Для множественного параллельного анализа специфичности связывания биологически активных соединений с нуклеиновыми кислотами разработан универсальный гексадезоксирибонуклеотидный биочип. Применение универсального биочипа продемонстрировано на примере анализа связывания Hoechst 33258 с различными 6-мерными дуплексами, анализа связывания белка HU с различными гексамерными дуплексами и анализа связывания белка HU с различными гексамерными одноцепочечными олигонуклеотидами.
В случае применения универсального гексадезоксирибонуклеотидного биочипа для множественного параллельного анализа связывания Hoechst 33258 с различными 6-мерными дуплексами был выполнен термодинамический анализ ряда таких комплексов и определены последовательности, ответственные за специфическое взаимодействие. Проведено их количественное сравнение с помощью статистического анализа большого числа кривых плавления дуплексов, сформирорванных на биочипе в присутствии и в отсутствии красителя. Такой подход может быть распространен на проведение скрининга специфичности связывания относительно последовательности пар оснований других лигандов, включая белки.
В случае применения универсального гексадезоксирибонуклеотидного биочипа для множественного параллельного анализа связывания белка HU с различными гексамерными дуплексами были получены 1024 экспериментальные кривые плавления двухцепочечных олигонуклеотидов в присутствии и в отсутствии белка HU. Ранее было предположено, что данный белок не обладает сильной и явно выраженной специфичностью связывания с определенными последовательностями двухцепочных ДНК, однако статистический анализ 1024 кривых плавления позволил выявить ряд закономерностей связывания и обнаружить последовательности ДНК, с которыми HU связывается предпочтительно.
В случае применения универсального гексадезоксирибонуклеотидного биочипа для множественного паралельного анализа связывания белка HU с различными гексамерными одноцепочечными олигонуклеотидами универсальный олигонуклеотидный биочип не переводили в двухцепочечное состояние путем добавления смеси из флуоресцентно меченных олигонуклеотидов, а флуоресцентно метили белок HU красителем FIТС. Затем измеряли прямое связывание флуоресцентно меченного белка с олигонуклеотидами, иммобилизованными на биочипе, после чего комплексы белка HU с одноцепочечными олигонуклеотидами подвергали плавлению. Полученные данные позволили определить специфичность связывания белка HU непосредственно с одноцепочечными последовательностями ДНК.
Примеры
Пример 1. Множественный параллельный анализ
специфичности связывания Hoechst 33258 с ДНК с использованием универсального
олигонуклеотидного
биочипа
Химические
соединения
Hoechst 33258 (Е345= 41000) был предоставлен
H.Loeve (Фирма "Hoechst", Германия) и использовался без дополнительной
очистки.
4096 октадезоксирибонуклеотидов (CyberSyn, США) были синтезированы с использованием стандартных процедур и 3'-С(7) аминомодификатора CPG (Glen Research, США) и использованы для приготовления универсальных биочипов. 8-меры имели следующую структуру: 5'-NH2-MNNNNNNM-3', где М - смесь четырех оснований в соотношении 1:1:1:1 как в 3'-, так и в 5'-концевой позиции; N - одно из четырех оснований, представляющих в срединной части все 4096 возможных 6-меров, NH2-аминолинкер, использованный для иммобилизации 8-меров в ячейках полиакриламидного геля на биочипе. Два вида смесей 8-меров 5'-MM(A/G)MM(A/G)MM-NH2-3' и 5'-MM(A/C)MM(A/C)MM-3'-NH2 были синтезированы при помощи ДНК/РНК синтезатора (Applied Biosystems, модель 394) с использованием стандартных процедур и 3'-С(7) аминомодификатора CPG (Glen Research, США). Смеси 8-меров были флуоресцентно помечены сульфонилхлоридным красителем Техасский красный (TexRed, Molecular Probes, USA) в соответствии с протоколом производителя.
Универсальный биочип
Универсальный биочип готовили в две стадии. Сначала при помощи процедуры
фотополимеризации (Guschin D., Yershov
G., Zaslavsky A., Gemmel A., Shick V. , Proudnikov D. , Arenkov P. and Mirzabekov A.
(1997) Anal. Biochem., 250, 203-211) готовили решетку из 4200 ячеек 5%-го
полиакриламидного геля (60х70 ячеек
размером 100х100х20 мкм), разделенных полосами гидрофобного стекла по 200 мкм. Затем на
каждую ячейку наносили капли 1 мМ водного раствора олигонуклеотида
объемом 1 нл (Yershov G., Barsky V.,
Belgovskiy A., Kirillov E., Kreindlin E. , Ivanov I., Parinov S., Guschin D., Drobishev A.,
Dubiley S. and Mirzabekov A. (1996) Proc. Nat. Acad. Sci. USA, 93,
4913-4918), олигонуклеотиды
иммобилизовали с помощью восстановительного аминирования их аминогрупп с альдегидными группами геля
(Timofeev E., Kochetkova S.V., Mirzabekov A.D. and Florentiev V.L.
(1996) Nucleic Acids Res., 24,
3142-3148).
Гибридизация и снятие кривых плавления
Гибридизацию
универсального биочипа со смесью флуоресцентно меченных 8-меров проводили в
гибридизационной кювете объемом
200 мкл при 0oС в течение 24 ч. Растворы для гибридизации содержали 200 мМ
олигонуклеотидов, 1 М NaCl, 10 мМ NaHP04, 5 mM EDTA, pH 6.8, 0.1% Tween-20.
После гибридизации биочип вместе с
кюветой помещали на термостатируемый столик микроскопа, где снимали неравновесные кривые
плавления для всех элементов биочипа при повышении температуры от 0oС до 50oС со
скоростью 2oС/мин (Fotin A.V., Drobyshev A. L. , Proudnikov D.Y., Perov A.N. and
Mirzabekov A.D. (1998) Nucleic Acids Res., 26, 1515-1521). После снятия
кривых плавления дуплексов при
отсутствии Hoechst 33258 биочип отмывали водой от флуоресцентно меченных олигонуклеотидов. Второй
раунд гибридизация-плавление проводили в тех же условиях, но в
присутствии 10 мМ Hoechst 33258.
Гибридизацию 8-мерного биочипа с 1 мМ раствором флуоресцентно меченного 20-мера тоже выполняли в одинаковых условиях как в отсутствии, так и в присутствии 2 мМ Hoechst 33258. Равновесные кривые плавления снимали на биочипе так, как это было описано (Fotin A.V., Drobyshev A.L., Proudnikov D.Y., Perov A.N. and Mirzabekov A.D. (1998) Nucleic Acids Res., 26, 1515-1521).
Все измерения при снятии кривых плавления производили с помощью автоматического эпифлуоресцентного микроскопа с полем 3.5х3.5 мм, снабженным CCD-камерой, термоэлементом Пелтье и датчиком температуры, соединенным с компьютером, оснащенным программой автоматической обработки данных (Fotin A.V. , Drobyshev A.L., Proudnikov D.Y., Perov A.N. and Mirzabekov A.D. (1998) Nucleic Acids Res., 26, 1515-1521). Интенсивность флуоресценции регистрировали для каждого значения температуры при сканировании универсального биочипа по нескольким полям, содержащим 100 ячеек геля. Сканирующая система состояла из двухкоординатного столика с направляющими, микромотора и контроллера (Newport, США). Для контроля хода эксперимента и обработки данных было разработано специальное программное обеспечение с использованием виртуального инструментального интерфейса Lab VIEW (National Instruments, США).
Множественные параллельные
измерения связывания Hoechst 33258 с
дуплексами ДНК на
универсальном биочипе
Универсальный 6-мерный биочип содержал все возможные 4096 (46=4096) одноцепочечные
гексадезоксирибонуклеотиды NNNNNN (N - одно из четырех
оснований),
иммобилизованные в индивидуальных ячейках геля. К этим 6-мерам, как к сердцевине, с обоих 3'- и
5'-концов примыкают по два 8-мера,
имеющих структуру типа 5'-гель-MNNNNNNM-3' и
составленных из смеси
четырех оснований М в соотношении 1:1:1:1.
Hoechst 33258 не связывается с одноцепочечной ДНК и не проявляет заметной специфичности при сильном связывании с двухцепочечной ДНК. Дуплексы ДНК в составе комплексов стабилизируются красителем и имеют более высокую температуру плавления Тпл. Специфичность связывания красителя можно оценить по увеличению Тпл у дуплексов при связывании лиганда. Для проведения этих измерений 4096 одноцепочечных олигонуклеотидов на универсальном биочипе должны быть достроены в двухспиральную форму. Это может быть достигнуто путем гибридизации биочипа со смесью 4096 флуоресцентно меченных 8-меров, имеющих структуру 5'-MNNNNNNM-TR-3'.
Было показано, что симметричные дуплексы, имеющие одинаковые пары оснований со стороны геля и со стороны красителя, например 5'-гель-AGTCTCGA-3'/3'-TR-TCAGAGCT-5' и 5'-AGTCTCGA-TR-3'/3'-TCAGAGCT-гель-5', имеют сходные кривые плавления как в отсутствии, так и в присутствии лиганда (данные не показаны). 6-мерные универсальные биочипы могут содержать 2080 различных 6-мерных дуплексов. Среди них есть 64 палиндромных самокомплементарных 6-мера, которые не могут быть получены на биочипе из-за их самогибридизации в растворе. Для того чтобы избежать конкурентной самогибридизации олигонуклеотидов в растворе и на биочипе, можно подобрать 6 смесей, содержащих несамокомплементарные олигонуклеотиды: MMM(A/G)(A/G)MMM, MMM(A/C)(A/C)MMM, MM(A/G)MM(A/G)MM, MM(A/C)MM(A/C)MM, M(A/G)MMMM(A/G)M, М(А/С)МMMМ(А/С)М. Такой набор смесей учитывает все возможные (2016) несамокомплементарные дуплексы.
Наиболее информативные данные могут быть получены с двумя несамокомплементарными смесями: 5'-MM(A/G)MM(A/G)MM-TR-3' и 5'-MM(A/C)MM(A/C)MM-TR-3'. Каждая из этих смесей 8-меров содержит 1024 различных центральных 6-мера, однако вместе они объединяют 1536 центральных 6-меров, так как структуры типа ММАММАММ присутствуют в обеих смесях. Эти две смеси включают АТ-богатые дуплексы, необходимые для детекции большей части последовательностей, специфичных относительно связывания Hoechst.
Биочип гибридизовали с каждой из указаннных смесей флуоресцентно меченных 8-меров. Неравновесные кривые плавления всех дуплексов, сформирорванных на биочипе, снимали в процессе повышения температуры. Процессы гибридизации и плавления повторяли на том же биочипе в присутствии Hoechst в буфере для гибридизации.
На фиг. 1 показаны две типичные кривые плавления, снятые для дуплекса в присутствии и в отсутствии Hoechst. Температуру, при которой сигнал гибридизации уменьшался в 10 раз по сравнению с исходным уровнем, определенным при 0oС, рассматривали как неравновесную температуру плавления Тпл . Присутствие лиганда сдвигает кривые плавления в сторону более высоких температур, давая увеличение температуры плавления ΔТпл, в зависимости от структуры дуплекса, до 13oС. Таким образом, величина Δ Тпл была использована для того, чтобы оценить степень сродства Hoechst к данному дуплексу.
На фиг.2 показаны величины Δ Тпл для 1680 олигонуклеотидов, которые сформировали дуплексы на универсальном биочипе при его гибридизации с двумя типами смесей флуоресцентно меченных олигонуклеотидов. 112 олигонуклеотидов были исключены из рассмотрения из-за слабого гибридизационного сигнала.
Анализ последовательностей,
специфичных для связывания Hoechst
Данные на фиг.2
показывают, что Hoechst 33258 имеет более сильное сродство
к
АТ-богатым последовательностям по сравнению с GC-богатыми. Характеристика
типов последовательностей, специфичных для связывания лиганда,
требует более детального анализа. Приведенный ниже подход
с
использованием компьютера был развит для анализа больших массивов
экспериментальных данных, полученных с помощью универсального биочипа.
Гистограмма большого числа дуплексов, имеющих характерные величины ΔТпл показана на фиг.3. Гистограмма содержит острый пик около 0oС, соответствующий дуплексам, которые не стабилизируются при связывании Hoechst, и широкий пик, начинающийся с 1,5oС с длинным плечом до 13oС. Таким образом, превышение величины ΔТпл выше уровня 1,5oС может рассматриваться как признак связывания Hoechst с данным дуплексом. Было выявлено 300 таких дуплексов.
Пример 2. Изучение специфичности связывания белка HU с двухспиральной ДНК путем множественного параллельного анализа на универсальном олигонуклеотидном биочипе.
Химические соединения.
Октадезоксирибонуклеотиды для универсального биочипа были изготовлены также, как описано в примере 1.
Смесь 8-меров 5'-MM(A/C)MM(A/C)MM-NH2-3' была синтезирована также, как описано в примере 1, и таким же образом была мечена красителем Техасский красный (TexRed).
Универсальный биочип был приготовлен также, как описано в примере 1.
Белок HU.
Белок HU выделяли из E. coli штамм JRY1 по ранее описанной методике (Rouviere-Yaniv, J. & Kjeldgaard, N.O. (1979) FEBS Letters, 106, 297-300). Концентрацию белка определяли по поглощению при 230 нм с использованием коэффициента экстинкции А230= 2.3, соответствующего концентрации белка HU 1мг/мл.
Гибридизация и снятие кривых плавления.
Гибридизацию универсального биочипа со смесью флуоресцентно меченных 8-меров проводили также, как в примере 1. Раствор для гибридизации содержал 200 мкМ олигонуклеотидов, 0.1 М NaCI, 20 mM Tris (pH 7.2), 5 mМ EDTA и 0.1% Tween 20.
Неравновесные кривые плавления снимали также, как описано в примере 1.
После снятия кривых плавления дуплексов в отсутствии белка HU биочип отмывали водой от флуоресцентно меченных олигонуклеотидов. Второй раунд гибридизации и плавления проводили в тех же условиях, но после гибридизации раствор олигонуклеотидов заменяли буфером, содержащим белок HU (0.55мг/мл) и инкубировали 12 часов при 0oС. Затем проводили плавление.
Все измерения на флуоресцентном микроскопе проводили также, как описано в примере 1.
Изучение специфичности связывания белка HU с двухцепочечной ДНК на универсальном биочипе.
Известно, что белок HU при связывании с двухцепочечной ДНК не проявляет заметной специфичности. Специфичность комплексов белок HU - ДНК оценивали с помощью статистического анализа множественных данных, содержащих кривые плавления дуплексов в отсутствии и в присутствии белка HU. Для проведения таких экспериментов 1024 октамера, иммобилизованных на биочипе, были переведены в двухцепочечную форму так, как это описано в примере 1. Для перевода олигонуклеотидов в двухцепочечную форму использовали несамокомплементарную смесь олигонуклеотидов с общей формулой: 5'-MM(A/C)MM(A/C)MM-NH2-3'-TR, содержащую 1024 олигонуклеотида, меченных Техасским красным.
Биочип гибридизовали со смесью флуоресцентно меченных октамеров в буфере, как это описано выше. Затем смесь октамеров в гибиридизационной камере заменили на чистый буфер. Неравновесные кривые плавления для всех дуплексов на биочипе получали в ходе плавления биочипа на термостолике.
После отмывки биочипа гибридизацию и плавление повторили еще раз. Но на этот раз после гибридизации смесь олигонуклеотидов заменяли на раствор белка HU (0.55мг/мл) в этом же буфере и инкубировали в течении 12 часов. После этого плавление проводили так же, как в первый раз.
Таким образом были получены 1024 кривые плавления
октамеров в отсутствии
белка HU и 1024 кривые плавления в присутствии белка HU. На фиг.4 показаны две такие кривые
плавления для одного олигонуклеотида AGTCTG. Для анализа кривых использовали
специальную компьютерную
программу. Все эксперментальные кривые плавления апроксимировали методом наименьших квадратов с
помощью следующей формулы:

где: f(T) - измеряемый сигнал,
А+В - начальный сигнал,
В - конечный сигнал,
Т - температура (К),
Тпл - температура плавления,
N - фактор неоперативности.
После апроксимации было получено 1024 значения температуры плавления То для всех дуплексов в присутствии и в отсутствии белка HU. Затем был создан массив из 1024 значений ΔТпл=Тпл (белка) - Тпл (без белка), где ΔТпл - сдвиг температуры плавления олигонуклеотида в присутствии белка. Данные по 14 дуплексам были исключены из рассмотрения из-за слабого сигнала. Массив из 1010 значений ΔТ0 был подвергнут статистическому анализу.
Значения ΔТпл были расположены в виде гистограммы, представленной на фиг.5. Гистограмма четко указывает на наличие двух типов комплексов белка HU с олигонуклеотидами. Первый, основной комплекс имеет положительное значение сдвига температуры плавления ΔТпл, средняя величина сдвига около +3oС. Второй, минорный комплекс описывается примерно 150 значениями ΔТпл и имеет средний отрицательный сдвиг около -3oС.
Для нахождения разницы между основным и минорным комплексами был проведен специальный анализ. Оказалось, что минорный комплекс содержит длинные А/Т последовательности, состоящие из 4, 5 и 6 оснований. В основном комплексе А/Т последовательности более короткие (2, 3 и 4 основания).
Наличие большого массива данных из 1010 значений ΔТ0 позволило нам исследовать специфичность связывания белка HU с двухцепочечной ДНК. Ранее считалось, что этот белок связывается с ДНК неспецифически.
Сдвиги температуры плавления комплексов HU с ДНК были вычислены для всех последовательностей из трех оснований на биочипе. Данные представлены на фиг.6.
Статистический анализ показал, что существуют три последовательности: AAG, AGA и ТАА, связывание с которыми белка HU является предпочтительным. Этот результат был получен исключительно благодаря использованию универсального олигонуклеотидного биочипа и возможности получить и обработать более 1000 кривых плавления одновременно.
Пример 3. Изучение специфичности связывания белка HU с одноцепочечными фрагментами ДНК путем множественного параллельного анализа на универсальном олигонуклеотидном биочипе.
Универсальный
биочип
Для изучения специфичности связывания белка HU с одноцепочечными фрагментами ДНК
использовали тот же самый
универсальный биочип, который описан
в примерах 1 и 2, причем флуоресцентно
меченные олигонуклеотиды, описанные в примерах 1 и 2, не применяли.
Белок HU
Использовали белок HU,
описанный в примере 2. Для
измерения связывания с одноцепочечными
последовательностями ДНК белок метили флуоресцентным красителем FITC в соответствии со стандартной
методикой (Hermanson G.T.(1996)
Bioconjugate Techniques,
Academic Press, Amsterdam).
Гибридизация и плавление
Гибридизацию и плавление белка проводили в такой же
гибридизационной камере, как описано в
примере 2. Гибридизационный
раствор содержал белок HU (0.55 мг/мл), 0.020 М
NaCl, 20mM Tris (pH 7.2), 5 mM EDTA и 0.1% Tween 20. Гибридизацию проводили при
0oС в течение 24 часов.
Все эксперименты по плавлению проводили так же, как описано в примерах 1 и 2, но для измерения интенсивности флуоресценции FIТС использовали специальный светофильтр.
Изучение специфичности связывания
белка HU с одноцепочечными
последовательностями ДНК на универсальном биочипе
Известно, что белок HU связывается с
одноцепочечными фрагментами ДНК. В
проведенных ранее работах по связыванию
белка с одноцепочечными ДНК
использовали фрагменты ДНК длиной 30-40 нуклеотидов. Такие фрагменты уже могут обладать
вторичной структурой и, соответственно,
будет измеряться константа связывания
белка не с одноцепочечной ДНК.
Только в случае иммобилизации на биочипе октамеров можно гарантировать, что белок будет связываться с действительно одноцепочечной ДНК.
Белок HU гибридизовали с универсальным биочипом в течение 24 часов в описанном выше буфере. Плавление комплекса белок-ДНК проводили так же, как это описано в примере 2. Однако в данном случае в ходе нагрева регистрировали не плавление двухцепочечных олигонуклеотидов, а термальный распад комплекса белок-ДНК. Регистрацию процесса такого плавления производили по флуоресценции FIТС, а не по флуоресценции Texas Red, как в случае олигонуклеотидов. На фиг. 7 представлены некоторые экспериментальные кривые плавления комплексов белка HU с одноцепочечными олигонуклеотидами.
Температуры плавления вычисляли для всех 4096 комплексов белок-ДНК с помощью метода, описанного в примере 2. Полученный массив данных был исследован с целью поиска специфичности связывания белка HU с различными последовательностями одноцепочечных олигонуклеотидов. На фиг.8 представлен результат статистической обработки этих данных.
Данные, приведенные на фиг. 8, показывают, что среди всех возможных гексамеров белок HU предпочтительно связывается с теми, где 6 или 5 оснований из 6 - это G или С. Все остальные последовательности имеют примерно одинаковые константы связывания с HU. Таким образом, применение универсального биочипа позволило обнаружить определенную специфичность связывания белка HU с одноцепочечными ДНК. Дальнейший анализ показал, что наиболее предпочтительной последовательностью связывания этого белка с ДНК является GCGC.
Реферат
Изобретение относится к молекулярной биологии, фармакологии, медицине и охране окружающей среды. Изготавливают биочип с иммобилизованными на нем олигонуклеотидами. Проводят гибридизацию этих олигонуклеотидов со смесью флуоресцентно меченных несамокомплементарных олигонуклеотидов. На биочипе образуются двухцепочечные олигонуклеотиды. Их плавят, одновременно регистрируя данные. Биочип отмывают. Проводят повторную гибридизацию с такой же смесью флуоресцентно меченных олигонуклеотидов, далее - инкубацию биочипа с изучаемым соединением. Повторно плавят на биочипе двухцепочечные олигонуклеотиды, находящиеся в комплексе с изучаемым биологически активным соединением. Данные регистрируют, определяют температуры плавления двухцепочечных олигонуклеотидов в присутствии и отсутствии изучаемого соединения. Вычисляют разность температур плавления. Определяют на основе массива данных специфичность связывания изучаемого соединения. Преимущественно используют универсальный биочип, в ячейках которого иммобилизованы все возможные гексануклеотиды. Флуоресцентный краситель может быть Техасский красный (Texas Red). Плавят олигонуклеотиды с помощью термостолика. Массив экспериментальных данных обрабатывают преимущественно с помощью компьютерной программы. Изучаемым соединением может быть краситель Hoechst 33258 или белок HU. По второму варианту после изготовления биочипа с иммобилизованными на нем олигонуклеотидами проводят сразу инкубацию биочипа с флуоресцирующим изучаемым соединением. Способ позволяет осуществить сравнительный экспериментальный анализ степени сродства химического соединения ко всем возможным последовательностям нуклеиновой кислоты в пределах участка связывания. 2 с. и 18 з.п. ф-лы, 8 ил.


Комментарии